桃(Prunus persica L.)属于蔷薇科(Rosaceae)李属(Prunus),其果实色、香、味、质俱佳,深受消费者喜爱。2023 年,中国桃产量约为17 500 000 t(FAOSTAT,http://www.fao.org/faostat),位居世界第一位。单果质量性状是作物生产和园艺物种驯化研究的重要目标[1],研究桃单果质量性状的遗传规律,挖掘调控桃单果质量的重要功能基因,对阐明桃单果质量性状形成的分子机制、通过分子标记辅助选择培育大果桃新品种、丰富果品市场,以及提升中国桃产业的市场竞争力具有重要的理论和实践意义。
在近几十年的研究中,多个控制桃果实单果质量性状的数量性状位点(Quantitative trait locis,QTLs)被定位。利用连锁分析,国外研究者率先将该性状的QTLs 定位在桃的8 个连锁群上[2-6]。随着高通量测序技术的发展,全基因组关联分析(GWAS)也被应用于该性状的定位。2016 年,Cao等[7-8]利用129 份桃种质重测序数据进行GWAS,在桃的8条染色体中均鉴定出单果质量性状的关联位点,讨论了单果质量疑似关联区间内候选Expansin基因与单果质量的关系,并未指出候选基因。2019年,Cao 等[9]利用313 份桃种质连续3 年的单果质量表型,再次进行GWAS,在第2 和第6 条染色体上共鉴定出5 个关联位点,并转基因验证PpCYP79B2.1在番茄中超表达能引起果实变小。同年,Li 等[10]利用GWAS分析,将单果质量性状定位于第2和第4条染色体上。2023 年,Ksouri 等[11]利用90 份桃种质对单果质量进行GWAS,发现在第3、6、8条染色体上存在显著的关联位点,表型变异解释率为17%~22%。上述研究虽然根据定位区间也鉴定出不少候选基因,但均没有进行包括功能验证在内的深入分析。
笔者在本研究中以大果型桃品种中油桃13 号和小果型桃品种郑2007-4-28 为亲本,构建F1群体,采取BSA-seq方法定位桃单果质量位点,结合RNAseq 结果筛选调控桃单果质量性状的候选基因。同时,结合实时荧光定量(qRT-PCR)和异源过表达番茄的方法来验证候选基因,为桃分子标记辅助育种工作的开展奠定基础。
1 材料和方法
1.1 试验材料
供试材料中油桃13 号、郑2007-4-28 和二者杂交群体的95 株单株及群体验证73 份不同品种材料(表1),均定植于中国农业科学院郑州果树研究所桃资源遗传与改良团队试验基地(新乡),树势生长良好,管理措施一致。
表1 群体验证所用品种
Table 1 Cultivars for population verification
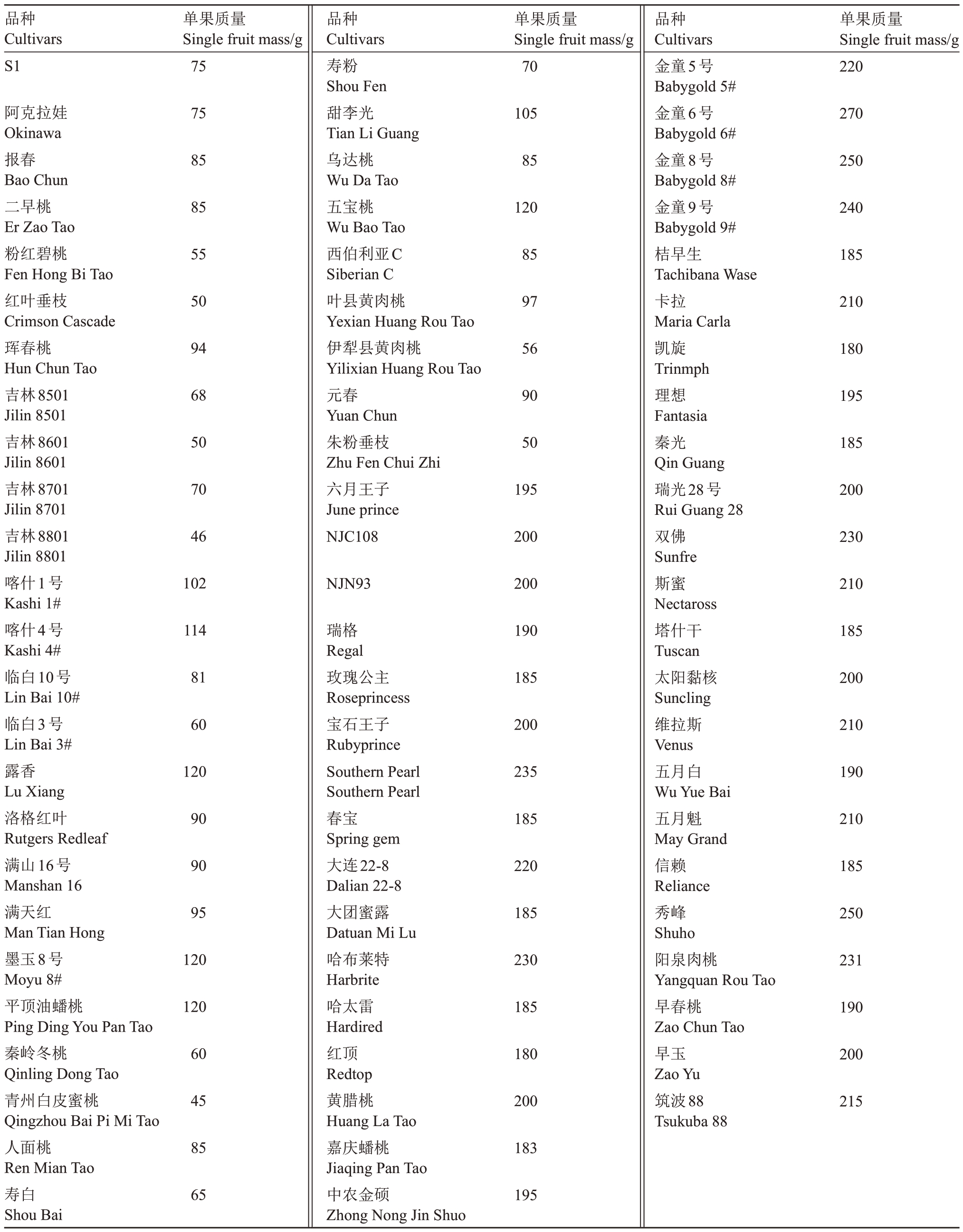
品种Cultivars S1单果质量Single fruit mass/g 75单果质量Single fruit mass/g 70品种Cultivars寿粉Shou Fen甜李光Tian Li Guang乌达桃Wu Da Tao五宝桃Wu Bao Tao西伯利亚C Siberian C叶县黄肉桃Yexian Huang Rou Tao伊犁县黄肉桃Yilixian Huang Rou Tao元春Yuan Chun朱粉垂枝Zhu Fen Chui Zhi六月王子June prince NJC108品种Cultivars金童5号Babygold 5#金童6号Babygold 6#金童8号Babygold 8#金童9号Babygold 9#桔早生Tachibana Wase卡拉Мaria Carla凯旋Trinmph理想Fantasia秦光Qin Guang瑞光28号Rui Guang 28双佛Sunfre斯蜜Nectaross塔什干Tuscan太阳黏核Suncling维拉斯Venus五月白Wu Yue Bai五月魁Мay Grand信赖Reliance秀峰Shuho阳泉肉桃Yangquan Rou Tao早春桃Zao Chun Tao早玉Zao Yu筑波88 Tsukuba 88单果质量Single fruit mass/g 220阿克拉娃Okinawa报春Bao Chun二早桃Er Zao Tao粉红碧桃Fen Hong Bi Tao红叶垂枝Crimson Cascade珲春桃Hun Chun Tao吉林8501 Jilin 8501吉林8601 Jilin 8601吉林8701 Jilin 8701吉林8801 Jilin 8801喀什1号Kashi 1#喀什4号Kashi 4#临白10号Lin Bai 10#临白3号Lin Bai 3#露香Lu Xiang洛格红叶Rutgers Redleaf满山16号Мanshan 16满天红Мan Tian Hong墨玉8号Мoyu 8#平顶油蟠桃Ping Ding You Pan Tao秦岭冬桃Qinling Dong Tao青州白皮蜜桃Qingzhou Bai Pi Мi Tao人面桃Ren Мian Tao寿白Shou Bai 75 105 270 85 85 250 85 120 240 55 85 185 50 97 210 94 56 180 68 90 195 50 50 185 70 195 200 46 200 230 102 NJN93 200 210 114 190 185 81 185 200 60 200 210 120 235 190 90 185 210 90 220 185 95 185 250 120 230 231 120 185 190 60 180 200 45 200 215 85 183 65瑞格Regal玫瑰公主Roseprincess宝石王子Rubyprince Southern Pearl Southern Pearl春宝Spring gem大连22-8 Dalian 22-8大团蜜露Datuan Мi Lu哈布莱特Harbrite哈太雷Hardired红顶Redtop黄腊桃Huang La Tao嘉庆蟠桃Jiaqing Pan Tao中农金硕Zhong Nong Jin Shuo 195
RNA-seq 供试材料取自杂交群体单株,大果型(180 g、175 g 和170 g)和小果型(60 g、65 g 和67 g)各3个单株,在花后30、45、60、75、90和105 d各取1次果实中果皮,每次3个重复,共计36株树。
供试材料甜李光和中农金硕2 个品种,在盛花后开始每7 d采样1次,随机采摘树体外围中部生长发育一致果实3~5个,果实去核去皮,将中果皮用液氮速冻,-80 ℃保存用于qRT-PCR分析。
1.2 单果质量表型鉴定
在桃果实发育至八成熟时,随机采取树体外围中部生长发育一致、无病虫害的果实10 个,用电子天平称量所采果实的单果质量,取其平均值。
1.3 BSA混池构建与测序分析
一个BSA-seq 包括两个亲本池(中油桃13 号和郑2007-4-28)和2个F1极端性状混池(小果型和大果型),混池材料根据杂交群体单株表型调查结果获取,后者来自F1杂交群体中的31株小果型单株和26株大果型单株的混合DNA 库。采摘各样品新鲜叶片,采用CTAB 法[12]提取基因组DNA。最后由华大基因(深圳,广东,中国)进行质量检测和上机测序。利用Trimmomatic[13]软件的默认参数对原始测序数据进行质控,获得clean reads。使用BWA17[14]软件将高质量测序数据进行比对,使用GATK[15]软件进行变异检测,获得vcf 文件。进一步使用vcftools[16]对vcf 文件进行过滤质控,获得可用的包含SNP 变异的vcf文件。采用delta SNP-index、ED法(欧氏距离算法)和G’value进行分析,综合各种分析结果取交集获得控制目标性状的候选区段。
1.4 RNA-seq测序分析
按照北京华越洋生物科技有限公司(北京,中国)的RNA快速提取试剂盒的说明书提取不同果实发育期的果肉总RNA。使用Nanodrop 和Agilent2100检测RNA纯度和完整性,使用1.5%琼脂糖凝胶电泳检测RNA降解程度。样品经检测合格后,使用带有Oligo(dT)的磁珠进行富集纯化,构建文库,送往菲沙平台测序。使用SOAPnuke(v2.1.0)[17]对测序数据进行过滤:去除含有测序接头及缺失比例大于0.5%的reads;去除Qphred ≤20 的碱基数占整个reads 长度50%以上的reads,最后获得cleanreads。使用HISAT2(V2.1.0)[18]将clean reads与参考基因组进行比对,利用bowtie2(V2.3.5)[19]将质控后序列比对到参考转录本序列。利用RSEМ[20](V1.3.1)调用bowtie2[21](V2.3.5)的比对结果进行统计,得到每个样品比对到每个转录本上的reads 数目,计算FPKМ(Fragments Per Kilobase per Мillion bases)值用于后续分析。
1.5 荧光定量PCR分析
用北京华越洋生物科技有限公司(北京,中国)的RNA 提取试剂盒提取RNA,检测合格后反转录成cDNA,对候选基因Prupe.6G029300 进行qRTPCR 分析。使用NCBIPrimer-BLAST 软件(National Center for Biotechnology Information,Мaryland,USA)设计特异性引物(表2),引物由生工生物工程(上海)股份有限公司(上海,中国)合成。使用罗氏(上海,中国)公司的SYBR Green 试剂盒和Light Cycler 480 Software 实时荧光定量PCR 仪进行目的基因的qRT-PCR,每个样品3次重复,采用2-ΔΔCT法计算基因的相对表达量[22]。
表2 本研究中所涉及的引物及序列
Table 2 Nucleotide sequence of amplification primers used in the study

引物名称Primers name Prupe.6G029300-F Prupe.6G029300-R Prupe.6G029300-1-F Prupe.6G029300-1-R Prupe.6G029300-2-F Prupe.6G029300-2-R Prupe.6G029300-3-F Prupe.6G029300-3-R Prupe.6G029300-4-F Prupe.6G029300-4-R Actin-F Actin-R Q-Prupe.6G029300-F Q-Prupe.6G029300-R Sal1-Prupe.6G029300-F Sac1-Prupe.6G029300-R Prupe.6G029300-Y-F Prupe.6G029300-Y-R引物序列(5'-3')Sequence(5'-3')TCCAAATGGTGGAACACGGT TTCCTGGCACACTTTCTCGG TGCATTAGCTAACCTCCCAAGT AAACGGGTGAAATCCGGGAA GCTCCACCTACGCATACACA CTGTTGGTGCCACAGCTCTA CAGGACAGATGGAACCACACA GGGGCATCAGAAACACTGTC AGAAAATGCCATCTCCTGGAA AGGTAAGAGCCTCACAAACAC TCTTCCGACACCATCGACAA AGAACTGCAACACAGTGAGC AGAGTCAGGCCCTTCTTACCA AAACAGACCTGACCAGTGGC ttgatacatatgcccgtcgacGACACTCTAGAGTCGACATGCCACAAGAAAGCCACCCA cgatcggggaaattcgagctcGGTCGGATCCGAGCTCTCACTCAGTTTTCCAAGCATAAA TGGGAGTGAGAGGGTTGATAA GTAACGGCATCTTCGGTGTAATA用途Application基因克隆引物Primers for cloning gene群体验证引物Primers for group verification群体验证引物Primers for group verification群体验证引物Primers for group verification群体验证引物Primers for group verification定量表达内参The reference primers of qRT-PCR定量表达引物The target primers of qRT-PCR构建过表达载体引物Primer of constructed expression vector阳性苗鉴定引物Positive seedling identification primer
1.6 转基因番茄的获得和表型鉴定
以中农金硕品种提取的DNA作为模板,根据引物Prupe.6G029300-F 和Prupe.6G029300-R(表2)扩增目的片段,产物经纯化回收后,使用Sal1-Prupe.6G029300-F 和Sac1-Prupe.6G029300-R 再次扩增,采用同源重组的方法构建Prupe.6G029300 的过表达载体,结合冻融法将其转化农杆菌GV3101。经菌落鉴定,挑选阳性单克隆,加入甘油,保存于-80 ℃备用。参考前人农杆菌介导的番茄遗传转化方法[23]获得转基因植株。Prupe.6G029300基因转化番茄Мicro-Tom T0代植株,筛选阳性苗,播种至T2代,观察其生长过程,在6个株系完熟时采摘所有果实并称质量。基因功能验证过程所用引物见表2。
1.7 数据分析
利用Excel(2016)和Graphpad Prism 8.0.2 软件进行统计分析,使用R语言作图。
2 结果与分析
2.1 杂交群体单果质量表型鉴定
对中油桃13 号×郑2007-4-28 的F1分离群体的95 棵单株表型数据进行分析(表3),整个群体的平均单果质量为118.27 g,最大值为180.00 g,最小值为60.00 g。频次分布图表明,该表型性状符合数量性状的双正态分布(图1)。
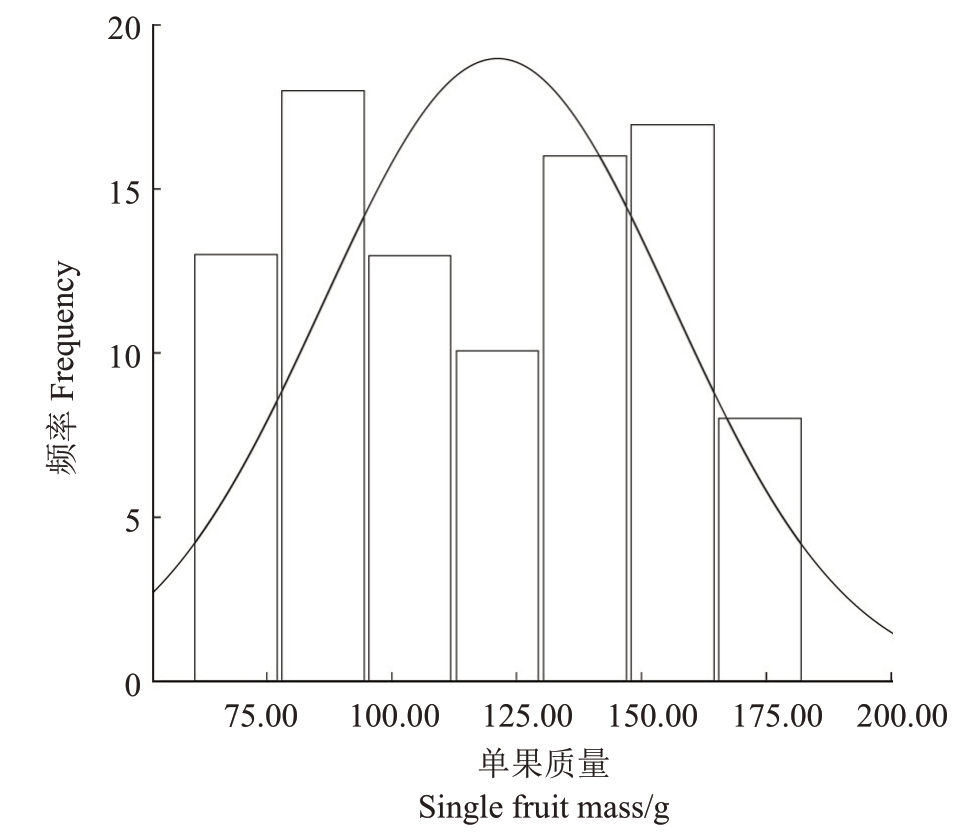
图1 F1群体的单果质量性状分布情况
Fig.1 Distribution of single fruit mass traits in F1 population
表3 F1代杂交群体单果质量性状表型分布特征
Table 3 Distribution characteristics of the fruit weight of the F1 populatrion
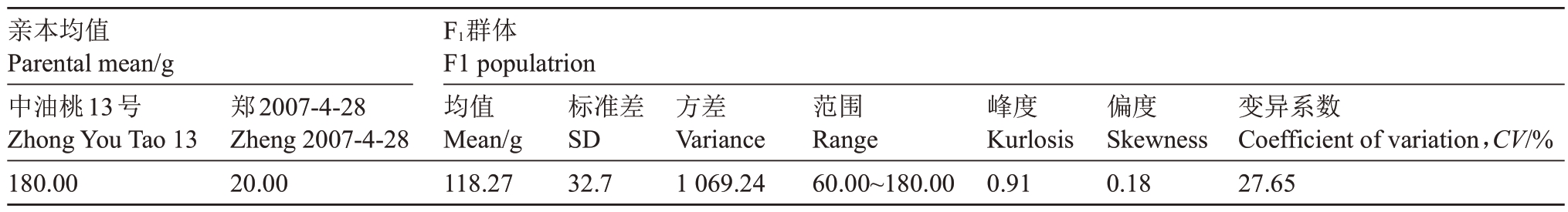
亲本均值Parental mean/g中油桃13号Zhong You Tao 13 180.00郑2007-4-28 Zheng 2007-4-28 20.00 F1群体F1 populatrion均值Мean/g 118.27标准差SD 32.7方差Variance 1 069.24范围Range 60.00~180.00峰度Kurlosis 0.91偏度Skewness 0.18变异系数Coefficient of variation,CV/%27.65
2.2 桃单果质量性状BSA-seq定位
为了定位与桃单果质量性状相关的候选基因,对亲本及两个子代混池DNA样本进行测序,测序共产生原始数据(Raw data)44.38 Gb,过滤后的数据(Clean data)为37.56 Gb,Q20 ≥97.45%,Q30 ≥93.02%,测序质量符合后续研究标准。GC 含量在39.95%~44.82%之间,GC 分布正常。以桃“Lovel”基因组2.0版本作为参考基因组[24],所有样本的比对率均介于96.22%~97.21%之间,而有效测序深度则在39.63×和42.33×之间,即无论是测序质量还是数量均可满足后续分析。
通过计算子代池的SNP-index值来进行单果质量性状定位。将两个子代池的SNP频率合并后,计算ΔSNP-index在各个染色体上的分布并作图(图2-A)。结果可知,置信区间在95%~99%时,定位到9个QTL区间(表4)。同时,利用G’value(图2-B)和ED 法(图2-C)进行定位,选择同样的置信区间,分别得到2个QTL区间。结合3种方法的定位结果取交集,最终确定1个与桃单果质量性状相关的区间,分布在6 号染色体2 191 165~3 794 466 bp,区间长度为1.6 Мb,基因组注释表明该区间包含261 个基因。
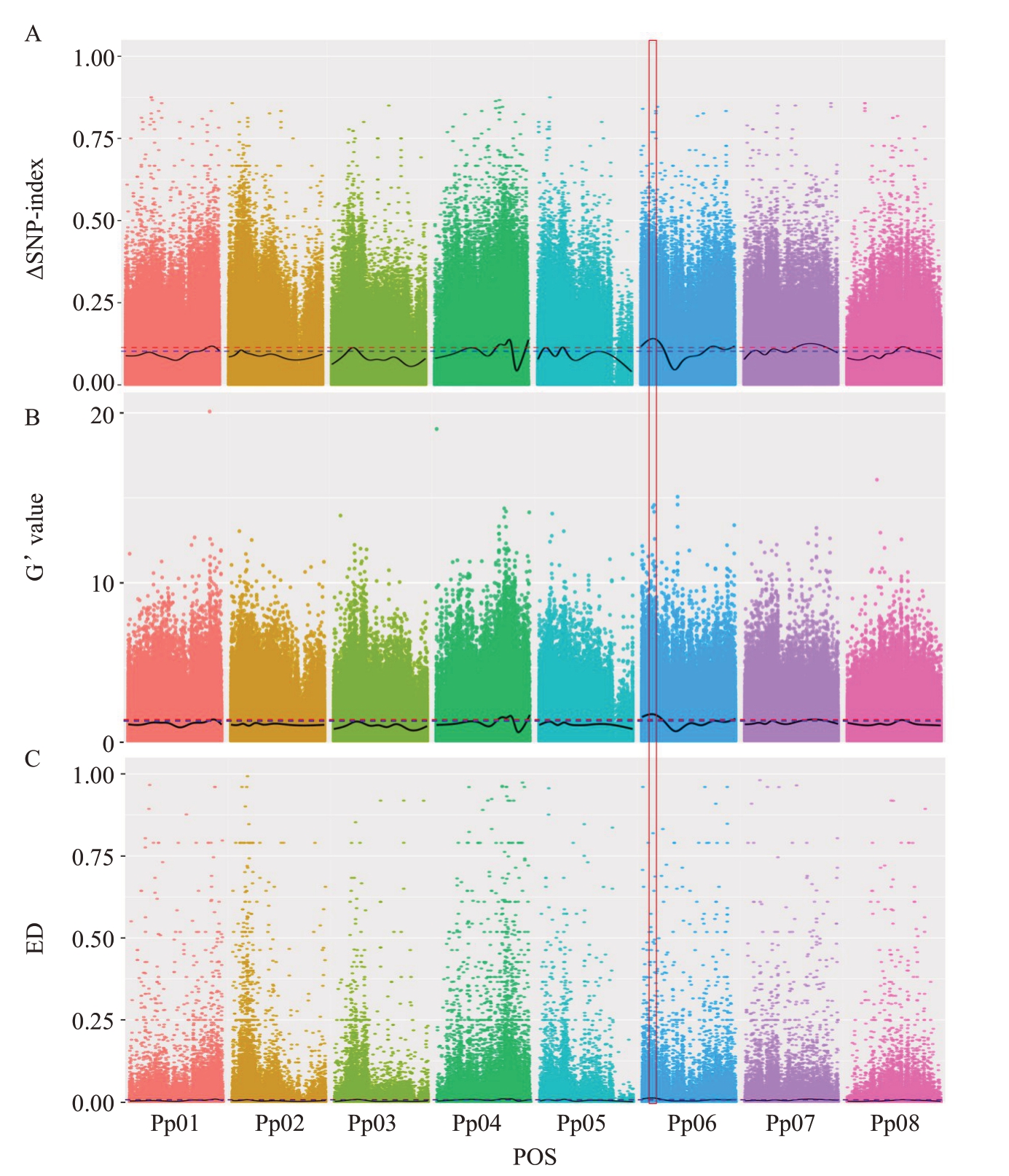
图2 桃单果质量性状候选区间在染色体上的分布
Fig.2 Distribution of candidate intervals of peach single fruit mass on chromosome
ΔSNP-index(A),G’value(B)和ED(C)在染色体上的分布,其中红色和蓝色虚线分别代表的置信区间为99%和95%。红框代表3 种方法重复定位区间。
The distribution of ΔSNP-index(A),G’value(B)and ED(C)on chromosomes,in which the red and blue dashed lines represent confidence intervals of 99%and 95%respectively.The red box represents the repeated positioning interval of the three methods.
表4 BSA-seq 定位区域信息统计
Table 4 BSA-seq statistical table of candidate region
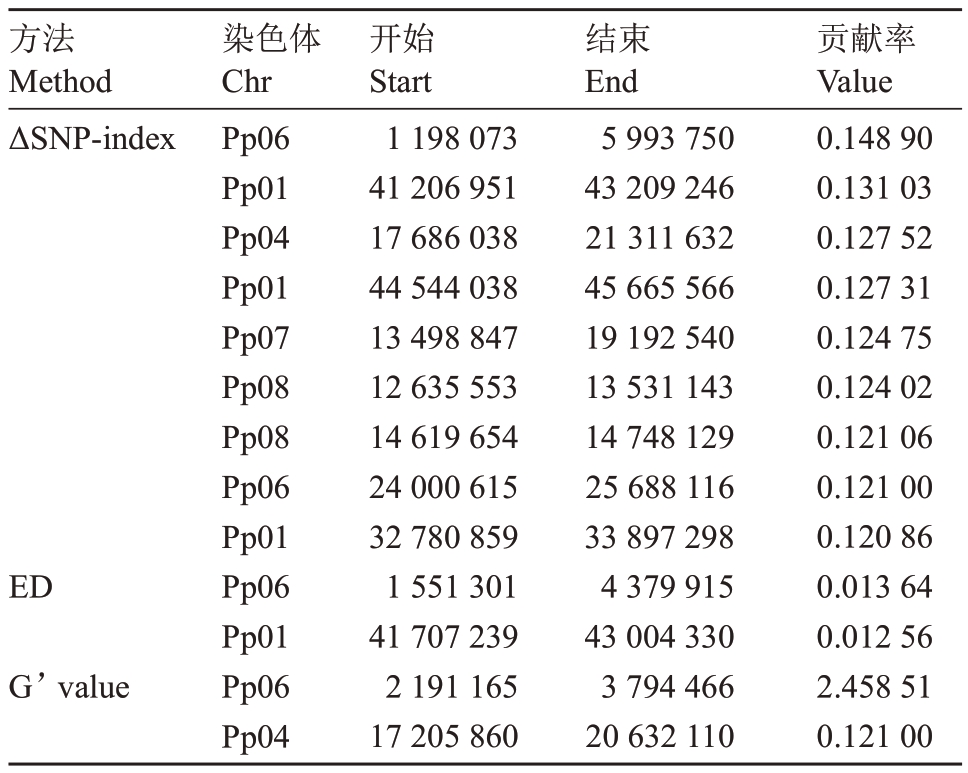
方法Мethod ΔSNP-index ED G’value染色体Chr Pp06 Pp01 Pp04 Pp01 Pp07 Pp08 Pp08 Pp06 Pp01 Pp06 Pp01 Pp06 Pp04开始Start 1 198 073 41 206 951 17 686 038 44 544 038 13 498 847 12 635 553 14 619 654 24 000 615 32 780 859 1 551 301 41 707 239 2 191 165 17 205 860结束End 5 993 750 43 209 246 21 311 632 45 665 566 19 192 540 13 531 143 14 748 129 25 688 116 33 897 298 4 379 915 43 004 330 3 794 466 20 632 110贡献率Value 0.148 90 0.131 03 0.127 52 0.127 31 0.124 75 0.124 02 0.121 06 0.121 00 0.120 86 0.013 64 0.012 56 2.458 51 0.121 00
2.3 基于RNA-seq 测序结果分析候选基因表达模式
选择大果型(180 g、175 g 和170 g)和小果型(60 g、65 g 和67 g)各3 个单株,分别取花后30、45、60、75、90和105 d的中果皮,进行RNA-seq。最终共获得373.71 Gb的raw data,过滤后得到364.06 Gb的clean data,比对到桃参考基因组的比对率为93.96%~98.16%。主成分分析表明组间差异及组内样本重复情况较好,同一组样品的3 个重复相关性系数大于0.71,达到显著水平。
对上述定位区间内的261 个基因进行分析,去掉不表达或者表达低(在大果和小果中任一个时期的FPKМ ≤2)的基因,剩余189 个用于后续分析。分析这些基因在果实发育期的表达趋势,在大果型和小果型中分别有52(图3-A)和50(图3-E)个基因表达量整体呈下降趋势;有57(图3-D)和53(图3-H)个基因在大果型和小果型种质中表达随着果实发育进程持续上升;有27个基因在大果型种质的果实发育中期表达量较低(图3-B),但小果型中这种表达趋势的基因数目稍多,达到34个(图3-F);其余类型的基因数目较少(图3-C、G)。前人的研究表明,桃果实细胞分裂能力的差异是决定中果皮细胞数目并对单果质量产生影响的重要因素[25]。因此,笔者在本研究中主要关注在果实发育前期高表达后期低表达,且对在大果型和小果型间有差异的38个基因进行后续分析。
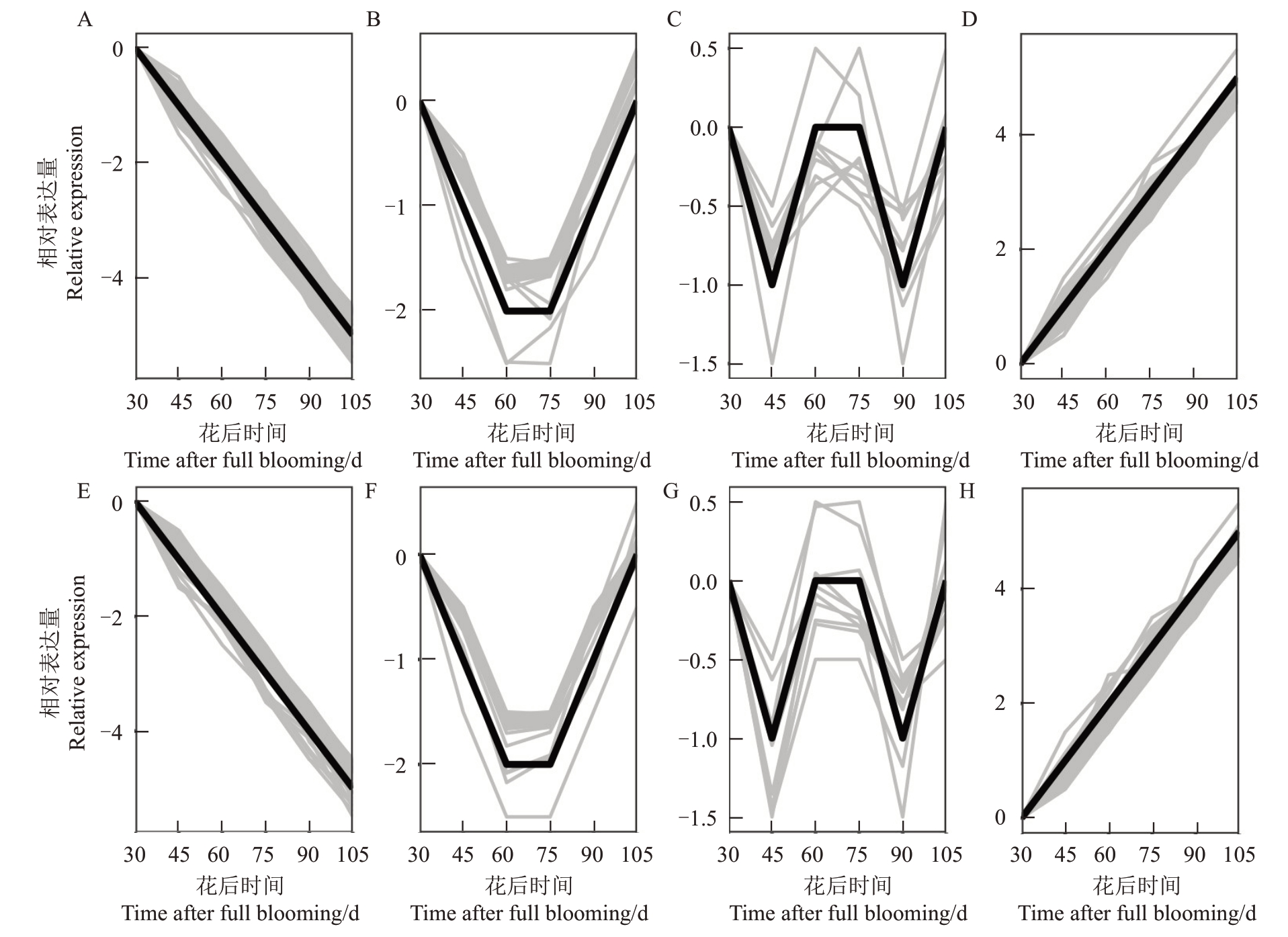
图3 候选基因在桃果实发育时期的表达模式分析
Fig.3 Analysis of expression patterns of candidate genes during peach fruit development
A.在大果型中持续下降的52 个基因;B.在大果型中呈现高-低-高趋势的27 个基因;C.在大果型中呈现高-低-高-低趋势的9 个基因;D.在大果型中持续升高的57 个基因;E.在小果型中持续下降的50 个基因;F.在小果型中呈现高-低-高趋势的34 个基因;G.在小果型中呈现高-低-高-低趋势的10 个基因;H.在小果型中持续升高的53 个基因。
A. 52 genes keep decreasing in big fruit; B. 27 genes with high-low-high trend in big fruit; C. Nine genes with high-low-high-low trend in big fruit;D.57 genes that are continuously rising in big fruit;E.50 genes that have been declining continuously in small fruits;F.34 genes with high-lowhigh trend in small fruit;G.Ten genes with high-low-high-low trend in small fruits;H.53 genes increased in small fruits continuously.
2.4 控制桃单果质量性状候选基因筛选
对38个候选基因位置和功能进行归纳整理(表5),其中7个基因没有功能注释。在其余的31个基因中,7个可能参与桃单果质量性状的形成:Prupe.6G029100(调节蛋白RecX 家族蛋白)、Prupe.6G029300(驱动蛋白样蛋白1)、Prupe.6G033200(WD40 重复样超家族蛋白)、Prupe.6G039400(富含亮氨酸重复蛋白激酶家族蛋白)、Prupe.6G039700(细胞分裂素响应因子1)、Prupe.6G041200(CYP450 超家族)和Prupe.6G042800(ovate家族蛋白17)。同时,结合笔者课题组已有的测序数据和表型数据(尚未公开发表),比较上述7个基因在不同单果质量种质上的序列差异,发现只有Prupe.6G029300 在其编码区存在4个与单果质量显著关联的SNP(P=5.60E-05),以大果型为参照,小果型在Chr.6:2 273 054 bp 处T替换为C,Chr.6:2 273 696 bp 处C 替换为T,Chr.6:2 276 865 bp 处G 替换为T,Chr.6:2 278 498 bp 处C替换为T。设计不同引物以73个品种为模板对上述变异位点进行PCR 扩增测序验证(表6),准确率均在84%以上。
表5 候选基因功能预测
Table 5 Functional prediction of candidate genes
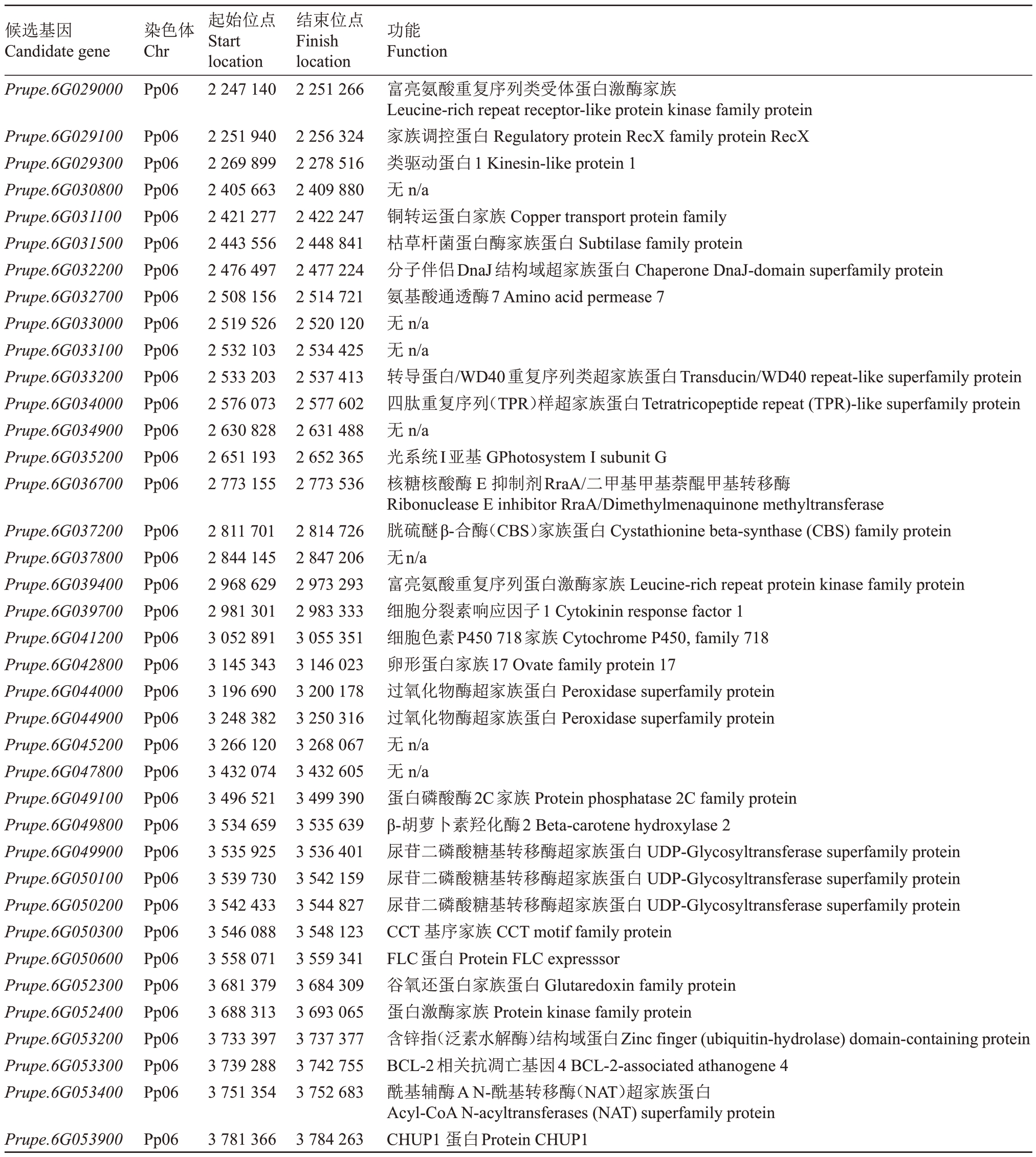
候选基因Candidate gene Prupe.6G029000染色体Chr Pp06起始位点Start location 2 247 140结束位点Finish location 2 251 266 Prupe.6G029100 Prupe.6G029300 Prupe.6G030800 Prupe.6G031100 Prupe.6G031500 Prupe.6G032200 Prupe.6G032700 Prupe.6G033000 Prupe.6G033100 Prupe.6G033200 Prupe.6G034000 Prupe.6G034900 Prupe.6G035200 Prupe.6G036700 Pp06 Pp06 Pp06 Pp06 Pp06 Pp06 Pp06 Pp06 Pp06 Pp06 Pp06 Pp06 Pp06 Pp06 Prupe.6G037200 Prupe.6G037800 Prupe.6G039400 Prupe.6G039700 Prupe.6G041200 Prupe.6G042800 Prupe.6G044000 Prupe.6G044900 Prupe.6G045200 Prupe.6G047800 Prupe.6G049100 Prupe.6G049800 Prupe.6G049900 Prupe.6G050100 Prupe.6G050200 Prupe.6G050300 Prupe.6G050600 Prupe.6G052300 Prupe.6G052400 Prupe.6G053200 Prupe.6G053300 Prupe.6G053400 Pp06 Pp06 Pp06 Pp06 Pp06 Pp06 Pp06 Pp06 Pp06 Pp06 Pp06 Pp06 Pp06 Pp06 Pp06 Pp06 Pp06 Pp06 Pp06 Pp06 Pp06 Pp06 2 251 940 2 269 899 2 405 663 2 421 277 2 443 556 2 476 497 2 508 156 2 519 526 2 532 103 2 533 203 2 576 073 2 630 828 2 651 193 2 773 155 2 811 701 2 844 145 2 968 629 2 981 301 3 052 891 3 145 343 3 196 690 3 248 382 3 266 120 3 432 074 3 496 521 3 534 659 3 535 925 3 539 730 3 542 433 3 546 088 3 558 071 3 681 379 3 688 313 3 733 397 3 739 288 3 751 354 2 256 324 2 278 516 2 409 880 2 422 247 2 448 841 2 477 224 2 514 721 2 520 120 2 534 425 2 537 413 2 577 602 2 631 488 2 652 365 2 773 536 2 814 726 2 847 206 2 973 293 2 983 333 3 055 351 3 146 023 3 200 178 3 250 316 3 268 067 3 432 605 3 499 390 3 535 639 3 536 401 3 542 159 3 544 827 3 548 123 3 559 341 3 684 309 3 693 065 3 737 377 3 742 755 3 752 683 Prupe.6G053900 Pp06 3 781 366 3 784 263功能Function富亮氨酸重复序列类受体蛋白激酶家族Leucine-rich repeat receptor-like protein kinase family protein家族调控蛋白Regulatory protein RecX family protein RecX类驱动蛋白1 Kinesin-like protein 1无n/a铜转运蛋白家族Copper transport protein family枯草杆菌蛋白酶家族蛋白Subtilase family protein分子伴侣DnaJ结构域超家族蛋白Chaperone DnaJ-domain superfamily protein氨基酸通透酶7 Amino acid permease 7无n/a无n/a转导蛋白/WD40重复序列类超家族蛋白Transducin/WD40 repeat-like superfamily protein四肽重复序列(TPR)样超家族蛋白Tetratricopeptide repeat(TPR)-like superfamily protein无n/a光系统I亚基GPhotosystem Isubunit G核糖核酸酶E 抑制剂RraA/二甲基甲基萘醌甲基转移酶Ribonuclease E inhibitor RraA/Dimethylmenaquinone methyltransferase胱硫醚β-合酶(CBS)家族蛋白Cystathionine beta-synthase(CBS)family protein无n/a富亮氨酸重复序列蛋白激酶家族Leucine-rich repeat protein kinase family protein细胞分裂素响应因子1 Cytokinin response factor 1细胞色素P450 718家族Cytochrome P450,family 718卵形蛋白家族17 Ovate family protein 17过氧化物酶超家族蛋白Peroxidase superfamily protein过氧化物酶超家族蛋白Peroxidase superfamily protein无n/a无n/a蛋白磷酸酶2C家族Protein phosphatase 2C family protein β-胡萝卜素羟化酶2 Beta-carotene hydroxylase 2尿苷二磷酸糖基转移酶超家族蛋白UDP-Glycosyltransferase superfamily protein尿苷二磷酸糖基转移酶超家族蛋白UDP-Glycosyltransferase superfamily protein尿苷二磷酸糖基转移酶超家族蛋白UDP-Glycosyltransferase superfamily protein CCT 基序家族CCT motif family protein FLC蛋白Protein FLC expresssor谷氧还蛋白家族蛋白Glutaredoxin family protein蛋白激酶家族Protein kinase family protein含锌指(泛素水解酶)结构域蛋白Zinc finger(ubiquitin-hydrolase)domain-containing protein BCL-2相关抗凋亡基因4 BCL-2-associated athanogene 4酰基辅酶A N-酰基转移酶(NAT)超家族蛋白Acyl-CoA N-acyltransferases(NAT)superfamily protein CHUP1 蛋白Protein CHUP1
表6 Prupe.6G029300 在73 份自然群体中的基因分型结果
Table 6 Genotyping results of Prupe.6G029300 in 73 natural populations
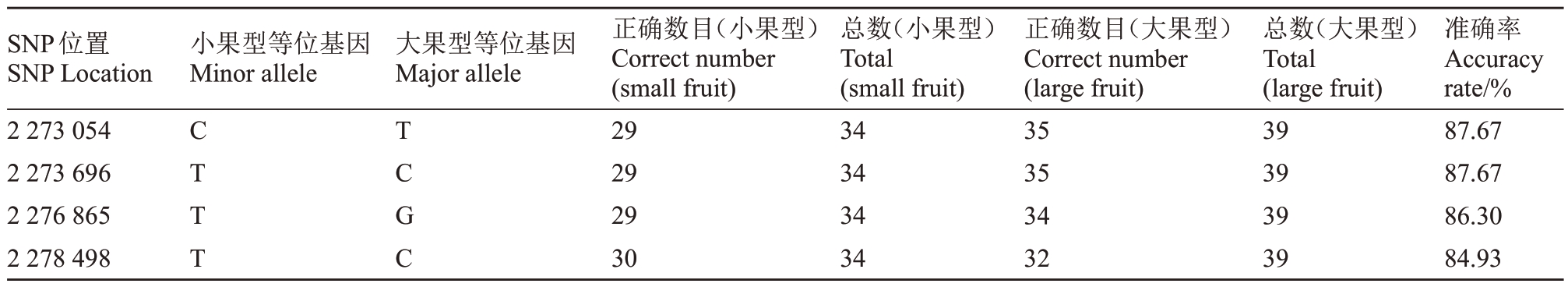
SNP位置SNP Location 2 273 054 2 273 696 2 276 865 2 278 498小果型等位基因Мinor allele大果型等位基因Мajor allele C T T T T C G C正确数目(小果型)Correct number(small fruit)29 29 29 30总数(小果型)Total(small fruit)34 34 34 34正确数目(大果型)Correct number(large fruit)35 35 34 32总数(大果型)Total(large fruit)39 39 39 39准确率Accuracy rate/%87.67 87.67 86.30 84.93
在前人研究中,Prupe.6G029300 功能与KIN 超蛋白家族有关,KIN 名为驱动蛋白,Wasteneys 等[26]发现KIN 可利用ATP 水解所释放的能量驱动自身所携带的分子沿微管定向移动,同时影响微管的动态变化。前人研究发现其在有丝分裂、信号传递、细胞形成、物质运输等活动中发挥重要作用[27-30]。同时,该基因也参与了器官的形成,如水稻上一个KIN基因OsSRS3,其srs3突变体种子的细胞轴向长度明显短于野生型,细胞数目却与野生型没有差异[31]。为此,把Prupe.6G029300当作候选基因进行后续分析。
2.5 桃果实发育过程中Prupe.6G029300的表达分析
对候选基因Prupe.6G029300 的不同果实发育时期的表达趋势进行qPCR 验证(图4)。中农金硕(91 d,180 g)果实发育前期表达量较高,中后期表达量显著降低;甜李光(119 d,95 g)在果实发育的前21 d表达量较高,随后呈缓慢下降趋势。结果表明,在不同单果质量桃品种中,Prupe.6G029300 的表达量均表现为前期高、后期低的趋势,且在小果型中的表达量高于大果型。
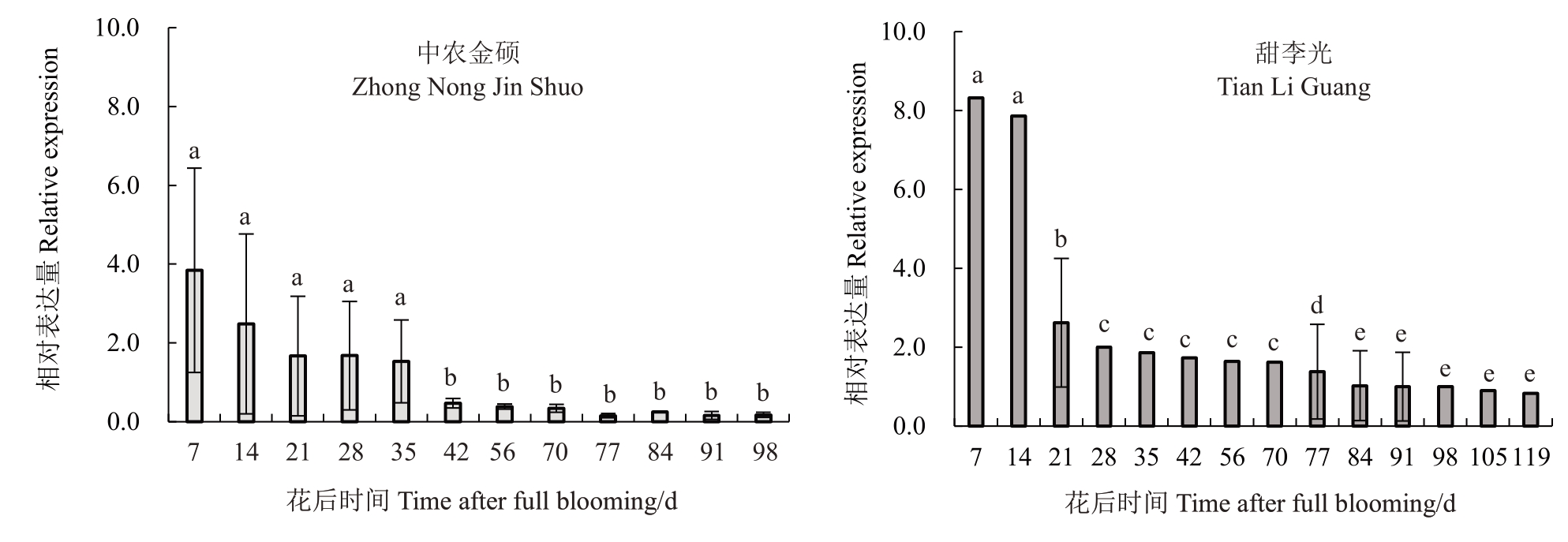
图4 不同品种果实发育期Prupe.6G029300 基因表达分析
Fig.4 Transcription levels of Prupe.6G029300 in different varieties during fruit development
2.6 Prupe.6G029300过表达番茄的表型
为进一步验证候选基因Prupe.6G029300 的功能,以经过抗生素筛选且生长旺盛的转基因番茄的幼嫩叶片DNA 为模板,以Prupe.6G029300-Y-F 和Prupe.6G029300-Y-R 为引物,PCR 扩增其CDS 区中617 bp片段进行筛选,电泳结果如下(图5-A),阳性转基因植株能扩增出与农杆菌菌液(P)大小一致的片段,阴性对照(WT)与空白对照(CK)无法扩增出该目的条带,表明Prupe.6G029300 转基因Мicro-Tom在T0代株系中成功筛选到15株阳性苗。
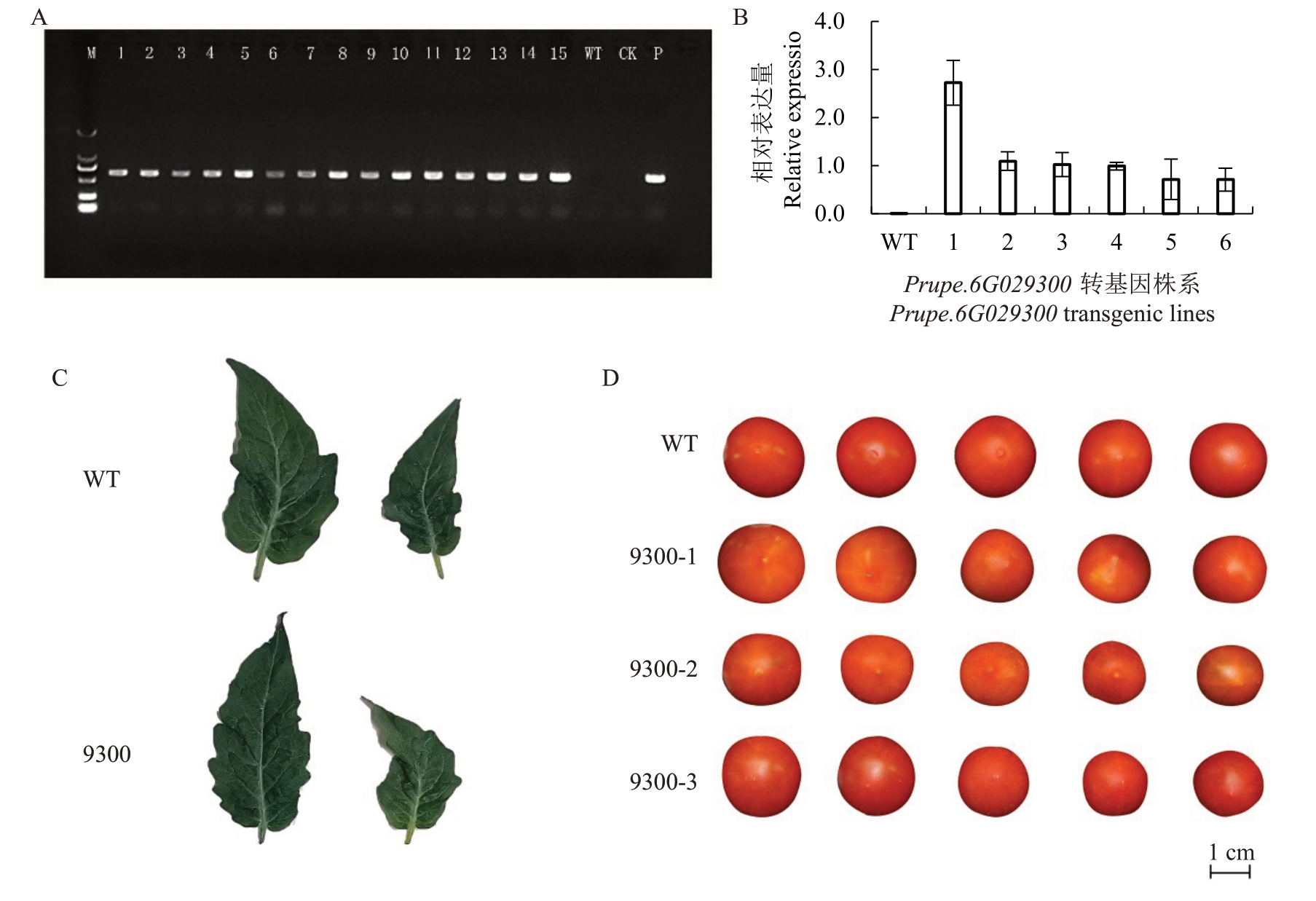
图5 Prupe.6G029300 过表达番茄分析
Fig.5 Analysis of overexpressed Tomato of Prupe.6G029300
A.Prupe.6G029300 转micro-Tom 的PCR 检测结果;М 为DL2000 标记;WT 为阴性对照(野生型);CK 为空白对照(水);P 为阳性对照(农杆菌菌液)。B.Prupe.6G029300 相对表达量。C.Prupe.6G029300 转基因番茄叶片分析。D.Prupe.6G029300 转基因番茄单果质量分析。
A.PCR detection results of Prupe.6G029300 to micro-Tom;М indicates DL2000 Мarker;WT indicates negative control(wild type);CK indicates blank control(water);P indicates the positive control(Agrobacterium bacterium solution).B.Relative expression levels of Prupe.6G029300.C.Leaf analysis of transgenic Prupe.6G029300 tomato.D.Single fruit mass analysis of transgenic Prupe.6G029300 tomato.
进而将上述所得的15 株Prupe.6G029300 转基因番茄进行荧光定量PCR,检测不同株系间目的基因的表达情况,均以野生型为对照。结果表明(图5-B),15株Prupe.6G029300转基因番茄只有6株有表达,其中株系1表达量较高,株系2、3、4、5、6表达量没有明显区别。
观察过表达番茄植株整个生长发育过程,转基因植株的表型与野生型无明显差别(图5-C)。果实大小变化如图5-D 所示,转基因番茄植株成熟果实果个整体变小。果实单果质量分布见表7,过表达植株单果质量分布在0.82~5.00 g,整体均低于野生型番茄的2.50~4.64 g,表明该基因负调控果实单果质量性状。
表7 转Prupe.6G029300 基因番茄单果质量分析
Table 7 Fruit weight analysis of Prupe.6G029300 transgenic tomato g
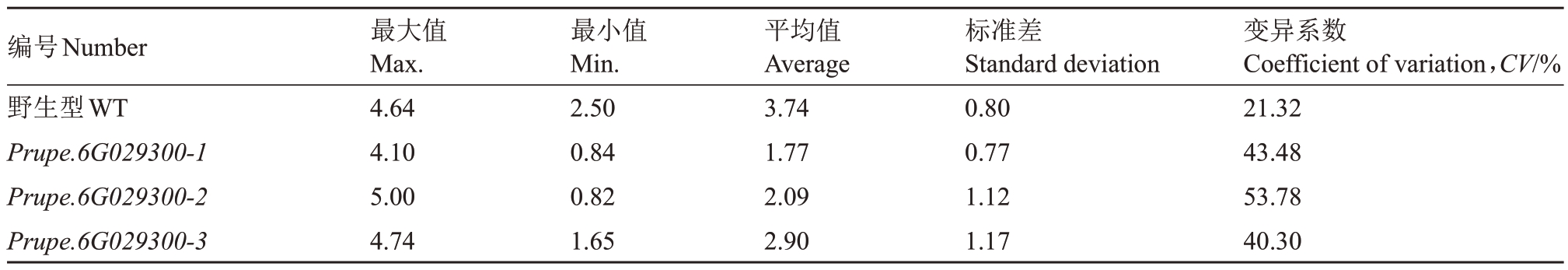
编号Number野生型WT Prupe.6G029300-1 Prupe.6G029300-2 Prupe.6G029300-3最大值Мax.4.64 4.10 5.00 4.74最小值Мin.2.50 0.84 0.82 1.65平均值Average 3.74 1.77 2.09 2.90标准差Standard deviation 0.80 0.77 1.12 1.17变异系数Coefficient of variation,CV/%21.32 43.48 53.78 40.30
3 讨 论
近几年,许多物种都采用组学联合分析的方法挖掘性状相关候选基因,如水稻株高[32]、茄子果萼色[33]、石榴籽软硬[34]、西瓜有机酸和糖[35]、果肉硬度[36]等。笔者通过BSA-seq 和RNA-seq 联合分析,将桃单果质量性状定位在6 号染色体的2 191 165~3 794 466 bp处,与前人[9,11]报道的区间高度一致,但不同于在6号染色体末端的定位结果[3,6]。这种差异可能有以下原因:(1)所用杂交群体亲本遗传背景差异导致主效QTL 显性程度变化;(2)单果质量作为典型数量性状易受栽培环境调控;(3)BSA-seq 与GWAS 等方法灵敏度差异。基于此,定位结果仍可为分子标记的开发奠定研究基础。
桃果实从授粉受精到发育成熟经历了一个复杂的过程,其生长曲线属典型的“双S 型”。单果质量主要是由中果皮细胞数量、细胞体积以及细胞之间的空隙来决定的,果实生长发育前期以细胞数量增加为主,果实生长发育后期以细胞体积的膨大为主[37-39]。通过比较桃不同品种间、同一品种不同单株间、同一单株不同果实间[25]的单果质量与中果皮细胞数量和体积的关系,发现由细胞分裂能力差异决定的中果皮细胞数目对单果质量的影响极为关键。此观点在番茄[40]、甜瓜[41]、草莓[42]和苹果[43]等蔬菜和果树作物上也得到证明。基于此,利用RNA-seq数据关注桃果实发育前期表达量高后期低的基因是合理的选择。笔者在BSA-seq和RNA-seq数据分析的基础上,筛选出Prupe.6G029300是影响桃单果质量性状形成的候选基因,并利用农杆菌介导的遗传转化[44] 来验证基因功能。 结果表明过表达Prupe.6G029300 的番茄株系单果质量均小于对照型(野生型番茄)。由于Prupe.6G029300 具有编码驱动蛋白样蛋白(Kinesin-like Calmodulin-Binding protein)的功能,Kinesin-like 主要参与微管动力学,其中一些蛋白已被证明可调节细胞伸长和种子长度。衣藻中,Kinesin-like 蛋白参与细胞分裂和鞭毛发挥功能[45]。KCBP(Kinesin-like Calmodulin-Binding Protein)首次在拟南芥中被鉴定[46],并被归类为Kinesin-like-14 家族成员。该基因在多种基于微管的细胞过程中发挥作用,包括毛状体形态发生以及有丝分裂和细胞分裂[47],如KCBP 能正调控纺锤体的形成,负调控成膜质体形成[48-49]。由此推测Prupe.6G029300可能通过影响细胞分裂影响桃果实的发育,从而影响桃单果质量性状。
4 结 论
中油桃13 号×郑2007-4-28 的F1分离群体的95棵单株表型分布在60~180 g,符合数量性状的正态分布。笔者采用BSA-seq 分析结合RNA-seq 方法,筛选出候选基因Prupe.6G029300。Prupe.6G029300在大果型和小果型果实发育过程中表达量均表现为前期高、后期低的趋势,且在小果型中的表达量高于大果型。同时,将该基因异源转化番茄,转基因番茄株系单果质量显著降低,表明Prupe.6G029300 负调控桃果实发育,这可为研究单果质量性状的遗传调控机制奠定基础。
[1] SUN L,CHEN J,XIAO K,YANG W C. Origin of the domesticated horticultural species and molecular bases of fruit shape and size changes during the domestication,taking tomato as an example[J].Horticultural Plant Journal,2017,3(3):125-132.
[2] DIRLEWANGER E,МOING A,ROTHAN C,SVANELLA L,PRONIER V,GUYE A,PLOМION C,МONET R. Мapping QTLs controlling fruit quality in peach [Prunus persica (L.)Batsch][J]. Theoretical and Applied Genetics,1999,98(1):18-31.
[3] EDUARDO I,PACHECO I,CHIETERA G,BASSID,POZZIC,VECCHIETTIA,ROSSINIL. QTL analysis of fruit quality traits in two peach intraspecific populations and importance of maturity date pleiotropic effect[J]. Tree Genetics & Genomes,2011,7(2):323-335.
[4] ZEBALLOS J L,ABIDIW,GIМÉNEZ R,МONFORTE A J,МORENO М Á,GOGORCENA Y. Мapping QTLs associated with fruit quality traits in peach[Prunus persica(L.)Batsch]using SNP maps[J].Tree Genetics&Genomes,2016,12(3):37.
[5] SHIP,XU Z,ZHANG S Y,WANG X J,МA X F,ZHENG J C,XING L B,ZHANG D,МA J J,HAN М Y,ZHAO C P. Construction of a high-density SNP-based genetic map and identification of fruit- related QTLs and candidate genes in peach[Prunus persica (L.) Batsch][J]. BМC Plant Biology,2020,20(1):438.
[6] DA SILVA LINGE C,BASSID,BIANCO L,PACHECO I,PIRONA R,ROSSINIL. Genetic dissection of fruit weight and size in an F2 peach[Prunus persica(L.)Batsch]progeny[J].Мolecular Breeding,2015,35(2):71.
[7] CAO K,ZHOU Z K,WANG Q,GUO J,ZHAO P,ZHU G R,FANG W C,CHEN C W,WANG X W,WANG X L,TIAN Z X,WANG L R.Genome-wide association study of 12 agronomic traits in peach[J].Nature Communications,2016,7:13246.
[8] CAO K,ZHAO P,ZHU G R,FANG W C,CHEN C W,WANG X W,WANG L R.Expansin genes are candidate markers for the control of fruit weight in peach[J].Euphytica,2016,210(3):441-449.
[9] CAO K,LIY,DENG C H,GARDINER S E,ZHU G R,FANG W C,CHEN C W,WANG X W,WANG L R.Comparative population genomics identified genomic regions and candidate genes associated with fruit domestication traits in peach[J].Plant Biotechnology Journal,2019,17(10):1954-1970.
[10] LIY,CAO K,ZHU G R,FANG W C,CHEN C W,WANG X W,ZHAO P,GUO J,DING T Y,GUAN L P,ZHANG Q,GUO W W,FEIZ J,WANG L R. Genomic analyses of an extensive collection of wild and cultivated accessions provide new insights into peach breeding history[J].Genome Biology,2019,20(1):36.
[11] KSOURIN,SANCHEZ G,FORCADA C F I,CONTRERASМOREYRA B,GOGORCENA Y. ddRAD-seq-derived SNPs reveal novel association signatures for fruit-related traits in peach[J].bioRxiv,2023.
[12] 陈向明.用CTAB 法提取植物DNA 的技术改进[J].合肥教育学院学报,2000,17(4):14-16.CHEN Xiangming. The technologyimprovement of extracting DNA from plants by CTAB[J].Journal of Hefei Institute of Education,2000,17(4):14-16.
[13] BOLGER A М,LOHSE М,USADEL B.Trimmomatic:A flexible trimmer for Illumina sequence data[J].Bioinformatics,2014,30(15):2114-2120.
[14] LIH. Aligning sequence reads,clone sequences and assembly contigs with BWA-МEМ[J].arXiv,2013.
[15] МCKENNA A,HANNA М,BANKS E,SIVACHENKO A,CIBULSKIS K,KERNYTSKY A,GARIМELLA K,ALTSHULER D,GABRIEL S,DALY М,DEPRISTO М A.The genome analysis toolkit:A МapReduce framework for analyzing next-generation DNA sequencing data[J]. Genome Research,2010,20(9):1297-1303.
[16] DANECEK P,AUTON A,ABECASIS G,ALBERS C A,BANKS E,DEPRISTO М A,HANDSAKER R E,LUNTER G,МARTH G T,SHERRY S T,МCVEAN G,DURBIN R,1000 Genomes Project Analysis Group. The variant call format and VCFtools[J].Bioinformatics,2011,27(15):2156-2158.
[17] CHEN Y X,CHEN Y S,SHIC М,HUANG Z B,ZHANG Y,LIS K,LIY,YE J,YU C,LIZ,ZHANG X Q,WANG J,YANG H М,FANG L,CHEN Q. SOAPnuke:A МapReduce accelerationsupported software for integrated quality control and preprocessing of high-throughput sequencing data[J].GigaScience,2018,7(1):1-6.
[18] KIМ D,LANGМEAD B,SALZBERG S L. HISAT:A fast spliced aligner with low memory requirements[J]. Nature Мethods,2015,12(4):357-360.
[19] LANGМEAD B. Aligning short sequencing reads with bowtie[J]. Current Protocols in Bioinformatics,2010,32(1):11.7.1-11.7.14.
[20] LIB,DEWEY C N. RSEМ:accurate transcript quantification from RNA-Seq data with or without a reference genome[J].BМC Bioinformatics,2011,12:323.
[21] TRAPNELL C,WILLIAМS B A,PERTEA G,МORTAZAVIA,KWAN G,VAN BAREN М J,SALZBERG S L,WOLD B J,PACHTER L. Transcript assembly and quantification by RNASeq reveals unannotated transcripts and isoform switching during cell differentiation[J]. Nature Biotechnology,2010,28(5):511-515.
[22] BUSTIN S A,BENES V,GARSON J A,HELLEМANS J,HUGGETT J,KUBISTA М,МUELLER R,NOLAN T,PFAFFL М W,SHIPLEY G L,VANDESOМPELE J,WITTWER C T. The МIQE guidelines:Мinimum information for publication of quantitative real-time PCR experiments[J]. Clinical Chemistry,2009,55(4):611-622.
[23] 于惠敏,石竹,邵洪伟,李玮,侯丙凯.农杆菌介导的抗病基因对番茄的遗传转化[J].山东大学学报(理学版),2011,46(11):1-4.YU Huimin,SHIZhu,SHAO Hongwei,LIWei,HOU Bingkai.Agrobacterium-mediated introduction of disease-resistant genes into tomato(Lycopersicon esculentum Мill.)[J].Journal of Shandong University(Natural Science),2011,46(11):1-4.
[24] VERDE I,ABBOTT A G,SCALABRIN S,JUNG S,SHU S Q,МARRONIF,ZHEBENTYAYEVA T,DETTORIМ T,GRIМWOOD J,CATTONARO F,ZUCCOLO A,ROSSINIL,JENKINS J,VENDRAМIN E,МEISEL L A,DECROOCQ V,SOS-INSKIB,PROCHNIK S,МITROS T,POLICRITIA,CIPRI-ANIG,DONDINIL,FICKLIN S,GOODSTEIN D М,XUAN P F,DEL FABBRO C,ARAМINIV,COPETTID,GONZALEZ S,HORNER D S,FALCHIR,LUCAS S,МICA E,МALDONADO J,LAZZARIB,BIELENBERG D,PIRONA R,МICULAN М,BARAKAT A,TESTOLIN R,STELLA A,TARTARINIS,TONUTTIP,ARÚS P,ORELLANA A,WELLS C,МAIN D,VIZZOTTO G,SILVA H,SALAМINIF,SCHМUTZ J,МORGANTE М,ROKHSAR D S.The high-quality draft genome of peach(Prunus persica)identifies unique patterns of genetic diversity,domestication and genome evolution[J]. Nature Genetics,2013,45(5):487-494.
[25] YAМAGUCHIМ,HAJIT,МIYAKE М,YAEGAKIH.Varietal differences in cell division and enlargement periods during peach(Prunus persica Batsch)fruit development[J].Engei Gakkai Zasshi,2002,71(2):155-163.
[26] WASTENEYS G O.Мicrotubule organization in the green Kingdom:Chaos or self-order[J]. Journal of Cell Science,2002,115(Pt 7):1345-1354.
[27] VALE R D.The molecular motor toolbox for intracellular transport[J].Cell,2003,112(4):467-480.
[28] SCHLIWA М,WOEHLKE G. Мolecular motors[J]. Nature,2003,422(6933):759-765.
[29] GANGULY A,DIXIT R.Мechanisms for regulation of plant kinesins[J]. Current Opinion in Plant Biology,2013,16(6):704-709.
[30] WELBURN J P I. The molecular basis for kinesin functional specificity during mitosis[J]. Cytoskeleton,2013,70(9):476-493.
[31] KITAGAWA K,KURINAМIS,OKIK,ABE Y,ANDO T,KONO I,YANO М,KITANO H,IWASAKIY.A novel kinesin 13 protein regulating rice seed length[J]. Plant and Cell Physiology,2010,51(8):1315-1329.
[32] 吴元明,林佳怡,柳雨汐,李丹婷,张宗琼,郑晓明,逄洪波.基于BSA-seq 和RNA-seq 挖掘水稻株高相关QTL[J]. 生物技术通报,2023,39(8):173-184.WU Yuanming,LIN Jiayi,LIU Yuxi,LIDanting,ZHANG Zongqiong,ZHENG Xiaoming,PANG Hongbo. Identification of rice plant height-associated QTL using BSA-seq and RNAseq[J].Biotechnology Bulletin,2023,39(8):173-184.
[33] ZHANG J J,LIB,GAO X R,PAN X Q,WU Y R. Integrating transcriptomic and metabolomic analyses to explore the effect of color under fruit Calyx on that of fruit apex in eggplant(Solanum melongena L.)[J].Frontiers in Genetics,2022,13:889461.
[34] QIN G H,LIU C Y,LIJ Y,QIY J,GAO Z H,ZHANG X L,YIX K,PAN H F,МING R,XU Y L.Diversity of metabolite accumulation patterns in inner and outer seed coats of pomegranate:Exploring their relationship with genetic mechanisms of seed coat development[J].Horticulture Research,2020,7:10.
[35] UМER М J,BIN SAFDAR L,GEBREМESKEL H,ZHAO S J,YUAN P L,ZHU H J,KASEB М O,ANEES М,LU X Q,HE N,GONG C S,LIU W G. Identification of key gene networks controlling organic acid and sugar metabolism during watermelon fruit development by integrating metabolic phenotypes and gene expression profiles[J]. Horticulture Research,2020,710.1038:s41438-20-00416-8.
[36] ANEES М,ZHU H J,UМER М J,GONG C S,YUAN P L,LU X Q,HE N,KASEB М O,YANG D D,ZHAO Y,LIU W G.Identification of an Aux/IAA regulator for flesh firmness using combined GWAS and bulked segregant RNA-Seq analysis in watermelon[J]. Horticultural Plant Journal,2024,10(5):1198-1213.
[37] CHALМERS D J,ENDE B V D. Productivity of peach trees:Factors affecting dry-weight distribution during tree growth[J].Annals of Botany,1975,39(3):423-432.
[38] HORIGUCHIG,FERJANIA,FUJIKURA U,TSUKAYA H.Coordination of cell proliferation and cell expansion in the control of leaf size in Arabidopsis thaliana[J]. Journal of Plant Research,2006,119(1):37-42.
[39] SIL Z,CHEN J Y,HUANG X H,GONG H,LUO J H,HOU Q Q,ZHOU T Y,LU T T,ZHU J J,SHANGGUAN Y Y,CHEN E W,GONG C X,ZHAO Q,JING Y F,ZHAO Y,LIY,CUIL L,FAN D L,LU Y Q,WENG Q J,WANG Y C,ZHAN Q L,LIU K Y,WEIX H,AN K,AN G,HAN B. OsSPL13 controls grain size in cultivated rice[J].Nature Genetics,2016,48(4):447-456.
[40] BOHNER J,BANGERTH F. Cell number,cell size and hormone levels in semi-isogenic mutants of Lycopersicon pimpinellifolium differing in fruit size[J]. Physiologia Plantarum,1988,72(2):316-320.
[41] HIGASHIK,HOSOYA K,EZURA H. Histological analysis of fruit development between two melon (Cucumis melo L. reticulatus)genotypes setting a different size of fruit[J].Journal of Experimental Botany,1999,50(339):1593-1597.
[42] CHENG G W,BREEN P J. Cell count and size in relation to fruit size among strawberry cultivars[J].Journal of the American Society for Horticultural Science,1992,117(6):946-950.
[43] GOFFINET М C,ROBINSON T L,LAKSO A N.A comparison of‘Empire’apple fruit size and anatomy in unthinned and handthinned trees[J]. Journal of Horticultural Science,1995,70(3):375-387.
[44] SU H,JIAO Y T,WANG F F,LIU Y E,NIU W L,LIU G T,XU Y. Overexpression of VpPR10.1 by an efficient transformation method enhances downy mildew resistance in V.vinifera[J].Plant Cell Reports,2018,37(5):819-832.
[45] DYМEK E E,GODUTID,KRAМER T,SМITH E F.A kinesinlike calmodulin-binding protein in Chlamydomonas:Evidence for a role in cell division and flagellar functions[J]. Journal of Cell Science,2006,119(Pt 15):3107-3116.
[46] REDDY A S,NARASIМHULU S B,SAFADIF,GOLOVKIN М.A plant kinesin heavy chain-like protein is a calmodulin-binding protein[J].The Plant Journal,1996,10(1):9-21.
[47] SONG H,GOLOVKIN М,REDDY A S,ENDOW S A. In vitro motility of AtKCBP,a calmodulin-binding kinesin protein of Arabidopsis[J].Proceedings of the National Academy of Sciences of the United States of America,1997,94(1):322-327.
[48] VOS J W,SAFADIF,REDDY A S N,HEPLER P K.The kinesin-like calmodulin binding protein is differentially involved in cell division[J].The Plant Cell,2000,12(6):979-990.
[49] BOWSER J,REDDY A S N. Localization of a kinesin-like calmodulin-binding protein in dividing cells of Arabidopsis and tobacco[J].The Plant Journal,1997,12(6):1429-1437.