CRISPR/Cas 基因编辑技术作为一种革命性的基因组编辑工具,凭借其高效性、灵活性和操作简便性等特点,在农作物和果树的产量提升、品质改良及抗病性增强等方面取得显著成效,展现出广阔的应用前景[1-2]。针对苹果、葡萄、桃等果树漫长的育种周期和复杂的遗传特性,CRISPR 技术能够直接靶向目标基因实现精准编辑,加速性状改良进程[3]。然而,苹果再生周期长,基因组信息不够全面,以及遗传转化体系尚未完全成熟,苹果基因编辑研究的发展仍相对滞后。为突破这一技术瓶颈,亟须构建高效稳定的基因编辑体系,从而推动苹果遗传改良和基础研究的深度发展[4]。
目前大多数植物基因编辑载体仅支持gRNA靶标序列的替换,而无法对启动子、Cas蛋白等核心功能元件灵活替换[5]。Golden Gate、Gibson Assembly等新兴克隆技术推动了植物表达载体工具箱的开发,例如MoClo、GreenGate 和GoldenBraid 等系统[6-7]。这些工具箱依赖特定的载体与模块库,其固定化的拼接方法难以满足果树复杂基因组编辑的需求。传统工具箱的模块库构建费时耗力,限制其在多样化应用中的灵活性,且缺乏对复杂转录单元的高效组装能力[8]。常规载体缺乏针对这些元件的快速替换策略,导致编辑效率难以系统性优化,严重制约了基因编辑技术体系的适应性和扩展性。
在CRISPR/Cas9 基因编辑系统中,sgRNA 的选择是影响编辑效率的关键因素。因此,在开展稳定遗传转化之前,需要初步评估sgRNA 的编辑效率。目前编辑效率的检测方法包括T7 endonuclease 1(T7E1)酶切法、同源重组检测法(HR)、Sanger 测序和高通量测序等技术体系[9-10]。近年来,瞬时表达系统被引入苹果基因编辑研究中,用于快速评估sgRNA 基因编辑效率[11-12]。这种方法通过农杆菌介导的瞬时转化技术,将CRISPR/Cas9 系统直接转化到苹果叶片中,利用分子标记(如GFP荧光蛋白)进行目标位点的实时监测,显著缩短了检测周期。相比稳定转化体系,瞬时转化系统操作简单、周期短,可在几天内获得编辑效率数据;此外,瞬时转化的结果具有较高的可重复性,有助于快速筛选高效的基因编辑工具和优化编辑条件[13]。
开发模块化高效的载体组装方法,并结合灵活可靠的基因编辑效率评价体系,是优化果树基因编辑工具的重要步骤。为解决上述问题,笔者构建了基于Golden Gate克隆技术的模块化载体组装系统,同时建立了高效的编辑效率评价体系。首先通过对传统载体进行改造使其兼容Golden Gate,构建了分级组装策略平台,实现Cas 蛋白、核定位信号(NLS)、启动子及sgRNA 表达盒等核心功能模块的灵活替换,突破了传统载体元件替换的局限性。其次,引入RUBY报告元件,通过颜色实现质粒转染的可视化检测[14]。此外,结合苹果叶片瞬时转化体系,笔者系统评估不同sgRNA对苹果PDS(Phytoene desaturase)基因的编辑效率。该研究不仅为苹果基因编辑技术的高效化和可操作性提供了全新的解决方案,更为其他果树的基因编辑技术开发提供了通用的工具和方法框架,为果树遗传改良和精准育种奠定了坚实基础。
1 材料和方法
1.1 载体骨架改造
pUC19-Esp3 Ⅰ-Bb:使用pUC19-F-Bbs Ⅰ和pUC19-R-BbsⅠ引物(表1),对pUC19 质粒(北京庄盟国际生物基因科技有限公司,ZK102)进行PCR反向扩增线性化。使用lacZ-F-BbsⅠ-Esp3Ⅰ和lacZR-BbsⅠ-Esp3Ⅰ引物,扩增pUC19 质粒中LacZ 基因。PCR 扩增体系为:KOD One™PCR Master Mix 25 μL,上下游引物各2 μL,质粒2 μL,无菌水补至50 μL。反应程序为:94 ℃预变性2 min;98 ℃变性10 s,58~60 ℃退火5 s,72 ℃延伸20 s,35 个循环;72 ℃终延伸2 min;4 ℃保存。上述PCR 产物纯化后,两者等量混合,使用BbsⅠ限制性内切酶和T4 DNA连接酶进行Golden Gate组装,反应体系为:线性化pUC19 片段100 ng,lacZ 片段100 ng,BbsⅠ内切酶20 U,T4 DNA连接酶400 U,10×连接酶缓冲液2 μL,无菌水补至20 μL。37 ℃酶切5 min,16 ℃连接5 min,重复30个循环,确保高效组装。最终将反应产物转化到大肠杆菌DH5α菌株,并接种Luria-Broth(LB)培养基(100 mg·L-1氨苄青霉素、24 mg·L-1 IPTG及40 mg·L-1 X-gal)。37 ℃倒置过夜培养,筛选蓝色菌落,获得目标克隆,并测序验证其正确性。
表1 载体改造引物序列
Table 1 Vector modification primer sequences

注:GAGACG表示Esp3Ⅰ识别位点,GAAGAC表示BbsⅠ识别位点,GAGACC表示BsaⅠ识别位点,下划线表示切割位点。
Note:GAGACG indicates Esp3Ⅰidentification site, GAAGAC indicates BbsⅠidentification site, GAGACC indicates BsaⅠidentification site and the underline indicates the cut site.
引物名称Primer name lacZ-F-BbsⅠ-Esp3ⅠlacZ-R-BbsⅠ-Esp3ⅠpUC19-F-BbsⅠpUC19-R-BbsⅠlacZ-pHK2-F-BbsⅠ-BsaⅠlacZ-pHK2-R-BbsⅠ-BsaⅠ引物序列Primer sequence(5'-3')GCGAAGACTCCATCAGTGAGACGAGACCGTCACAGCTTGTCTG构建Construct pUC19-Esp3Ⅰ-Bb GCGAAGACTCTTTCGCTGAGACGGACCGCAGCTGGCACGACAG GCGAAGACTCGAAAAGGGCCTCGTGATACGCCTA GCGAAGACTCGATGCCCGCTTTCCAGTCGGGA GCGAAGACTCAGCTCGATTGAGACCAGACCGTCACAGCTTGTCTG pHK2-BsaⅠ-Bb GCGAAGACTCAATTACTCTGAGACCGACCGCAGCTGGCACGACAG
pHK2-BsaⅠ-Bb:使用lacZ-pHK2-F-BbsⅠ-BsaⅠ和lacZ-pHK2-R-BbsⅠ-BsaⅠ引物,扩增pUC19 质粒,获得lacZ基因片段,PCR扩增体系和反应程序同上。PCR产物纯化后用限制性内切酶BbsⅠ单酶切进一步纯化。随后,将植物双元表达质粒pHK2-Cas9-AtU6(武汉伯远,REC43-Ⅰ)用限制性内切酶Hind Ⅲ和EcoRⅠ双酶切,产物切胶并回收纯化。将纯化的lacZ 基因片段与pHK2 骨架等量混合,按照T4 DNA 连接酶反应流程(Takara Tomoy,2011A),16 ℃连接4 h。最终将反应产物转化到大肠杆菌DH5α 菌株,并接种LB 培养基(50 mg·L-1卡那霉素、24 mg·L-1 IPTG及40 mg·L-1 X-gal)。37 ℃倒置过夜培养,筛选蓝色菌落,获得目标克隆,并测序验证其正确性。
1.2 Cas9-AtU6-gRNA-RUBY质粒构建
使用Golden Gate 引物设计在线网站(https://goldengate.neb.com/)简化引物设计。 首先将pUC19-Esp3Ⅰ-Bb 载体序列输入网站,参数中选择Esp3Ⅰ作为酶切位点。将增强型35S 启动子、spy-Cas9以及NOS终止子等片段序列,按照顺序依次添加至软件,自动生成带有Esp3Ⅰ酶切位点的3 对引物,用以扩增启动子、Cas 蛋白以及终止子序列。3个扩增产物纯化后,等量混合,酶切连接体系为:pUC19-Esp3Ⅰ-Bb载体100 ng,PCR混合产物300 ng,Esp3Ⅰ内切酶20 U,T4 DNA连接酶400 U,10×连接酶缓冲液2 μL,无菌水补至20 μL。37 ℃酶切5 min,16 ℃连接5 min,重复30个循环,确保高效组装。最终将反应产物转化到大肠杆菌DH5α 菌株,并接种LB 培养基(100 mg·L-1氨苄青霉素、24 mg·L-1 IPTG及40 mg·L-1 X-gal)。37 ℃倒置过夜培养,筛选白色菌落,获得目标克隆,并测序验证其正确性,生成e35S-Cas9-NOSter表达元件(pUC19-Module A)。
参照前人报道[1],构建靶向苹果PDS(MD04G0021400)基因的AtU6-gRNA 表达元件,并设计附加BsaⅠ酶切位点引物(表2),将AtU6-gRNA从启动子到poly T区域扩增,产生AtU6-gRNA表达元件(Module B)。以质粒35S:RUBY(Addgene#160908)为模板,设计附加BsaⅠ酶切位点引物,扩增e35S-RUBY-HSPter 区域,产生RUBY 表达元件(Module C)。上述Module A/B/C,三者等量混合,使用BsaⅠ限制性内切酶和T4 DNA 连接酶进行Golden Gate组装,反应体系:ModuleA/B/C各100 ng,pHK2-BsaⅠ-Bb 质粒100 ng,BsaⅠ内切酶20 U,T4DNA 连接酶400 U,10×连接酶缓冲液2 μL,无菌水补至20 μL。37 ℃酶切5 min,16 ℃连接5 min,重复30个循环,确保高效组装。最终将反应产物转化到大肠杆菌DH5α菌株,并接种LB培养基(100 mg·L-1氨苄青霉素、24 mg·L-1 IPTG 及40 mg·L-1 X-gal)。37 ℃倒置过夜培养,筛选白色菌落,获得目标克隆,并测序验证其正确性。
表2 质粒构建引物序列
Table 2 Plasmid construction primer sequences

注:CGTCTC表示Esp3Ⅰ识别位点,GGTCTC表示BsaⅠ识别位点,下划线表示切割位点。
Note:CGTCTC indicates Esp3Ⅰidentification site,GGTCTC indicates BsaⅠidentification site and the underline indicates the cut site.
引物名称Primer name e35s-F引物序列Primer sequence(5'-3')CGTCTCTTCGCGGTCTCTACTCTGAGACTTTT模板Template pCAMBIA1303扩增产物Amplification e35S promoter模块Module A来源Source庄盟ZK860 Zoman ZK860 e35s-R CAACAAAGGGTAATATC CGTCTCATACTTCTTGTCCATTCAGCGTGTC SpyCas-F SpyCas-R Nos-F Nos-R CTCTCCAAATG CGTCTCAAGTACTCCATTGGGCTCG Addgene#78931 SpyCas9 CGTCTCACCTTCCTCTTCTTCTTGGG CGTCTCAAAGGTGTGAGATCGTTCAAACATTTGG pHDE-35SCas9-mCherry pCAMBIA1303 CGTCTCGTCAGGGTCTCCTTACGATCTAGTAACAT庄盟ZK860 Zoman ZK860 NOS terminator AtU6-F gRNA-R Ruby-F Ruby-R AGATGACAC GGTCTCAGTAAGGCGCGCCAAGCTTC pPTG [15]AtU6-gRNA GGTCTCCAAAGTCTCAGCATGCGGCCGCTAGCTC GGTCTCCCTTTTCAACAAAGGGTAATATC 35S:RUBY Addgene#160908 RUBY B C GGTCTCGCGATCTTATCTTTAATCATATTCCATAGTC
1.3 苹果叶片瞬时表达系统优化
为了构建苹果叶片的瞬时转化体系,选取25 d Gala苹果组培苗作为试验材料。首先将基因编辑质粒转化至GV3101(pSoup-p19)农杆菌,后续挑选阳性克隆接种于YEB液体培养基(50 mg·L-1卡那霉素,30 mg·L-1利福平),28 ℃过夜培养(200 r·min-1)。将培养液按10%比例转接至新鲜培养基中,继续扩大培养至OD600为0.8~1.0。离心收集菌体后,用等体积10 mmol·L-1 MES(pH=5.6)缓冲液重悬,同时可加入200 μmol·L-1 乙酰丁香酮和0.01% Silwet L-77。将整棵植株浸泡到侵染液中,采用真空渗透法,-0.086 MPa 真空处理5 min 并快速释放压力,3次重复[16]。经无菌滤纸吸取表面残留液体后,将植株转入组培瓶中,在26 ℃黑暗条件下静置24 h,继而转至弱光(16 h光/8 h暗)条件下培养72 h,随后提取叶片RNA和DNA用于后续试验。每次试验设置3个组间重复,每个组别包括5株生物学重复。
1.4 植物RNA提取以及检测分析
分别采集瞬时转化后的烟草和苹果叶片的新鲜组织样本,使用RNA Easy Fast 植物组织RNA 快速提取试剂盒(北京天根生化科技有限公司)提取总RNA,并使用PrimeScript™Ⅳ1st strand cDNA Synthesis Mix(TaKaRa)反转录为cDNA。利用qRTPCR 对烟草叶片中RUBY 报告元件进行表达量分析。qRT-PCR 反应体系为:cDNA 1 μL,2×ChamQ Universa SYBR qPCR Master mix 5 μL,上下游引物各1 μL,无菌水补至10 μL。RUBY报告元件主要由CYP76AD1 基因(CYP)、DODA 基因、Glucosyltransferase 基因(Glu)三部分组成。以烟草Actin 为内参基因,采用2-△△CT法进行RUBY 的定量分析。使用RT-PCR检测苹果叶片中Cas9蛋白、gRNA以及RUBY 的表达情况。设计Cas9 基因mRNA 检测引物Cas9-F、Cas9-R;gRNA 检测引物gRNA1-F、gRNA2-R;RUBY 报告元件mRNA 检测引物DODA-F、DODA-R(表3)。扩增产物通过1.5%琼脂糖凝胶电泳检测,采用GelRed 染料染色并在紫外光下观察条带,确定各个基因的表达情况。
表3 引物序列及片段长度
Table 3 Primer sequences and fragment length

引物名称Primer name Cas9-F Cas9-R gRNA1-F gRNA2-R DODA-F DODA-R CYP-F CYP-R Glu-F Glu-R引物序列Primer sequence(5'-3')ACCTGTCAGACGCCATTCTG TGGGGGATGCTTCCATTGTC GCAGAAGCGAGAGATGTTTTAGG AAAACTGACAGTACTGCTAATTTCT GACAGCCGAGACAGACCAAA GGGGCAACACCATCGAAGTA CTGACGATGGGCGAGATCAA GGCAGGTTGATGATGTCGGA CCGATAAGCTCACACTCCCG GTGAACACCACGCCTGTAGA片段长度Fragment length/bp 355 201 246 194 190
1.5 基因编辑效率检测
使用植物基因组DNA提取试剂盒(北京庄盟国际生物基因科技有限公司,ZP309),对叶片进行基因组DNA 提取。使用NanoDrop 分光光度计测定DNA 质量浓度,稀释至20 ng·μL-1。稀释后的DNA样品使用2×Rapid Taq Plus Master Mix 聚合酶(诺唯赞生物科技股份有限公司,P223)和PDS 特异检测引物进行PCR 反应(PDS-F:GTGTTTGCCCCTT-GAAGGTT;PDS- R:TGTCTCTGTTCCTTATGTTTCCTTT)。PCR 产物纯化后送往生工生物工程(上海)股份有限公司进行Sanger 测序,测序数据使用TIDE(https://tide.nki.nl/)软件进行分析。
1.6 数据分析
使用Excel 2019 对试验数据进行统计,利用IBM SPSS 23.0 软件对试验数据进行差异显著性分析,使用GraphPad Prism v 9.0绘图。
2 结果与分析
2.1 模块化载体实现多个表达单元无缝整合
笔者在本研究中的思路是通过改造常规质粒载体,在选定的位置插入一对Esp3Ⅰ或BsaⅠ等ⅡS型限制性内切酶位点,将传统质粒改造为兼容Golden Gate技术的升级版质粒。ⅡS型限制性内切酶可在远离其识别序列的位置裂解DNA,并产生黏性末端,从而有序并同时无缝组装多个片段。pUC19 是一种常见的DNA克隆质粒,含有LacZ基因,提供蓝白斑筛选功能,便于插入片段的鉴定。pHK2 是基于pCAMBIA1300改造的适用于双子叶植物基因编辑载体,虽然已携带Cas9 和gRNA 表达元件,但是无法对启动子、Cas 蛋白、核定位信号、终止子等元件灵活替换。因此,笔者对pUC19、pHK2 进行改造,使其与Golden Gate 兼容,构建中间载体和植物表达载体的基础骨架。
pUC19 质粒改造流程:首先,设计附加Esp3Ⅰ和BbsⅠ酶切位点引物,用以扩增pUC19 质粒的lacZ基因;其次,设计附加BbsⅠ酶切位点引物反向扩增pUC19的骨架;两者PCR产物经过限制性内切酶BbsⅠ酶切连接以后,成功将Esp3Ⅰ酶切位点插入lacZ 基因两侧;最终将中间载体骨架命名为pUC19-Esp3Ⅰ-Bb(图1-A),此载体原核抗性为氨苄青霉素,兼容Golden Gate 连接技术,同时可利用蓝白斑筛选提高阳性克隆成功率。
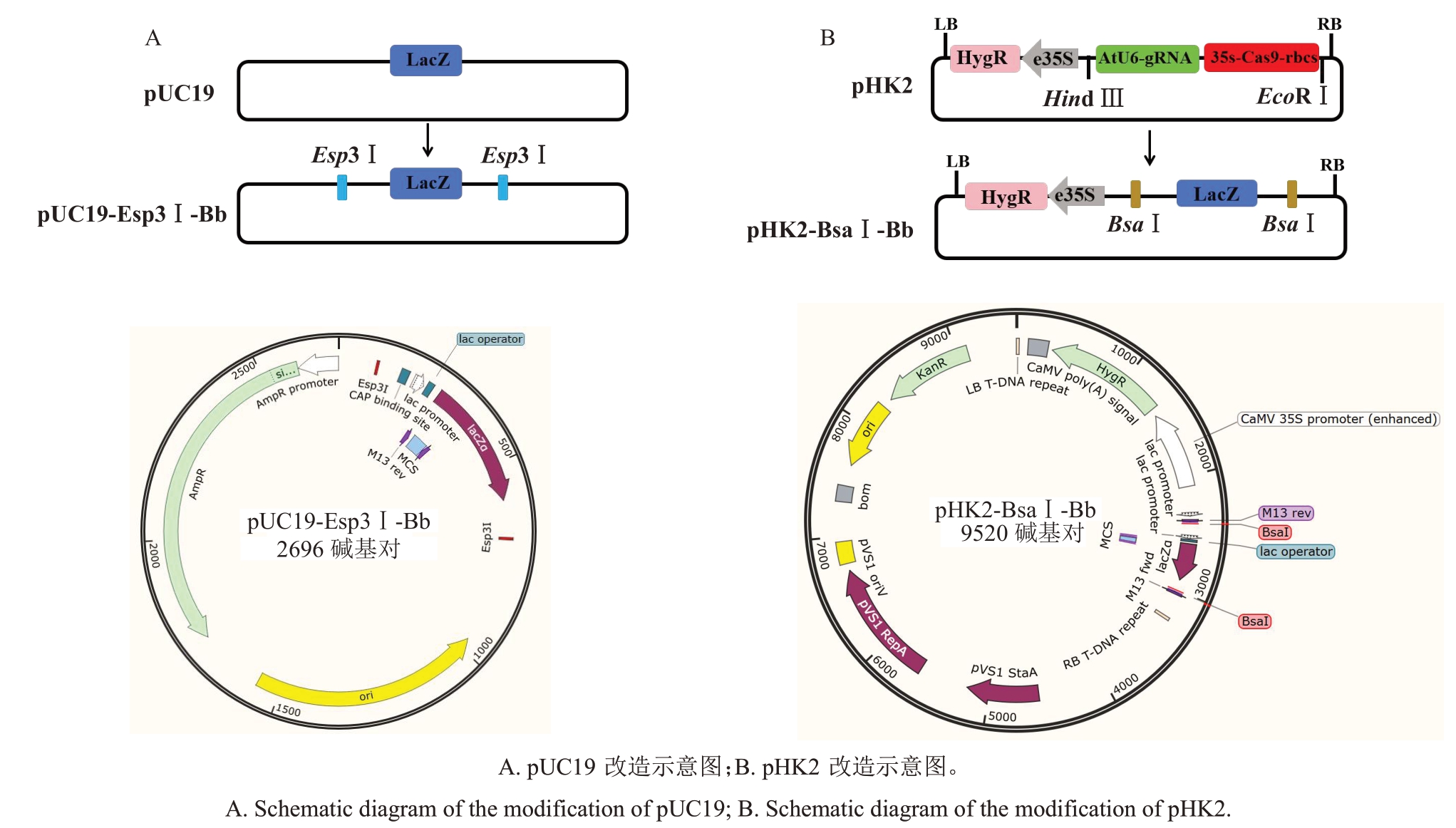
图1 载体改造示意图
Fig.1 Schematic diagram of the modification of vector
pHK2 质粒改造流程:设计附加BsaⅠ和BbsⅠ酶切位点引物,扩增pUC19 质粒的lacZ 基因,随后使用限制性内切酶BbsⅠ酶切扩增产物,在扩增子两端形成黏性末端;使用限制性内切酶Hind Ⅲ和EcoRⅠ酶切pHK2 质粒,释放出AtU6-gRNA 和35s-Cas9-rbcs表达元件,并产生与lacZ模块具有相同黏性末端的骨架分子;最终将lacZ与pHK2骨架连接,生成植物表达骨架载体pHK2-BsaⅠ-Bb(图1-B)。该质粒具备双元表达载体元件,以及卡那霉素(原核)和潮霉素(真核)抗性,插入BsaⅠ-lacZ-BsaⅠ模块,使之兼容Golden Gate 连接技术,同时可利用蓝白斑筛选提高阳性克隆成功率。
本系统开发的中间载体pUC19-Esp3Ⅰ-Bb,支持将多个片段整合进一个转录模块(Module),形成中间载体pUC19-Module A/B/C,并进一步将多个Module 无缝装载到植物表达载体pHK2-BsaⅠ-Bb,用于后续植物遗传转化(图2)。为此,用于扩增的引物被设计为含有Esp3Ⅰ酶切位点,以实现无缝连接,可以通过Golden Gate引物设计在线网站简化引物设计。此外,每个Module的第一个片段的正向引物和最后一个片段的反向引物,应在Esp3Ⅰ酶切位点和连接序列之间包含一个BsaⅠ酶切位点。在这些引物中,相邻片段之间的融合位点互补,组装体中第一个和最后一个融合位点与pHK2-BsaⅠ-Bb载体位点互补。通过在引物中附加BsaⅠ酶切位点,第二轮组装时只需一个pHK2-BsaⅠ-Bb就能装载多个pUC19-Module,并形成一个完整的表达质粒。
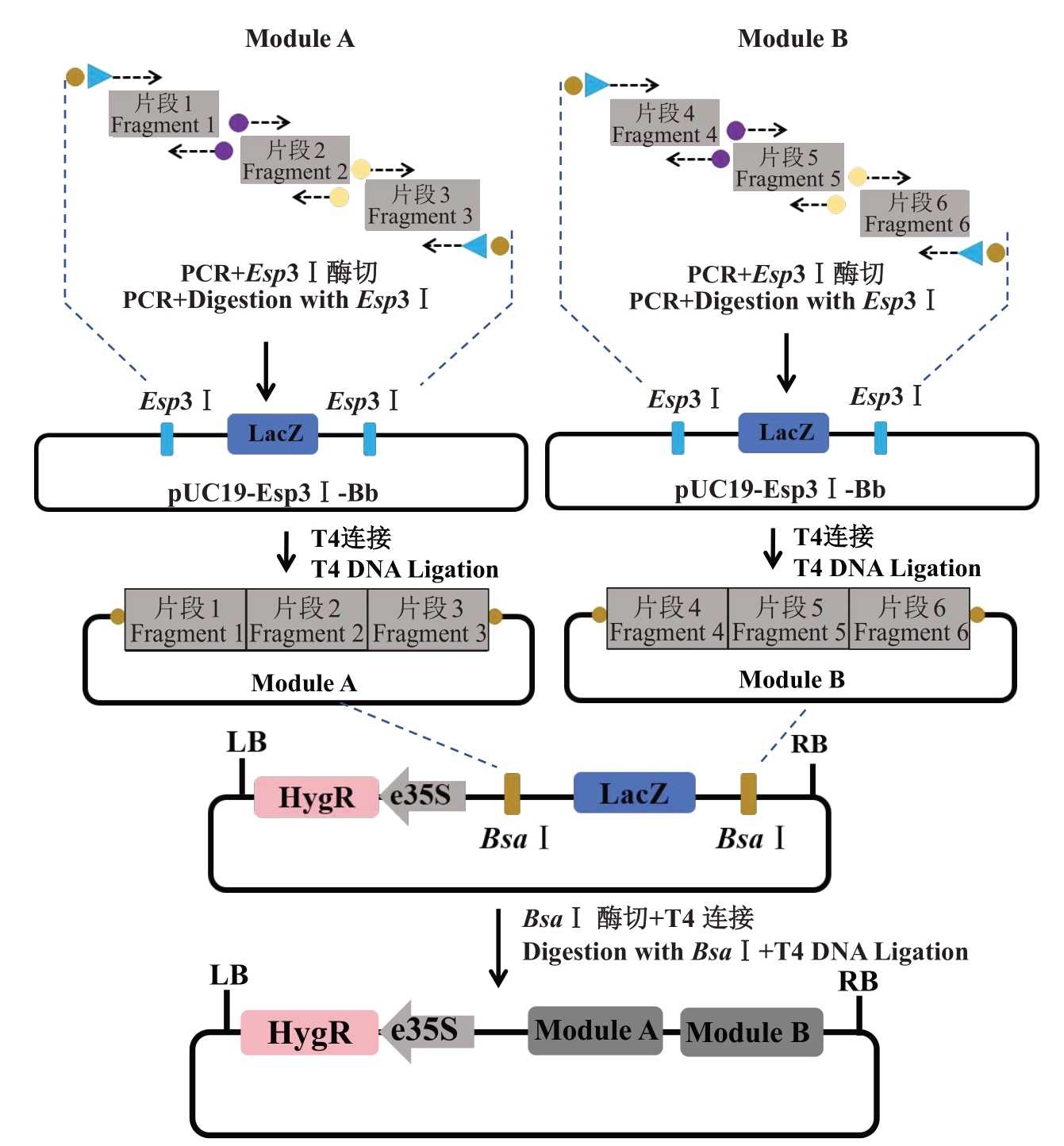
图2 pUC19-Module 构建示意图
Fig.2 Schematic diagram for producing the pUC19-Module construct
2.2 植物基因编辑载体e35S-Cas9-AtU6-gRNARUBY的构建
笔者在本研究中构建的载体骨架,能够将不同的功能元件灵活组合,可以作为不同植物基因编辑工具优化的基础载体。为了确认开发的方法有效性,笔者尝试利用pUC19-Esp3Ⅰ-Bb 和pHK2-BsaⅠ-Bb 构建全新基因编辑质粒。首先,将e35S 启动子、SpyCas9和NOS终止子分别从不同质粒中扩增,并无缝组装到pUC19-Esp3Ⅰ-Bb中,形成e35S-Cas9-NOS表达模块A。构建靶向苹果PDS基因的AtU6-gRNA表达元件,并设计附加BsaⅠ酶切位点引物,将AtU6-gRNA扩增,产生表达模块B。其次,设计附加BsaⅠ酶切位点引物,扩增RUBY报告系统的完整表达元件,产生表达模块C。最终,将模块A、B、C 与pHK2-BsaⅠ-Bb 混合,在BsaⅠ限制性内切酶和T4 DNA连接酶作用下进行一体化无缝连接,得到基因编辑载体e35S-Cas9-AtU6-gRNA-RUBY(图3-A)。此载体包含基因编辑元件和RUBY 可视化报告系统,可通过颜色初步识别遗传转化事件。瞬时转化烟草叶片显示,e35S-Cas9-AtU6-gRNA-RUBY 使得烟草叶片变红(图3-B),与原始质粒35S:RUBY的表现一致。进一步对两组叶片中RUBY报告元件的表达量进行分析,结果表明,在这两组叶片中均检测到RUBY的表达,且表达水平无显著差异(图3-C)。以上结果表明,此质粒在遗传转化系统中具备可视化指示功能。
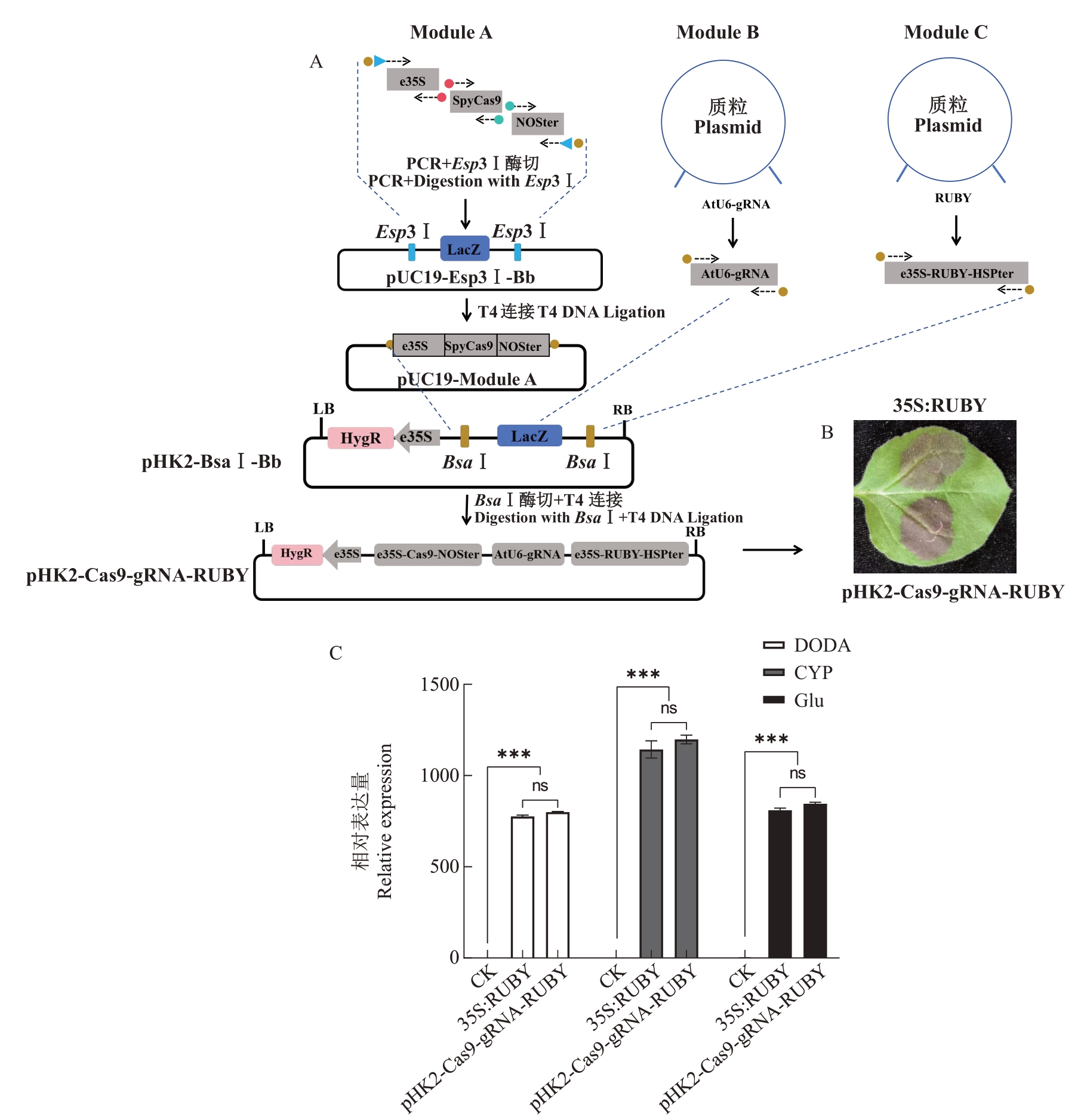
图3 e35S-Cas9-AtU6-gRNA-RUBY 载体在烟草中实现瞬时表达
Fig.3 The e35S-Cas9-AtU6-gRNA-RUBY vector enables transient expression in N.benthamiana
A.e35S-Cas9-AtU6-gRNA-RUBY 构建示意图;B.烟草叶片瞬时转化表型;C.RUBY 相对表达量分析。DODA.DODA,CYP.CYP76AD1,Glu.Glucosyltransferase。***表示在P<0.001 差异极显著。
A.Schematic diagram for producing the e35S-Cas9-AtU6-gRNA-RUBYconstruct;B.Transient transformation Phenotype of tobacco leaves;C.Analysis of relative expression of RUBY. DODA. DODA, CYP. CYP76AD1, Glu. Glucosyltransferase. ***indicate extremely significant difference at P<0.001.
2.3 苹果瞬时表达系统
植物稳定转化需要较长时间筛选以及再生,使基因编辑难度与时间成本大幅增加。对于植物而言,在成功构建好CRISPR 载体后,先进行瞬时转化,在较短时间内迅速检测CRISPR 载体系统和gRNA的有效性,随后开展稳定转化,大大降低试验难度与时间成本。
由于苹果叶片蜡质层较厚,无法采用针管注射法瞬时转化,笔者采用真空渗透法进行苹果瞬时表达。如图4-A 所示,苹果组培苗完全浸泡在农杆菌悬浮液中,-0.086 MPa 真空辅助侵染。暗培养1 d后,在弱光下继续培养。植株在侵染4 d 后,叶片出现明显的红色,表明质粒成功转染进植物细胞并表达RUBY 报告元件(图4-B)。为了检测质粒中Cas9、gRNA及RUBY元件是否成功表达,提取叶片RNA 并采用特异性引物对Cas9 蛋白、gRNA 以及RUBY的mRNA进行扩增检测。RT-PCR显示,侵染后的红色叶片具有明显的Cas9、gRNA 以及RUBY的特异性条带(图4-C),后续测序进一步确认序列正确。以上结果表明,该瞬时转化方法可以将质粒导入苹果叶肉细胞,并且质粒中基因编辑和RUBY功能元件均在叶片中成功表达。
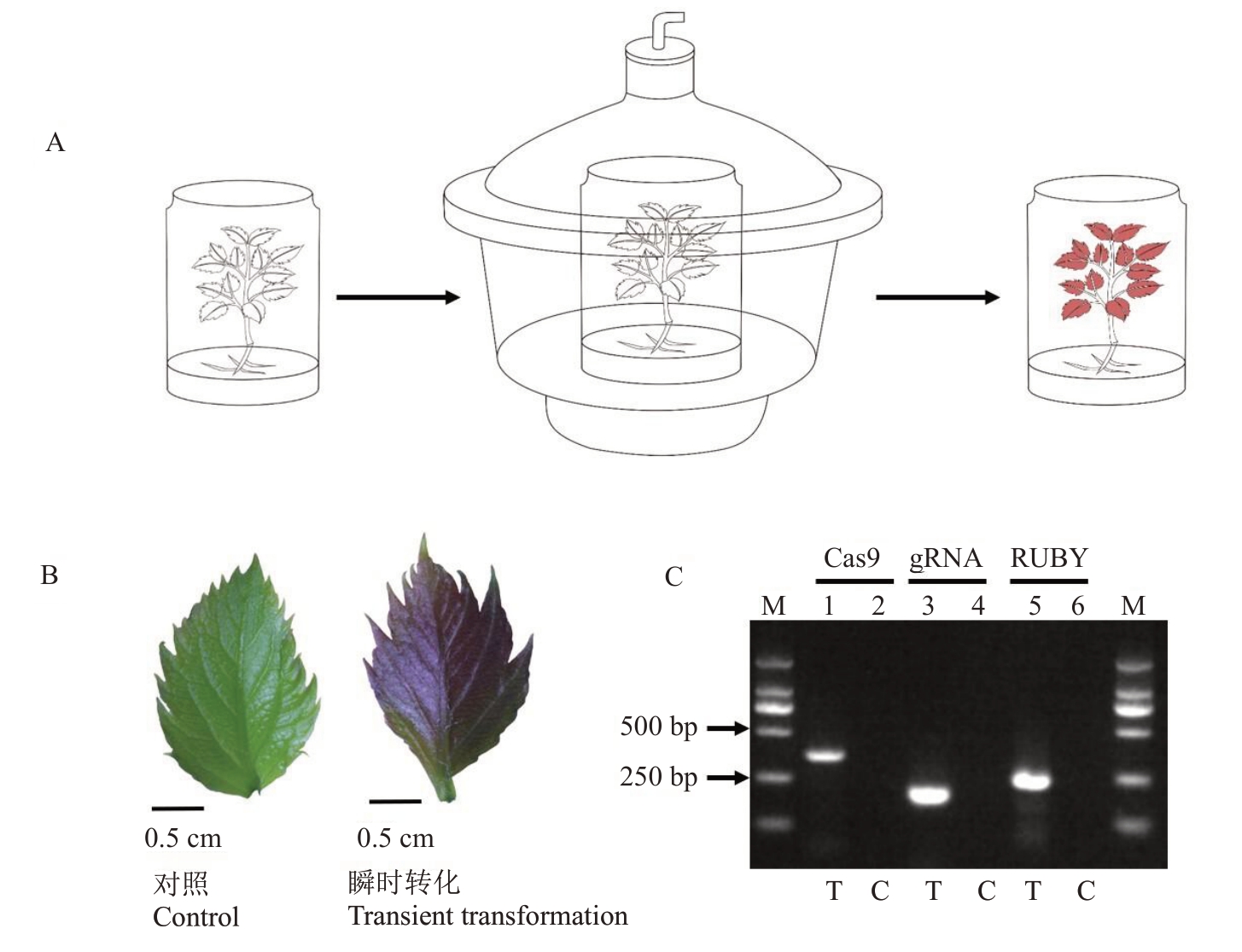
图4 农杆菌介导的苹果植株瞬时转化试验
Fig.4 Agrobacterium-mediated transient delivery assay in apple plantlets
A.苹果植株真空渗透瞬时转化流程;B.苹果植株瞬时转化表型;C.通过RT-PCR 检测苹果植株中T-DNA 插入。M.Marker;T.瞬时转化叶片;C.对照叶片。
A.Transient expression protocol with vacuum infiltration in apple plantlets;B.Transient transformation Phenotype of apple plantlets;C.Confirmation of T-DNA insertion in apple plantlets by RT-PCR analysis.M.Marker;T.The transiently transformed leaves;C.The control leaves.
2.4 苹果瞬时表达系统验证基因编辑质粒的功能
为了进一步验证构建的质粒具有基因编辑功能,笔者采用瞬时表达的方法,对叶肉细胞中PDS基因进行敲除试验。两个gRNA 靶标(gRNA-1/2)分别位于苹果PDS基因的第2个和第3个外显子区域,gRNA-1靶向负义链,gRNA-2靶向正义链(图5-A)。两个gRNA 由共同的AtU6 启动子驱动,中间tRNA-Gly 序列连接,转录后会被细胞内的tRNA 加工酶切割,将gRNA从同一转录本中分开,释放出功能性单个gRNA(图5-B)。
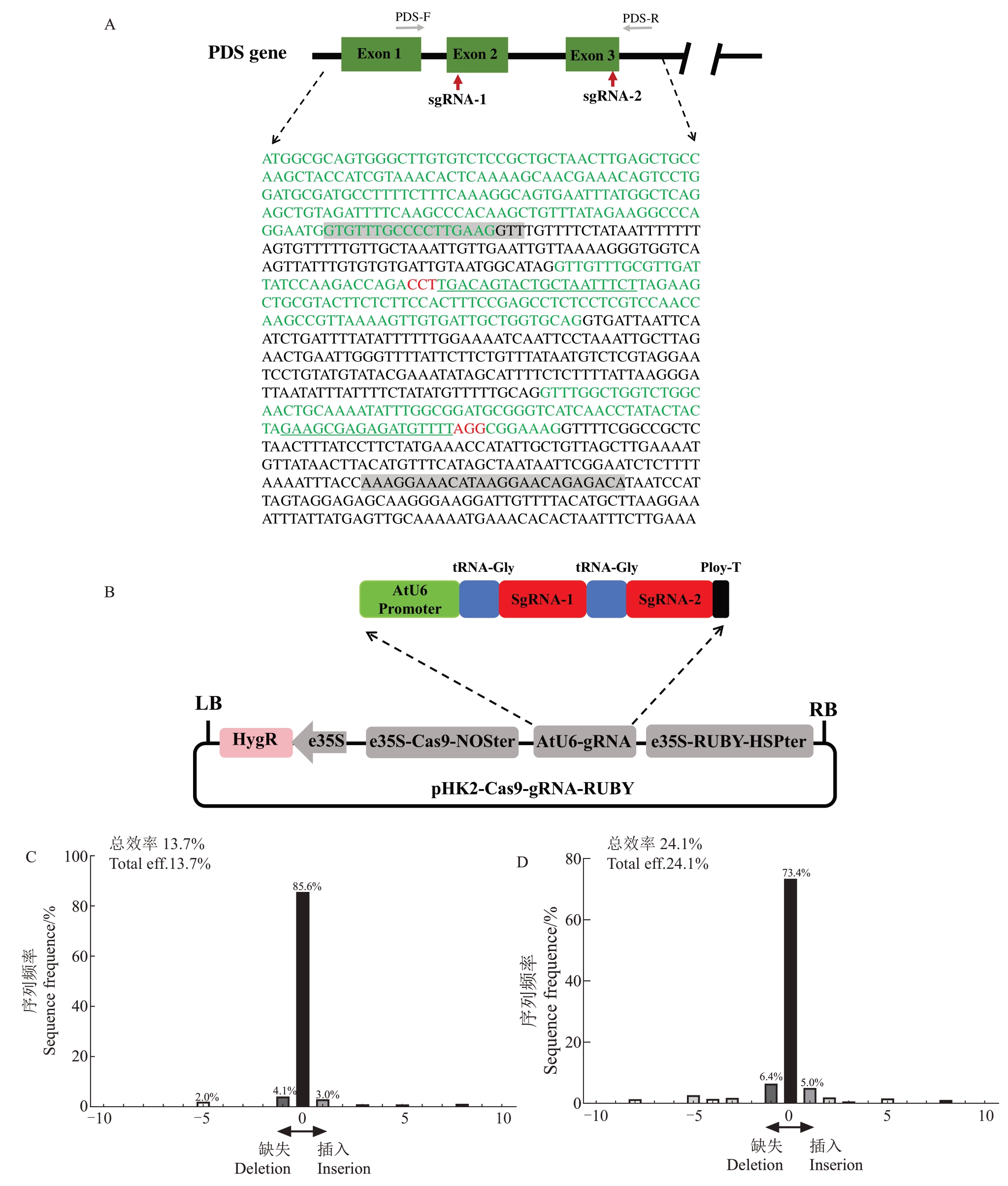
图5 苹果PDS 基因CRISPR/Cas9 载体构建及gRNA 基因编辑频率检测
Fig.5 Schematic diagram of CRISPR/Cas9 vector of apple PDS gene and gRNA gene editing frequency detection
A.靶向苹果PDS 基因组序列gRNA 的靶点示意图。绿色为外显子序列,下划线是gRNA 的序列(红色是PAM 序列);灰色为PDS 检测引物序列;黑色为内含子序列。B.苹果PDS 基因CRISPR/Cas9 载体示意图;C.gRNA-1 的基因编辑类型及频率;D.gRNA-2 的基因编辑类型及频率。
A. Schematic position of gRNA targeting the apple PDS genome sequence. The green is the exon sequence, the underline is the gRNA sequence(the red is the PAM sequence); the gray is PDS detection primer sequence; the black is the intron sequence. B. Schematic diagram of CRISPR/Cas9 vector of apple PDS gene;C.Types and frequency of gRNA-1 gene editing;D.Types and frequency of gRNA-2 gene editing.
使用TIDE在线工具可以分析目标区域PCR产物的Sanger测序数据,从而解析野生型/编辑细胞群体中插入/缺失突变(indel)的大小频率。通过比较gRNA-1 和gRNA-2 位点的Sanger 测序数据,TIDE可以识别不同indel 的种类和相对频率,gRNA-2 位点的基因效率高于gRNA-1(24.1% vs 13.7%),gRNA1 与gRNA2 之间存在显著差异(P<0.05),碱基类型多为1 bp的插入和缺失(图5-C~D)。
3 讨 论
笔者基于Golden Gate Assembly 技术开发的模块化载体构建系统,成功解决了植物基因编辑中常见的多元件整合的技术难题。通过对传统载体pUC19 和pHK2 进行改造,实现了多元件的精准整合,提供了一种高效且灵活的基因组整合方案。相较于传统克隆技术,本系统在构建效率和灵活性方面展现出明显优势。与Gibson Assembly相比,本系统无需依赖序列重叠区设计,减少了同源臂的设计及长片段PCR扩增需求,避免了序列依赖性组装偏差,通过Type ⅡS限制性内切酶(如BsaⅠ、Esp3Ⅰ)和T4 DNA连接酶的协同作用,可实现多片段DNA的一次性精准高效组装。与MoClo系统相比,本系统克服了其对单一限制性内切酶的过度依赖,采用更灵活的模块化设计。相比之下本系统“一锅”反应简化了操作流程,减少了步骤和时间[17],允许根据试验需求自由组合Cas蛋白、启动子及gRNA等元件,显著提升了基因编辑载体的构建效率与精准度。此外,相较于传统抗生素筛选标记,RUBY可视化报告元件的整合将表型确认时间从2~3周缩短至4~6 d,为基因编辑的实时监控提供了创新性解决方案。
相关研究表明,基于Golden Gate 和CRISPR/Cas9 的基因编辑平台提供了高效且可重复的模块化编辑工具,具备多基因同时编辑、无疤痕拼接和快速构建目标载体三大技术特征[18]。基于此,笔者在本研究中构建的pUC19-Esp3Ⅰ-Bb和pHK2-BsaⅠ-Bb载体展现出优异的模块化兼容性,能够使多个表达模块无缝整合,且具有较强的适应性,可以根据需求快速调整载体的功能元件。该平台不仅提供了植物基因编辑载体的优化方案,还可以在后续的基因编辑研究中灵活地调整Cas 蛋白、gRNA、启动子等关键元件,以满足不同植物的编辑需求。在功能验证方面,笔者成功地将e35S-Cas9、AtU6-gRNA 和RUBY 报告系统整合,构建了e35S-Cas9-AtU6-gRNA-RUBY基因编辑质粒。该质粒在烟草叶片中成功表达,并表现出良好的可视化指示功能,与以往的基因编辑体系相比[1],该体系为基因编辑效率的监控提供了便利。在苹果叶片的瞬时转化体系中,利用优化的真空渗透法应对了苹果叶片转化的挑战,成功实现了PDS 基因的靶向编辑和gRNA 编辑效率的初步评价,并通过RT-PCR 和测序确认了编辑效果。
然而本系统存在以下局限性:首先,仅验证了苹果瞬时转化体系的编辑效率,尚未通过稳定遗传转化评估基因编辑的能力,后续将通过苹果遗传转化体系进行验证;其次,当前试验样本量可能不足以全面评估编辑效率的稳定性,特别是在CRISPR/Cas9系统常见的嵌合体形成方面;最后,本系统对TypeⅡS 限制性内切酶的依赖性可能限制大片段(>15 kb)或高CG含量模块的组装效率,未来可结合Gibson Assembly 技术进行互补优化。此外,本系统尚未在单子叶植物中进行验证,其应用范围仍需进一步验证。
综上所述,笔者建立的模块化载体系统具有较高的转化效率和强大的编辑能力。通过适当设计引物和限制性内切酶位点,能够实现快速组装,减少了传统克隆过程中的时间和工作量,并有效提高了组装成功率。这为不同植物的基因编辑系统提供了一个标准化且易于调整的构建平台,极大地促进了植物功能基因组学研究的深入,为果树基因编辑研究提供了可靠的工具。
4 结 论
笔者基于Golden Gate 克隆技术成功构建模块化载体系统,实现了多个基因模块的高效精准组装,并通过苹果瞬时转化体系验证其编辑效率。针对果树转化难题,优化的真空渗透法解决了苹果遗传转化效率低的问题,成功实现了对PDS基因的靶向编辑。该系统在基因编辑工具多元化、载体的灵活性以及转化效率等方面提供了新的思路与技术支持,其模块化设计支持CRISPR元件、核定位信号、启动子及sgRNA表达盒的灵活组合,为果树等难转化植物的高效精准编辑及遗传改良提供了技术支撑。未来,基于此平台的进一步优化和应用有望推动更多植物品种的遗传改良,尤其是在经济作物和果树领域,带来更高效、精准的基因组编辑技术。
[1] ZHANG Y,ZHOU P,BOZOROV T A,ZHANG D Y.Application of CRISPR/Cas9 technology in wild apple (Malus sieverii)for paired sites gene editing[J].Plant Methods,2021,17(1):79.
[2] NISHITANI C,HIRAI N,KOMORI S,WADA M,OKADA K,OSAKABE K,YAMAMOTO T,OSAKABE Y. Efficient genome editing in apple using a CRISPR/Cas9 system[J].Scientific Reports,2016,6:31481.
[3] 何玉坤,欧阳嫣惟,张秀梅,林文秋,潘晓璐,张红娜.CRISPR/Cas9 基因编辑技术在果树作物中的应用研究进展[J].果树学报,2022,39(5):870-881.HE Yukun,OUYANG Yanwei,ZHANG Xiumei,LIN Wenqiu,PAN Xiaolu,ZHANG Hongna. Application of CRISPR/Cas9 gene editing technology in fruit trees[J]. Journal of Fruit Science,2022,39(5):870-881.
[4] XU X,YUAN Y J,FENG B H,DENG W. CRISPR/Cas9-mediated gene-editing technology in fruit quality improvement[J].Food Quality and Safety,2020,4(4):159-166.
[5] DEVELTERE W,DECAESTECKER W,ROMBAUT D,ANDERS C,CLICQUE E,VUYLSTEKE M,JACOBS T B. Continual improvement of CRISPR-induced multiplex mutagenesis in Arabidopsis[J].The Plant Journal,2024,119(2):1158-1172.
[6] RUKAVTSOVA E B,ZAKHARCHENKO N S,LEBEDEV V G,SHESTIBRATOV K A.CRISPR-Cas genome editing for horticultural crops improvement:Advantages and prospects[J].Horticulturae,2023,9(1):38.
[7] PAK S,LI C H. Progress and challenges in applying CRISPR/Cas techniques to the genome editing of trees[J]. Forestry Research,2022,2:6.
[8] HASSAN M M,ZHANG Y X,YUAN G L,DE K,CHEN J G,MUCHERO W,TUSKAN G A,QI Y P,YANG X H. Construct design for CRISPR/Cas- based genome editing in plants[J].Trends in Plant Science,2021,26(11):1133-1152.
[9] ZHAO Y M,WANG F,WANG C H,ZHANG X B,JIANG C Z,DING F,SHEN L,ZHANG Q F. Optimization of CRISPR/Cas system for improving genome editing efficiency in Plasmodium falciparum[J].Frontiers in Microbiology,2021,11:625862.
[10] JOHNSON R A,GUREVICH V,FILLER S,SAMACH A,LEVY A A. Comparative assessments of CRISPR-Cas nucleases’cleavage efficiency in planta[J]. Plant Molecular Biology,2015,87(1):143-156.
[11] ZHANG Y,LIANG Z,ZONG Y,WANG Y P,LIU J X,CHEN K L,QIU J L,GAO C X. Efficient and transgene-free genome editing in wheat through transient expression of CRISPR/Cas9 DNA or RNA[J].Nature Communications,2016,7:12617.
[12] CHEN L Z,LI W,KATIN-GRAZZINI L,DING J,GU X B,LI Y J,GU T T,WANG R,LIN X C,DENG Z N,MCAVOY R J,GMITTER F G,DENG Z N,ZHAO Y D,LI Y. A method for the production and expedient screening of CRISPR/Cas9-mediated non-transgenic mutant plants[J].Horticulture Research,2018,5:13.
[13] NISHIZAWA-YOKOI A,TOKI S. A piggyBac-mediated transgenesis system for the temporary expression of CRISPR/Cas9 in rice[J].Plant Biotechnology Journal,2021,19(7):1386-1395.
[14] HE Y B,ZHANG T,SUN H,ZHAN H D,ZHAO Y D.A reporter for noninvasively monitoring gene expression and plant transformation[J].Horticulture Research,2020,7(1):152.
[15] WANG Z P,WANG S B,LI D W,ZHANG Q,LI L,ZHONG C H,LIU Y F,HUANG H W.Optimized paired-sgRNA/Cas9 cloning and expression cassette triggers high-efficiency multiplex genome editing in kiwifruit[J].Plant Biotechnology Journal,2018,16(8):1424-1433.
[16] HAN P L,LI R,YUE Q Y,LI F D,NIE J J,YIN Z Y,XU M,GUAN Q M,HUANG L L. The apple receptor-like kinase Md-SRLK3 positively regulates resistance against pathogenic fungus Valsa mali by affecting the Ca2+signaling pathway[J].Phytopathology,2022,112(10):2187-2197.
[17] ANDREOU A I,NAKAYAMA N.Mobius Assembly:A versatile Golden-Gate framework towards universal DNA assembly[J].PLoS One,2018,13(1):e0189892.
[18] MA X L,ZHANG Q Y,ZHU Q L,LIU W,CHEN Y,QIU R,WANG B,YANG Z F,LI H Y,LIN Y R,XIE Y Y,SHEN R X,CHEN S F,WANG Z,CHEN Y L,GUO J X,CHEN L T,ZHAO X C,DONG Z C,LIU Y G.A robust CRISPR/Cas9 system for convenient,high-efficiency multiplex genome editing in monocot and dicot plants[J].Molecular Plant,2015,8(8):1274-1284.