余甘子(Phyllanthus emblica L.)属大戟科(Euphorbiaceae)叶下珠属(Phyllanthus)多年生灌木或乔木[1],是中国卫生部颁布的药食同源植物,也是世界卫生组织在全世界推广种植的3 种保健植物之一[2],并载入《中国药典》。现代药理学研究表明余甘子具有抗氧化、抗血脂异常、抗胆固醇、抗糖尿病、抗病毒、肝保护、心脏保护和抗腹泻等作用[3-5]。中国余甘子分布于东经98.5~122.0°,北纬18.0~29.0°,包括福建、广东、云南、广西、四川、贵州、海南和台湾等省份的热带或亚热带区域[1,6],种质资源储量十分丰富。遗传多样性研究是生物保护学的核心,也是植物遗传变异、品种选育和性状改良研究的基础[7],中国余甘子分布区域广阔,野生资源丰富,表型性状复杂,遗传变异丰富,开展遗传多样性和核心种质研究对其资源保护、新品种创制和遗传改良具有重要意义。随着现代分子生物技术的发展,分子标记技术已广泛应用于植物遗传多样性的鉴定[8]。目前,国内外学者先后利用少量余甘子种质资源开展了RAPD[9-13]、SRAP[14]和ISSR[15-16]等DNA 分子标记,但简单重复序列(SSR,simple sequence repeat)在余甘子种质资源的遗传多样性鉴定中鲜有报道,有较广阔的研究空间。SSR 广泛存在于基因组序列中,具有重复性好、数量丰富、共显性和操作简单等特点,是开展遗传多样性研究的可靠工具。此外,利用分子标记数据开展余甘子核心种质构建研究亦未见报道。福建省农业科学院果树研究所建立了中国第一个国家级余甘子种质资源保护平台国家热带植物种质资源库-余甘子种质资源分库,收集保存了中国大部分地方品种和国内外重要的遗传材料,可为余甘子的遗传多样性和核心种质研究提供丰富研究材料,为余甘子种质资源保护和优良种质筛选利用提供保障;前期项目组已开发余甘子SSR 分子标记技术,筛选高多态性SSR 引物10 对,为余甘子遗传多样性研究奠定了基础[17]。笔者在本研究中利用10对高多态性SSR 引物,对193 份国家热带植物种质资源库-余甘子种质资源分库保存的资源材料开展遗传多样性分析,明确余甘子的遗传差异程度和亲缘关系远近,并进一步利用SSR 分子标记数据探讨余甘子核心种质的构建方法,为余甘子种质资源的保存、遗传育种和高效利用提供科学依据。
1 材料和方法
1.1 植物材料
试验材料来源于国家热带植物种质资源库-余甘子种质资源分库(并被选认为福建省特色果树种质资源圃-余甘子圃)(图1)的192 份余甘子和1 份余甘子的近缘种材料小果叶下珠(P. reticulatus Poir.)(样品信息见表1,样品编号No. 1~No. 193)。国家热带植物种质资源库-余甘子种质资源分库地址位于福建省漳州市龙文区朝阳镇福建省农科院闽南分院(E:119°19′59ʺ,N:26°07′48ʺ,海拔20 m),该地区属南亚热带海洋性季风气候,平均气温21.4 ℃,平均降水量1453 mm,平均相对湿度82%,年平均日照2 185.2 h。于2022 年4 月采集193 份种质资源的新鲜幼嫩叶片冷冻保存备用。
表1 样品信息
Table 1 The sample information

序号No.1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 33 34 35 36 37 38 39 40 41 42 43 44 45 46 47 48 49种质名称Germplasm name青皮实生01 Qingpi seeding 01青皮01 Qingpi 01广东野生01 Guangdong wild 01青皮实生02 Qingpi seeding 02揭西种01 Jiexizhong 01广东野生02 Guangdong wild 02广东野生03 Guangdong wild 03广东野生04 Guangdong wild 04广东野生05 Guangdong wild 05广东野生06 Guangdong wild 06广玻甘01 Guangbogan 01广东野生07 Guangdong wild 07广东野生08 Guangdong wild 08福建野生01 Fujian wild 01甜甘Tiangan甜种01 Tianzhong 01饼甘05 Binggan 05广东野生09 Guangdong wild 09福建野生02 Fujian wild 02福建野生03 Fujian wild 03广东野生10 Guangdong wild 10甜甘实生01 Tiangan seeding 01广东野生11 Guangdong wild 11广东野生12 Guangdong wild 12饼甘实生01 Binggan seeding 01广玻甘02 Guangbogan 02广玻甘03 Guangbogan 03福建野生04 Fujian wild 04饼甘01 Binggan 01赤皮Chipi青皮02 Qingpi 02特甜Tetian福建野生05 Fujian wild 05福建野生06 Fujian wild 06福建野生07 Fujian wild 07青皮03 Qingpi 03广东野生13 Guangdong wild 13福建野生08 Fujian wild 08印引01 India introduce 01青皮实生03 Qingpi seeding 03广东野生14 Guangdong wild 14甜种02 Tianzhong 02福建野生09 Fujian wild 09广东野生15 Guangdong wild 15福建野生10 Fujian wild 10广玻甘04 Guangbogan 04福建野生11 Fujian wild 11福建野生12 Fujian wild 12青皮04 Qingpi 04来源Origin广东Guangdong广东Guangdong广东Guangdong福建Fujian广东Guangdong广东Guangdong广东Guangdong广东Guangdong广东Guangdong广东Guangdong广东Guangdong广东Guangdong广东Guangdong福建Fujian广东Guangdong广东Guangdong广东Guangdong广东Guangdong福建Fujian福建Fujian广东Guangdong福建Fujian广东Guangdong广东Guangdong福建Fujian广东Guangdong广东Guangdong福建Fujian广东Guangdong广东Guangdong广东Guangdong广东Guangdong福建Fujian福建Fujian福建Fujian广东Guangdong广东Guangdong福建Fujian印度India福建Fujian广东Guangdong广东Guangdong福建Fujian广东Guangdong福建Fujian广东Guangdong福建Fujian福建Fujian广东Guangdong序号No.50 51 52 53 54 55 56 57 58 59 60 61 62 63 64 65 66 67 68 69 70 71 72 73 74 75 76 77 78 79 80 81 82 83 84 85 86 87 88 89 90 91 92 93 94 95 96 97 98种质名称Germplasm name广东野生16 Guangdong wild 16广东野生17 Guangdong wild 17福建野生13 Fujian wild 13福建野生14 Fujian wild 14福建野生15 Fujian wild 15福建野生16 Fujian wild 16广东野生18 Guangdong wild 18广玻甘05 Guangbogan 05福诏野生Fuzhao wild福建野生17 Fujian wild 17福建野生18 Fujian wild 18甜甘实生02 Tiangan seeding 02甜甘实生03 Tiangan seeding 03福建野生19 Fujian wild 19广玻甘实生01 Guangbogan seeding 01甜种03 Tianzhong 03福建野生20 Fujian wild 20福建野生21 Fujian wild 21福建野生22 Fujian wild 22广玻甘06 Guangbogan 06广玻甘实生02 Guangbogan seeding 02福建野生23 Fujian wild 23福建野生24 Fujian wild 24广东野生19 Guangdong wild 19福饼5号Fubing 5 hao广玻甘07 Guangbogan 07广玻甘实生03 Guangbogan seeding 03饼甘02 Binggan 02福建野生25 Fujian wild 25福建野生26 Fujian wild 26福建野生27 Fujian wild 27狮头01 Shitou 01福建野生28 Fujian wild 28福建野生29 Fujian wild 29福建野生30 Fujian wild 30福建野生31 Fujian wild 31福建野生32 Fujian wild 32福建野生33 Fujian wild 33泰引01 Thailand introduce 01福建野生34 Fujian wild 34甜种04 Tianzhong 04缅引01 Myanmar introduce 01福建野生35 Fujian wild 35大玉子Dayuzi福莆1号Fupu 1 hao福建野生36 Fujian wild 36福建野生37 Fujian wild 37广玻甘08 Guangbogan 08福建野生38 Fujian wild 38来源Origin广东Guangdong广东Guangdong福建Fujian福建Fujian福建Fujian福建Fujian广东Guangdong广东Guangdong福建Fujian福建Fujian福建Fujian福建Fujian福建Fujian福建Fujian福建Fujian广东Guangdong福建Fujian福建Fujian福建Fujian广东Guangdong福建Fujian福建Fujian福建Fujian广东Guangdong福建Fujian广东Guangdong福建Fujian广东Guangdong福建Fujian福建Fujian福建Fujian广东Guangdong福建Fujian福建Fujian福建Fujian福建Fujian福建Fujian福建Fujian泰国Thailand福建Fujian广东Guangdong缅甸Myanmar福建Fujian广西Guangxi福建Fujian福建Fujian福建Fujian广东Guangdong福建Fujian
表1 (续) Table 1 (Continued)

序号No.99 100 101 102 103 104 105 106 107 108 109 110 111 112 113 114 115 116 117 118 119 120 121 122 123 124 125 126 127 128 129 130 131 132 133 134 135 136 137 138 139 140 141 142 143 144 145 146种质名称Germplasm name福建野生39 Fujian wild 39福建野生40 Fujian wild 40福建野生41 Fujian wild 41缅引02 Myanmar introduce 02福建野生42 Fujian wild 42缅引03 Myanmar introduce 03广东野生20 Guangdong wild 20福建野生43 Fujian wild 43甜种05 Tianzhong 05甜种06 Tianzhong 06福建野生44 Fujian wild 44福建野生45 Fujian wild 45缅引04 Myanmar introduce 04缅引05 Myanmar introduce 05福建野生46 Fujian wild 46福建野生47 Fujian wild 47缅引06 Myanmar introduce 06缅引07 Myanmar introduce 07广东野生21 Guangdong wild 21甜种07 Tianzhong 07福建野生48 Fujian wild 48福建野生49 Fujian wild 49甜种08 Tianzhong 08福建野生50 Fujian wild 50福建野生51 Fujian wild 51福建野生52 Fujian wild 52福建野生53 Fujian wild 53青皮05 Qingpi 05福建野生54 Fujian wild 54福建野生55 Fujian wild 55福建野生56 Fujian wild 56甜种09 Tianzhong 09福建野生57 Fujian wild 57福建野生58 Fujian wild 58青皮06 Qingpi 06福仙野生Fuxian wild秋白01 Qiubai 01扁甘01 Biangan 01福建野生59 Fujian wild 59饼甘03 Binggan 03饼甘实生02 Binggan seeding 02福建野生60 Fujian wild 60福建野生61 Fujian wild 61福建野生62 Fujian wild 62粉甘Fengan蓝丰Lanfeng枣甘Zaogan福建野生63 Fujian wild 63来源Origin福建Fujian福建Fujian福建Fujian缅甸Myanmar福建Fujian缅甸Myanmar广东Guangdong福建Fujian广东Guangdong广东Guangdong福建Fujian福建Fujian缅甸Myanmar缅甸Myanmar福建Fujian福建Fujian缅甸Myanmar缅甸Myanmar广东Guangdong广东Guangdong福建Fujian福建Fujian广东Guangdong福建Fujian福建Fujian福建Fujian福建Fujian广东Guangdong福建Fujian福建Fujian福建Fujian广东Guangdong福建Fujian福建Fujian广东Guangdong福建Fujian福建Fujian福建Fujian福建Fujian广东Guangdong福建Fujian福建Fujian福建Fujian福建Fujian福建Fujian福建Fujian福建Fujian福建Fujian序号No.147 148 149 150 151 152 153 154 155 156 157 158 159 160 161 162 163 164 165 166 167 168 169 170 171 172 173 174 175 176 177 178 179 180 181 182 183 184 185 186 187 188 189 190 191 192 193种质名称Germplasm name福建野生64 Fujian wild 64青皮07 Qingpi 07福建野生65 Fujian wild 65福建野生66 Fujian wild 66红果实生01 Red fruit seeding 01秋白02 Qiubai 02六月白Liuyuebai福建野生67 Fujian wild 67饼甘04 Binggan 04福建野生68 Fujian wild 68福建野生69 Fujian wild 69福建野生70 Fujian wild 70缅引08 Myanmar introduce 08扁甘02 Biangan 02福建野生71 Fujian wild 71福建野生72 Fujian wild 72甜种10 Tianzhong 10福建野生73 Fujian wild 73福建野生74 Fujian wild 74云南野生01 Yunnan wild 01云南野生02 Yunnan wild 02盈玉Yingyu福建野生75 Fujian wild 75福建野生76 Fujian wild 76福建野生77 Fujian wild 77小果叶下珠P.reticulatus印引02 India introduce 02泰引02 Thailand introduce 02福建野生78 Fujian wild 78饼甘实生03 Binggan seeding 03红河野生02 Honghe wild 02红果实生02 Red fruit seeding 02云南野生03 Yunnan wild 03云南野生04 Yunnan wild 04云南大果Yunnan Daguo福建野生79 Fujian wild 79狮头02 Shitou 02玻璃种Bolizhong土尖Tujian土饼Tubing尖屁股Jianpigu缅引09 Myanmar introduce 09广大甘Guangdagan糯橄榄Nuoganlan福建野生80 Fujian wild 80广玻甘实生04 Guangbogan seeding 04广西野生Guangxi wild来源Origin福建Fujian广东Guangdong福建Fujian福建Fujian云南Yunnan福建Fujian福建Fujian福建Fujian广东Guangdong福建Fujian福建Fujian福建Fujian缅甸Myanmar福建Fujian福建Fujian福建Fujian广东Guangdong福建Fujian福建Fujian云南Yunnan云南Yunnan云南Yunnan福建Fujian福建Fujian福建Fujian泰国Thailand印度India泰国Thailand福建Fujian福建Fujian云南Yunnan云南Yunnan云南Yunnan云南Yunnan云南Yunnan福建Fujian广东Guangdong广东Guangdong广东Guangdong广东Guangdong广东Guangdong缅甸Myanmar广东Guangdong云南Yunnan福建Fujian福建Fujian广西Guangxi

图1 国家热带植物种质资源库-余甘子种质资源分库俯视图与植株生长情况
Fig.1 Phyllanthus emblica Germplasm Resource Sub-Center,National Tropical Plants Germplasm Resource Center top view and plant growth situation
1.2 试验方法
1.2.1 DNA 的提取与检测 提取余甘子基因组DNA 参考CTAB 法[18]。选择DNA 质量浓度为100~200 ng·μL-1、A260/A280比值为1.8~2.2、1%琼脂糖凝胶电泳条带完整性较好的DNA 样品,将DNA 浓度稀释为20 ng·μL-1,-20 ℃保存。
1.2.2 PCR 扩增体系及程序 PCR 反应体系为20 μL:上、下游引物各0.4 μL,DNA模板(20 ng·μL-1)1 μL,10×Buffer 2 μL,dNTPs 1.6 μL,Easy Taq DNA Polymerase 0.2 μL,用ddH2O 补足至20 μL。PCR 扩增程序:95 ℃预变性5 min;94 ℃变性45 s,55 ℃退火1 min,72 ℃延伸1 min,30 个循环;最后72 ℃延伸1 min。
1.2.3 8%非变性聚丙烯酰胺凝胶电泳与银染 8%非变性聚丙烯酰胺凝胶电泳参考周雨晴等[19]的方法。电泳结束后,将做好标记的凝胶放入装有超纯水的保鲜盒中清洗,微沥干水分;加入500 mL 银染液,置于脱色摇床摇动15 min后,超纯水漂洗并微沥干水分;加入500 mL显色液置于脱色摇床直至显示出清晰的条带,用超纯水漂洗后,凝胶平铺于灯箱观测拍照。
1.2.4 余甘子遗传多样性分析 使用项目组前期筛选出的10对高多态性SSR引物(表2)分别对193份样品进行PCR 扩增,扩增产物于8%非变性聚丙烯酰胺凝胶电泳,银染显色后拍照,对不同引物电泳条带赋值后进行遗传多样性分析。
表2 10 对多态性SSR 引物信息
Table 2 10 Pairs of polymorphic SSR primer information

引物编号Primer number PE1 PE2 PE3 PE4 PE5 PE6 PE7 PE8 PE9 PE10重复基元Repeat motif(TCCCT)5(GAAAA)6(TTC)11(CT)21(AG)25(AG)17(AG)22(TC)20(CT)21(CT)20正向引物序列Positive primer sequence(5'-3')AGAAGAAGACAGCGACCAAA TTGATTGTTCGTTGGTCGGC AGTCTGCCATCCGATGAAGC CGAAGCAATTGCCCAAACCT ACGGAGAGGGAACACAAACC ATATGATGGCCACCGTCACC GCAGCAAAGAGAGCTTCTTCA AGCACTACACACCAACCTTCT TGGTTTGTAGCGAACATGCTG GCCACTTGAAAAGGTGAAGCA反向引物序列Reverse primer sequence(5'-3')GGAGATTAGGTGGCGGGATG AGAAGGGAAAAGTTGGGACGA GCCTCTGGCCTTAAACCTGA GGGGCAAAGAAGTTCCCTGA TGAGGGTCAAAAAGGGTTCTCA AGGTGTCTCTCTCTTGGACTCA GAGGTTGTAAGAGGGGCTTGT GTGGCGGTCTATGGTTTTGC CAGCACAACAAGAGGCCAAC GAGGACGATGTGGAGTTCCC产物长度Product length/bp 236 238 127 254 255 237 270 265 137 186
1.2.5 核心种质构建 根据SSR 分子标记数据,在25%的取样比例下构建初级核心种质,对聚类方法、取样策略和取样比例方法进行筛选。
(1)合理聚类方法的选择。分别采用SM 遗传距离、Jaccard遗传距离和Nei&Li遗传距离进行类平均法(UPGMA)聚类。
(2)合理取样策略的选择。优先取样策略:在不同构建方法的逐步聚类图中,最先聚在一起的各组株系,优先选择具有多态位点数较多的株系进入下一轮聚类,若各组的2个株系多态位点数目相等,则随机选择株系进入下一轮聚类,若某个组内只有一个株系,则直接抽取,直至最终抽取的材料数量达到核心种质取样比例标准。
随机取样策略:在不同构建方法的逐步聚类图中,最先聚在一起的各组株系,随机选择某个株系进入下一轮聚类,若某个组内只有一个株系,则直接被选择进入下一轮聚类,直至最终抽取的材料数量达到核心种质取样比例标准。
(3)合理取样比例的选择。应用筛选的最佳构建方案,分别按照10%、15%、20%、25%、30%、35%和40%的取样比例构建出7个核心种质组合。
(4)评价。筛选出核心种质后,分析核心种质与原始种质遗传多样性各参数的差异[20],并进行主成分分析,评价核心种质的代表性和异质性。
1.3 数据统计
根据PCR扩增结果,记录目标范围内扩增的条带,同一水平上可清晰分辨条带记“1”,无条带或不易分辨记“0”,Microsoft Excel 2016软件记录。利用POPGENE 32 计算等位基因数(Na)、有效等位基因数(Ne)、杂合度(h)和香农信息指数(I)等遗传多样性参数;利用PIC-CAL软件计算每对引物的多态性信息量(PIC,polymorphism information content)。利用DPS 7.05多元分析系统的“0~1系统聚类”模块计算获得各样品之间的遗传距离;利用MEGA 6.4 采用UPGMA法进行聚类分析并绘制聚类环形图。核心种质构建中t检验、数据标准化和主成分分析利用SPSS 27.0,主成分得分图根据主成分分析结果应用OriginPro 2022绘制。
2 结果与分析
2.1 余甘子种质资源遗传多样性分析
10 对引物在193 个样本中共检测到85 条多态性条带(目标区域内),平均每条引物扩增8.5条,多态性为100%,部分引物(PE8)对193 份材料的扩增结果见图2。遗传多样性相关参数可知(表3),有效等位基因数(Ne)变化范围为1.091 0(PE4)~1.696 5(PE1),平均值为1.321 6,有效率为65.63%。杂合度(h)变化范围为0.083 0(PE4)~0.373 8(PE1),平均值为0.194 6。香农信息指数(I)变化范围为0.177 5(PE4)~0.535 1(PE1),平均值为0.325 7。多态信息含量(PIC)变化范围为0.69(PE2 和PE3)~0.87(PE7),平均值为0.77;10 对引物的PIC 均大于0.5,表明10条引物的多态位点均为高度多态性,标记提供的信息合理,表现丰富的遗传多样性。
表3 10 对SSR 引物在193 个样本中的多样性参数
Table 3 Genetic diversity parameters of 10 pairs of SSR markers in 193 samples
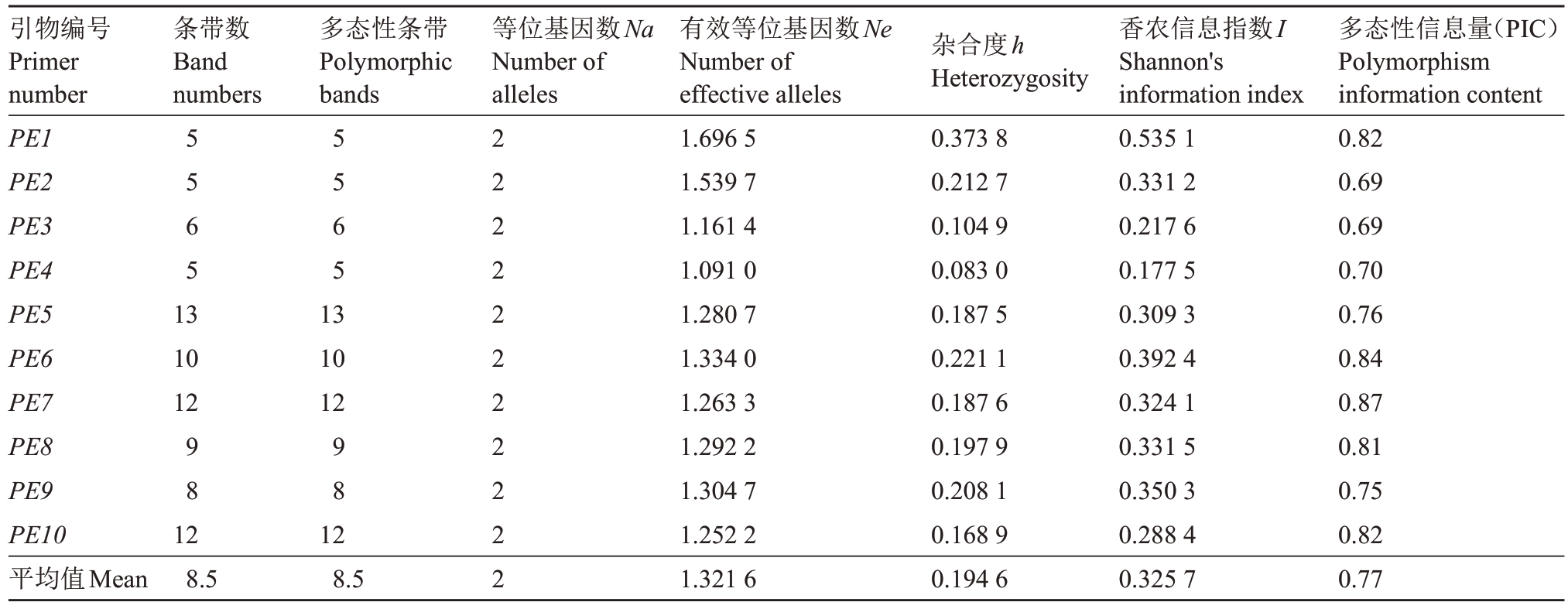
引物编号Primer number PE1 PE2 PE3 PE4 PE5 PE6 PE7 PE8 PE9 PE10平均值Mean条带数Band numbers多态性条带Polymorphic bands等位基因数Na Number of alleles 5 5 6 5 5 5 6 5 13 10 12 13 10 12 9 8 9 8 12 8.5 12 8.5 2 2 2 2 2 2 2 2 2 22有效等位基因数Ne Number of effective alleles 1.696 5 1.539 7 1.161 4 1.091 0 1.280 7 1.334 0 1.263 3 1.292 2 1.304 7 1.252 2 1.321 6杂合度h Heterozygosity 0.373 8 0.212 7 0.104 9 0.083 0 0.187 5 0.221 1 0.187 6 0.197 9 0.208 1 0.168 9 0.194 6香农信息指数I Shannon's information index 0.535 1 0.331 2 0.217 6 0.177 5 0.309 3 0.392 4 0.324 1 0.331 5 0.350 3 0.288 4 0.325 7多态性信息量(PIC)Polymorphism information content 0.82 0.69 0.69 0.70 0.76 0.84 0.87 0.81 0.75 0.82 0.77

图2 引物PE8 对193 份样品的扩增结果
Fig.2 The amplification results of primers PE8 in 193 samples
图中仅展示目标区域条带,1~193 对应样品编号No.1~No.193,M 为DNA Marker。
Only the bands in the target area were shown in the figure, and the corresponding samples from 1 to 193 were numbered No. 1-No. 193, and M was DNA Marker.
利用10 对SSR 引物扩增样品,根据电泳结果,计算193份样品间的Nei&Li遗传距离,结果表明,192 份余甘子种质资源的变异范围为0~0.958 3,表明余甘子种质资源间的遗传分化水平高,遗传变异范围大,遗传差异显著,多样性丰富。No. 107 与No.108的遗传距离为0,种质间差异为0,结合表型可进一步判定是否为同种;No.116 和No.115 遗传距离为0.001,No.20 和No.19 遗传距离为0.006,表明这几组种质的亲缘关系非常相近,样品间的遗传变异小,可能为相同或相近种质;遗传距离较大的有No.158与No.2(30.958 3)、No.158与No.3(90.957 4)、No. 188 与No. 149(0.944 4)、No. 159 与No. 121(0.904 8)说明这几份种质的亲缘关系较远;近缘种No. 172 与192 份余甘子种质资源的遗传距离范围为0.818 2~1,与No.190(广大甘,广东)亲缘关系最近;193份种质中近缘种No.172与66份余甘子种质的遗传距离为1,其中来源福建和广东的种质占96.97%。
2.2 聚类分析
193 份样品的UPGWA 聚类结果见图3,除近缘种No. 172 单独聚类外,其余192 份在遗传距离为0.6可分为3个组别,第1组主要为福建、广东和广西等来源地的地方品种和野生种质(170 份),该区域同属相似的湿润区生态条件,最先聚在一起;第2组为云南种质和境外种质(21份),同属相似的干热区生态条件,与前期表型遗传多样性研究的聚类结果一致,该区域种质表现结果枝上叶片数量多,叶面积和周长小等特点[17];第3组为来自福建的No.158野生种质,该种质与其他种质亲缘关系均较远,可作为对开展余甘子起源和遗传基因历史演变等研究潜在试验材料。

图3 193 份样品的遗传聚类分析
Fig.3 Genetic clustering map of 193 samples
序号对应种质名称同表1。下同。
The germplasm name corresponding to the number are same Table 1.The same below.
根据聚类图,第1组别170份资源又可分为8个亚类。第1 亚类聚集了99 份样品,主要为来源广东(54份)和福建(40份)的种质,种质类型以地方品种(系)和野生种质为主,广东的甜种、青皮、狮头和饼甘等,福建的粉甘和六月白等品系在此聚类,此外还有两地的野生种质,表明生态环境相似、地理来源相近地区的余甘子种质资源遗传关系较近,存在频繁基因交流。第2 亚类聚集了49 份样品,主要是来源福建的野生资源和地方品种(系),占93.88%,包括蓝丰、秋白和扁甘等地方品种(系),表明在此聚类的福建野生资源和主要的地方品种遗传差异较小、亲缘关系较近,福建地区种质表现出一定的地理区域特性。第3亚类聚集了3份样品,其中No.70和No.132 均为福建种,No.70 是广玻甘的实生后代与No.132遗传差异较小,遗传距离为0.379 3。第4亚类聚集了3份样品,分别为福建野生资源2份和广东地方品系青皮05,3 份种质遗传差异较小,遗传距离为0.404 3~0.647 1。第5 亚类聚集了3 份样品,遗传距离为0.476 2~0.523 8,其中包括2 份福建野生种和1份云南野生种,表明在漫长的自然环境发展过程中,生态条件差异大的地区种质之间也可能存在基因的交流。第6亚类聚集了4份样品,均为福建的野生资源,相互之间的遗传距离为0.404 3~0.523 8。第7 亚类聚集了6份样品,其中来源广东的No.184玻璃种与No.148 青皮07 的遗传距离为0.463 4,两个品系遗传信息相似度较高。第8亚类福建种质3份,分别为福建地方品系枣甘与2 份野生种,遗传距离为0.523 8~0.617 0。
2.3 主成分分析
根据SSR 分子标记结果进行主成分分析,以PC1 和PC2 构建193 份样品的二维主成分散点图(图4)。图中样品位置近则亲缘关系近、位置远则亲缘关系远[21],从PCA图中可以明显地看出,193份材料中第2 组类别(云南及境外种质,图中圈内种质)与其他类别分开,其他类别含有不同地理来源的余甘子种质资源,PCA 主成分分析与UPGMA 聚类分析结果较一致,不同生态分布区的种质相互分开,相同生态分布区的种质亲缘关系相对较近。
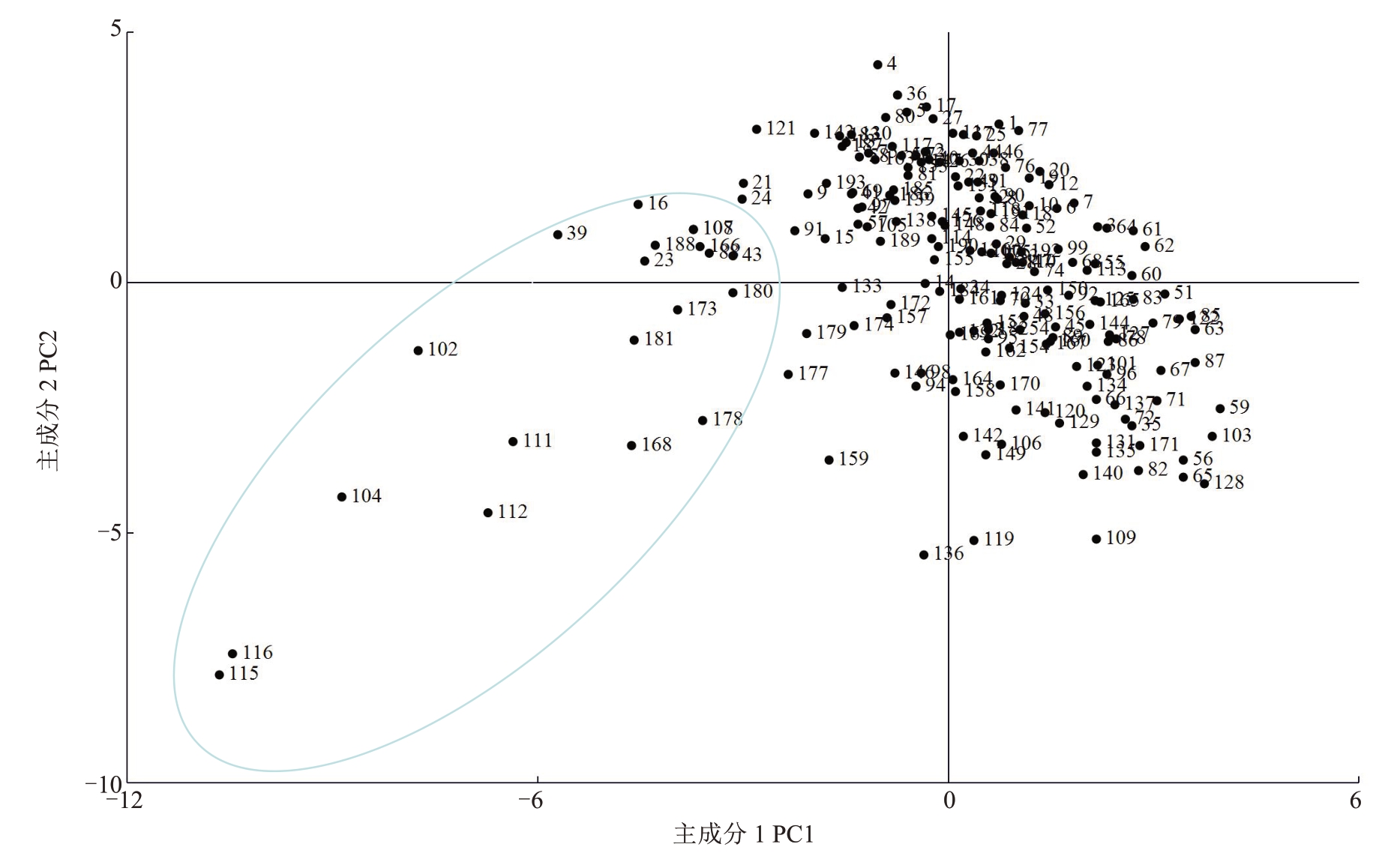
图4 193 份样品主成分分析二维散点图
Fig.4 Two-dimensional scatter plot of PCA of 193 samples
2.4 核心种质构建方法的确定
2.4.1 聚类方法与取样策略的确定 计算不同遗传距离结合不同取样策略下核心种质与原种质遗传多样性参数差异(表4),结果表明,优先取样策略下,以3 种遗传距离聚类时,SM 遗传距离的多态位点、多态位点百分率和等位基因数均小于Jaccard 和Nei&Li;以Nei&Li聚类时,其有效等位基因、Nei’s基因多样性指数和香农信息指数均小于Jaccard 和SM;以Jaccard 聚类的香农信息指数高于Nei&Li 和SM。随机取样策略下,Jaccard 和SM 的多态位点、多态位点百分率、等位基因、有效等位基因、Nei’s基因多样性指数和香农信息指数均相同,且有效等位基因、Nei’s基因多样性指数和香农信息指数均高于Nei&Li 遗传距离。综上所述,选择Jaccard 遗传距离进行多次UPGMA 聚类,取样策略则采用优先取样策略。
表4 不同构建方法核心种质与原种质的遗传参数差异比较
Table 4 Comparison of genetic parameter between core and original germplasm based on different construction methods
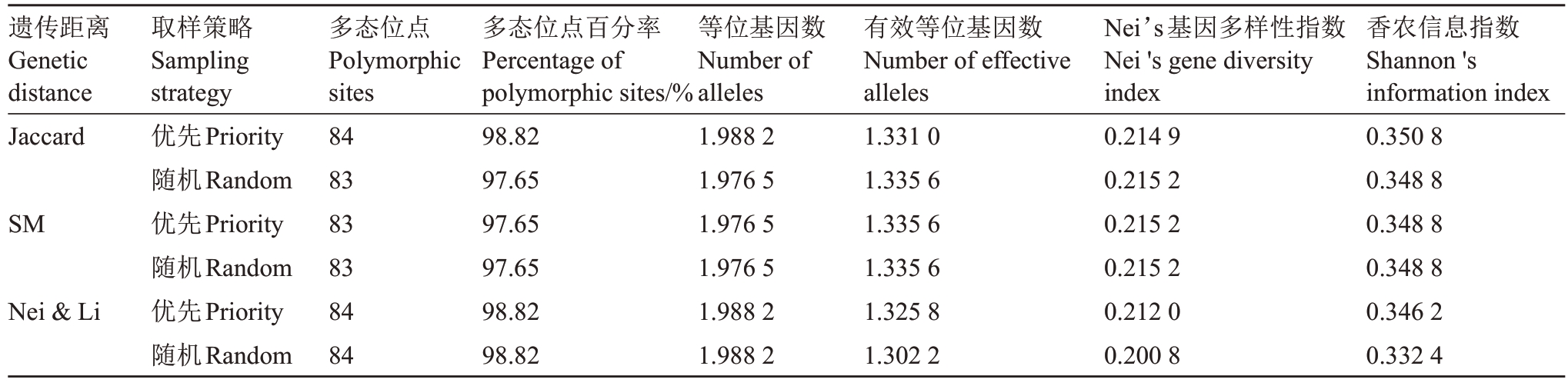
遗传距离Genetic distance Jaccard SM Nei&Li取样策略Sampling strategy优先Priority随机Random优先Priority随机Random优先Priority随机Random多态位点Polymorphic sites 84 83 83 83 84 84多态位点百分率Percentage of polymorphic sites/%98.82 97.65 97.65 97.65 98.82 98.82等位基因数Number of alleles 1.988 2 1.976 5 1.976 5 1.976 5 1.988 2 1.988 2有效等位基因数Number of effective alleles 1.331 0 1.335 6 1.335 6 1.335 6 1.325 8 1.302 2 Nei’s基因多样性指数Nei's gene diversity index 0.214 9 0.215 2 0.215 2 0.215 2 0.212 0 0.200 8香农信息指数Shannon's information index 0.350 8 0.348 8 0.348 8 0.348 8 0.346 2 0.332 4
2.4.2 取样比例确定 根据筛选出的最佳聚类和取样方案(Jaccard遗传距离UPWAG聚类,结合优先取样策略),以取样比例为10%、15%、20%、25%、30%、35%和40%共7个比例抽取核心种质,核心种质与原种质的评价参数见表5。结果可知,随着取样比例的增大,多态位点、多态位点百分率和等位基因数均呈增长趋势,多态位点81~85,多态位点百分率95.29%~100%,等位基因1.952 9~2.000 0。10%取样比例时,有效等位基因(1.339 6)、Nei’s 基因多样性指数(0.223 8)和香农信息指数(0.363 7)均为最高,表明该比例下很好地保留原种质群体基因位点的情况下,较好地保留了遗传多样性,所以选择10%为最适取样比例(真实比例为10.42%),最终构建包含20份余甘子种质资源的初级核心种质。
表5 不同取样比例下核心种质与原种质遗传参数差异比较
Table 5 Comparison of genetic parameter between core and original germplasm of different sampling ratios
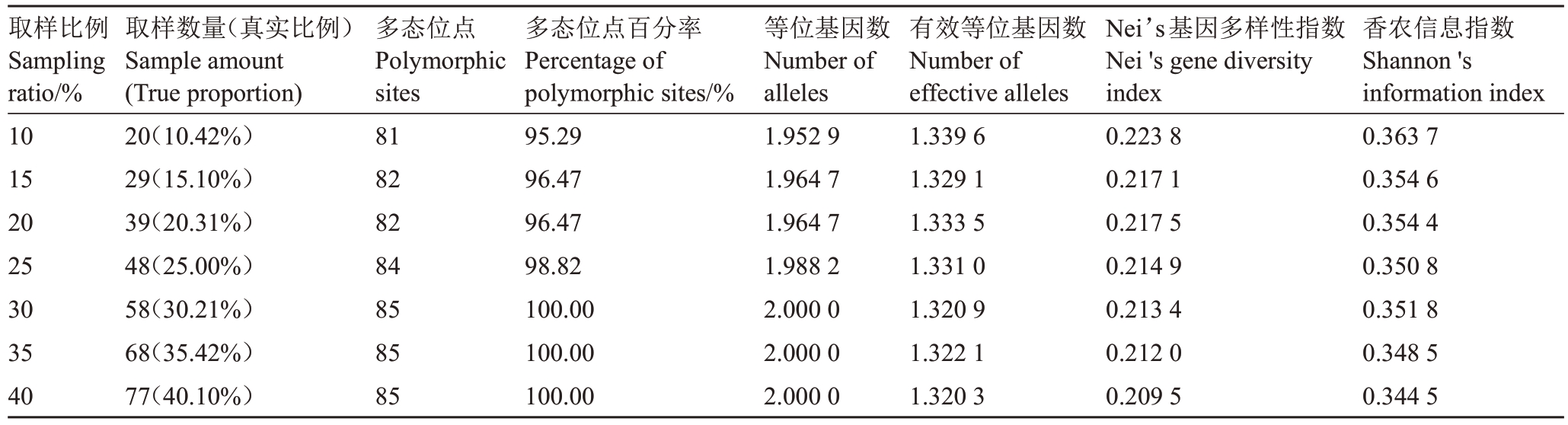
取样比例Sampling ratio/%10 15 20 25 30 35 40取样数量(真实比例)Sample amount(True proportion)20(10.42%)29(15.10%)39(20.31%)48(25.00%)58(30.21%)68(35.42%)77(40.10%)多态位点Polymorphic sites 81 82 82 84 85 85 85多态位点百分率Percentage of polymorphic sites/%95.29 96.47 96.47 98.82 100.00 100.00 100.00等位基因数Number of alleles 1.952 9 1.964 7 1.964 7 1.988 2 2.000 0 2.000 0 2.000 0有效等位基因数Number of effective alleles 1.339 6 1.329 1 1.333 5 1.331 0 1.320 9 1.322 1 1.320 3 Nei’s基因多样性指数Nei's gene diversity index 0.223 8 0.217 1 0.217 5 0.214 9 0.213 4 0.212 0 0.209 5香农信息指数Shannon's information index 0.363 7 0.354 6 0.354 4 0.350 8 0.351 8 0.348 5 0.344 5
2.4.3 核心种质评价 核心种质与初始种质遗传参数比较(表6)可知,核心种质保留了原种质10.42%的种质,多态性位点保留率95.29%,多态百分率保留率95.29%,等位基因保留率97.65%,有效等位基因保留率102.70%,Nei’s 基因多样性指数保留率112.41%,香农信息指数保留率110.48%,遗传多样性参数有效等位基因、Nei’s基因多样性指数和香农信息指数均高于原种质和保留种质,表明核心种质能很好地代表原种质。以SSR分子标记的遗传多样性数据构建余甘子初级核心种质20份,种质编号为4、28、56、77、83、87、94、98、102、112、121、135、150、163、169、170、176、177、178和182。
表6 核心种质与原种质遗传参数比较
Table 6 Comparison of genetic parameters between core and original germplasm
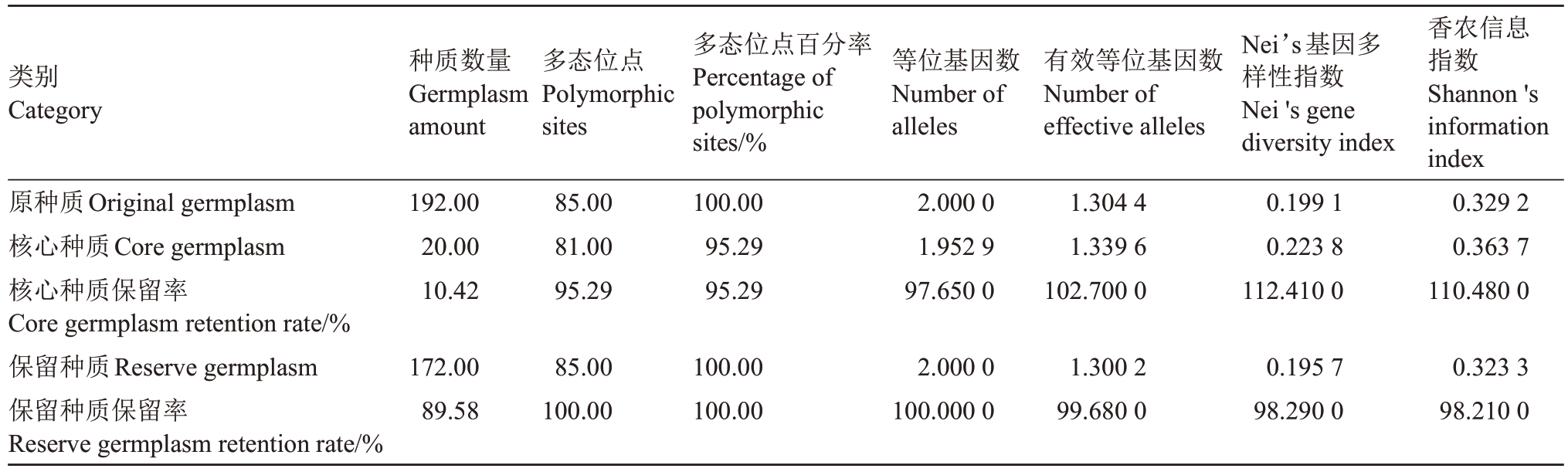
类别Category种质数量Germplasm amount多态位点Polymorphic sites等位基因数Number of alleles有效等位基因数Number of effective alleles原种质Original germplasm核心种质Core germplasm核心种质保留率Core germplasm retention rate/%保留种质Reserve germplasm保留种质保留率Reserve germplasm retention rate/%192.00 20.00 10.42 85.00 81.00 95.29多态位点百分率Percentage of polymorphic sites/%100.00 95.29 95.29 2.000 0 1.952 9 97.650 0 1.304 4 1.339 6 102.700 0 Nei’s基因多样性指数Nei's gene diversity index 0.199 1 0.223 8 112.410 0香农信息指数Shannon's information index 0.329 2 0.363 7 110.480 0 172.00 89.58 85.00 100.00 100.00 100.00 2.000 0 100.000 0 1.300 2 99.680 0 0.195 7 98.290 0 0.323 3 98.210 0
进一步分别对20份核心种质和192份余甘子原种质的SSR 分子数据进行主成分分析,图5 为两份种质集合的主成分得分散点图。可知,核心种质散点分布区域和形状与原种质散点分布区域和形状重合度较高,外围的个体也被选入核心种质库中,遗传相似性高的株系中仅有部分入选,排除遗传冗余,同时又确保了核心种质的代表性和异质性。综上所述,最终构建的核心种质能够很好地代表原始群体,具有很好的异质性,在一定程度上剔除了遗传重复。余甘子核心种质的建立为资源的保存评价、特异资源挖掘和高效利用提供基础和保障,在今后的种质资源保护、评价和创新利用工作中给予充分的重视。
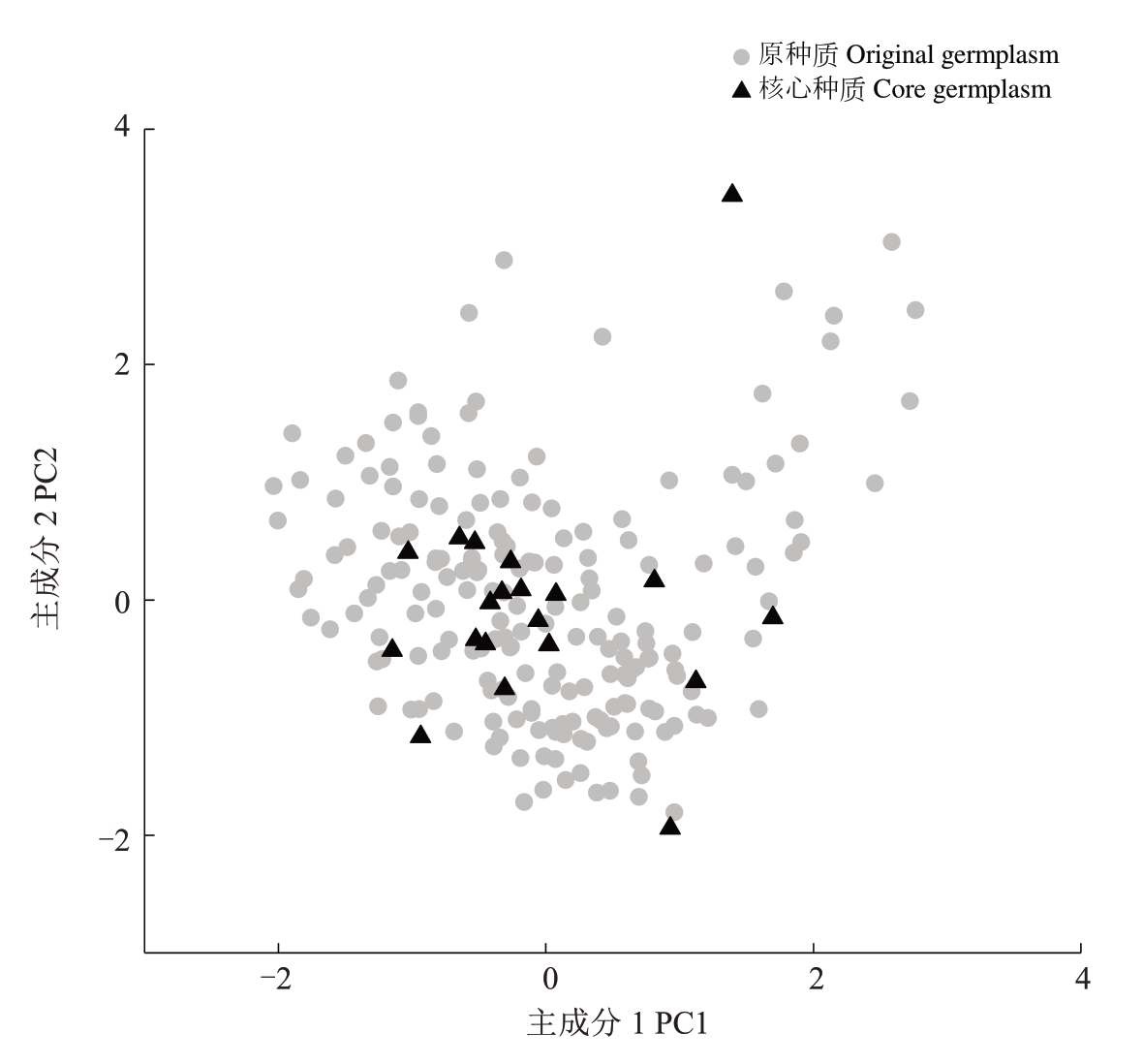
图5 核心种质与原种质主成分得分图
Fig.5 The PCA scores of between core and original germplasm
3 讨 论
3.1 余甘子遗传多样性分析
利用SSR分子标记开展余甘子遗传多样性分析的结果表明,192 份余甘子种质资源的变异范围为0~0.958 3,余甘子种质资源间的遗传分化水平高,遗传变异范围大,表现出丰富的遗传多样性。两份云南野生种质(No. 107 和No. 108)的遗传距离为0,种质间差异为0,结合前期研究结果,两份种质表型数据基本相当,可进一步判定为同种资源。聚类分析已普遍应用于植物的遗传分类、亲缘关系和遗传多样性评价中[22-23],中国野生余甘子长期对多样性气候的适应,造就了资源分布的多样性[24]。笔者在本研究中通过UPGMA聚类,将192份余甘子种质资源分为3个组别,第1组别可分为8个亚类,类群的划分与生态分布有较大关联,余甘子的湿润分布区和干热分布区得到较好区分,生态区内不同省份的种质在聚类中并没有很明显的分开,多个省份的资源分散在不同的类群中,说明相同生态分布区域内的种质分类与地域来源相关性不明显,与普天磊等[25]研究结果一致。福建地区一部分种质单独聚为一类(第2 亚类),该部分余甘子种质的亲缘关系远近与福建省的地理分布有一定的相关性,较能反映福建省的种质特点,福建省地域立体气候复杂,余甘子种子传播范围有限,而且群体间地理隔离较大,群体间进行基因交流的机会少,导致余甘子群体间存在较大遗传差异。
试验材料来源于中国云南、缅甸、泰国和印度的种质聚类在一起,前人研究将云南划分为中国余甘子的干热分布区[13],与其生态气候类似的缅甸、泰国和印度等地区种质的亲缘关系较近,表明余甘子的遗传变异与气候的相关性高于地域分布,研究结果与余甘子分布的海拔和降水量与遗传多样性有显著相关性的结果[26]较一致。在本研究中,两个大果余甘子种质资源(No. 168 和No. 173)的遗传距离为0.733 3,两份种质的遗传距离较大,亲缘关系较远,是不同的两份种质,与应用ISSR 分析结果一致[27]。No.158 是来源福建的野生种质,单独聚为一类,与其他191份种质的遗传距离范围为0.550 0~0.958 3,是与来源福建的种质也有较远亲缘关系的特殊资源,可能由福建省独特地理环境因素、局部地理隔离导致,可作为余甘子起源和遗传基因历史演变等方面研究的潜在试验材料。笔者在本研究中开发的SSR分子标记技术可较好地反映余甘子的遗传差异和亲缘关系,但传统SSR 分析只针对微卫星区域的多态性,无法捕捉单核苷酸多态性(SNP)等其他类型变异,在今后的研究中可开发多态信息量大、自动化程度高的SNP 标记,为余甘子的资源鉴定、分类和选育种提供参考。
3.2 余甘子核心种质构建
核心种质的概念是1984 年澳大利亚科学家Frankle提出的,后经Brown扩展[28],核心种质是以最小资源量和重复性反映整体的遗传多样性,未被选择的资源则作为保留资源。开展余甘子核心种质构建不仅有利于种质资源的保存与鉴定评价,而且可大大提高资源利用效率[29]。在构建核心种质过程中,逐步聚类是常用方法[30],聚类结果直接受遗传距离和聚类方法的影响。构建核心种质方法中,常用的遗传距离是欧氏和马氏距离[31]。笔者在本研究中利用SSR 分子标记数据构建核心种质的遗传距离采用Jaccard 遗传距离+类平均法,与显性分子标记多使用SM遗传距离,而共显性分子标记利用Jaccard 和Nei&Li 较为理想的前人研究结果较一致[30]。徐海明等[32]筛选出马氏距离结合最短距离法最适合构建陆地棉核心种质;高志红等[33]利用欧氏距离和类平均法构建中国果梅核心种质;董玉慧[34]构建枣核心种质认为欧氏距离结合类平均法和离差平方与聚类效果较好;刘遵春等[31]采用欧氏距离结合最短距离法聚类构建新疆野苹果的核心种质,张春雨等[35]则认为离差平方法是最适宜方法;马玉敏[36]研究表明构建中国板栗最适宜的聚类方法为离差平方和法;刘娟[37]构建栽培杏和野杏核心种质时,认为最短距离法和离差平方和法的构建效果较好。综上所述,因不同类型数据或物种的独特性,对遗传距离与聚类方法不能一概而论,应由具体的对象经分析研究而定。
从取样比例看,笔者在本研究中构建核心种质的真实取样比例为10.42%,符合前人总结的大多数园艺作物构建核心种质取样比例为10%~30%的研究结果[38],前人总结原种质总量的5%~10%取样比例构建核心种质可保留70%以上的遗传变异[39]。李秀诗等[40]基于248份薏仁构建核心种质发现最适取样比例为25%,胡建斌等[41]构建甜瓜核心种质发现15%的取样比例时遗传保留和原种质的代表性达到最佳。在核心种质的进一步研究中,需尽量增加表型性状的数量包括植株、叶片、花和果实品质等的数据,此外还包括诸如抗性、产量等农艺性状数据,避免重要遗传性状的种质丢失,将更多的变异类型提取至核心种质库内,对全面构建余甘子核心种质有重要意义。核心种质建成后需动态管理,在实践中进行适当调整,并及时补充稀有、有用的材料,扩大保存变异类型,确保保存的全面性、长期性和安全性,为余甘子的育种和创新利用提供保障。
4 结 论
利用10 对SSR 引物对193 份样品扩增后进行遗传多样性分析,结果表明余甘子种质资源的遗传分化水平高,遗传变异范围大,遗传多样性丰富,开发的SSR分子标记技术,可较好地反映余甘子的遗传差异和亲缘关系。筛选出构建余甘子核心种质的最佳方案为Jaccard 遗传距离+类平均法+优先取样策略+10%取样比例,构建了包含20 份余甘子的核心种质,核心种质有效地排除了遗传冗余,同时又确保了原种质的代表性和异质性。
[1] 中国科学院中国植物志编辑委员会.中国植物志-余甘子[M].北京:科学出版社,2004.Chinese Flora Committee,Chinese Academy of Sciences. Flora reipublicae popularis sinicae-Phyllanthus emblica Linn.[M].Beijing:Science Press,2004.
[2] 吴玲芳,叶婷,梁林金,梁文仪,陈文静,李师,崔亚萍,亓旗,张兰珍.藏药余甘子鞣质部位主要药效成分在人工胃肠液中的稳定性研究[J]. 世界科学技术-中医药现代化,2017,19(8):1386-1390.WU Lingfang,YE Ting,LIANG Linjin,LIANG Wenyi,CHEN Wenjing,LI Shi,CUI Yaping,QI Qi,ZHANG Lanzhen. Research on stability of tannin part in Phyllanthus emblica L.in artificial gastric and intestinal juice[J].Modernization of Traditional Chinese Medicine and Materia Medica-World Science and Technology,2017,19(8):1386-1390.
[3] ZHAO H J,LIU T,MAO X,HAN S X,LIANG R X,HUI L Q,CAO C Y,YOU Y,ZHANG L Z.Fructus phyllanthi tannin fraction induces apoptosis and inhibits migration and invasion of human lung squamous carcinoma cells in vitro via MAPK/MMP pathways[J].Acta Pharmacologica Sinica,2015,36(6):758-768.
[4] CHAPHALKAR R,APTE K G,TALEKAR Y,OJHA S K,NANDAVE M.Antioxidants of Phyllanthus emblica L. bark extract provide hepatoprotection against ethanol-induced hepatic damage:A comparison with silymarin[J]. Oxidative Medicine and Cellular Longevity,2017,2017(1):3876040.
[5] 王建超,张小艳,谢丽雪,张立杰,李韬.余甘子药用价值研究进展[J].东南园艺,2024,12(6):536-546.WANG Jianchao,ZHANG Xiaoyan,XIE Lixue,ZHANG Lijie,LI Tao.Research progress on medicinal value of Phyllanthus emblica L.[J].Southeast Horticulture,2024,12(6):536-546.
[6] 王建超,陈志峰,郭林榕.我国余甘子种质资源生态分布区域综述[J].东南园艺,2020,8(2):57-60.WANG Jianchao,CHEN Zhifeng,GUO Linrong. Summary on ecological distribution of Phyllanthus emblica germplasm resources in China[J].Southeast Horticulture,2020,8(2):57-60.
[7] MAO Y F,BOTELLA J R,LIU Y G,ZHU J K. Gene editing in plants:Progress and challenges[J]. National Science Review,2019,6(3):421-437.
[8] 胡文舜,陈秀萍,郑少泉.龙眼EST-SSR 标记开发及无患子科5 个属种质遗传多样性分析[J].园艺学报,2019,46(7):1359-1372.HU Wenshun,CHEN Xiuping,ZHENG Shaoquan. EST- SSR markers developed from Dimocarpus longan and their application in genetic diversity analysis of five genera of Sapindaceae[J].Acta Horticulturae Sinica,2019,46(7):1359-1372.
[9] CHAURASIA A K,SUBRAMANIAM V R,KRISHNA B,SANE P V.RAPD based genetic variability among cultivated varieties of Aonla (Indian Gooseberry,Phyllanthus emblica L.)[J].Physiology and Molecular Biology of Plants,2009,15(2):169-173.
[10] DNYANESHWAR W,PREETI C,KALPANA J,BHUSHAN P.Development and application of RAPD-SCAR marker for identification of Phyllanthus emblica Linn.[J]. Biological & Pharmaceutical Bulletin,2006,29(11):2313-2316.
[11] THILAGA S,NAIR R R,KANNAN M R,GANESH D. RAPD markers for screening shoot gall maker(Betousa stylophora Swinhoe)tolerant genotypes of amla(Phyllanthus emblica L.)[J].Journal of Genetic Engineering and Biotechnology,2017,15(2):323-330.
[12] 蔡英卿,赖钟雄,陈义挺,郭玉琼,潘东明.福建余甘子遗传资源的RAPD 分析[J].热带作物学报,2007,28(2):74-79.CAI Yingqing,LAI Zhongxiong,CHEN Yiting,GUO Yuqiong,PAN Dongming. RAPD analysis of emblic (Phyllanthus emblica L.) genetic resources in Fujian Province[J]. Chinese Journal of Tropical Crops,2007,28(2):74-79.
[13] 熊仪俊.余甘子不同生态型特征与分化初步研究[D].北京:中国林业科学研究院,2003.XIONG Yijun. Preliminary study on the characteristics and differentiations of ecological types of Phyllanthus emblica[D].Beijing:Chinese Academy of Forestry,2003.
[14] 郭林榕,周平,陈志峰.22 份余甘子核心种质资源遗传多样性的SRAP 分析[J].热带作物学报,2014,35(7):1382-1387.GUO Linrong,ZHOU Ping,CHEN Zhifeng.Analysis of genetic diversity of 22 Phyllanthus emblica germplasms with SRAP markers[J]. Chinese Journal of Tropical Crops,2014,35(7):1382-1387.
[15] 刘晓生,郑道序,周春娟,马瑞君,詹潮安,杨海东,杨培奎,庄东红.潮汕余甘子种质资源遗传多样性与亲缘关系的ISSR分析[J].中国南方果树,2014,43(1):18-22.LIU Xiaosheng,ZHENG Daoxu,ZHOU Chunjuan,MA Ruijun,ZHAN Chaoan,YANG Haidong,YANG Peikui,ZHUANG Donghong. Analysis of genetic diversity and genetic relationship of Phyllanthus emblica L. germplasm in Chaoshan area with ISSR[J].South China Fruits,2014,43(1):18-22.
[16] 王建超,何银莺,黄旭萍,沈朝贵,陈发兴,郭林榕.响应面法优化余甘子种质资源ISSR 反应体系[J]. 福建农业学报,2021,36(7):759-765.WANG Jianchao,HE Yinying,HUANG Xuping,SHEN Chaogui,CHEN Faxing,GUO Linrong. Response surface optimization of ISSR-PCR reaction for genetic study on Phyllanthus emblica[J].Fujian Journal of Agricultural Sciences,2021,36(7):759-765.
[17] 王建超.余甘子(Phyllanthus emblica L.)遗传多样性分析与核心种质构建[D].福州:福建农林大学,2024.WANG Jianchao. Genetic diversity analysis and core collection constrution of Phyllanthus emblica L.[D]. Fuzhou:Fujian Agriculture and Forestry University,2024.
[18] 李金璐,王硕,于婧,王玲,周世良.一种改良的植物DNA 提取方法[J].植物学报,2013,48(1):72-78.LI Jinlu,WANG Shuo,YU Jing,WANG Ling,ZHOU Shiliang.A modified CTAB protocol for plant DNA extraction[J]. Chinese Bulletin of Botany,2013,48(1):72-78.
[19] 周雨晴,戴文婧,杨羽清,姚森洋,周圆,程智慧,潘玉朋.基于SSR 标记的126 份甜瓜种质遗传多样性分析[J].西北农业学报,2025,34(1):54-62.ZHOU Yuqing,DAI Wenjing,YANG Yuqing,YAO Senyang,ZHOU Yuan,CHENG Zhihui,PAN Yupeng. Genetic diversity analysis of 126 melon germplasms based on SSR markers[J].Acta Agriculturae Boreali-occidentalis Sinica,2025,34(1):54-62.
[20] ESCRIBANO P,VIRUEL M A,HORMAZA J I.Comparison of different methods to construct a core germplasm collection in woody perennial species with simple sequence repeat markers.A case study in cherimoya (Annona cherimola,Annonaceae),an underutilised subtropical fruit tree species[J].Annals of Applied Biology,2008,153(1):25-32.
[21] 陈越,陈玲,李春花,张敦宇,付坚,程在全.中国南方地区水稻资源SSR 指纹数据库的构建及遗传多样性分析[J].分子植物育种,2020,18(19):6502-6517.CHEN Yue,CHEN Ling,LI Chunhua,ZHANG Dunyu,FU Jian,CHENG Zaiquan. Construction of SSR fingerprint database and genetic diversity analysis of rice resources in Southern China[J].Molecular Plant Breeding,2020,18(19):6502-6517.
[22] 赵青,都真真,李锡香,宋江萍,张晓辉,阳文龙,贾会霞,王海平.利用SSRseq 分子标记的大蒜种质资源遗传多样性研究[J].园艺学报,2021,48(7):1397-1408.ZHAO Qing,DU Zhenzhen,LI Xixiang,SONG Jiangping,ZHANG Xiaohui,YANG Wenlong,JIA Huixia,WANG Haiping.Genetic diversity of garlic germplasm resources based on SSRseq molecular markers[J]. Acta Horticulturae Sinica,2021,48(7):1397-1408.
[23] 徐婉,林雅君,赵荘,周庄.兰属植物资源与育种研究进展[J].园艺学报,2022,49(12):2722-2742.XU Wan,LIN Yajun,ZHAO Zhuang,ZHOU Zhuang.Advances in genetic resources and breeding research of Cymbidium[J].Acta Horticulturae Sinica,2022,49(12):2722-2742.
[24] 陈进,陈贵清,胡建湘,杨坤,李其惠.滇南野生余甘子果实性状变异规律[J].福建果树,1991(1):63-64.CHEN Jin,CHEN Guiqing,HU Jianxiang,YANG Kun,LI Qihui. Variation law of fruit traits of wild Phyllanthus emblica in southern Yunnan[J].Fujian Fruits,1991(1):63-64.
[25] 普天磊,金杰,何璐,瞿文林,廖承飞,袁建民,罗会英,赵琼玲.基于SNP 和InDel 标记的余甘子群体遗传分析[J].果树学报,2023,40(5):875-883.PU Tianlei,JIN Jie,HE Lu,QU Wenlin,LIAO Chengfei,YUAN Jianmin,LUO Huiying,ZHAO Qiongling.Population and genetic analysis of Phyllanthus emblica by SNP and InDel markers[J].Journal of Fruit Science,2023,40(5):875-883.
[26] LIU X F,MA Y P,WAN Y M,LI Z H,MA H.Genetic diversity of Phyllanthus emblica from two different climate type areas[J].Frontiers in Plant Science,2020,11:580812.
[27] 王建超,何银莺,黄旭萍,陈发兴,郭林榕.余甘子种质资源的遗传多样性分析[J].森林与环境学报,2021,41(4):396-401.WANG Jianchao,HE Yinying,HUANG Xuping,CHEN Faxing,GUO Linrong. Genetic diversity analysis of Phyllanthus emblica germplasm resources[J]. Journal of Forest and Environment,2021,41(4):396-401.
[28] 张咏琦,王超,许林林,吴娥娇,李天红,赵密珍,袁华招.野生二倍体草莓的SSR 分子标记开发及核心种质的构建[J].园艺学报,2023,50(11):2365-2375.ZHANG Yongqi,WANG Chao,XU Linlin,WU Ejiao,LI Tianhong,ZHAO Mizhen,YUAN Huazhao. Development of SSR molecular markers and construction of core collection of wild diploid strawberry[J]. Acta Horticulturae Sinica,2023,50(11):2365-2375.
[29] MALOSETTI M,ABADIE T. Sampling strategy to develop a core collection of Uruguayan maize landraces based on morphological traits[J]. Genetic Resources and Crop Evolution,2001,48(4):381-390.
[30] ZIETKIEWICZ E,RAFALSKI A,LABUDA D.Genome fingerprinting by simple sequence repeat (SSR)-anchored polymerase chain reaction amplification[J]. Genomics,1994,20(2):176-183.
[31] 刘遵春,张春雨,张艳敏,张小燕,吴传金,王海波,石俊,陈学森.利用数量性状构建新疆野苹果核心种质的方法[J].中国农业科学,2010,43(2):358-370.LIU Zunchun,ZHANG Chunyu,ZHANG Yanmin,ZHANG Xiaoyan,WU Chuanjin,WANG Haibo,SHI Jun,CHEN Xuesen. Study on method of constructing core collection of Malus sieversii based on quantitative traits[J].Scientia Agricultura Sinica,2010,43(2):358-370.
[32] 徐海明,邱英雄,胡晋,王建成.不同遗传距离聚类和抽样方法构建作物核心种质的比较[J].作物学报,2004,30(9):932-936.XU Haiming,QIU Yingxiong,HU Jin,WANG Jiancheng.Methods of constructing core collection of crop germplasm by comparing different genetic distances,cluster methods and sampling strategies[J].Acta Agronomica Sinica,2004,30(9):932-936.
[33] 高志红,章镇,韩振海,房经贵.中国果梅核心种质的构建与检测[J].中国农业科学,2005,38(2):363-368.GAO Zhihong,ZHANG Zhen,HAN Zhenhai,FANG Jinggui.Development and evaluation of core collection of Japanese apricot germplasms in China[J]. Scientia Agricultura Sinica,2005,38(2):363-368.
[34] 董玉慧.枣树农艺性状遗传多样性评价与核心种质构建[D].保定:河北农业大学,2008.DONG Yuhui. Evaluation on genetic diversity of agronomic characters and core collection construction in Ziziphus jujuba Mill.[D].Baoding:Hebei Agricultural University,2008.
[35] 张春雨,陈学森,张艳敏,苑兆和,刘遵春,王延龄,林群.采用分子标记构建新疆野苹果核心种质的方法[J].中国农业科学,2009,42(2):597-604.ZHANG Chunyu,CHEN Xuesen,ZHANG Yanmin,YUAN Zhaohe,LIU Zunchun,WANG Yanling,LIN Qun.A method for constructing core collection of Malus sieversii using molecular markers[J].Scientia Agricultura Sinica,2009,42(2):597-604.
[36] 马玉敏.中国野生板栗(Castanea mollissim Blume)群体遗传结构和核心种质构建方法[D].泰安:山东农业大学,2009.MA Yumin. Population genetic structure and method of constructing core collection for Castanea mollissim Blume[D].Taian:Shandong Agricultural University,2009.
[37] 刘娟.新疆杏种质资源遗传多样性及核心种质构建[D].乌鲁木齐:新疆农业大学,2015.LIU Juan. Analysis of genetic diversity and establishment of core collection of apricot germplasm resources in Xinjiang[D].Urumqi:Xinjiang Agricultural University,2015.
[38] 缪黎明,王神云,邹明华,李建斌,孔李俊,余小林.园艺作物核心种质构建的研究进展[J].植物遗传资源学报,2016,17(5):791-800.MIAO Liming,WANG Shenyun,ZOU Minghua,LI Jianbin,KONG Lijun,YU Xiaolin.Review of the studies on core collection for horticultural crops[J]. Journal of Plant Genetic Resources,2016,17(5):791-800.
[39] BROWN A H D.Core collections:A practical approach to genetic resources management[J].Genome,1989,31(2):818-824.
[40] 李秀诗,付瑜华,周祥,黎青,刘凡值,杨成龙,周明强.基于表型性状的薏苡初级核心种质库构建[J].热带作物学报,2020,41(4):669-675.LI Xiushi,FU Yuhua,ZHOU Xiang,LI Qing,LIU Fanzhi,YANG Chenglong,ZHOU Mingqiang. Establishment of Coix lacryma-jobi L. core germplasm collection based on phenotypic characters[J]. Chinese Journal of Tropical Crops,2020,41(4):669-675.
[41] 胡建斌,马双武,王吉明,苏艳,李琼.基于表型性状的甜瓜核心种质构建[J].果树学报,2013,30(3):404-411.HU Jianbin,MA Shuangwu,WANG Jiming,SU Yan,LI Qiong.Establishment of a melon (Cucumis melo) core collection based on phenotypic characters[J]. Journal of Fruit Science,2013,30(3):404-411.