板栗(Castanea mollissima)是壳斗科(Fagaceae)栗属(Castanea)树种,是中国重要的木本粮食树种,也是中国五大优势经济林树种之一。板栗在东亚地区广泛分布,并已成为北半球重要的食物来源,具有重要的经济和生态价值[1-2]。板栗坚果不仅营养丰富,而且具有独特的风味和药用价值[3]。板栗生长发育过程中需要应对各种环境胁迫,如极端温度、干旱、盐胁迫以及病虫害[4]。开展板栗发育和环境胁迫相关基因家族的鉴定和全面表征对使用分子手段进行优质抗逆板栗新品种培育具有重要意义。
转录因子是一类能够调控基因转录的蛋白质,可以结合到DNA的特定序列上,通过与RNA聚合酶或其他转录因子相互作用,调控基因的转录速率[5]。在植物中,转录因子扮演着关键的调控角色,其功能涵盖了植物生长发育的各个方面,包括对外界逆境胁迫的响应[6]。大量转录因子,如WRKY[7]、MYB[8]、NAC[9]、bHLH[10]和bZIP[11],在植物生长发育及应对环境胁迫中的关键作用已经被广泛证实和报道。
生长调控因子(growth- regulating factors,GRFs)作为一个重要的转录因子基因家族,在植物生长发育和胁迫响应中发挥着重要作用[12]。GRF蛋白的N 端区域包含两个保守的基序,Gln-X3-Leu-X2-Gln(QLQ)和Trp-Arg-Cys(WRC)[13]。QLQ 对GRF和其他蛋白的相互作用至关重要,WRC含有核定位信号和DNA结合基序[14-15]。此外,有些GRF转录因子的C端区域还含有TQL、GGPL和FFD共3个保守结构域[15-17]。GRF 转录因子家族广泛存在于各种植物中,并且可能在不同植物种类之间具有一定的功能差异性,这为进一步研究其在植物生物学中的功能提供了重要线索[18-19]。自从水稻(Oryza sativa)OsGRF1 作为第一个成员被鉴定出在赤霉素(gibberellic acid,GA)诱导的茎伸长中发挥着重要的调控作用[15],后续的研究发现大量GRFs参与植物的生长发育调控[20-21]。AtGRF4 是拟南芥(Arabidopsis thaliana)叶细胞增殖、子叶胚胎发育和茎尖分生组织发育所必需的[22]。AtGRF7 可以抑制渗透胁迫响应基因的表达来防止植物在胁迫条件下的生长受影响[17]。OsGRF6通过生长素(auxin,IAA)途径调控花序构型,通过GA途径调控水稻植株高度[23-24]。生长调节因子互作因子(GRF-interacting factors,GIFs)是GRF 的转录共激活因子,其在植物发育中起重要作用,特别是在增强GRF 蛋白的活性方面[25]。GIF蛋白属于SSXT 基因超家族,发挥植物转录共激活因子的作用[26]。GIF 蛋白包含一个SNH(SYTN 末端同源性)结构域,该结构域介导与GRF的QLQ 结构域的相互作用形成功能性转录复合物。在这类复合物中,GIF 将SWI/SNF(Switch defective/sucrose non-fermentable)染色质重塑复合物招募到其靶基因,通过被GRF转录激活或抑制来调控植物的生长发育和再生效率[27-28]。GRFs 和GIFs转录因子在植物生长发育和应对环境胁迫中协同发挥作用已经被广泛报道[29-30]。小麦中的GRF4 和GIF1 蛋白结合生成嵌合体可以显著提高小麦和水稻的再生效率[31]。在柑橘(Citrus reticulata)和草莓(Fragaria ananassa)中的研究表明,不同组合的GRF-GIF 嵌合物均能在不同程度上提高愈伤组织的芽再生效率,显著增强再生能力[31]。与野生型相比,ZmGRF11-ZmGIF2 和ZmGRF2-ZmGIF3 的过表达加速了玉米(Zea mays)花序茎的生长[32]。橡胶(Hevea brasiliensis)中过表达HbGRF4-GIF1后可以显著提高细胞的再生效率[33]。西瓜(Citrullus lanatus)ClGRF4-GIF1 还被用于构建高效再生体系,并与CRISPR/Cas9 系统结合实现了基因编辑[34]。然而,GRF 和GIF 基因家族作为调控植物生长发育和环境胁迫应答的转录因子,尚未在板栗基因组中被鉴定,其生物学功能和作用机制也尚未被报道。

图1 板栗与其他物种GRF 和GIF 蛋白的系统发育树Fig.1 Phylogenetic tree of the members of the GRF and GIF proteins among Chinese chestnut and other species
A.板栗(Cm)、拟南芥(At)、水稻(Os)、毛果杨(Pt)、葡萄(Vv)的GRF 蛋白构建的系统发育树;B.板栗(Cm)、拟南芥(At)、番茄(Sl)、马铃薯(St)、毛果杨(Pt)、美洲黑杨(Pd)、红皮柳(SPU)、大豆(Gm)、水稻(Os)、玉米(Zm)的GIF 蛋白构建的系统发育树。
A.A phylogenetic tree constructed from GRF proteins of C. mollissima (Cm),A. thaliana (At), O. sativa (Os), P. trichocarpa (Pt), and V. vinifera(Vv);B.A phylogenetic tree constructed from GIF proteins of C.mollissima(Cm),A.thaliana(At),S.lycopersicum(Sl),S.tuberosum(St),P.trichocarpa(Pt),P.deltoides(Pd),S.sinopurpurea(SPU),G.max(Gm),O.sativa(Os),and Z.mays(Zm).
笔者在本研究中通过分析板栗的基因组,成功鉴定了GRF 和GIF 基因家族成员,并进行系统分析,包括系统发育关系、基因结构、蛋白保守基序、共线性和进化关系、染色体分布、复制类型,预测其启动子顺式元件以及与转录因子的相互作用。此外,还结合RNA-Seq数据和RT-qPCR实验,分析这些基因在不同组织的不同发育阶段中的表达模式,以及对冷冻、热休克、干旱胁迫和栗瘿蜂(Drycosmusk kuriphilus)侵染的响应,以期为进一步研究CmGRF和CmGIF的功能提供参考。
1 材料和方法
1.1 板栗GRFs和GIFs成员鉴定
从栗属基因组数据库(Castanea Genome Database:http://castaneadb.net/#/)下载板栗基因组数据,包括基因组文件(核苷酸序列)、蛋白质文件(氨基酸序列)、基因结构注释文件(General Feature Format Version 3,GFF3 file)。从Pfam 数据库中下载QLQ(PF08880)、WRC(PF08879)和SNH(PF05030)的隐马尔可夫模型(Hidden Markov Model,HMM)。利用HMMER 3.0 软件对板栗蛋白文件进行搜索,同时含有QLQ 和WRC 结构域的蛋白序列被认为是GRF 蛋白的候选成员,含有SNH结构域的蛋白序列被认为是GIF蛋白的候选成员。此外,利用已报道的拟南芥中的GRFs和GIFs的蛋白序列作为参考序列对板栗所有蛋白序列进行blast检索[35]。以上结果全部上传至NCBI-CDD进一步筛选,以确保GRF成员均含有QLQ和WRC两个结构域,GIF成员含有SNH结构域。基于最终确定的CmGRFs和CmGIFs在染色体上的相对位置被分别命名为CmGRF1~CmGRF10和CmGIF1~CmGIF3。CmGRFs 和CmGIFs 蛋白序列上传至Expasy(https://www.expasy.org/)获取CmGRFs和CmGIFs蛋白的理化性质数据。
1.2 系统发育分析
拟南芥GRFs 和GIFs 蛋白序列下载自TAIR 网站(http://www.arabidopsis.org)。水稻GRFs 和GIFs蛋白序列下载自MSU-RGAP 网站(http://rice.uga.edu/index.shtml)。其他物种的蛋白序列来自Phytozome v13(https://phytozome-next.jgi.doe.gov/)。利用MEGA 7.0 软件分别对GRFs 和GIFs 蛋白序列进行多序列比对,采用最大似然法来构建GRFs 和GIFs的系统发育树。
1.3 基因结构和保守结构域
CmGRFs 和CmGIFs 的蛋白序列被上传至NCBI-CDD 以获取含有保守结构域的hitdata 文件。CmGRFs 和CmGIFs 的蛋白序列被上传至MEME(https://meme-suite.org/meme/)以获取保守基序信息。CmGRFs 和CmGIFs 的基因结构信息(外显子-内含子)来源于板栗基因组的结构注释文件(General Feature Format Version 3,GFF3 file)。
1.4 启动子顺式作用原件
提取CmGRFs 和CmGIFs 基因起始密码子上游2000 bp 的序列,并利用PlantCARE 数据库(http://bioinformatics.psb.ugent.be/webtools/plantcare/html/)预测其中的顺式作用元件,对结果进行汇总整理,以确定顺式作用元件的种类、数量及位置,并利用TBtools软件进行可视化分析[36]。
1.5 染色体分布和共线性
CmGRFs 和CmGIFs 的染色体分布信息从板栗基因组的结构注释文件(GFF3 file)获取,之后利用TBtools 进行可视化分析。使用Multiple Collinearity Scan toolkit(MCScanX)来分析板栗基因组内部的共线性,以及板栗基因组与拟南芥、栎树(Quercus robur)、葡萄(Vitis vinifera)、水稻和玉米的共线性,并且通过板栗基因组内部的共线性分析获取CmGRFs和CmGIFs的复制类型[37]。
1.6 表达谱分析和热图绘制
从NCBI 数据库(https://www.ncbi.nlm.nih.gov/)中收集了公开可用的5 种板栗组织的不同发育时期,以及在3 种非生物胁迫(干旱、冷冻、热休克)和栗瘿蜂(Drycosmusk kuriphilus)侵染下的RNA-seq数据。这些数据来源于9 个项目,项目号分别为:PRJNA512447、PRJNA731244、PRJNA791965、PRJNA818064、PRJNA862256、PRJNA883560、PRJNA885276、PRJNA957852、PRJNA1166987[4,38-41]。使用Sratoolkit 3.0将RNA-seq文件从SRA格式转换为Fastq 格式[42]。使用Tophat2 将这些Fastq 文件映射到参考基因组以确定表达模式,参考基因组为来自Castanea 基因组数据库(http://castaneadb.net/#/)的N11-1[43]。使用TBtools 中的“scale method”对表达数据进行归一化处理,并基于对数转换[log2(FPKM+1)]值生成表示基因表达水平的热图[36]。
1.7 CmGRFs 和CmGIFs 与转录因子互作网络预测
使用Plant Transcriptional Regulatory Map(https://plantregmap.gao-lab.org/)对可能作用于CmGRFs 和CmGIFs 上游(2000 bp)区域的转录因子进行预测,并使用Cytoscape(v3.9.1)软件可视化分析转录因子与CmGRFs和CmGIFs的相互作用。
1.8 RT-qPCR分析
RT-qPCR试验材料采自河北科技师范学院板栗种植基地(N 40°06′,E 119°22′)长势一致、生长健壮的5 年生燕山早丰板栗,砧木为燕紫。分别在花后60、70、80、90和100 d采集燕山早丰板栗的坚果,每个时间点采集坚果后,立即用枝剪和手术刀去除刺苞和种皮,确保去除过程中种仁未受损伤。之后迅速将样品放入已经标记的50 mL 离心管中,并迅速置于液氮中直至无气泡产生,确保样品已被完全冷冻。最后将样品存储于-80 ℃超低温冰箱中,用于后续对CmGRFs 和CmGIFs 的RT-qPCR 实验。使用RNAprep Pure 多糖多酚植物总RNA 提取试剂盒(DP441,天根生化科技北京有限公司,北京)提取板栗种仁总RNA。使用PrimeScript 第一链cDNA 合成试剂盒(TaKaRa,日本)反转录合成单链cDNA。根据CmGRFs 和CmGIFs 的基因序列,使用Primer Premier 5 设计特异引物,以18S 为内参基因(表1)。使用TB Green Premix Ex Taq(TaKaRa)试剂盒在ABI 7500 qRT-PCR(Applied Biosystems,USA)上进行qRT-PCR 实验。采用2-△△CT方法计算CmGRFs 和CmGIFs的相对表达量。
表1 实时荧光定量PCR 引物
Table 1 Primers for real-time fluorescent quantitative PCR

基因名称Gene name CmGRF1 CmGRF2 CmGRF3 CmGRF4 CmGRF5 CmGRF6 CmGRF7 CmGRF8 CmGRF9 CmGRF10 CmGIF1 CmGIF2 CmGIF3 18S上游引物(5'→3')Forward primer(5'→3')GTGCAGCAGGGATGTGGT ACTTGCAGCCCAGTGGTG TCGGAGAAGAGCCTGGCT CTGGCTCACCCCTGCTTC AAACTGGCTCACCCCTGC TGCTGCCAGAAGGTGTCG GCTTCAGTGGTGGTGCCT ACGCGTACCCGGACTCTA CGGCTGCACTAACCCCAA TGGACGTGGGTCTGGACA CCCATCCACAGCCACCAG GACACTCGGGGTTGGTGG TTGGAGGTGGCAGCAGTG CGGCTACCACATCCAAGGAA下游引物(5'→3')Reverse primer(5'→3')GGAGGCTACCACACGCTT AGAGGCCCCGTGGATCTT TCAATGCCTCCGCTAGCG CGGCAGTTGGAGACGAGG CCGGCAGTTGGAGACGAG GGCGGCCTCTGTTCATGT GGTGGCTGCGGAAGGATT TCCCATTCCCACTGCTGC TGAAGGTGGCTGTGATGGG TGATGTGGCACGGGAAGC TGTTGCCTGGGGATGCTG TCTTGCTTGCTCCCACCG CCACTTCCATGCCCAGCA GCTGGAATTACCGCGGCT
2 结果与分析
2.1 CmGRFs和CmGIFs的鉴定和理化性质
在板栗基因组中共鉴定到10 个GRFs 基因,10个CmGRFs基因不均匀地分布在5、8、9、10、11和12号染色体上。CmGRFs 基因编码236 个到602 个氨基酸,分子质量介于25.78~64.97 ku。CmGRFs蛋白的不稳定系数均在40以上,并且亲水性指数均小于0,表明CmGRFs 属于不稳定的亲水性蛋白。CmGRFs 的等电点在7.74(CmGRF8)到9.41(CmGRF10)之间,表明CmGRFs均为碱性蛋白。亚细胞定位预测显示,10个CmGRFs蛋白均定位于细胞核(表2)。3 个CmGRFs 基因分别位于4、5 和10条染色体上。在板栗基因组中共鉴定到3 个GIFs基因。CmGIFs 蛋白的长度和分子质量分别介于216到223个氨基酸,23.44~24.55 ku。CmGIFs蛋白的不稳定指数和亲水性指数分别介于60.84~80.47和-1.159~-0.647,这说明CmGIFs 是不稳定的亲水性蛋白。CmGIF2 和CmGIF3 蛋白等电点为5.93,CmGIF1 蛋白的等电点为6.1,这表明CmGIFs 是酸性蛋白。亚细胞定位预测显示,3个CmGIFs蛋白定位于细胞核(表2)。
表2 CmGRF 和CmGIF 转录因子家族理化特征
Table 2 Physicochemical properties of CmGRF 和CmGIF transcription factor families
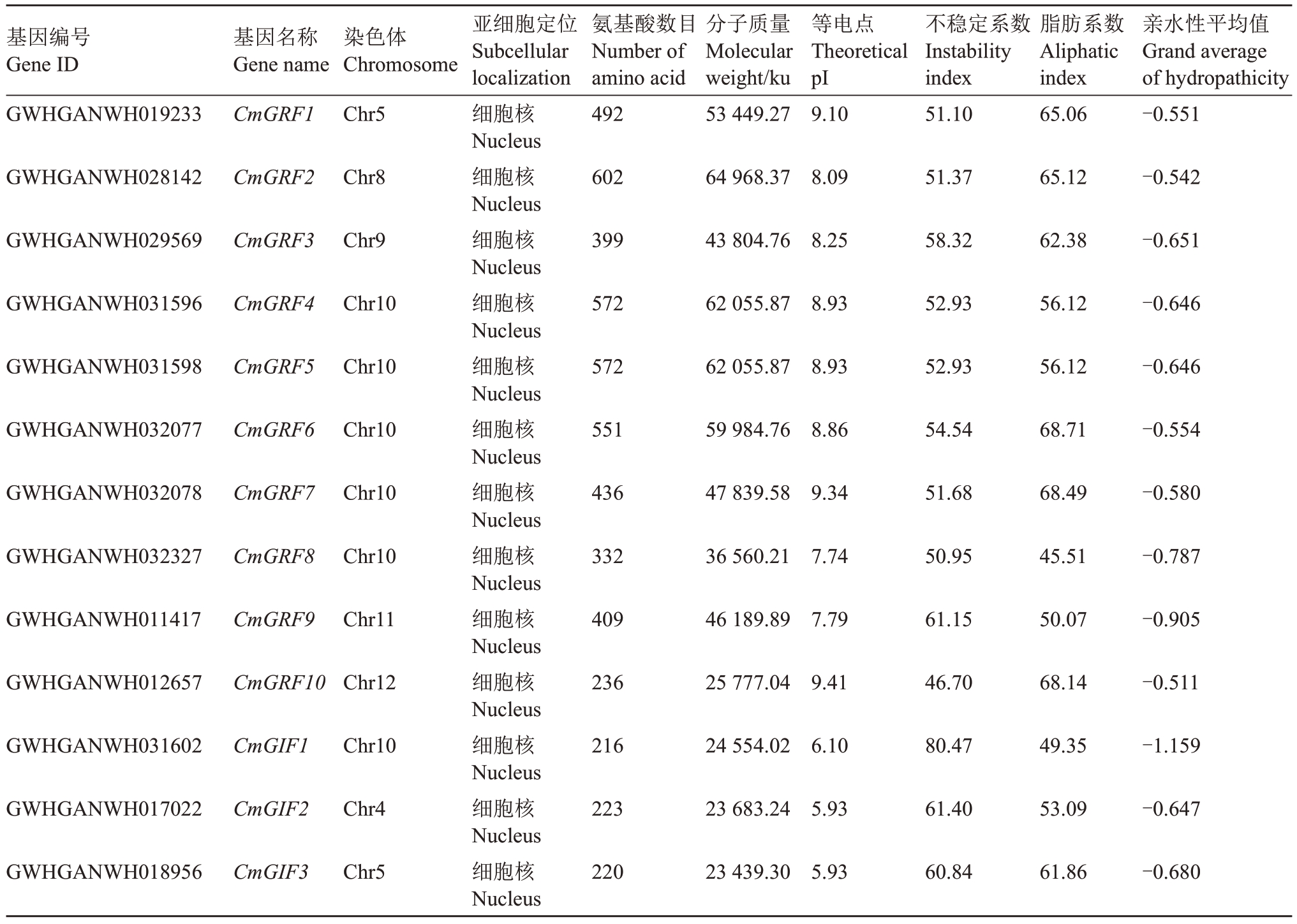
基因编号Gene ID GWHGANWH019233基因名称Gene name CmGRF1染色体Chromosome Chr5氨基酸数目Number of amino acid 492分子质量Molecular weight/ku 53 449.27等电点Theoretical pI 9.10不稳定系数Instability index 51.10脂肪系数Aliphatic index 65.06亲水性平均值Grand average of hydropathicity-0.551 GWHGANWH028142CmGRF2Chr860264 968.378.0951.3765.12-0.542 GWHGANWH029569CmGRF3Chr939943 804.768.2558.3262.38-0.651 GWHGANWH031596CmGRF4Chr1057262 055.878.9352.9356.12-0.646 GWHGANWH031598CmGRF5Chr1057262 055.878.9352.9356.12-0.646 GWHGANWH032077CmGRF6Chr1055159 984.768.8654.5468.71-0.554 GWHGANWH032078CmGRF7Chr1043647 839.589.3451.6868.49-0.580 GWHGANWH032327CmGRF8Chr1033236 560.217.7450.9545.51-0.787 GWHGANWH011417CmGRF9Chr1140946 189.897.7961.1550.07-0.905 GWHGANWH012657CmGRF10Chr1223625 777.049.4146.7068.14-0.511 GWHGANWH031602CmGIF1Chr1021624 554.026.1080.4749.35-1.159 GWHGANWH017022CmGIF2Chr422323 683.245.9361.4053.09-0.647 GWHGANWH018956CmGIF3Chr5亚细胞定位Subcellular localization细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus 22023 439.305.9360.8461.86-0.680
2.2 CmGRFs和CmGIFs的系统发育分析
基于系统发育树,来自板栗(10)、拟南芥(9)、水稻(12)、毛果杨(Populus trichocarpa)(19)和葡萄(8)的共58个GRF蛋白被分为4个分支(Group1~Group4)(图1-A)。在Group3(CmGRF2、CmGRF6、CmGRF7)和Group4(CmGRF3、CmGRF8、CmGRF9)中分别有3个CmGRFs 蛋白。Group1(CmGRF4 和CmGRF5)和Group2(CmGRF1 和CmGRF10)分支分别包括2 个CmGRF成员。Group1仅包含双子叶植物的GRF成员,其余3个分支均包含双子叶和单子叶植物的GRF蛋白,表明GRF蛋白可能在双子叶和单子叶植物分化后形成了新的进化分支。
基于系统发育树,来自板栗(3)、拟南芥(3)、番茄(Solanum lycopersicum)(4)、马铃薯(Solanum tuberosum)(4)、毛果杨(4)、美洲黑杨(Populus deltoides)(6)、红皮柳(Salix sinopurpurea)(4)、大豆(Glycine max)(8)、水稻(3)和玉米(3)的共42 个GIF 蛋白被分为3 个分支,Ⅲ分支含有2 个CmGIF 成员(CmGIF1 和CmGIF3),Ⅱ分支仅含有CmGIF2。Ⅰ分支中只含有拟南芥(AtGIF2和AtGIF3)、美洲黑杨(PdGIF2和PdGIF3)和大豆(GmGIF4),没有CmGIF成员(图1-B)。
2.3 CmGRFs 和CmGIFs 的基因结构及其编码蛋白的保守基序
每个CmGRF 包含2~9 个蛋白保守基序,并且位于同一分支中的CmGRF成员具有相似的基序组成(图2-A)。例如,一些基序则仅分布在特定亚家族中,如基序10 仅存在于Group 4 分支的CmGRFs成员。基序6、7、8、9仅存在于Group 1分支。此外,所有CmGRFs 都包含基序1 和基序2(图2-A)。总体来说,基序组成特征支持CmGRFs 的系统发育关系。3个CmGIF蛋白均只包含一个基序(图2-C)。
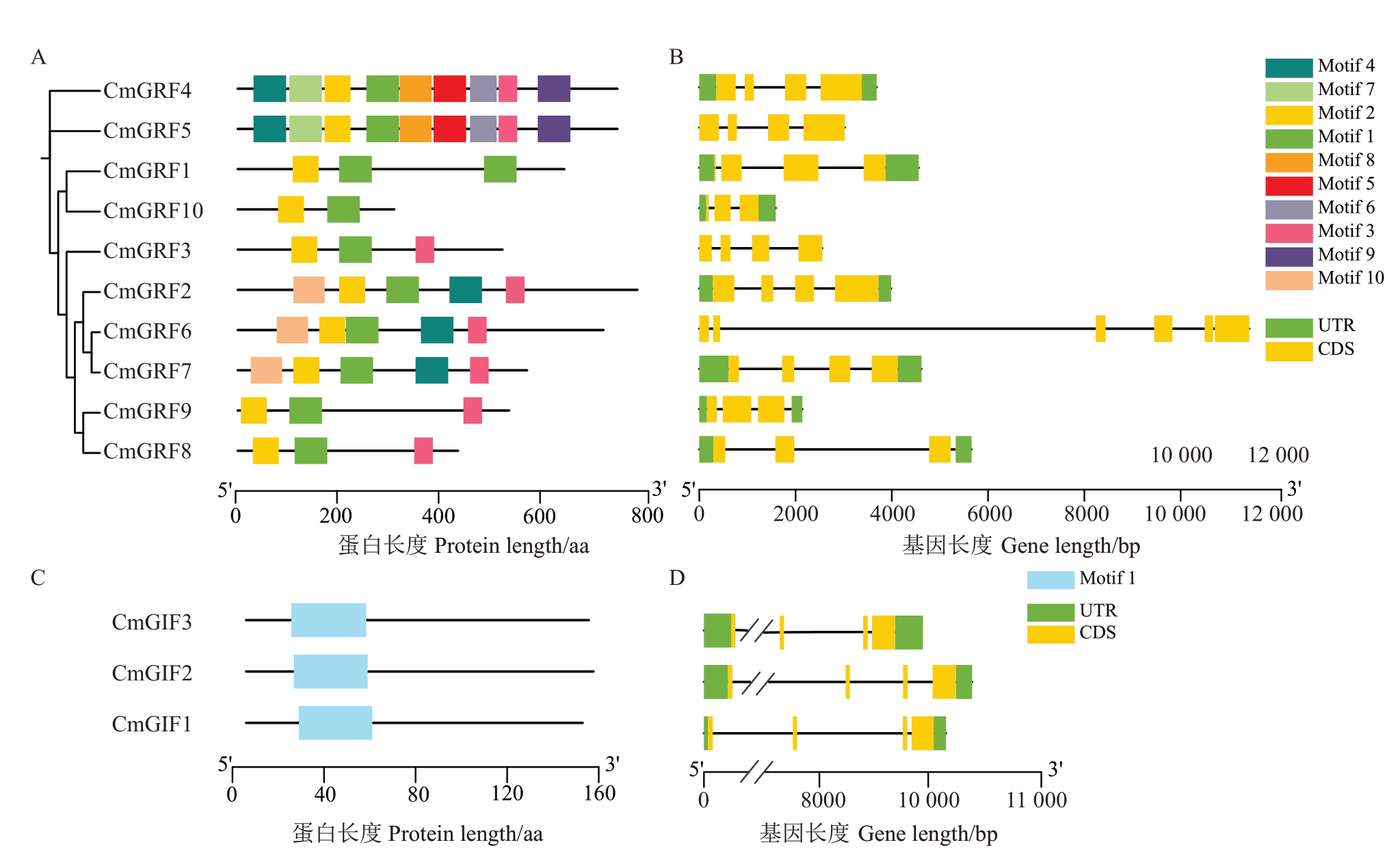
图2 板栗GRF 和GIF 的基因结构及其编码蛋白保守基序
Fig.2 Gene structures of Castanea mollissima GRF and GIF and the conserved motifs of their coding protein
A.CmGRF 蛋白的保守基序;B.CmGRF 基因的结构;C.CmGIF 蛋白的保守基序;D.CmGIF 基因的结构。
A.Conserved motifs of CmGRF proteins;B.Structure of CmGRF genes;C.Conserved motifs of CmGIF proteins;D.Structure of CmGIF genes.
与基序组成类似,同一分支中的CmGRFs 成员具有相似的基因结构(图2-B)。CmGRFs 基因结构显示,板栗GRF家族成员基因包含2~5个内含子,相应的包含3~6个外显子。CmGRF6含有6个外显子,CmGRF10 含有3 个外显子,其他CmGRF 成员均含有4 个外显子,同一分支中的外显子和内含子的数量几乎相同。此外,3 个CmGIFs 均只含有3 个内含子和4个外显子(图2-D)。
2.4 CmGRFs 和CmGIFs 的顺式作用元件及互作转录因子预测
启动子区域中的顺式作用元件对基因的转录和功能表达至关重要[44]。分析所有CmGRFs 和Cm-GIFs起始密码子上游2000 bp的序列,以研究其顺式作用元件。CmGRFs启动子区域中鉴定到的顺式作用元件主要分为4类:与光响应有关,与环境胁迫有关,与发育有关,与植物激素有关(图3-A)。光响应元件主要包括Box-4、G-Box、GT1-motif、TCT-motif、MRE、AE-box。鉴定到大量与生长发育密切相关的激素相关的作用元件,如ABRE 与脱落酸有关;AuxRR-core 响应生长素;CGTCA-motif 和TGACGmotif 参与MeJA 反应;GARE-motif、P-box 和TATCbox 与赤霉素反应有关;TCA-element 和TGA-element分别为水杨酸响应元件、生长素反应元件。此外,一些参与防御和应激反应元件,如ARE(与抗氧反应有关)、LTR(与冷胁迫有关)、MBS(与干旱有关)、MBSI(与类黄酮形成有关)也大量存在于CmGRFs的启动子区域中。其中CmGRF1启动子区域中存在最多的顺式作用元件,而CmGRF8 启动子区域含有最少的顺式作用元件。这些结果表明CmGRFs在板栗生长发育和应对环境胁迫方面发挥重要作用,这与后续RNA-seq 数据分析结果一致。类似的,也预测CmGIFs的启动子区域的顺式作用元件(图3-B)。CmGIFs的顺式作用元件也主要分为4类,分别是与光响应有关,与环境胁迫有关,与发育有关,与植物激素有关。在CmGRFs 和CmGIFs 的启动子区域中鉴定到相似种类的顺式作用元件,表明其可能与相似的顺式作用元件存在相互作用。
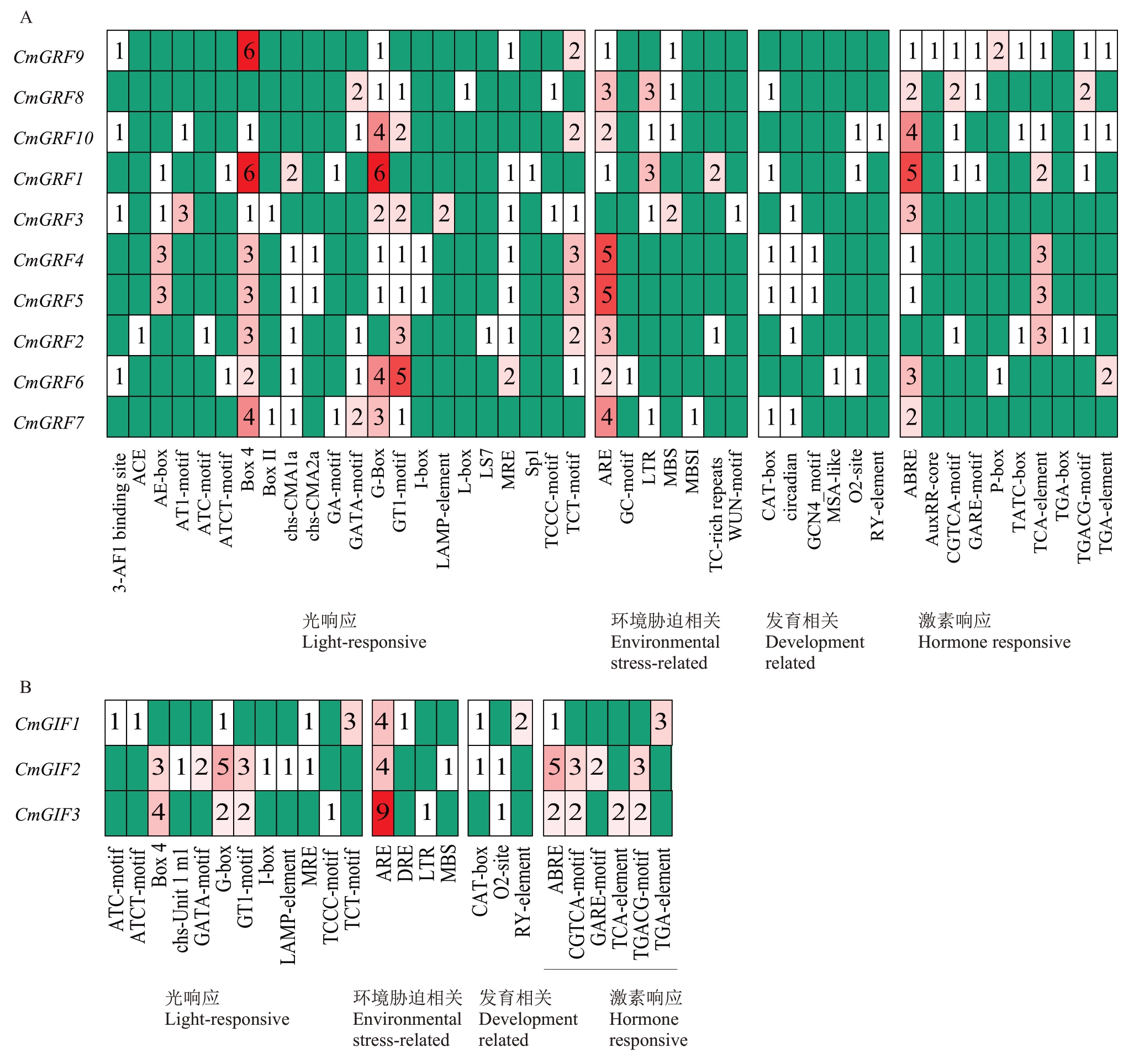
图3 板栗GRF 和GIF 家族启动子顺式作用元件
Fig.3 Cis-elements of the promoters Chinese chestnut GRF and GIF gene families
A.CmGRFs 的顺式作用元件;B.CmGIFs 的顺式作用元件。A.Cis-elements of CmGRFs;B.Cis-elements of CmGIFs.
对可能作用于CmGRFs和CmGIFs上游2000 bp区域的转录因子进行预测(图4),共有126个转录因子可能作用于CmGRFs,属于34 个转录因子家族(图4-A)。在这些转录因子家族中,数量最多的家族是MYB(19),随后是NAC(11),C2H2(10),Dof(9)和ERF(9);数量最少的家族只有1 个成员,如ARF、GRAS、HD-ZIP、SRS、Trihelix和WOX。此外,CmGRF1启动子区域可以被37个转录因子识别,其次是CmGRF7(35)、CmGRF6(30)和CmGRF9(29)。类似的,共预测到有199 个转录因子可能作用于CmGIFs,属于22个转录因子家族(图4-B)。数量最多的家族是MYB(9),其次是Dof/bHLH(9)和C2H2(7);数量最少的家族只有一个成员,如GRAS、HD-ZIP、NAC、WRKY 和bZIP。预测到Cm-GIF1和CmGIF2与28个转录因子存在相互作用,而14 个转录因子可以作用于CmGIF3。总体而言,作用于CmGRFs 和CmGIFs 最多的5 个基因家族分别是MYB、Dof、bHLH、C2H2和TCP,表明其可能在调节CmGRFs和CmGIFs的表达中发挥重要作用。

图4 CmGRFs 和CmGIFs 的转录因子调控网络分析
Fig.4 Transcription factor regulatory network analysis of CmGRFs and CmGIFs
A.CmGRFs 的转录因子调控网络分析;B.CmGIFs 的转录因子调控网络分析。
A.Transcription factor regulatory network analysis of CmGRFs;B.Transcription factor regulatory network analysis of CmGIFs.
2.5 CmGRFs 和CmGIFs 的染色体分布和共线性分析
可视化分析CmGRFs 和CmGIFs 在染色体中的分布(图5-A),CmGRFs 位于6 条染色体上,其中Chr10 含有最多的CmGRFs(CmGRF4、CmGRF5、CmGRF6、CmGRF7和CmGRF8),另外5个CmGRFs分别位于Chr5(CmGRF1)、Chr8(CmGRF2)、Chr9(CmGRF3)、Chr11(CmGRF9)和Chr12(CmGRF10)上。3 个CmGIFs 均匀分布在3 条染色体上,Cm-GIF1 位于Chr10,CmGIF2 位于Chr4,CmGIF3 位于Chr5 上(图5-A)。这些结果表明CmGRFs 和Cm-GIFs分布在板栗的不同染色体上,并且分布在每条染色体上的基因数量不同。

图5 CmGRFs 和 CmGIFs 的染色体分布及共线性分析
Fig. 5 Chromosomal distribution and collinear analysis of CmGRFs and CmGIFs
板栗基因组内的共线性分析发现两个CmGRF基因对(CmGRF2/CmGRF6、CmGRF1/CmGRF10)分别在Chr8/Chr10 和Chr5/Chr12 上形成两个节段重复。1 个CmGIF 基因对(CmGIF1/CmGIF3)在Chr5和Chr10 上形成一个节段重复(图5-B)。将板栗基因组与代表性双子叶植物(拟南芥、葡萄)、单子叶植物(水稻、玉米)以及同科植物(栎树)成员之间的共线性关系进行分析(图5-C)。对于CmGRFs,板栗与5个物种(拟南芥、栎树、葡萄、水稻、玉米)之间的基因对数量分别为8、4、9、5和1,这表明CmGRFs与葡萄和拟南芥的亲缘关系更为密切。板栗与本研究中的双子叶植物之间具有更多的共线同源基因对,这表明CmGRFs可能在双子叶植物中发生过额外的复制事件。此外,一些CmGRFs 与其他物种的基因形成两个直系同源基因对,如CmGRF2 与拟南芥中的AT2G36400、AT3G52910,以及水稻中的Os03g51970和Os03g47140。这些结果表明,在进化过程中CmGRF2在拟南芥和水稻中的直系同源基因保留有更多的拷贝数。对于CmGIFs,板栗与拟南芥、栎树和葡萄之间的基因对数量分别为2、2、6,与水稻和玉米之间无共线基因对。这一结果可能表明,在双子叶植物与单子叶植物分化后,GIF 基因家族可能经历了独立的复制事件。
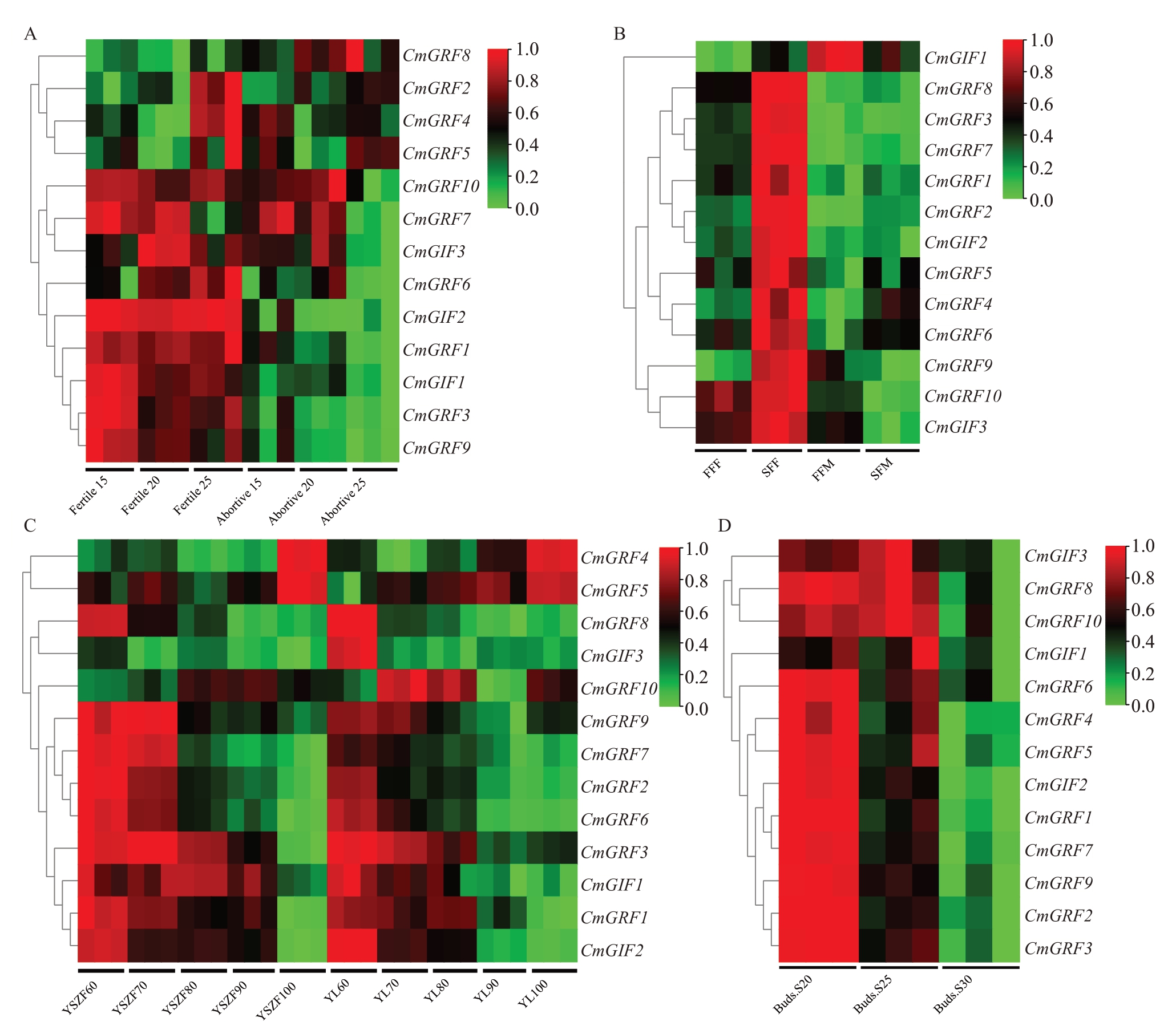
图6 CmGRFs 和CmGIFs 在不同组织中的表达Fig.6 Expression of CmGRFs and CmGIFs in different tissues of C.mollissima
A.CmGRFs 和CmGIFs 在可育(Fertile)胚珠和败育(abortive)胚珠3 个发育阶段的表达(7 月15 日、7 月20 日和7 月25 日);B.CmGRFs和CmGIFs 在雄花和雌花中的表达(FFF:雌花一次花,SFF:雌花二次花,FFM:雄花一次花,SFM:雄花二次花);C.CmGRFs 和CmGIFs 在燕山早丰(YSZF)和燕龙(YL)板栗种仁5 个发育阶段的表达(花后60、70、80、90、100 d);D.CmGRFs 和CmGIFs 在芽(Buds)3 个发育阶段的表达(花后20、25、30 d)。
A.Expression of CmGRFs and CmGIFs at three developmental stages(July 15,July 20,and July 25)of fertile and abortive ovules;B.Expression of CmGRFs and CmGIFs in male and female flowers (FFF:First flowering (female), SFF:Secondary flowering (female), FFM:First flowering(male),SFM:Secondary flowering(male);C.Expression of CmGRFs and CmGIFs at five developmental stages(60,70,80,90,100 days after flowering) of C. mollissima kernels of Yanshanzaofeng and Yanlong; D. Expression of CmGRFs and CmGIFs at three developmental stages (20, 25, and 30 days after flowering)of buds.
2.6 CmGRFs 和CmGIFs 在板栗不同组织中的表达
分析CmGRFs 和CmGIFs 在不同组织中的表达谱,包括胚珠(可育胚珠和败育胚珠的3 个发育时期)、雄花(一次花和二次花)、雌花(一次花和二次花)、种仁(两个品种的5 个发育时期)、芽(3 个发育时期)(图6)。CmGRF1 在可育胚珠中的表达水平显著高于败育胚珠,表明其可能与胚珠的育性有关(图6-A)[40]。CmGRF3 在雌花中持续高表达(FPKM>20),而在雄花中几乎不表达(FPKM<1)(图6-B),暗示了CmGRF3 可能与雌雄花分化或发育有关[45]。这表明其可能与板栗种仁发育关系密切[41]。有趣的是,CmGRF2 在花芽发育的早期阶段(花后20 d)表现出较高的表达水平(FPKM>60),然后在花芽发育的后期阶段(花后30 d)显著下调(FPKM<10)(图6-D)。总体而言,CmGRFs 在板栗的不同组织或同一组织的不同发育阶段中表现出不同的表达水平。类似的,CmGIF2 在可育胚珠中的表达显著高于败育胚珠(图6-A)[40],CmGIF3在板栗的雌花和雄花中持续高表达(图6-B)[45]。CmGIF3 在燕山早丰和燕龙种仁的5 个发育时期中持续高表达,而CmGIF1 在种仁中表达水平较低(FPKM<5)(图6-C),这暗示了在板栗种仁发育中的功能差异[41]。CmGIF3 在花发育的早期阶段(花后20 d)到中期阶段(花后25 d)的表达水平逐渐升高,然后在花芽发育的后期阶段(花后30 d)表达缓慢下调(FPKM<10)(图6-D)。总体来看,CmGRFs和CmGIFs的表达存在组织特异性,表明了其可能参与板栗不同组织的生物学过程。
2.7 CmGRFs和CmGIFs在不同胁迫下的表达
分析CmGRFs和CmGIFs在冷冻、热休克、干旱胁迫,以及在栗瘿蜂侵染下的表达谱,以探讨其在板栗应对环境胁迫中的潜在功能(图7)。结果表明,CmGRF3的表达水平在热休克处理4 h后显著上调,CmGRF2 在冷冻胁迫下的表达持续升高,而CmGRF4在冷冻和热休克处理下均几乎不表达(FPKM<2)[4]。其余CmGRFs 的表达水平在冷冻和热休克胁迫下均显著下调(图7-A)[4]。在不同程度干旱胁迫下,燕山早丰和大板红板栗中的CmGRFs 表达几乎无差异(FPKM<2)(图7-B)。栗瘿蜂是影响板栗生产的主要害虫之一,幼虫会危害芽和叶片,导致形成各种虫瘿[38]。与对照相比,CmGRF2 在栗瘿蜂形成虫瘿的初期阶段(4月7日)表达水平较高,随后显著下调(图7-C)[38]。此外,在栗瘿蜂形成虫瘿的初期阶段,10个CmGRFs在Hongli(HL:易受栗瘿蜂侵染)中的表达明显高于Shuhe_Wuyingli(SW:对栗瘿蜂侵染有部分抗性)的表达(图7-D)[39]。值得注意的是,CmGRF2、CmGRF3和CmGRF7在栗瘿蜂侵染的叶片中的表达显著高于在虫瘿中的表达(图7-E)。
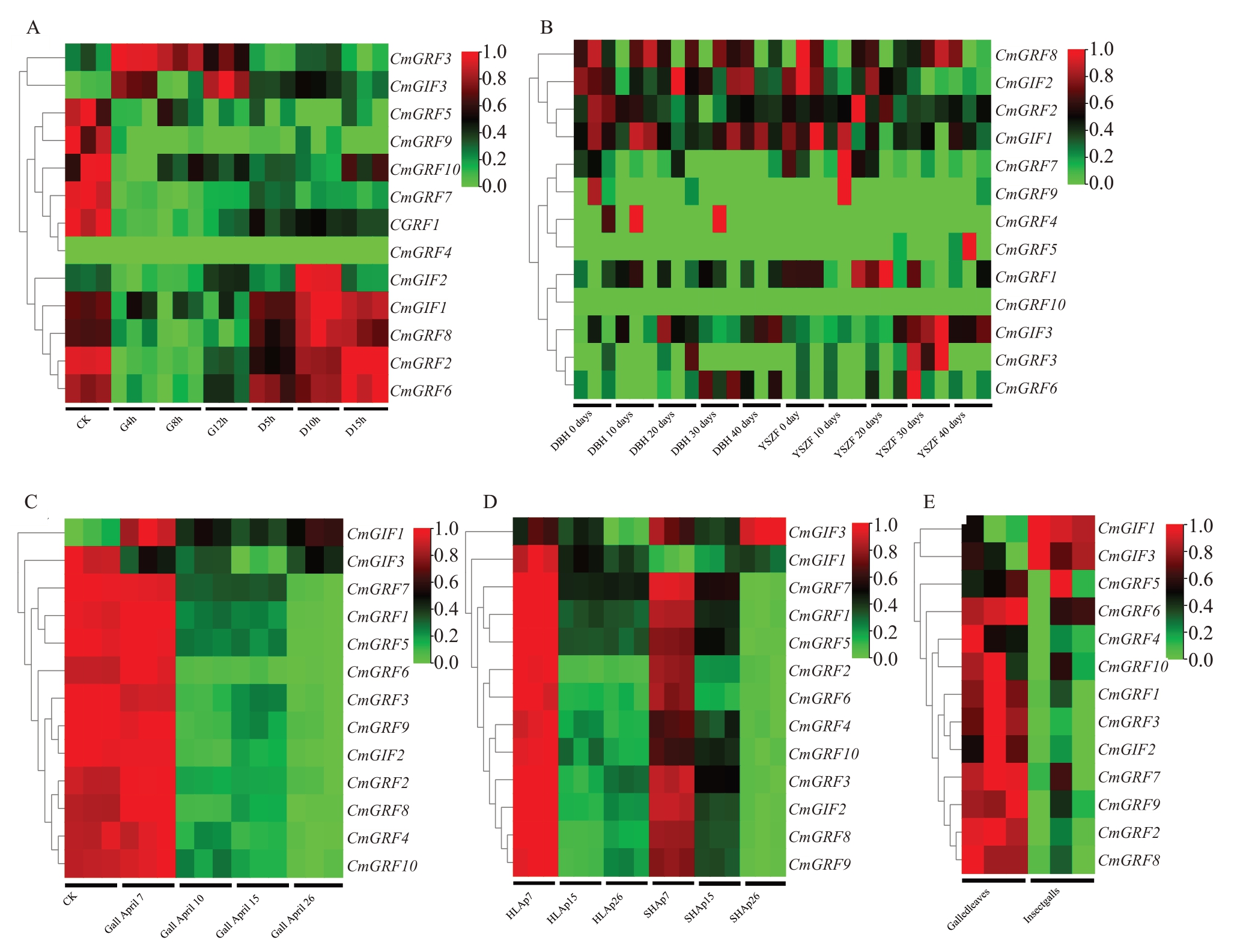
图7 CmGRFs 和CmGIFs 在不同胁迫下的表达
Fig.7 Expression of CmGRFs and CmGIFs in C.mollissima under different stresses
A.CmGRFs 和CmGIFs 在不同冷冻(-15 ℃)和热休克(45 ℃)胁迫时间的表达;CK 为对照,D5h、D10h 和D15h 分别为冷冻(-15 ℃)5、10和15 h,G4h、G8h 和G12h 分别为热休克(45 ℃)4、8 和12 h;B.CmGRFs 和CmGIFs 在干旱胁迫0、10、20、30、40 d 的大板红(DBH)和燕山早丰(YSZF)中的表达;C.CmGRFs 和CmGIFs 在虫瘿(Gail)(由板栗叶片遭受栗瘿蜂浸染形成)不同阶段(4 月7 日、4 月10 日、4 月15 日、4 月26 日)的表达;D.CmGRFs 和CmGIFs 在Hongli(HL:易受栗瘿蜂侵染)和Shuhe_Wuyingli(SW:对栗瘿蜂侵染有部分抗性)遭受栗瘿蜂侵染叶片不同阶段(4 月7 日、4 月15 日、4 月26 日)中的表达;E.CmGRFs 和CmGIFs 在栗瘿蜂侵染的叶片(Galledleaves)和诱导形成虫瘿(Insectgalls)的表达。
A. Expression of CmGRFs and CmGIFs at different freezing (-15 ℃) and heat shock (45 ℃) stress times. CK serves as the control, D5h, D10h,and D15h are frozen (-15 ℃) for 5, 10, and 15 hours, respectively, while G4h, G8h, and G12h are subjected to heat shock (45 ℃) for 4, 8, and 12 hours, respectively; B. Expression of CmGRFs and CmGIFs in Dabanghong (DBH) and Yanshanzaofeng (YSZF) under drought stress for 0, 10, 20,30, and 40 days; C. Expression of CmGRFs and CmGIFs in different stages (April 7th,April 10th,April 15th,April 26th) of Chinese chestnut galls(Gail:Formed by Chinese chestnut leaves being infected by chestnut gall wasps); D. Expression of CmGRFs and CmGIFs in the leaves of Hongli(HL:Susceptible to Drycosmusk kuriphilus infection) and Shuhe-Wuyingli (SH:Partially resistant to Drycosmusk kuriphilus infection) at different stages(April 7th,April 15th,April 26th)of chestnut gall wasp infection;E.Expression of CmGRFs and CmGIFs in leaves(Galledleaves)and insect galls(Insectgalls)induced by Drycosmusk kuriphilus.
CmGIFs 的表达在上述几种环境胁迫下表现出不同的趋势。例如,CmGIF3 在冷冻胁迫下的表达显著上调,而在热休克处理4 h到12 h内,表达水平先上升后下降(图7-A)[4]。CmGIF3在燕山早丰和大板红板栗的表达随着干旱胁迫的持续而显著上调(图7-B)。与对照相比,CmGIF1在栗瘿蜂侵染初期阶段(4 月7 日)形成虫瘿中的表达显著上调(图7-C)[38]。此外,在栗瘿蜂形成虫瘿的初期阶段,Cm-GIF1在Hongli(HL:易受栗瘿蜂侵染)中的表达显著高于Shuhe_Wuyingli(SW:对栗瘿蜂侵染有部分抗性)(图7-D)[39]。另外,CmGIF1 在虫瘿中的表达水平显著高于在栗瘿蜂侵染的叶片中(图7-E)。
2.8 CmGRFs和CmGIFs的RT-qPCR分析
通过RT-qPCR 实验验证CmGRFs 和CmGIFs 在板栗种仁不同发育时期的表达水平(图8)。从结果来看,CmGRF1/2/6/7/8 的表达水平随板栗种仁的发育而下调,并在花后100 d达到最小值。CmGRF3/9/10的表达水平在板栗种仁发育过程中呈先升高后降低的趋势。其中,CmGRF3/9在花后70 d达到峰值后开始逐渐下调,CmGRF10 在花后80 d 达到峰值。CmGRF4/5 在种仁发育过程的表达变化相似,都在花后100 d 达到峰值。此外,CmGIF2 和CmGIF3 的表达水平在种仁发育过程中逐渐下调,而CmGIF1的表达水平没有显著变化。总的来说,RT-qPCR 实验结果与RNA-seq 分析结果具有相似的变化趋势,验证了RNA-seq数据的可靠度。
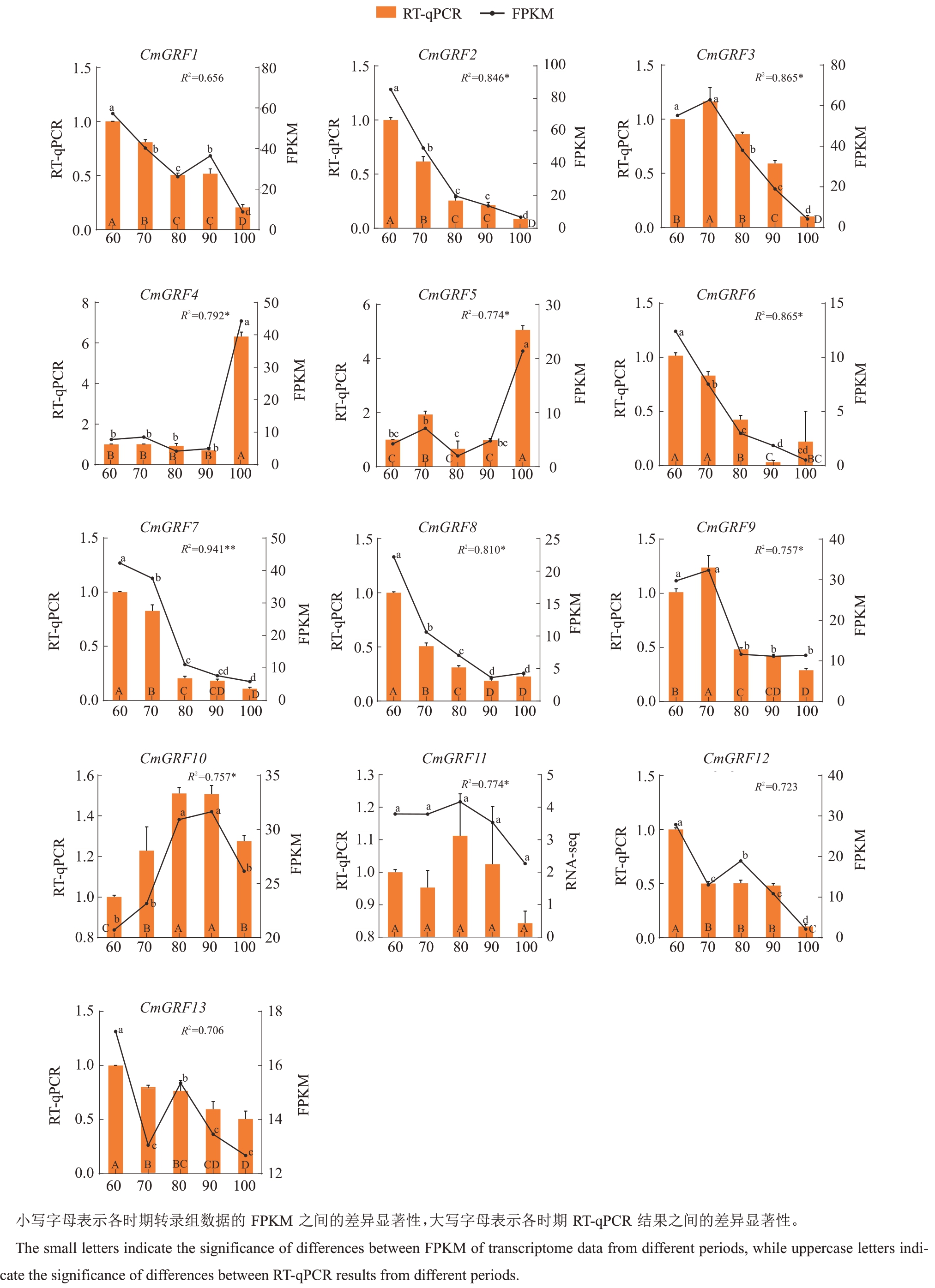
图8 CmGRFs 和CmGIFs 在花后60、70、80、90 和100 d 的表达分析
Fig.8 Expression analysis of CmGRFs and CmGIFs at 60,70,80,90,and 100 days after flowering
3 讨 论
GRF 是植物特异性转录因子家族,在植物生长和发育中起关键作用,包括细胞增殖、器官发生和胁迫反应[46-47]。GRF 基因家族已在众多植物中被鉴定和系统表征,如拟南芥[48]、水稻[16]、大豆[49]和蒺藜苜蓿(Medicago truncatula)[50]。GIF 充当转录共激活因子,可与GRF形成蛋白复合物共同发挥作用调控植物的生长发育和响应环境胁迫[47]。然而,GRF 和GIF尚未在板栗中被鉴定和系统表征。
笔者在本研究中发现,板栗基因组中有10 个CmGRFs 和3 个CmGIFs 成员。这些成员符合GRF和GIF 基因家族的基本特征,所有CmGRFs 编码的蛋白均包含WRC和QLQ结构域,所有CmGIFs编码的蛋白均包含SNH结构域。SNH结构域与QLQ结构域结合形成GRF-GIF复合体,共同作用于下游靶基因,这是CmGRFs 和CmGIFs 相互作用影响基因表达的分子基础[29]。系统发育分析将10个CmGRFs分成4 个分支,所有CmGRFs 均与双子叶植物的GRFs基因相距较近,而与水稻GRFs相距较远,这体现了双子叶植物中GRFs具有更近的遗传距离。高度相似的序列和结构往往意味着发挥相似的功能[51]。Group 1分支中的AtGRF9过表达会限制拟南芥早期发育叶片中的细胞增殖[52],而AtGRF7作为多种渗透应激反应基因的抑制因子发挥作用,以防止生长抑制[17]。CmGRF4/5 与AtGRF7/9 位于同一分支,并且具有相似的序列信息,这表明CmGRF4/5可能通过增强细胞活性来促进生长发育。Group 2 中AtGRF4 可以抑制拟南芥THALIANA2(KNAT2)启动子活性[21],OsGRF1/10在茎尖分生组织、叶原基以及刚出现的叶中有较高表达,在表皮以及节间分生组织的维管束周围有较高表达[16],OsGRF6 是水稻生长和耐冷性之间平衡的重要调节因子[53]。因此,位于Group 2中的CmGRF1/10可能在幼嫩组织及冷胁迫上发挥重要作用。Group 3 中AtGRF1 的过表达导致叶片增大[48],以及花序茎的延迟抽薹,OsGRF2在成熟植株的伸长叶片中有较高表达[16],OsGRF7主要在叶片、节间、腋芽和幼嫩的花序中表达[54]。位于Group 3 中的CmGRF3/8/9 可能与花发育有关。类似的,系统发育分析将3个CmGIFs分到3个分支中。OsGIF1 在营养和生殖发育过程中起重要作用[55],AtGRF1 和AtGIF1 分别充当转录激活因子和共激活因子,并且是参与调节叶片和花瓣生长的复合物的一部分[14]。因此,CmGIF2 可能在板栗的生殖和营养生长上也发挥相似功能。
基因复制不仅有助于基因家族的扩张,还对基因的功能分化以及植物对环境变化的适应具有重要的意义[56-58]。串联重复和节段重复被认为是基因家族扩张的主要复制模式[59]。由于全基因组加倍(whole-genome duplication,WGD)在植物基因组中普遍存在,并且WGD 后通常伴随着大量染色体重排,这导致了节段重复的频繁发生[60]。复制类型分析发现10个CmGRFs中的4个,以及3个CmGIFs中的2 个是通过节段复制形成的,这表明节段复制是CmGRF和CmGIF家族的主要驱动力。启动子区域的顺式作用元件通过与转录因子相互作用在调控植物基因表达方面发挥重要作用[61]。在预测到的CmGRFs 和CmGIFs 的启动子区域顺式作用元件中,ARE 最丰富,其次是G-box 和ABRE。此外,一些重复基因的顺式作用元件的数量和类型存在差异。例如,CmGRF1 启动子区域存在3 个温度胁迫响应(LTR),而其节段复制基因CmGRF10 仅有1个;CmGRF10 的启动子区域有1 个干旱响应元件(MBS),而其节段复制基因CmGRF1则没有。这表明节段复制基因启动子区域的顺式作用元件并不一致,可能导致了在不同发育阶段和不同环境胁迫下基因表达的特异性调控。在CmGRFs和CmGIFs的启动子区域还预测到大量其他顺式作用元件,主要涉及生长发育、胁迫响应以及激素响应,如Box-4、G-box、ARE、LTR、ABRE元件等。这些结果暗示了CmGRFs 和CmGIFs 在板栗生长发育和环境胁迫响应方面的多样化功能。笔者在本研究中预测到可能作用于上述顺式作用元件的126 个转录因子,涉及34 个转录因子家族,数量最多的5 个转录因子家族是MYB、Dof、bHLH、C2H2和TCP。MYB是植物中最大的转录因子家族之一,在植物生长发育与响应环境胁迫中扮演重要角色[61-62]。拟南芥中的At-MYB96被干旱胁迫显著诱导,而过表达该基因可以显著提高拟南芥的耐旱性[63]。苹果中的MdMYB23在冷胁迫下显著诱导,其过表达增强了转基因苹果愈伤组织和拟南芥植物的耐寒性[64]。其余预测到的转录因子家族,如Dof、bHLH、C2H2和TCP,也被广泛报道参与调控植物的生长发育和环境胁迫响应[65-68]。然而,就本研究结果而言,预测到的转录因子是否作用于顺式作用元件,以及是否调控CmGRFs 和CmGIFs 的表达来影响板栗的生长发育和对环境胁迫的响应,仍需进一步研究。
大量研究报道了GRFs和GIFs通常在发生细胞增殖的生长区域中高表达,如发芽的种子、愈伤组织和芽[17,69-71]。CmGRFs和CmGIFs在板栗各组织中具有不同的表达模式,这些组织特异性的表达模式表明CmGRFs 和CmGIFs 可能参与组织特异性发育和信号转导。一些CmGRFs成员在组织发育早期表现出比发育后期更高的表达水平,如CmGRF2 在花芽发育的早期表达水平显著高于花发育晚期。实际上,GRF基因在活跃生长组织中的表达水平显著高于成熟组织,并且GRF转录水平随着植物衰老而降低[72-73]。AtGRF 的表达水平随着拟南芥的生长发育而降低[72]。水稻中的GRF基因在芽和未成熟叶片中具有较高的表达水平[73]。CmGRF3在板栗雌花中高表达,而在雄花中几乎不表达(FPKM<1),暗示了其可能与雌雄花分化或发育有关。大量研究报道了GRF 和GIF 调控网络对花器官发育的重要作用[74-75]。水稻中OsGRF6 的反义抑制或OsmiR396d(靶向OsGRF6转录产物)的超表达会导致花器官发育畸形[76]。拟南芥gif1/2/3 三突变体植株的花器官严重发育不良,表现为雌蕊分裂、胚囊缺失和花药发育不全[35]。GRF和GIF在植物响应环境胁迫中同样发挥重要作用[72,77]。拟南芥中敲除AtGRF7 显著增强了植株的耐盐和抗旱性[78]。本研究中CmGRFs在两个板栗品种干旱胁迫中表达水平几乎无变化,而CmGIF3则在两种板栗干旱胁迫下均显著上调表达,这暗示了CmGIF3可能未通过与CmGRFs协同响应干旱胁迫。模式植物的研究中已经证明了一些GRF和GIF在响应环境胁迫中的作用。例如,在渗透胁迫下,AtGRF7蛋白可以与脱水反应元件结合形成特定蛋白以减少损伤[78]。水稻OsGRF4、OsGRF6 和Os-GRF8参与调控氮同化过程,可以增强水稻对缺氮条件下的适应性以保持产量[79]。在玉米中过表达ZmrGRF1则显著提高了叶片对紫外线抑制生长的抵抗能力[80]。笔者在本研究中使用转录组数据充分表征了CmGRFs 和CmGIFs 在5 种板栗组织中的不同发育时期以及4种环境胁迫下的表达模式,为后续研究板栗GRF和GIF基因的生物学功能提供了参考。
4 结 论
笔者在本研究中首次对板栗中的GRF 和GIF转录因子家族进行了全基因组鉴定和系统表征。同时,利用RNA-seq数据和RT-qPCR技术分析了其在不同板栗组织以及不同环境胁迫下的表达谱,研究结果将为进一步研究CmGRF和CmGIF家族基因的生物学功能提供理论参考。
[1] NIE X H,WANG Z H,LIU N W,SONG L,YAN B Q,XING Y,ZHANG Q,FANG K F,ZHAO Y L,CHEN X,WANG G P,QIN L,CAO Q Q. Fingerprinting 146 Chinese chestnut (Castanea mollissima Blume) accessions and selecting a core collection using SSR markers[J]. Journal of Integrative Agriculture,2021,20(5):1277-1286.
[2] ZHOU P F,ZHANG P,GUO M L,LI M S,WANG L Q,ADEEL M,SHAKOOR N,RUI Y K.Effects of age on mineral elements,amino acids and fatty acids in Chinese chestnut fruits[J].European Food Research and Technology,2021,247(8):2079-2086.
[3] HU G L,CHENG L L,CHENG Y H,MAO W T,QIAO Y J,LAN Y P. Pan-genome analysis of three main Chinese chestnut varieties[J].Frontiers in Plant Science,2022,13:916550.
[4] YU L Y,TIAN Y J,WANG X Y,CAO F,WANG H F,HUANG R M,GUO C L,ZHANG H E,ZHANG J Z.Genome-wide identification,phylogeny,evolutionary expansion,and expression analyses of ABC gene family in Castanea mollissima under temperature stress[J]. Plant Physiology and Biochemistry,2025,219:109450.
[5] LANDMAN J,VERDUYN LUNEL S M,KEGEL W K. Transcription factor competition facilitates self-sustained oscillations in single gene genetic circuits[J].PLoS Computational Biology,2023,19(9):e1011525.
[6] YOON Y,SEO D H,SHIN H,KIM H J,KIM C M,JANG G.The role of stress-responsive transcription factors in modulating abiotic stress tolerance in plants[J].Agronomy,2020,10(6):788.
[7] CHENG Z Y,LUAN Y T,MENG J S,SUN J,TAO J,ZHAO D Q. WRKY transcription factor response to high- temperature stress[J].Plants,2021,10(10):2211.
[8] MA R,LIU B W,GENG X,DING X,YAN N,SUN X,WANG W L,SUN X Z,ZHENG C S. Biological function and stress response mechanism of MYB transcription factor family genes[J].Journal of Plant Growth Regulation,2023,42(1):83-95.
[9] ZHOU C C,BO W H,EL-KASSABY Y A,LI W.Transcriptome profiles reveal response mechanisms and key role of PsNAC1 in Pinus sylvestris var. mongolica to drought stress[J]. BMC Plant Biology,2024,24(1):343.
[10] QIAN Y C,ZHANG T Y,YU Y,GOU L P,YANG J T,XU J,PI E X. Regulatory mechanisms of bHLH transcription factors in plant adaptive responses to various abiotic stresses[J]. Frontiers in Plant Science,2021,12:677611.
[11] LIU W,WANG M,ZHONG M,LUO C,SHI S Q,QIAN Y L,KANG Y Y,JIANG B. Genome-wide identification of bZIP gene family and expression analysis of BhbZIP58 under heat stress in wax gourd[J].BMC Plant Biology,2023,23(1):598.
[12] OMIDBAKHSHFARD M A,PROOST S,FUJIKURA U,MUELLER-ROEBER B. Growth-regulating factors (GRFs):A small transcription factor family with important functions in plant biology[J].Molecular Plant,2015,8(7):998-1010.
[13] FONINI L S,LAZZAROTTO F,BARROS P M,CABREIRACAGLIARI C,MARTINS M A B,SAIBO N J M,TURCHETTO-ZOLET A C,MARGIS-PINHEIRO M. Molecular evolution and diversification of the GRF transcription factor family[J].Genetics and Molecular Biology,2020,43(3):20200080.
[14] KIM J H,KENDE H. A transcriptional coactivator,AtGIF1,is involved in regulating leaf growth and morphology in Arabidopsis[J]. Proceedings of the National Academy of Sciences of the United States of America,2004,101(36):13374-13379.
[15] VAN DER KNAAP E,KIM J H,KENDE H.A novel gibberellininduced gene from rice and its potential regulatory role in stem growth[J].Plant Physiology,2000,122(3):695-704.
[16] CHOI D,KIM J H,KENDE H. Whole genome analysis of the OsGRF gene family encoding plant-specific putative transcription activators in rice (Oryza sativa L.)[J]. Plant and Cell Physiology,2004,45(7):897-904.
[17] KIM J S,MIZOI J,KIDOKORO S,MARUYAMA K,NAKAJIMA J,NAKASHIMA K,MITSUDA N,TAKIGUCHI Y,OHME-TAKAGI M,KONDOU Y,YOSHIZUMI T,MATSUI M,SHINOZAKI K,YAMAGUCHI-SHINOZAKI K.Arabidopsis GROWTH-REGULATING FACTOR7 functions as a transcriptional repressor of abscisic acid- and osmotic stress-responsive genes,including DREB2A[J]. The Plant Cell,2012,24(8):3393-3405.
[18] CHEN X H,ZHANG J,WANG S J,CAI H Y,YANG M S,DONG Y. Genome-wide molecular evolution analysis of the GRF and GIF gene families in plantae (Archaeplastida) [J].BMC Genomics,2024,25(1):74.
[19] CHENG Z H,WEN S Q,WU Y K,SHANG L N,WU L,LYU D Q,YU H T,WANG J C,JIAN H J. Comparatively evolution and expression analysis of GRF transcription factor genes in seven plant species[J].Plants,2023,12(15):2790.
[20] HORIGUCHI G,KIM G T,TSUKAYA H.The transcription factor AtGRF5 and the transcription coactivator AN3 regulate cell proliferation in leaf primordia of Arabidopsis thaliana[J]. The Plant Journal,2005,43(1):68-78.
[21] KUIJT S J H,GRECO R,AGALOU A,SHAO J X,‘T HOEN C C J,ÖVERNÄS E,OSNATO M,CURIALE S,MEYNARD D,VAN GULIK R,DE FARIA MARASCHIN S,ATALLAH M,DE KAM R J,LAMERS G E M,GUIDERDONI E,ROSSINI L,MEIJER A H,OUWERKERK P B F. Interaction between the GROWTH-REGULATING factor and KNOTTED1-LIKE HOMEOBOX families of transcription factors[J].Plant Physiology,2014,164(4):1952-1966.
[22] KIM J H,LEE B H. GROWTH-REGULATING FACTOR4 of Arabidopsis thaliana is required for development of leaves,cotyledons,and shoot apical meristem[J].Journal of Plant Biology,2006,49(6):463-468.
[23] TANG Y Y,LIU H H,GUO S Y,WANG B,LI Z T,CHONG K,XU Y Y. OsmiR396d affects gibberellin and brassinosteroid signaling to regulate plant architecture in rice[J].Plant Physiology,2018,176(1):946-959.
[24] GAO F,WANG K,LIU Y,CHEN Y P,CHEN P,SHI Z Y,LUO J,JIANG D Q,FAN F F,ZHU Y G,LI S Q. Blocking miR396 increases rice yield by shaping inflorescence architecture[J].Nature Plants,2015,2:15196.
[25] DUAN P G,NI S,WANG J M,ZHANG B L,XU R,WANG Y X,CHEN H Q,ZHU X D,LI Y H. Regulation of OsGRF4 by OsmiR396 controls grain size and yield in rice[J].Nature Plants,2015,2:15203.
[26] ZAN T,ZHANG L,XIE T T,LI L Q. Genome-wide identification and analysis of the growth-regulating factor (GRF) gene family and GRF-interacting factor family in Triticum aestivum L.[J].Biochemical Genetics,2020,58(5):705-724.
[27] LUO G B,PALMGREN M. GRF-GIF chimeras boost plant regeneration[J].Trends in Plant Science,2021,26(3):201-204.
[28] FU M K,HE Y N,YANG X Y,TANG X,WANG M,DAI W S.Genome-wide identification of the GRF family in sweet orange(Citrus sinensis) and functional analysis of the CsGRF04 in response to multiple abiotic stresses[J]. BMC Genomics,2024,25(1):37.
[29] WANG P,XIAO Y,YAN M,YAN Y,LEI X J,DI P,WANG Y P. Whole- genome identification and expression profiling of growth- regulating factor (GRF) and GRF- interacting factor(GIF) gene families in Panax ginseng[J]. BMC Genomics,2023,24(1):334.
[30] KHISTI M,AVUTHU T,YOGENDRA K,KUMAR VALLURI V,KUDAPA H,REDDY P S,TYAGI W.Genome-wide identification and expression profiling of growth- regulating factor(GRF) and GRF-interacting factor (GIF) gene families in chickpea and pigeonpea[J].Scientific Reports,2024,14:17178.
[31] DEBERNARDI J M,TRICOLI D M,ERCOLI M F,HAYTA S,RONALD P,PALATNIK J F,DUBCOVSKY J.A GRF-GIF chimeric protein improves the regeneration efficiency of transgenic plants[J].Nature Biotechnology,2020,38(11):1274-1279.
[32] ZHANG D F,LI B,JIA G Q,ZHANG T F,DAI J R,LI J S,WANG S C. Isolation and characterization of genes encoding GRF transcription factors and GIF transcriptional coactivators in maize(Zea mays L.)[J].Plant Science,2008,175(6):809-817.
[33] LUO X M,ZHANG Y,ZHOU M M,LIU K Y,ZHANG S M,YE D,TANG C R,CAO J. Overexpression of HbGRF4 or Hb-GRF4-HbGIF1 chimera improves the efficiency of somatic embryogenesis in Hevea brasiliensis[J]. International Journal of Molecular Sciences,2024,25(5):2921.
[34] FENG Q,XIAO L,HE Y Z,LIU M,WANG J F,TIAN S J,ZHANG X,YUAN L. Highly efficient,genotype-independent transformation and gene editing in watermelon (Citrullus lanatus) using a chimeric ClGRF4-GIF1 gene[J]. Journal of Integrative Plant Biology,2021,63(12):2038-2042.
[35] LEE B H,WYNN A N,FRANKS R G,HWANG Y S,LIM J,KIM J H.The Arabidopsis thaliana GRF-INTERACTING FACTOR gene family plays an essential role in control of male and female reproductive development[J]. Developmental Biology,2014,386(1):12-24.
[36] CHEN C J,CHEN H,ZHANG Y,THOMAS H R,FRANK M H,HE Y H,XIA R. TBtools:An integrative toolkit developed for interactive analyses of big biological data[J]. Molecular Plant,2020,13(8):1194-1202.
[37] WANG Y P,TANG H B,DEBARRY J D,TAN X,LI J P,WANG X Y,LEE T H,JIN H Z,MARLER B,GUO H,KISSINGER J C,PATERSON A H. MCScanX:A toolkit for detection and evolutionary analysis of gene synteny and collinearity[J]. Nucleic Acids Research,2012,40(7):e49.
[38] ZHU C C,SHI F H,CHEN Y,WANG M,ZHAO Y Q,GENG G M.Transcriptome analysis of Chinese chestnut(Castanea mollissima Blume) in response to Dryocosmus kuriphilus yasumatsu infestation[J]. International Journal of Molecular Sciences,2019,20(4):855.
[39] ZHU C C,WANG W,CHEN Y,ZHAO Y Q,ZHANG S J,SHI F H,KHALIL-UR-REHMAN M,NIEUWENHUIZEN N J.Transcriptomics and antioxidant analysis of two Chinese chestnut(Castanea mollissima Bl.)varieties provides new insights into the mechanisms of resistance to gall wasp Dryocosmus kuriphilus infestation[J]. Frontiers in Plant Science,2022,13:874434.
[40] DU B S,ZHANG Q,CAO Q Q,XING Y,QIN L,FANG K F.Morphological observation and protein expression of fertile and abortive ovules in Castanea mollissima[J]. PeerJ,2021,9:e11756.
[41] HUANG R M,PENG F,WANG D S,CAO F,GUO C L,YU L Y,ZHANG J Z,YANG Y D.Transcriptome analysis of differential sugar accumulation in the developing embryo of contrasting two Castanea mollissima cultivars[J]. Frontiers in Plant Science,2023,14:1206585.
[42] GOLDBERG D H,VICTOR J D,GARDNER E P,GARDNER D.Spike train analysis toolkit:Enabling wider application of information-theoretic techniques to neurophysiology[J]. Neuroinformatics,2009,7(3):165-178.
[43] KIM D,PERTEA G,TRAPNELL C,PIMENTEL H,KELLEY R,SALZBERG S L. TopHat2:Accurate alignment of transcriptomes in the presence of insertions,deletions and gene fusions[J].Genome Biology,2013,14(4):R36.
[44] WITTKOPP P J,KALAY G. Cis-regulatory elements:Molecular mechanisms and evolutionary processes underlying divergence[J].Nature Reviews Genetics,2011,13(1):59-69.
[45] QIN C,DU T T,ZHANG R Y,WANG Q J,LIU Y,WANG T Y,CAO H Y,BAI Q,ZHANG Y,SU S C. Integrated transcriptome,metabolome and phytohormone analysis reveals developmental differences between the first and secondary flowering in Castanea mollissima[J]. Frontiers in Plant Science,2023,14:1145418.
[46] LIU Y T,GUO P,WANG J,XU Z Y.Growth-regulating factors:Conserved and divergent roles in plant growth and development and potential value for crop improvement[J].The Plant Journal,2023,113(6):1122-1145.
[47] DEBERNARDI J M,MECCHIA M A,VERCRUYSSEN L,SMACZNIAK C,KAUFMANN K,INZE D,RODRIGUEZ R E,PALATNIK J F.Post-transcriptional control of GRF transcription factors by microRNA miR396 and GIF co-activator affects leaf size and longevity[J]. The Plant Journal,2014,79(3):413-426.
[48] KIM J H,CHOI D,KENDE H. The AtGRF family of putative transcription factors is involved in leaf and cotyledon growth in Arabidopsis[J].The Plant Journal,2003,36(1):94-104.
[49] CHEN F,YANG Y Z,LUO X F,ZHOU W G,DAI Y J,ZHENG C,LIU W G,YANG W Y,SHU K.Genome-wide identification of GRF transcription factors in soybean and expression analysis of GmGRF family under shade stress[J]. BMC Plant Biology,2019,19(1):269.
[50] LI H,QIU T T,ZHOU Z S,KANG L Q,CHEN R R,ZENG L M,YU H Y,WANG Y H,SONG J B.Genome-wide analysis of the growth-regulating factor family in Medicago truncatula[J].Journal of Plant Growth Regulation,2023,42(4):2305-2316.
[51] LIU B L,SUN Y,XUE J N,JIA X Y,LI R Z. Genome-wide characterization and expression analysis of GRAS gene family in pepper(Capsicum annuum L.)[J].PeerJ,2018,6:e4796.
[52] OMIDBAKHSHFARD M A,FUJIKURA U,OLAS J J,XUE G P,BALAZADEH S,MUELLER-ROEBER B.GROWTH-REGULATING FACTOR 9 negatively regulates Arabidopsis leaf growth by controlling ORG3 and restricting cell proliferation in leaf primordia[J].PLoS Genetics,2018,14(7):e1007484.
[53] LI Z T,WANG B,ZHANG Z Y,LUO W,TANG Y Y,NIU Y D,CHONG K,XU Y Y. OsGRF6 interacts with SLR1 to regulate OsGA2ox1 expression for coordinating chilling tolerance and growth in rice[J]. Journal of Plant Physiology,2021,260:153406.
[54] CHEN Y P,DAN Z W,GAO F,CHEN P,FAN F F,LI S Q.Rice GROWTH-REGULATING FACTOR7 modulates plant architecture through regulating GA and indole-3-acetic acid metabolism[J].Plant Physiology,2020,184(1):393-406.
[55] HE Z S,ZENG J,REN Y,CHEN D,LI W J,GAO F Y,CAO Y,LUO T,YUAN G Q,WU X H,LIANG Y Y,DENG Q M,WANG S Q,ZHENG A P,ZHU J,LIU H N,WANG L X,LI P,LI S C. OsGIF1 positively regulates the sizes of stems,leaves,and grains in rice[J].Frontiers in Plant Science,2017,8:1730.
[56] PANCHY N,LEHTI-SHIU M,SHIU S H.Evolution of gene duplication in plants[J].Plant Physiology,2016,171(4):2294-2316.
[57] VISION T J,BROWN D G,TANKSLEY S D.The origins of genomic duplications in Arabidopsis[J].Science,2000,290(5499):2114-2117.
[58] MAGADUM S,BANERJEE U,MURUGAN P,GANGAPUR D,RAVIKESAVAN R.Gene duplication as a major force in evolution[J].Journal of Genetics,2013,92(1):155-161.
[59] KONG H Z,LANDHERR L L,FROHLICH M W,LEEBENSMACK J,MA H,DEPAMPHILIS C W.Patterns of gene duplication in the plant SKP1 gene family in angiosperms:Evidence for multiple mechanisms of rapid gene birth[J]. The Plant Journal,2007,50(5):873-885.
[60] YU J,WANG J,LIN W,LI S G,LI H,ZHOU J,NI P X,DONG W,HU S N,ZENG C Q,ZHANG J G,ZHANG Y,LI R Q,XU Z Y,LI S T,LI X R,ZHENG H K,CONG L J,LIN L,YIN J N,GENG J N,LI G Y,SHI J P,LIU J,LV H,LI J,WANG J,DENG Y J,RAN L H,SHI X L,WANG X Y,WU Q F,LI C F,REN X Y,WANG J Q,WANG X L,LI D W,LIU D Y,ZHANG X W,JI Z D,ZHAO W M,SUN Y Q,ZHANG Z P,BAO J Y,HAN Y J,DONG L L,JI J,CHEN P,WU S M,LIU J S,XIAO Y,BU D B,TAN J L,YANG L,YE C,ZHANG J F,XU J Y,ZHOU Y,YU Y P,ZHANG B,ZHUANG S L,WEI H B,LIU B,LEI M,YU H,LI Y Z,XU H,WEI S L,HE X M,FANG L J,ZHANG Z J,ZHANG Y Z,HUANG X G,SU Z X,TONG W,LI J H,TONG Z Z,LI S L,YE J,WANG L S,FANG L,LEI T T,CHEN C,CHEN H,XU Z,LI H H,HUANG H Y,ZHANG F,XU H Y,LI N,ZHAO C F,LI S T,DONG L J,HUANG Y Q,LI L,XI Y,QI Q H,LI W J,ZHANG B,HU W,ZHANG Y L,TIAN X J,JIAO Y Z,LIANG X H,JIN J,GAO L,ZHENG W M,HAO B L,LIU S Q,WANG W,YUAN L P,CAO M L,MCDERMOTT J,SAMUDRALA R,WANG J,WONG G K,YANG H M.The genomes of Oryza sativa:A history of duplications[J].PLoS Biology,2005,3(2):e38.
[61] CHEN J S,LYU C C,JIANG Y J,LIU R L,LIU S Q,QU W J,HOU K,XU D B,FENG D J,WU W. Genome-wide identification of MYB genes and analysis of their expression under different abiotic stress conditions in Stevia rebaudiana[J]. Industrial Crops and Products,2024,216:118803.
[62] QIAN Y L,YAN J Q,LUO C,LI Y,WU Y G,LIU W R,LIU W,XIE D S,JIANG B. Genome-wide analysis of the MYB gene family and functional analysis of BhMYB79 in wax gourd[J].Horticultural Plant Journal,2025,11(2):788-803.
[63] SEO P J,XIANG F N,QIAO M,PARK J Y,LEE Y N,KIM S G,LEE Y H,PARK W J,PARK C M.The MYB96 transcription factor mediates abscisic acid signaling during drought stress response in Arabidopsis[J]. Plant Physiology,2009,151(1):275-289.
[64] AN J P,LI R,QU F J,YOU C X,WANG X F,HAO Y J.R2R3-MYB transcription factor MdMYB23 is involved in the cold tolerance and proanthocyanidin accumulation in apple[J]. The Plant Journal,2018,96(3):562-577.
[65] LIU J,MENG Q L,XIANG H T,SHI F M,MA L G,LI Y C,LIU C L,LIU Y,SU B H. Genome-wide analysis of Dof transcription factors and their response to cold stress in rice (Oryza sativa L.)[J].BMC Genomics,2021,22(1):800.
[66] SUN H,FAN H J,LING H Q. Genome-wide identification and characterization of the bHLH gene family in tomato[J]. BMC Genomics,2015,16(1):9.
[67] JIAO Z C,WANG L P,DU H,WANG Y,WANG W X,LIU J J,HUANG J H,HUANG W,GE L F.Genome-wide study of C2H2 zinc finger gene family in Medicago truncatula[J]. BMC Plant Biology,2020,20(1):401.
[68] MARTÍN-TRILLO M,CUBAS P.TCP genes:A family snapshot ten years later[J].Trends in Plant Science,2010,15(1):31-39.
[69] BAO M L,BIAN H W,ZHA Y L,LI F Y,SUN Y Z,BAI B,CHEN Z H,WANG J H,ZHU M Y,HAN N.miR396a-mediated basic helix-loop-helix transcription factor bHLH74 repression acts as a regulator for root growth in Arabidopsis seedlings[J].Plant and Cell Physiology,2014,55(7):1343-1353.
[70] LIANG G,HE H,LI Y,WANG F,YU D Q. Molecular mechanism of microRNA396 mediating pistil development in Arabidopsis[J].Plant Physiology,2014,164(1):249-258.
[71] PAJORO A,MADRIGAL P,MUIÑO J M,MATUS J T,JIN J,MECCHIA M A,DEBERNARDI J M,PALATNIK J F,BALAZADEH S,ARIF M,Ó'MAOILÉIDIGH D S,WELLMER F,KRAJEWSKI P,RIECHMANN J L,ANGENENT G C,KAUFMANN K. Dynamics of chromatin accessibility and gene regulation by MADS-domain transcription factors in flower development[J].Genome Biology,2014,15(3):R41.
[72] BELTRAMINO M,ERCOLI M F,DEBERNARDI J M,GOLDY C,ROJAS A M L,NOTA F,ALVAREZ M E,VERCRUYSSEN L,INZÉ D,PALATNIK J F,RODRIGUEZ R E.Robust increase of leaf size by Arabidopsis thaliana GRF3-like transcription factors under different growth conditions[J]. Scientific Reports,2018,8:13447.
[73] LU Y Z,MENG Y L,ZENG J,LUO Y,FENG Z,BIAN L Y,GAO S Y. Coordination between GROWTH-REGULATING FACTOR1 and GRF-INTERACTING FACTOR1 plays a key role in regulating leaf growth in rice[J]. BMC Plant Biology,2020,20(1):200.
[74] KIM J H. Biological roles and an evolutionary sketch of the GRF-GIF transcriptional complex in plants[J]. BMB Reports,2019,52(4):227-238.
[75] SUN Q W,ZHOU D X. Rice jmjC domain-containing gene JMJ706 encodes H3K9 demethylase required for floral organ development[J]. Proceedings of the National Academy of Sciences of the United States of America,2008,105(36):13679-13684.
[76] LIU H H,GUO S Y,XU Y Y,LI C H,ZHANG Z Y,ZHANG D J,XU S J,ZHANG C,CHONG K. OsmiR396d-regulated Os-GRFs function in floral organogenesis in rice through binding to their targets OsJMJ706 and OsCR4[J]. Plant Physiology,2014,165(1):160-174.
[77] LAZZARA F E,RODRIGUEZ R E,PALATNIK J F. Molecular mechanisms regulating GROWTH-REGULATING FACTORS activity in plant growth,development,and environmental responses[J].Journal of Experimental Botany,2024,75(14):4360-4372.
[78] WANG Q Y,LI Y H,LIN D D,FENG X X,WANG Y J,WANG T Y,DING H Y,ZHANG J S. A growth-regulating factor 7(GRF7)-mediated gene regulatory network promotes leaf growth and expansion in sugarcane[J]. The Crop Journal,2024,12(2):422-431.
[79] ZHANG J S,ZHOU Z Y,BAI J J,TAO X P,WANG L,ZHANG H,ZHU J K. Disruption of MIR396e and MIR396f improves rice yield under nitrogen-deficient conditions[J]. National Science Review,2020,7(1):102-112.
[80] CASATI P. Analysis of UV-B regulated miRNAs and their targets in maize leaves[J]. Plant Signaling & Behavior,2013,8(10):e26758.