桃(Prunus persica L.)是我国重要的经济作物之一,种植面积和产量均居世界首位[1]。桃果实风味浓郁,营养丰富,备受消费者喜欢。随着社会经济的发展,人们消费观念不断发生转变,消费者对鲜食桃品质的要求更高、更多元。在桃的品质评价指标中,甜酸风味是消费者最为关注的感官品质之一[2-3]。“甜”是桃优质育种的首要目标[4]。研究表明,甜酸风味主要取决于可溶性糖和有机酸的含量及比例,有机酸含量对甜酸风味的影响高于糖组分[5]。揭示桃果实中糖组分和酸组分代谢调控机制对桃品质育种至关重要。现阶段桃育种仍主要通过常规杂交育种方式进行桃果实品质改良,周期长,随机性大,成本高,急需利用现代分子手段建立高效的桃育种体系。生产中过分追求桃产量和果个大小,栽培管理措施不合理,导致果实内在品质欠佳[6]。结合前人的研究,笔者从桃果实糖酸变化、糖酸组分合成、降解与转运和影响糖酸的因子(遗传背景、外界因子、栽培措施)等方面展开论述,以期为利用栽培技术和现代分子育种手段提升桃果实品质提供理论基础。
1 桃果实糖酸组分积累规律
桃果实中的可溶性糖包括蔗糖、葡萄糖、果糖和山梨醇等,其中蔗糖是主要的碳水化合物[7]。大多数研究表明,在桃果实发育进程中总糖含量不断增加,其中蔗糖含量呈上升趋势,在果实成熟后期下降,果糖含量随桃果实发育呈下降趋势,葡萄糖含量变化与果糖含量基本一致[8]。桃果实的山梨醇含量较低,且随桃果实发育呈先上升后下降的趋势[9]。Ⅴizzotto等[10]利用同位素示踪法分析的结果表明,桃幼果中葡萄糖和果糖含量高,蔗糖和山梨醇含量低,花后88 d蔗糖含量开始显著增加。沈志军等[11]分析21个桃品种果实发育过程中糖组分的变化规律,不同品种桃果实中蔗糖表现出相同的变化趋势,即在成熟前30~45 d快速积累,而不同品种中葡萄糖、果糖和山梨醇的变化差异较大。在桃果实成熟后期,伴随乙烯释放,果实中蔗糖含量降低,葡萄糖和果糖含量增加,而山梨醇含量持续降低,且不受果实成熟后其他因子改变的影响[12]。
桃果实中最重要的有机酸有3类,分别为苹果酸、柠檬酸和奎尼酸。在201份桃种质果实中,苹果酸含量最高,占总有机酸的62.0%,柠檬酸含量次之,占总有机酸的22.6%,二者对桃总酸度的贡献最大[13]。Zheng等[14]对75份桃种质成熟果实中有机酸含量分析的结果表明,苹果酸、柠檬酸和奎尼酸平均占比分别为60.7%、25.6%和10.7%,其中高酸品种有柠檬酸主导型和苹果酸主导型。徐子媛等[5]分析了73份桃种质果实中有机酸含量,其中安农水蜜、陈圃蟠桃、宣城甜桃和春蜜等以奎尼酸为主,含量分别占总酸含量的66.4%、52.2%、48.3%和40.8%。大多数研究表明,在桃果实发育进程中有机酸含量呈下降趋势。在不同品种间,随桃果实发育苹果酸含量变化有两种趋势,分别为整体下降和缓慢积累,而奎尼酸和柠檬酸整体上均呈下降趋势。沈志军等[11]分析21个桃品种果实发育过程中酸组分的变化规律,发现柠檬酸含量在不同品种间表现相似的变化规律,在花后10 d果实中柠檬酸含量最高,花后10~30 d柠檬酸含量大幅下降,而苹果酸和奎尼酸含量无明显变化规律。Jiang等[15]比较了5份桃种质果实中酸组分的差异,山桃果实的总酸含量最高,主要由苹果酸和奎尼酸组成,奎尼酸含量在授粉75 d后缓慢下降,而苹果酸从花后50 d开始持续积累,此外油桃和毛桃之间果实酸含量差异发生在果实第2次膨大期和成熟期。Wang等[16]比较了低酸品种金凤和酸品种天津水蜜的有机酸含量,发现有机酸含量差异发生在花后100~120 d。上述结果表明,成熟期是决定桃果实酸风味品质形成的关键期。
2 桃果实糖组分合成与降解的分子机制
2.1 蔗糖的合成与降解
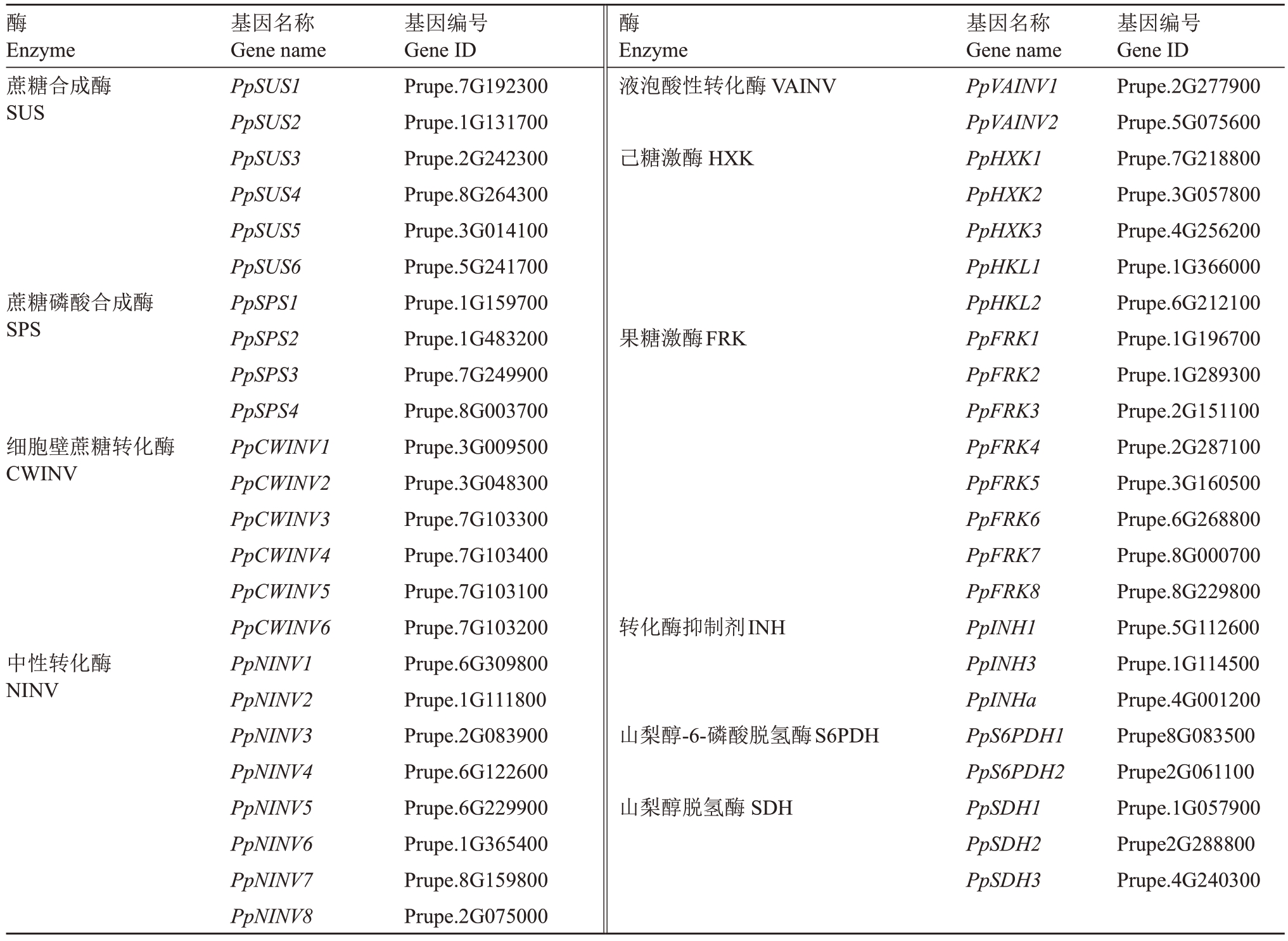
蔗糖是大多数桃种质果实中的主要糖组分,参与蔗糖代谢的酶主要有蔗糖合成酶(SS和SUS)、蔗糖磷酸合成酶(SPS)和转化酶(IⅤR)(表1)。Zhang等[17-18]在桃基因组中鉴定到6 个蔗糖合成酶基因(PpSUS1-6),分属于3 类,且都在果实中表达,其中PpSUS1 在花后105 d 果实中表达量最高。PpSUS4在桃果实中的表达水平与糖积累呈显著正相关[19]。SPS 不仅是植物光合“源”组织中蔗糖合成的关键酶,而且在积累蔗糖的“库”组织中也起重要作用。Aslam 等[12]在桃基因组中鉴定到4 个PpSPSs,PpSPS2 的转录水平在花后65 d 开始持续升高。PpSPS3 在成熟果实中的表达量显著高于未成熟果实[7],但在霞晖6号中,PpSPS3的表达无显著变化[18],说明不同品种之间调控蔗糖合成的SPS基因存在差异。转化酶包括细胞壁蔗糖转化酶(CWINⅤ)、中性转化酶(NINⅤ)和液泡酸性转化酶(ⅤAINⅤ)。在细胞中,蔗糖通过转化酶裂解成为葡萄糖和果糖。Ⅴimolmangkang 等[7]在桃基因组中鉴定到6 个PpCWINVs、8 个PpNINVs 和2 个PpVAINVs,其中PpNINV8的转录丰度与蔗糖含量呈显著正相关。PpCWINV2和PpNINV2的转录水平与蔗糖含量无显著相关性,但NINⅤ活性与蔗糖含量呈显著负相关[18]。PpNINV3 表达量与NINⅤ活性呈正相关[20]。过表达PpVAINV2 使桃果实中总糖和蔗糖含量下降[21]。上述研究表明,PpNINV3、PpNINV8 和PpVAINV2 可能是参与桃果实中蔗糖代谢的关键基因。20 世纪60年代,研究者发现了一种抑制转化酶活性的内源性蛋白质,称之为转化酶抑制剂(INH)[22],它与ⅤAINⅤ相互作用并调节其活性,从而在糖信号转导和碳分配中发挥至关重要的作用。研究表明,PpINH1通过抑制桃果实采后PpVAINV2 表达来维持蔗糖水平[23]。过表达PpINH3使桃果实中蔗糖含量增加,而过表达PpINHa 的桃果实中蔗糖含量却下降,但是二者都不与PpⅤAINⅤ2 互作[24],相关的调控路径有待揭示。
表1 桃果实糖代谢关键基因信息
Table 1 Key genes involved in the sugar metabolism of peach fruit
酶酶Enzyme蔗糖合成酶SUS Enzyme液泡酸性转化酶ⅤAINⅤ己糖激酶HXK蔗糖磷酸合成酶SPS 果糖激酶FRK细胞壁蔗糖转化酶CWINⅤ转化酶抑制剂INH中性转化酶NINⅤ基因名称Gene name PpSUS1 PpSUS2 PpSUS3 PpSUS4 PpSUS5 PpSUS6 PpSPS1 PpSPS2 PpSPS3 PpSPS4 PpCWINV1 PpCWINV2 PpCWINV3 PpCWINV4 PpCWINV5 PpCWINV6 PpNINV1 PpNINV2 PpNINV3 PpNINV4 PpNINV5 PpNINV6 PpNINV7 PpNINV8基因编号Gene ID Prupe.7G192300 Prupe.1G131700 Prupe.2G242300 Prupe.8G264300 Prupe.3G014100 Prupe.5G241700 Prupe.1G159700 Prupe.1G483200 Prupe.7G249900 Prupe.8G003700 Prupe.3G009500 Prupe.3G048300 Prupe.7G103300 Prupe.7G103400 Prupe.7G103100 Prupe.7G103200 Prupe.6G309800 Prupe.1G111800 Prupe.2G083900 Prupe.6G122600 Prupe.6G229900 Prupe.1G365400 Prupe.8G159800 Prupe.2G075000山梨醇-6-磷酸脱氢酶S6PDH山梨醇脱氢酶SDH基因名称Gene name PpVAINV1 PpVAINV2 PpHXK1 PpHXK2 PpHXK3 PpHKL1 PpHKL2 PpFRK1 PpFRK2 PpFRK3 PpFRK4 PpFRK5 PpFRK6 PpFRK7 PpFRK8 PpINH1 PpINH3 PpINHa PpS6PDH1 PpS6PDH2 PpSDH1 PpSDH2 PpSDH3基因编号Gene ID Prupe.2G277900 Prupe.5G075600 Prupe.7G218800 Prupe.3G057800 Prupe.4G256200 Prupe.1G366000 Prupe.6G212100 Prupe.1G196700 Prupe.1G289300 Prupe.2G151100 Prupe.2G287100 Prupe.3G160500 Prupe.6G268800 Prupe.8G000700 Prupe.8G229800 Prupe.5G112600 Prupe.1G114500 Prupe.4G001200 Prupe8G083500 Prupe2G061100 Prupe.1G057900 Prupe2G288800 Prupe.4G240300
2.2 葡萄糖和果糖的合成与降解
葡萄糖和果糖是桃果实中最重要的己糖。己糖激酶(HXK)和果糖激酶(FRK)是催化己糖磷酸化的两种关键酶(表1)。HXK催化糖酵解过程的第一步。Xu 等[24]在桃基因组中鉴定到5 个己糖激酶基因,包括PpHXK1-3和PpHKL1-2。FRK是磷酸果糖激酶B型家族(pfkB)的一员,在6-磷酸果糖(F6P)的合成中起着至关重要的作用。桃基因组中有8 个PpFRK 家族成员[25]。在桃果实发育过程中,花后85~90 d PpHXK2 和PpHKL1 的转录水平提高,花后80 d 到成熟期PpFRK1、PpFRK4 和PpFRK5 的表达水平降低[12]。此外,研究认为高糖桃品种果实中,PpHXK2 和PpFRK5 在低温贮藏过程中调控糖代谢[26]。
2.3 山梨醇的合成与降解
在桃果实生长过程中,山梨醇含量变化较小且含量相对较低,占总糖含量的2.63%~10.39%。山梨醇转化为果糖和葡萄糖是果实发育和成熟过程中甜味逐渐增加的原因[27]。山梨醇合成代谢酶包括山梨醇-6-磷酸脱氢酶(S6PDH)和山梨醇-6-磷酸磷酸酶(SorPP)。山梨醇分解代谢酶包括山梨醇氧化酶(SOX)、NAD-山梨醇脱氢酶(NAD+-SDH)和NADP+-山梨醇脱氢酶(NADP-SDH)(表1)。Li等[28]在桃基因组中鉴定到2个PpS6PDHs和1个PpSDH,其中PpS6PDH2和PpSDH2在桃叶片中的表达量高于果实,这可能与山梨醇的合成发生在叶片有关。Yamada 等[29]研究表明,在未成熟桃果实中NADSDH 活性高,之后下降,然后随着果实成熟再次升高。1 ℃贮藏条件下桃果实中S6PDH 活性升高促进山梨醇的合成,同时SOX、NAD+-SDH 和NADP+-SDH活性降低减缓山梨醇的降解[30]。
除上述直接参与糖代谢的酶以外,其他非直接参与代谢过程的结构基因和转录因子也被报道。蔗糖非酵解型蛋白激酶(SnRKs)有3 个亚家族,包括SnRK1s、SnRK2s 和SnRK3s。PpSnRK1α 可以磷酸化修饰PpSDH增强山梨醇代谢活性,同时调控SUS和SPS活性,促进蔗糖的积累[31]。Zhang等[32]研究表明,过表达PpNAC1 和PpNAC5 显著提高桃糖含量。PpNAC050过表达显著提高桃果糖和葡萄糖含量[33]。Wang等[26]利用高通量测序的方法筛选到8个转录因子,其中PpMYB1/3、PpMYB- related1、PpWRKY4、PpbZIP1/2/3 和PpbHLH2 与糖代谢有关。此外,Shang 等[34]发现转录因子PpTCP13、硝酸盐转运蛋白PpNRT1、推定转录延伸因子PpSTP5 和F-box家族成员PpFBX与桃果实山梨醇含量的变异密切相关。
3 桃果实酸组分合成与降解的分子机制
3.1 苹果酸的合成与降解
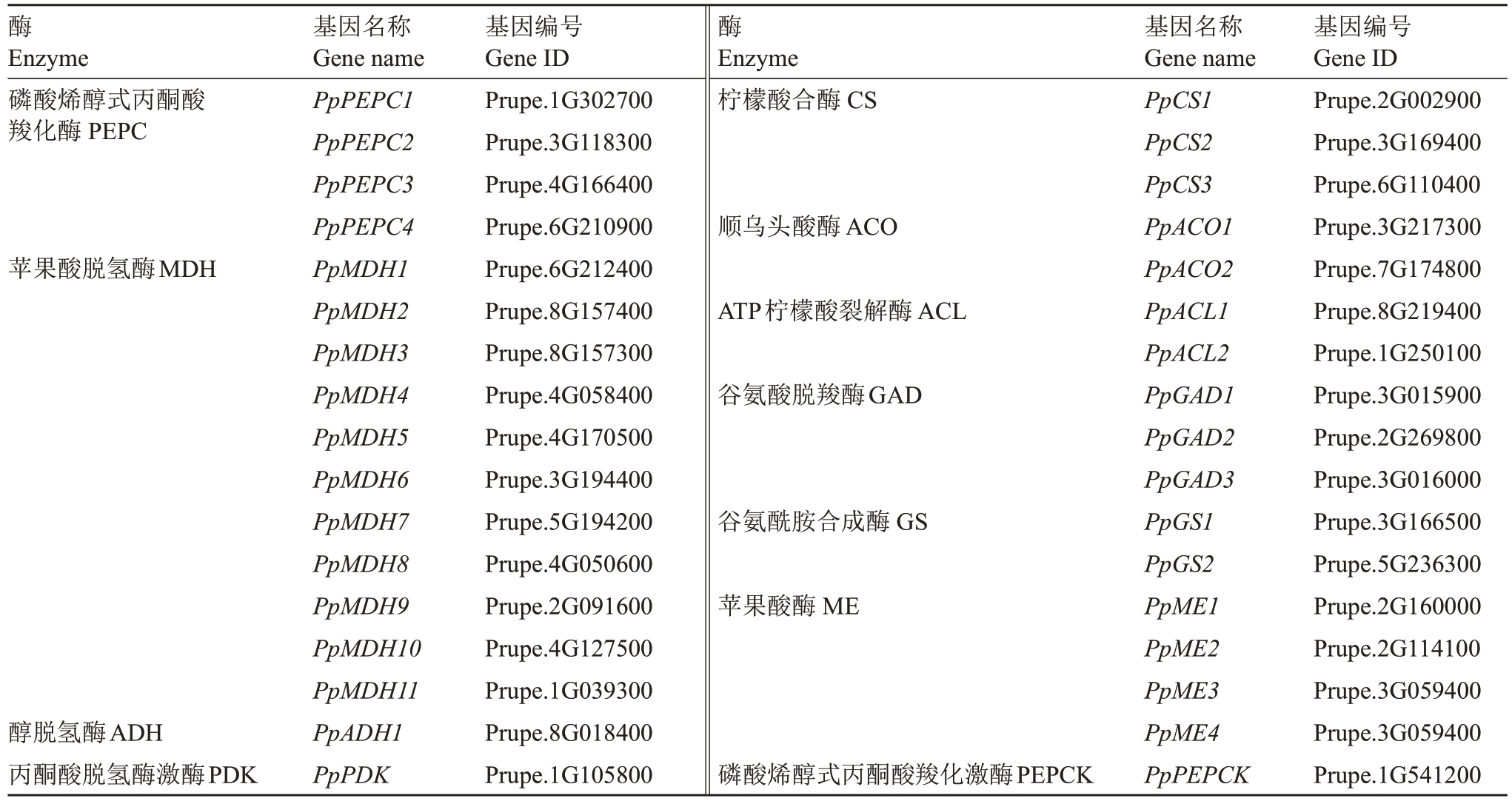
参与苹果酸代谢的酶有磷酸烯醇式丙酮酸羧化酶(PEPC)、苹果酸脱氢酶(MDH)、苹果酸酶(ME)和磷酸烯醇式丙酮酸羧化激酶(PEPCK)(表2)。PEPC 催化磷酸烯醇式丙酮酸(PEP)羧化合成草酰乙酸(OAA)和其他无机盐。Zhi 等[35]研究表明,桃基因组中有3 个PpPEPCs,成员间表达模式存在差异。高酸品种桃果实幼果期PpPEPC2 的表达量高于低酸品种,在成熟期表达量无显著差异[36]。苹果酸脱氢酶(MDH)负责催化OAA和苹果酸之间的可逆反应。Ma 等[37]在桃基因组中鉴定到11 个编码MDH 的基因。Zheng 等[14]研究发现PpMDH3 在霞脆、霞晖和瓦罗兰特等桃果实中的表达水平与苹果酸含量变化一致。李依尘[8]报道了PpMDH3、PpMDH5、PpMDH9 和PpMDH10 在不同品种和不同成熟期桃果实中的表达特征与苹果酸积累的相关性规律各异。细胞质NADP依赖性苹果酸酶(NADP-cytME)催化苹果酸羧化生成丙酮酸(PK)。NADP-cytME 与果实成熟过程中苹果酸含量的降低有关,且因不同物种而异。研究表明,随桃果实发育苹果酸含量降低的品种中,PpME1 与苹果酸的降解相关[8]。仓方早生及其早熟芽变桃中苹果酸含量差异受PpMDH3 和PpME2 表达调控[38]。PEPCK 催化OAA 转化为PEP 的可逆反应。在桃果实发育过程中,PpPEPCK 的转录水平显著提高,花后87 d 时的表达水平是花后37 d 的约50 倍,在采后成熟过程中,PpPEPCK 的转录水平较高,参与果实中有机酸的分解代谢,从而影响苹果酸积累[39]。以上结果表明,不同基因型桃之间调控苹果酸代谢的基因存在差异。
表2 有机酸代谢关键基因信息
Table 2 Key genes involved in the organic acid metabolism of peach fruit
酶酶Enzyme磷酸烯醇式丙酮酸羧化酶PEPC Enzyme柠檬酸合酶CS顺乌头酸酶ACO苹果酸脱氢酶MDH ATP柠檬酸裂解酶ACL谷氨酸脱羧酶GAD谷氨酰胺合成酶GS苹果酸酶ME醇脱氢酶ADH丙酮酸脱氢酶激酶PDK基因名称Gene name PpPEPC1 PpPEPC2 PpPEPC3 PpPEPC4 PpMDH1 PpMDH2 PpMDH3 PpMDH4 PpMDH5 PpMDH6 PpMDH7 PpMDH8 PpMDH9 PpMDH10 PpMDH11 PpADH1 PpPDK基因编号Gene ID Prupe.1G302700 Prupe.3G118300 Prupe.4G166400 Prupe.6G210900 Prupe.6G212400 Prupe.8G157400 Prupe.8G157300 Prupe.4G058400 Prupe.4G170500 Prupe.3G194400 Prupe.5G194200 Prupe.4G050600 Prupe.2G091600 Prupe.4G127500 Prupe.1G039300 Prupe.8G018400 Prupe.1G105800磷酸烯醇式丙酮酸羧化激酶PEPCK基因名称Gene name PpCS1 PpCS2 PpCS3 PpACO1 PpACO2 PpACL1 PpACL2 PpGAD1 PpGAD2 PpGAD3 PpGS1 PpGS2 PpME1 PpME2 PpME3 PpME4 PpPEPCK基因编号Gene ID Prupe.2G002900 Prupe.3G169400 Prupe.6G110400 Prupe.3G217300 Prupe.7G174800 Prupe.8G219400 Prupe.1G250100 Prupe.3G015900 Prupe.2G269800 Prupe.3G016000 Prupe.3G166500 Prupe.5G236300 Prupe.2G160000 Prupe.2G114100 Prupe.3G059400 Prupe.3G059400 Prupe.1G541200
3.2 柠檬酸的合成与降解
桃果实柠檬酸合成有两种途径,一种为茎和叶中合成的糖转运到果实细胞,通过糖酵解和三羧酸循环(TCA)转换合成柠檬酸,另一种为果实细胞直接进行碳固定合成柠檬酸。MDH、ME、PEPC 和柠檬酸合酶(CS)等参与柠檬酸合成[40](表2)。在线粒体NAD-mtMDH和NAD-mtME的共同作用下,苹果酸分别生成OAA 和丙酮酸,中断TCA 循环进而影响果实柠檬酸含量。CS催化OAA和乙酰辅酶A合成柠檬酸。PpCS1和PpCS2在桃果实中的表达模式差异明显,其中PpCS2 在不同品种中的表达变化与其柠檬酸含量变化规律一致,可能是桃果实柠檬酸合成的关键基因[8]。Zheng等[14]研究发现,在高酸和低酸品种之间PpCSs 表达水平无显著差异;丙酮酸脱氢酶激酶基因PpPDK 表达下调促进高酸桃品种中乙酰辅酶A的合成,从而促进柠檬酸的积累;醇脱氢酶基因PpADH1在低酸品种中的高表达对丙酮酸在细胞质中的积累产生负面效应,影响柠檬酸积累。
异柠檬酸脱氢酶(IDH)和顺乌头酸酶(ACO)负责线粒体中柠檬酸的降解反应(表2)。研究表明,PpACO1在不同桃品种果实柠檬酸积累过程中整体上呈上调表达趋势,PpACO2 在不同品种间差异较大且与成熟过程中柠檬酸积累没有显著关联[8]。ATP柠檬酸裂解酶(ACL)催化柠檬酸转化为草酰乙酸和乙酰辅酶。其中,PpACL1 可能是参与柠檬酸降解的关键基因[8]。此外,γ-氨基丁酸(GABA)途径也是柠檬酸降解的主要途径之一,包括谷氨酸脱羧酶(GAD)和谷氨酰胺合成酶(GS)(表2)。在湖景蜜露、丹霞玉露、玉露和白丽桃果实成熟过程中,Pp-GAD1 和PpGAD2 的表达量均呈下降趋势,与柠檬酸含量变化相似[8]。Zheng等[32]研究表明,低酸品种桃果实中PpGAD3表达与柠檬酸的降解相关[14]。过表达PpGAD3降低了桃果实中的柠檬酸含量。李依尘[8]研究表明,PpGS2 在桃果实发育过程中呈上升表达模式,从而促进柠檬酸降解。
除上述参与酸代谢的结构基因以外,Wang等[16]认为转录因子PpWRKY50 和PpMYB62 与桃果实苹果酸含量相关,PpbHLH1与柠檬酸含量相关。Jiang等[15]通过WGCNA 分析认为MYB、MADS-box、GRAS和HSF等转录因子参与调控桃果实发育过程中的酸代谢。Zhang 等[32]研究表明,PpNAC1 和Pp-NAC5 可以激活有机酸降解基因PpGAD3 的表达。这些成果丰富了桃果实酸合成与降解的调控网络。
4 桃果实糖酸转运的分子机制
4.1 糖分子转运
液泡是成熟期桃果实细胞糖沉积的主要场所。蔗糖通过蔗糖转运蛋白(SUT)、液泡膜糖转运蛋白(TST)和液泡葡萄糖转运蛋白(ⅤGT)等转运到液泡中(表3)。Aslam 等[12]从桃基因组上鉴定到3 个PpSUTs 和3 个PpVGTs,其中PpSUT2、PpVGT1、PpVGT2和PpVGT3参与调控蔗糖转运。Zanon等[41]研究发现,Redhaven中PpSUT2和PpSUT3在果肉中高表达。然而,Zhang 等[18]研究表明,霞晖6 号中PpSUT1 在果实发育后期表达量显著提高。液泡糖转运蛋白(TSTs)的功能是把细胞质中的葡萄糖、果糖和蔗糖转运至液泡中。桃基因组中共有4 个PpTST 成员,PpTST2 与蔗糖含量呈显著正相关,与葡萄糖和果糖含量呈显著负相关[7]。在中桃13 中,PpTST1和PpTST2的转录水平在果实成熟过程中整体呈上升趋势[12]。研究表明,PpTST1在桃果实成熟期的表达水平最高,与可溶性糖积累模式一致,基因沉默后总糖、蔗糖、葡萄糖和果糖的含量显著降低,山梨醇含量则无显著变化[42]。PpTST1 表达受ABA响应转录因子PpABRE1的激活,调控可溶性糖的含量[43]。早期响应干旱类似蛋白(ERD6L)介导液泡中葡萄糖/H+的同向输出以及非特异介导糖分的易化扩散。PpERDL16 属于单糖转运蛋白ERD6-like 亚家族成员,过表达PpERDL16 降低了桃果肉中葡萄糖和果糖含量[44]。SWEET 蛋白也是一类广泛存在于不同类型真核单细胞生物、高等植物以及动物中的糖运输蛋白。贾梦霄[45]在桃基因组上共鉴定到19个PpSWEETs,分为4 个分支,其中Clade Ⅲ中的PpSWEET11 和PpSWEET12 具有蔗糖转运蛋白活性。在桃果实发育过程中,PpSWEET4/6/8/7/15/16/19表达量与蔗糖含量显著相关[46]。山梨醇转运属于主动运输,主要由山梨醇转运蛋白(SOT)协助完成。余彩云等[47]在桃基因组中鉴定到9 个PpSOTs。PpSOT1、PpSOT4 和PpSOT9 在果肉中表达量较高[48],可能调控山梨醇的转运过程。
表3 糖酸分子转运关键基因信息
Table 3 Key genes involved in the sugar and organic acid transportation of peach fruit

酶酶Enzyme蔗糖转运蛋白SUT Enzyme液泡葡萄糖转运蛋白ⅤGT液泡膜糖转运蛋白TST SWEET山梨醇转运蛋白SOT铝离子激活的苹果酸转运蛋白ALMT基因名称Gene name PpSUT1 PpSUT2 PpSUT3 PpTST1 PpTST2 PpTST3 PpTST4 PpSOT1 PpSOT2 PpSOT3 PpSOT4 PpSOT5 PpSOT6 PpSOT7 PpSOT8 PpSOT9 PpALMT1 PpALMT2 PpALMT3 PpALMT4 PpALMT5 PpALMT6 PpALMT7 PpALMT8 PpALMT9 PpALMT10 PpALMT11基因编号Gene ID Prupe.8G052700 Prupe.1G542000 Prupe.1G271500 Prupe.5G006300 Prupe.8G180600 Prupe.7G186000 Prupe.7G185700 Prupe.8G100700 Prupe.8G100900 Prupe.8G101000 Prupe.8G101200 Prupe.8G105300 Prupe.8G105400 Prupe.8G105500 Prupe.8G105600 Prupe.8G101500 Prupe.6G144200 Prupe.2G192500 Prupe.8G242400 Prupe.1G308100 Prupe.5G220200 Prupe.1G308300 Prupe.5G110600 Prupe.5G040200 Prupe.8G253500 Prupe.7G094400 Prupe.5G127200液泡型质子焦磷酸酶Ⅴ-PPase二羧酸转运蛋白TDT早期响应干旱类似蛋白ERDL基因名称Gene name PpALMT12 PpVGT1 PpVGT2 PpVGT3 PpSWEET1 PpSWEET3 PpSWEET4 PpSWEET5 PpSWEET6 PpSWEET7 PpSWEET8 PpSWEET9 PpSWEET10 PpSWEET11 PpSWEET12 PpSWEET13 PpSWEET14 PpSWEET15 PpSWEET16 PpSWEET17 PpSWEET18 PpSWEET19 PpVp1 PpVp2 PpTDT PpERDL16基因编号Gene ID Prupe.6G144000 Prupe.2G325500 Prupe.2G100000 Prupe.6G070700 Prupe.1G133300 Prupe.2G118600 Prupe.2G245600 Prupe.2G307700 Prupe.2G307800 Prupe.3G034900 Prupe.3G283400 Prupe.4G072300 Prupe.4G155700 Prupe.5G146500 Prupe.1G220700 Prupe.5G146400 Prupe.5G125100 Prupe.5G175500 Prupe.6G355900 Prupe.8G017400 Prupe.8G017500 Prupe.8G076100 Prupe.3G091900 Prupe.6G313800 Prupe.4G009400 Prupe.1G378500
4.2 酸分子转运
苹果酸的积累除受代谢水平的调控外,还与苹果酸的转运密切相关。苹果酸主要储存于液泡中,其从细胞质到液泡的转移主要有两种方式,一是液泡型质子泵Ⅴ-ATPase和Ⅴ-PPase驱动H+进入液泡维持酸性环境,质子化苹果酸,使其保持电化学势梯度并连续输送到液泡;二是在二羧酸转运蛋白(DT)和铝离子激活的苹果酸转运蛋白(ALMT)作用下进入液泡中积累[49](表3)。Etienne等[50]研究表明,PpVp2在桃果实发育过程中的表达模式与有机酸积累一致,参与果实中有机酸积累的调控。Xu等[51]在桃基因组中鉴定到12 个PpALMTs。PpALMT4 的表达趋势与果实苹果酸的积累变化一致[8]。Zheng 等[14]研究表明,低酸桃品种果实中PpALMT6表达量下调与苹果酸由胞质向液泡转运的减少有关。Yu 等[44]过表达PpALMT1提高了桃果肉中苹果酸含量。
此外,研究表明,PpTST1 除参与转运糖分子以外,还与桃果实有机酸的积累密切相关。PpTST1HIS过表达减少了总有机酸、苹果酸、柠檬酸和奎尼酸含量,其对质子泵相关基因的表达无显著影响,但降低了参与有机酸转运基因的表达水平,如质膜二羧酸转运蛋白基因PpTDT 和3 个PpALMTs(Prupe.5G110600,Prupe.5G127200和Prupe.6G144100)[52]。
5 影响桃果实糖酸含量的因子及其调控机制
5.1 遗传因素
桃种质资源多样,其复杂的遗传背景形成了丰富的桃果实糖酸性状。朱更瑞等[53]对6类生态品种群118 份桃地方品种糖酸组分的分析表明,糖酸组分差异形成源自不同生态区选择导致的遗传物质差异。不同类型桃之间糖酸存在差异,蟠桃对可溶性固形物含量有11.20%的遗传增效性,油蟠桃对可溶性固形物含量有27.64%的遗传增效性[4]。Wu 等[54]连续两年的试验表明,母性遗传对果实糖酸性状并没有显著影响,蟠桃后代的蔗糖和总糖含量高于圆形果后代,苹果酸、柠檬酸和总酸含量与果形无显著相关性。表明桃自身携带的遗传信息与果实糖酸性状密切相关。蔗糖性状定位在桃基因组5号染色体(LG5)上,其中PpTST1 调控蔗糖含量,该基因第三外显子中的非同义G/T变异引起组氨酸取代谷氨酰胺,G/T或T/T基因型桃果实的蔗糖和总糖含量显著高于G/G 基因型的桃品种[42],但其机制仍待进一步揭示。Wang 等[52]利用GWAS 和RNA-Seq 将有机酸积累性状也定位在LG5上,PpTST1参与调控可滴定酸、苹果酸和柠檬酸含量。Yu 等[44]通过564 份桃种质的重测序结果把蔗糖相关的QTL定位在LG5上,PpTST1 位于该位点附近;葡萄糖性状定位在LG1、LG3、LG4 和LG8 上;果糖性状定位在LG1 上,其中PpERDL16 负调控果糖积累;LG5 上的QTL 位点与可滴定酸、苹果酸和柠檬酸性状相关。在控制酸性状的LG5 上,编码一种假定小蛋白的候选基因PpRPH 的表达与果实总酸含量变化一致[55]。Hernandez-Mora 等[56]研究表明,LG1 上的PpPDK 也参与调控桃可滴定酸性状。
5.2 外界因素
诸多植物生长调节剂等在调控果实代谢过程中扮演重要角色。1.25 mmol·L-1 GA4+7促进PpSPS2表达,并显著提高锦绣黄桃果实的蔗糖含量;0.5 mmol·L-1 NAA 处理显著降低了果实蔗糖含量[57]。1 μmol·L-1水杨酸显著促进贮藏期PpSPS4 的表达,抑制Pp-SUS2 和PpNINV3 的表达,从而提高果实蔗糖含量[58]。桃叶面喷施150 μmol·L-1 褪黑素显著提高SUS、SPS、NINⅤ和ⅤAINⅤ活性,降低SOX活性,提高可溶性总糖和蔗糖含量,增加成熟期葡萄糖和山梨醇含量。此外,褪黑素可以显著降低PpCS3 和PpPEPC4 的表达水平,促进PpME3 和PpMDH11 的转录,降低桃果实成熟期总有机酸、苹果酸和柠檬酸含量[59]。5-氨基乙酰丙酸(ALA)显著促进桃果实幼果期淀粉积累和成熟期降解为蔗糖[46]。桃树体喷施山梨醇对果实糖组分含量的影响并不显著,但是糖组分间的比值显著降低,糖代谢酶基因PpSDH、PpHXK2、PpSUS1/4、PpNINV2 和糖转运基因PpSOT1/9、PpSWEET9/15 参与调控糖组分积累[48]。β-氨基丁酸(BABA)、茉莉酸甲酯(MeJA)或苯并噻唑(BTH)诱导PpWRKY40 表达,增强蔗糖磷酸合成酶活性,促进蔗糖的合成;另外,诱导桃果实山梨醇分解代谢酶S6PDH、NAD-SDH 和NADP-SDH 活性增强,促进山梨醇转化为葡萄糖和果糖[60]。在贮藏期,使用1-MCP 后桃果实中苹果酸合成基因Pp-PEPC2、PpPEPC3和PpMDH11的表达无显著变化,但抑制了苹果酸降解基因PpME4的表达,延缓了苹果酸降解。此外,1-MCP 处理也推迟了桃果实PpCS1 的转录丰度增加,第7 天时柠檬酸盐含量增加0.5倍[61]。
温度是影响果实代谢的重要环境因子。诸多研究表明,在低温环境下桃果实为避免冷冻伤害,启动调控糖代谢进程。Chen等[62]研究发现,低温诱导编码锌指蛋白的基因PpZAT10 的表达并与PpVAINV2启动子直接结合,PpZAT10虽未显著影响PpVAINV2的转录水平但降低了ⅤIN 活性,调控了蔗糖代谢过程。Cao 等[63]研究表明,低温诱导C-重复结合因子(CBF)基因PpCBF6 的表达,PpCBF6 与PpVAINV2启动子结合并负调控其表达,进而延缓蔗糖的降解。低温诱导桃果实乙烯响应转录因子PpRAP2.12的表达,并与PpVAINV2 互作,从而提高ⅤIN 活性,降低了蔗糖水平[64]。油菜素内酯信号通路中参与低温响应的BES/BZR 转录因子PpBZR1 通过抑制PpVAINV2的表达和ⅤIN活性,提高蔗糖含量[65]。多聚半乳糖醛酸酶抑制蛋白PpPGIP1与PpⅤAINⅤ2蛋白相互作用正向调节桃果实液泡转化酶活性,进而加速蔗糖降解[66]。上述基因均通过调控果实蔗糖降解基因PpVAINV2,改变了桃果实应对低温环境的能力。
适宜的光照对优质水果生产至关重要。紫外线b可以调节植物的生长发育、光合作用、抗氧化系统和内源激素,并影响植物的产量。Wang 等[67]研究表明,1.44 kJ·m-2·d-1 UⅤ-B 增强了叶片中单糖转运体功能,导致蔗糖向果实的转运增加,使第2次膨大期果实的蔗糖和总糖含量增加。UⅤ-C 照射显著抑制了PpVAINV1 的表达,促进了蔗糖合成基因PpSPS2和酸代谢相关基因PpACL2 和PpMDH11 的表达上调,使桃果实蔗糖、柠檬酸和苹果酸含量显著增加[68]。γ 辐射显著促进桃果实中苹果酸和蔗糖的积累[69]。
5.3 栽培管理
优良的栽培管理模式是影响桃果实糖酸风味的重要外因,包括砧木的选择、果实负载量、水肥管理等。Font i forcada等[70]比较了嫁接在6种砧木(Adafuel、Adarcias、Felinem、Garnem、GF677 和Cadaman)上的桃果实糖酸含量,其中嫁接在砧木Adarcias 上的桃果实单糖和总糖含量最高。Nuñez 等[71]研究表明,疏果通过影响叶片和果实中PpSUT1 和PpSUT2的转录水平,从而影响果实的糖组分含量。Toumi等[72]研究表明,与充分灌溉相比,亏缺灌溉和根系分区灌溉不影响桃果实山梨醇和蔗糖含量,但葡萄糖含量提高23%和21.5%。充分灌溉下桃果实可滴定酸含量显著高于亏缺灌溉和根系分区灌溉处理,主要原因是苹果酸的积累,而奎尼酸和柠檬酸含量显著低于亏缺灌溉和根系分区灌溉处理。与不套袋+化肥处理相比,在黑色果袋+生物有机肥栽培模式下,桃果实糖含量提高了9.66%,参与蔗糖合成的PpSUS4 和PpSPS2 差异表达[73],但是光和肥水为核心的影响因子尚不明确。与设施栽培相比,露地桃果实中蔗糖、果糖和葡萄糖积累量更高,其中PpSDH1、PpSDH2和PpSDH3的高表达使更多山梨醇转化为葡萄糖和果糖[9]。因此,考虑栽培管理因子的复杂性,有待开展更多研究明确栽培措施在调控果实糖酸风味中的关键作用。
6 展 望
桃果实糖酸代谢调控极其复杂,每个组分的代谢均涉及合成、降解和转运等过程,各个过程又受到遗传背景、发育过程、环境因子以及栽培管理措施等诸多方面的影响。目前,桃果实中糖酸组分、含量及其代谢调控机制取得了一系列的进展。但是,鉴于果实糖酸代谢的复杂性以及糖酸平衡在调控果实风味中的重要作用,仍有诸多问题有待深入研究。基于前人的研究成果,对于今后开展研究的几点建议:1)挖掘特异桃种质资源,鉴定参与调控糖酸单一性状和复合性状的关键基因,利用转基因手段明确基因在调控果实风味中的作用;2)开展优势杂交群体桃果实糖酸遗传规律分析,利用现代高通量测序手段进行功能基因定位,为分子育种体系构建提供借鉴;3)加强外界因子特别是轻简化栽培管理方式在提升果实糖酸品质方面的应用基础研究,构建绿色安全高品质桃果实生产技术体系。
[1] 王力荣.我国桃产业现状与发展建议[J].中国果树,2021(10):1-5.WANG Lirong. Current situation and development suggestions of peach industry in China[J].China Fruits,2021(10):1-5.
[2] 叶正文,李雄伟,马亚萍,刘盼,苏明申,周京一,杜纪红.桃果实糖代谢研究进展[J].上海农业学报,2019,35(4):144-150.YE Zhengwen,LI Xiongwei,MAYaping,LIU Pan,SU Mingshen,ZHOU Jingyi,DU Jihong. Research progress of sugar metabolism in peach[J].Acta Agriculturae Shanghai,2019,35(4):144-150.
[3] DELGADO C,CRISOSTO G M,HEYMANN H,CRISOSTO C H. Determining the primary drivers of liking to predict consumers’acceptance of fresh nectarines and peaches[J]. Journal of Food Science,2013,78(4):S605-S614.
[4] 王力荣. 中国桃品种改良历史回顾与展望[J]. 果树学报,2021,38(12):2178-2195.WANG Lirong. History and prospect of peach breeding in China[J].Journal of Fruit Science,2021,38(12):2178-2195.
[5] 徐子媛,严娟,蔡志翔,孙朦,宿子文,沈志军,马瑞娟,俞明亮.桃果实糖酸和酚类物质与口感风味的相关性[J].江苏农业学报,2022,38(1):190-199.XU Ziyuan,YAN Juan,CAI Zhixiang,SUN Meng,SU Ziwen,SHEN Zhijun,MA Ruijuan,YU Mingliang. Correlation between soluble sugar,organic acid and phenolic substances with tasted flavor in peach fruit[J]. Jiangsu Journal of Agricultural Sciences,2022,38(1):190-199.
[6] 贾云云,王越辉,白瑞霞,李建明,马之胜.光照对桃果实内在品质的影响研究进展[J].江西农业学报,2020,32(12):30-36.JIA Yunyun,WANG Yuehui,BAI Ruixia,LI Jianming,MA Zhisheng. Research progress in effects of sun light on internal quality of peach fruit[J]. Acta Agriculturae Jiangxi,2020,32(12):30-36.
[7] ⅤIMOLMANGKANG S,ZHENG H Y,PENG Q,JIANG Q,WANG H L,FANG T,LIAO L,WANG L,HE H P,HAN Y P.Assessment of sugar components and genes involved in the regulation of sucrose accumulation in peach fruit[J].Journal of Agricultural and Food Chemistry,2016,64(35):6723-6729.
[8] 李依尘.11 个水蜜桃品种果实糖酸和芳香品质分析评价[D].杭州:浙江大学,2023.LI Yichen.Analysis and evaluation of fruit sugar,acid and aroma quality of 11 peach varieties[D].Hangzhou:Zhejiang University,2023.
[9] XU G X,LI C,QIN S J,XIAO W,FU X L,CHEN X D,LI L,LI D M. Changes in sucrose and sorbitol metabolism cause differences in the intrinsic quality of peach fruits cultivated in field and greenhouse environments[J].Agronomy,2022,12(11):2877.
[10] ⅤIZZOTTO G,PINTON R,ⅤARANINI Z,COSTA G. Sucrose accumulation in developing peach fruit[J]. Physiologia Plantarum,1996,96(2):225-230.
[11] 沈志军,马瑞娟,俞明亮,蔡志翔,宋宏峰,李晓.桃果实发育过程中主要糖及有机酸含量的变化分析[J].华北农学报,2007,22(6):130-134.SHEN Zhijun,MA Ruijuan,YU Mingliang,CAI Zhixiang,SONG Hongfeng,LI Xiao. Regularity analysis of main sugar and acid in fruit development of peach[J].Acta Agriculturae Boreali-Sinica,2007,22(6):130-134.
[12] ASLAM M M,DENG L,WANG X B,WANG Y,PAN L,LIU H,NIU L,LU Z H,CUI G C,ZENG W F,WANG Z Q.Expression patterns of genes involved in sugar metabolism and accumulation during peach fruit development and ripening[J]. Scientia Horticulturae,2019,257:108633.
[13] BACCICHET I,CHIOZZOTTO R,BASSI D,GARDANA C,CIRILLI M,SPINARDI A. Characterization of fruit quality traits for organic acids content and profile in a large peach germplasm collection[J].Scientia Horticulturae,2021,278:109865.
[14] ZHENG B B,ZHAO L,JIANG X H,CHERONO S,LIU J J,OGUTU C,NTINI C,ZHANG X J,HAN Y P.Assessment of organic acid accumulation and its related genes in peach[J]. Food Chemistry,2021,334:127567.
[15] JIANG X H,LIU K C,PENG H X,FANG J,ZHANG A D,HAN Y P,ZHANG X J. Comparative network analysis reveals the dynamics of organic acid diversity during fruit ripening in peach(Prunus persica L.Batsch)[J].BMC Plant Biology,2023,23(1):16.
[16] WANG Q,CAO K,LI Y,WU J L,FAN J Q,DING T Y,KHAN I A,WANG L R. Identification of co-expressed networks and key genes associated with organic acid in peach fruit[J].Scientia Horticulturae,2023,307:111496.
[17] ZHANG C H,YU M L,MA R J,SHEN Z J,ZHANG B B,KORIR N K. Structure,expression profile,and evolution of the sucrose synthase gene family in peach (Prunus persica)[J]. Acta Physiologiae Plantarum,2015,37(4):81.
[18] ZHANG C H,SHEN Z J,ZHANG Y P,HAN J,MA R J,KORIR N K,YU M L. Cloning and expression of genes related to the sucrose-metabolizing enzymes and carbohydrate changes in peach[J].Acta Physiologiae Plantarum,2013,35(2):589-602.
[19] LI C H,WANG K T,XU F,LEI C Y,JIANG Y B,ZHENG Y H.Sucrose metabolism and sensory evaluation in peach as influenced by β-aminobutyric acid (BABA)-induced disease resistance and the transcriptional mechanism involved[J]. Postharvest Biology and Technology,2021,174:111465.
[20] HE X X,WEI Y Y,KOU J Y,XU F,CHEN Z H,SHAO X F.PpVIN2,an acid invertase gene family member,is sensitive to chilling temperature and affects sucrose metabolism in postharvest peach fruit[J]. Plant Growth Regulation,2018,86(2):169-180.
[21] MOLLAH M D A,ZHANG X,ZHAO L,JIANG X H,OGUTU C O,PENG Q,BELAL M A A,YANG Q R,CAI Y M,NISHAWY E,CHERONO S,WANG L,HAN Y P.Two vacuolar invertase inhibitors PpINHa and PpINH3 display opposite effects on fruit sugar accumulation in peach[J]. Frontiers in Plant Science,2022,13:1033805.
[22] SCHWIMMER S,MAKOWER R U,ROREM E S. Invertase &invertase inhibitor in potato[J]. Plant Physiology,1961,36(3):313-316.
[23] WANG X X,CHEN Y,JIANG S,XU F,WANG H F,WEI Y Y,SHAO X F. PpINH1,an invertase inhibitor,interacts with vacuolar invertase PpⅤIN2 in regulating the chilling tolerance of peach fruit[J].Horticulture Research,2020,7:168.
[24] XU W J,WEI Y Y,WANG X X,HAN P P,CHEN Y,XU F,SHAO X F. Molecular cloning and expression analysis of hexokinase genes in peach fruit under postharvest disease stress[J].Postharvest Biology and Technology,2021,172:111377.
[25] CAO Y P,LI S M,HAN Y H,MENG D D,JIAO C Y,ABDULLAH M,LI D H,JIN Q,LIN Y,CAI Y P.A new insight into the evolution and functional divergence of FRK genes in Pyrus bretschneideri[J]. Royal Society Open Science,2018,5(7):171463.
[26] WANG L F,ZHENG X L,YE Z W,SU M S,ZHANG X N,DU J H,LI X W,ZHOU H J,HUAN C. Transcriptome co-expression network analysis of peach fruit with different sugar concentrations reveals key regulators in sugar metabolism involved in cold tolerance[J].Foods,2023,12(11):2244.
[27] GU C,WU R F,YU C Y,QI K J,WU C,ZHANG H P,ZHANG S L. Spatio-temporally expressed sorbitol transporters cooperatively regulate sorbitol accumulation in pear fruit[J].Plant Science,2021,303:110787.
[28] LI L T,LI M,WU J Y,YIN H,DUNWELL J M,ZHANG S L.Genome-wide identification and comparative evolutionary analysis of sorbitol metabolism pathway genes in four Rosaceae species and three model plants[J].BMC Plant Biology,2022,22(1):341.
[29] YAMADA K,NIWA N,SHIRATAKE K,YAMAKI S. cDNA cloning of NAD-dependent sorbitol dehydrogenase from peach fruit and its expression during fruit development[J].The Journal of Horticultural Science and Biotechnology,2001,76(5):581-587.
[30] ZHOU H J,SU M S,DU J H,ZHANG X N,LI X W,ZHANG M H,HU Y,HUAN C,YE Z W.Crucial roles of sorbitol metabolism and energy status in the chilling tolerance of yellow peach[J].Plant Physiology and Biochemistry,2023,204:108092.
[31] YU W,PENG F T,WANG W R,LIANG J H,XIAO Y S,YUAN X F. SnRK1 phosphorylation of SDH positively regulates sorbitol metabolism and promotes sugar accumulation in peach fruit[J].Tree Physiology,2021,41(6):1077-1086.
[32] ZHANG R X,LIU Y D,ZHANG X,CHEN X M,SUN J L,ZHAO Y,ZHANG J Y,YAO J L,LIAO L,ZHOU H,HAN Y P.Two adjacent NAC transcription factors regulate fruit maturity date and flavor in peach[J].New Phytologist,2024,241(2):632-649.
[33] 刘建豪,荆彦付,刘月芯,徐摇光,于洋,葛秀秀,谢华.桃NAC基因家族的鉴定及PpNAC050 促进果实果糖积累研究[J].园艺学报,2024,51(9):1983-1996.LIU Jianhao,JING Yanfu,LIU Yuexin,XU Yaoguang,YU Yang,GE Xiuxiu,XIE Hua. Identification of peach NAC gene family and role of PpNAC050 in promoting fruit fructose accumulation[J].Acta Horticulturae Sinica,2024,51(9):1983-1996.
[34] SHANG X L,ZHANG J P,MA Y H,WANG L R. Preliminary identification of candidate genes associated with the peach fruit sorbitol content based on comparative transcriptome analysis[J].Scientia Horticulturae,2020,263:109151.
[35] ZHI C,ALI M M,ALAM S M,GULL S,ALI S,YOUSEF A F,AHMED M A A,MA S F,CHEN F X. Genome-wide in silico analysis and expression profiling of phosphoenolpyruvate carboxylase genes in loquat,apple,peach,strawberry and pear[J].Agronomy,2022,12(1):25.
[36] MOING A,ROTHAN C,SⅤANELLA L,JUST D,DIAKOU P,RAYMOND P,GAUDILLÈRE J P,MONET R. Role of phosphoenolpyruvate carboxylase in organic acid accumulation during peach fruit development[J]. Physiologia Plantarum,2000,108(1):1-10.
[37] MA B Q,YUAN Y Y,GAO M,XING L B,LI C Y,LI M J,MA F W. Genome-wide identification,classification,molecular evolution and expression analysis of malate dehydrogenases in apple[J]. International Journal of Molecular Sciences,2018,19(11):3312.
[38] 寇单单,张叶,王朋飞,李东东,张学英,陈海江.‘仓方早生’桃及其早熟芽变果实蔗糖和苹果酸积累与相关基因表达[J].园艺学报,2019,46(12):2286-2298.KOU Dandan,ZHANG Ye,WANG Pengfei,LI Dongdong,ZHANG Xueying,CHEN Haijiang. Differences in sucrose and malic acid accumulation and the related gene expression in‘Kurakato Wase’peach and its early-ripening mutant[J]. Acta Horticulturae Sinica,2019,46(12):2286-2298.
[39] LOMBARDO ⅤA,OSORIO S,BORSANI J,LAUXMANN M A,BUSTAMANTE C A,BUDDE C O,ANDREO C S,LARA M Ⅴ,FERNIE A R,DRINCOⅤICH M F. Metabolic profiling during peach fruit development and ripening reveals the metabolic networks that underpin each developmental stage[J]. Plant Physiology,2011,157(4):1696-1710.
[40] 赵永,朱红菊,杨东东,龚成胜,刘文革.果实柠檬酸代谢研究进展[J].园艺学报,2022,49(12):2579-2596.ZHAO Yong,ZHU Hongju,YANG Dongdong,GONG Chengsheng,LIU Wenge. Research progress of citric acid metabolism in the fruit[J]. Acta Horticulturae Sinica,2022,49(12):2579-2596.
[41] ZANON L,FALCHI R,SANTI S,ⅤIZZOTTO G.Sucrose transport and phloem unloading in peach fruit:Potential role of two transporters localized in different cell types[J].Physiologia Plantarum,2015,154(2):179-193.
[42] PENG Q,WANG L,OGUTU C,LIU J J,LIU L,MOLLAH M D A,HAN Y P. Functional analysis reveals the regulatory role of PpTST1 encoding tonoplast sugar transporter in sugar accumulation of peach fruit[J]. International Journal of Molecular Sciences,2020,21(3):1112.
[43] 王宁,孟祥光,文滨滨,和华杰,陈修德,李玲.桃糖转运蛋白基因PpTST2 的功能初探[J]. 山东农业大学学报(自然科学版),2022,53(2):180-187.WANG Ning,MENG Xiangguang,WEN Binbin,HE Huajie,CHEN Xiude,LI Ling. Preliminary study on the function of peach sugar transporter gene PpTST2[J]. Journal of Shandong Agricultural University (Natural Science Edition),2022,53(2):180-187.
[44] YU Y,GUAN J T,XU Y G,REN F,ZHANG Z Q,YAN J,FU J,GUO J Y,SHEN Z J,ZHAO J B,JIANG Q,WEI J H,XIE H.Population-scale peach genome analyses unravel selection patterns and biochemical basis underlying fruit flavor[J]. Nature Communications,2021,12(1):3604.
[45] 贾梦霄.桃SWEET 基因家族鉴定及其蔗糖转运功能研究[D].重庆:西南大学,2021.JIA Mengxiao. Identification of members of SWEET gene family of peach and their function in sucrose transport[D]. Chongqing:Southwest University,2021.
[46] LIANG R L,WANG L J,WANG X Q,ZHANG J T,GAN X.Effects of exogenous ALA on leaf photosynthesis,photosynthate transport,and sugar accumulation in Prunus persica L.[J]. Forests,2023,14(4):723.
[47] 余彩云,谷超,张绍铃.白梨SOT 基因家族成员组织表达特性的分析[J].植物学研究,2018,7(5):496-506.YU Caiyun,GU Chao,ZHANG Shaoling.Analysis of tissue specific expression of SOT gene family in white pear[J]. Botanical Research,2018,7(5):496-506.
[48] 周平,郭瑞,颜少宾,金光.外源山梨醇影响桃叶片和果实糖代谢的分子机制研究[J].园艺学报,2023,50(5):959-971.ZHOU Ping,GUO Rui,YAN Shaobin,JIN Guang. Molecular mechanism study of exogenous sorbitol effects on sugar metabolism in peach leaves and fruits[J]. Acta Horticulturae Sinica,2023,50(5):959-971.
[49] 张立华,徐玉,郑丽桐,王长智,祝令成,马百全,李明军.酸转运蛋白与果实酸积累关系的研究进展[J].园艺学报,2024,51(7):1474-1488.ZHANG Lihua,XU Yu,ZHENG Litong,WANG Changzhi,ZHU Lingcheng,MA Baiquan,LI Mingjun. The relevance research between acid transporters and fruit acidity[J].Acta Horticulturae Sinica,2024,51(7):1474-1488.
[50] ETIENNE C,MOING A,DIRLEWANGER E,RAYMOND P,MONET R,ROTHAN C. Isolation and characterization of six peach cDNAs encoding key proteins in organic acid metabolism and solute accumulation:Involvement in regulating peach fruit acidity[J].Physiologia Plantarum,2002,114(2):259-270.
[51] XU L L,QIAO X,ZHANG M Y,ZHANG S L. Genome-wide analysis of aluminum-activated malate transporter family genes in six Rosaceae species,and expression analysis and functional characterization on malate accumulation in Chinese white pear[J].Plant Science,2018,274:451-465.
[52] WANG Q,CAO K,CHENG L L,LI Y,GUO J,YANG X W,WANG J,KHAN I A,ZHU G R,FANG W C,CHEN C W,WANG X W,WU J L,XU Q,WANG L R. Multi-omics approaches identify a key gene,PpTST1,for organic acid accumulation in peach[J].Horticulture Research,2022,9:uhac026.
[53] 朱更瑞,王新卫,曹珂,方伟超,陈昌文,王力荣.不同生态品种群桃果实糖酸及其组分含量分析[J]. 植物遗传资源学报,2017,18(5):891-904.ZHU Gengrui,WANG Xinwei,CAO Ke,FANG Weichao,CHEN Changwen,WANG Lirong. Analysis of sugars and acid contents and its compositions in different ecological peach cultivar groups[J]. Journal of Plant Genetic Resources,2017,18(5):891-904.
[54] WU B H,ZHAO J B,CHEN J,XI H F,JIANG Q,LI S H. Maternal inheritance of sugars and acids in peach [P. persica (L.)Batsch]fruit[J].Euphytica,2012,188(3):333-345.
[55] WANG L,JIANG X H,ZHAO L,WANG F R,LIU Y D,ZHOU H,HE H P,HAN Y P.A candidate PpRPH gene of the D locus controlling fruit acidity in peach[J]. Plant Molecular Biology,2021,105(3):321-332.
[56] HERNÁNDEZ-MORA J R,MICHELETTI D,BINK M,ⅤAN DE WEG E,CANTÍN C,NAZZICARI N,CAPRERA A,DETTORI M T,MICALI S,BANCHI E,CAMPOY J A,DIRLEWANGER E,LAMBERT P,PASCAL T,TROGGIO M,BASSI D,ROSSINI L,ⅤERDE I,QUILOT-TURION B,LAURENS F,ARÚS P,ARANZANA M J. Integrated QTL detection for key breeding traits in multiple peach progenies[J]. BMC Genomics,2017,18(1):404.
[57] LI X W,LIU P,ZHOU J Y,SU M S,MA Y P,JIA H J,DU J H,GAO Z S,YE Z W. Effects of exogenous application of GA4+7 and NAA on sugar accumulation and related gene expression in peach fruits during developing and ripening stages[J].Journal of Plant Growth Regulation,2021,40(3):962-973.
[58] ZHAO Y Y,SONG C C,BRUMMELL D A,QI S N,LIN Q,BI J F,DUAN Y Q.Salicylic acid treatment mitigates chilling injury in peach fruit by regulation of sucrose metabolism and soluble sugar content[J].Food Chemistry,2021,358:129867.
[59] ZHOU K X,CHENG Q,DAI J T,LIU Y,LIU Q,LI R,WANG J Y,HU R P,LIN L J. Effects of exogenous melatonin on sugar and organic acid metabolism in early-ripening peach fruits[J].PLoS One,2023,18(10):e0292959.
[60] WANG K T,LEI C Y,TAN M L,WANG J S,LI C H,ZOU Y Y. Increased soluble sugar accumulation in postharvest peaches in response to different defense priming elicitors[J]. Horticulture,Environment,and Biotechnology,2023,64(1):115-131.
[61] CAI H F,HAN S,WANG Q W,LIU X Y,YU Z F. Transcriptomic and metabolite analyses provided a new sight of 1-MCP on organic acid metabolism in peach during storage[J]. Journal of Food Science,2023,88(8):3323-3331.
[62] CHEN Y,SUN J C,WEI Y Y,CAO K F,JIANG S,SHAO X F.PpZAT10 negatively regulates peach cold resistance predominantly mediated by enhancing ⅤIN activity[J]. Postharvest Biology and Technology,2022,190:111952.
[63] CAO K F,WEI Y Y,CHEN Y,JIANG S,CHEN X Y,WANG X X,SHAO X F. PpCBF6 is a low-temperature-sensitive transcription factor that binds the PpVIN2 promoter in peach fruit and regulates sucrose metabolism and chilling injury[J].Postharvest Biology and Technology,2021,181:111681.
[64] CAO K F,ZHANG S Y,CHEN Y,YE J F,WEI Y Y,JIANG S,SHAO X F. ERF transcription factor PpRAP2.12 activates PpⅤIN2 expression in peach fruit and reduces tolerance to cold stress[J]. Postharvest Biology and Technology,2023,199:112276.
[65] ZHANG S Y,CAO K F,WEI Y Y,JIANG S,YE J F,XU F,CHEN Y,SHAO X F. PpBZR1,a BES/BZR transcription factor,enhances cold stress tolerance by suppressing sucrose degradation in peach fruit[J]. Plant Physiology and Biochemistry,2023,202:107972.
[66] WEI Y Y,MAO Y H,GAO Y L,CHEN Y,SUN J C,WANG X X,JIANG S,XU F,WANG H F,SHAO X F.The polygalacturonase-inhibiting protein PpPGIP1,positively regulates vacuolar invertase activity via a protein-protein interaction with PpⅤIN2 in peach fruit[J].Scientia Horticulturae,2023,320:112209.
[67] WANG X X,FU X L,CHEN M,HUAN L,LIU W H,QI Y H,GAO Y G,XIAO W,CHEN X D,LI L,GAO D S.Ultraviolet B irradiation influences the fruit quality and sucrose metabolism of peach (Prunus persica L.)[J]. Environmental and Experimental Botany,2018,153:286-301.
[68] ZHOU D D,ZHANG Q,LI P X,PAN L Q,TU K. Combined transcriptomics and proteomics analysis provides insight into metabolisms of sugars,organic acids and phenols in UⅤ-C treated peaches during storage[J]. Plant Physiology and Biochemistry,2020,157:148-159.
[69] PIERONI Ⅴ,OTTAⅤIANO F G,SOSA M,GABILONDO J,BUDDE C,COLLETTI A C,DENOYA G,POLENTA G,BUSTAMANTE C,MÜLLER G,PACHADO J,ANDRES S C,CARDINAL P,RODRIGUEZ G,GARITTA L. Effects of gamma irradiation on the sensory and metabolic profiles of two peach cultivars[J]. Journal of the Science of Food and Agriculture,2023,103(13):6362-6372.
[70] FONT I FORCADA C,GOGORCENA Y,MORENO M A.Fruit sugar profile and antioxidants of peach and nectarine cultivars on almond × peach hybrid rootstocks[J]. Scientia Horticulturae,2013,164:563-572.
[71] NUÑEZ C,DUPRÉ G,MUJICA K,MELET L,MEISEL L,ALMEIDAA M.Thinning alters the expression of the PpeSUT1 and PpeSUT4 sugar transporter genes and the accumulation of translocated sugars in the fruits of an early season peach variety[J].Plant Growth Regulation,2019,88(3):283-296.
[72] TOUMI I,ZARROUK O,GHRAB M,NAGAZ K. Improving peach fruit quality traits using deficit irrigation strategies in Southern Tunisia arid area[J].Plants,2022,11(13):1656.
[73] QIU Y X,ZHANG Z X,LIU D J,LIU J L,WANG Q,YU Q,ZHONG L,XIE H F. Transcriptomic insights into the development of olecranon honey peach fruits using two different planting methods[J].Agronomy,2023,13(7):1833.