甜瓜(Cucumis melo L.)是葫芦科二倍体植物(2n=2x=24),广泛栽培于全球,尤其在西方国家作为水果食用[1]。甜瓜富含维生素C、维生素A、钾和膳食纤维等营养成分,能保护心血管健康并促进消化。甜瓜含有约90%的水分,是天然的补水食物,同时具有抗氧化作用,有助于减少自由基损伤并延缓衰老。在传统医学中,甜瓜具有清热解毒、利尿等保健功效。甜瓜经济价值较高,尤其在中国新疆,是重要的生产基地。新疆甜瓜品种多样,哈密、吐鲁番、库尔勒和昌吉等地为甜瓜的主要种植区。近年来,甜瓜种植面积不断扩大,甜瓜产量占全国的三分之一。新疆甜瓜产业同质化、育种集中在少数亲本上、选育效率低等问题。对甜瓜种质资源进行遗传多样性分析,发掘重要性状基因位点可为制定遗传改良策略提供重要信息,促进品种多样性并提升市场竞争力。
甜瓜果实糖度、酸度、果肉颜色及硬度等是重要的品质特性,一直是遗传改良的关键研究领域。近年来,随着分子标记技术的发展,已经鉴定了许多与甜瓜品质相关的基因和数量性状位点(QTL),为提升甜瓜的市场品质和消费者接受度提供了理论基础和实践依据。Harel-Beja等[2]通过使用来自两个Cucumis melo L.亚种(野生亚种PI 414723和甜瓜亚种Dulce)的重组自交系群体,构建了一个富含果实性状的遗传图谱。该图谱鉴定了6个与果实糖含量(特别是主要影响甜度的蔗糖)相关的QTLs,以及3个与果实肉质颜色和类胡萝卜素含量相关的QTLs。Cohen等[3]基于Dulce(非酸性)和PI 414323(酸性)品种构建了重组自交系群体,鉴定了多个与pH、柠檬酸和苹果酸含量相关的QTLs。研究发现,一个与pH 相关的主要QTL与柠檬酸和苹果酸含量的QTL共定位,在这些QTLs中,酸性特性竟然来自非酸性亲本。
Sherman 等[4]结合大规模分离群体法与自主设计的微阵列技术,成功识别出与pH性状紧密相关的标记,且这些标记在所有种质资源中与pH性状显著关联。Cohen等[5]基于图位克隆的方法,从甜瓜中鉴定出一个对果实酸度有重大影响的特异性基因家族-C.melo pH 基因(CmpH),该基因在甜瓜品种中表现出高酸度和低酸度的自然遗传变异。通过在黄瓜和番茄中功能性沉默CmpH 的同源基因,成功获得了低酸度的果实,证明CmpH 基因调控甜瓜的果实酸度。Tzuri 等[6]发现,甜瓜果肉中类胡萝卜素的积累与CmOr基因的多态性相关联,CmOr基因与果肉颜色共同分离,且在种质资源中呈现两种单倍型:一种与橙色果肉相关,另一种则与白色或绿色果肉相关。形态学研究表明,甜瓜果肉硬度受多种因素的影响[7],其中果肉细胞大小扮演着关键角色。细胞越小,甜瓜果肉质地越趋于坚硬[8]。杨丽萍等[9]的研究表明,在花后的35~40 d 期间,是软质与脆质甜瓜质地特征形成的关键阶段。此阶段内,两种甜瓜的果肉细胞面积、细胞间隙大小以及细胞壁酶活性均表现出显著差异。进一步表达分析证实细胞壁扩展酶基因(如CmEXP3、CmEXP5、CmEXP9)在软肉与脆肉甜瓜果肉中的表达模式存在明显区别。此外,Chen 等[10]通过QTL 定位技术,成功识别出一个控制甜瓜果肉硬度的主效位点(ff2.1),进一步利用F2群体将ff2.1位点精细定位到了一个包含3个功能基因的28.3 kb 区域内。这些发现不仅加深了对甜瓜果肉硬度遗传基础的理解,也强调了深入探究与甜瓜果肉硬度相关基因的重要性,对改良甜瓜质地至关重要。
随着高通量测序技术的进步,甜瓜双单倍体品系DHL92 的基因组已完成测序,组装的N50 scaffold大小为4.68 Mb,N90指数为78[11]。然而,由于标记覆盖不足和重组缺失,部分大且富含基因的scaffold未能被锚定,且已锚定的scaffold未定向。为解决这一问题,Argyris等[12]利用DHL92亲本品系的重测序数据,从未锚定的scaffold中开发了新的SNP标记,构建了由580 个单核苷酸多态性(SNP)组成的高分辨率遗传图谱。该图谱将354.8 Mb的序列(包含141 个scaffold,平均大小为2.5 Mb)锚定至12 条甜瓜染色体,且90%的组装结果得到了定向。此外,Lyu 等[13]对来自地方品种甜瓜(Cucumis melo ssp.agrestis)样本进行了染色体级别的基因组组装,并通过整合多个已测序的甜瓜基因组数据,构建了一个甜瓜泛基因组图谱。比较基因组分析揭示了340万个遗传变异,主要为缺失/存在变异(PAV),并与蔗糖代谢相关基因的功能调控密切相关。这些研究为甜瓜群体遗传多样性分析及果肉硬度的全基因组关联分析(GWAS)提供了依据。
Zhao等[14]基于1175个甜瓜样本的重测序数据,构建了全面的基因组变异图谱,并采用GWAS方法对16 个农艺性状进行了研究,发现了208个位点与果实质量、品质和形态显著相关,验证了GWAS 在发掘关键性状遗传变异中的有效性。基于这些研究,笔者收集了292份甜瓜种质资源,其中76%来自新疆地区,旨在通过群体重测序技术进一步揭示群体基因组变异和遗传多样性特征,采用GWAS方法鉴定与果肉硬度相关的基因,以期为甜瓜的遗传改良奠定基础。
1 材料和方法
1.1 材料及表型鉴定
本研究所用甜瓜种质资源群体,由新疆哈密市农业农机技术推广服务中心提供。甜瓜果肉硬度鉴定以所取瓜中心部位为观测对象,用品尝的方法鉴定果肉质地。根据鉴定结果,参照《西瓜种质资源描述规范和数据标准》[15]做以下分类:软(果肉质地松,含水量多)、脆(果肉质地较实,含水量适中)、柔(果肉质地实,含水量较少)、硬(果肉质地实,含水量较多)。
1.2 DNA提取及基因型分析
采用CTAB 法从新鲜幼叶中提取基因组DNA[16]。每份种质至少提取5 μg基因组DNA,将提取好的DNA 送华大基因公司进行文库构建和测序。每份种质的DNA 文库构建采用约500 bp 的插入片段大小,并按照制造商说明书(Illumina Inc.,9885 Towne Centre Drive, San Diego, CA 92121,USA)进行操作。所有种质均使用Illumina HiSeq 2500测序仪进行测序。
对下机数据Raw reads进行过滤,去除接头和低质量的Reads,以此保证信息分析质量。将过滤后的Clean reads 应用于后续分析。使用比对软件BWA[17]将Reads比对到参考基因组上。进一步使用SAMtools 过滤掉非唯一和未比对上的Reads[18]。Picard 包(http://broadinstitute.github.io/picard/,version:1.87)被用来过滤重复Reads。基因组分析工具包GATK被用来检测全基因组SNP变异[19]。在群体中等位基因频率低于1%、缺失率大于80%、杂合位点频率大于5%的SNP被剔除。
1.3 遗传多样性和群体结构分析
使用邻接法计算种质群体距离矩阵,构建系统发育树,并在MEGA 5.0[20]中展示。使用PLINK软件(版本1.90)计算连锁不平衡(LD),参数设置为–ldwindow-r2 0、–ld-window 99999、–ld-window-kb 1000。
1.4 全基因组关联分析和单倍型分析
仅使用群体中最小等位基因频率(MAF)≥0.05且缺失率<0.1 的SNP 进行全基因组关联分析。使用EMMAX(版本beta)软件包基于混合线性模型进行关联分析。关联分析结果可视化采用qqman包进行[21]。通过Bonferroni 测试的显著性阈值(1/SNP 数目)定义了全基因组的显著性临界值。采用gene-HapR包进行单倍型及可视化分析[22]。
2 结果与分析
2.1 甜瓜种质群体基因分型
本研究聚焦于292份来自全球多个区域的甜瓜种质资源,包含157 个地方品种和135 个栽培品种,涵盖的分布区域包括中国、加拿大、美国、南非、苏联、日本、土耳其、匈牙利、印度和伊朗等,其中约76%的样本来源于中国新疆。利用新一代高通量测序技术,成功获得了3.57 T碱基的序列数据,平均每个样本的测序量为12.25 Gb,且平均测序深度为33 X,已覆盖甜瓜基因组的33 倍[12]。通过将测序数据读取比对至甜瓜参考基因组[12],共识别了2 280 089 个SNP 标记(图1-A~B),平均每千碱基包含6.3 个SNP,显示出该群体中广泛的基因组变异。通过这一大规模基因组数据集的构建,将有助于甜瓜遗传多样性研究、关键性状基因的发掘以及群体遗传结构的深入解析。

图1 甜瓜群体SNP 基因分型及遗传多样性分析
Fig.1 SNP genotyping and genetic diversity analysis of melon population
2.2 甜瓜种质群体结构和连锁不平衡分析
基于全基因组SNP 数据,构建了甜瓜种质群体的系统发育树(图1-C),发现该群体可分为两个显著的分支:分支Ⅰ(Pop1)和分支Ⅱ(Pop2)。Pop1主要聚集了来自中国新疆、日本和苏联的地方品种,其中新疆地区的地方品种占绝大多数(90.84%,图2-A)。而Pop2 主要由栽培品种组成(图2-B),这些栽培品种分布广泛,包括中国(新疆、陕西、台湾、甘肃、内蒙古、辽宁)、日本、苏联、美国、加拿大、伊朗、土耳其、匈牙利、南非和印度等国家,其中新疆的栽培品种占67.5%。种质资源群体按果肉硬度性状可以分为脆、柔、软、硬4种,其中以脆和软为主(图2-C)。这一群体结构揭示了甜瓜种质在地理栽培环境以及果肉硬度上的高度分化,反映了地方品种和栽培品种之间的显著遗传差异,尤其是新疆甜瓜可能具有多样化的地理来源和果肉性状变异。
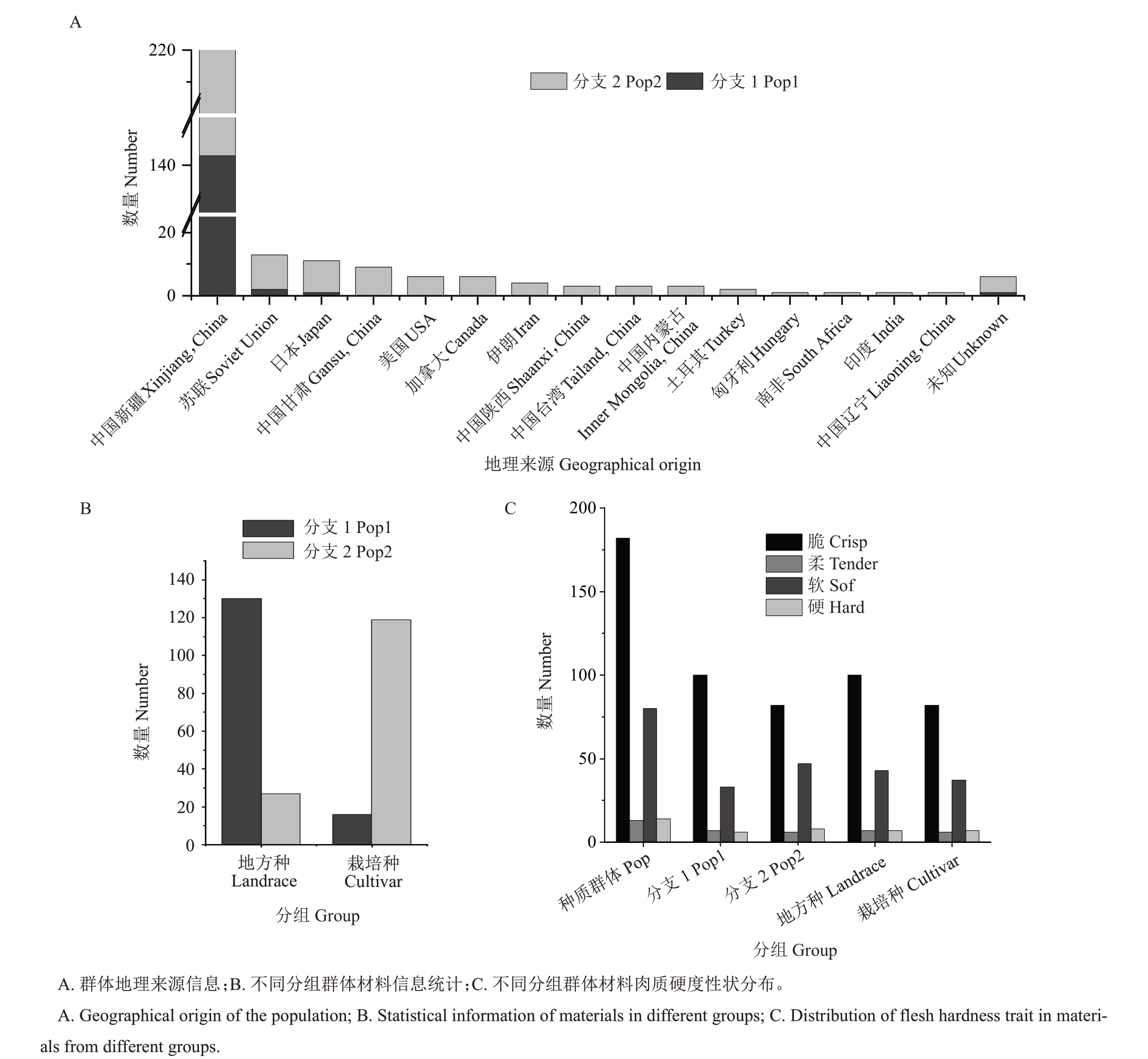
图2 甜瓜群体特征信息及结构分析
Fig.2 Characteristics and structure analysis of the melon population
全基因组LD 分析进一步表明,整个甜瓜种质群体的LD 在衰减至一半(r²=0.33)时,标记对之间的距离为49.2 kb(图3)。然而,当分析地方品种和栽培品种时,LD 衰减至一半的距离分别为57.0 kb和45.9 kb,显示出栽培品种的LD 衰减速率较地方品种更快,提示栽培品种经历了更大的选择压力和遗传漂变。此外,核酸多态性分析表明,栽培品种的核酸多态性(2.537e-3)显著(p<0.01)高于地方品种(1.989e-3),进一步证明了栽培品种在遗传多样性上的快速演化。
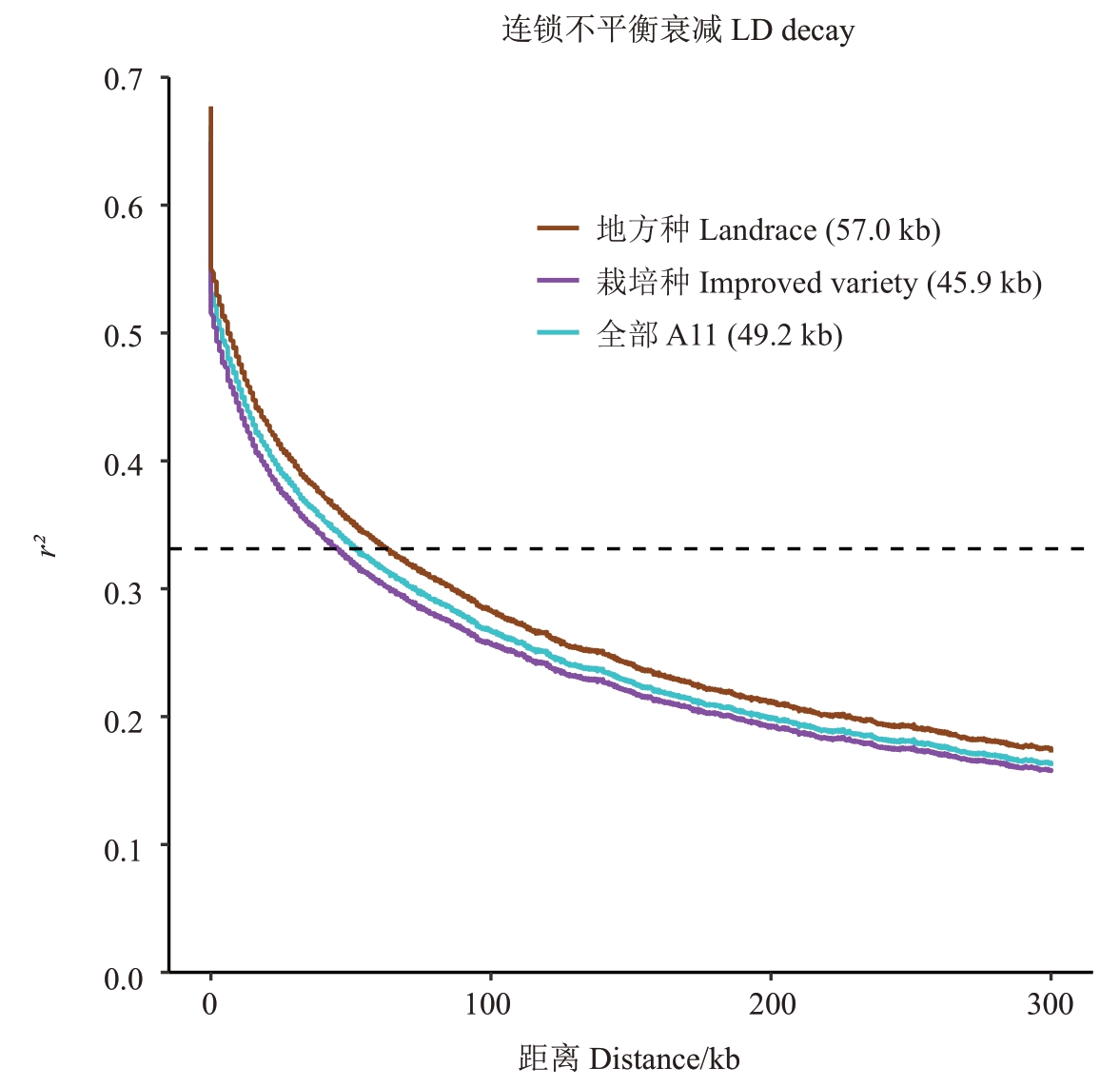
图3 不同分组材料的连锁不平衡分析
Fig.3 Linkage disequilibrium analysis of materials from different groups
2.3 甜瓜种质群体果肉硬度变异及GWAS分析
基于表型性状(表1)和基因型数据,在甜瓜基因组的8 号染色体上发现了一个显著的关联信号(Chr08:2815957~2831112;p=2.36e-07,图4-A~B)。该区域包含11个显著的关联SNPs,能够解释9.04%的表型变异。
表1 群体不同单倍型肉质性状表型分布
Table 1 Phenotypic distribution of flesh hardness trait in different haplotypes of the population

注:NA表示缺失。
Note:NA means the date is missing.
编号ID 地理来源Location性状Trait 单倍型Haplotype编号ID 地理来源Location性状Trait 单倍型Haplotype s81 s83 s95 s96 s101 s104 s111 s120 s269 s273 s139 s97 s75 s134 s129 s92 s118 s103 s57 s71 s65 s136 s131 s280 s119 s281 s98 s94 s85 s132 s259 s275 s89 s90 s133 s130 s99 s87 s100 s252 s182 s93 s283 s278 s105日本Japan中国台湾Tailand,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国台湾Tailand,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国陕西Shaanxi,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China苏联Soviet Union中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国甘肃Gansu,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China软Soft软Soft脆Crisp脆Crisp脆Crisp脆Crisp脆Crisp脆Crisp脆Crisp脆Crisp脆Crisp硬Hard软Soft脆Crisp脆Crisp脆Crisp软Soft脆Crisp脆Crisp硬Hard脆Crisp柔Tender脆Crisp脆Crisp脆Crisp脆Crisp脆Crisp脆Crisp软Soft脆Crisp脆Crisp软Soft脆Crisp软Soft脆Crisp脆Crisp脆Crisp脆Crisp柔Tender脆Crisp柔Tender脆Crisp脆Crisp脆Crisp脆Crisp单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1 s164 s106 s151 s159 s160 s91 s50 s168 s276 s153 s174 s56 s257 s192 s222 s152 s202 s217 s8 s274 s270 s24 s162 s150 s4 s140 s169 s9 s163 s58 s69 s21 s142 s48 s138 s72 s224 s228 s149 s147 s122 s1 s27 s144 s253伊朗Iran中国新疆Xinjiang,China未知Unknown苏联Soviet Union苏联Soviet Union中国新疆Xinjiang,China中国新疆Xinjiang,China日本Japan中国新疆Xinjiang,China中国新疆Xinjiang,China加拿大Canada中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国内蒙古Inner Mongolia,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China日本Japan中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China美国USA中国新疆Xinjiang,China苏联Soviet Union中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国陕西Shaanxi,China中国新疆Xinjiang,China中国新疆Xinjiang,China苏联Soviet Union中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China软Soft脆Crisp脆Crisp脆Crisp脆Crisp脆Crisp脆Crisp柔Tender脆Crisp软Soft软Soft脆Crisp脆Crisp柔Tender软Soft柔Tender软Soft软Soft软Soft软Soft脆Crisp软Soft柔Tender脆Crisp软Soft硬Hard软Soft脆Crisp脆Crisp脆Crisp脆Crisp脆Crisp硬Hard软Soft脆Crisp软Soft脆Crisp软Soft NA脆Crisp脆Crisp软Soft硬Hard脆Crisp脆Crisp单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1
表1 (续) Table 1 (Continued)
编号ID 地理来源Location性状Trait 单倍型Haplotype编号ID 地理来源Location性状Trait 单倍型Haplotype s241 s60 s107 s282 s156 s135 s284 s84 s30 s23 s237 s143 s73 s62 s88 s246 s245 s173 s137 s197 s271 s167 s146 s110 s213 s223 s227 s32 s158 s124 s42 s292 s236 s254 s31 s255 s194 s178 s59 s54 s279 s161 s49 s206 s29 s285 s235 s35中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China苏联Soviet Union中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China美国USA中国新疆Xinjiang,China土耳其Turkey中国新疆Xinjiang,China日本Japan中国新疆Xinjiang,China中国新疆Xinjiang,China未知Unknown中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China苏联Soviet Union中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China印度India未知Unknown中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China苏联Soviet Union中国新疆Xinjiang,China苏联Soviet Union中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China脆Crisp脆Crisp脆Crisp软Soft脆Crisp脆Crisp脆Crisp软Soft脆Crisp脆Crisp脆Crisp脆Crisp脆Crisp脆Crisp软Soft脆Crisp脆Crisp软Soft软Soft脆Crisp脆Crisp柔Tender脆Crisp脆Crisp软Soft软Soft软Soft软Soft脆Crisp脆Crisp脆Crisp脆Crisp硬Hard脆Crisp软Soft脆Crisp软Soft软Soft脆Crisp软Soft脆Crisp脆Crisp脆Crisp脆Crisp脆Crisp脆Crisp脆Crisp脆Crisp单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1 s226 s230 s39 s14 s200 s102 s66 s67 s179 s188 s176 s114 s177 s214 s121 s287 s128 s70 s77 s195 s165 s216 s181 s64 s268 s295 s296 s55 s231 s61 s20 s220 s52 s126 s16 s240 s7 s11 s13 s18 s19 s22 s36 s41 s43 s44 s46 s47中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China未知Unknown日本Japan中国新疆Xinjiang,China中国新疆Xinjiang,China中国内蒙古Inner Mongolia,China未知Unknown中国新疆Xinjiang,China中国内蒙古Inner Mongolia,China中国新疆Xinjiang,China中国陕西Shaanxi,China日本Japan中国新疆Xinjiang,China加拿大Canada中国甘肃Gansu,China中国甘肃Gansu,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China美国USA中国新疆Xinjiang,China中国新疆Xinjiang,China软Soft脆Crisp脆Crisp软Soft柔Tender脆Crisp硬Hard脆Crisp软Soft脆Crisp脆Crisp脆Crisp软Soft软Soft软Soft软Soft脆Crisp软Soft脆Crisp柔Tender脆Crisp软Soft NA脆Crisp脆Crisp脆Crisp脆Crisp脆Crisp脆Crisp脆Crisp脆Crisp脆Crisp脆Crisp软Soft脆Crisp脆Crisp软Soft脆Crisp软Soft脆Crisp脆Crisp脆Crisp脆Crisp脆Crisp脆Crisp脆Crisp软Soft脆Crisp单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1
表1 (续) Table 1 (Continued)

编号ID 地理来源Location性状Trait 单倍型Haplotype编号ID 地理来源Location性状Trait 单倍型Haplotype s199 s286 s127 s261 s68 s300 s297 s290 s51 s289 s145 s141 s28 s248 s25 s242 s5 s299 s2 s264 s294 s201 s26 s17 s251 s113 s250 s33 s63 s249 s244 s37 s109 s34 s12 s115 s117 s229 s211 s10 s116 s15 s232 s265 s108 s247中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China苏联Soviet Union中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China脆Crisp脆Crisp软Soft脆Crisp脆Crisp软Soft脆Crisp脆Crisp硬Hard软Soft柔Tender脆Crisp脆Crisp脆Crisp软Soft脆Crisp软Soft脆Crisp软Soft脆Crisp脆Crisp脆Crisp软Soft脆Crisp脆Crisp脆Crisp脆Crisp软Soft脆Crisp脆Crisp脆Crisp脆Crisp软Soft软Soft脆Crisp脆Crisp脆Crisp脆Crisp脆Crisp脆Crisp脆Crisp脆Crisp脆Crisp脆Crisp柔Tender脆Crisp单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1 s53 s112 s123 s207 s208 s209 s210 s225 s233 s243 s256 s258 s262 s263 s266 s267 s291 s293 s80 s78 s170 s189 s171 s74 s212 s183 s205 s203 s238 s148 s218 s219 s239 s190 s184 s221 s172 s185 s166 s191 s215 s76 s180 s157 s196 s38中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China苏联Soviet Union中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China中国新疆Xinjiang,China伊朗Iran日本Japan美国USA日本Japan美国USA中国新疆Xinjiang,China伊朗Iran中国甘肃Gansu,China苏联Soviet Union加拿大Canada中国新疆Xinjiang,China中国新疆Xinjiang,China加拿大Canada日本Japan中国新疆Xinjiang,China日本Japan中国甘肃Gansu,China中国新疆Xinjiang,China美国USA中国甘肃Gansu,China南非South Africa日本Japan中国甘肃Gansu,China匈牙利Hungary未知Unknown加拿大Canada中国新疆Xinjiang,China中国新疆Xinjiang,China脆Crisp脆Crisp软Soft脆Crisp脆Crisp脆Crisp脆Crisp软Soft脆Crisp脆Crisp脆Crisp脆Crisp脆Crisp脆Crisp脆Crisp脆Crisp脆Crisp脆Crisp软Soft硬Hard软Soft软Soft软Soft软Soft脆Crisp软Soft软Soft软Soft脆Crisp NA软Soft硬Hard软Soft脆Crisp硬Hard硬Hard硬Hard软Soft软Soft脆Crisp脆Crisp软Soft软Soft软Soft脆Crisp脆Crisp单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型1 Hap1单倍型2 Hap2单倍型2 Hap2单倍型2 Hap2单倍型2 Hap2单倍型2 Hap2单倍型2 Hap2单倍型2 Hap2单倍型2 Hap2单倍型2 Hap2单倍型2 Hap2单倍型2 Hap2单倍型2 Hap2单倍型2 Hap2单倍型2 Hap2单倍型2 Hap2单倍型3 Hap3单倍型3 Hap3单倍型3 Hap3单倍型3 Hap3单倍型3 Hap3单倍型3 Hap3单倍型3 Hap3单倍型3 Hap3单倍型4 Hap4单倍型4 Hap4单倍型4 Hap4单倍型4 Hap4单倍型1 Hap1
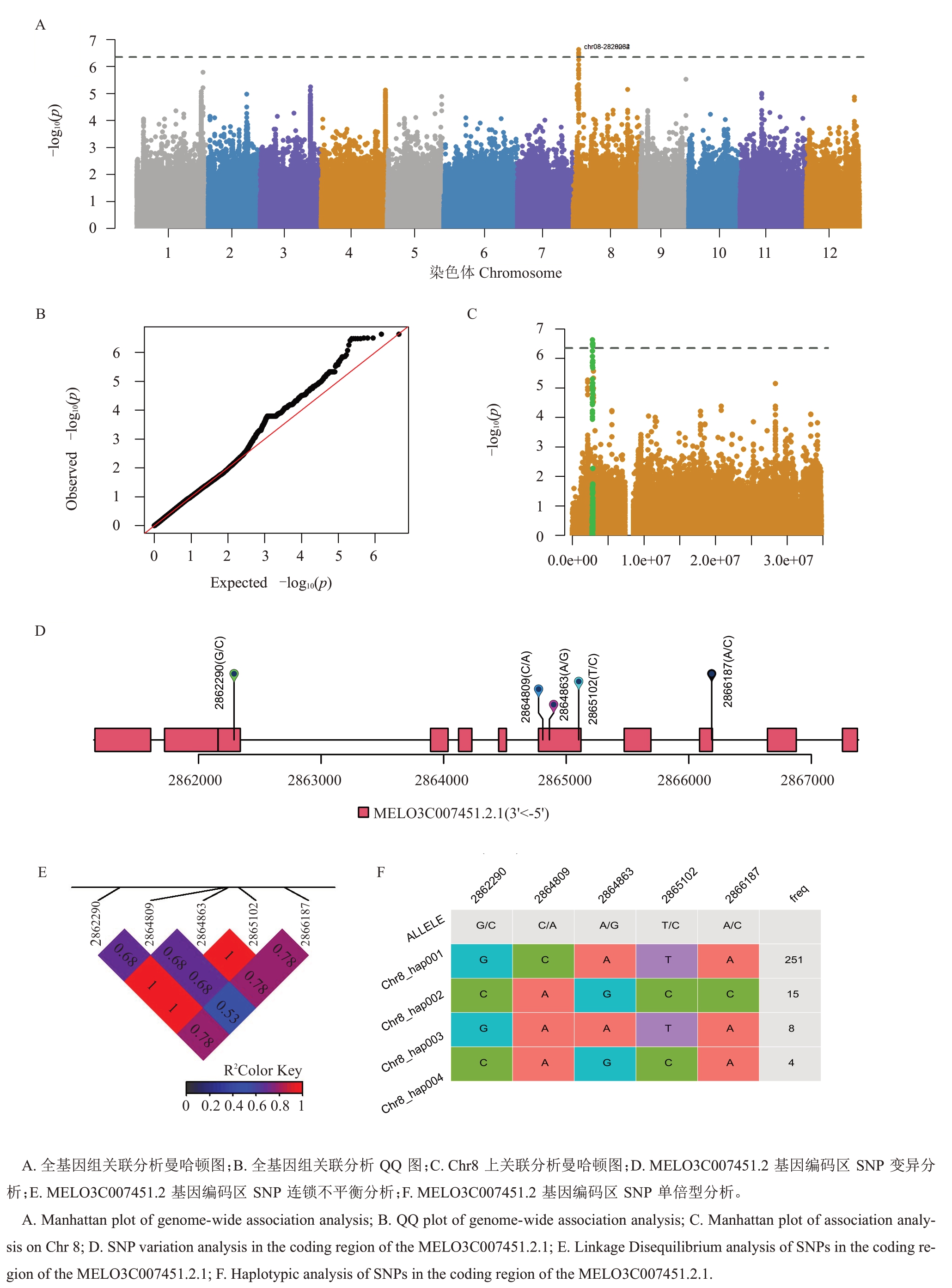
图4 群体肉质性状全基因组关联分析
Fig.4 Genome-wide association analysis of flesh hardness trait in the melon population
2.4 果肉硬度相关候选基因鉴定及单倍型分析
为了探究肉质相关的候选基因,参考群体LD衰减距离,笔者在最强关联SNP(chr08-2828062)上下游50 kb范围(Chr08:2768094~2867376)内共鉴定到了18个基因。基因组注释信息表明,在该关联区域鉴定到的基因主要为编码蛋白基因和酶,如硫酸腺苷转移酶(MELO3C007436.2),纤维鞘CABYR结合蛋白(MELO3C007438.2),肌醇转运蛋白(MELO3C007442.2),花粉绒毡层决定因子1 蛋白(MELO3C007447.2),核苷酸- 糖转运蛋白(MELO3C007449.2),E3 泛素蛋白连接酶(MELO3C007439.2) ,肌 醇 单 磷 酸 酶(MELO3C007440.2),环形E3 泛素转移酶(MELO3C007441.2),双功能3-脱氢奎尼酸脱水酶/莽草酸脱氢酶(MELO3C007451.2)等。转录组分析表明,有9个编码蛋白或酶基因在绿瓤和黄瓤的种质果肉中高表达(图5)。因此,推测这些蛋白和酶可能通过调控关键代谢生物合成路径来影响果肉硬度。
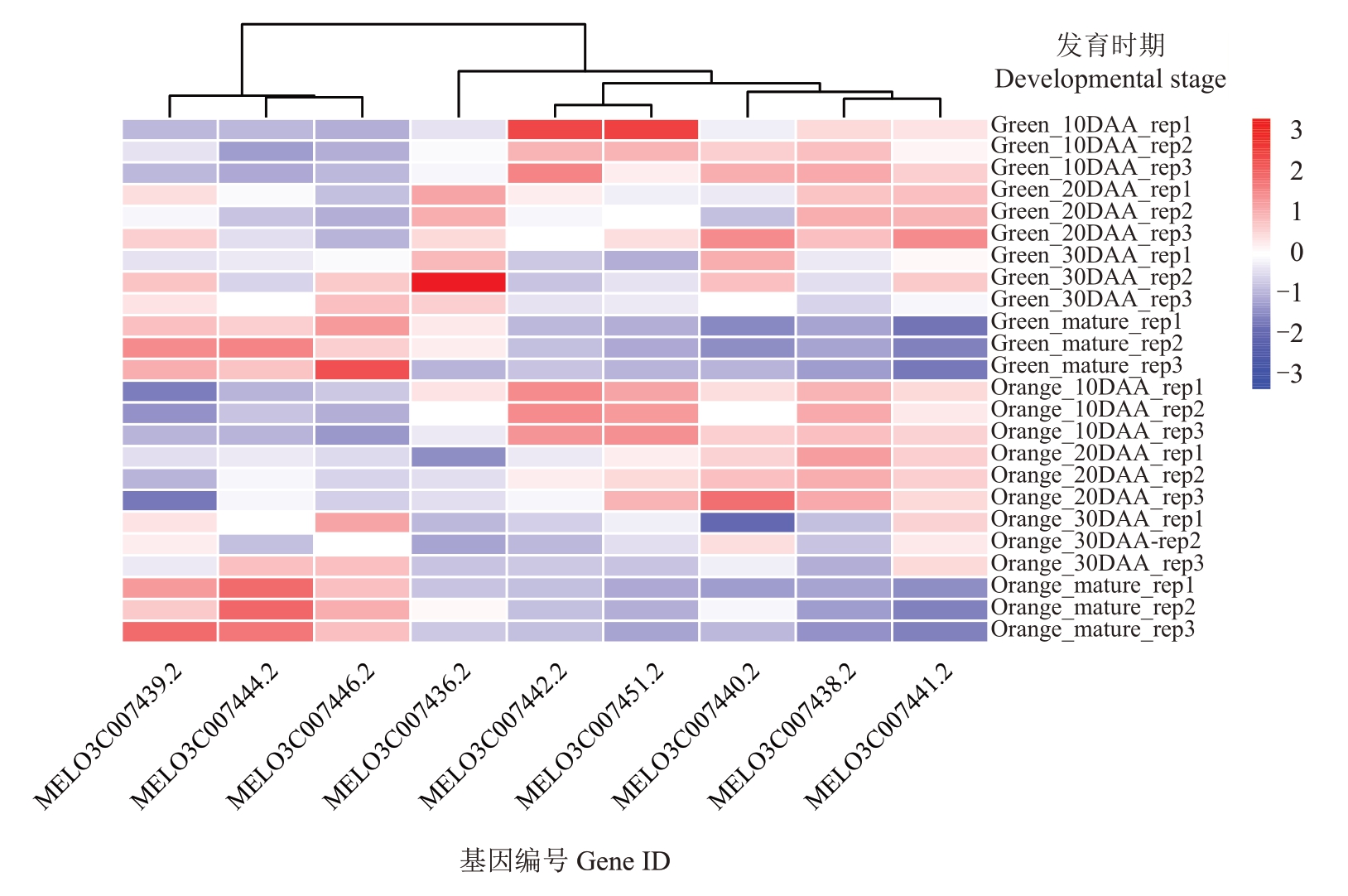
图5 候选基因在果肉发育过程中的表达分析
Fig.5 Expression analysis of candidate genes during flesh development
肌醇单磷酸酶(MELO3C007440.2)位于显著关联SNP(Chr08:2820234)上游24.98 kb 处,主要调控肌醇代谢,参与植物细胞壁多糖(尤其是果胶)合成和重塑[23]。显著关联SNP(Chr08:2830666)下游14 kb处鉴定到一个编码核苷酸-糖转运蛋白基因MELO3C007449.2。核苷酸糖是细胞壁多糖(包括果胶和半纤维素)合成的关键底物[24],MELO3C007449.2有可能负责核苷酸糖从胞质转运到高尔基体,在细胞壁多糖合成中发挥重要作用。显著关联SNP(Chr08:2830666)下游30.49 kb 处鉴定到一个双功能3-脱氢奎尼酸脱水酶/莽草酸脱氢酶合成基因MELO3C007451.2(图4-C)。莽草酸途径是合成芳香族氨基酸的关键代谢通路,这些氨基酸是木质素和其他细胞壁相关次生代谢物的前体[25],推测MELO3C007451.2 可能参与细胞壁强化和重塑过程。在MELO3C007451.2 编码区存在5 个SNPs,这些SNPs 处于强连锁不平衡水平,可以将群体划分为4个单倍型(图4-D~E)。98.05%的地方品种都享有hap001 单倍型。hap004 单倍型材料表现为软和脆,而硬质材料享有hap001,hap002 和hap003 单倍型(图4-F)。这些候选基因和单倍型为研究甜瓜种质资源群体分化和表型变异提供了新的视角。
3 讨 论
甜瓜是一种重要的经济果蔬作物,已经栽培了几千年。甜瓜作为一种形态多样的异交物种,普遍认为其起源于非洲[26-27]。Sebastian等[28]研究表明,甜瓜和黄瓜实际上都起源于亚洲。Zhao 等[14]研究表明,甜瓜经历了3次独立的驯化事件,其中两次发生在印度,一次发生在非洲。Luan等[29]评估了来自印度、非洲、克里特岛/希腊、日本、欧洲、美国、西班牙和中国68个栽培品种的遗传多样性,证实中国的甜瓜品种是甜瓜改良的丰富遗传多样性来源。笔者通过对292份来自全球多个区域的甜瓜种质资源进行高通量测序,成功构建了一个大规模的遗传变异图谱,为甜瓜基因组学研究和育种提供了数据支持。研究结果表明,甜瓜种质群体可分为两个显著的分支(Pop1和Pop2),其中Pop1主要由地方品种组成,Pop2则由栽培品种占主导。地理来源上分析发现,来自新疆的种质群体分散在了两个不同的亚群。来自美国、印度的资源和中国资源分布在了Pop2 中,支持了Luan等[29]的结论,即美国品种资源与中国厚皮甜瓜在形态学性状上非常相似。这一群体结构的差异反映了甜瓜在不同地理区域和栽培环境中的适应性演化,进一步加深了对甜瓜驯化和遗传演化的理解,尤其是新疆甜瓜的独特性,可能揭示了该地区丰富的地理多样性和栽培历史。
通过全基因组关联分析,笔者首次在甜瓜基因组中识别了与果肉硬度相关的重要SNP位点,并发现这些位点位于8 号染色体上的一个显著区域(Chr08:2815957~2831112)。该区域上下游50 kb区域内包含18 个基因,其中MELO3C007434、MELO3C007435、MELO3C007436、MELO3C007437、MELO3C007438、MELO3C007439、MELO3C007440、MELO3C007441 被证实为受选择基因,与野生种和栽培种分化相关[14]。笔者猜测这些基因可能在果肉硬度方面从野生种到栽培种驯化过程中扮演了重要角色。进一步基因注释结果表明,这些关联基因可能通过影响植物细胞壁的合成与重塑过程,进而调控果肉硬度性状。 肌醇单磷酸酶(MELO3C007440.2)和核苷酸- 糖转运蛋白(MELO3C007449.2)等候选基因,参与了果胶和细胞壁多糖的合成,可能是影响果肉硬度的重要调控因子。该研究结果和曾文芳等[30]的综述结论一致,即果实质地软化主要由细胞壁结构的改变和细胞壁组分的降解所引起。GWAS结合转录组分析数据进一步揭示了影响果肉硬度的细胞壁酶基因,特别是双功能3-脱氢奎尼酸脱水酶/莽草酸脱氢酶合成基因MELO3C007451.2。对口感不同的甜瓜材料细胞壁酶活性的研究结果表明,果实质地的变化受多个细胞壁扩展酶基因共同调控,并且花后35 d为细胞壁扩展酶基因表达的关键时期[9],进一步支持了笔者的结论。因此,基于GWAS 结果,不仅有利于揭示果肉硬度的遗传基础,还为后续的分子机制研究提供了新的视角。这些发现为未来甜瓜育种中果肉硬度等性状的分子标记辅助选择提供了有力支持。
此外,笔者在群体中还发现了多个与果肉硬度相关的单倍型,这些单倍型的差异与果肉质地的表型变异密切相关。特别是在硬质和软质果肉的样本中,单倍型分化表现出明显的关联,突出了基因型与表型之间的联系。值得注意的是,硬质甜瓜普遍携带特定的单倍型(如hap001),而软质或脆性品种则主要表现为hap004等单倍型。这一结果为揭示甜瓜果肉硬度的遗传背景提供了有价值的线索,也为后续通过单倍型选择来进行品种改良提供了重要依据。
随着基因组学、表型分析和育种技术的持续发展,甜瓜在品质改良方面将迎来更加精准和高效的育种方法。通过深入解析甜瓜基因组的结构变异、PAVs以及功能变异,有望发现更多与果肉硬度等品质性状相关的关键基因。结合现代技术,如基因编辑和基因组选择,可以加速甜瓜品种的改良,特别是在提高消费者偏好的品质方面。
4 结 论
笔者研究分析了新疆甜瓜种质资源的遗传多样性,定位了与甜瓜果肉硬度相关的候选基因,发掘了与果肉硬度相关单倍型,为甜瓜果肉硬度及其他品质性状的遗传改良提供了新的分子标记,并为甜瓜分子标记辅助选择育种奠定了基础。
[1] BURGER Y,PARIS H,COHEN R,KATZIR N,TADMOR Y,LEWINSOHN E,SCHAFFER A. Genetic diversity of Cucumis melo[J].Horticultural Reviews,2009,36:165-198.
[2] HAREL-BEJA R,TZURI G,PORTNOY V,LOTAN-POMPAN M,LEV S,COHEN S,DAI N,YESELSON L,MEIR A,LIBHABER S E,AVISAR E,MELAME T,VAN KOERT P,VERBAKEL H,HOFSTEDE R,VOLPIN H,OLIVER M,FOUGEDOIRE A,STALH C,FAUVE J,COPES B,FEI Z,GIOVANNONI J,ORI N,LEWINSOHN E,SHERMAN A,BURGER J,TADMOR Y,SCHAFFER A A,KATZIR N. A genetic map of melon highly enriched with fruit quality QTLs and EST markers,including sugar and carotenoid metabolism genes[J]. Theoretical and Applied Genetics,2010,121(3):511-533.
[3] COHEN S,TZURI G,HAREL-BEJA R,ITKIN M,PORTNOY V,SA’AR U,LEV S,YESELSON L,PETRIKOV M,ROGACHEV I,AHARONI A,OPHIR R,TADMOR Y,LEWINSOHN E,BURGER Y,KATZIR N,SCHAFFER A A. Comapping studies of QTLs for fruit acidity and candidate genes of organic acid metabolism and proton transport in sweet melon(Cucumis melo L.)[J]. Theoretical and Applied Genetics,2012,125(2):343-353.
[4] SHERMAN A,ESHED R,HAREL-BEJA R,TZURI G,PORTNOY V,COHEN S,RUBINSTEIN M,SCHAFFER A A,BURGER J,KATZIR N,OPHIR R. Combining bulk segregation analysis and microarrays for mapping of the pH trait in melon[J].Theoretical and Applied Genetics,2013,126(2):349-358.
[5] COHEN S,ITKIN M,YESELSON Y,TZURI G,PORTNOY V,HAREL-BAJA R,LEV S,SA’AR U,DAVIDOVITZ-RIKANATI R,BARANES N,BAR E,WOLF D,PETREIKOV M,SHEN S,BEN-DOR S,ROGACHEV I,AHARONI A,AST T,SCHULDINER M,BELAUSOV E,ESHED R,OPHIR R,SHERMAN A,FREI B,NEUHAUS H E,XU Y M,FEI Z J,GIOVANNONI J,LEWINSOHN E,TADMOR Y,PARIS H S,KATZIR N,BURGER Y,SCHAFFER A A.The PH gene determines fruit acidity and contributes to the evolution of sweet melons[J].Nature Communications,2014,5:4026.
[6] TZURI G,ZHOU X J,CHAYUT N,YUAN H,PORTNOY V,MEIR A,SA’AR U,BAUMKOLER F,MAZOUREK M,LEWINSOHN E,FEI Z J,SCHAFFER A A,LI L,BURGER J,KATZIR N,TADMOR Y. A‘Golden’SNP in CmOr governs the fruit flesh color of melon(Cucumis melo)[J].The Plant Journal,2015,82(2):267-279.
[7] 潘好斌,刘东,邵青旭,高歌,齐红岩.不同品种薄皮甜瓜成熟期果实质地品质分析及综合评价[J].食品科学,2019,40(21):35-42.PAN Haobin,LIU Dong,SHAO Qingxu,GAO Ge,QI Hongyan.Analysis and comprehensive evaluation of textural quality of ripe fruits from different varieties of oriental melon (Cucumis melo var. makuwa Makino)[J]. Food Science,2019,40(21):35-42.
[8] 李三培,华德平,高星,徐伟欣,杨旭辉,刘莉.不同类型甜瓜成熟过程中果肉质地及其细胞显微结构的变化[J].西北植物学报,2017,37(6):1118-1125.LI Sanpei,HUA Deping,GAO Xing,XU Weixin,YANG Xuhui,LIU Li. Variation characteristics of flesh texture and cell microstructure of different types of melon during ripening[J].Acta Botanica Boreali-Occidentalia Sinica,2017,37(6):1118-1125.
[9] 杨丽萍,马荣雪,周永海,程登虎,魏春华,张显,张勇.甜瓜质地差异及相关细胞壁酶活性变化[J].中国瓜菜,2020,33(5):12-17.YANG Liping,MA Rongxue,ZHOU Yonghai,CHENG Denghu,WEI Chunhua,ZHANG Xian,ZHANG Yong. Study on the difference of melon texture and related cell wall enzyme[J].China Cucurbits and Vegetables,2020,33(5):12-17.
[10] CHEN K X,DAI D Y,WANG L,YANG L M,LI D D,WANG C,JI P,SHENG Y Y.SLAF marker based QTL mapping of fruitrelated traits reveals a major-effect candidate locus ff2.1 for flesh firmness in melon[J]. Journal of Integrative Agriculture,2023,22(11):3331-3345.
[11] GARCIA-MAS J,BENJAK A,SANSEVERINO W,BOURGEOIS M,MIR G,GONZÁLEZ V M,HÉNAFF E,CÂMARA F,COZZUTO L,LOWY E,ALIOTO T,CAPELLA- GUTIÉRREZ S,BLANCA J,CAÑIZARES J,ZIARSOLO P,GONZALEZ-IBEAS D,RODRÍGUEZ-MORENO L,DROEGE M,DU L,ALVAREZ- TEJADO M,LORENTE- GALDOS B,MELÉ M,YANG L M,WENG Y Q,NAVARRO A,MARQUES-BONET T,ARANDA M A,NUEZ F,PICÓ B,GABALDÓN T,ROMA G,GUIGÓ R,CASACUBERTA J M,ARÚS P,PUIGDOMÈNECH P. The genome of melon (Cucumis melo L.)[J].Proceedings of the National Academy of Sciences of the United States of America,2012,109(29):11872-11877.
[12] ARGYRIS J M,RUIZ-HERRERA A,MADRIZ-MASIS P,SANSEVERINO W,MORATA J,PUJOL M,RAMOS-ONSINS S E,GARCIA-MAS J. Use of targeted SNP selection for an improved anchoring of the melon(Cucumis melo L.)scaffold genome assembly[J].BMC Genomics,2015,16(1):4.
[13] LYU X L,XIA Y L,WANG C H,ZHANG K J,DENG G C,SHEN Q H,GAO W,ZHANG M Y,LIAO N Q,LING J,BO Y M,HU Z Y,YANG J H,ZHANG M F. Pan-genome analysis sheds light on structural variation-based dissection of agronomic traits in melon crops[J]. Plant Physiology,2023,193(2):1330-1348.
[14] ZHAO G W,LIAN Q,ZHANG Z H,FU Q S,HE Y H,MA S W,RUGGIERI V,MONFORTE A J,WANG P Y,JULCA I,WANG H S,LIU J P,XU Y,WANG R Z,JI J B,XU Z H,KONG W H,ZHONG Y,SHANG J L,PEREIRA L,ARGYRIS J,ZHANG J,MAYOBRE C,PUJOL M,OREN E,OU D D,WANG J M,SUN D X,ZHAO S J,ZHU Y C,LI N,KATZIR N,GUR A,DOGIMONT C,SCHAEFER H,FAN W,BENDAHMANE A,FEI Z J,PITRAT M,GABALDÓN T,LIN T,GARCIA-MAS J,XU Y Y,HUANG S W.A comprehensive genome variation map of melon identifies multiple domestication events and loci influencing agronomic traits[J].Nature Genetics,2019,51(11):1607-1615.
[15] 马双武,刘君璞.西瓜种质资源描述规范和数据标准[M].北京:中国农业出版社,2005.MA Shuangwu,LIU Junpu. Descriptors and data standard for watermelon[M].Beijing:China Agriculture Press,2005.
[16] MURRAY M G,THOMPSON W F.Rapid isolation of high molecular weight plant DNA[J]. Nucleic Acids Research,1980,8(19):4321-4326.
[17] LI H,DURBIN R. Fast and accurate short read alignment with Burrows-Wheeler transform[J]. Bioinformatics,2009,25(14):1754-1760.
[18] LI H,HANDSAKER B,WYSOKER A,FENNELL T,RUAN J,HOMER N,MARTH G,ABECASIS G,DURBIN R,1000 Genome Project Data Processing Subgroup. The sequence alignment/map format and SAMtools[J]. Bioinformatics,2009,25(16):2078-2079.
[19] MCKENNA A,HANNA M,BANKS E,SIVACHENKO A,CIBULSKIS K,KERNYTSKY A,GARIMELLA K,ALTSHULER D,GABRIEL S,DALY M,DEPRISTO M A.The genome analysis toolkit:A mapReduce framework for analyzing next-generation DNA sequencing data[J]. Genome Research,2010,20(9):1297-1303.
[20] TAMURA K,PETERSON D,PETERSON N,STECHER G,NEI M,KUMAR S. MEGA5:Molecular evolutionary genetics analysis using maximum likelihood,evolutionary distance,and maximum parsimony methods[J].Molecular Biology and Evolution,2011,28(10):2731-2739.
[21] TURNER S D.Qqman:An R package for visualizing GWAS results using Q-Q and Manhattan plots[J]. The Journal of Open Source Software,2018,3(25):731.
[22] ZHANG R L,JIA G Q,DIAO X M. GeneHapR:An R package for gene haplotypic statistics and visualization[J]. BMC Bioinformatics,2023,24(1):199.
[23] 张梦,谢益民,杨海涛,姚兰.肌醇在植物体内的代谢概述:肌醇作为细胞壁木聚糖和果胶前驱物的代谢途径[J].林产化学与工业,2013,33(5):106-114.ZHANG Meng,XIE Yimin,YANG Haitao,YAO Lan.Myo-inositol metamolism as the precursor of xylan and pectin in plants[J].Chemistry and Industry of Forest Products,2013,33(5):106-114.
[24] 王夏青,宋伟,张如养,陈怡凝,孙轩,赵久然.玉米茎秆抗倒伏遗传的研究进展[J].中国农业科学,2021,54(11):2261-2272.WANG Xiaqing,SONG Wei,ZHANG Ruyang,CHEN Yining,SUN Xuan,ZHAO Jiuran. Genetic research advances on maize stalk lodging resistance[J]. Scientia Agricultura Sinica,2021,54(11):2261-2272.
[25] 章霄云,郭安平,贺立卡,孔华.木质素生物合成及其基因调控的研究进展[J].分子植物育种,2006,4(3):431-437.ZHANG Xiaoyun,GUO Anping,HE Lika,KONG Hua.Advances in study of lignin biosynthesis and its genetic manipulation[J].Molecular Plant Breeding,2006,4(3):431-437.
[26] JR KIRKBRIDE J H. Biosystematics monograph of the genus Cucumis (Cucurbitaceae)[M]. Boone,NC,USA:Parkway Publishers,1993.
[27] ROBINSON R W,DECKER-WALTERS D S. Cucurbits[M].Wallingford,Oxon,UK;New York,N. Y:CAB International,1997
[28] SEBASTIAN P,SCHAEFER H,TELFORD I R H,RENNER S S. Cucumber (Cucumis sativus) and melon (C. Melo) have numerous wild relatives in Asia and Australia,and the sister species of melon is from Australia[J]. Proceedings of the National Academy of Sciences of the United States of America,2010,107(32):14269-14273.
[29] LUAN F S,DELANNAY I,STAUB J E. Chinese melon (Cucumis melo L.)diversity analyses provide strategies for germplasm curation,genetic improvement,and evidentiary support of domestication patterns[J].Euphytica,2008,164(2):445-461.
[30] 曾文芳,王志强,牛良,潘磊,丁义峰,鲁振华,崔国朝.桃果实肉质研究进展[J].果树学报,2017,34(11):1475-1482.ZENG Wenfang,WANG Zhiqiang,NIU Liang,PAN Lei,DING Yifeng,LU Zhenhua,CUI Guochao. Research process on peach fruit flesh texture[J]. Journal of Fruit Science,2017,34(11):1475-1482.