休眠是植物在进化过程中形成的一种对季节性气候变化相适应的生物学特性,被称为“隐蔽的生命现象”。休眠使芽在冬季低温条件下得以生存,这对物种繁衍具有重要意义[1]。休眠是落叶果树在秋季停止分生组织活动的一种现象,其间对促生信号不敏感。休眠解除后,植株继续生长和进行组织分化,这是对周围环境和季节性气候变化的适应性现象[1]。休眠大致分为类休眠(para-dormancy)、内休眠(endo-dormancy)和生态休眠(eco-dormancy)三种类型(图1),通常所说的休眠是指内休眠。类休眠是由植物自身因素导致,如顶端优势、内源激素变化等,除去相关器官的限制后植株可以恢复生长;内休眠是多年生植物都必需经历的阶段,需要一定时间的低温积累才能使休眠解除;生态休眠是由环境条件变化所致,限制因素一般为营养缺乏、光照或氧气不足、温度及水分胁迫等,限制因素解除后植株即可恢复生长[2]。芽休眠不是单一的固定状态,而是个体在活动-休眠(activity-dormancy)周期过程中可以变化的一系列状态[2]。芽休眠需要一种动态平衡的机制调控,而相关机制在很大程度上是未知的。科研人员早期通过需冷量(chilling requirement,CR)预测模型、DAM-like 基因、植物激素脱落酸(abscisic acid,ABA)和赤霉素(gibberellin,GA)以及表观遗传调控等方面对休眠调控进行研究[3-5]。CR 作为打破休眠最重要的因素,若CR不能得到满足,则会导致花期延长,落花落果严重,甚至影响果实的商品特性。DAM-like 作为蔷薇科果树控制休眠的明星基因,不仅通过自身表达水平,还通过介导不同的信号通路调控休眠。DAM-like 为果树休眠期控制及相关分子育种提供了重要的基因资源,在全球气候急剧变化的背景下,对蔷薇科落叶果树芽休眠的相关进展进行梳理,有利于解析果树芽休眠的调控机制及为分子育种提供参考。
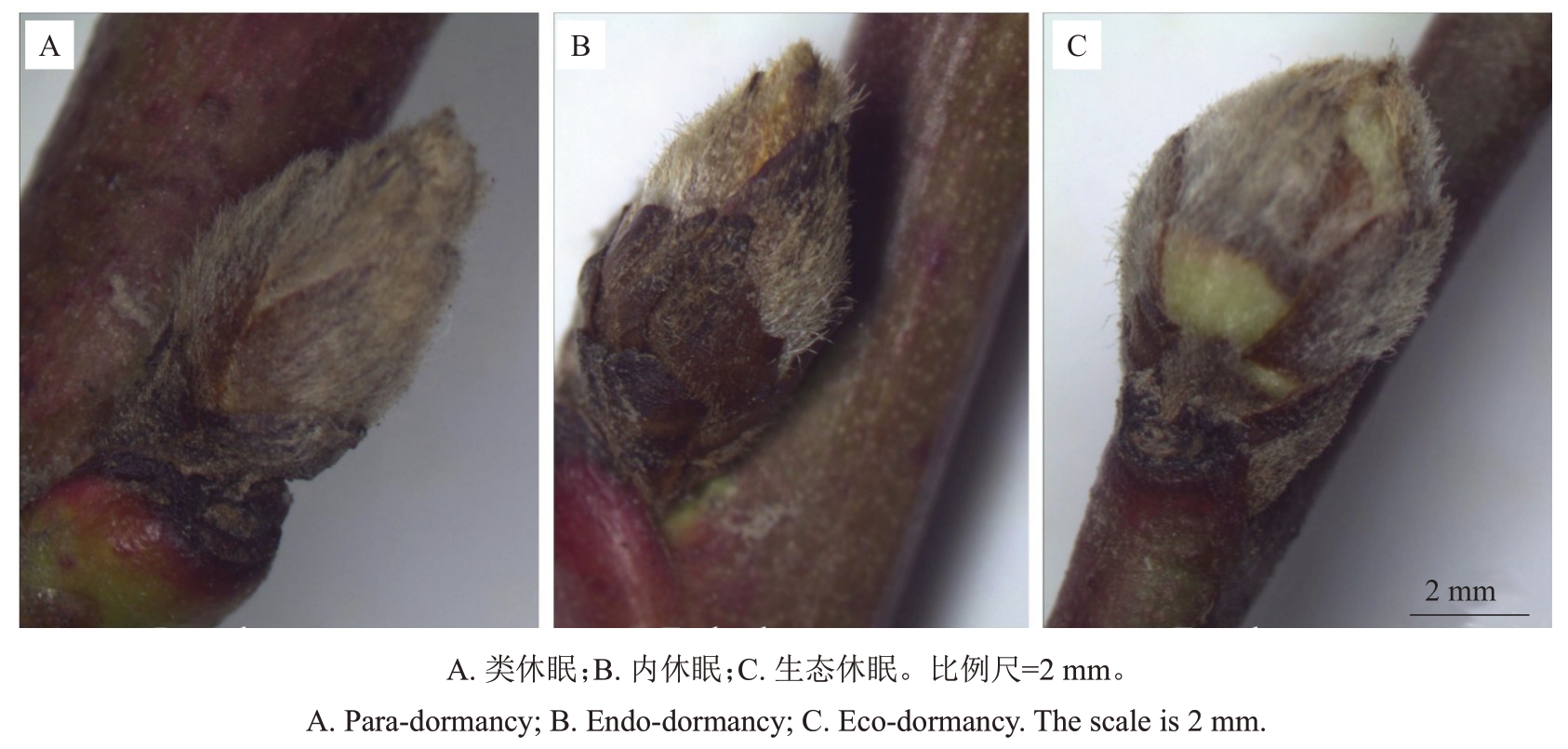
图1 桃树休眠过程中不同物候期变化
Fig.1 Changes in different phenological periods during dormancy in peach trees
1 DAM 基因鉴定及在休眠调控中的作用
1994年,研究人员在墨西哥发现了桃的非休眠突变体evergrowing(evg)。在休眠诱导条件下,桃evg 突变体不能形成顶芽和进入休眠状态,这种表型由一个隐性等位基因控制[6]。Bielenberg 等[7]在EVG 中鉴定到6 个串联排列的MADS-box 转录因子,它们位于一号染色体末端,且在evg 中不表达,所以被命名为与休眠相关的DAM(DORMANCY ASSOCIATED MADS- box)基因(DAM1~6)。DAM1~6 属于MIKCC转录因子家族,能够调节花器官和分生组织的发育及从营养生长到生殖生长的转变[8]。Fan 等[9]发现了一个与CR 和开花时间紧密相关的QTL位点,且该位点与EVG共定位,重叠区域包括PpDAM5 和PpDAM6。在桃、扁桃和杏中,与开花、CR相关的QTL位均与DAM基因共定位于1号染色体末端[10]。Yamane等[11]鉴定到了在休眠花芽中优先表达的MADS-box基因,序列分析结果表明,其和SHORT VEGETATIVE PHASE(SVP)及拟南芥AGAMOUS-LIKE 24(AGL24)同源,而后两者是编码多年生植物花芽休眠和萌发的关键作用因子。此外,鄢馨卉[12]在砀山酥梨基因组中共鉴定了5 个DAM 基因,将其命名为PpyMADS28~31 和PpyMADS71;Gao 等[13]运用PacBio 和Hi-C 技术组装脆冠梨基因组,同样鉴定到了受选择的DAM基因。笔者团队前期基于345个不同CR桃自然群体的重测序数据,利用GWAS(genome-wide association studies)定位并验证了控制桃CR的关键基因PpDAM6[5]。
在休眠进程中,DAM-like 的表达模式受光周期和低温信号的影响,也受到破眠剂单氰胺的调控[14]。位于桃EVG 位点的6 个DAM 基因的表达具有周期性,它们能够响应光周期的变化[15]。DAM1/2/4/5/6 对光周期的减少有响应,而DAM3 则可能受到低温调节。DAM5/~6 的表达量在侧芽休眠状态下最高,而在休眠解除时降至最低[14,16];Li 等[15]在桃中证实了DAM1、DAM2和DAM4的表达均与生长停止和芽的形成有关,而DAM3、DAM5 和DAM6 均表现出与休眠进程相关的表达模式。Zhu 等[3]认为低温对桃花芽休眠中DAM1和DAM3~6的表达具有明显的下调作用,另外,DAM 基因中DAM4 的表达量最高。DAM基因在休眠进程中表现出高CR品种高表达、低CR品种低表达的特性;DAM基因的表达水平在休眠解除时降至最低[13]。DAM 在营养芽和花芽中均有表达,表达量随CR的增加而逐渐降低,表现出不同品种对CR的需求差异[15,17-18]。用单氰胺处理内休眠和生态休眠的花芽,导致PpDAM5~6 的表达量下调,同时诱导花芽早萌发,表明DAM 可能通过抑制芽生长来调节低温需求[14]。
在桃中,PpDAM3和PpDAM5能够调控ABA信号途径响应基因ABI5 的表达[19]。PpTCP20(Teosinte branched1/Cycloidea/Proliferating cell factors 20)形成同源二聚体后与PpDAM5和PpDAM6基因的启动子GCCCR(R=A or G)元件结合,负调控PpDAM5和PpDAM6 的表达;PpTCP20 还与PpABF2(ABREbinding factor 2)相互作用,然后间接调控DAM基因的表达[20];PpBPCs(BASIC PENTACYSTEINE PROTEINs)通过结合PpDAM6 启动子的GA-repeat 元件,正调控PpDAM6 的表达[21]。PpDAM6 基因在上游通过参与ABA信号通路基因的诱导,促进花芽中胼胝质的生物合成,进而调控休眠的进程[5](图2)。在梅中,PmuDAM6和PmuSOC1(suppressor of overexpression of co1)发生互作[22];甜樱桃中也发现PavDAM1/5 在体内和体外均可与PavSOC1 互作[23]。在梅中,PmuDAM1、PmuDAM5 和Pmu-DAM6 之间能够形成蛋白复合物共同调控休眠[24]。在梨(Pyrus pyrifolia)中,Niu等[25]发现秋季短期低温激活冷响应基因CBFs(C-repeat binding factors)的表达并导致其积累,继而诱导DAM 基因表达;随后DAM 抑制FT(FLOWERING LOCUS T)表达,诱导休眠发生;miR6390 靶向降解DAM 基因,使休眠解除。持续低温使DAMs 的表达水平降低,从而解除对FT 的抑制,促使生长通路重新打开,进而得以休眠解除和花芽萌发。然而,在日本梨中则没有出现类似DAM 和FT 的互作现象[26],表明不同物种间DAM-like 基因通过参与不同的调控途径影响休眠进程。梨休眠花芽中,PpyABF3与PpyDAM3的表达呈正相关,进一步实验表明PpyABF3 直接与Ppy-DAM3 启动子中的第二个ABRE 元件结合,激活其表达;同时ABA 响应基因PpyABF2 与PpyABF3 发生互作,能够阻断PpyABF3对PpyDAM3的激活,使得DAM的表达受到精确控制[27]。在另一项研究中,PpABF2 抑制PpDAM1 的表达,后者可激活ABA 生物合成酶基因PpNCED3(9-cis-epoxycarotenoid dioxygenase)的表达。DAM-like 基因介导ABA 信号通路调控休眠进程,DAM 基因表达与ABA 信号途径之间存在负反馈调控机制[28]。

图2 落叶果树中DAM-like 基因参与的休眠调控模式图
Fig.2 Dormancy regulation patterns involving DAM-like genes in deciduous fruits
2 休眠进程中DAM-like 基因与激素的关系
ABA 含量在芽休眠时升高,休眠解除时降低,外源施用ABA 延缓了休眠进程和芽破裂的时间[5,29]。ABA在植物体内的稳态是植物正常生长发育所必需的,芽既是ABA作用的靶点,也是ABA新陈代谢的主要部位。ABA 对芽的抑制作用随着休眠程度的加深而减弱,休眠解除后抑制作用消失[30]。NCEDs 在休眠建立时上调表达,在休眠解除时下调表达,这在桃、梨和葡萄(Vitis vinifera)中都得到了证实[27,30-31]。与DAMs 的表达特性相似,在不同物种休眠期间,NCEDs 也遵循不同的表达模式。例如:NCED1 在桃营养芽中表达水平高于花芽,而NCED2在花芽中表达量较高[30];NCED2和NCED3在梨休眠开始时上调表达,而NCED1则在休眠结束时达到峰值[29];只有NCED1 在葡萄休眠期间被检测到[32]。表明ABA 生物合成存在一个复杂的调控网络,不同的NCEDs可能受到相对独立的调控,并以器官特异性的表达方式呈现。对休眠芽进行转录组测序分析,发现一个过程的改变往往伴随着另一个过程的相反变化,两个过程密切协同控制。例如:在芽休眠早期VvNCEDs 的表达量较高,而VvCYC707A4 的表达量很低;相反,在休眠后期,随着VvCYC707A4的快速增加,VvNCEDs的表达量开始下降[32]。
在植物中,细胞间的物质运输依赖于相邻细胞间的胞间连丝特殊通道的通透性。细胞间的分子移动受到胞间连丝中胼胝质(β-1,3-葡聚糖聚合物)的沉积和降解控制,其合成由胼胝质合成酶和葡聚糖酶共同催化完成[33]。胼胝质沉积引起的共质体闭合是植物抵御病原菌入侵的主要机制,这也是建立休眠的关键步骤。短光周期(short day,SD)事件引起胞间连丝的通透性降低是植物进行防御和休眠控制的共同表征[34]。DAM-like 基因介导下游CALS(callose synthase)的表达,使胼胝质在细胞膜之间的胞间连丝中沉积,导致细胞间信号传导受阻,进而控制休眠进程[5,35],这表明ABA 可间接作用于细胞间信号传导(图2)。
GA 通过调节细胞间分子或物质的运输控制休眠进程,对休眠解除发挥重要作用。在李中,Pp-DAM6 通过下调赤霉素生物合成通路基因KAO2-like(ENT-kaurenoic acid oxidase 2-like)、CPS1-like(ENT- copalyl diphosphate synthase 1- like)和GA20OX2-like(GA20-oxidase 2-like)的表达,同时上调赤霉素代谢基因GA2OX8-like(GA2-oxidase 8-like)的表达来控制赤霉素的含量[21]。Singh 等[35]提出了一个GA 与ABA 共同介导胞间连丝闭合而控制休眠的模型:在缺乏ABA时,PKL(PICKLE)抑制SVL(SVP-LIKE)的表达,后者正调控CALS1 和GA分解代谢基因GA2ox 的表达;ABA 水平升高时,抑制PKL的表达,后者负调节SVL的表达,导致GA分解和胼胝质积累。SVL 可作为中枢基因,通过连接ABA和GA以及低温感知途径来决定休眠建立和解除[36]。GA 可促进活性氧(reactive oxygen species,ROS)的产生。葡萄破芽率与ROS的快速积累密切相关,快速积累亚致死水平的ROS能够诱导细胞壁松动和芽萌发[37]。GA也通过糖代谢途径介导休眠解除。在梅中,GA4处理导致糖代谢途径增强[38]。可溶性糖被认为是休眠解除过程中维持芽缓慢生长的重要能量来源,其作为一种潜在的信号物质,间接增强与细胞分裂和细胞周期等相关基因的表达能力[39]。
细胞分裂素、乙烯和生长素等其他植物激素也参与了休眠周期的调节[2,40-42],但目前针对这些激素的研究较少,且都未与DAM-like相互联系起来。细胞分裂素在调节细胞分裂中起着核心作用,但在活动-休眠周期方面还未得到广泛研究。梨早期落叶可引发生长素重新分布,降低芽内的生长素含量,促进顶芽和侧芽类休眠解除,这一过程不受顶端优势中顶端组织来源的生长素影响,而受叶片的作用[40]。该生长素分配特性的提出有助于深入理解多年生木本植物周期性生长的调控机制,并为抑制早期落叶引起的侧芽反季节萌发提供理论依据。
3 表观遗传参与DAM-like 基因的调控
植物长期暴露在低温下,DAM-like 基因发生的染色质修饰与FLOWERING LOCUS C(FLC)在春化作用中发生的修饰类似,表明春化和休眠过程之间可能存在相似的机制[43]。与春化一样,休眠是通过长期暴露在低温环境中解除的,低温可以恢复生长潜力,但不能促进生长[2]。在乳浆草休眠芽中,DAM-like 受到表观遗传调控,DAM1 在不同花芽休眠阶段表现出H3K27me3 和H3K4me3 沉积水平的变化,伴随着DAM1 的表达下调和由内休眠向生态休眠的转换[44]。
在拟南芥中,H3K4me3 和H3K27me3 在开花抑制基因FLC 处沉积以应对低温胁迫[45],它们分别与基因表达的促进和抑制显著相关。在桃休眠花芽中,DAMs 的表达也与H3K4me3 和H3K27me3 的修饰有关,同时也伴随着在该基因转录起始位点区域H3ac 的降低[16],这表明染色质修饰通过调节DAMlike的表达变化进而影响花芽休眠状态。PpBPCs与存在于PpDAM6 上游H3K27me3 富集区域的两个GA-repeat基序互作,进一步支持了DAM的表达可通过H3K27me3标记进行调控[21]的论点。休眠解除时,组蛋白乙酰化修饰水平下降以及H3K27me3显著富集,这些变化参与了DAM-like基因在休眠期间的差异转录[46]。Leida等[16]和Zhu等[3]分别发现在DAM1/4/6基因区域H3K27me3 水平升高和H3K4me3 水平降低。然而,在桃休眠花芽中DAM-like基因区域则没有发现H3K27me3修饰的存在[47]。在甜樱桃和桃的非休眠芽中也发现了H3K27me3修饰的存在[3,48],这表明H3K27me3在DAM-like位点的沉积可能存在物种或品种依赖性以及环境条件的差异。
相比H3K27me3,在苹果休眠花芽中H3K4me3对基因表达谱的影响更广泛[49]。在秋季,DAMs的高表达与H3K4me3 的高水平显著相关,导致休眠建立;在冬季,DAM-like 和GA 代谢基因的低表达和H3K4me3 修饰水平有关,导致花芽休眠解除[49]。在梨中,ABA 响应的bZIP 类型转录因子PpyABF3 通过与COMPASS- like 复合体共同作用,招募H3K4me3 添加到PpyDAM4 和GA2ox 中,激活它们的表达,从而控制休眠;PpyABF3 和PpyWDR5a 互作,这两者都能够抑制梨愈伤组织的生长,且过表达PpyWDR5a 后提高了在DAM4 位点的H3K4me3 水平;ChIP-qPCR分析表明,GA2OX1在PpyABF3的下游发挥作用[50]。在桃中,仅在PpDAM4 和PpDAM5位点中发现H3K4me3 修饰水平的变化[47]。对苹果中H3K4me3 和H3K27me3 标记基因的基序进行分析,发现了可能介导组蛋白甲基化的转录因子。具体来说,在H3K4me3标记基因的2-kb启动子区域中有较多的GAGA(a trithorax-group protein)、TCP 和DAM-like 转录调控因子的结合位点,而ABI3/VP1 B3-type 转录因子家族成员FUS3 和AP2/ERF 的结合位点在H3K27me3 标记基因中富集[49]。表明DAM-like基因是组蛋白修饰的目标,但编码的蛋白质也可能与SVP 结合形成有助于组蛋白修饰的复合物(如与PpyABF3)。因此,H3K4me3对调节休眠芽中ABA诱导的GA代谢至关重要(图2)。
组蛋白修饰沉积在与休眠相关的代谢途径基因上。在桃中,蔗糖和山梨醇含量增加,山梨醇-6-磷酸脱氢酶生物合成基因在休眠期间大量表达,分别与高的H3K4me3 和低的H3K27me3 修饰水平相关[51]。在苹果的休眠营养芽中,脂质体在休眠期间积累,一种甘油分解代谢相关基因LIP2A 的下调表达与染色质可及性有关,这表明染色质修饰会影响休眠期间的脂质积累[52]。事实上,梅中的DAM6 通过抑制脂质体分解代谢基因的表达促进休眠营养分生组织的脂质积累[53]。Chen 等[49]发现,影响脂质分解代谢的GDSL LIPASE 的表达量下降伴随着H3K4me3水平的降低。以上这些结果表明,组蛋白修饰不仅影响植物激素的代谢水平,还可以通过影响脂质代谢来控制休眠进程。
除了组蛋白修饰以外,DNA甲基化以及长度通常为21~24 nt 的小分子RNA(microRNA,miRNA)也参与基因表达调控。在拟南芥中,长链非翻译RNA 参与抑制开花基因FLC 以应对低温胁迫[45]。在整个DAM 基因区域,CG 甲基化水平在低温期间保持相对恒定,但在转向暖温后下降[3]。在沙梨中,miR6390 靶向PpyDAM,与PpCBF 和PpFT2 一起参与休眠调控[25]。DAM1 和DAM5 的表达受到H3K27me3 的影响,而DAM3 和DAM4 则分别受到21-nt sRNA 和ncRNA 调控,DAM 基因被高度甲基化与24-nt的sRNAs的产生有关[3]。因此,不同表观遗传修饰及相互作用可能决定了DAM 基因的表达丰度和下调模式。在低温后的暖温条件下,5 个DAM 基因(DAM1,DAM3~6)的表达保持不变或持续下调,这种调控状态与sRNA的增加相关,尤其是DAM4[3]。降低DAM 基因表达的表观遗传变化,可能与花发育密切相关,这对了解低温需求和休眠解除的调控机制具有重要意义。然而,当满足甜樱桃CR 时,PavMADS1 启动子中DNA 甲基化水平与siRNAs(small interference RNAs)的丰度增加有关;而对PavMADS2的表达调控只取决于CG位点DNA甲基化[54],表明DNA甲基化参与了甜樱桃低温积累和休眠解除过程中PavMADS基因的沉默。
4 总结和展望
CR 与休眠的诱导、维持和解除密切相关,影响果树的萌芽、开花[55],因此准确地评价CR 积累对预测休眠进程非常重要。基于此,多种CR 预测模型已被开发出来。Weinberger[56]提出了7.2 ℃以下低温积累模型,该模型已经使用多年,对打破桃芽休眠是有效的,但不同果树品种可能有特定的最低温度,因此它的应用范围受到了限制。Richardson 等[57]提出了Uath模型,该模型常应用于动态温度范围变化条件下(小于1.4 ℃赋值为0 Chilling Units(CU),1.4~2.5 ℃赋值为0.5 CU,2.5~9.2 ℃赋值为1.0 CU,9.2~12.4 ℃赋值为0.5 CU,12.4~16 ℃赋值为0 CU,16~18 ℃赋值为-0.5 CU,大于18 ℃赋值为-1 CU)。受气候和其他环境因素的影响,在不同年份间CR差别很大。利用Uath 模型预测的翠冠梨休眠解除的CR 单位为113 CU 低于之前0~7.2 ℃模型预测的300 CU[58]。Uath 模型多适用于果树等园艺作物,但计算较为繁琐,在实际应用中可能需要考虑气候类型和植物种类的影响。在这两种模型的基础上又衍生出了0~7.2 ℃模型、0~14 ℃模型以及低温模型等,以适用特殊地理位置的气候特征[59]。通过不同CR模型对郑州地区桃品种进行评价分析,发现以0~7.2 ℃累积低温值作为CR 的评价标准比较适宜[60];在此模型的基础上,将桃芽的萌动标准划分为了五个级别和七个不同的生态型CR品种群[61]。根据这些模型预测休眠的进程固然有一定的依据,但从实际操作来看,这些判定方法都依赖视觉观察来评估芽的休眠状态,如以花芽的发育程度和萌芽率超过50%的时间作为休眠解除的临界点等。因为不同地区的环境温度差别很大,而且细胞分裂和新叶原体的形成可能发生在没有任何可见的芽生长活动或芽形态变化的情况下,所以这类观察方法有较大的局限性[2]。依据上述观点,这些模型并不能准确预测休眠的起始和结束时间,因此有必要从分子水平加以探索。
在杨树叶芽中关于休眠的研究较为深入。在DAM-like参与的休眠调控机制方面,ABA促进休眠并激活SVL 的表达,SVL 正调控NCED3 和TCP18/BRC1的表达,该作用方式促进ABA积累;此外SVL负调控FT1,同时抑制GA 的产生,多条途径共同作用于休眠进程[62-63],在休眠周期中,这一反馈调节通路解释了叶芽中ABA 含量变化的原因。在DAMlike 介导的激素途径方面,创制的DAM-like 沉默株系中GA生物合成基因上调表达,表明GA信号通路基因作为DAM-like 的下游靶点,使芽破裂时间提前[62]。在DAM-like 组蛋白表观修饰方面,在杨树休眠叶芽中的DAM-like基因区域没有发现H3K27me3修饰的存在[62]。综上,作者认为今后在蔷薇科果树上的研究可参考以下方面:
(1)梨和桃DAMs基因启动子区域的变异和需冷量的高低密切相关,且围绕桃PpDAM6 基因启动子区域的30-bp InDel已开发出了需冷量标记[5,27];考虑到不同需冷量预测模型受到较大的区域限制,建议将DAM-like分子标记或表达水平与需冷量预测模型结合起来,从生理和分子水平共同预测休眠进程。
(2)休眠的诱导、维持和解除受到光周期和低温的共同作用,但自然条件下导致落叶果树生长停止和芽休眠诱导的机制尚不明确。DAM-like 作为一个与需冷量和休眠联系起来的明星基因,其感知光周期变化和低温积累的机制仍需深入探究。
(3)虽然DAM-like在部分果树中已被证明参与休眠维持和解除,但仍需更多的遗传证据来证实它们在其他物种芽休眠控制中的作用。梨PpyDAM蛋白能够抑制FT2 的表达[25],但未发现桃PpDAM6 能够介导FT 基因的表达(未发表数据)。此外,DAMlike 充当生长抑制剂的作用机制仍不清楚,未来可通过研究其介导的靶基因功能进行阐明。
(4)表观遗传调控参与休眠周期的转换,但这些表观修饰(如组蛋白修饰、DNA甲基化等)在休眠过程中是如何丰富或逆转的仍不清楚。表观遗传标记是如何通过低温积累被招募到特定基因(DAM 和SVP)的,对冬小麦或拟南芥的春化响应机制研究可能为此提供更多参考。
[1] LANG G A,EARLY J D,MARTIN G C,DARNELL R L. Endo-,para-,and ecodormancy:Physiological terminology and classification for dormancy research[J]. HortScience,1987,22(3):371-377.
[2] COOKE J E K,ERIKSSON M E,JUNTTILA O. The dynamic nature of bud dormancy in trees:Environmental control and molecular mechanisms[J]. Plant,Cell & Environment,2012,35(10):1707-1728.
[3] ZHU H,CHEN P Y,ZHONG S L,DARDICK C,CALLAHAN A,AN Y Q,VAN KNOCKER S,YANG Y Z,ZHONG G Y,ABBOTT A,LIU Z R.Thermal-responsive genetic and epigenetic regulation of DAM cluster controlling dormancy and chilling requirement in peach floral buds[J]. Horticulture Research,2020,7:114.
[4] VEERABAGU M,VAN DER SCHOOT C,TUREČKOVÁ V,TARKOWSKÁ D,STRNAD M,RINNE P L H.Light on perenniality:Para-dormancy is based on ABA-GA antagonism and endo-dormancy on the shutdown of GA biosynthesis[J]. Plant,Cell&Environment,2023,46(6):1785-1804.
[5] ZHAO Y L,LI Y,CAO K,YAO J L,BIE H L,KHAN I A,FANG W C,CHEN C W,WANG X W,WU J L,GUO W W,WANG L R. MADS-box protein PpDAM6 regulates chilling requirement-mediated dormancy and bud break in peach[J]. Plant Physiology,2023,193(1):448-465.
[6] RODRIGUEZ A J,SHERMAN W B,SCORZA R,WISNIEWSKI M,OKIE W R.‘Evergreen’peach,its inheritance and dormant behavior[J]. Journal of the American Society for Horticultural Science,1994,119(4):789-792.
[7] BIELENBERG D G,WANG Y,LI Z G,ZHEBENTYAYEVA T,FAN S H,REIGHARD G L,SCORZA R,ABBOTT A G. Sequencing and annotation of the evergrowing locus in peach[Prunus persica (L.) Batsch] reveals a cluster of six MADS-box transcription factors as candidate genes for regulation of terminal bud formation[J]. Tree Genetics & Genomes,2008,4(3):495-507.
[8] CAI Y M,WANG L,OGUTU C O,YANG Q R,LUO B W,LIAO L,ZHENG B B,ZHANG R X,HAN Y P. The MADS-box gene PpPI is a key regulator of the double-flower trait in peach[J].Physiologia Plantarum,2021,173(4):2119-2129.
[9] FAN S H,BIELENBERG D G,ZHEBENTYAYEVA T N,REIGHARD G L,OKIE W R,HOLLAND D,ABBOTT A G.Mapping quantitative trait loci associated with chilling requirement,heat requirement and bloom date in peach (Prunus persica)[J].New Phytologist,2010,185(4):917-930.
[10] SÁNCHEZ-PÉREZ R,CUETO J D,DICENTA F,MARTÍNEZGÓMEZ P. Recent advancements to study flowering time in almond and other Prunus species[J]. Frontiers in Plant Science,2014,5:334.
[11] YAMANE H,TAKEUCHI T,MATSUSHITA M,BANNO K,TAO R. Expression analysis of apple DORMANCY-ASSOCIATED MADS-box genes in‘Fuji’dormant flower buds during flower bud development[J].Acta Horticulturae,2019(1261):143-148.
[12] 鄢馨卉.梨休眠相关DAM 基因的鉴定及功能分析[D].杭州:浙江大学,2019.YAN Xinhui. Identification and functional analysis of pear dormancy associated DAM genes[D]. Hangzhou:Zhejiang University,2019.
[13] GAO Y H,YANG Q S,YAN X H,WU X Y,YANG F,LI J Z,WEI J,NI J B,AHMAD M,BAI S L,TENG Y W.High-quality genome assembly of‘Cuiguan’pear(Pyrus pyrifolia)as a reference genome for identifying regulatory genes and epigenetic modifications responsible for bud dormancy[J]. Horticulture Research,2021,8:197.
[14] YAMANE H,OOKA T,JOTATSU H,HOSAKA Y,SASAKI R,TAO R. Expressional regulation of PpDAM5 and PpDAM6,peach (Prunus persica) dormancy-associated MADS-box genes,by low temperature and dormancy-breaking reagent treatment[J].Journal of Experimental Botany,2011,62(10):3481-3488.
[15] LI Z G,REIGHARD G L,ABBOTT A G,BIELENBERG D G.Dormancy-associated MADS genes from the EVG locus of peach [Prunus persica (L.) Batsch] have distinct seasonal and photoperiodic expression patterns[J]. Journal of Experimental Botany,2009,60(12):3521-3530.
[16] LEIDA C,CONESA A,LLÁCER G,BADENES M L,RÍOS G.Histone modifications and expression of DAM6 gene in peach are modulated during bud dormancy release in a cultivar-dependent manner[J].New Phytologist,2012,193(1):67-80.
[17] YAMANE H,TAO R,OOKA T,JOTATSU H,SASAKI R,YONEMORI K. Comparative analyses of dormancy- associated MADS-box genes,PpDAM5 and PpDAM6,in low- and highchill peaches(Prunus persica L.)[J].Journal of the Japanese Society for Horticultural Science,2011,80(3):276-283.
[18] JIMÉNEZ S,REIGHARD G L,BIELENBERG D G. Gene expression of DAM5 and DAM6 is suppressed by chilling temperatures and inversely correlated with bud break rate[J].Plant Molecular Biology,2010,73(1/2):157-167.
[19] 郇蕾,王旭旭,陈修淼,文滨滨,谭秋平,陈修德,高东升,李玲,付喜玲.桃ABA 信号关键基因PpABI5 酵母单杂交文库构建及其上游转录因子的筛选[J].植物生理学报,2017,53(7):1259-1266.HUAN Lei,WANG Xuxu,CHEN Xiumiao,WEN Binbin,TAN Qiuping,CHEN Xiude,GAO Dongsheng,LI Ling,FU Xiling.Constructing yeast one-hybrid library and screening the potential regulator of PpABI5 in peach (Prunus persica)[J]. Plant Physiology Journal,2017,53(7):1259-1266.
[20] WANG Q J,XU G X,ZHAO X H,ZHANG Z J,WANG X X,LIU X,XIAO W,FU X L,CHEN X D,GAO D S,LI D M,LI L.Transcription factor TCP20 regulates peach bud endodormancy by inhibiting DAM5/DAM6 and interacting with ABF2[J].Journal of Experimental Botany,2020,71(4):1585-1597.
[21] LLORET A,QUESADA- TRAVER C,CONEJERO A,ARBONA V,GÓMEZ-MENA C,PETRI C,SÁNCHEZ-NAVARRO J A,ZURIAGA E,LEIDA C,BADENES M L,RÍOS G.Regulatory circuits involving bud dormancy factor PpeDAM6[J].Horticulture Research,2021,8(1):261.
[22] KITAMURA Y,TAKEUCHI T,YAMANE H,TAO R. Simultaneous down-regulation of DORMANCY-ASSOCIATED MADSbox6 and SOC1 during dormancy release in Japanese apricot(Prunus mume)flower buds[J].The Journal of Horticultural Science and Biotechnology,2016,91(5):476-482.
[23] WANG J Y,GAO Z,LI H,JIU S T,QU Y T,WANG L,MA C,XU W P,WANG S P,ZHANG C X. Dormancy-associated MADS-box(DAM)genes influence chilling requirement of sweet cherries and co-regulate flower development with SOC1 gene[J].International Journal of Molecular Sciences,2020,21(3):921.
[24] ZHAO K,ZHOU Y Z,AHMAD S,YONG X,XIE X H,HAN Y,LI Y S,SUN L D,ZHANG Q X. PmCBFs synthetically affect PmDAM6 by alternative promoter binding and protein complexes towards the dormancy of bud for Prunus mume[J].Scientific Reports,2018,8(1):4527.
[25] NIU Q F,LI J Z,CAI D Y,QIAN M J,JIA H M,BAI S L,HUSSAIN S,LIU G Q,TENG Y W,ZHENG X Y. Dormancy-associated MADS-box genes and microRNAs jointly control dormancy transition in pear (Pyrus pyrifolia white pear group)flower bud[J]. Journal of Experimental Botany,2016,67(1):239-257.
[26] SAITO T,TUAN P A,KATSUMI-HORIGANE A,BAI S L,ITO A,SEKIYAMA Y,ONO H,MORIGUCHI T. Development of flower buds in the Japanese pear (Pyrus pyrifolia) from late autumn to early spring[J].Tree Physiology,2015,35(6):653-662.
[27] YANG Q S,YANG B,LI J Z,WANG Y,TAO R Y,YANG F,WU X Y,YAN X H,AHMAD M,SHEN J Q,BAI S L,TENG Y W. ABA-responsive ABRE-BINDING FACTOR3 activates DAM3 expression to promote bud dormancy in Asian pear[J].Plant,Cell&Environment,2020,43(6):1360-1375.
[28] TUAN P A,BAI S L,SAITO T,ITO A,MORIGUCHI T. Dormancy-associated MADS-box (DAM) and the abscisic acid pathway regulate pear endodormancy through a feedback mechanism[J].Plant&Cell Physiology,2017,58(8):1378-1390.
[29] LI J Z,XU Y,NIU Q F,HE L F,TENG Y W,BAI S L.Abscisic acid(ABA)promotes the induction and maintenance of pear(Pyrus pyrifolia white pear group)flower bud endodormancy[J].International Journal of Molecular Sciences,2018,19(1):310.
[30] WANG D L,GAO Z Z,DU P Y,XIAO W,TAN Q P,CHEN X D,LI L,GAO D S. Expression of ABA metabolism-related genes suggests similarities and differences between seed dormancy and bud dormancy of peach (Prunus persica)[J]. Frontiers in Plant Science,2016,6:1248.
[31] ZHENG C L,ACHEAMPONG A K,SHI Z W,MUGZECH A,HALALY- BASHA T,SHAYA F,SUN Y F,COLOVA V,MOSQUNA A,OPHIR R,GALBRAITH D W,OR E. Abscisic acid catabolism enhances dormancy release of grapevine buds[J].Plant,Cell&Environment,2018,41(10):2490-2503.
[32] ZHENG C L,HALALY T,ACHEAMPONG A K,TAKEBAYASHI Y,JIKUMARU Y,KAMIYA Y,OR E.Abscisic acid(ABA) regulates grape bud dormancy,and dormancy release stimuli may act through modification of ABA metabolism[J].Journal of Experimental Botany,2015,66(5):1527-1542.
[33] WU S W,KUMAR R,ISWANTO A B B,KIM J Y.Callose balancing at plasmodesmata[J]. Journal of Experimental Botany,2018,69(22):5325-5339.
[34] RINNE P L,VAN DER SCHOOT C. Plasmodesmata at the crossroads between development,dormancy,and defense[J].Canadian Journal of Botany,2003,81(12):1182-1197.
[35] SINGH R K,MISKOLCZI P,MAURYA J P,BHALERAO R P.A tree ortholog of short vegetative phase floral repressor mediates photoperiodic control of bud dormancy[J].Current Biology,2019,29(1):128-133.e2.
[36] BUSOV V B. Plant development:Dual roles of poplar SVL in vegetative bud dormancy[J].Current Biology,2019,29(2):R68-R70.
[37] SUDAWAN B,CHANG C S,CHAO H F,KU M S B,YEN Y F.Hydrogen cyanamide breaks grapevine bud dormancy in the summer through transient activation of gene expression and accumulation of reactive oxygen and nitrogen species[J]. BMC Plant Biology,2016,16(1):202.
[38] ZHUANG W B,GAO Z H,WEN L H,HUO X M,CAI B H,ZHANG Z. Metabolic changes upon flower bud break in Japanese apricot are enhanced by exogenous GA4[J]. Horticulture Research,2015,2:15046.
[39] RUAN Y L,JIN Y,YANG Y J,LI G J,BOYER J S. Sugar input,metabolism,and signaling mediated by invertase:Roles in development,yield potential,and response to drought and heat[J].Molecular Plant,2010,3(6):942-955.
[40] WEI J,YANG Q S,NI J B,GAO Y H,TANG Y X,BAI S L,TENG Y W. Early defoliation induces auxin redistribution,promoting paradormancy release in pear buds[J].Plant Physiology,2022,190(4):2739-2756.
[41] CORBINEAU F,XIA Q,BAILLY C,EL-MAAROUF-BOUTEAU H. Ethylene,a key factor in the regulation of seed dormancy[J].Frontiers in Plant Science,2014,5:539.
[42] SHI Z W,HALALY- BASHA T,ZHENG C L,SHARABISCHWAGER M,WANG C,GALBRAITH D W,OPHIR R,PANG X Q,OR E. Identification of potential post-ethylene events in the signaling cascade induced by stimuli of bud dormancy release in grapevine[J].The Plant Journal,2020,104(5):1251-1268.
[43] BASTOW R,MYLNE J S,LISTER C,LIPPMAN Z,MARTIENSSEN R A,DEAN C.Vernalization requires epigenetic silencing of FLC by histone methylation[J]. Nature,2004,427(6970):164-167.
[44] HORVATH D P,SUNG S,KIM D,CHAO W,ANDERSON J.Characterization,expression and function of dormancy associated mads-box genes from leafy spurge[J].Plant Molecular Biology,2010,73(1/2):169-179.
[45] TAMADA Y,YUN J Y,WOO S C,AMASINO R M. ARABIDOPSIS TRITHORAX-RELATED7 is required for methylation of lysine 4 of histone H3 and for transcriptional activation of FLOWERING LOCUS C[J]. The Plant Cell,2009,21(10):3257-3269.
[46] DE LA FUENTE L,CONESA A,LLORET A,BADENES M L,RÍOS G.Genome-wide changes in histone H3 lysine 27 trimethylation associated with bud dormancy release in peach[J]. Tree Genetics&Genomes,2015,11(3):45.
[47] CANTON M,FORESTAN C,MARCONI G,CARRERA E,BONGHI C,VAROTTO S.Evidence of chromatin and transcriptional dynamics for cold development in peach flower bud[J].New Phytologist,2022,236(3):974-988.
[48] VIMONT N,QUAH F X,SCHÖEPFER D G,ROUDIER F,DIRLEWANGER E,WIGGE P A,WENDEN B,CORTIJO S.ChIPseq and RNA-seq for complex and low-abundance tree buds reveal chromatin and expression co-dynamics during sweet cherry bud dormancy[J].Tree Genetics&Genomes,2019,16(1):9.
[49] CHEN W X,TAMADA Y,YAMANE H,MATSUSHITA M,OSAKO Y,GAO-TAKAI M,LUO Z R,TAO R. H3K4me3 plays a key role in establishing permissive chromatin states during bud dormancy and bud break in apple[J].The Plant Journal,2022,111(4):1015-1031.
[50] YANG Q S,WU X Y,GAO Y H,NI J B,LI J J,PEI Z Q,BAI S L,TENG Y W. PpyABF3 recruits the COMPASS-like complex to regulate bud dormancy maintenance via integrating ABA signaling and GA catabolism[J]. New Phytologist,2023,237(1):192-203.
[51] LLORET A,MARTÍNEZ-FUENTES A,AGUSTÍ M,BADENES M L,RÍOS G. Chromatin-associated regulation of sorbitol synthesis in flower buds of peach[J]. Plant Molecular Biology,2017,95(4/5):507-517.
[52] SAITO T,WANG S S,OHKAWA K,OHARA H,IKEURA H,OGAWA Y,KONDO S. Lipid droplet-associated gene expression and chromatin remodelling in LIPASE 5'-upstream region from beginning- to mid-endodormant bud in‘Fuji’apple[J].Plant Molecular Biology,2017,95(4/5):441-449.
[53] HSIANG T F,YAMANE H,GAO-TAKAI M,TAO R. Regulatory role of Prunus mume DAM6 on lipid body accumulation and phytohormone metabolism in the dormant vegetative meristem[J].Horticulture Research,2024,11(6):uhae102.
[54] ROTHKEGEL K,SÁNCHEZ E,MONTES C,GREVE M,TAPIA S,BRAVO S,PRIETO H,ALMEIDA A M. DNA methylation and small interference RNAs participate in the regulation of MADS-box genes involved in dormancy in sweet cherry(Prunus avium L.)[J].Tree Physiology,2017,37(12):1739-1751.
[55] 王力荣,朱更瑞,左覃元.桃需冷量遗传特性的研究[J].果树科学,1996,13(4):237-240.WANG Lirong,ZHU Gengrui,ZUO Qinyuan. Study on genetic characteristics of chilling requirement in peach[J]. Journal of Fruit Science,1996,13(4):237-240.
[56] WEINBERGER J. Chilling requirements of peach varieties[J].Proceedings of the American Society for Horticultural Science 1950,13(4):46-51.
[57] RICHARDSON E A,SEELEY S D,WALKER D R. A model for estimating the completion of rest for‘Redhaven’and‘Elberta’peach trees[J].HortScience,1974,9(4):331-332.
[58] YANG Q S,GAO Y H,WU X Y,MORIGUCHI T,BAI S L,TENG Y W. Bud endodormancy in deciduous fruit trees:Advances and prospects[J].Horticulture Research,2021,8(1):139.
[59] 庄维兵,章镇,侍婷,王培培,邵静,罗晓燕,高志红.落叶果树需冷量及其估算模型研究进展[J].果树学报,2012,29(3):447-453.ZHUANG Weibing,ZHANG Zhen,SHI Ting,WANG Peipei,SHAO Jing,LUO Xiaoyan,GAO Zhihong.Advance on chilling requirement and its chilling models in deciduous fruit crops[J].Journal of Fruit Science,2012,29(3):447-453.
[60] 王力荣,朱更瑞,方伟超,左覃元.桃品种需冷量评价模式的探讨[J].园艺学报,2003,30(4):379-383.WANG Lirong,ZHU Gengrui,FANG Weichao,ZUO Qinyuan.Estimating models of the chilling requirement for peach[J].Acta Horticulturae Sinica,2003,30(4):379-383.
[61] 王力荣,朱更瑞,左覃元.中国桃品种需冷量的研究[J].园艺学报,1997,24(2):194-196.WANG Lirong,ZHU Gengrui,ZUO Qinyuan. Studies on the chilling requirement of peach varieties[J].Acta Horticulturae Sinica,1997,24(2):194-196.
[62] SINGH R K,MAURYA J P,AZEEZ A,MISKOLCZI P,TYLEWICZ S,STOJKOVIČ K,DELHOMME N,BUSOV V,BHALERAO R P. A genetic network mediating the control of bud break in hybrid aspen[J]. Nature Communications,2018,9(1):4173-4182.
[63] TYLEWICZ S,PETTERLE A,MARTTILA S,MISKOLCZI P,AZEEZ A,SINGH R K,IMMANEN J,MÄHLER N,HVIDSTEN T R,EKLUND D M,BOWMAN J L,HELARIUTTA Y,BHALERAO R P. Photoperiodic control of seasonal growth is mediated by ABA acting on cell-cell communication[J]. Science,2018,360(6385):212-215.