WRKY 家族是一类在植物界特有的、数量庞大的转录因子[1]。其名称源于他们高度保守的WRKY结构域,该结构域大约包含60 个氨基酸残基,其中氨基(N)端有高度保守的七肽序列WRKYGQK,而在结构域的羧基(C)端则是一个C2H2 型或C2HC型的锌指结构[2]。根据WRKY结构域的数量以及锌指结构的特征,WRKY 转录因子一般被分为Ⅰ、Ⅱ和Ⅲ组[2-4]。WRKY 转录因子通过WRKY 结构域识别并结合DNA 序列中的顺式作用元件W-box[(C/T)TGAC(C/T)],进而调控靶基因的表达水平,发挥相应的生物学功能[1]。自第一个WRKY基因SPF1于1994 年从甘薯(Dioscorea esculenta)中被克隆[5],WRKY 基因家族相继在拟南芥[6]、水稻[7]、草莓[8]、香樟[9]、绿豆[10]、板栗[11]等多种植物中被鉴定。
植物在生长过程中面临着各种逆境胁迫[12],其中干旱是严重影响植物生长发育和产量的重要因素之一[13]。近年来,越来越多的研究表明,WRKY 转录因子在植物响应干旱胁迫中起着关键作用[1,14]。研究表明,大豆中的GmWRKY27基因可以通过两种方式增强大豆对干旱的抵抗力:一是直接抑制GmNAC29基因的启动子活性,二是与GmMYB174 基因协同作用,进一步抑制GmNAC29 的表达[15]。拟南芥的At-WRKY1通过调节膜转运蛋白以保持水分,从而调节其对干旱胁迫的响应[16]。尤其在拟南芥中过表达小麦的TaWRKY2 和TaWRKY19 或水稻的OsWRKY45基因,可提高植株对干旱胁迫的耐受性[17-18]。茶树过表达CsWRKY26可通过激活脱落酸信号通路或调节乙烯、生长素及丝裂原活化蛋白激酶信号转导,增强胁迫相关基因的表达,从而提高茶树的抗旱能力[19]。棉花的GhWRKY68 通过调节ABA 信号传导以及细胞活性氧来响应干旱胁迫和盐胁迫[20]。
富民枳(Poncirus polyandra)是芸香科枳属常绿小乔木,为云南省特有的极小种群野生植物[21-22]。富民枳抗逆性强,是一种优秀的柑橘矮化砧[23-24],尤其在前期研究中发现,该物种具有一定的抗旱性(研究结果尚未发表)。枳(Poncirus trifoliata)是枳属富民枳唯一的近缘种,耐旱耐寒,是柑橘最重要的砧木之一,也是最有价值的抗性和耐性基因来源[22,25-26]。虽然笔者课题组前期研究已发现富民枳基因组富含多个抗逆性相关基因[27],但富民枳的WRKY 基因在干旱胁迫下是否发挥功能仍不得而知。
因此,笔者在本研究中基于富民枳的全基因组数据,通过筛选和鉴定PpWRKYs 基因家族成员,分析其蛋白理化性质、进化关系、保守基序和顺式作用元件等,并基于近缘种枳在干旱胁迫下的转录组数据,在富民枳中筛选同源抗旱候选WRKY 基因,最后通过qRT-PCR 验证PpWRKYs 在干旱胁迫下的表达水平,为进一步研究WRKY 基因家族成员在富民枳响应干旱胁迫应答过程中的作用提供理论基础。
1 材料和方法
1.1 富民枳WRKY基因家族鉴定及理化性质分析
富民枳基因组由笔者课题组完成测序并发表[27],WRKY 保守结构域序列(PF03106)文件从InterPro(https://www.ebi.ac.uk/interpro/)数据库下载。通过HMMER 软件构建隐马尔可夫模型,在富民枳的氨基酸序列中检索。检索到的蛋白序列利用NCBI-CDD(https://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi)和SMART(http://smart.embl-heidelberg.de/)数据库进行结构域分析,剔除无WRKY 结构域的蛋白,最终确定富民枳的WRKY 基因家族成员。利用ExPASy(https://web.expasy.org/protparam/)网站中的ProtParam 工具分析富民枳WRKY 蛋白的序列长度、相对分子质量及等电点。利用Euk-mPLoc2.0(http://www.csbio.sjtu.edu.cn/bioinf/Cell-PLoc-2/)在线网站进行亚细胞定位分析。
1.2 富民枳WRKY 家族蛋白的序列比对和系统发育分析
利用MEGA11 软件中的MUSCLE 对富民枳和拟南芥WRKY 转录因子的氨基酸序列进行多序列比对,构建邻接系统发育树,bootstrap 复制为1000,最后使用ggtree 对进化树进行美化[28]。利用DNAMAN 软件对PpWRKYs 蛋白各亚组成员的WRKY结构域进行多序列比对及可视化。
1.3 PpWRKY基因的染色体定位和共线性分析
从Phytozome (https://phytozome- next.jgi.doe.gov/info/Ptrifoliata_v1_3_1)网站下载枳的全基因数据和基因注释文件,富民枳基因组数据由笔者课题组完成测序并注释,通过TBtools 的MCScanX 功能对富民枳进行种内与种间共线性分析,并计算富民枳中片段重复基因对的Ka/Ks比值。
1.4 PpWRKY 家族基因结构、保守基序及结构域分析
利用MEME(http:∥meme suite.org/tools/meme)在线网站预测富民枳WRKY 家族蛋白的保守基序,设置motif 数量为10;通过TBtools 软件从富民枳基因组注释文件中提取富民枳WRKY家族基因的外显子和内含子位置信息;利用NCBI-CDD 获取PpWRKY 家族蛋白的保守结构域位置信息。最后通过TBtools 软件的Gene Structure View 工具进行可视化。
1.5 富民枳WRKY基因启动子顺式作用元件分析
利用TBtools 软件提取PpWRKYs 基因上游2000 bp 的启动子序列,将其提交到Plant CARE(http://bioinformatics.psb.ugent.be/webtools/plantcare/html/)网站进行顺式作用元件预测,通过手动整理后利用TBtools软件进行可视化。
1.6 富民枳同源抗旱WRKY候选基因的鉴定
从Phytozome 获取枳的基因组数据,从NCBI(https://www.ncbi.nlm.nih.gov/)下载枳在干旱胁迫下的转录组数据,登记号是PRJNA905931。再利用HMMER 软件鉴定枳的WRKY 家族基因,方法如1.1,并通过枳WRKY基因ID提取在模拟干旱胁迫处理后0、1.5、6 和24 h 的转录组数据,利用TBtools 软件进行可视化。最后通过TBtools 软件把枳中表达量高且显著上调的WRKY基因与富民枳基因组进行Blast,初步鉴定到富民枳抗旱候选基因。
1.7 实时荧光定量PCR(qRT-PCR)表达分析
以云南省林业与草原科学院温室大棚培育的一年生富民枳作为试验材料。选取长势一致的富民枳幼苗,用20%的PEG-6000 溶液处理模拟干旱胁迫,每个处理3 个重复,分别于处理0、3、6、12 和24 h 后采集叶片,保存于-80 ℃冰箱。利用HiPure Plant RNA Plus Kit 试剂盒(美基生物,北京)提取富民枳叶片总RNA,然后用江苏百时美生物科技有限公司(连云港)的All-in-One First-Strand Synthesis Master-Mix (with dsDNase)反转录试剂盒将总RNA 反转录成cDNA。通过Vector NTI 设计引物(表1),然后在生工生物工程股份有限公司(上海)合成。以Actin基因作为内参,使用F488 SYBR qPCR Mix 荧光定量试剂盒(江苏百时美,连云港)进行qRT-PCR 检测。反应体系10 μL:5 μL F488 SYBR qPCR Mix,0.25 μL 正向引物,0.25 μL 反向引物,1 μL cDNA,3.5 μL ddH2O。扩增程序:95 ℃预变性30~60 s;95 ℃变性10 s,60 ℃退火30 s,40 个循环。采用2-ΔΔCT方法测定相对表达量[29]。
表1 用于qRT-PCR 的引物
Table 1 Primers for qRT-PCR

基因名称Gene name Actin PpWRKY8 PpWRKY12 PpWRKY13 PpWRKY19 PpWRKY23 PpWRKY27 PpWRKY28 PpWRKY30 PpWRKY41正向引物(5'-3')Forward primer (5'-3')CCGACCGTATGAGCAAGGAAA ACGAAGGGCCACCTTCTGAT TCGACCGGACAAACCATCACTC AAGCACAACCATGATGTTCCGG AGTCTGAGCAATCTGCCGTACATC AGCTTTGTGACCGAATGCCAAG ATGCTGTCTCCTTCCCTTCTGG AGCTACTACCGATGCACGCATC ATGTTGAGAGAGCATCTACGGACCC ATCAAGGGCTCACCATACCCAC反向引物(5'-3')Reverse primer (5'-3')TTCCTGTGGACAATGGATGGA TGGTTATGGCTAGAGGTGTAGGTG TTCCCAGAAATTGCCGCAGC TGTTAGGATGATGAGCCACCGC AGTCTCTGACTCGCAATTCGATTG ATGCACTGAGCCTCAGGACCTTC TCAGGAACCTGATGACCAAGTTG TGCATCTGGTTCAAGATGTGCTC TGTGGCTACTATTCTTACCAGCCGG AATGACGGTGCTCTCCTTCGTAC
1.8 PpWRKY蛋白互作网络预测
利用string 11.5(http://string-db.org/)数据库预测PpWRKY 蛋白与调控植物耐旱性的MYB 蛋白(AtMYB15、AtMYB44 和AtMYB96)的相互作用网络,选定模式植物拟南芥为物种参数,去除不成簇和单个节点的蛋白得到蛋白互作网络图。
2 结果与分析
2.1 富民枳WRKY基因家族鉴定及理化性质分析
在富民枳基因组中共搜索到49条具有WRKY结构域的蛋白序列,剔除3 条(evm.model.LG09.1378、evm.model.LG05.2450 和evm.model.LG05.872)不含有典型WRKY 结构域的假阳性序列后,最终从富民枳基因组中鉴定到46 个PpWRKY 基因,按照其在染色体上的位置顺序命名为PpWRKY1~PpWRKY46(表2)。理化性质分析结果表明,鉴定到的46 个PpWRKY 蛋白的氨基酸数量为116~1103,其中PpWRKY21 分子质量最低,为13 213.48 kDa;PpWRKY23 分子质量最高,为120 199.00 kDa。蛋白等电点(pI)为4.93 (PpWRKY24)~9.80(PpWRKY17),其中61%的蛋白等电点低于7.0,说明富民枳WRKY 蛋白大部分是酸性。亚细胞定位预测结果显示,除PpWRKY23 定位于溶酶体和液泡外,其余45个PpWRKY基因均定位于细胞核中。
表2 PpWRKY 基因相关信息
Table 2 Information related to PpWRKY genes

基因ID Gene ID evm.model.LG01.326 evm.model.LG01.3345 evm.model.LG01.3451 evm.model.LG01.3503 evm.model.LG01.369 evm.model.LG01.510 evm.model.LG01.572 evm.model.LG01.594 evm.model.LG01.944 evm.model.LG02.1969 evm.model.LG02.452 evm.model.LG03.1430 evm.model.LG03.1603 evm.model.LG03.1900 evm.model.LG03.1927 evm.model.LG03.424 evm.model.LG04.829 evm.model.LG05.1194 evm.model.LG05.1295 evm.model.LG05.1970 evm.model.LG05.22 evm.model.LG05.2392 evm.model.LG05.2539基因名称Gene name PpWRKY1 PpWRKY2 PpWRKY3 PpWRKY4 PpWRKY5 PpWRKY6 PpWRKY7 PpWRKY8 PpWRKY9 PpWRKY10 PpWRKY11 PpWRKY12 PpWRKY13 PpWRKY14 PpWRKY15 PpWRKY16 PpWRKY17 PpWRKY18 PpWRKY19 PpWRKY20 PpWRKY21 PpWRKY22 PpWRKY23氨基酸数量Number of amino acid 342 602 307 336 359 501 323 274 353 393 359 320 595 230 202 363 358 599 488 492 116 265 1103等电点pI 6.32 6.23 5.77 5.20 5.02 7.23 8.78 5.18 9.69 5.88 9.30 8.35 7.09 9.10 7.18 9.38 9.80 6.45 6.27 6.07 9.67 5.20 6.10分子质量Mw/Da 37 421.21 65 179.94 34 362.27 37 632.48 40 495.95 54 333.76 35 708.04 31 021.30 39 621.87 43 909.87 38 791.88 35 519.57 65 006.34 25 906.22 23 128.21 40 112.03 40 365.76 65 236.13 53 121.33 53 073.03 13 213.48 30 068.16 120 199.00组/亚组Group/SubgroupⅡeⅡbⅡe染色体ChromosomeⅢⅢⅡbⅡcⅡeⅡdⅡbⅡdⅡaⅠaⅡcⅡcⅡdⅡdⅡbⅠaⅡeⅡcⅡeⅡb evm.model.LG06.1687 evm.model.LG06.1727 evm.model.LG06.91 evm.model.LG07.1123 evm.model.LG07.1630 evm.model.LG07.1989 evm.model.LG07.2082 evm.model.LG07.2104 evm.model.LG07.279 evm.model.LG07.280 evm.model.LG07.926 evm.model.LG08.140 evm.model.LG08.141 evm.model.LG08.1427 evm.model.LG08.1495 evm.model.LG08.1635 evm.model.LG08.2185 evm.model.LG08.66 evm.model.LG09.1053 evm.model.LG09.1115 evm.model.LG09.135 evm.model.LG09.1507 evm.model.LG09.37 PpWRKY24 PpWRKY25 PpWRKY26 PpWRKY27 PpWRKY28 PpWRKY29 PpWRKY30 PpWRKY31 PpWRKY32 PpWRKY33 PpWRKY34 PpWRKY35 PpWRKY36 PpWRKY37 PpWRKY38 PpWRKY39 PpWRKY40 PpWRKY41 PpWRKY42 PpWRKY43 PpWRKY44 PpWRKY45 PpWRKY46 405 382 372 305 193 568 468 291 356 309 154 257 321 569 563 721 485 341 331 569 588 469 497 4.93 5.35 6.00 7.00 9.12 6.64 7.71 6.19 6.57 6.02 9.64 8.41 7.17 6.60 6.47 5.78 6.31 9.56 5.48 6.48 5.96 8.42 7.00 44 907.20 42 079.37 40 961.72 33 912.31 21 656.09 62 894.97 50 962.14 32 079.37 39 395.82 35 007.62 17 280.34 28 673.11 35 906.14 62 543.49 60 903.47 77 868.67 53 220.25 36 671.40 36 852.64 62 646.18 63 697.51 52 130.91 54 874.39ⅡeⅢⅡcⅡcⅡcⅡbⅠaⅡcⅢⅢⅡcⅡaⅡaⅡbⅠaⅠaⅠaⅡdⅢⅠaⅡbⅠaⅠa 1 1 1 1 1 1 1 1 1 2 2 3 3 3 3 3 4 5 5 5 5 5 5 6 6 6 7 7 7 7 7 7 7 7 8 8 8 8 8 8 8 9 9 9 9 9亚细胞定位预测Prediction of subcellular localization细胞核 Nucleus细胞核 Nucleus细胞核 Nucleus细胞核 Nucleus细胞核 Nucleus细胞核 Nucleus细胞核 Nucleus细胞核 Nucleus细胞核 Nucleus细胞核 Nucleus细胞核 Nucleus细胞核 Nucleus细胞核 Nucleus细胞核 Nucleus细胞核 Nucleus细胞核 Nucleus细胞核 Nucleus细胞核 Nucleus细胞核 Nucleus细胞核 Nucleus细胞核 Nucleus细胞核 Nucleus溶酶体、液泡Lysosome, vacuole细胞核 Nucleus细胞核 Nucleus细胞核 Nucleus细胞核 Nucleus细胞核 Nucleus细胞核 Nucleus细胞核 Nucleus细胞核 Nucleus细胞核 Nucleus细胞核 Nucleus细胞核 Nucleus细胞核 Nucleus细胞核 Nucleus细胞核 Nucleus细胞核 Nucleus细胞核 Nucleus细胞核 Nucleus细胞核 Nucleus细胞核 Nucleus细胞核 Nucleus细胞核 Nucleus细胞核 Nucleus细胞核 Nucleus
2.2 富民枳WRKY 家族蛋白的序列比对和系统发育分析
对富民枳和拟南芥的WRKY 蛋白序列构建系统发育树,结果显示,46 个PpWRKY 蛋白分为Ⅰ、Ⅱ、Ⅲ 3个组(图1)。其中,Ⅱ组的成员数量最多,有31 个,可进一步分为5 个亚组(Ⅱa~Ⅱe),依次含有3、8、9、6和5个PpWRKY成员;Ⅰ和Ⅲ组分别含有9个和6 个成员。富民枳WRKY 结构域的多序列比对结果显示,Ⅰ组9 个蛋白均是C2H2 型锌指基序,属于Ⅰa 亚组,未在PpWRKY 蛋白中发现含C2HC型锌指基序的Ⅰb亚组成员(图2)。除了Ⅱc亚组的PpWRKY15 和PpWRKY21 蛋白在WRKY 结构域的七肽序列上出现了单个氨基酸变异,PpWRKY15 中的Q 被K 替换,PpWRKY21 中的R 被K 替换,其余44 个PpWRKY 基因家族成员均具有典型的WRKY结构域特征序列WRKYGQK,说明PpWRKYs 基因家族保守性较强,暗示该基因家族编码的蛋白质在生物体中承担着关键的生物学功能。

图1 富民枳和拟南芥WRKY 家族成员的系统发育树
Fig. 1 Phylogenetic trees of WRKY family members of Poncirus polyandra and Arabidopsis thaliana

图2 PpWRKYs 保守结构域多序列比对(MSA)
Fig. 2 PpWRKYs conserved domain multiple sequence alignment (MSA)
2.3 染色体定位和共线性分析
染色体定位分析发现,46 个富民枳的WRKY 基因在9 条染色体上的分布是不均匀的,尤其是1号染色体,上面聚集了最多的PpWRKY基因,共有9个,其次是7号染色体,有8个,4号染色体分布的PpWRKY基因最少,只有1 个(图3)。位于1 号染色体的PpWRKY7 和PpWRKY8,7 号染色体的PpWRKY30 和PpWRKY31,PpWRKY32 和PpWRKY33,8 号染色体的PpWRKY35 和PpWRKY36 为串联重复基因;PpWRKY 基因家族还存在20 对片段复制基因,如PpWRKY13 和PpWRKY43。为了探究PpWRKY 基因在进化中是否经历自然选择,将计算其片段复制的Ka/Ks 比值进行分析,结果显示,在富民枳中发生片段复制的WRKY 基因的Ka/Ks 值均小于1(表3),说明这些基因经历了纯化选择作用。通过对富民枳和枳的WRKY 基因进行共线性分析发现,富民枳和枳有88对共线性基因(图4)。
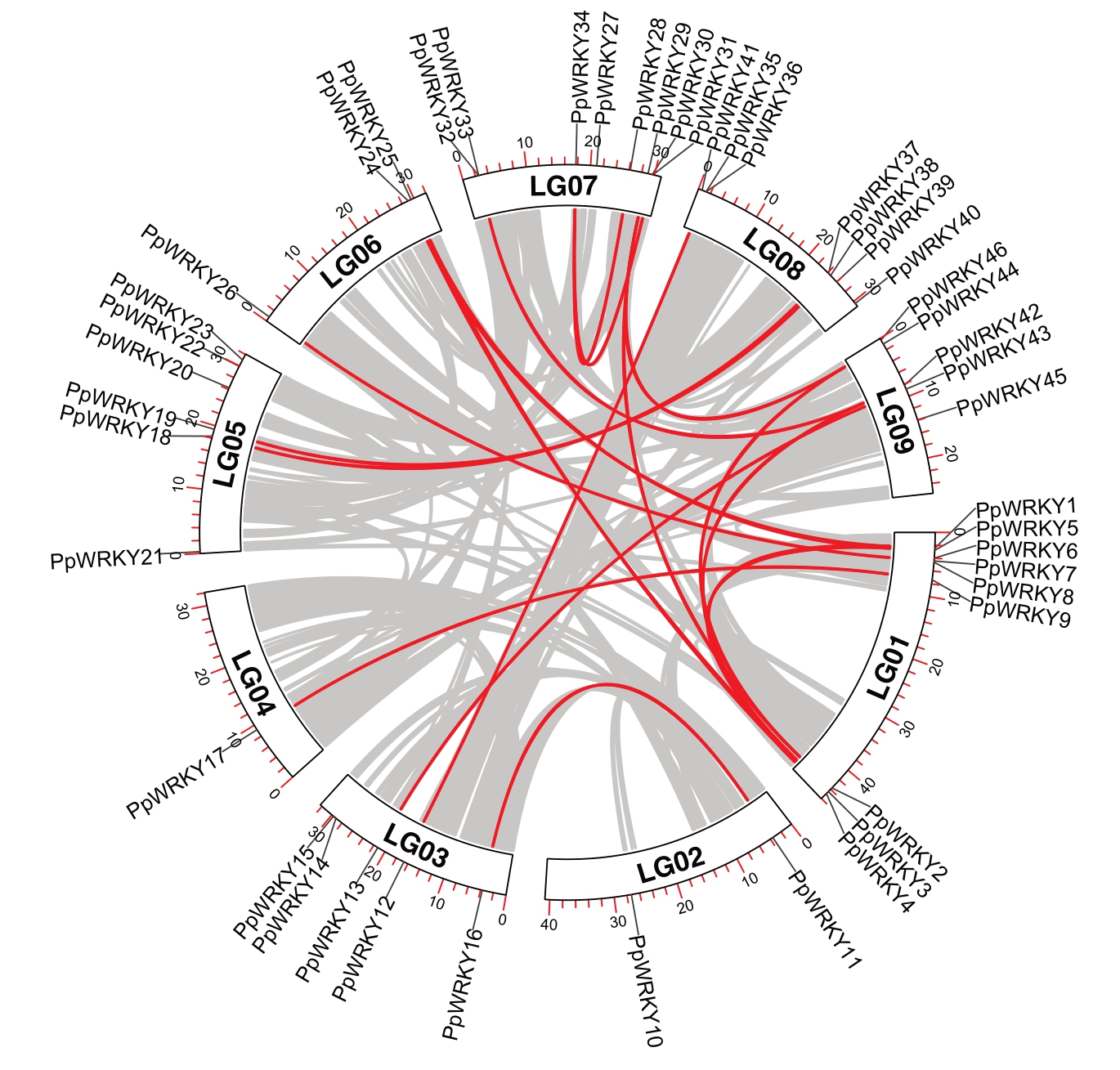
图3 富民枳WRKY 基因家族染色体定位和共线性分析
Fig. 3 Chromosome localization and collinearity analysis of WRKY gene family of Poncirus polyandra

图4 富民枳和枳WRKY 基因家族的共线性分析
Fig. 4 Collinearity analysis of WRKY gene families of Poncirus polyandra and Poncirus trifoliata
表3 富民枳WRKY 基因家族进化选择压力分析
Table 3 Analysis of evolutionary selection pressure of WRKY gene family in Poncirus polyandra

注:NaN 表示两个序列差距太大,计算无意义。
Note:NaN indicates that the two sequences are too far apart for the calculation to be meaningful.
基因对Gene pair PpWRKY1-PpWRKY3 PpWRKY1-PpWRKY24 PpWRKY2-PpWRKY29 PpWRKY2-PpWRKY44 PpWRKY3-PpWRKY24 PpWRKY5-PpWRKY4 PpWRKY4-PpWRKY25 PpWRKY4-PWRKY42 PpWRKY5-PpWRKY25 PpWRKY7-PpWRKY26 PpWRKY9-PpWRKY17 PpWRKY11-PpWRKY16 PpWRKY12-PpWRKY35 PpWRKY13-PpWRKY43 PpWRKY18-PpWRKY37 PpWRKY19-PpWRKY38 PpWRKY34-PpWRKY28 PpWRKY29-PpWRKY44 PpWRKY34-PpWRKY31 PpWRKY33-PpWRKY42有效长度Effective length/bp 891 936 1617 1641 843 972 969 792 1014 915 987 1026 714 1569 1521 1257 456 1593 450 873非同义突变频率Ka 0.472 743 091 0.544 214 695 0.381 015 570 0.369 024 580 0.552 613 297 0.413 011 094 0.394 458 768 0.629 533 389 0.406 770 881 0.351 616 092 0.301 116 399 0.353 671 882 0.482 939 687 0.301 185 505 0.394 549 307 0.568 454 942 0.332 928 003 0.442 097 469 0.603 872 571 0.558 069 516同义突变频率Ks 1.693 329 527 1.705 310 132 1.484 446 351 1.182 774 268 3.330 629 746 2.339 242 867 2.249 854 199 NaN 2.408 042 46 2.180 353 963 2.806 189 938 1.528 342 258 NaN 1.585 841 657 2.173 707 703 1.456 897 398 1.911 064 164 1.709 517 499 NaN 1.872 714 226非同义突变频率/同义突变频率Ka/Ks 0.279 179 618 0.319 129 456 0.256 671 836 0.311 999 162 0.165 918 562 0.176 557 595 0.175 326 369 NaN 0.168 921 806 0.161 265 601 0.107 304 354 0.231 408 822 NaN 0.189 921 550 0.181 509 826 0.390 181 864 0.174 210 793 0.258 609 502 NaN 0.298 000 362
2.4 PpWRKY 家族基因结构、保守基序及结构域分析
通过MEME 软件对46 个PpWRKY 蛋白的10个保守基序进行分析发现,Motif 1 和Motif 3 包括WRKY 七肽结构域,Motif 2 和Motif 10 是锌指基序,Motif 3 和Motif 10 组成了N 端WRKY 结构域,Motif 1 和Motif 2 组成了C 端WRKY 结构域(图5)。46 个富民枳WRKY 蛋白中都包含Motif 1 和Motif 2,而Motif 3 只存在于Ⅰa 亚组,说明Motif 1和Motif 2 在PpWRKY 蛋白中高度保守,可能具有重要的功能。不同组或亚组成员的基序组成有不同的特点。Ⅱa 亚组和Ⅱb 亚组基序组成相似,都具有Motif 1、Motif 2、Motif 5、Motif 6 和Motif 8 5 个基序,此外Ⅱb 亚组还特有一个Motif 9;Ⅱc 亚组成员除PpWRKY21 只有Motif 1 和Motif 2 外,其余成员皆还有Motif 4;Ⅱd、Ⅱe 亚组和Ⅲ组成员基序组成较为相似,除了 PpWRKY3、PpWRKY32 和PpWRKY33 缺失Motif 7 外,其余成员都由Motif 1、Motif 2 和Motif 7 组成。这些结果反映了WRKY 转录因子在富民枳进化过程中功能的多样性和保守性。
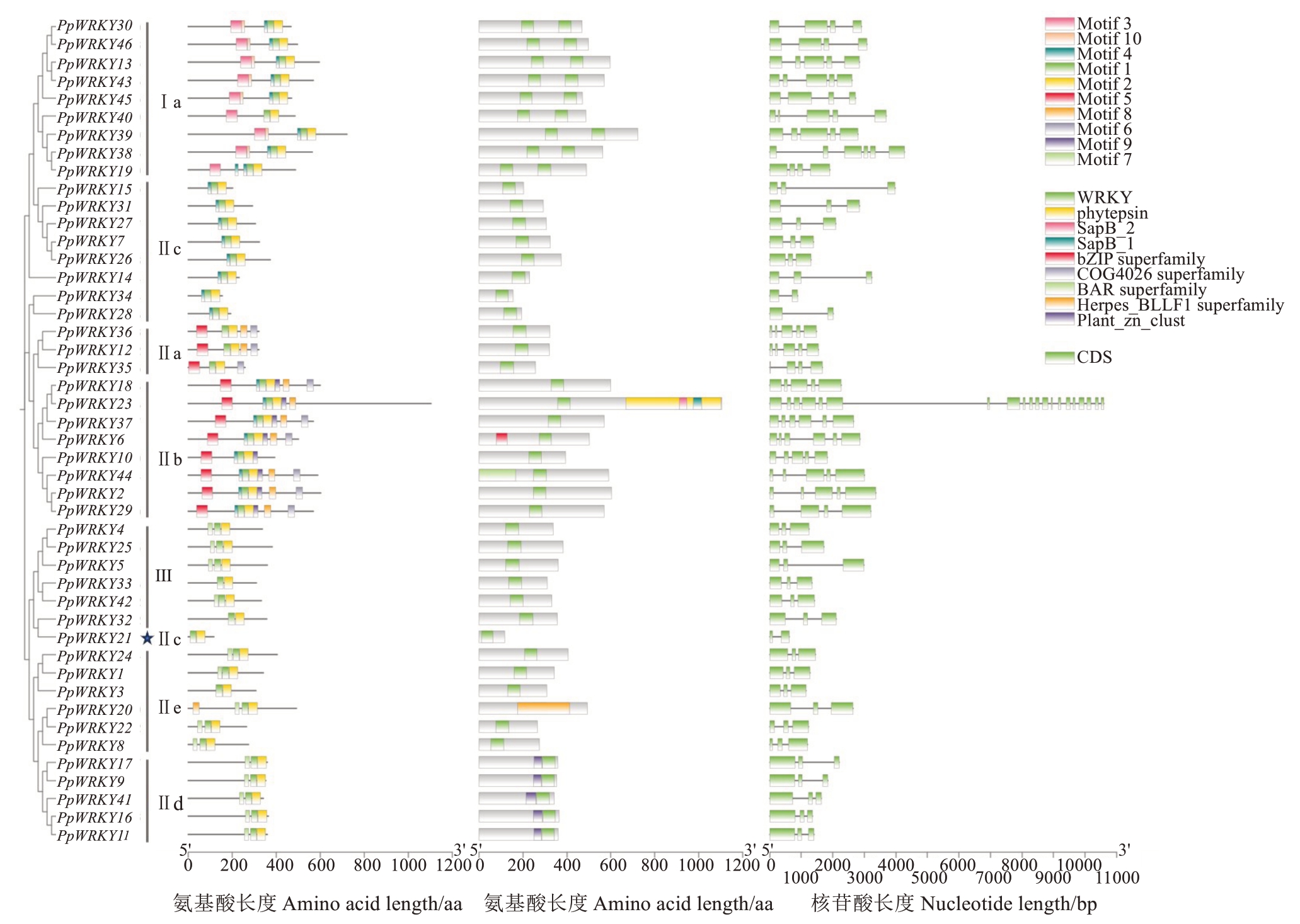
图5 PpWRKYs 的保守基序、保守结构域和基因结构
Fig. 5 Conserved motif, conserved domain and gene structure of PpWRKYs
保守结构域分析显示,46个PpWRKY蛋白中至少含有一个WRKY结构域,其中有9个PpWRKY蛋白还含有其他结构域,值得注意的是Ⅱd亚组的蛋白(PpWRKY9、PpWRKY17、PpWRKY41、PpWRKY11和 PpWRKY16)都含有植物锌簇结构域(Plant_zn_clust)(图5),但其功能尚不清楚。PpWRKY 家族基因结构分析显示,除PpWRKY23 基因的外显子数量(20 个)较多外,其余45 个PpWRKY基因的外显子数量为2 到6 个不等。其中,Ⅰa、Ⅱa和Ⅱb亚组WRKY 基因的外显子数量为4~6个;Ⅱd、Ⅱe 亚组和Ⅲ组WRKY 基因的外显子数量均为3 个;Ⅱc 亚组WRKY 基因的外显子数量为2~3 个,其中含有3 个外显子的基因占大部分。上述结果表明,WRKY 家族每个亚组的成员在基因结构上具有多样性和特异性。
2.5 富民枳WRKY基因启动子顺式作用元件分析
对富民枳WRKY 基因上游2000 bp 的启动子区域顺式作用元件进行预测的结果显示,PpWRKY 启动子中存在8 种与激素类和胁迫类响应相关的元件(图6)。其中,37 个PpWRKY 基因的启动子中含有184 个茉莉酸甲酯响应元件(TGACG-motif/CGTCA-motif);21 个PpWRKY 基因的启动子中有24 个胁迫响应元件(TC-rich repeats);26个PpWRKY基因含有38 个干旱诱导元件(MBS);37 个PpWRKY 基因含有132 个脱落酸响应元件(ABRE);15 个PpWRKY 基因含有18 个水杨酸响应元件(TCA-element);17 个PpWRKY 基因的启动子含有25 个低温响应元件(LTR)。推测PpWRKY 基因在富民枳激素调控和逆境胁迫中发挥着重要作用。
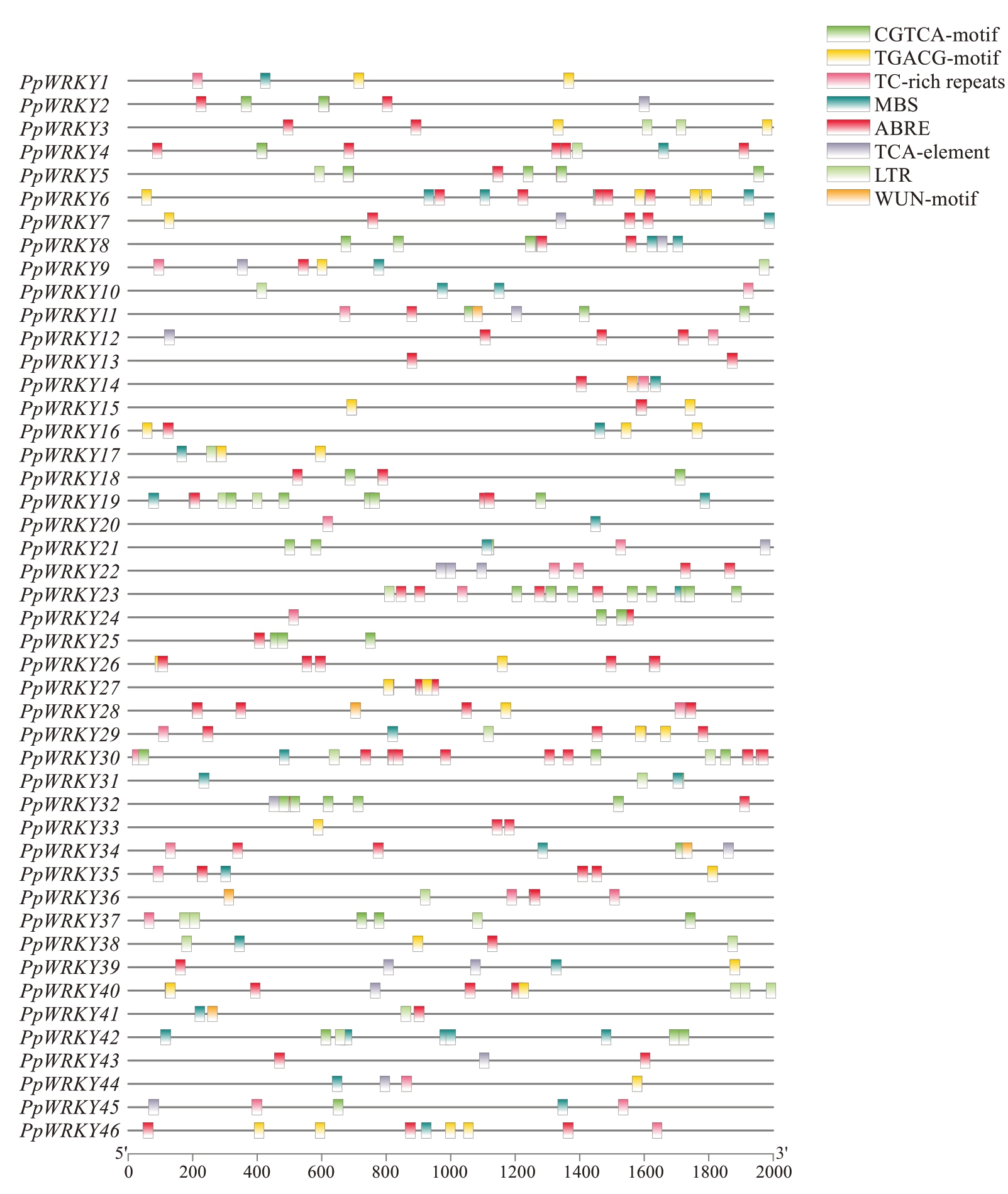
图6 PpWRKYs 家族成员启动子区域顺式作用元件分析
Fig. 6 Analysis of cis-acting elements in promoter regions of PpWRKYs family members
2.6 富民枳同源抗旱WRKY候选基因的鉴定
在枳中鉴定出53个WRKY基因后,对其在PEG-6000 模拟干旱处理后0、1.5、6 和24 h 的表达情况进行分析。结果显示,53 个枳的WRKY 基因按照其对干旱胁迫的应答模式可分为3 组,第1 组为Ptrif.0001s2122.1 等8 个基因,表达量随着模拟干旱胁迫时间的延长逐渐下降;第2 组为Ptrif.0006s0664.1等8 个基因,在干旱处理前期(1.5 h)表达量上升,后期(24 h)表达量下降;第3 组为Ptrif.0004s1310.1 等37 个基因,表达量随着干旱处理时间的延长而上升(图7),表明这些WRKY基因在干旱胁迫中发挥重要作用。 值得注意的是,Ptrif.0001s0079.1、Ptrif.0002s1584.1、Ptrif.0002s3086.1、Ptrif.0003s3779.1、Ptrif.0004s1310.1、Ptrif.0004s1919.1、Ptrif.0004s242-6.1、Ptrif.0007s1695.1 和Ptrif.0007s1914.1 基因表达水平较高且干旱处理后显著上调表达,表明这些基因在干旱胁迫中极为关键。将这9 个基因编码的氨基酸序列比对到富民枳基因组数据库,分别在富民枳中搜索到同源基因PpWRKY41、PpWRKY19、PpWRKY23、PpWRKY8、PpWRKY27、PpWRKY28、PpWRKY30、PpWRKY12 和PpWRKY13,且同源性都在95%以上,推测这9 个PpWRKY 基因也受干旱诱导表达,可能参与富民枳对干旱胁迫的防御反应。

图7 枳的WRKY 基因干旱胁迫下的表达模式
Fig. 7 Expression pattern of WRKY genes in Poncirus trifoliata under drought stress
2.7 实时荧光定量PCR(qRT-PCR)表达分析
为了进一步研究PpWRKY8、PpWRKY12、PpWRKY13、PpWRKY19、PpWRKY23、PpWRKY27、PpWRKY28、PpWRKY30 和PpWRKY41 基因在干旱胁迫下的表达特征,利用20%的PEG-6000模拟干旱试验,分别检测这9 个基因在干旱胁迫处理后0、3、6、12和24 h的表达水平。结果显示,与对照(0 h)相比,PpWRKY8 在干旱处理后3 h 表达量开始显著上调,在12 h 后达到峰值,为对照的2.60 倍;PpWRKY12、PpWRKY27、PpWRKY28 和PpWRKY30在干旱处理后3 h 表达量开始显著上调,在6 h 后达到峰值,分别为对照的3.63、2.63、3.22 和2.89 倍;PpWRKY19 和PpWRKY23 在干旱处理后6 h 和12 h表达量显著上调,其中在6 h 时表达量最高,分别为对照的2.25 倍和1.48 倍;PpWRKY13 在干旱处理后6 h 表达量开始显著上调,并在12 h 后达到峰值,为对照的2.90倍;PpWRKY41在干旱处理后6 h表达量开始显著上调,在24 h后达到峰值,为对照的2.92倍(图8)。以上结果表明,PpWRKY8、PpWRKY12、PpWRKY13、PpWRKY19、PpWRKY23、PpWRKY27、PpWRKY28、PpWRKY30 和PpWRKY41 基因均受干旱胁迫诱导表达,进一步证明这9 个WRKY 基因可能参与富民枳对干旱胁迫的防御反应。

图8 PpWRKYs 基因在干旱胁迫下的表达分析
Fig. 8 Expression analysis of PpWRKYs gene under drought stress
2.8 PpWRKY蛋白互作网络预测
为了探索PpWRKY 蛋白之间潜在的调控网络,笔者利用STRING 11.5 在线软件构建46 个PpWRKY 蛋白与调控植物耐旱性的MYB 蛋白(At-MYB15、AtMYB44 和AtMYB96)之间的蛋白质-蛋白质相互作用网络。预测结果显示,共有41 个PpWRKY 蛋白相互作用,其中AtWRKY40(PpWRKY12,36)属于网络中的中心节点,与17 个AtWRKY 蛋白相互作用(图9)。 AtWRKY33(PpWRKY13,43)、AtWRKY28(PpWRKY27)、At-WRKY11(PpWRKY41)和AtWRKY22(PpWRKY3,8)等蛋白均至少与8 个蛋白相互作用。此外,还预测到AtWRKY22(PpWRKY3,8)和AtWRKY41(PpWRKY4,25)与AtMYB44 蛋白相互作用,At-WRKY46(PpWRKY5)和AtWRKY48(PpWRKY26)与AtMYB15 蛋白相互作用,而AtWRKY33(PpWRKY13,43)、AtWRKY40(PpWRKY12,36)和AtWRKY70(PpWRKY33,42)蛋白与AtMYB44 和AtMYB15 蛋白均可相互作用。上述结果表明,PpWRKY 蛋白在富民枳对干旱胁迫的调控中发挥重要作用。
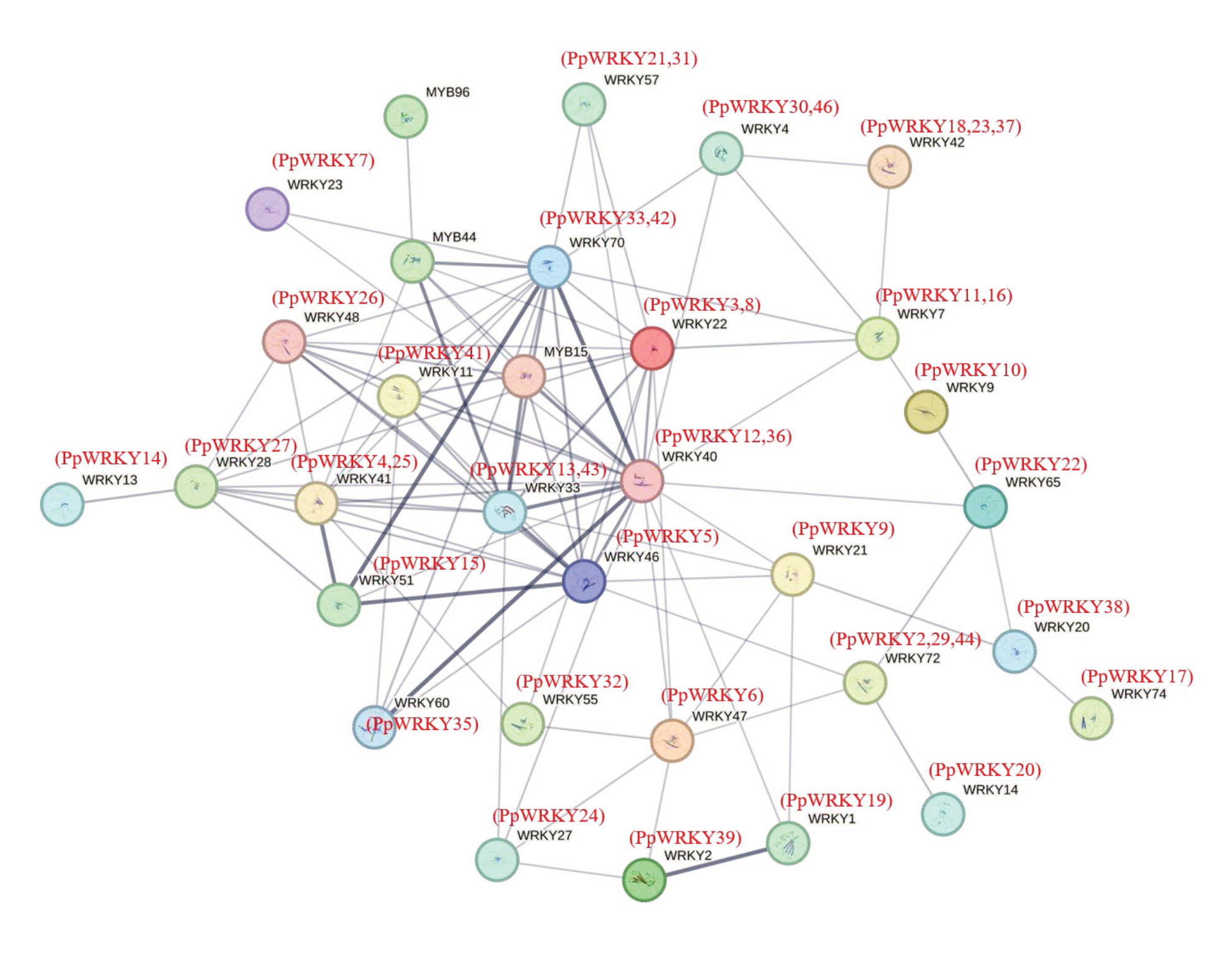
图9 PpWRKYs 蛋白互作网络
Fig. 9 PpWRKYs protein interaction network
3 讨 论
3.1 PpWRKY基因家族的结构特征
WRKY 基因家族大小具有物种特异性,尤其是草本植物中的WRKY 基因数量比木本植物高[30]。例如,茶树中只有50个WRKY基因[30],而在假俭草中却有92 个[31]。本研究对象富民枳为常绿小乔木,共鉴定出46 个WRKY 基因家族成员,与黄连(41 个)[32]、栀子(47 个)[33]及杜仲(51 个)[34]等木本植物中的WRKY 基因数量差距不大,但与拟南芥(74 个)[6]、水稻(98 个)[7]和大豆(188 个)[35]等草本植物相比,富民枳中的WRKY 基因数量明显减少,此结果在一定程度上支持Wu 等[30]的观点。Ⅱ组WRKY 转录因子在植物中的数量相对较多,且在进化上显示出较高的多样性,可使植物具有更强的环境适应性[36]。在本研究中,富民枳的46 个PpWRKY 被分为Ⅰ、Ⅱ和Ⅲ共3 组,其中Ⅱ组成员同样最多,占比最大(67%),并可分为5 个亚组,这有可能是富民枳抵抗干旱胁迫的重要原因。研究发现,短舌野生稻(Oryza barthii A. Chev.)、南方野生稻(Oryza meridionalis Ng.)和普通野生稻(Oryza rufipogon Grif.)等植物的WRKY 转录因子中都含有Ⅰb 亚组成员[37],但富民枳中未鉴定到Ⅰb 亚组的WRKY 转录因子,推测富民枳的WRKY基因家族进化速度较为缓慢。
相同或相似的基序是维系蛋白质结构域进而发挥生物学功能的重要前提[38]。富民枳PpWRKY 蛋白同一亚组基序分布基本一致,而各组间存在差异。基因结构分析发现,各亚组的WRKY 基因的外显子数量有所不同,但Ⅱd、Ⅱe 亚组和Ⅲ组WRKY基因的外显子数量是相同的,而Ⅰa、Ⅱa 和Ⅱb 亚组的外显子数量也较为相似。因此,推测这些外显子数量相同或相似的基因在功能上可能相同。WRKYGQK 序列的变异可能会导致WRKY 转录因子与DNA 结合能力的改变[39]。保守结构域多序列比对分析发现,富民枳46 个WRKY 蛋白都含有完整的WRKY 结构域,具备与DNA 序列中的顺式作用元件W-box 结合的能力。然而,PpWRKY 家族中Ⅱc亚组的2 个蛋白的WRKYGQK 七肽序列分别变异为 WRKYGKK(PpWRKY15)和 WKKYGQK(PpWRKY21),这两个基因与DNA 结合的能力可能会发生变化。在本研究中,PpWRKY 基因家族存在4 对串联重复基因和20 对片段复制基因,说明片段复制是富民枳WRKY 基因家族的主要扩增方式,这与香樟和绿豆WRKY 基因家族的扩增方式相似[9-10]。Ka/Ks>1 表示正选择,Ka/Ks=1 表示中性选择,Ka/Ks<1 表示纯化选择。富民枳20 对片段复制基因的Ka/Ks均低于1,证明这些基因在其进化历程中,可能借助纯化选择消除了有害的突变位点[40]。
3.2 PpWRKY基因家族的功能分析及表达特点
植物遭受干旱胁迫时会引起脱落酸(ABA)水平的增高[41-42]。ABA 作为响应逆境胁迫的内源激素,在植物抵抗干旱胁迫中起到重要作用[43]。ABA依赖性途径是植物响应干旱胁迫的信号转导途径之一[44]。在ABA 依赖性途径中,启动子区存在ABRE的基因通常会被ABA诱导,从而调控植物对干旱胁迫的响应[44]。在富民枳中,80.4%的PpWRKY基因启动子区含有132 个ABRE,说明大部分PpWRKY 基因可能通过ABA 依赖性途径参与干旱响应。启动子区含有干旱诱导元件(MBS)的基因可参与植物对干旱胁迫的响应[45]。例如,启动子区含有MBS 元件的AtMYC2基因,其表达受干旱和ABA的诱导,并且过表达AtMYC2的转基因植物对ABA具有更高的敏感性。在富民枳WRKY 基因启动子区存在38 个MBS 元件,进而推测富民枳通过MBS 元件响应干旱胁迫。
植物受到干旱胁迫时,WRKY 基因可被快速诱导表达,从而触发一个信号级联网络,最终增强植物的抗逆性[46]。如玉米ZmWRKY40基因的表达会受干旱、盐害、高温和ABA 诱导[47]。甘蔗ScWRKY5 基因能够被盐胁迫和干旱胁迫诱导表达[48]。杜梨PbrWRKY53 基因被干旱和ABA 诱导大幅上调表达,并且在烟草和杜梨中过表达PbrWRKY53基因都能显著增强对干旱胁迫的抗性[49]。在本研究中,富民枳近缘种枳的大部分WRKY基因在干旱胁迫下表达量上调,表明这些WRKY 基因在枳对干旱胁迫的响应中可能起到了正向调控的作用。虽然在本研究中富民枳未测干旱胁迫下的转录组数据,但枳与富民枳亲缘关系近[22],因此利用枳同源WRKY 基因在干旱胁迫下的表达情况筛选富民枳的抗旱基因成为一种可能。PpWRKY12、PpWRKY13 和PpWRKY23分别与拟南芥中的AtWRKY40(AT1G80840)、At-WRKY33(AT2G38470)和AtWRKY6(AT1G62300)聚在一起。研究发现,AtWRKY40 在干旱胁迫下会被诱导表达,并且能够通过调节植株的抗氧化能力和渗透调节能力来参与拟南芥对干旱胁迫的响应过程[50];AtWRKY33受渗透胁迫和ABA 诱导表达,并参与调控包括干旱在内的多种逆境胁迫[51];AtWRKY6的表达受到ABA 的诱导,并且其编码蛋白可通过直接抑制RAV1基因的表达来增强ABA 信号转导途径中的关键基因ABI3、ABI4 和ABI5 的表达[52]。因此,推测富民枳中的PpWRKY12、PpWRKY13 和PpWRKY23也具有响应干旱胁迫的功能。笔者在本研究中发现,PpWRKY12、PpWRKY13 和PpWRKY23基因的启动子中均存在ABA 响应元件,其中,PpWRKY12 和PpWRKY23 还分别含有胁迫响应元件和干旱诱导元件,并且qRT-PCR 试验证明这3 个基因均受干旱胁迫诱导表达。这些结果说明PpWRKY12、PpWRKY13 和PpWRKY23 基因可参与富民枳抵御干旱胁迫的过程。此外,PpWRKY8、PpWRKY19、PpWRKY27、PpWRKY28、PpWRKY30和PpWRKY41基因分别与拟南芥中的AtWRKY22、AtWRKY1、AtWRKY71、AtWRKY75、AtWRKY4 和AtWRKY17 基因的序列相似性较高,但拟南芥的这6 个基因在抗旱方面的研究未见报道。笔者在本研究中发现,PpWRKY8、PpWRKY19、PpWRKY27、PpWRKY28、PpWRKY30和PpWRKY41基因均含脱落酸响应元件,除此以外,PpWRKY8、PpWRKY19、PpWRKY30 和PpWRKY41 基因还存在干旱诱导元件,而PpWRKY28 基因还含有胁迫响应元件。因此,推测这6 个基因可能直接或间接被干旱诱导表达。为验证这6个基因是否能被干旱胁迫诱导,检测了他们在干旱胁迫处理后的表达水平,结果发现这6个基因确实受干旱胁迫诱导表达,其中PpWRKY19、PpWRKY27、PpWRKY28 和PpWRKY30 基因在干旱处理6 h后表达量达到峰值,说明这4个PpWRKY基因可能在干旱胁迫早期发挥关键作用。
3.3 PpWRKY蛋白互作预测
蛋白互作预测有助于研究PpWRKY蛋白的调控机制和功能。AtWRKY40(PpWRKY12,36)属于网络中的中心节点,与多个蛋白相互作用,可能在富民枳生长发育及生物和非生物胁迫中发挥关键作用。前期研究发现,拟南芥的MYB44和MYB15蛋白通过ABA信号可以促进气孔关闭,提高植物对干旱的耐受性[53]。在本研究中,蛋白互作预测发现AtWRKY22(PpWRKY3,8)和AtWRKY41(PpWRKY4,25)可与AtMYB44 蛋白相互作用,AtWRKY46(PpWRKY5)和AtWRKY48(PpWRKY26)可与AtMYB15 蛋白相互作用,而AtWRKY33(PpWRKY13,43)、AtWRKY40(PpWRKY12,36)和AtWRKY70(PpWRKY33,42)蛋白与这两个MYB 蛋白均可相互作用,推测PpWRKY3、PpWRKY4、PpWRKY5、PpWRKY8、PpWRKY12、 PpWRKY13、 PpWRKY25和PpWRKY26、PpWRKY33、PpWRKY36、PpWRKY42和PpWRKY43 可能与MYB 蛋白互作,从而通过ABA 信号参与调控气孔运动来影响植物的耐旱性。AtMYB96 的过表达可以促进表皮蜡质生物合成上调,从而提高拟南芥的耐旱性[54]。AtMYB96 虽然未与WRKY 蛋白直接互作,但通过与AtMYB44互作,间接参与WRKY 蛋白对干旱的调控。综上所述,本研究为进一步探索WRKY 转录因子在富民枳中的抗旱机制了提供理论依据。
4 结 论
笔者从富民枳中鉴定到46 个WRKY 基因,其不均匀地分布在9 条染色体上,以片段复制作为基因家族扩张的主要方式。PpWRKY基因家族可能与植物抵御逆境胁迫密切相关。qRT-PCR 试验证明,PpWRKY8、PpWRKY12、PpWRKY13、PpWRKY19、PpWRKY23、PpWRKY27、PpWRKY28、PpWRKY30 和PpWRKY41可能参与富民枳对干旱胁迫的响应。蛋白互作结果表明,有12 个PpWRKY 蛋白可能通过MYB 转录因子介导ABA 信号参与调控气孔运动来影响植物的耐旱性。研究结果可为后续进行PpWRKY基因的抗旱功能和调控机制研究提供参考依据。
[1] 杜超. WRKY 转录因子家族在植物响应逆境胁迫中的功能及应用[J]. 草业科学,2021,38(7):1287-1300.DU Chao. Function and application of the WRKY transcription factor superfamily in plant response to stresses[J]. Pratacultural Science,2021,38(7):1287-1300.
[2] EULGEM T,RUSHTON P J,ROBATZEK S,SOMSSICH I E.The WRKY superfamily of plant transcription factors[J]. Trends in Plant Science,2000,5(5):199-206.
[3] WU K L,GUO Z J,WANG H H,LI J. The WRKY family of transcription factors in rice and Arabidopsis and their origins[J].DNA Research,2005,12(1):9-26.
[4] 阮孙美,张攀,张敏,曾千春,张慧,罗琼. 疣粒野生稻WRKY基因家族全基因组鉴定和分析[J]. 植物遗传资源学报,2024,25(4):629-646.RUAN Sunmei,ZHANG Pan,ZHANG Min,ZENG Qianchun,ZHANG Hui,LUO Qiong. Genome-wide identification and bioinformatics analysis of WRKY gene family in wild rice species,Oryza granulata[J]. Journal of Plant Genetic Resources,2024,25(4):629-646.
[5] ISHIGURO S,NAKAMURA K. Characterization of a cDNA encoding a novel DNA-binding protein,SPF1 that recognizes SP8 sequences in the 5' upstream regions of genes coding for sporamin and beta-amylase from sweet potato[J]. Molecular & General Genetics,1994,244(6):563-571.
[6] EULGEM T,SOMSSICH I E. Networks of WRKY transcription factors in defense signaling[J]. Current Opinion in Plant Biology,2007,10(4):366-371.
[7] ROSS C A,LIU Y,SHEN Q J. The WRKY gene family in rice(Oryza sativa)[J]. Journal of Integrative Plant Biology,2007,49(6):827-842.
[8] GARRIDO-GALA J,HIGUERA J J,RODRÍGUEZ-FRANCO A,MUÑOZ-BLANCO J,AMIL-RUIZ F,CABALLERO J L. A comprehensive study of the WRKY transcription factor family in strawberry[J]. Plants,2022,11(12):1585.
[9] 倪辉,孙维红,丁乐,曾伟伟,邹双全. 香樟全基因组WRKY 基因家族的鉴定与分析[J]. 植物科学学报,2022,40(4):513-523.NI Hui,SUN Weihong,DING Le,ZENG Weiwei,ZOU Shuangquan. Identification and analysis of the WRKY gene family in whole genome of Cinnamomum camphora (L.) Presl.[J]. Plant Science Journal,2022,40(4):513-523.
[10] 郝青婷,高伟,闫虎斌,张泽燕,朱慧珺,王茜,赵雪英,张耀文. 绿豆WRKY 基因家族的全基因组鉴定及生物信息学分析[J]. 西北农林科技大学学报(自然科学版),2023,51(5):59-71.HAO Qingting,GAO Wei,YAN Hubin,ZHANG Zeyan,ZHU Huijun,WANG Qian,ZHAO Xueying,ZHANG Yaowen. Genome-wide identification and bio-informatics analysis of WRKY gene family in Vigna radiata[J]. Journal of Northwest A & F University (Natural Science Edition),2023,51(5):59-71.
[11] 赵爽,葛朝红,石鹤飞,闵卓,王广鹏,李伟明. 板栗WRKY 基因家族鉴定及其在干旱胁迫下的表达分析[J]. 华北农学报,2024,39(1):72-82.ZHAO Shuang,GE Chaohong,SHI Hefei,MIN Zhuo,WANG Guangpeng,LI Weiming. Identification and expression analysis of the chestnut WRKY gene family under drought stress[J]. Acta Agriculturae Boreali-Sinica,2024,39(1):72-82.
[12] 欧阳梦真,朱磊,孙治强,李胜利,吴帼秀,李阳,何富豪,李严曼. 西瓜ClWRKY54 基因的克隆、亚细胞定位及表达分析[J].中国瓜菜,2019,32(12):8-14.OUYANG Mengzhen,ZHU Lei,SUN Zhiqiang,LI Shengli,WU Guoxiu,LI Yang,HE Fuhao,LI Yanman. Cloning,subcellular localization and expression analysis of ClWRKY54 in Citrullus lanatus[J]. China Cucurbits and Vegetables,2019,32(12):8-14.
[13] SHAFFIQUE S,KHAN M A,IMRAN M,KANG S M,PARK Y S,WANI S H,LEE I J. Research progress in the field of microbial mitigation of drought stress in plants[J]. Frontiers in Plant Science,2022,13:870626.
[14] ZHANG J W,HUANG D Z,ZHAO X J,ZHANG M,WANG Q,HOU X Y,DI D L,SU B B,WANG S K,SUN P. Drought-responsive WRKY transcription factor genes IgWRKY50 and Ig-WRKY32 from Iris germanica enhance drought resistance in transgenic Arabidopsis[J]. Frontiers in Plant Science,2022,13:983600.
[15] CAI H Y,YANG S,YAN Y,XIAO Z L,CHENG J B,WU J,QIU A L,LAI Y,MOU S L,GUAN D Y,HUANG R H,HE S L. CaWRKY6 transcriptionally activates CaWRKY40,regulates Ralstonia solanacearum resistance,and confers high-temperature and high-humidity tolerance in pepper[J]. Journal of Experimental Botany,2015,66(11):3163-3174.
[16] QIAO Z,LI C L,ZHANG W. WRKY1 regulates stomatal movement in drought-stressed Arabidopsis thaliana[J]. Plant Molecular Biology,2016,91(1/2):53-65.
[17] QIU Y P,YU D Q. Over-expression of the stress-induced Os-WRKY45 enhances disease resistance and drought tolerance in Arabidopsis[J]. Environmental and Experimental Botany,2009,65(1):35-47.
[18] NIU C F,WEI W,ZHOU Q Y,TIAN A G,HAO Y J,ZHANG W K,MA B,LIN Q,ZHANG Z B,ZHANG J S,CHEN S Y.Wheat WRKY genes TaWRKY2 and TaWRKY19 regulate abiotic stress tolerance in transgenic Arabidopsis plants[J]. Plant,Cell& Environment,2012,35(6):1156-1170.
[19] CHEN W,ZHENG C,YAO M Z,CHEN L. The tea plant Cs-WRKY26 promotes drought tolerance in transgenic Arabidopsis plants[J]. Beverage Plant Research,2021,1:3.
[20] JIA H H,WANG C,WANG F,LIU S C,LI G L,GUO X Q. Gh-WRKY68 reduces resistance to salt and drought in transgenic Nicotiana benthamiana[J]. PLoS One,2015,10(3):e0120646.
[21] 张珊珊,甘云浩,杨文忠,段宗亮,丁红茜,康红梅,诺苏那玛.富民枳种质资源的SSR 遗传多样性研究[J]. 西部林业科学,2018,47(4):14-20.ZHANG Shanshan,GAN Yunhao,YANG Wenzhong,DUAN Zongliang,DING Hongqian,KANG Hongmei,NUO Sunama.Genetic diversity of Poncirus polyandra resources[J]. Journal of West China Forestry Science,2018,47(4):14-20.
[22] 张余,龚洵,冯秀彦. 利用DNA 片段测序方法探究枳属和富民枳的分类地位[J]. 广西植物,2021,41(1):114-122.ZHANG Yu,GONG Xun,FENG Xiuyan. Phylogenetic position of Poncirus and Poncirus polyandra by DNA sequencing[J].Guihaia,2021,41(1):114-122.
[23] 周开兵,夏仁学. 中国柑橘砧木选择研究进展与展望[J]. 中国农学通报,2005,21(1):213-218.ZHOU Kaibing,XIA Renxue. The proceedings and tendencies in the study on the choice of rootstocks for citrus in China[J].Chinese Agricultural Science Bulletin,2005,21(1):213-218.
[24] 吴玉,陶晓宁,袁河清,易敏,王辉,杨利平. 基于4 个核基因系统发育树分析华中枳和富民枳的分类地位[J]. 果树学报,2024,41(10):1979-1989.WU Yu,TAO Xiaoning,YUAN Heqing,YI Min,WANG Hui,YANG Liping. Taxonomic status of Citrus × pubinervia and Citrus × polytrifolia based on the phylogenetic trees reconstructed by four nuclear genes[J]. Journal of Fruit Science,2024,41(10):1979-1989.
[25] PENG Z,BREDESON J V,WU G A,SHU S Q,RAWAT N,DU D L,PARAJULI S,YU Q B,YOU Q,ROKHSAR D S,Jr GMITTER F G,DENG Z N. A chromosome-scale reference genome of trifoliate orange (Poncirus trifoliata) provides insights into disease resistance,cold tolerance and genome evolution in Citrus[J]. The Plant Journal,2020,104(5):1215-1232.
[26] YU X,REN Y L,LIU X F,JIANG D,ZHU S P,SHEN W X,ZHAO W T,ZHAO X C,CAO L. Whole transcriptome analysis of trifoliate orange [Poncirus trifoliata (L.) Raf.] under osmotic stress[J]. Scientia Horticulturae,2023,317:112045.
[27] ZHANG S S,CHEN J,ZHANG C G,ZHANG S Q,ZHANG X L,GAO L J,YANG W Z. Insights into identifying resistance genes for cold and disease stresses through chromosome-level reference genome analyses of Poncirus polyandra[J]. Genomics,2023,115(3):110617.
[28] YU G C. Using ggtree to visualize data on tree-like structures[J].Current Protocols in Bioinformatics,2020,69(1):e96.
[29] LIVAK K J,SCHMITTGEN T D. Analysis of relative gene expression data using real-time quantitative PCR and the 2 (-Delta Delta C(T)) method[J]. Methods,2001,25(4):402-408.
[30] WU Z J,LI X H,LIU Z W,LI H,WANG Y X,ZHUANG J.Transcriptome-wide identification of Camellia sinensis WRKY transcription factors in response to temperature stress[J]. Molecular Genetics and Genomics,2016,291(1):255-269.
[31] 于元平,蒋宇佳,孙向一,吴春妍,周敏,刘明稀. 假俭草WRKY家族基因鉴定及其响应干旱胁迫表达分析[J]. 草地学报,2024,32(5):1378-1391.YU Yuanping,JIANG Yujia,SUN Xiangyi,WU Chunyan,ZHOU Min,LIU Mingxi. Identification of the WRKY family genes and thier expression analysis in response to drought stress in centipedegrass[J]. Acta Agrestia Sinica,2024,32(5):1378-1391.
[32] 刘微,蒋莉萍,池玉洁,刘义飞,陈士林,刘迪. 黄连WRKY 基因家族鉴定及表达分析[J]. 世界科学技术-中医药现代化,2022,24(4):1369-1380.LIU Wei,JIANG Liping,CHI Yujie,LIU Yifei,CHEN Shilin,LIU Di. Identification and expression analysis of WRKY gene family in Coptis chinensis[J]. Modernization of Traditional Chinese Medicine and Materia Medica-World Science and Technology,2022,24(4):1369-1380.
[33] 陈啸,陈淑颖,朱渊铭,倪林,邹双全. 栀子WRKY 基因家族鉴定及其响应盐胁迫的表达模式[J]. 西南农业学报,2024,37(3):503-512.CHEN Xiao,CHEN Shuying,ZHU Yuanming,NI Lin,ZOU Shuangquan. Identification of WRKY gene family in Gardenia jasminoides and its expression patterns in response to salt stress[J].Southwest China Journal of Agricultural Sciences,2024,37(3):503-512.
[34] 庆军,杜庆鑫,朱利利,何凤,杜红岩. 杜仲全基因组WRKY 转录因子基因家族鉴定及表达分析[J]. 基因组学与应用生物学,2021,40(增刊1):2276-2289.QING Jun,DU Qingxin,ZHU Lili,HE Feng,DU Hongyan. Genome-wide identification and expression analysis of WRKY transcription factor family in Eucommia ulmoides[J]. Genomics and Applied Biology,2021,40(Suppl. 1):2276-2289.
[35] 陈林英,李佳佳,王博,杜婉清,高梦雪,刘慧,檀淑琴,邱丽娟,王晓波. WRKY 转录因子在大豆响应生物和非生物胁迫中的功能研究进展[J]. 植物遗传资源学报,2022,23(2):323-332.CHEN Linying,LI Jiajia,WANG Bo,DU Wanqing,GAO Mengxue,LIU Hui,TAN Shuqin,QIU Lijuan,WANG Xiaobo.Research progress on the function of WRKY transcription factor response to biotic and abiotic stresses in soybean[J]. Journal of Plant Genetic Resources,2022,23(2):323-332.
[36] GOYAL P,DEVI R,VERMA B,HUSSAIN S,ARORA P,TABASSUM R,GUPTA S. WRKY transcription factors:Evolution,regulation,and functional diversity in plants[J]. Protoplasma,2023,260(2):331-348.
[37] VILLACASTIN A J,ADAMS K S,BOONJUE R,RUSHTON P J,HAN M,SHEN J Q. Dynamic differential evolution schemes of WRKY transcription factors in domesticated and wild rice[J].Scientific Reports,2021,11(1):14887.
[38] 吴圳,张明英,闫锋,李依民,高静,颜永刚,张岗. 掌叶大黄(Rheum palmatum L.)WRKY 基因家族鉴定与分析[J]. 生物技术通报,2024,40(1):250-261.WU Zhen,ZHANG Mingying,YAN Feng,LI Yimin,GAO Jing,YAN Yonggang,ZHANG Gang. Identification and analysis of WRKY gene family in Rheum palmatum L.[J]. Biotechnology Bulletin,2024,40(1):250-261.
[39] CHEN F,HU Y,VANNOZZI A,WU K C,CAI H Y,QIN Y,MULLIS A,LIN Z G,ZHANG L S. The WRKY transcription factor family in model plants and crops[J]. Critical Reviews in Plant Sciences,2017,36(5/6):311-335.
[40] 郭纯,宋桂梅,闫艳,邸鹏,王英平. 西洋参bZIP 基因家族全基因组鉴定和表达分析[J]. 生物技术通报,2024,40(4):167-178.GUO Chun,SONG Guimei,YAN Yan,DI Peng,WANG Yingping. Genome wide identification and expression analysis of the bZIP gene family in Panax quinquefolius[J]. Biotechnology Bulletin,2024,40(4):167-178.
[41] ZHANG J H,JIA W S,YANG J C,ISMAIL A M. Role of ABA in integrating plant responses to drought and salt stresses[J].Field Crops Research,2006,97(1):111-119.
[42] LATA C R,PRASAD M. Role of DREBs in regulation of abiotic stress responses in plants[J]. Journal of Experimental Botany,2011,62(14):4731-4748.
[43] ZHU J K. Abiotic stress signaling and responses in plants[J].Cell,2016,167(2):313-324.
[44] 王冰,程宪国. 干旱、高盐及低温胁迫下植物生理及转录因子的应答调控[J]. 植物营养与肥料学报,2017,23(6):1565-1574.WANG Bing,CHENG Xianguo. Physiological responses and regulatory pathways of transcription factors in plants under drought,high-salt,and low temperature stresses[J]. Journal of Plant Nutrition and Fertilizer,2017,23(6):1565-1574.
[45] ABE H,URAO T,ITO T,SEKI M,SHINOZAKI K,YAMAGUCHI-SHINOZAKI K. Arabidopsis AtMYC2 (bHLH) and At-MYB2 (MYB) function as transcriptional activators in abscisic acid signaling[J]. The Plant Cell,2003,15(1):63-78.
[46] JIANG J J,MA S H,YE N H,JIANG M,CAO J S,ZHANG J H. WRKY transcription factors in plant responses to stresses[J].Journal of Integrative Plant Biology,2017,59(2):86-101.
[47] WANG C T,RU J N,LIU Y W,YANG J F,LI M,XU Z S,FU J D. The maize WRKY transcription factor ZmWRKY40 confers drought resistance in transgenic Arabidopsis[J]. International Journal of Molecular Sciences,2018,19(9):2580.
[48] WANG D J,WANG L,SU W H,REN Y J,YOU C H,ZHANG C,QUE Y X,SU Y C. A class III WRKY transcription factor in sugarcane was involved in biotic and abiotic stress responses[J].Scientific Reports,2020,10(1):20964.
[49] LIU Y,YANG T Y,LIN Z K,GU B J,XING C H,ZHAO L Y,DONG H Z,GAO J Z,XIE Z H,ZHANG S L,HUANG X S. A WRKY transcription factor PbrWRKY53 from Pyrus betulaefolia is involved in drought tolerance and AsA accumulation[J].Plant Biotechnology Journal,2019,17(9):1770-1787.
[50] 车永梅,孙艳君,卢松冲,赵方贵,侯丽霞,刘新. AtWRKY40参与拟南芥干旱胁迫响应过程[J]. 植物生理学报,2018,54(3):456-464.CHE Yongmei,SUN Yanjun,LU Songchong,ZHAO Fanggui,HOU Lixia,LIU Xin. AtWRKY40 functions in drought stress response in Arabidopsis thaliana[J]. Plant Physiology Journal,2018,54(3):456-464.
[51] CHEN Y H,ZHANG J. Multiple functions and regulatory networks of WRKY33 and its orthologs[J]. Gene,2024,931:148899.
[52] HUANG Y,FENG C Z,YE Q,WU W H,CHEN Y F. Arabidopsis WRKY6 transcription factor acts as a positive regulator of abscisic acid signaling during seed germination and early seedling development[J]. PLoS Genetics,2016,12(2):e1005833.
[53] WANG X P,NIU Y L,ZHENG Y. Multiple functions of MYB transcription factors in abiotic stress responses[J]. International Journal of Molecular Sciences,2021,22(11):6125.
[54] LEE S B,KIM H,KIM R J,SUH M C. Overexpression of Arabidopsis MYB96 confers drought resistance in Camelina sativa via cuticular wax accumulation[J]. Plant Cell Reports,2014,33(9):1535-1546.