梨亚科(Pomoideae)中梨属(Pyrus L.)、苹果属(Malus Mill.)、枇杷属(Eriobotrya Lindl.)、山楂属(Crataegus L.)和榅桲属(Cydonia Mill.)中有许多人们喜欢食用的康养果品,如梨(Pyrus L.)[1]、苹果(Malus domestica Borkh.)[2]、枇杷[Eriobotrya japonica(Thunb.) Lindl.][3]、山楂(Crataegus pinnatifida Bge.)[4]和榅桲(Cydonia oblonga Mill.)[5]等。这些果树在进化过程中为了避免自花授粉,普遍存在自交不亲和现象[6-7],其自交不亲和现象由雌蕊S-RNase和花粉F-box(SFBB)相互作用实现[6-8]。
根据已有梨亚科5 个属S-RNase 基因一级结构特征[9],其N 端到C 端分别为信号肽(Signal peptide,SP)、保守区(Conserved region)C1、保守区C2、高变区HV(High variable region,包含内含子intron)、保守区C3、保守区C4、保守区C5。除了SP 和保守区C1 区外,其他相邻区域间以及C5 后面还存在其他序列,其中HV区被公认为是S-RNase基因特异识别的重要区域,同时该区域也是鉴定梨亚科5 个属SRNase 新基因的重要指标[9]。近些年,随着梨亚科5个属越来越多的S-RNase 基因被分离鉴定,其基因进化也逐渐成为研究的热点之一。目前,关于梨亚科5个属S-RNase基因进化的相关研究较少,且研究多以S-RNase 基因序列片段为基础[10]。近些年随着测序技术的不断完善,梨亚科5个属的S-RNase基因CDS(Coding Sequence)全长序列被分离鉴定;有误序列得到了更正[11]。因此,有必要利用最新数据对现有梨亚科5 个属的S-RNase 基因序列特征和进化情况进行研究,以便反映更真实的情况。笔者在本研究中收集整理梨亚科5 个属S-RNase 基因CDS 全长序列、大于330 bp的S-RNase基因外显子片段序列和intron 序列,分析其序列特征和密码子使用情况。同时,利用其基因CDS 全长、intron 序列和密码子使用偏好分别构建进化树,从3 个方面反映其进化情况。通过对梨亚科5个属最新数据的整理和分析,可以为研究者提供精准的S-RNase基因数据;研究发现新的序列位点符合参与自交不亲和识别的特征,为目前陷入停滞的梨亚科自交不亲和研究提供参考。
1 材料和方法
1.1 试验材料
2024 年4 月10 日前NCBI 的GenBank 数据库中收录的梨亚科苹果属、梨属、榅桲属、山楂属、枇杷属S-RNase基因CDS全长序列、大于330 bp的S-RNase基因外显子片段序列以及HV区内含子序列。
1.2 试验方法
1.2.1 序列获取 全长序列分别采用关键词Malus S-RNase complete cds,Pyrus S-RNase complete cds,Cydonia S-RNase complete cds,Crataegus S-RNase complete cds 和Eriobotrya S-RNase complete cds 进行搜索,并利用Blast工具进行搜索补充;大于330 bp的外显子片段序列也采用上述方法,其中的complete替换为partial。
1.2.2 基因序列分析 利用VectorNTI11.5.3软件进行基因序列分析,并人工加以纠正。
1.2.3 梨亚科5 个属S-RNase 基因分化及多态性利用MEGA11 软件分别筛选出梨亚科5 个属SRNase 基因CDS 全长序列、片段序列和intron 序列适合的最佳数学模型,选择该模型计算各序列分化数据,同时利用Tajima’s Test 计算各序列的遗传多态性。
1.2.4 密码子使用偏好分析 利用MEGA11 软件自带Compute nucleotide composition 和Compute codon usage bias程序分别计算序列碱基组成比例情况和RSCU(Relative synonymous codon usage)值。
1.2.5 系统进化分析 用MEGA11软件的ClustalW程序对梨亚科5 个属S-RNase 基因CDS 全长序列和HV 区内含子序列分别作序列比对,利用邻接法分别构建二者的进化树;利用SPSS22 对其CDS 全长序列RSCU 以欧式平方距离作为基因间进化距离进行聚类分析。
2 结果与分析
2.1 基因序列分析
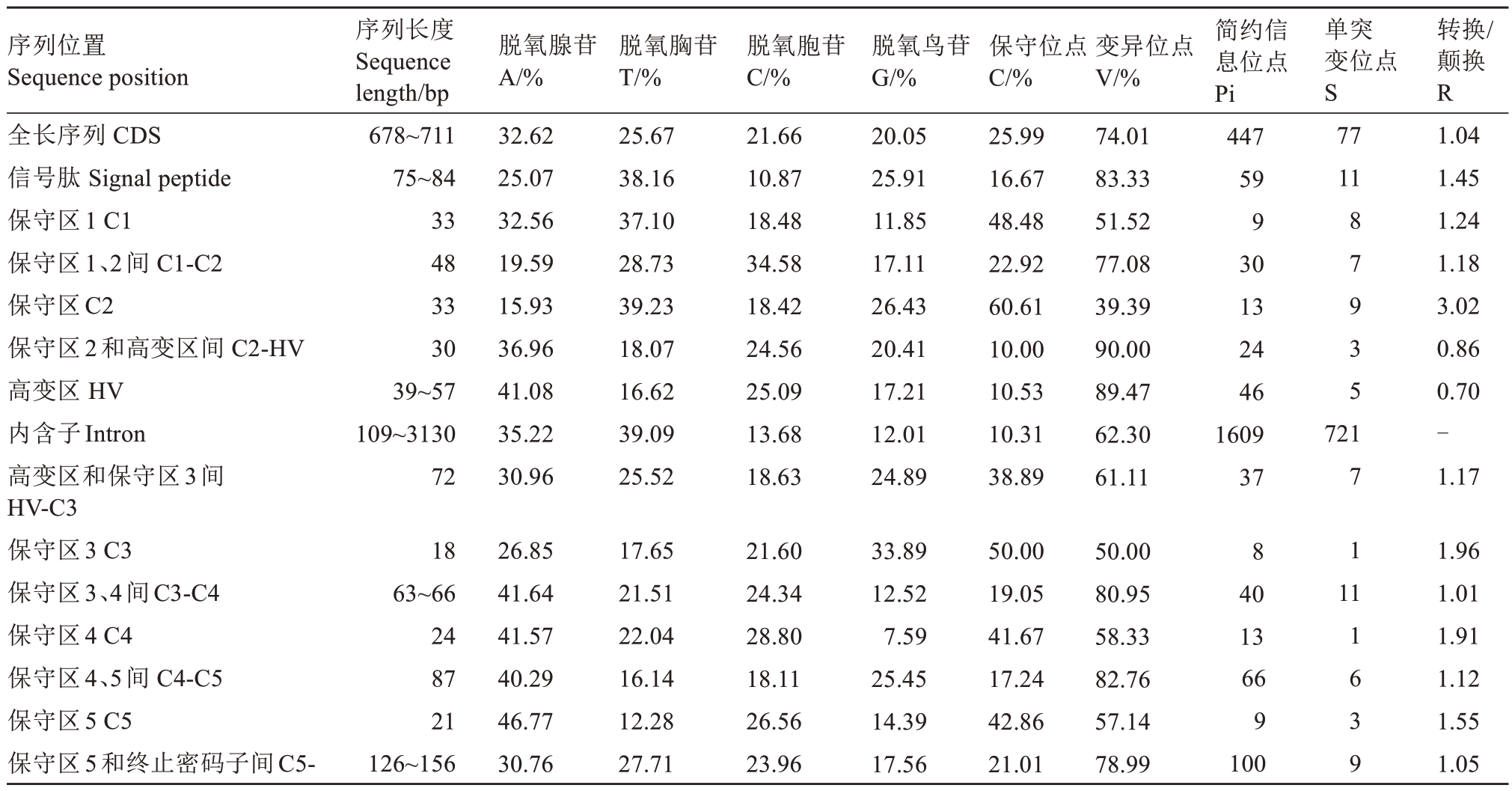
2.1.1 外显子序列分析 GenBank数据库中收录梨亚科5个属S-RNase基因CDS全长序列120条,剔除重复序列后共90条。GenBank数据库中收录梨亚科5 个属S-RNase 基因大于330 bp 的外显子片段序列140条,其中梨属51条,苹果属35条,榅桲属5条,枇杷属29条,山楂属20条。分析梨亚科5个属90条SRNase基因CDS全长序列,结果(表1)显示其长度为678~711 bp。其中678 bp 1 条,681 bp 17 条,684 bp 26 条,687 bp 33 条,690 bp 6 条,699 bp 7 条,711 bp 1 条,平均长度为686 bp。由表1可以看出,梨亚科5个属S-RNase基因SP、HV、intron、C3-C4(C3、C4间)和C5-区域序列长度存在多态性,且以intron 变化最大,其次为HV、C5-、SP、C3-C4,其余部位序列长度均无变化。SP 序列长度:81 bp 77 条,84 bp 2 条,78 bp 9 条,75 bp 2 条,SP 区域主要发生替换,其次为缺失和插入;HV 区序列长度:57 bp 7 条,48 bp 5条,45 bp 53条,42 bp 13条,39 bp 12条,该区域发生缺失的现象远大于插入的情况;分布在HV 区的intron 序列大小为109~3130 bp,具有丰富的长度多态性;C5-序列长度:126 bp 4 条,129 bp 16 条,132 bp 60条,135 bp 9 条,156 bp 1条,主要发生为缺失和插入;另外C3-C4也出现一处插入的情况。
表1 梨亚科5 个属S-RNase 序列特征
Table 1 Characteristics of S-RNase sequences among five genera Pomoideae
注:“-”表示不适用项。
Note:“-” indicates not applicable.
序列位置Sequence position全长序列 CDS信号肽 Signal peptide保守区1 C1保守区1、2间 C1-C2保守区C2保守区2和高变区间 C2-HV高变区 HV内含子Intron高变区和保守区3间HV-C3保守区3 C3保守区3、4间C3-C4保守区4 C4保守区4、5间 C4-C5保守区5 C5保守区5和终止密码子间C5-序列长度Sequence length/bp 678~711 75~84 33 48 33 30 39~57 109~3130 72脱氧腺苷A/%32.62 25.07 32.56 19.59 15.93 36.96 41.08 35.22 30.96脱氧胸苷T/%25.67 38.16 37.10 28.73 39.23 18.07 16.62 39.09 25.52脱氧胞苷C/%21.66 10.87 18.48 34.58 18.42 24.56 25.09 13.68 18.63脱氧鸟苷G/%20.05 25.91 11.85 17.11 26.43 20.41 17.21 12.01 24.89保守位点C/%25.99 16.67 48.48 22.92 60.61 10.00 10.53 10.31 38.89变异位点V/%74.01 83.33 51.52 77.08 39.39 90.00 89.47 62.30 61.11简约信息位点Pi 447 59 9 30 13 24 46 1609 37单突变位点S 77 11 8 7 9 3 5 721转换/颠换R 1.04 1.45 1.24 1.18 3.02 0.86 0.70-1.17 1.96 1.01 1.91 1.12 1.55 1.05 18 63~66 24 87 21 126~156 26.85 41.64 41.57 40.29 46.77 30.76 17.65 21.51 22.04 16.14 12.28 27.71 21.60 24.34 28.80 18.11 26.56 23.96 33.89 12.52 7.59 25.45 14.39 17.56 50.00 19.05 41.67 17.24 42.86 21.01 50.00 80.95 58.33 82.76 57.14 78.99 8 40 13 66 9 100 7 1 1 1 1 6 3 9
梨亚科5 个属S-RNase基因各区域的A、T、C、G组成有较大差异,其转换/颠换比值仅有C2-HV 和HV 区小于1,其余位置均大于1,综合保守位点、变异位点、简约信息位点以及单突变位点情况,C2-HV和HV 区数值最接近。5 个保守区保守位点占比由大到小分别为:C2、C3、C1、C5、C4;外显子各部位序列变异位点占比由大到小分别为C2-HV、HV、SP、C4-C5、C3-C4、C5-、C1-C2、HV-C3。
2.1.2 intron 序列分析 梨亚科5 个属90 条SRNase 基因全长CDS 序列共登录HV 区内含子序列83 条,其余7 条未登录内含子信息的基因均为苹果属S-RNase 基因mRNA 序列;140 条片段序列对应121 条内含子序列,包含梨属51 条,苹果属34 条,榅桲属5 条,枇杷属29 条,山楂属2 条,其余为mRNA序列,未登录内含子序列。分析梨亚科5 个属SRNase 基因HV 区内含子序列(图1),结果显示其长度为109~3130 bp,平均长度为505 bp,具有丰富的长度多态性。其中151~200 bp 占比最大,为31.33%;其次为100~150 bp,占比26.51%;其余长度区间占比均较小。内含子均符合GT-AG 法则,其中全长序列对应的内含子序列还符合GTAA-CAG。内含子序列T、C、A、G平均含量分别为T:39.1%,C:13.7%,A:35.2%,G:12.0%,内含子保守位点占比仅为10.31%。

图1 梨亚科5 个属S-RNase 基因HV 区内含子长度
Fig. 1 Distribution of the length of the offspring in the HV region of S-RNase gene in 5 genera of Pomoideae
2.2 梨亚科5个属S-RNase基因序列分化及多态性
由表2 可以看出,梨亚科5 个属S-RNase 基因CDS 全长序列阳性位点数(dN-dS>0 位点)占比小于50%,不同部位区域中,大于全长序列阳性位点占比,由大到小分别为HV、SP、C4-C5;其中连续阳性位点数,C5-、C4-C5、SP 超过HV,HV-C3 与HV 持平。
表2 梨亚科5 个属S-RNase 基因核苷酸非同义突变率与同义突变率
Table 2 Nucleotide non-synonymous mutation rate and synonymous mutation rate of S-RNase gene in 5 genera of Pomoideae
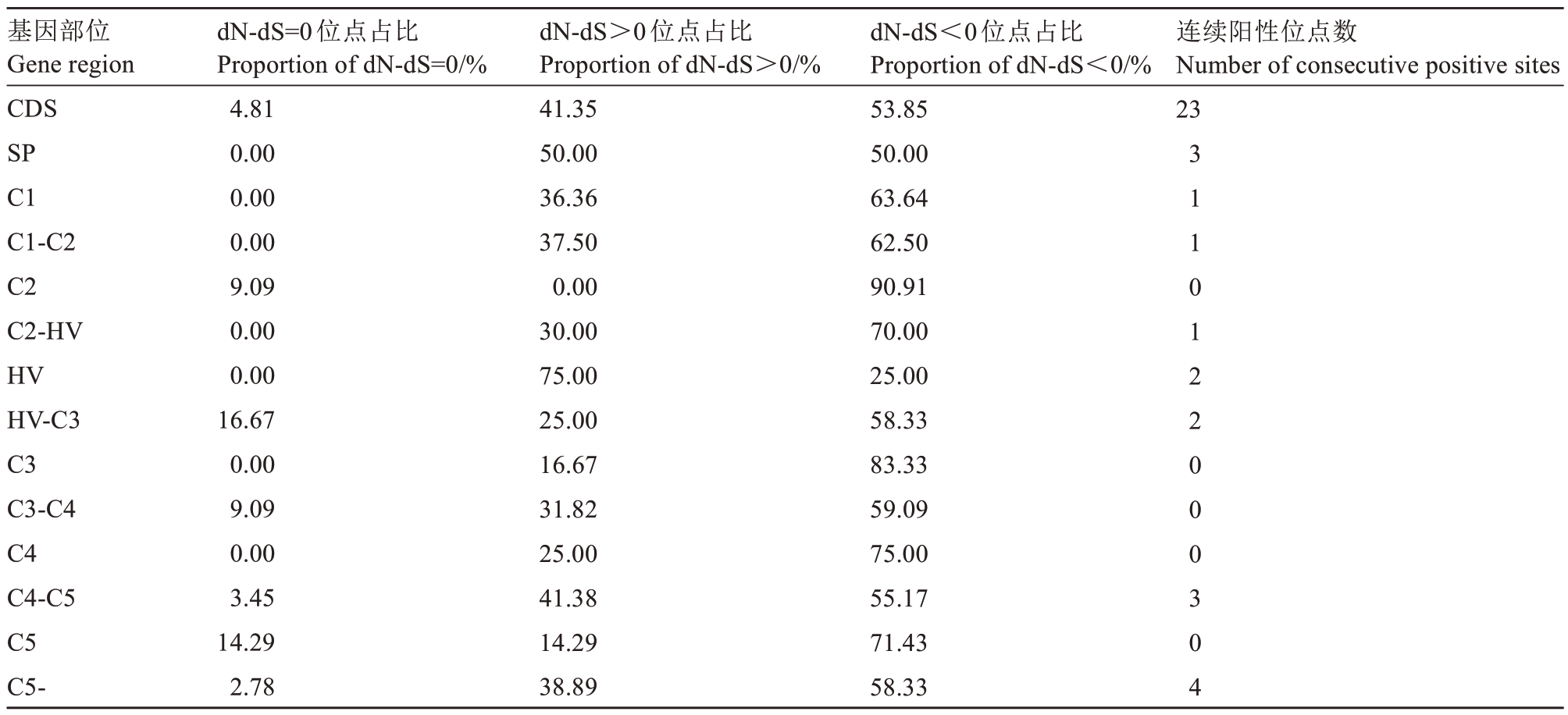
基因部位Gene region CDS SP C1 C1-C2 C2 C2-HV HV HV-C3 C3 C3-C4 C4 C4-C5 C5 C5-dN-dS=0位点占比Proportion of dN-dS=0/%4.81 0.00 0.00 0.00 9.09 0.00 0.00 16.67 0.00 9.09 0.00 3.45 14.29 2.78 dN-dS>0位点占比Proportion of dN-dS>0/%41.35 50.00 36.36 37.50 0.00 30.00 75.00 25.00 16.67 31.82 25.00 41.38 14.29 38.89 dN-dS<0位点占比Proportion of dN-dS<0/%53.85 50.00 63.64 62.50 90.91 70.00 25.00 58.33 83.33 59.09 75.00 55.17 71.43 58.33连续阳性位点数Number of consecutive positive sites 23 3 1 1 0 1 2 2 0 0 0 3 0 4
由表3 反映的遗传多态性的两个指标Θ 和π 数值来看,梨亚科5 个属S-RNase 基因的C2-HV、HV、HV-C3、C3-C4、C4-C5、C5-核苷酸多样性指标π大于整条序列平均值,且其Tajima 检验统计量D>0,证明这些位置受平衡选择的影响。综合考虑Θ、π数值以及S 值来看,C2-HV 和HV 最接近,其次是C4-C5。其中5 个保守区以及intron、SP、C1-C2 区D<0,表明这些区域受定向选择的影响。HV 区π 值最大,其次为C2-HV、C4-C5、C3-C4、C5-、HV-C3,这些区域的π 均大于全长序列平均π 值,5 个保守区的Θ和π数值均偏小,π值越小说明DNA序列差异越小。
表3 梨亚科5 个属 S-RNase 基因遗传多态性
Table 3 Genetic polymorphisms of S-RNase in 5 genera of Pomoideae
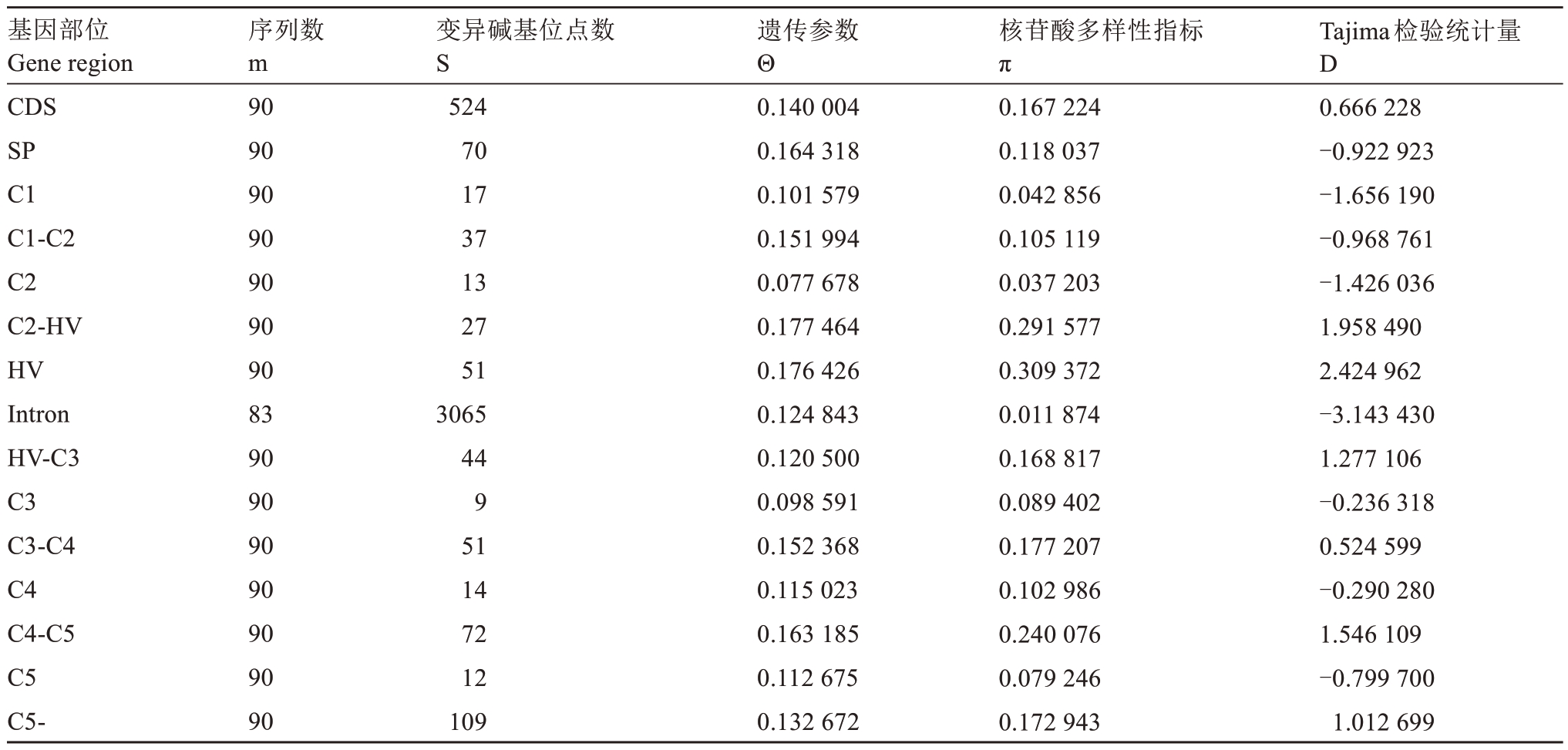
基因部位Gene region CDS SP C1 C1-C2 C2 C2-HV HV Intron HV-C3 C3 C3-C4 C4 C4-C5 C5 C5-序列数m 90 90 90 90 90 90 90 83 90 90 90 90 90 90 90变异碱基位点数S 524 70 17 37 13 27 51 3065 44 9 51 14 72 12 109遗传参数Θ 0.140 004 0.164 318 0.101 579 0.151 994 0.077 678 0.177 464 0.176 426 0.124 843 0.120 500 0.098 591 0.152 368 0.115 023 0.163 185 0.112 675 0.132 672核苷酸多样性指标π 0.167 224 0.118 037 0.042 856 0.105 119 0.037 203 0.291 577 0.309 372 0.011 874 0.168 817 0.089 402 0.177 207 0.102 986 0.240 076 0.079 246 0.172 943 Tajima检验统计量D 0.666 228-0.922 923-1.656 190-0.968 761-1.426 036 1.958 490 2.424 962-3.143 430 1.277 106-0.236 318 0.524 599-0.290 280 1.546 109-0.799 700 1.012 699
2.3 梨亚科5个属不同类群S-RNase基因位点遗传多态性和遗传分化
由表4 可以看出,梨亚科5 个属S-RNase 基因CDS 全长序列、序列片段和内含子序列分化均没有形成明显的以属为单位的界限,苹果属、梨属、枇杷属、榅桲属、山楂属序列均出现属间分化距离小于属内的情况,CDS 全长序列属间分化距离0.198~0.275,属内分化距离为0.165~0.214;对应intron序列属间分化距离0.145~0.323,属内分化距离为0.140~0.374;其中苹果属、梨属S-RNase 基因CDS 全长序列均和山楂属距离最近,而二者intron 序列均与枇杷属距离最近。梨亚科5 个属S-RNase 基因也没有形成明显的以种为单位的界限,山楂、苹果、海棠(M. spectabilis)、花叶海棠(M. transitoria)、砂梨(P.pyrifolia)、白梨(P. bretschneideri)、西洋梨(P. communis)、秋子梨(P. ussuriensis)CDS 全长序列和intron 序列均出现种间遗传分化距离小于种内的情况,其中CDS 全长序列种间距离为0.160~0.275,种内距离为0.165~0.228,intron 种间距离为0.145~0.567,种内距离为0.139~0.359。intron 序列的平均分化距离是对应CDS 全长分化距离的1.5 倍。其中,花叶海棠和新疆梨,海棠和新疆梨,苹果和山楂,砂梨和山楂,白梨和新疆野苹果,秋子梨和海棠,西洋梨CDS 全长序列和山楂距离较近,该结果和常规意义上植物亲缘关系远近不一致,表明梨亚科5 个属S-RNase 基因分化早于属的分化。内含子的遗传分化距离和编码区在某些属或种之间存在一定的相关性,但在另一些属或种之间又呈现相互独立的关系。序列片段的遗传分化距离和全长序列的遗传分化距离不存在严格的相关性,在有CDS 全长序列的情况下,应优先使用全长序列数据作为分析依据。
表4 梨亚科5 个属S-RNase 基因属间及属内、种间及种内的遗传分化
Table 4 Genetic differentiation of S-RNase genes within group average and between group average in genus and species in 5 genera of Pomoideae

注:“/”前数字为全长CDS 序列或对应内含子序列遗传分化距离;“/”后数字为片段序列或对应内含子序列遗传分化距离;n/c. 不适用 。
Note:The number before “/” is the pairwise distance of the full-length CDS sequence or corresponding intron sequence; The number after “/” is the pairwise distance of the fragment sequence sequence or corresponding intron sequence; n/c. Not applicable.
内含子遗传分化距离 Intron pairwise distance 0.228/0.289 0.299/0.351 0.407/0.574 n/c /0.277 n/c /0.290 0.286/0.332 0.268/0.289 0.270/0.256 0.218/0.278 0.287/0.313 0.433/0.523 n/c /0.266 n/c /0.210 0.270/0.284 0.270/0.294 0.257/0.244 0.246/0.350 0.229/0.310 0.357/0.377 0.574/0.692 n/c /0.281 n/c /0.312 0.328/0.363 0.308/0.364 0.275/0.289 0.187/0.185 0.206/0.207 0.206/0.241 0.222/0.261 0.378/0.451 n/c /0.231 n/c /0.218 0.227/0.244 0.240/0.254 0.245/0.257 0.239/0.264 0.139/0.141 n/c /0.222 n/c /0.235 0.359/0.374 n/c /n/c n/c /0.228 n/c /n/c 0.296/0.324 0.283/0.346 n/c /n/c 0.351/0.402 0.210/0.226属1 Genus 1 Crataegus Crataegus Cydonia Crataegus Cydonia Eriobotrya Crataegus Cydonia Eriobotrya Malus Crataegus Cydonia Eriobotrya Malus Pyrus种1 Species 1 C. pinnatifida C. pinnatifida C. oblonga C. pinnatifida C. oblonga E. japonica C. pinnatifida C. oblonga E. japonica M. domestica C. pinnatifida C. oblonga E. japonica M. domestica M. sieversii C. pinnatifida C. oblonga E. japonica M. domestica M. sieversii M. spectabilis C. pinnatifida C. oblonga E. japonica M. domestica M. sieversii M. spectabilis M. transitoria C. pinnatifida C. oblonga属2 Genus 2 Cydonia Eriobotrya Eriobotrya Malus Malus Malus Pyrus Pyrus Pyrus Pyrus Crataegus Cydonia Eriobotrya Malus Pyrus种2 Species 2 C. oblonga E. japonica E. japonica M. domestica M. domestica M. domestica M. sieversii M. sieversii M. sieversii M. sieversii M. spectabilis M. spectabilis M. spectabilis M. spectabilis M. spectabilis M. transitoria M. transitoria M. transitoria M. transitoria M. transitoria M. transitoria P. pyrifolia P. pyrifolia P. pyrifolia P. pyrifolia P. pyrifolia P. pyrifolia P. pyrifolia P. bretschneideri P. bretschneideri编码区遗传分化距离 CDS pairwise distance 0.225/0.289 0.208/0.310 0.275/0.294 0.198/0.306 0.242/0.297 0.210/0.313 0.199/0.309 0.242/0.299 0.216/0.319 0.209/0.305 0.165/0.316 n/c /0.294 n/c /0.302 0.210/0.310 0.214/0.312编码区遗传分化距离 CDS pairwise distance 0.225/0.248 0.208/0.286 0.275/0.294 0.202/0.278 0.238/0.292 0.210/0.310 0.222/0.272 0.270/0.300 0.215/0.304 0.204/0.273 0.160/0.243 0.246/0.293 0.206/0.315 0.204/0.303 0.227/0.302 0.217/0.382 0.273/0.372 0.220/0.359 0.212/0.351 0.230/0.327 0.201/0.334 0.196/0.285 0.230/0.301 0.216/0.321 0.209/0.307 0.208/0.274 0.200/0.309 0.205/0.340 0.214/0.289 0.261/0.301内含子遗传分化距离Intron pairwise distance 0.166/0.194 0.145/0.183 0.214/0.236 0.276/0.253 0.237/0.328 0.215/0.284 0.245/0.237 0.237/0.312 0.226/0.288 0.323/0.344 0.140/0.140 n/c /0.222 n/c /0.235 0.374/0.359 0.298/0.335内含子遗传分化距离 Intron pairwise distance 0.166/0.194 0.145/0.183 0.214/0.236 0.266/0.251 0.231/0.335 0.211/0.288 0.556/0.599 0.382/0.695 0.343/0.499 0.567/0.639 n/c /0.187 n/c /0.226 n/c /0.228 n/c /0.282 n/c /0.524 n/c /0.291 n/c /0.276 n/c /0.268 n/c /0.304 n/c /0.537 n/c /0.248 0.255/0.248 0.230/0.304 0.228/0.281 0.315/0.349 0.448/0.566 n/c /0.271 n/c /0.270 0.230/0.224 0.248/0.317种1 Species 1 E. japonica M. domestica M. sieversii M. spectabilis M. transitoria P. pyrifolia C. pinnatifida C. oblonga E. japonica M. domestica M. sieversii M. spectabilis M. transitoria P. pyrifolia P. bretschneideri C. pinnatifida C. oblonga E. japonica M. domestica M. sieversii M. spectabilis M. transitoria P. pyrifolia P. bretschneideri P. sinkiangensis C. pinnatifida C. oblonga E. japonica M. domestica M. sieversii M. spectabilis M. transitoria P. pyrifolia P. bretschneideri P. sinkiangensis P. communis C. pinnatifida C. oblonga E. japonica M. domestica M. sieversii M. spectabilis M. transitoria P. pyrifolia P. bretschneideri P. sinkiangensis P. communis P. ussuriensis种2 Species 2 P. bretschneideri P. bretschneideri P. bretschneideri P. bretschneideri P. bretschneideri P. bretschneideri P. sinkiangensis P. sinkiangensis P. sinkiangensis P. sinkiangensis P. sinkiangensis P. sinkiangensis P. sinkiangensis P. sinkiangensis P. sinkiangensis P. communis P. communis P. communis P. communis P. communis P. communis P. communis P. communis P. communis P. communis P. ussuriensis P. ussuriensis P. ussuriensis P. ussuriensis P. ussuriensis P. ussuriensis P. ussuriensis P. ussuriensis P. ussuriensis P. ussuriensis P. ussuriensis C. pinnatifida C. oblonga E. japonica M. domestica M. sieversii M. spectabilis M. transitoria P. pyrifolia P. bretschneideri P. sinkiangensis P. communis P. ussuriensis编码区遗传分化距离 CDS pairwise distance 0.214/0.316 0.213/0.300 0.193/0.255 0.212/0.311 0.224/0.361 0.218/0.312 0.220/0.354 0.275/0.344 0.215/0.352 0.204/0.313 0.220/0.330 0.176/0.277 0.177/0.251 0.197/0.311 0.204/0.312 0.192/0.255 0.243/0.290 0.216/0.316 0.209/0.297 0.208/0.274 0.202/0.298 0.220/0.365 0.212/0.309 0.215/0.303 0.209/0.326 0.205/0.304 0.239/0.307 0.216/0.325 0.213/0.314 0.228/0.297 0.199/0.309 0.204/0.336 0.207/0.317 0.223/0.323 0.201/0.306 0.215/0.318 0.165/0.200 n/c /0.294 n/c /0.302 0.213/0.307 n/c /n/c 0.198/0.328 0.228/0.368 0.212/0.323 0.226/0.314 n/c /n/c 0.214/0.301 0.227/0.360
从图2 可以看出,梨亚科5 个属S-RNase 基因遗传分化距离在0.000~0.330 之间,除HV 和C2-HV 各区域遗传分化距离在0.000~0.600 之间,不同位置的区间分布不同;5个保守区中,C2遗传分化距离最为集中,0.000~0.150 占100% ;在此区间C1 占比98.38%,C5 占比77.85%,C4 占比76.03%,C3 占比80.60%,其他区域分布相对比较分散(图2-A)。HV、C2-HV 和intron 区的遗传分化距离较大,HV 区的分化距离在-214 748 364.000~2.400,C2-HV 区的分化距离在-214 748 364.000~1.200(图2-B),intron区的分化距离在-214 748 364.000~2.196(图2-C)。出现负值的情况是由MEGA11 的算法所导致的,说明遗传分化距离较大。相比较而言,HV 区距离分布较为分散,C2-HV和intron分布则较为集中。

图2 梨亚科5 个属S-RNase 基因编码区序列各位置以及内含子所有碱基成对遗传距离分布
Fig. 2 Distribution of pairs of genetic distance of all bases at each position and introns of S-RNase gene coding region in 5 genera of Pomoideae
2.4 梨亚科5个属S-RNase基因密码子使用偏好
某一具体密码子在编码相应氨基酸的同义密码子中的相对概率即为相对同义密码子使用度(RSCU),是密码子使用偏好性的直观反映。当值为1时,密码子无使用偏好;大于1,使用频率较高;小于1,使用频率较低。在梨亚科5 个属S-RNase 基因密码子RSCU 表中(表5),26 个密码子RSCU 值大于1,其中密码子TTG、TCT、TCA、GCC、TAA 的RSCU值大于1.5,是高频密码子,20 个以T 或A 结尾的密码子TTT、TTA、ATT、ATA、GTT、GTA、TCT、TCA、CCT、CCA、TAT、TAA、CAT、AAT、AAA、GAT、GAA、CGA、AGA、GGA为偏好密码子;而结尾为C、G 的密码子RSCU 值多数小于1,显示此类密码子利用率较低,TAA为终止密码子。
表5 梨亚科5 个属S-RNase 基因的RSCU
Table 5 RSCU of S-RNase gene in 5 genera of Pomoideae

密码子Codon TTT(F)TTC(F)TTA(L)TTG(L)CTT(L)CTC(L)CTA(L)CTG(L)ATT(I)ATC(I)相对概率RSCU 1.21 0.79 1.40 2.17 0.50 1.14 0.30 0.50 1.14 0.60密码子Codon TCT(S)TCC(S)TCA(S)TCG(S)CCT(P)CCC(P)CCA(P)CCG(P)ACT(T)ACC(T)相对概率RSCU 1.58 0.68 1.75 0.50 1.29 0.85 1.16 0.71 0.84 0.86密码子Codon TAT(Y)TAC(Y)TAA(*)TAG(*)CAT(H)CAC(H)CAA(Q)CAG(Q)AAT(N)AAC(N)相对概率RSCU 1.25 0.75 2.87 0.13 1.32 0.68 0.94 1.06 1.15 0.85密码子Codon TGT(C)TGC(C)TGA(*)TGG(W)CGT(R)CGC(R)CGA(R)CGG(R)AGT(S)AGC(S)相对概率RSCU 0.56 1.44 0.00 1.00 0.61 0.95 1.18 0.64 0.62 0.86密码子Codon ATG(M)GTT(V)GTC(V)GTA(V)GTG(V)AGG(R)GGT(G)GGG(G)ACA(T)AGA(R)相对概率RSCU 1.00 1.07 0.81 1.48 0.64 1.48 0.83 1.29 0.94 1.14密码子Codon ACG(T)GCT(A)GCC(A)GCA(A)GCG(A)GGC(G)GGA(G)ATA(I)AAA(K)相对概率RSCU 1.35 0.68 1.55 0.80 0.97 0.68 1.20 1.26 1.04
由表6可以看出:梨亚科5个属90条S-RNase基因,其属间密码子T、C、A、G 平均含量分别为25.7%、21.7%、32.6%、20.1%;种间T-1、C-1、A-1、G-1分别为22.4%、19.8%、36.8%、21.0%;种间T-2、C-2、A-2、G-2 分别为27.0%、22.5%、35.3%、15.3%;种间T-3、C-3、A-3、G-3 分别为27.7%、22.6%、25.8%、23.9%。3个位置的碱基含量均符合A+T大于C+G,A、T、C、G 碱基排列在梨亚科5个属S-RNase基因中呈现一定的偏好。这种差异没有以种或属为单位的明显区别。
表6 梨亚科5 个属不同种或属S-RNase 基因碱基分布
Table 6 Base distribution of S-RNase gene in different species or genera in 5 genera of Pomoideae
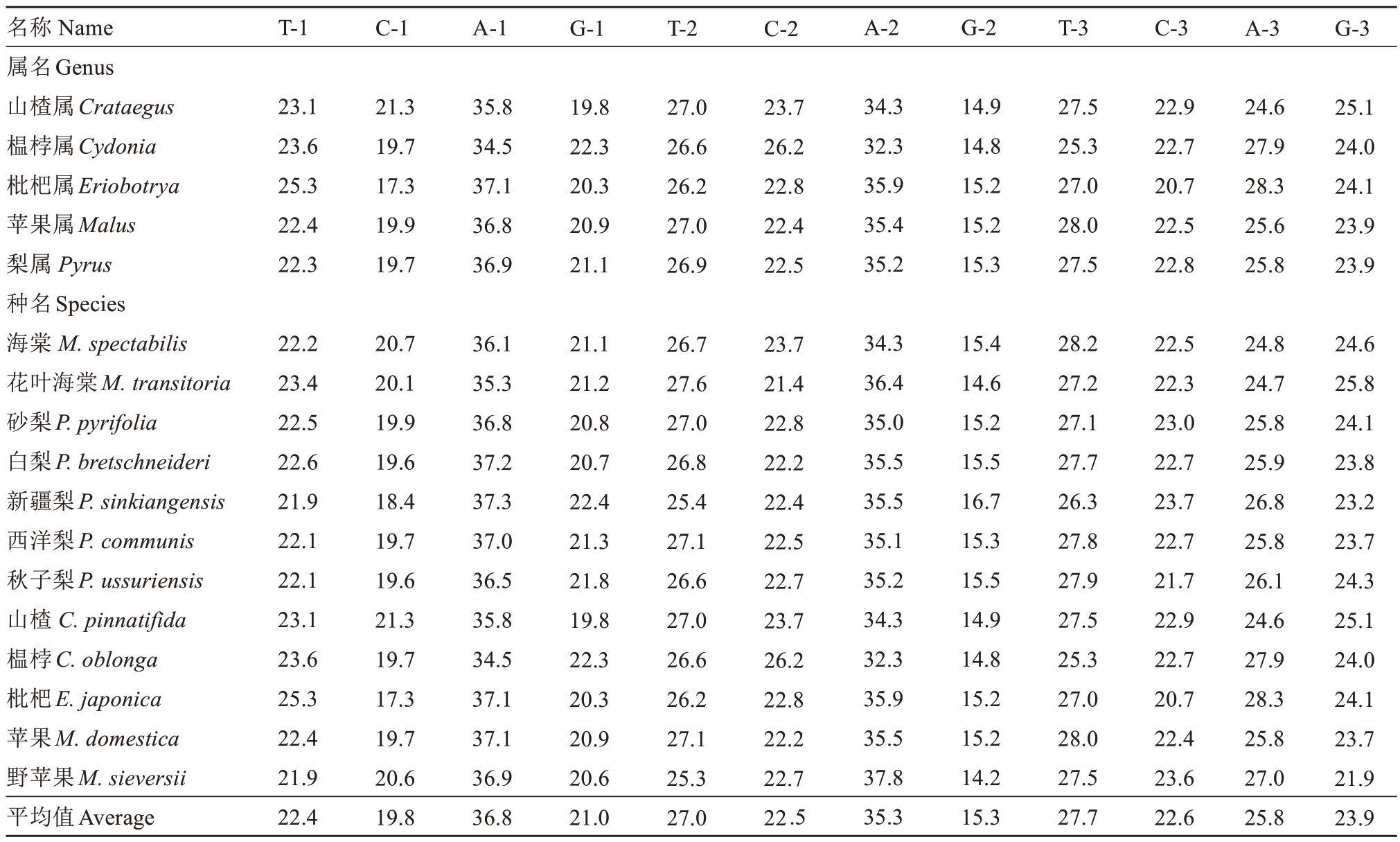
名称 Name属名Genus山楂属Crataegus榅桲属Cydonia枇杷属Eriobotrya苹果属Malus梨属 Pyrus种名Species海棠 M. spectabilis花叶海棠M. transitoria砂梨P. pyrifolia白梨P. bretschneideri新疆梨P. sinkiangensis西洋梨P. communis秋子梨P. ussuriensis山楂 C. pinnatifida榅桲C. oblonga枇杷E. japonica苹果M. domestica野苹果M. sieversii平均值Average T-1 C-1 A-1 G-1 T-2 C-2 A-2 G-2 T-3 C-3 A-3 G-3 23.1 23.6 25.3 22.4 22.3 21.3 19.7 17.3 19.9 19.7 35.8 34.5 37.1 36.8 36.9 19.8 22.3 20.3 20.9 21.1 27.0 26.6 26.2 27.0 26.9 23.7 26.2 22.8 22.4 22.5 34.3 32.3 35.9 35.4 35.2 14.9 14.8 15.2 15.2 15.3 27.5 25.3 27.0 28.0 27.5 22.9 22.7 20.7 22.5 22.8 24.6 27.9 28.3 25.6 25.8 25.1 24.0 24.1 23.9 23.9 22.2 23.4 22.5 22.6 21.9 22.1 22.1 23.1 23.6 25.3 22.4 21.9 22.4 20.7 20.1 19.9 19.6 18.4 19.7 19.6 21.3 19.7 17.3 19.7 20.6 19.8 36.1 35.3 36.8 37.2 37.3 37.0 36.5 35.8 34.5 37.1 37.1 36.9 36.8 21.1 21.2 20.8 20.7 22.4 21.3 21.8 19.8 22.3 20.3 20.9 20.6 21.0 26.7 27.6 27.0 26.8 25.4 27.1 26.6 27.0 26.6 26.2 27.1 25.3 27.0 23.7 21.4 22.8 22.2 22.4 22.5 22.7 23.7 26.2 22.8 22.2 22.7 22.5 34.3 36.4 35.0 35.5 35.5 35.1 35.2 34.3 32.3 35.9 35.5 37.8 35.3 15.4 14.6 15.2 15.5 16.7 15.3 15.5 14.9 14.8 15.2 15.2 14.2 15.3 28.2 27.2 27.1 27.7 26.3 27.8 27.9 27.5 25.3 27.0 28.0 27.5 27.7 22.5 22.3 23.0 22.7 23.7 22.7 21.7 22.9 22.7 20.7 22.4 23.6 22.6 24.8 24.7 25.8 25.9 26.8 25.8 26.1 24.6 27.9 28.3 25.8 27.0 25.8 24.6 25.8 24.1 23.8 23.2 23.7 24.3 25.1 24.0 24.1 23.7 21.9 23.9
由表7 可以看出梨亚科5 个属S-RNase 基因CG含量C1 区最低,为30.3%,C3 最高,为55.5%,其中HV 区为42.3%。从具体分布看,SP、C2、HV、C5-均符合CG1<CG2<CG3。HV 区CG 分布较为一致,在40.1%~45.4%,与C5-、C2-HV、C4-C5数值接近。
表7 梨亚科5 个属S-RNase 基因不同位置CG 分布
Table 7 CG distribution at different locations of S-RNase gene in 5 genera of Pomoideae
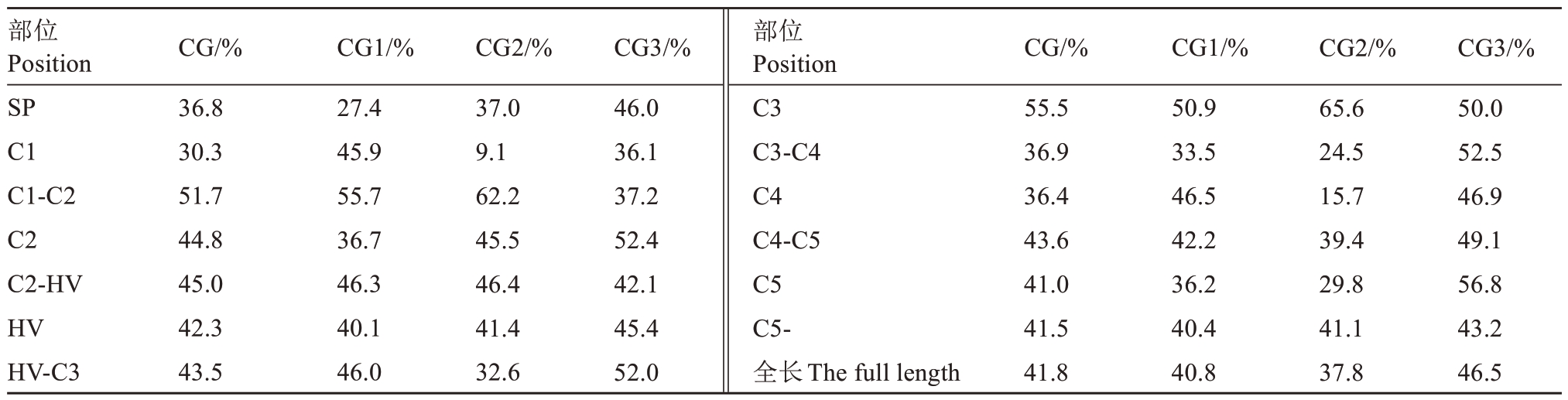
部位Position SP C1 C1-C2 C2 C2-HV HV HV-C3 CG/%36.8 30.3 51.7 44.8 45.0 42.3 43.5 CG1/%27.4 45.9 55.7 36.7 46.3 40.1 46.0 CG2/%37.0 9.1 62.2 45.5 46.4 41.4 32.6 CG3/%46.0 36.1 37.2 52.4 42.1 45.4 52.0部位Position C3 C3-C4 C4 C4-C5 C5 C5-全长The full length CG/%55.5 36.9 36.4 43.6 41.0 41.5 41.8 CG1/%50.9 33.5 46.5 42.2 36.2 40.4 40.8 CG2/%65.6 24.5 15.7 39.4 29.8 41.1 37.8 CG3/%50.0 52.5 46.9 49.1 56.8 43.2 46.5
2.5 梨亚科5个属S-RNase进化分析
由梨亚科5 个属S-RNase 基因CDS 全长序列和其密码子使用偏好分别构建的进化树显示(图3):砂梨、白梨、秋子梨、西洋梨、山楂、苹果、海棠、花叶海棠等同属或同种的S-RNase 基因并没有出现明显的聚类。两个进化树中,山楂的两条S-RNase 基因均先与苹果属、梨属的S-RNase 聚成一类,苹果属S-RNase 出现多次和梨属聚成一类的现象,以种为单位的S-RNase 基因聚类也是如此。不同属或同属不同种间的S-RNase 基因相似性高于同属或同种间基因相似性(图3-A)。该结果表明梨亚科5 个属SRNase的分化要早于各属和各种的分化。进化树分支结构存在一些相同的地方,密码子使用偏好是物种进化过程中基因经历选择和突变呈现的结果,根据其构建的进化树体现了基因特定的进化历程,是基因编码序列进化树良好的补充(图3-B)。由梨亚科5 个属S-RNase 基因HV 区内含子序列构成的进化树(图3-C)和其CDS 全长序列进化树更接近,二者出现多处的分枝结构基本一致的现象,说明这些基因的CDS序列与HV区内含子序列进化具有相关性。

图3 梨亚科S-RNase 基因进化树
Fig. 3 Evolution tree of S-RNase genes in Pomoideae
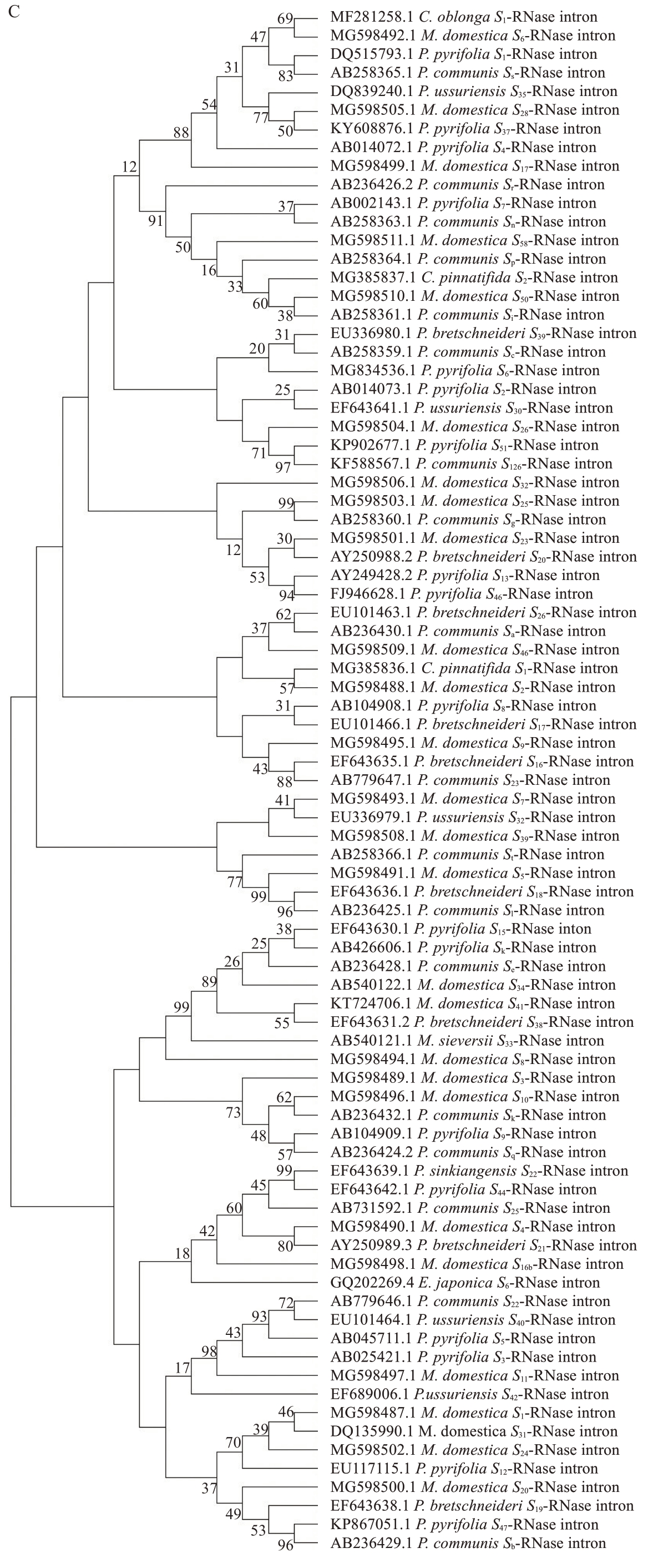
图3 (续) Fig. 3 (Continued)
3 讨 论
近年来,随着分子生物学的发展,人们对梨亚科梨属[1]、苹果属[2]、枇杷属[3]、山楂属[4]和榅桲属[5]等果树自交不亲和系统的分子基础进行了广泛研究。作为其雌蕊S位点决定因子的S-RNase基因,其序列特征和进化机制一直是研究热点之一[12-13]。根据研究梨亚科5 个属S-RNase 基因结构由5 个保守区和1个包含长度高度多态性的内含子在内的HV 区组成[1-3],HV 区域长期以来被认为是雌蕊S-RNase基因识别雄蕊花粉S基因的关键位置[11-12]。梨亚科5个属S-RNase基因HV 区域阳性位点占比、连续阳性位点数以及Tajima检验统计量D也支持此观点。近年来在肯定此观点的同时,也逐渐有新的观点,认为其他区域在花粉S 基因识别中也可能起到重要作用。如西洋梨Sn 和Si 在HV 区域有相同的氨基酸序列,但Sn 花粉管的生长不受Si-RNase 的抑制[12],表明HV区域可能不足以单独与花粉S蛋白进行选择性相互作用,还可能存在其他区域在相互识别过程中发挥重要作用。一般认为这些区域显示出非同义氨基酸取代多于同义取代,同时具有阳性选择位点。在对梨亚科5 个属S-RNase 基因各区域阳性位点和Tajima 检验值分析时也发现了一些区域符合这一特征。还有学者研究发现西洋梨等位基因S6a与S6b的HV 区完全一致,但二者其他区域存在14 处氨基酸差异,认为二者应该定义为不同的S 基因[11]。在苹果中也发现了一对功能不同的S-RNase 等位基因共享相同的HV区域,表明存在HV区域外的位置参与识别雄蕊花粉S 基因的可能性[14]。梨亚科5 个属SRNase 基因的功能区域,特别是与花粉识别和反应相关的区域,可能受到正选择的作用,以维持其在自交不亲和反应中的有效性[14]。同时,基因的其他区域可能更加保守,以保持基因的基本结构和功能。基于最新的梨亚科5 个属S-RNase 基因CDS 全长序列中5 个保守区的分析也支持这一观点,其中C2 区高度保守,90 条序列中仅出现梨S32- RNase(EU336979.1)一处V 变为I 的现象。研究还发现梨亚科5 个属S-RNase 基因C2-HV、C4-C5、C5-无论是阳性位点、反映遗传多态性的两个指标Θ 和π、Tajima检验统计量D值、CG含量及分布情况均与HV区上述指标接近。以已有的X-射线衍射获得的梨S3-RNase 三维晶体结构作为模型[15],通过SWISS-Model 和AlphaFold Protein Structure Database 构建梨亚科5 个属S-RNase 三维结构,分析HV 区域位于分子的表面,C4-C5和C5-分别在三维结构中靠近活性位点裂缝的两侧,均是最容易和花粉S 基因发生相互作用的有利位置。另外研究发现梨亚科5 个属SRNase 基因信号肽区域有较高的阳性位点和连续阳性位点,这令人疑惑。信号肽主要作用是引导SRNase 向细胞质基质分泌,并在受粉过程中进入花粉细胞,一般来说,信号肽在蛋白质成熟以前就已经被信号肽酶切除掉,最新的研究显示信号肽会在蛋白的空间排列上起直接的作用[16-17],是否梨亚科5 个属S-RNase 的信号肽也有此作用需要进一步的研究来验证。
5 个属均有一些自交亲和的品种,自交亲和的原因有多种[18-20]。枇杷S6-RNase 基因C5-区与现有梨亚科S-RNase基因该区域存在1个24 bp的核苷酸序列插入,根据现有研究,含有该基因的枇杷品种均是自交亲和品种,且有证据表明是由该插入导致了自交亲和[21]。闫庄梨中的S21-RNase 基因C2 区1 个保守的G 被V 所取代,改变活性位点附近的空间结构,使得S-RNase 的核酸酶活性降低,从而导致亲和[22]。苹果品种Vered 的S39sm-RNase 基因与正常S39-RNase基因相比,编码序列中有两个非同义替换和3个同义替换,以及一个205 bp碱基对的缺失,其仅编码121 个氨基酸残基,导致S-RNase 异构体的截短,从而导致亲和[23]。
梨亚科S-RNase 基因HV 区内含子序列长期被认为不参与自交不亲和的过程[6-7,24]。分析发现梨亚科5 个属S-RNase 基因内含子序列成对总平均遗传距离0.312,仅为编码区序列成对总平均遗传距离0.210 的1.5 倍,其分属和分种遗传分化距离与对应CDS 序列遗传分化距离也呈现一定的相关性,因此,HV 区的内含子序列在进化过程中存在受到编码区特别是HV 区影响的可能性。同时,通过分析内含子序列发现一些S-RNase 基因的登录信息有误:P. communis Si-RNase(AB258361.1)登录的内含子5'端GTGAATTCTCACAGG 序列应为外显子序列,内含子序列应为该序列后面的GTAA 开始;P. communis S25-RNase(AB731592.1)内含子序列长3030 bp,而不是3031 bp。另有P. pyrifolia S46-RNase(FJ946628.1)基因序列提交信息的外显子序列比真实序列长36 bp,该36 bp 应为内含子序列。研究还发现,5个属中外显子序列相同的两条S-RNase基因内含子序列存在内含子不一致的情况:P.s S28-RNase(EF566872.1)和P.p S8-RNase(AB104908.1),前者比后者存在7 bp核苷酸的缺失,另有4处核苷酸突变;P. b S20-RNase(AY250988.2)和P.b S29-RNase(EU101462.1),前者比后者存在一个20 bp核苷酸插入,另有6处核苷酸突变;P.c S210-RNase(EU477840.1)和P.c S21-RNase(EU477839.1),前者比后者存在1 个长度为561 bp序列的插入、另外还有1个碱基突变[25]。这些S-RNase 外显子序列一致而内含子序列存在差异,是否为同一基因进化过程中不同时期的形态还有待进一步研究。
蔷薇科基于S-RNase 的配子体自交不亲和性,被认为是开花植物中最广泛和最古老的自交不亲和系统之一[26]。关于S-RNase 基因进化目前主流的观点是:单一位点起源,多次分化[27-28]。笔者在本研究中表明梨亚科5 个属S-RNase 基因CDS 全长序列和基因密码子偏好性进化树均显示未形成以种或属为单位的聚类,说明梨亚科S-RNase 基因分化至少早于5个属的分化时间。
基因结构特征以及进化与基因功能密不可分。自从证实了S-RNase 就是梨亚科果树自交不亲和的雌性决定因子以来,学者们普遍认为其主要通过降解相应的花粉RNA来完成自交不亲和[29-31]。长时间以来学者们也在不断探寻其分子机制。Matsuura等[15]报告了日本梨S3-RNase 在1.5 Å 分辨率下的晶体结构,并给出了S3-RNase 存在的识别位点及其酶活性的结构基础。Meng 等[29]研究认为苹果花粉ABCF转运蛋白可以将S-RNase运输至花粉管;Yuan等[30]研究认为进入花粉管中的苹果S-RNase 会被不同基因型的花粉SCF(SKP1-Cullin1-F-box)复合体和26S 蛋白酶降解导致亲和,同基因型的花粉因不能降解S-RNase 而导致自交不亲和。Chen 等[31]研究认为梨S-RNase直接作用于PbrAct1 蛋白,诱导其花粉管细胞凋亡,无法完成受精。Tang 等[32]研究发现梨S-RNase 可以介导无机焦磷酸酶通过抑制果胶甲基化酶诱导花粉管顶端膨胀。由于梨亚科雄性自交不亲和因子还未最终确定,其S-RNase 基因的功能以及如何与雄性S 基因互作来完成自交不亲和还有待进一步研究。
4 结 论
目前,NCBI 中GenBank 共收录梨亚科5 个属SRNase 基因CDS 全长序列,剔除重复序列90 条。大于330 bp 的片段序列140 条。研究发现除了公认的HV 区具备和花粉S基因识别的特征外,其他区域也表现出了相似的特征;基因编码序列、内含子序列和密码子偏好生成的进化树均未形成以属或种为单位的聚类,表明梨亚科5个属S-RNase基因的分化早于各属之间的分化。研究显示梨亚科5 个属S-RNase基因中的密码子存在一定的偏好性,并且偏好以A/T结尾的密码子。
[1] 吕文娟,冯建荣,刘小芳,刘海楠,李文慧,钟颖. ‘库尔勒香梨’自交不亲和S-RNase 等位基因全长的克隆与分析[J]. 分子植物育种,2017,15(5):1639-1647.LÜ Wenjuan,FENG Jianrong,LIU Xiaofang,LIU Hainan,LI Wenhui,ZHONG Ying. Cloning and analysis of self-incompatibility S-RNase allelic genes in Korla fragrant pear[J]. Molecular Plant Breeding,2017,15(5):1639-1647.
[2] SHEICK R,SERRA S,TILLMAN J,LUBY J,EVANS K,MUSACCHI S. Characterization of a novel S-RNase allele and genotyping of new apple cultivars[J]. Scientia Horticulturae,2020,273:109630.
[3] CARRERA L,SANZOL J,HERRERO M,HORMAZA J I. Genomic characterization of self-incompatibility ribonucleases (SRNases) in loquat (Eriobotrya japonica Lindl.) (Rosaceae,Pyrinae)[J]. Molecular Breeding,2009,23(4):539-551.
[4] MA J Z,WANG H J,WANG D S,LI X Y,LIU J Z,XIAO X,ZHANG L B,WU J K. Self-compatibility of Crataegus pinnatifida Bge. ‘Zizhenzhu’ is associated with the mutation of a pistilpart non-S factor[J]. Scientia Horticulturae,2020,268:109362.
[5] SADEGHNEJAD S,ABDOLLAHI H,DAVOODI D,TATARI M,KHOSROSHAHLI M. Identification of self-incompatibility alleles in quince (Cydonia oblonga Mill.)[J]. PLoS One,2024,19(2):e0297595.
[6] EREZ K,JANGID A,FELDHEIM O N,FRIEDLANDER T.The role of promiscuous molecular recognition in the evolution of RNase-based self-incompatibility in plants[J]. Nature Communications,2024,15(1):4864.
[7] SASSA H. Molecular mechanism of the S-RNase-based gametophytic self-incompatibility in fruit trees of Rosaceae[J]. Breeding Science,2016,66(1):116-121.
[8] 梁文杰,乌云塔娜,谭晓风. 梨花粉自交不亲和基因克隆及其评价分析[J]. 果树学报,2022,39(6):905-919.LIANG Wenjie,WU Yuntana,TAN Xiaofeng. Cloning and evaluation of S locus F-box brother genes in genus Pyrus plants[J].Journal of Fruit Science,2022,39(6):905-919.
[9] HE M,LI L F,XU Y,MU J X,XIE Z H,GU C,ZHANG S L.Identification of S-genotypes and a novel S-RNase in 84 native Chinese pear accessions[J]. Horticultural Plant Journal,2022,8(6):713-726.
[10] MA R C,OLIVEIRA M M. Evolutionary analysis of S-RNase genes from Rosaceae species[J]. Molecular Genetics and Genomics,2002,267(1):71-78.
[11] DE FRANCESCHI P,BIANCO L,CESTARO A,DONDINI L,VELASCO R. Characterization of 25 full-length S-RNase alleles,including flanking regions,from a pool of resequenced apple cultivars[J]. Plant Molecular Biology,2018,97(3):279-296.
[12] ZISOVICH A H,STERN R A,SAPIR G,SHAFIR S,GOLDWAY M. The RHV region of S-RNase in the European pear (Pyrus communis) is not required for the determination of specific pollen rejection[J]. Sexual Plant Reproduction,2004,17(3):151-156.
[13] 何敏,谷超,吴巨友,张绍铃. 果树自交不亲和机制研究进展[J].园艺学报,2021,48(4):759-777.HE Min,GU Chao,WU Juyou,ZHANG Shaoling. Recent advances on self-incompatibility mechanism in fruit trees[J]. Acta Horticulturae Sinica,2021,48(4):759-777.
[14] MATSUMOTO S,YAMADA K,SHIRATAKE K,OKADA K,ABE K. Structural and functional analyses of two new S-RNase alleles,Ssi5 and Sad5,in apple[J]. The Journal of Horticultural Science and Biotechnology,2010,85(2):131-136.
[15] MATSUURA T,SAKAI H,UNNO M,IDA K,SATO M,SAKIYAMA F,NORIOKA S. Crystal structure at 1.5-a resolution of Pyrus pyrifolia pistil ribonuclease responsible for gametophytic self-incompatibility[J]. Journal of Biological Chemistry,2001,276(48):45261-45269.
[16] HE X Y,LI Y J,KALYANARAMAN C,QIU L L,CHEN C,XIAO Q,LIU W X,ZHANG W,YANG J J,CHEN G Q,JACOBSON M P,SHI Y S. GluA1 signal peptide determines the spatial assembly of heteromeric AMPA receptors[J]. Proceedings of the National Academy of Sciences of the United States of America,2016,113(38):E5645-E5654.
[17] DUAN G F,YE Y X,XU S,TAO W C,ZHAO S P,JIN T C,NICOLL R A,SHI Y S,SHENG N Y. Signal peptide represses GluK1 surface and synaptic trafficking through binding to amino-terminal domain[J]. Nature Communications,2018,9(1):4879.
[18] ABDALLAH D,BEN MUSTAPHA S,BALTI I,SALHI-HANNACHI A,BARAKET G. Self-(in) compatibility in Tunisian apple accessions [Malus domestica. Borkh]:S-genotypes identification and pollen tube growth analysis[J]. Planta,2024,259(6):137.
[19] CLAESSEN H,PALMERS H,KEULEMANS W,VAN DE POEL B,DE STORME N. The influence of the pollination compatibility type on the pistil S-RNase expression in European pear(Pyrus communis)[J]. Frontiers in Genetics,2024,15:1360332.
[20] WU C,ZHANG Y,WANG H K,YAN M Y,WANG S H. Selfcompatibility of ‘Zaohuang’ loquat is attributed to the lack of a 52 bp fragment in the S10-RNase promoter[J]. Scientia Horticulturae,2022,301:111093.
[21] NYSKA R,RAZ A,BARAS Z,SHAFIR S,GOLDWAY M,SCHNEIDER D. Self-compatibility in loquat (Eriobotrya japonica Lindl.) is possibly due to S6-RNase mutation[J]. Scientia Horticulturae,2013,161:43-48.
[22] LI Y,WU J K,WU C B,YU J,LIU C S,FAN W Q,LI T Z,LI W. A mutation near the active site of S-RNase causes self-compatibility in S-RNase-based self-incompatible plants[J]. Plant Molecular Biology,2020,103(1/2):129-139.
[23] OKADA K,SHIMIZU T,MORIYA S,WADA M,ABE K,SAWAMURA Y. Alternative splicing and deletion in S-RNase confer stylar- part self- compatibility in the apple cultivar‘Vered’[J]. Plant Molecular Biology,2024,114(6):113.
[24] LI W,YANG Q,GU Z Y,WU C B,MENG D,YU J,CHEN Q J,LI Y,YUAN H,WANG D M,LI T Z. Molecular and genetic characterization of a self-compatible apple cultivar,‘CAU-1’[J].Plant Science,2016,252:162-175.
[25] SANZOL J. Pistil-function breakdown in a new S-allele of European pear,S21*,confers self-compatibility[J]. Plant Cell Reports,2009,28(3):457-467.
[26] ZHAO H,ZHANG Y,ZHANG H,SONG Y Z,ZHAO F,ZHANG Y E,ZHU S H,ZHANG H K,ZHOU Z D,GUO H,LI M M,LI J H,GAO Q,HAN Q Q,HUANG H Q,COPSEY L,LI Q,CHEN H,COEN E,ZHANG Y J,XUE Y B. Origin,loss,and regain of self-incompatibility in angiosperms[J]. The Plant Cell,2022,34(1):579-596.
[27] 赵洪,薛勇彪. 显花植物自交不亲和性的分子与演化机制[J].遗传,2024,46(1):3-17.ZHAO Hong,XUE Yongbiao. Molecular and evolutionary mechanisms of self-incompatibility in angiosperms[J]. Hereditas (Beijing),2024,46(1):3-17.
[28] LV S Z,QIAO X,ZHANG W,LI Q H,WANG P,ZHANG S L,WU J Y. The origin and evolution of RNase T2 family and gametophytic self-incompatibility system in plants[J]. Genome Biology and Evolution,2022,14(7):evac093.
[29] MENG D,GU Z Y,LI W,WANG A D,YUAN H,YANG Q,LI T Z. Apple MdABCF assists in the transportation of S-RNase into pollen tubes[J]. The Plant Journal,2014,78(6):990-1002.
[30] YUAN H,MENG D,GU Z Y,LI W,WANG A D,YANG Q,ZHU Y D,LI T Z. A novel gene,MdSSK1,as a component of the SCF complex rather than MdSBP1 can mediate the ubiquitination of S-RNase in apple[J]. Journal of Experimental Botany,2014,65(12):3121-3131.
[31] CHEN J Q,WANG P,DE GRAAF B H J,ZHANG H,JIAO H J,TANG C,ZHANG S L,WU J Y. Phosphatidic acid counteracts S-RNase signaling in pollen by stabilizing the actin cytoskeleton[J]. The Plant Cell,2018,30(5):1023-1039.
[32] TANG C,WANG P,ZHU X X,QI K J,XIE Z H,ZHANG H,LI X Q,GAO H R,GU T T,GU C,LI S,DE GRAAF B H J,ZHANG S L,WU J Y. Acetylation of inorganic pyrophosphatase by S-RNase signaling induces pollen tube tip swelling by repressing pectin methylesterase[J]. The Plant Cell,2023,35(9):3544-3565.