西瓜(Citrullus lanatus)果实汁多味甜,营养丰富,是盛夏季节消暑、解渴的佳品。中国是世界第一大西瓜生产国和消费国,西瓜的栽培面积和产量均居世界首位[1]。西瓜幼苗根系柔弱,对盐胁迫敏感,土壤盐含量(w,后同)达0.3%时即会显著抑制幼苗生长,造成西瓜产量和品质严重下降[2]。在西瓜生产中,为了获得较高产量常进行土壤漫灌、盲目地过量施用化肥和多年连作,导致土壤次生盐渍化逐年加重;另外,近年来中国设施农业迅速发展,全国设施农业面积达266.67万hm2[3],随着西瓜主栽区反季节保护地的栽培面积不断扩大,土壤因长期得不到雨水淋洗致使盐分聚集,引起土壤次生盐渍化,进而严重影响西瓜的生长和发育。培育耐盐西瓜新品种是解决这一问题行之有效的方法。探究西瓜应答盐胁迫的机制、发掘关键耐盐基因是培育耐盐西瓜新品种的重要基础,对西瓜产业的安全和可持续发展具有重要意义。目前,西瓜耐盐胁迫研究多集中在外源物质的利用、耐盐品种的筛选以及砧木的应用等方面[4-7]。在耐盐基因的挖掘方面,主要开展了转录组、代谢组、耐盐相关基因表达模式等工作[8-10]。同时,也有研究发现,多倍体西瓜的耐盐能力强于同源二倍体西瓜[8],但具体机制不明确。因此,目前调控西瓜耐盐的分子机制仍不清晰,急需继续挖掘调控西瓜耐盐性的关键基因。
近年来,通过高质量的西瓜基因组组装结合大规模基因组重测序阐明了西瓜果实品质和抗性的选择驯化过程[11-13]。耐盐是由多个基因控制的涉及多种分子和生物学过程的复杂数量性状[14]。在研究西瓜复杂表型性状时,越来越多的研究人员选择利用高通量测序数据开展与西瓜性状相关的GWAS 分析,这极大方便了西瓜育种工作。Dou等[15]利用315份西瓜材料的测序数据,关联到与果实形状相关的主效位点,并通过F2 群体精细定位确定ClFS1(Cla011257)为控制果实形状的候选基因。王学征等[16]利用62 份西瓜种质资源对种子大小性状进行了GWAS 分析,检测到7 个与种子长度相关的SNP位点。高美玲等[17]利用144 份西瓜材料关联到3 个与种子百粒质量相关的QTL 位点。Gong 等[18]利用197 份西瓜种质关联到4 个与种子大小显著相关的SNP 位点,并筛选到2 个与种子大小相关的候选基因Cla97C05G104360 和Cla97C05G104380。 Guo等[11]通过414 份西瓜种质筛选到与果实糖含量、果肉颜色、果实形状、条纹形状和种皮颜色相关的SNP位点。Ren 等[19]利用135 份西瓜资源关联到与棉子糖显著相关的SNP 位点,筛选到关键的碱性α-半乳糖苷酶基因ClAGA2。
然而,利用GWAS 鉴定西瓜耐盐基因的研究还未见报道。因此,在西瓜中利用GWAS 方法筛选与耐盐性状相关的SNP位点,进而挖掘耐盐相关的关键基因具有重大潜力。笔者利用本团队前期发表的西瓜核心种质材料的重测序结果和鉴定得到的SNP变异位点,结合121 份核心种质的耐盐性相关生理生化指标进行GWAS分析,挖掘与根表面积、根K+、根Na+和根可溶性糖含量显著相关的SNP 位点,并在区间内筛选与耐盐相关的候选基因,以期为解析西瓜耐盐性的分子机制、开发分子标记以及选育耐盐西瓜新品种奠定基础。
1 材料和方法
1.1 材料
本试验用于GWAS分析的121份西瓜核心种质材料包括15份黏籽西瓜(C.mucosospermus),3份饲用西瓜(C.amarus),1 份缺须西瓜(C.ecirrhosus),4份药西瓜(C.colocynthis),10份籽瓜(C.megalospermus)和88份栽培西瓜(C.lanatus),具体信息参考高博文等[7]的报道。用于GWAS分析的SNP变异数据源于Guo等[11]已发表的文章。用于候选基因表达量分析的耐盐材料中石红和盐敏感材料PI186489(图1)以及盐处理方法参考Zhu 等[20]和高博文[21]的报道。以上西瓜种质材料均来自中国农业科学院郑州果树研究所国家西甜瓜中期库。

图1 150 mmol·L-1 NaCl 处理前后耐盐和盐敏感西瓜材料的表型
Fig.1 Phenotypes of salt-tolerant and salt-sensitive materials before and after 150 mmol·L-1 NaCl treatment
1.2 表型数据的测定
2021年6月对表型数据进行测定,其中,地上部鲜质量、地上部干质量、根长、根表面积、叶绿素含量的测定方法参考高博文等[7]的报道;根脯氨酸含量测定方法参考高博文[21]的报道;根钾离子(K+)含量和钠离子(Na+)含量的测定方法参考Zhu等[20]的报道;根可溶性糖含量采用南京建成生物工程研究所试剂盒(货号:A145-1-1)测定,使用分光光度计读数。
1.3 全基因组关联分析
采用FaST- LMM(Factored Spectrally Transformed Linear Mixed Models)方法[22]对表型数据进行GWAS分析,定位与表型数据相关的SNP变异位点,并由曼哈顿(Manhattan)图显示关联位点。横坐标代表染色体位置,纵坐标代表p 值取以10 为底的负对数[-log10(p)],图上散点(或线条)代表每个SNP 位点对应的-log10(p)。蓝色水平线代表0.01·标记量-1对应的值,红色水平线代表0.1·标记量-1对应的值。超过阈值线以上的散点(或线条)即为候选位点,并选择显著SNP的上下游100 kb区间作为候选区间[11]。
1.4 候选基因功能注释和表达量分析
使用C. lanatus subsp. vulgaris cv. 97103 V2 参考基因组(http://cucurbitgenomics.org/organism/21)进行基因表达量分析和基因功能注释。FPKM(fragments per kilobase million,每千碱基对每百万对应基因的读取数)用于计算基因表达水平。基于KEGG(http://www.genome.jp/kegg/)数据库和GO(http://www.geneontology.org/)数据库进行基因注释和功能分析。DEGs(differentially expressed genes,差异表达基因):|差异倍数|≥2.00,FDR(false discovery rate,错误发现率)≤0.001。
用于基因表达量分析的数据来源于耐盐材料中石红和盐敏感材料PI186489的转录组数据(NCBI数据库登录号PRJNA844416)[20]。采用TBtools[23]作图。
2 结果与分析
2.1 121份西瓜核心种质资源表型性状差异分析
9个数量性状的变异分析结果如表1所示,其中根可溶性糖含量的变异系数最大,为91.41%,变异范围为0.03~7.06 mg·g-1,平均值为1.08 mg·g-1,说明这个性状的遗传多样性是最丰富的;叶绿素含量(SPAD 值)的变异系数最小,为9.05%,变异范围为31.35~59.87,平均值为44.51,表明其遗传变异程度相对较低。
表1 西瓜9 个耐盐性状的描述统计
Table 1 Descriptive statistics of nine salt tolerance traits in watermelon
性状Trait地上部鲜质量Shoot fresh mass/g地上部干质量Shoot dry mass/g根长Root length/mm根表面积Root surface area/mm2叶绿素含量(SPAD值)Chlorophyll content(SPAD value)w(根脯氨酸)Root proline content/(μg·g-1)w(根钾离子)Root K+content/(mg·g-1)w(根钠离子)Root Na+content/(mg·g-1)w(根可溶性糖)Root soluble sugar content/(mg·g-1)平均值Mean 9.02 0.77 397.40 106.03 44.51 191.98 68.53 266.88 1.08最大值Maximum 36.18 3.30 1 520.77 538.83 59.87 628.89 108.54 1 090.27 7.06最小值Minimum 1.60 0.09 162.09 20.08 31.35 5.55 2.97 23.39 0.03标准差Standard deviation 6.98 0.60 227.57 91.51 4.03 143.57 24.98 206.83 0.99变异系数Coefficient of variation/%77.41 77.26 57.26 86.31 9.05 74.78 36.45 77.50 91.41
2.2 西瓜耐盐性状的GWAS分析
为了进一步确定121份西瓜材料中与耐盐性状相关的SNP 位点,笔者采用Fast-LMM 算法开展关联分析。结果表明,与地上部鲜质量、地上部干质量、根长、叶绿素含量和根脯氨酸含量均没有显著相关的SNP位点,而与根表面积及根K+、根Na+和根可溶性糖含量均有显著相关的SNP位点。
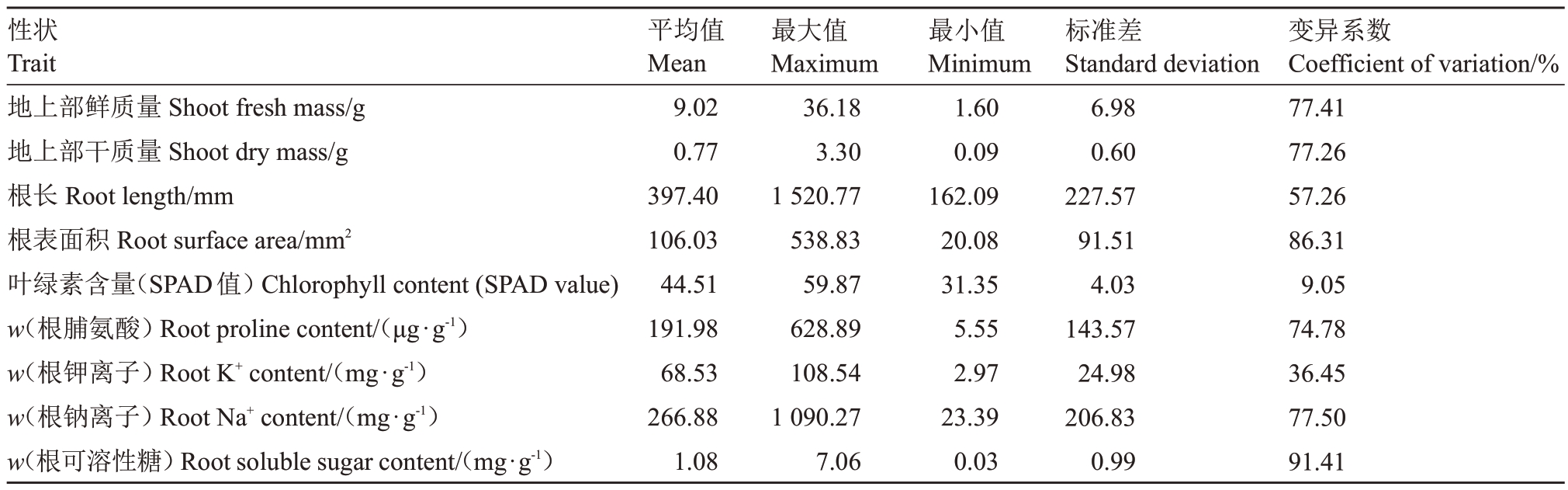
对于根表面积,鉴定出1 个显著的SNP 位点(SNP31480842),位于2 号染色体上(图2-A),在该SNP 位点附近(前后各100 kb 范围内)获得23 个基因(表2)。利用盐处理前后的耐盐材料和盐敏感材料对这23个基因的表达水平进行分析,发现只有20个基因具有表达量,并且Cla97C02G043360、Cla97C02G043200、Cla97C02G043190、Cla97C02G-043250、Cla97C02G043350、Cla97C02G043290 和Cla97C02G043320 在两份材料中均受盐胁迫的诱导上调表达(图2-B),表明这些基因可能响应盐胁迫或与根表面积的大小相关。
表2 根表面积指标下GWAS 分析区间内候选基因
Table 2 Candidate genes in GWAS analysis interval for root surface area

基因编号Gene ID Cla97C02G043190 Cla97C02G043200 Cla97C02G043210 Cla97C02G043220 Cla97C02G043230 Cla97C02G043240 Cla97C02G043250 Cla97C02G043260 Cla97C02G043270 Cla97C02G043280 Cla97C02G043290 Cla97C02G043300 Cla97C02G043310 Cla97C02G043320 Cla97C02G043330 Cla97C02G043340 Cla97C02G043350 Cla97C02G043360 Cla97C02G043370 Cla97C02G043380 Cla97C02G043390 Cla97C02G043400 Cla97C02G043410基因功能注释Gene function annotation information未知蛋白Uncharacterized(LOC101222318)(RS)-去甲乌药碱6位氧甲基转移酶(RS)-norcoclaurine 6-O-methyltransferase-like(LOC103490512)(RS)-去甲乌药碱6位氧甲基转移酶(RS)-norcoclaurine 6-O-methyltransferase-like(LOC103490512)含有ELMO结构域的蛋白A ELMO domain-containing protein A-like(LOC103490510)分泌肽类激素表皮模式因子1 Protein EPIDERMAL PATTERNING FACTOR 1(LOC102625234)锌指蛋白MAGPIE-like Zinc finger protein MAGPIE-like未知蛋白Uncharacterized(LOC103490509)乙烯响应转录因子ERF054 Ethylene-responsive transcription factor ERF054-like(LOC101227522)乙烯响应转录因子ERF054 Ethylene-responsive transcription factor ERF054-like(LOC101227522)原叶绿素酸酯还原酶Protochlorophyllide reductase(LOC103490507)可能的多元醇转运体4 Probable polyol transporter 4(LOC103490506)网格蛋白轻链1 Clathrin light chain 1(LOC103490517)扩张蛋白B3 Expansin-B3(LOC103490518)四次跨膜蛋白19 Tetraspanin-19(LOC103490521)未知蛋白Uncharacterized LOC101203471(LOC101203471)假定蛋白Hypothetical protein Csa_6G128020未知蛋白Uncharacterized LOC103490523 E3泛素蛋白连接酶RMA1H1 E3 ubiquitin-protein ligase RMA1H1-like未知蛋白Uncharacterized(LOC101203963)未知蛋白Uncharacterized(LOC103490624)核黄素合酶Riboflavin synthase-like(LOC101204942)胼胝质合成酶12 Callose synthase 12-like甘露聚糖内切-1,4-β-甘露糖苷酶2 Mannan endo-1,4-beta-mannosidase 2(LOC103490531)
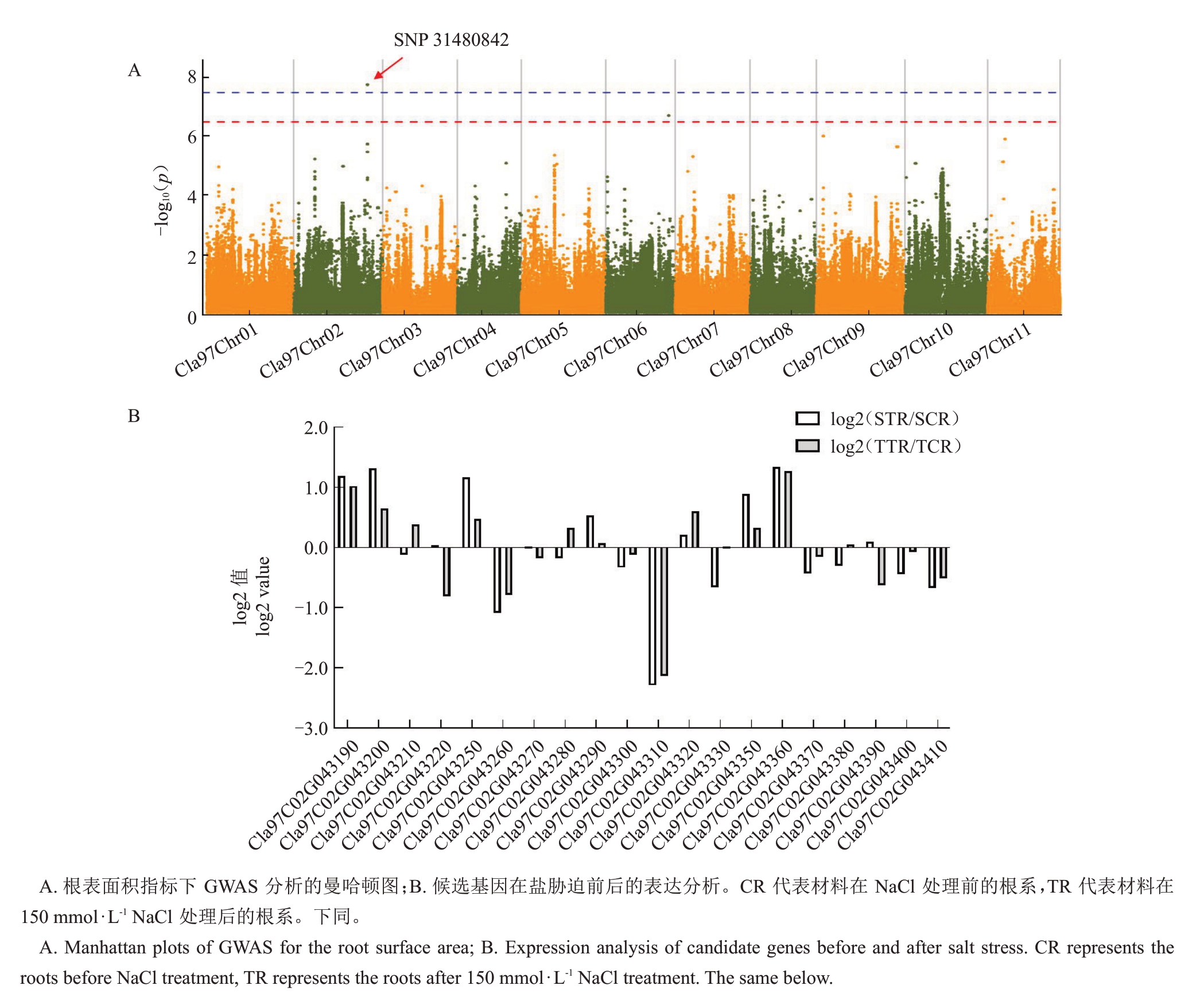
图2 与根表面积相关的SNP 位点及候选基因分析
Fig.2 Analysis of SNP locus and candidate genes related to root surface area
对于根K+含量,鉴定出25个显著SNP位点,其中在8号染色体上鉴定出4个SNP位点(SNP1534021、SNP1534034、SNP1543221和SNP1591030),在10号染色体上鉴定出21 个SNP 位点(SNP15499967、SNP15500235、SNP15579732、SNP15725425、SNP1-5763480、SNP15764890、SNP15812170、SNP15826-966、SNP15828491、SNP15875980、SNP15903887、SNP15925585、SNP15941986、SNP15942088、SNP1-5991712、SNP16079486、SNP16151054、SNP161793-09、SNP16250312、SNP16271845 和SNP16324947)(图3-A)。在以上SNP 位点附近(前后各100 kb 范围内)获得25 个基因(表3)。利用盐处理前后的耐盐材料和盐敏感材料对这23 个基因的表达水平进行分析,发现只有12 个基因具有表达量,并且Cla97C08G145130、Cla97C10G191810、Cla97C08G-145090、Cla97C08G145150 和Cla97C08G145120 在两份材料中均受盐胁迫的诱导上调表达(图3-B),表明这些基因可能响应盐胁迫或与根系对K+的转运相关。
表3 根K+含量指标下GWAS 分析区间内候选基因
Table 3 Candidate genes in GWAS analysis interval for root K+content

注:“-”代表该基因没有功能注释。下同。
Note:“-”indicates that the gene has no functional annotation.The same below.
基因号Gene ID Cla97C08G145000 Cla97C08G145010 Cla97C08G145020 Cla97C08G145030 Cla97C08G145040 Cla97C08G145050 Cla97C08G145060 Cla97C08G145090 Cla97C08G145100 Cla97C08G145110 Cla97C08G145120 Cla97C08G145130 Cla97C08G145140 Cla97C08G145070 Cla97C08G145080 Cla97C08G145150 Cla97C08G145160 Cla97C08G145170 Cla97C08G145180 Cla97C08G145190 Cla97C08G145200 Cla97C10G191790 Cla97C10G191800 Cla97C10G191810 Cla97C10G191820基因功能注释Gene function annotation information β-半乳糖苷酶15-like Beta-galactosidase 15-like丝氨酸/苏氨酸蛋白激酶Serine/threonine-protein kinase At1g09600(LOC103498696)-未知蛋白Uncharacterized(LOC101208468)羟基类固醇11-β-脱氢酶1样蛋白Hydroxysteroid 11-beta-dehydrogenase 1-like protein-like(LOC101206377)未知蛋白Uncharacterized(LOC101230421)-未知蛋白Uncharacterized(LOC103498665)钼酸盐合成酶硫载体亚基Molybdopterin synthase sulfur carrier subunit-SCY1样蛋白2 SCY1-like protein 2(LOC103498664)甘露聚糖内切-1,4-β-甘露糖苷酶1 Mannan endo-1,4-beta-mannosidase 1-like(LOC103498662)核糖体RNA加工蛋白36同源物Ribosomal RNA processing protein 36 homolog(LOC103496541)未知蛋白Uncharacterized(LOC103492844)-含五肽重复序列蛋白Pentatricopeptide repeat-containing protein At3g13880(LOC103498661)含五肽重复序列蛋白Pentatricopeptide repeat-containing protein At3g13880-like克隆BAC 393-16,完整序列Clone BAC 39-3-16,complete sequence-内切葡聚糖酶9 Endoglucanase 9-like(LOC101222401)克隆BAC 66-O16 Clone BAC 66-O16木葡聚糖内转糖基化酶,部分Xyloglucan endotransglycosylase,partial木葡聚糖内转葡萄糖基化酶/水解酶蛋白Xyloglucan endotransglucosylase/hydrolase protein 6-like(LOC101219554)同源异型域-亮氨酸拉链蛋白HAT22 Homeobox-leucine zipper protein HAT22-like(LOC103491835)1-氨基环丙烷-1-羧酸合酶CMA101 1-aminocyclopropane-1-carboxylate synthase CMA101-like(LOC101205326)
图3 与根K+含量相关的SNP 位点及候选基因分析
Fig.3 Analysis of SNP locus and candidate genes related to root K+content
对于根Na+含量,鉴定出2 个显著SNP 位点(SNP11293147 和SNP11301987),均位于1 号染色体上(图4-A)。在SNP 位点附近(前后各100 kb 范围内)获得10 个基因(表4)。利用盐处理前后的耐盐材料和盐敏感材料对这10 个基因的表达水平进行分析,发现只有7 个基因具有表达量,并且Cla97C01G009540、Cla97C01G009490和Cla97C01-G009510在两份材料中均受盐胁迫的诱导上调表达(图4-B),表明这些基因可能响应盐胁迫。
表4 根Na+含量指标下GWAS 分析区间内候选基因
Table 4 Candidate genes in GWAS analysis interval for root Na+content

基因编号Gene ID Cla97C01G009540 Cla97C01G009450 Cla97C01G009460 Cla97C01G009470 Cla97C01G009480 Cla97C01G009490基因功能注释Gene function annotation information韧皮部蛋白2样A9 Phloem protein 2-like A9(LOC103498278)假定蛋白Hypothetical protein Csa_1G707120--Cla97C01G009500 Cla97C01G009510 Cla97C01G009520 Cla97C01G009530囊泡相关膜蛋白722 Vesicle-associated membrane protein 722-like(LOC103498283)肽基脯氨酸顺式反式异构酶FKBP17-1,叶绿体Peptidyl-prolyl cis-trans isomerase FKBP17-1,chloroplastic-like(LOC101222010)葫芦分离的Tularosa_Cave叶绿体,部分基因组Lagenaria siceraria isolate Tularosa_Cave chloroplast,partial genome锌指蛋白ZPR1 Zinc finger protein ZPR1-like(LOC103498281)未知蛋白Uncharacterized(LOC101222251)核糖核酸酶H2亚基C Ribonuclease H2 subunit C-like(LOC101208243)

图4 与根Na+含量相关的SNP 位点及候选基因分析
Fig.4 Analysis of SNP locus and candidate genes related to root Na+content
对于根可溶性糖含量,鉴定出1 个显著SNP 位点(SNP20908124),位于4 号染色体上(图5-A)。在SNP 位点附近(前后各100 kb 范围内)获得18 个基因(表5)。利用盐处理前后的耐盐材料和盐敏感材料对这18 个基因的表达水平进行分析,发现有17 个基因具有表达量,并且Cla97C04G073310、Cla97C04G073300、Cla97C04G073240、Cla97C04G-073230、Cla97C04G073290、Cla97C04G073280、Cla9-7C04G073190、Cla97C04G073210和Cla97C04G073-270 在两份材料中均受盐胁迫的诱导上调表达(图5-B),表明这些基因可能响应盐胁迫或与可溶性糖的积累相关。
表5 根可溶性糖含量指标下GWAS 分析区间内候选基因
Table 5 Candidate genes in GWAS analysis interval for root soluble sugar content

基因编号Gene ID Cla97C04G073340 Cla97C04G073330 Cla97C04G073320 Cla97C04G073310 Cla97C04G073300 Cla97C04G073290 Cla97C04G073280 Cla97C04G073270 Cla97C04G073260 Cla97C04G073250 Cla97C04G073240 Cla97C04G073230 Cla97C04G073220 Cla97C04G073210 Cla97C04G073200 Cla97C04G073190 Cla97C04G073180 Cla97C04G073170基因功能注释Gene function annotation information-毛状体双折射蛋白34 Trichome birefringence-like 34(LOC103502160)双折射蛋白35 Birefringence-like 35(LOC103502161)MATE外排家族蛋白2 MATE efflux family protein 2(LOC103502163)干旱应答元件的结合蛋白2A Dehydration-responsive element-binding protein 2A BRCA1-A复合亚基BRCA1-A complex subunit Abraxas(LOC103502164)PRA1家族蛋白B2 PRA1 family protein B2巨噬细胞迁移抑制因子同源物Macrophage migration inhibitory factor homolog(LOC101227626)巨噬细胞迁移抑制因子同源物Macrophage migration inhibitory factor homolog(LOC103502168)一种具有SEC14蛋白结构域的脂质转移蛋白Patellin-6(LOC103502169)-蛋白磷酸酶2C 65 Protein phosphatase 2C 65(LOC103502171)未知蛋白Uncharacterized(LOC101209474)未知蛋白Uncharacterized(LOC101209719)溶质载体家族40个成员2-like Solute carrier family 40 member 2-like(LOC101223571)未知蛋白Uncharacterized LOC101215386(LOC101215386)创面诱导蛋白Wound-induced protein 1-like(LOC101228392)未知蛋白Uncharacterized(LOC103502180)
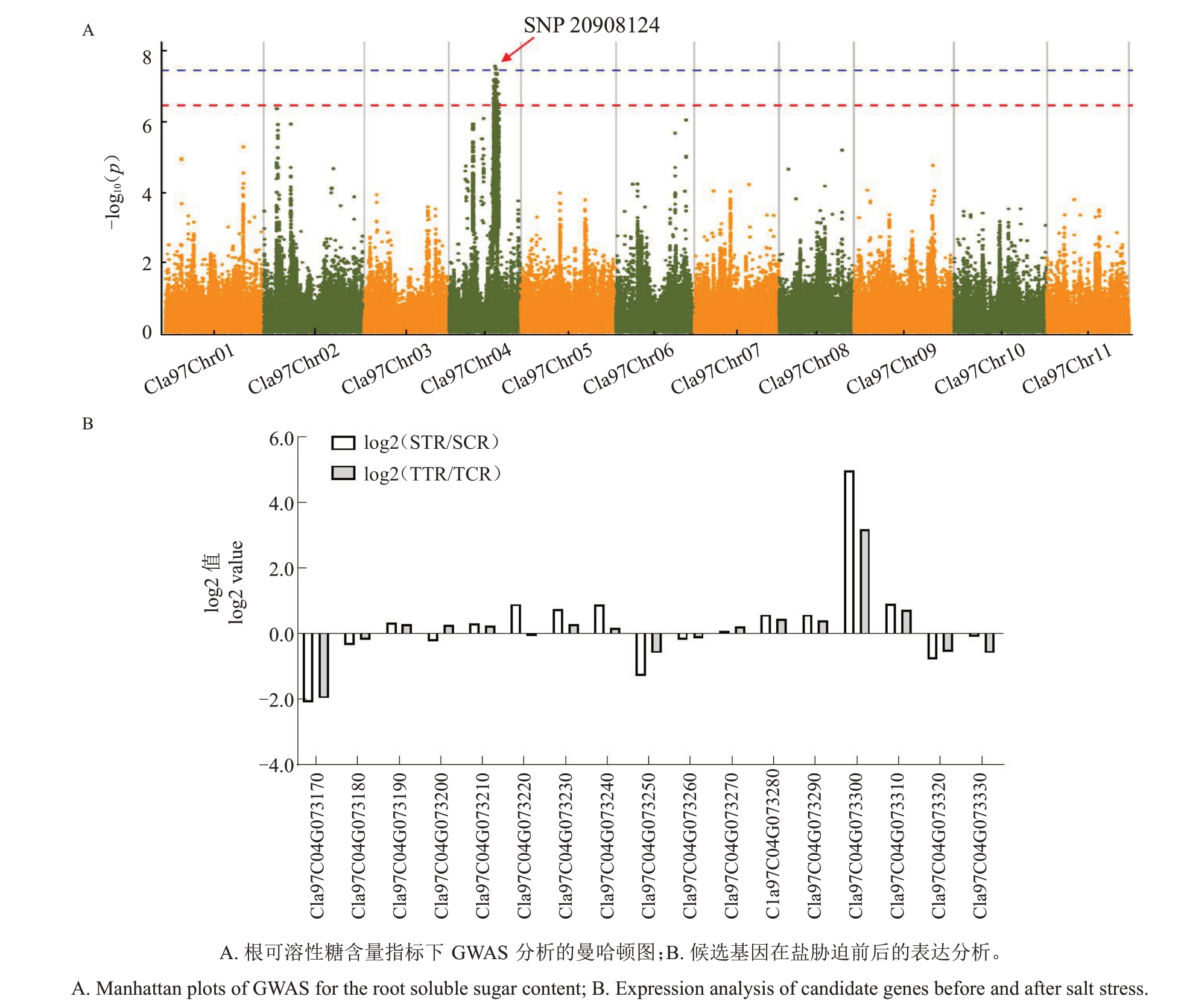
图5 与根可溶性糖含量相关的SNP 位点及候选基因分析
Fig.5 Analysis of SNP locus and candidate genes related to root soluble sugar content
2.3 西瓜耐盐相关关键候选基因的筛选
利用上述区间内获得的具有表达量的56 个基因与SCR-vs-STR 和TCR-vs-TTR 组合获得的4870个共有差异表达基因[20]作韦恩图分析,以筛选耐盐相关的关键候选基因。结果表明,共得到9 个共有的基因(图6-A),表明区间内有9个差异表达基因。为了进一步明确这9个基因在西瓜响应盐胁迫中的作用,分析了其在盐胁迫下耐盐材料和盐敏感材料中的表达水平,发现Cla97C08G145130、Cla97C04G073300、Cla97C01G009540、Cla97C10G191810、Cla97C02G0-43360、Cla97C02G043190和Cla97C04G073310受盐胁诱导显著上调表达,而Cla97C04G073170 和Cla97C02G043310受盐胁诱导显著下调表达。根据基因的表达趋势可将他们分为两类(图6-B),其中Ⅰ类包含4个基因,Ⅱ类包含5个基因。值得注意的是,Ⅰ类中Cla97C04G073300(dehydration-responsive element-binding protein 2A,DREB2A)变化最显著,在盐敏感材料和耐盐材料中分别上调31.63和9.18倍;Ⅱ类中Cla97C08G145130(mannan endo-1,4-betamannosidase 1- like,ManA1)和Cla97C01G009540(phloem protein 2-like A9,PP2A9)在盐敏感材料和耐盐材料中分别上调255.82和7.80倍、13.10和3.56倍。推测他们可能是西瓜耐盐相关的关键候选基因,在西瓜响应盐胁迫过程中具有重要作用。

图6 西瓜耐盐相关关键候选基因的筛选
Fig.6 Screening of key candidate genes related to salt tolerance in watermelon
3 讨 论
在植物中,盐胁迫一般通过施加几个主要的限制性因素来抑制植物的生长和发育。第一个限制是渗透胁迫(降低外部水势),主要抑制植物吸收水分的能力[24-26]。在宏观水平上,根细胞的扩张由于膨胀压力的降低而立即被阻止,为了解决这一问题,植物必须进行渗透调节[27]。
Chen等[28]研究表明,可溶性糖、K+、Na+含量等指标在葫芦科作物耐盐中具有重要作用。在本研究中,在可溶性糖、K+、Na+含量指标下均获得与耐盐相关的显著SNP 位点,表明可溶性糖、K+、Na+在西瓜响应盐胁迫中发挥着重要作用。可溶性糖不仅为有机物的合成提供物质和能量,而且参与渗透调节和细胞失水后的恢复过程以及维持蛋白质结构的稳定。姚铭榕等[29]研究发现,盐处理后番茄叶片中的可溶性糖含量显著高于对照。石婧等[30]在棉花上的研究表明,盐胁迫下棉花叶片中的可溶性糖含量显著上升,并且耐盐品种中可溶性糖含量显著高于盐敏感品种。
外源添加可溶性糖可直接或者间接地提高植物对非生物胁迫的抵抗能力[31]。施加外源糖可以显著降低小黑麦的相对电导率,缓解小黑麦受到的盐胁迫[32]。另外,在小麦中,低浓度的葡萄糖处理,能够促进盐胁迫下种子的萌发以及胚芽鞘和胚根的生长[33]。外源葡萄糖处理可缓解盐胁迫下叶绿素含量的下降,保持离子平衡和积累渗透调节物质Pro,以减少水分的散失,激活抗氧化酶活性,最终提高盐胁迫下植物的干质量[33]。此外,外源葡萄糖能够抑制盐胁迫下小麦幼苗细胞中的Na+积累,同时促进K+的吸收,有利于盐胁迫下幼苗中的离子平衡[34]。在盐胁迫下,葡萄糖还具有渗透保护剂和自由基清除剂的功能,能够提高水稻对盐胁迫的抵抗能力[35]。海藻糖作为可溶性糖的一种,在保护植物免受非生物胁迫方面发挥了重要作用,通过减少活性氧的积累减轻高盐浓度下的氧化应激[36]。20 mmol·L-1外源海藻糖显著改善了盐胁迫下西瓜幼苗生理状态,提高了过氧化物酶、超氧化物歧化酶、过氧化氢酶等酶活性以及西瓜根部K+/Na+比值[6]。15 mmol·L-1外源海藻糖能够提高盐胁迫下黄秋葵的株高、干质量、鲜质量和K+含量,降低Na+含量和Na+/K+比值[37]。10 mmol·L-1的海藻糖通过DNA 去甲基化、增强抗氧化能力和积累脱落酸来增强番茄幼苗的耐盐性[38]。徐婷等[39]对薄皮甜瓜的研究发现,叶面喷施0.4%海藻糖通过增强抗氧化酶活性来缓解盐胁迫对甜瓜幼苗造成的伤害。在本研究中,可溶性糖含量变异最为丰富,并且在该指标下筛选到1 个显著的SNP位点,盐胁迫后,区间内候选基因DREB2A(Cla97C04G073300)在耐盐材料和盐敏感材料中的表达水平显著上调,且变化最为明显,表明DREB2A响应盐胁迫或与可溶性糖的积累相关。研究表明,在玉米中,ZmDREB2A 通过与ZmGOLS2 启动子结合直接调控ZmGOLS2 的表达,促进棉子糖积累,进而提高玉米的耐盐性[40];在大豆中,过表达水稻Os-DREB2A能够调控一些胁迫响应转录因子和关键基因的表达水平,积累棉子糖来增强大豆的耐盐性[41]。
盐胁迫施加的第二个限制因子是离子失衡,通常称为“离子胁迫”或“离子毒性”[25,42-43]。在大多数情况下,这种限制与细胞内Na+的过度积累有关。虽然Na+会损害植物的代谢,并可能导致植物死亡,但Na+在植物中的靶标尚不清楚[44]。Na+毒性体现在对酶活性具有抑制作用,如细胞质中包含的许多参与初级代谢、卡尔文循环、苯丙烷途径、糖酵解、多胺和淀粉合成的酶。在Na+指标下鉴定出2 个显著性SNP 位点,区间内基因PP2A9(Cla97C01G009540)的表达量在耐盐材料和盐敏感材料中显著上调。研究表明,PP2 家族成员编码的蛋白质具有抗逆功能。在高浓度盐胁迫下,过表达NtPP2A9L1 烟草的抗氧化酶活性、脯氨酸和叶绿素含量显著提高,丙二醛和过氧化氢含量显著降低。另外,过表达NtPP2A9L1显著上调活性氧清除相关基因和应激反应相关基因的转录水平[45]。在黄瓜中,CsPP2-A1-RNAi 植株表现出较弱的耐盐性,而CsPP2-A1 过表达植株始终表现出较强的耐盐性,验证了CsPP2-A1通过渗透调节和活性氧稳态增强黄瓜的耐盐能力[46]。在柽柳中,过表达ThPP2 的植株中过氧化氢酶活性、超氧化物酶活性及电解质和丙二醛含量降低,超氧化物歧化酶、过氧化物酶和过氧化氢酶活性升高。相比之下,RNAi介导的ThPP2的瞬时沉默在柽柳中具有相反的效果,表明ThPP2 通过减少活性氧积累和增强抗氧化酶活性来调节柽柳耐盐性[47]。因此,推测西瓜PP2A9 基因对西瓜耐盐性同样具有重要作用,其可能通过活性氧稳态、渗透调节提高西瓜耐盐性。但是其如何参与细胞中Na+的吸收和转运还需要进一步深入研究。
细胞质中众多酶的活性除了受Na+调控外,有许多同时受K+控制[48]。作为一种主要的无机渗透物,K+对细胞渗透调节和膨胀维持至关重要[49]。Chakraborty 等[50]研究表明,外源K+的施用改善了花生的水分状况,使其在盐胁迫下具有更高的生物量和更强的耐盐性。Na+和K+具有拮抗效应,Na+显著抑制植株对K+的吸收和转运,导致高浓度盐条件下K+缺乏[51]。K+含量被认为是耐盐性的关键指标,其在胁迫信号转导、离子稳态中起至关重要的作用[52]。在本研究中,K+含量指标下鉴定出的显著SNP位点最多(25个),一方面证实了K+在耐盐性方面的重要性,另一方面也表明了K+可能参与了西瓜响应盐胁迫的多条调控途径。区间内基因ManA1(Cla97C08G145130)在耐盐材料和盐敏感材料中的上调倍数较高,该基因编码一个甘露聚糖内切-1,4-β甘露糖苷酶,能够催化甘露聚糖聚合物中内部-1,4-β-甘露糖苷键的随机水解,释放短链β-1,4-甘露聚糖和甘露聚糖。前人研究表明,该类基因在植物上的作用主要与果实开裂和成熟相关[52-53]。但是该基因是如何参与细胞中K+的吸收和转运以及提高耐盐性的研究还未见报道,需要进一步探索。
4 结 论
笔者利用121份西瓜核心种质材料进行GWAS分析,在根表面积及根K+、根Na+和根可溶性糖含量指标下筛选到与耐盐相关的显著SNP变异位点,并在候选区间内获得多个候选基因。研究结果为解析提高西瓜耐盐性的分子机制及开发分子标记用于辅助选择育种奠定了基础。
[1] FAO.https://www.fao.org/faostat/zh/#data/QCL.2024.
[2] 陈敏,李海云,吕福堂.植物耐盐性研究进展[J].聊城大学学报(自然科学版),2011,24(3):47-50.CHEN Min,LI Haiyun,LÜ Futang.Research advances in mechanisms of plant salinity tolerance[J]. Journal of Liaocheng University(Natural Science Edition),2011,24(3):47-50.
[3] 贾丽娟.粮食主产区设施农业用地管控机制研究:以河南省为例[D].郑州:郑州大学,2022.JIA Lijuan. Study on the control mechanism of land for facility agriculture in the main grain producingareas:An example from Henan province[D].Zhengzhou:Zhengzhou University,2022.
[4] LI H,CHANG J J,CHEN H J,WANG Z Y,GU X R,WEI C H,ZHANG Y,MA J X,YANG J Q,ZHANG X. Exogenous melatonin confers salt stress tolerance to watermelon by improving photosynthesis and redox homeostasis[J]. Frontiers in Plant Science,2017,8:295.
[5] YAN Y Y,WANG S S,WEI M,GONG B,SHI Q H. Effect of different rootstocks on the salt stress tolerance in watermelon seedlings[J].Horticultural Plant Journal,2018,4(6):239-249.
[6] YUAN G P,SUN D X,AN G L,LI W H,SI W J,LIU J P,ZHU Y C. Transcriptomic and metabolomic analysis of the effects of exogenous trehalose on salt tolerance in watermelon (Citrullus lanatus)[J].Cells,2022,11(15):2338.
[7] 高博文,孙德玺,袁高鹏,安国林,李卫华,刘君璞,朱迎春.121 份西瓜材料幼苗期耐盐性鉴定[J].果树学报,2022,39(9):1597-1606.GAO Bowen,SUN Dexi,YUAN Gaopeng,AN Guolin,LI Weihua,LIU Junpu,ZHU Yingchun.Identification of salt tolerance of 121 watermelon(Citrullus lanatus L.)germplasm resources[J]. Journal of Fruit Science,2022,39(9):1597-1606.
[8] ZHU H J,ZHAO S J,LU X Q,HE N,GAO L,DOU J L,BIE Z L,LIU W G.Genome duplication improves the resistance of watermelon root to salt stress[J]. Plant Physiology and Biochemistry,2018,133:11-21.
[9] YUAN G P,LIU J P,AN G L,LI W H,SI W J,SUN D X,ZHU Y C.Genome-wide identification and characterization of the trehalose-6-phosphate synthetase (TPS) gene family in watermelon(Citrullus lanatus) and their transcriptional responses to salt stress[J]. International Journal of Molecular Sciences,2021,23(1):276.
[10] LIU Y,ZHANG W H,ELANGO D,LIU H X,JIN D D,WANG X Y,WU Y.Metabolome and transcriptome analysis reveals molecular mechanisms of watermelon under salt stress[J]. Environmental and Experimental Botany,2023,206:105200.
[11] GUO S G,ZHAO S J,SUN H H,WANG X,WU S,LIN T,REN Y,GAO L,DENG Y,ZHANG J,LU X Q,ZHANG H Y,SHANG J L,GONG G Y,WEN C L,HE N,TIAN S W,LI M Y,LIU J P,WANG Y P,ZHU Y C,JARRET R,LEVI A,ZHANG X P,HUANG S W,FEI Z J,LIU W G,XU Y. Resequencing of 414 cultivated and wild watermelon accessions identifies selection for fruit quality traits[J]. Nature Genetics,2019,51(11):1616-1623.
[12] DENG Y,LIU S C,ZHANG Y L,TAN J S,LI X P,CHU X,XU B H,TIAN Y,SUN Y D,LI B S,XU Y B,DENG X W,HE H,ZHANG X P.A telomere-to-telomere gap-free reference genome of watermelon and its mutation library provide important resources for gene discovery and breeding[J]. Molecular Plant,2022,15(8):1268-1284.
[13] WU S,SUN H H,GAO L,BRANHAM S,MCGREGOR C,RENNER S S,XU Y,KOUSIK C,WECHTER W P,LEVI A,FEI Z J. A Citrullus genus super-pangenome reveals extensive variations in wild and cultivated watermelons and sheds light on watermelon evolution and domestication[J]. Plant Biotechnology Journal,2023,21(10):1926-1928.
[14] YIN W J,LU T Q,CHEN Z G,LU T,YE H F,MAO Y J,LUO Y T,LU M,ZHU X D,YUAN X,RAO Y C,WANG Y X.Quantitative trait locus mapping and candidate gene analysis for salt tolerance at bud stage in rice[J]. Frontiers in Plant Science,2023,13:1041081.
[15] DOU J L,ZHAO S J,LU X Q,HE N,ZHANG L,ALI A,KUANG H H,LIU W G. Genetic mapping reveals a candidate gene (ClFS1) for fruit shape in watermelon (Citrullus lanatus L.)[J].Theoretical and Applied Genetics,2018,131(4):947-958.
[16] 王学征,刘硕,徐雅菲,高启帆,修洪超,李燕,李亚平,齐国安,刘识.西瓜种子大小全基因组关联分析[J].东北农业大学学报,2019,50(5):26-34.WANG Xuezheng,LIU Shuo,XU Yafei,GAO Qifan,XIU Hongchao,LI Yan,LI Yaping,QI Guoan,LIU Shi. Genome-wide association analysis of seed size characters in watermelon[J].Journal of Northeast Agricultural University,2019,50(5):26-34.
[17] 高美玲,胡创然,袁成志,郭宇,刘秀杰,刘继秀,高越. 基于GWAS 的西瓜种子性状候选QTL 鉴定[J]. 四川农业大学学报,2021,39(6):721-728.GAO Meiling,HU Chuangran,YUAN Chengzhi,GUO Yu,LIU Xiujie,LIU Jixiu,GAO Yue. Identification of candidate QTL for watermelon seed traits based on GWAS[J]. Journal of Sichuan Agricultural University,2021,39(6):721-728.
[18] GONG C S,ZHAO S J,YANG D D,LU X Q,ANEES M,HE N,ZHU H J,ZHAO Y,LIU W G. Genome-wide association analysis provides molecular insights into the natural variation of watermelon seed size[J]. Horticulture Research,2022,9:uhab074.
[19] REN Y,LI M Y,GUO S G,SUN H H,ZHAO J Y,ZHANG J,LIU G M,HE H J,TIAN S W,YU Y T,GONG G Y,ZHANG H Y,ZHANG X L,ALSEEKH S,FERNIE A R,SCHELLER H V,XU Y. Evolutionary gain of oligosaccharide hydrolysis and sugar transport enhanced carbohydrate partitioning in sweet watermelon fruits[J].The Plant Cell,2021,33(5):1554-1573.
[20] ZHU Y C,YUAN G P,GAO B W,AN G L,LI W H,SI W J,SUN D X,LIU J P. Comparative transcriptome profiling provides insights into plant salt tolerance in watermelon (Citrullus lanatus)[J].Life,2022,12(7):1033.
[21] 高博文.西瓜幼苗耐盐鉴定体系的初步建立及转录组分析[D].北京:中国农业科学院,2022.GAO Bowen. Preliminary establishment of salt tolerance evaluation system and transcriptomic analysis in watermelon seedlings[D]. Beijing:Chinese Academy of Agricultural Sciences,2022.
[22] LIPPERT C,LISTGARTEN J,LIU Y,KADIE C M,DAVIDSON R I,HECKERMAN D. FaST linear mixed models for genome-wide association studies[J].Nature Methods,2011,8(10):833-835.
[23] CHEN C J,WU Y,LI J W,WANG X,ZENG Z H,XU J,LIU Y L,FENG J T,CHEN H,HE Y H,XIA R.TBtools-II:A“one for all,all for one”bioinformatics platform for biological big-data mining[J].Molecular Plant,2023,16(11):1733-1742.
[24] YANG Y Q,GUO Y.Elucidating the molecular mechanisms mediating plant salt-stress responses[J].New Phytologist,2018,217(2):523-539.
[25] ZHAO C Z,ZHANG H,SONG C P,ZHU J K,SHABALA S.Mechanisms of plant responses and adaptation to soil salinity[J].The Innovation,2020,1(1):100017.
[26] 薛洋,赵胜杰,何玉敏,王方方,李杰,张林龙,徐志红,王平勇.瓜菜作物耐盐性研究进展[J].中国瓜菜,2023,36(12):1-8.XUE Yang,ZHAO Shengjie,HE Yumin,WANG Fangfang,LI Jie,ZHANG Linlong,XU Zhihong,WANG Pingyong.Research progress on salt tolerance of cucurbits and vegetables[J]. China Cucurbits and Vegetables,2023,36(12):1-8.
[27] MUNNS R,PASSIOURA J B,COLMER T D,BYRT C S. Osmotic adjustment and energy limitations to plant growth in saline soil[J].New Phytologist,2020,225(3):1091-1096.
[28] CHEN C Y,YU W C,XU X R,WANG Y H,WANG B,XU S Y,LAN Q K,WANG Y. Research advancements in salt tolerance of Cucurbitaceae:From salt response to molecular mechanisms[J]. International Journal of Molecular Sciences,2024,25(16):9051.
[29] 姚铭榕,谢凯玲,张文静,张文杰,史庆华,刘杰.两个番茄品种幼苗耐盐性评价[J].中国瓜菜,2021,34(8):21-25.YAO Mingrong,XIE Kailing,ZHANG Wenjing,ZHANG Wenjie,SHI Qinghua,LIU Jie.Evaluation of salt tolerance of two tomato varieties[J]. China Cucurbits and Vegetables,2021,34(8):21-25.
[30] 石婧,刘东洋,张凤华.棉花幼苗对盐胁迫的生理响应与耐盐机理[J].浙江农业学报,2020,32(7):1141-1148.SHI Jing,LIU Dongyang,ZHANG Fenghua. Physiological response and salt tolerance mechanism of cotton seedlings to salt stress[J]. Acta Agriculturae Zhejiangensis,2020,32(7):1141-1148.
[31] 陈宏艳,李小二,李忠光.糖信号及其在植物响应逆境胁迫中的作用[J].生物技术通报,2022,38(7):80-89.CHEN Hongyan,LI Xiaoer,LI Zhongguang. Sugar signaling and its role in plant response to environmental stress[J].Biotechnology Bulletin,2022,38(7):80-89.
[32] 王丽华,李改玲,李晶,左师宇,曹鑫波,佟昊阳,魏湜.外源糖对盐胁迫下小黑麦幼苗糖代谢的影响[J]. 麦类作物学报,2017,37(4):548-553.WANG Lihua,LI Gailing,LI Jing,ZUO Shiyu,CAO Xinbo,TONG Haoyang,WEI Shi. Effect of exogenous sugar on the sugar metabolism in triticale seedling under salt stress[J]. Journal of Triticeae Crops,2017,37(4):548-553.
[33] HU M Y,SHI Z G,ZHANG Z B,ZHANG Y J,LI H.Effects of exogenous glucose on seed germination and antioxidant capacity in wheat seedlings under salt stress[J]. Plant Growth Regulation,2012,68(2):177-188.
[34] NEMATI I,MORADI F,GHOLIZADEH S,ESMAEILI M A,BIHAMTA M R.The effect of salinity stress on ions and soluble sugars distribution in leaves,leaf sheaths and roots of rice (Oryza sativa L.)seedlings[J].Plant,Soil and Environment,2011,57(1):26-33.
[35] PATTANAGUL W,THITISAKSAKUL M. Effect of salinity stress on growth and carbohydrate metabolism in three rice(Oryza sativa L.) cultivars differing in salinity tolerance[J]. Indian Journal of Experimental Biology,2008,46(10):736-742.
[36] MOSTOFA M G,HOSSAIN M A,FUJITA M. Trehalose pretreatment induces salt tolerance in rice (Oryza sativa L.) seedlings:Oxidative damage and co-induction of antioxidant defense and glyoxalase systems[J]. Protoplasma,2015,252(2):461-475.
[37] 刘畅,程玉静,王小秋,仇亮,刘岩,戴志刚,谢冬微,孙健.海水胁迫下外源海藻糖对黄秋葵幼苗耐盐性的影响[J].中国瓜菜,2023,36(8):84-91.LIU Chang,CHENG Yujing,WANG Xiaoqiu,QIU Liang,LIU Yan,DAI Zhigang,XIE Dongwei,SUN Jian. Effects of exogenous trehalose on salt tolerance of okra seedlings under seawater stress[J].China Cucurbits and Vegetables,2023,36(8):84-91.
[38] YAO Y D,YANG Y,PAN Y,LIU Z S,HOU X M,LI Y H,ZHANG H S,WANG C L,LIAO W B. Crucial roles of trehalose and 5-azacytidine in alleviating salt stress in tomato:Both synergistically and independently[J]. Plant Physiology and Biochemistry,2023,203:108075.
[39] 徐婷,周传余,周超,赵索,武琳琳,谭可菲.海藻糖对盐胁迫下薄皮甜瓜幼苗抗氧化系统的影响[J].北方园艺,2014(19):28-30.XU Ting,ZHOU Chuanyu,ZHOU Chao,ZHAO Suo,WU Linlin,TAN Kefei.Effect of trehalose on antioxidant system of melon seedling under salt stress[J]. Northern Horticulture,2014(19):28-30.
[40] GU L,ZHANG Y M,ZHANG M S,LI T,DIRK L M A,DOWNIE B,ZHAO T Y. ZmGOLS2,a target of transcription factor ZmDREB2A,offers similar protection against abiotic stress as ZmDREB2A[J]. Plant Molecular Biology,2016,90(1/2):157-170.
[41] ZHANG X X,TANG Y J,MA Q B,YANG C Y,MU Y H,SUO H C,LUO L H,NIAN H. OsDREB2A,a rice transcription factor,significantly affects salt tolerance in transgenic soybean[J].PLoS One,2013,8(12):e83011.
[42] HERNÁNDEZ J A,FERRER M A,JIMÉNEZ A,BARCELÓ A R,SEVILLA F.Antioxidant systems and O2.-/H2O2 production in the apoplast of pea leaves:Its relation with salt-induced necrotic lesions in minor veins[J]. Plant Physiology,2001,127(3):817-831.
[43] BARBA-ESPÍN G,CLEMENTE-MORENO M J,ÁLVAREZ S,GARCÍA-LEGAZ M F,HERNÁNDEZ J A,DÍAZ-VIVANCOS P. Salicylic acid negatively affects the response to salt stress in pea plants[J].Plant Biology,2011,13(6):909-917.
[44] CHEESEMAN J M.The integration of activity in saline environments:Problems and perspectives[J]. Functional Plant Biology,2013,40(9):759-774.
[45] XING M G,MA Y Y,HOU H,ZHAO C M,XUE R G.Overexpression of Nicotiana tomentosiformis phloem protein 2-like A9-likegene NTPP2A9L enhances tolerance to abiotic stress in transgenic tobacco[J/OL]. Social Science Research Network,2022.
[46] SI Y Y,FAN H F,LU H J,LI Y P,GUO Y T,LIU C,CHAI L A,DU C X. Cucumis sativus PHLOEM PROTEIN 2-A1 like gene positively regulates salt stress tolerance in cucumber seedlings[J].Plant Molecular Biology,2023,111(6):493-504.
[47] 王培龙,刘中原,张腾倩,唐绯绯,曲冠证,高彩球.刚毛柽柳ThPP2C 基因的克隆和表达分析[J]. 植物研究,2017,37(3):395-401.WANG Peilong,LIU Zhongyuan,ZHANG Tengqian,TANG Feifei,QU Guanzheng,GAO Caiqiu. Cloning and expression analysis of ThPP2C gene from Tamarix hispida[J]. Bulletin of Botanical Research,2017,37(3):395-401.
[48] WU H H,ZHANG X C,GIRALDO J P,SHABALA S. It is not all about sodium:Revealing tissue specificity and signalling roles of potassium in plant responses to salt stress[J]. Plant and Soil,2018,431(1):1-17.
[49] SHABALA S.Regulation of potassium transport in leaves:From molecular to tissue level[J].Annals of Botany,2003,92(5):627-634.
[50] CHAKRABORTY K,BHADURI D,MEENA H N,KALARIYA K.External potassium(K(+))application improves salinity tolerance by promoting Na(+)-exclusion,K(+)-accumulation and osmotic adjustment in contrasting peanut cultivars[J]. Plant Physiology and Biochemistry,2016,103:143-153.
[51] ABBASI H,JAMIL M,HAQ A,ALI S,AHMAD R,MALIK Z,PARVEEN. Salt stress manifestation on plants,mechanism of salt tolerance and potassium role in alleviating it:A review[J].Zemdirbyste-Agriculture,2016,103(2):229-238.
[52] XUE L Z,SUN M T,WU Z,YU L,YU Q H,TANG Y P,JIANG F L.LncRNA regulates tomato fruit cracking by coordinating gene expression via a hormone-redox-cell wall network[J].BMC Plant Biology,2020,20(1):162.
[53] CHEN Y T,CHENG C Z,FENG X,LAI R L,GAO M X,CHEN W G,WU R J. Integrated analysis of lncRNA and mRNA transcriptomes reveals the potential regulatory role of lncRNA in kiwifruit ripening and softening[J]. Scientific Reports,2021,11(1):1671.