转录因子是植物生长发育和外部(非)生物胁迫响应的主要调控因子[1]。目前已知植物中存在的转录因子有58 种,其中,NAC 转录因子是超大转录因子家族之一,是植物特有的基因家族[2]。NAC 这一命名源自矮牵牛(Petunia hybrida)的NAM(no apical meristem)基因、拟南芥(Arabipopsis thaliana)的ATAF1/ATAF2(actiation factor 1/2)基因和CUC2(cup-shaped cotyledon 2)基因的首字母[3]。NAC 蛋白的N 末端区域含有约150 个高度保守氨基酸的NAC结构域,负责DNA结合和二聚体形成,可分为A~E 5个亚结构,其中A、C和D高度保守,B和E较为多变。NAC 蛋白的C 端由一些简单氨基酸重复序列构成高度可变的调控区域(transcription regulation region,TRR),具有蛋白结合活性,能够转录激活或者抑制[4]。
植物基因组中NAC转录因子成员众多,目前已在多种植物鉴定出这一家族成员,包括模式植物拟南芥(Arabidopsis thaliana,117 个)[5]和毛果杨(Populus trichocarpa,170 个)[6]、作物水稻(Oryza sativa,151)[5]、玉米(Zea mays,148 个)[7]和大豆(Glycine max,152 个)[8]、蔬菜作物番茄(Solanum lycopersicum,93个)[9]、辣椒(Capsicum annuum,104个)[10]、大白菜(Brassica rapa L.ssp.pekinensis,188个)[11]和果树作物苹果(Malus domestica,180 个)[12]、梨(Pyrus bretschneideri,185)[13]、火龙果(Hylocereus undatus,64个)[14]、菠萝(Ananas comosus,73个)[15]、欧李(Cerasus humilis,76个)[16]。
研究表明,NAC转录因子广泛参与植物多种生命代谢活动,包括在植物调控生长发育、响应逆境胁迫[17-20]、花器官形成[21]、器官边界和植物形态建成[22-23]、次生细胞壁形成与增厚[24-25]、芽和根尖分生组织形成[1,26]、侧根发育[20,27]、纤维发育[24]、植株衰老调节[28]、果实生长发育[29]、果实风味形成[30-31]、果实成熟[1,32]等方面发挥着重要作用。
皇冠李(Prunus salicina Lindl. var. cordata‘Huangguan’)是笔者团队选育的一种适宜福建地区栽培的优质柰李新品种。该品种于2018 年12 月通过福建省林木良种审定,编号为闽S-SV-PS-2018。皇冠李的果实呈鲜亮的黄色,口味鲜甜,风味浓郁,成熟期在5 月底至6 月初,是早熟柰李的优新品种。然而,皇冠李是属于典型的柰李类,存在的突出问题是果实内部的核顶端常与果肉分离,形成蛀孔状(似虫蛀而非虫蛀)的空腔(hollowness,or cavity)。笔者课题组前期的长期观察发现,这种现象使得空腔周围的果肉表面变得粗糙呈结晶状,同时出现木质化(lignification)褐变(化)(browning),降低了果实内在品质与商品价值。这种空腔褐变特征,可能会让消费者误认为是烂果,从而对产业造成严重的经济损失。
果肉组织的木质化过程是木质素积累的结果,受到木质素生物合成和转录调控的相关基因调控[33-34]。木质素生物合成是一个复杂的过程,其中转录因子如NAC、MYB、WRKY 和bHLH 等在调控木质素生物合成中发挥着关键作用。在这些转录因子中,NAC 和MYB 被认为是木质素生物合成调控网络的关键上游转录因子,担任主调节开关角色[37]。NAC 转录因子特别被视为植物次生壁生物合成转录调控的一级开关[35-37]。
本研究利用生物信息学手段,鉴定李PsNAC基因家族成员,并系统分析PsNAC家族基因的理化性质、基因结构、染色体定位、系统进化、亚细胞定位等特征,采用qRT-PCR技术分析PsNAC家族基因在皇冠李不同发育时期的空腔褐变果和非空腔褐变果的基因表达模式。研究结果将为进一步研究PsNAC家族基因的生物学功能、寻找可能参与木质素合成调控柰李果实空腔褐变的PsNAC 候选基因提供重要参考,并有望为未来制定柰李果实空腔褐变防控技术方案、品种改良和种质创新提供重要依据。
1 材料和方法
1.1 基因家族成员鉴定、染色体定位和理化性质分析
从Pfam 数据库(http://pfam.xfam.org/)下载NAC.hmm(PF01849)和NAM.hmm(PF02365)隐马尔结构域模型,利用TBtools 软件的Simple HMM search功能鉴定李NAC家族蛋白序列(E-value设定为1e-5)。利用NCBI 在线CD-search 工具,检测候选蛋白序列的保守结构域,剔除冗余和无效基因模型,最终获得PsNAC基因家族成员及其序列信息。
基于三月李(P. salicina Lindl.)基因组注释的GFF 文件,采用TBtools 对PsNAC 基因家族成员进行染色体定位分析,并根据它们在染色体上的位置顺序进行重命名。
利用ExPASy 网站(https://web.expasy.org/protparam/)分析PsNAC家族成员的理化性质,包括等电点、分子质量、不稳定系数、氨基酸数目等。使用SignaIP4.1(https://services.healthtech.dtu.dk/services/SignalP-4.1/)预测这些PsNAC家族成员是否存在信号肽。采用TMHMM 2.0(https://dtu.biolib.com/DeepTMHMM)预测PsNAC 家族成员的跨膜结构、利用WoLF PSORT 网站(https://www.genscript.com/wolfpsort.html)预测PsNAC家族成员的亚细胞定位。
1.2 基因家族成员多序列比对与系统进化树的构建
从PlantTFDB(http://planttfdb.gao-lab.org/)获取拟南芥NAC 基因(125 个)的蛋白序列,采用MEGA 11 软件的ClustalW 进行李和拟南芥NAC 蛋白序列比对,去除差异较大的氨基酸序列。采用Neihgbor-Joining 方法构建系统进化树(Bootstrap 设置为1000 次)。利用iTOL 网站(https://itol.embl.de/tree/)美化系统进化树。最后参考已知的拟南芥NAC 基因家族分类信息,对构建好的系统发育树进行亚族分类。
1.3 基因家族成员保守基序和基因结构分析
根据亚族分类结果,利用TBtools 的Simple MEME Wrapper 功能对PsNAC 家族的保守基序进行分析,最大motif 数设置为20,其他参数为默认值。根据生成的XML文件和家族成员ID,使用TBtools 将PsNAC 亚族成员的保守基序进行可视化绘制。根据三月李基因注释文件,使用TBtools 将PsNAC 亚族成员的编码区和非编码区的基因结构进行可视化绘制。
1.4 基因家族成员的启动子顺式作用元件预测
根据三月李基因组文件和基因结构注释信息文件,使用TBtools 筛选PsNAC 家族成员基因转录起始位点上游2000 bp 的序列,并将这些序列上传到Plant TFDB 网站,进行启动子区域顺式作用元件的预测与分析。最后,通过TBtools 的Simple BioSequence Viewer 功能,将筛选出的启动子顺式作用元件数据进行可视化展示。
1.5 基因家族成员共线性分析
从拟南芥基因组数据库(http://www.arabidopsis.org/)下载拟南芥基因组和基因注释文件,从三月李 基 因 组(https://www.rosaceae.org/Analysis/9450778)下载全基因组文件和注释文件,利用MCScanX软件对这2个物种进行共线性比较。
1.6 PsNAC基因家族成员的表达模式分析
以皇冠李不同发育时期,包括膨大期、转色期、成熟期和后熟期的果实作为试验材料,采自福建省宁德市古田县旸谷村李园(E 118°49′4″,N 26°39′3″,海拔280 m)。选取树龄为5年,株行距3.5 m×4.0 m,生长一致、长势良好,水肥管理水平一致的植株为采样树。采样时,选择树体东、南、西、北四个方向高度一致、外围花束状果枝上果形均匀一致的果实,每3株树的样本作为1 个生物学重复,用冰盒放置带回实验室。用手术刀将果实切开,判断是否为空腔褐变果实。
采用qRT-PCR 技术分析PsNAC 家族基因的相对表达量。采用RNAprep Pure 多糖多酚植物总RNA提取试剂盒(DP441,天根生化科技北京有限公司,北京)提取RNA,M5 Super plus qPCR RT kit with gDNA remover试剂盒(北京聚合美生物科技有限公司,北京)合成cDNA 第一链。使用Primer Premier 5 设计实时荧光定量引物,选用Actin 为内参基因(表1),引物由擎科伟业生物技术有限公司进行合成。qRT-PCR 反应使用LightCycler 480 SYBR Green I Master 试剂盒(Roche),通过2-ΔΔCT法计算基因的相对表达量。
表1 李PsNAC 基因家族表达分析的实时荧光定量引物
Table 1 Primers for expression analysis of plum PsNAC gene family

基因Gene PsNAC95 PsNAC77 PsNAC74 PsNAC57 PsNAC54 PsNAC22 PsNAC23 PsNAC26 PsNAC107 PsNAC51 PsActin上游引物(5'-3')Forward primer(5'-3')AGAAGCCCCAGGAAAGTTGA CATCAACAACAGAGCTCGCA AGATGAAGAGCTGGTGGACT AAATGCTGCTCGATCGTCTG AGCGAAGGAAGGTTCACACT TTCCACCACTCATCACCAGA GACGTTGTGATTTGTCGGCT GCCCCAATCTCCCAACTTTT GGGGCATATGTTGAAGCGTT TCATCCTTCGTATCGGCTAA CTGGACCTTGCTGGTCGT下游引物(5'-3')Reverse primer(5'-3')TAGAAATCAGGCCACGACGT ACATTGAGACGCACGACCT ATCCCATGGCTCGAACTTGA GCCCGGCCATTCTGATAAAA ACCCTTAACTGCTCCACCAA GTATTTTCTTGCCGGCGTCT ATTCATAATCTGCGGCCCCA GCTGTGTCTGTTGTGCTTTG AGTTGCTCGTCTCCCCATAA TCACCACCGTTGATCAAGGA ATTTCCCGCTCAGCAGTG
2 结果与分析
2.1 PsNAC基因家族成员的鉴定及理化性质特征
通过三月李NAC 转录因子特异隐马可夫模型文件的构建及对三月李蛋白数据库的二次搜索,共提取到117个PsNAC转录因子家族成员。经过NCBI CD-search 功能验证蛋白序列的保守结构域,剔除冗余和无效基因模型,最终获得了115 个PsNAC转录因子基因。根据其在染色体位置的排列顺序,依次编号为PsNAC01~PsNAC115。
对PsNAC 家族基因蛋白序列进行基本理化性质分析,如表2 所示,PsNACs 编码蛋白质的氨基酸序列长度范围是182(PsNAC98)至861(PsNAC95)个,平均氨基酸数量366 个。蛋白理论等电点值(pI)分布在4.43~9.55 之间,其中86 个为酸性蛋白,29 个为碱性蛋白。相对分子质量在20 980.52(PsNAC98)~95 967.63(PsNAC95)之间。不稳定指数范围在27.64(PsNAC60)~61.36(PsNAC093),其中,108个PsNAC家族成员不稳定指数大于35,7个PsNAC 家族成员不稳定指数则小于35。PsNAC 家族成员的GRAVY 值均为负数,说明这些PsNAC 家族成员蛋白均为亲水性蛋白。
表2 PsNAC 转录因子家族理化特征分析
Table 2 Physicochemical properties of PsNAC transcription factor family

基因编号Gene ID基因名称Gene name氨基酸数量Number of amino acids相对分子质量Molecular mass/ku等电点Theoretical pI不稳定指数Instability index亲水指数Hydrophobicity score(GRAVY)亚细胞定位Subcellar localization PsSY0028742 PsSY0026484 PsSY0007631 PsSY0017418 PsSY0022568 PsSY0014229 PsSY0021220 PsSY0023045 PsSY0024302 PsSY0025630 PsSY0008631 PsSY0017528 PsSY0022852 PsSY0010290 PsSY0004695 PsSY0006766 PsSY0017158 PsSY0011487 PsSY0013949 PsSY0013465 PsSY0012107 PsSY0013234 PsSY0025091 PsSY0006726 PsSY0014952 PsSY0024912 PsSY0020064 PsSY0026548 PsSY0017862 PsSY0018284 PsSY0027731 PsSY0005034 PsSY0000769 PsSY0003225 PsSY0026419 PsSY0022165 PsSY0024469 PsSY0006447 PsSY0015074 PsSY0001009 PsSY0017649 PsSY0001124 PsSY0007275 PsSY0014760 PsSY0008940 PsSY0015468 PsNAC1 PsNAC2 PsNAC3 PsNAC4 PsNAC5 PsNAC6 PsNAC7 PsNAC8 PsNAC9 PsNAC10 PsNAC11 PsNAC12 PsNAC13 PsNAC14 PsNAC15 PsNAC16 PsNAC17 PsNAC18 PsNAC19 PsNAC20 PsNAC21 PsNAC22 PsNAC23 PsNAC24 PsNAC25 PsNAC26 PsNAC27 PsNAC28 PsNAC29 PsNAC30 PsNAC31 PsNAC32 PsNAC33 PsNAC34 PsNAC35 PsNAC36 PsNAC37 PsNAC38 PsNAC39 PsNAC40 PsNAC41 PsNAC42 PsNAC043 PsNAC44 PsNAC45 PsNAC46 321 330 372 252 237 348 445 457 298 357 284 373 255 389 355 461 425 207 295 345 280 383 442 264 225 288 485 256 258 353 352 353 307 321 367 478 292 381 360 363 374 362 349 358 295 254 37 017.25 37 567.05 43 076.79 29 578.21 27 449.07 39 717.71 50 530.61 52 034.23 34 315.34 41 217.73 32 817.57 43 174.54 29 162.70 43 572.52 40 346.24 52 127.43 47 098.06 23 690.33 33 898.11 38 258.01 31 554.47 42 472.49 50 215.67 30 280.41 25 749.80 33 177.55 53 902.07 29 159.05 29 352.30 40 095.44 40 146.64 40 172.71 34 785.63 36 628.23 42 570.98 53 627.83 33 651.07 42 431.65 40 047.17 41 232.57 41 847.26 40 318.29 39 152.99 39 674.39 33 800.70 29 401.98 6.09 5.13 4.65 6.70 9.44 8.18 4.43 4.49 5.16 4.73 5.14 4.6 5.67 5.84 7.95 6.47 4.57 5.15 8.09 8.95 8.06 6.47 4.87 5.31 4.68 7.01 7.93 9.12 9.12 4.64 4.78 4.70 4.84 6.01 4.74 4.87 6.19 4.70 4.89 4.72 4.62 8.34 8.10 7.16 4.79 5.43 42.10 34.35 47.59 46.91 37.24 37.25 40.83 42.99 39.51 46.78 37.45 40.61 49.28 45.11 41.33 49.76 41.88 49.39 61.09 32.66 51.20 55.10 44.72 46.24 44.23 37.02 47.31 46.89 47.87 42.89 44.21 44.59 43.81 40.03 45.91 50.74 45.55 47.57 48.29 40.82 46.26 43.63 38.94 55.28 43.46 43.36-0.824-0.646-0.787-0.840-0.765-0.672-0.780-0.833-0.766-0.845-0.812-0.886-0.749-0.630-0.745-0.818-0.622-0.698-0.861-0.711-0.514-0.644-0.931-0.381-0.600-0.692-0.655-0.707-0.695-0.754-0.774-0.746-0.783-0.998-0.956-0.746-0.749-0.714-0.674-0.817-0.610-0.746-0.598-0.622-0.715-0.810过氧化物酶体Peroxisome叶绿体Chloroplast细胞核Nucleus细胞核Nucleus线粒体Mitochondria细胞核Nucleus细胞核Nucleus细胞核Nucleus叶绿体Chloroplast细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus液泡Vacuole细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞骨架Cytoskeleton细胞核Nucleus细胞核Nucleus细胞质Cytoplasm细胞质Cytoplasm细胞骨架Cytoskeleton细胞核Nucleus细胞质Cytoplasm细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus叶绿体Chloroplast细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞质Cytoplasm细胞质Cytoplasm
表2 (续) Table 2 (Continued)

基因编号Gene ID基因名称Gene name氨基酸数量Number of amino acids相对分子质量Molecular mass/ku等电点Theoretical pI不稳定指数Instability index亲水指数Hydrophobicity score(GRAVY)亚细胞定位Subcellar localization PsSY0012082 PsSY0019957 PsSY0009323 PsSY0016103 PsSY0002281 PsSY0016233 PsSY0024290 PsSY0018074 PsSY0001699 PsSY0014297 PsSY0021550 PsSY0019581 PsSY0013638 PsSY0001933 PsSY0002320 PsSY0017909 PsSY0006858 PsSY0018753 PsSY0005654 PsSY0006576 PsSY0007670 PsSY0022170 PsSY0025975 PsSY0020086 PsSY0023204 PsSY0024339 PsSY0027895 PsSY0012520 PsSY0022316 PsSY0026601 PsSY0012725 PsSY0004600 PsSY0003387 PsSY0028882 PsSY0021976 PsSY0021124 PsSY0024583 PsSY0000423 PsSY0016599 PsSY0010260 PsSY0010208 PsSY0010297 PsSY0027682 PsSY0002886 PsSY0015905 PsSY0021606 PsSY0010930 PsNAC47 PsNAC48 PsNAC49 PsNAC50 PsNAC51 PsNAC52 PsNAC53 PsNAC54 PsNAC55 PsNAC56 PsNAC57 PsNAC58 PsNAC59 PsNAC60 PsNAC61 PsNAC62 PsNAC63 PsNAC64 PsNAC65 PsNAC66 PsNAC67 PsNAC68 PsNAC69 PsNAC70 PsNAC71 PsNAC72 PsNAC73 PsNAC74 PsNAC75 PsNAC76 PsNAC77 PsNAC78 PsNAC79 PsNAC80 PsNAC81 PsNAC82 PsNAC83 PsNAC84 PsNAC85 PsNAC86 PsNAC87 PsNAC88 PsNAC89 PsNAC90 PsNAC91 PsNAC92 PsNAC93 253 321 324 354 375 482 302 317 368 237 389 394 352 305 374 681 403 242 322 449 505 456 467 453 451 380 504 191 448 437 213 185 184 539 284 290 284 271 577 306 422 397 428 378 438 344 362 29 574.07 36 506.42 36 467.39 40 368.58 42 546.59 56 167.15 34 734.30 35 812.27 40 532.08 27 190.66 42 781.55 43 936.15 40 169.07 35 315.07 41 854.15 77 211.69 46 055.38 27 611.42 35 696.89 50 804.13 56 058.04 51 255.43 52 846.77 51 139.41 52 028.34 44 037.27 58 318.07 22 513.61 52 203.43 49 531.24 25 252.89 22 033.89 22 018.89 60 419.46 32 847.21 33 751.64 32 704.57 30 898.07 65 388.75 35 495.61 47 309.30 44 406.02 47 751.40 42 887.64 49 498.64 39 180.21 42 246.97 5.31 4.49 8.32 4.74 6.94 8.27 8.82 8.47 7.92 9.55 5.24 6.54 6.56 8.54 4.56 5.51 6.47 8.75 5.05 4.66 4.91 4.44 4.70 5.47 4.80 4.70 6.08 7.81 6.91 5.82 6.55 5.35 8.41 5.03 5.64 7.72 5.67 8.66 4.90 6.17 5.87 5.43 4.53 7.71 6.38 5.34 6.39 48.76 44.21 55.55 40.57 36.61 56.80 34.40 50.56 42.18 53.04 40.04 42.55 49.74 27.64 36.39 37.09 51.13 48.58 51.35 55.90 60.18 55.28 57.29 55.29 55.23 58.91 50.45 38.80 43.60 40.10 56.24 58.82 44.04 41.11 42.51 46.84 53.47 39.66 48.17 56.37 44.12 35.03 35.92 37.86 35.80 38.01 61.36-0.868-0.768-0.600-0.799-0.682-0.492-0.719-0.876-0.714-0.832-0.597-0.654-0.717-0.801-0.638-0.537-0.736-0.558-0.634-0.776-0.754-0.717-0.754-0.777-0.896-0.941-0.827-0.583-0.852-0.720-0.730-0.875-0.895-0.545-0.674-0.668-0.811-0.555-0.594-0.806-0.448-0.479-0.411-0.948-0.639-0.820-0.932细胞核Nucleus细胞质Cytoplasm细胞核Nucleus细胞核Nucleus细胞核Nucleus过氧化物酶体Peroxisome细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus线粒体Mitochondria细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus质膜Plasma membrane细胞核Nucleus细胞骨架Cytoskeleton细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus叶绿体Chloroplast细胞骨架Cytoskeleton高尔基体Golgi apparatus细胞核Nucleus细胞骨架Cytoskeleton细胞骨架Cytoskeleton细胞核Nucleus叶绿体Chloroplast叶绿体Chloroplast细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus线粒体Mitochondria叶绿体Chloroplast细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus
表2 (续) Table 2 (Continued)

基因编号Gene ID基因名称Gene name氨基酸数量Number of amino acids相对分子质量Molecular mass/ku等电点Theoretical pI不稳定指数Instability index亲水指数Hydrophobicity score(GRAVY)亚细胞定位Subcellar localization PsSY0013892 PsSY0023673 PsSY0003939 PsSY0024811 PsSY0002811 PsSY0027226 PsSY0021398 PsSY0026579 PsSY0019871 PsSY0003865 PsSY0002691 PsSY0010897 PsSY0010774 PsSY0001641 PsSY0027282 PsSY0009363 PsSY0021588 PsSY0018873 PsSY0006710 PsSY0006233 PsSY0026633 PsSY0012482 PsNAC94 PsNAC95 PsNAC96 PsNAC97 PsNAC98 PsNAC99 PsNAC100 PsNAC101 PsNAC102 PsNAC103 PsNAC104 PsNAC105 PsNAC106 PsNAC107 PsNAC108 PsNAC109 PsNAC110 PsNAC111 PsNAC112 PsNAC113 PsNAC114 PsNAC115 395 861 233 363 182 353 261 457 383 459 383 418 485 590 632 425 316 423 376 285 335 486 45 510.50 95 967.63 26 331.53 40 626.78 20 980.52 39 619.25 29 885.22 50 894.25 42 830.21 51 077.45 42 796.20 47 376.36 54 152.06 66 114.13 68 967.68 47 698.99 36 132.78 48 680.55 43 058.75 32 107.54 38 645.14 54 722.66 5.94 5.02 8.33 6.63 6.80 6.96 5.07 6.03 7.75 6.03 7.75 4.80 4.79 4.53 5.71 4.92 5.92 6.01 6.75 6.51 5.00 6.63 54.35 49.05 57.01 41.98 46.42 47.97 48.98 35.99 44.28 32.58 43.96 38.19 55.42 28.26 32.72 53.79 47.47 40.90 42.69 59.94 57.01 53.27-0.880-0.839-0.664-0.585-0.696-0.692-0.827-0.786-0.587-0.777-0.584-0.508-0.745-0.573-0.789-0.716-0.933-1.009-0.087-0.856-0.782-0.979细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞质Cytoplasm细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus高尔基体Golgi apparatus细胞核Nucleus叶绿体Chloropast细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus质膜Plasma membrane细胞核Nucleus细胞核Nucleus细胞核Nucleus
跨膜结构分析显示,94%的PsNAC 家族成员没有跨膜结构域,而PsNAC14、PsNAC80、PsNAC85、PsNAC95、PsNAC105 和PsNAC107 则各含有1 个跨膜结构域,仅PsNAC112 具有3 个跨膜结构域。亚细胞定位的预测结果显示,91 个PsNAC 家族成员位于细胞核,其余分布于细胞质、高尔基体、过氧化物酶体、细胞骨架、线粒体、叶绿体、质膜和液泡等结构中。
2.2 PsNAC基因染色体定位
染色体定位结果(图1)显示,115 个PsNAC 基因不均匀地定位到8 对染色体上,其中,第2 号和3号染色体上分布的PsNAC 家族成员最多(占17.4%),其次是第5 号染色体上(占15.7%),第6 号和7号染色体上分布的基因数量最少,仅含有10个PsNACs。
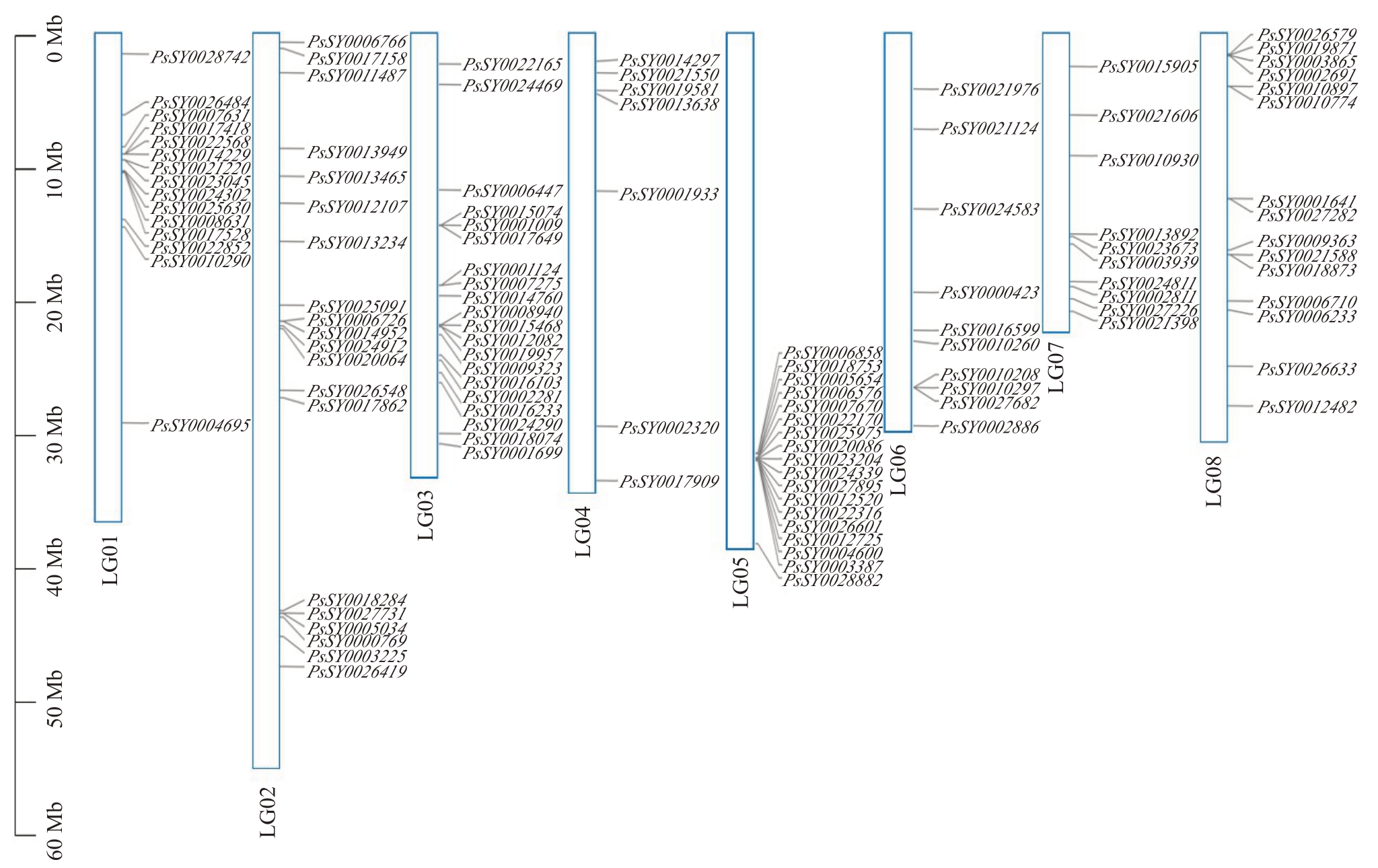
图1 李PsNAC 家族成员在染色体上的位置分布
Fig.1 The location distribution of PsNAC gene family members across the chromosomes of Prunus salicina Lindl.
2.3 PsNAC多序列比对与系统发育树构建
利用拟南芥NAC 基因序列作为对照,可将PsNACs分为17个亚族,不同亚族在功能和进化关系上存在差异。OsNAC8和ANC063亚族不含PsNAC家族成员,其余亚族均含有数量不等的PsNAC家族成员。NAC-C和TIP亚族的PsNAC家族成员数量超过20个,其中NAC-C亚族包含PsNAC家族成员数量最多,为26个。OsNAC003亚族含6个PsNAC家族成员,分别是PsNAC22、PsNAC23、PsNAC54、PsNAC56、PsNAC57和PsNAC115;OsNAC7亚族含10个PsNAC家族成员,分别是PsNAC1、PsNAC51、PsNAC63、PsNAC64、PsNAC84、PsNAC86、PsNAC90、PsNAC93、PsNAC94和PsNAC114(图2)。

图2 李PsNAC 和拟南芥AtNAC 基因家族系统进化树
Fig.2 Phylogenetic tree of NAC gene family in Prunus salicina Lindl.and Arabidopsis thaliana
2.4 PsNAC家族成员保守基序和基因结构
PsNAC 基因的CDS 片段数量在1~8 个不等,多数PsNAC 基因具有3~6 个CDS 片段。基于基因注释文件的分析,共识别出10个保守motifs,它们的分布位置和数量在PsNAC 基因中并不完全一致。其中,motif 1、motif 2、motif 3 和motif 6 出现的次数最多,且通常是依次分布在N 端。同一个亚族的PsNAC成员具有相似的motif分布和基因结构(包括CDS 和UTR 区域),这说明每个亚族的PsNAC 成员可能具有类似的功能。PsNAC33 和PsNAC95 所具有的motif数量最少,仅有3个(图3)。

图3 PsNAC 家族基因的保守基序(A)和基因结构(B)
Fig.3 Schematic diagram of conserved motif elements(A)and gene structure characteristics(B)of PsNACs
2.5 PsNAC家族成员启动子顺式作用元件
为便于预测PsNAC基因所具有的潜在功能,对PsNAC基因起始密码子ATG上游2000 bp的序列进行分析,共预测到3069个功能不同的顺式作用元件(图4)。其中,最主要的响应元件是植物激素响应元件,包括茉莉酸甲酯响应元件(MeJA-responsive element,320 个)、脱落酸响应元件(abscisic acid-responsive element,296 个)、赤霉素响应元件(gibberellin-responsive element,127 个)和生长素响应元件(auxin-responsive element,78 个)。此外,在非生物胁迫中,PsNAC 家族还含有87、64 和49 个响应元件,分别参与干旱诱导反应的MYB 结合位点(MYB binding site involved in drought-inducibility)、参与低温(Cis-acting element involved in low-temperature responsiveness)和光(MYB binding site involved in light responsiveness)响应。
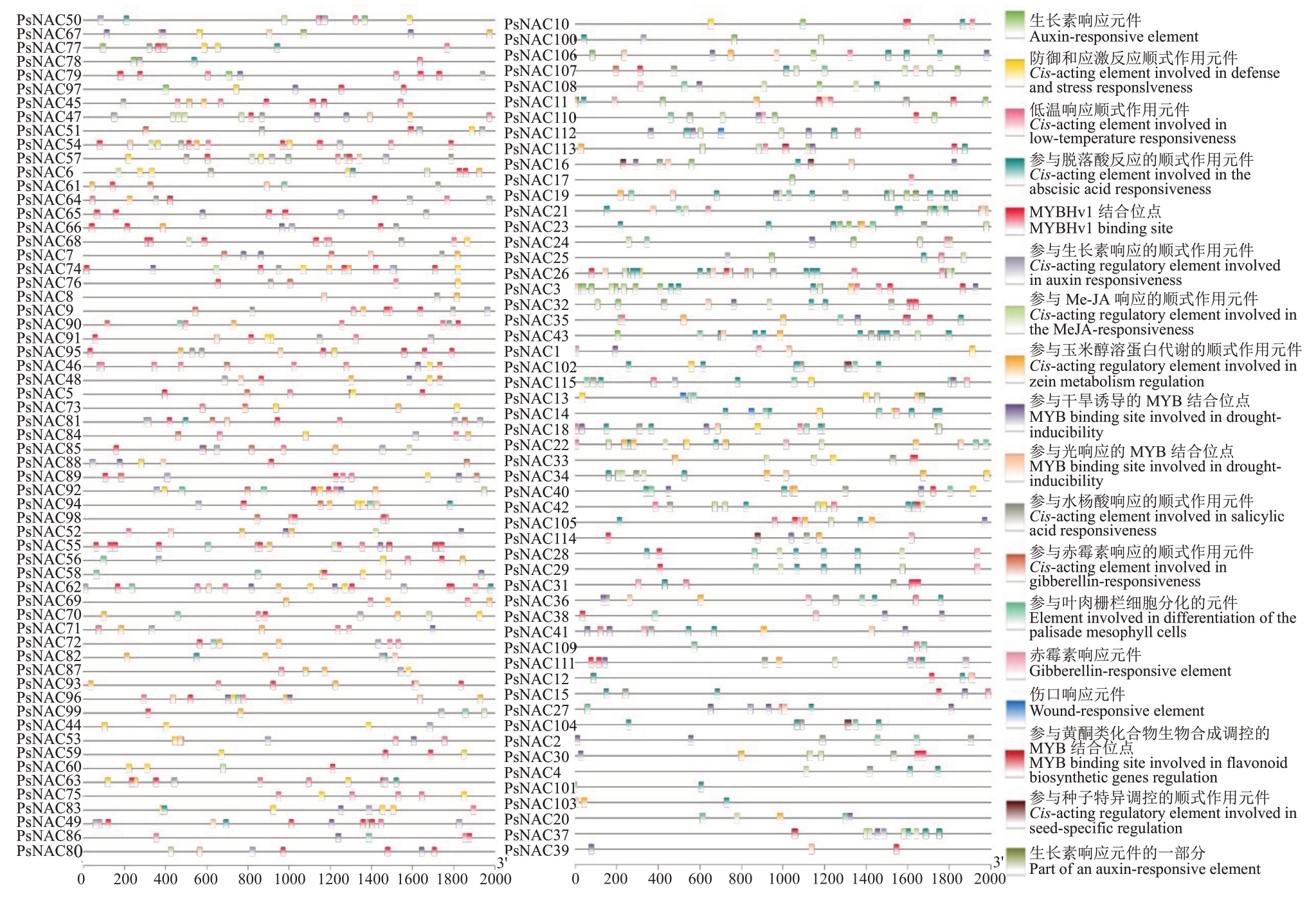
图4 PsNAC 基因启动子区域的顺式作用元件
Fig.4 Cis-acting elements in promoter region of PsNAC genes
2.6 PsNAC基因家族种内共线性
为了鉴定PsNAC 基因家族内是否存在基因复制现象,通过MCScanX共线性分析,总共鉴定出16对片段重复基因复制事件(图5,红线相连的是基因复制事件的基因对),片段重复是PsNAC基因家族进化重要事件。其中,PsNAC37与PsNAC19、PsNAC29与PsNAC56、PsNAC28 与PsNAC96、PsNAC26 与PsNAC58、PsNAC96 与 PsNAC56、PsNAC80 与PsNAC94、PsNAC93 与 PsNAC1、PsNAC94 与PsNAC71 发生的是两两基因片段复制,PsNAC97、PsNAC59 与 PsNAC58,PsNAC79、PsNAC94 与PsNAC95,PsNAC29、PsNAC28与PsNAC96发生的是3个基因之间的基因片段复制。PsNAC13、PsNAC14、PsNAC113与PsNAC114发生的是4个基因之间的基因片段复制。根据片段重复的基因结构分析和系统发育树构建,推测这些片段重复基因的功能可能存在相似性。
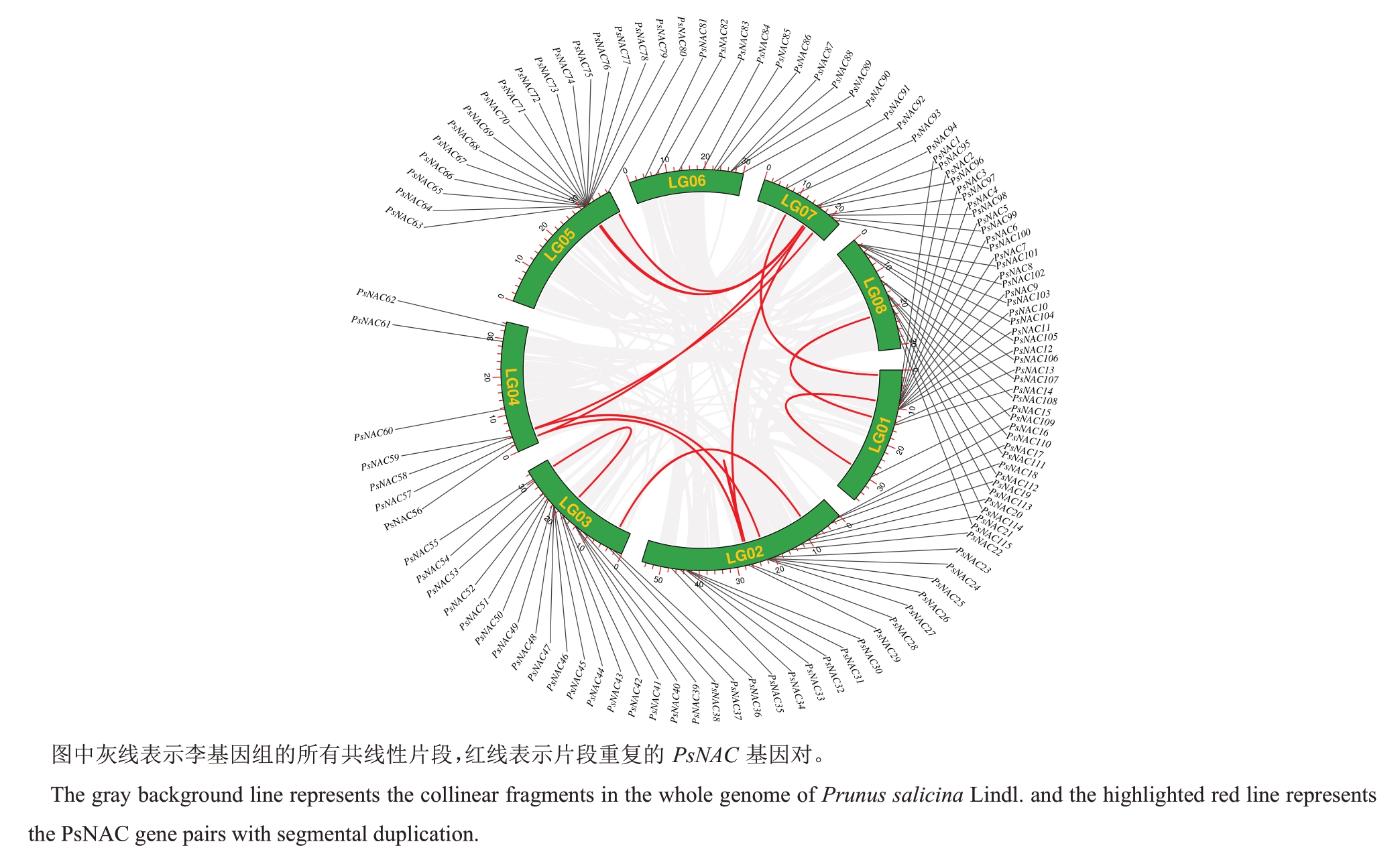
图5 PsNAC 基因的共线性分析
Fig.5 The collinearity analysis of PsNAC genes
2.7 PsNAC 基因家族在空腔褐变皇冠李果实中的表达模式分析
植物次生细胞壁加厚过程包含纤维素、半纤维素和木质素的合成,同时结合PsNAC 基因家族成员的顺式作用元件预测结果,对10 个PsNAC 基因家族成员的基因表达量进行了分析(图6)。其中,PsNAC57、PsNAC54 和PsNAC51 既是OsNAC7 和OsNAC003 的亚族成员,又同时具有生长素和赤霉素响应元件,在激素参与木质素生物合成的转录调控网络中具有重要作用。PsNAC26、PsNAC57、PsNAC77 和PsNAC95 在果实膨大期的基因相对表达量最高,随果实发育逐渐降低,在空腔褐变果实中的表达量高于非空腔褐变果。PsNAC54和PsNAC74在果实膨大期的表达量最低,随果实发育逐渐上升,在空腔褐变果实中的表达量高于非空腔褐变果。PsNAC22 和PsNAC23 在果实膨大期基因相对表达量最高,随果实发育呈现先下降后上升的趋势。PsNAC51 基因相对表达量随果实发育呈现先下降后上升的波动变化,后熟期的相对表达量最低,而PsNAC107在各时期的相对表达量基本保持不变。
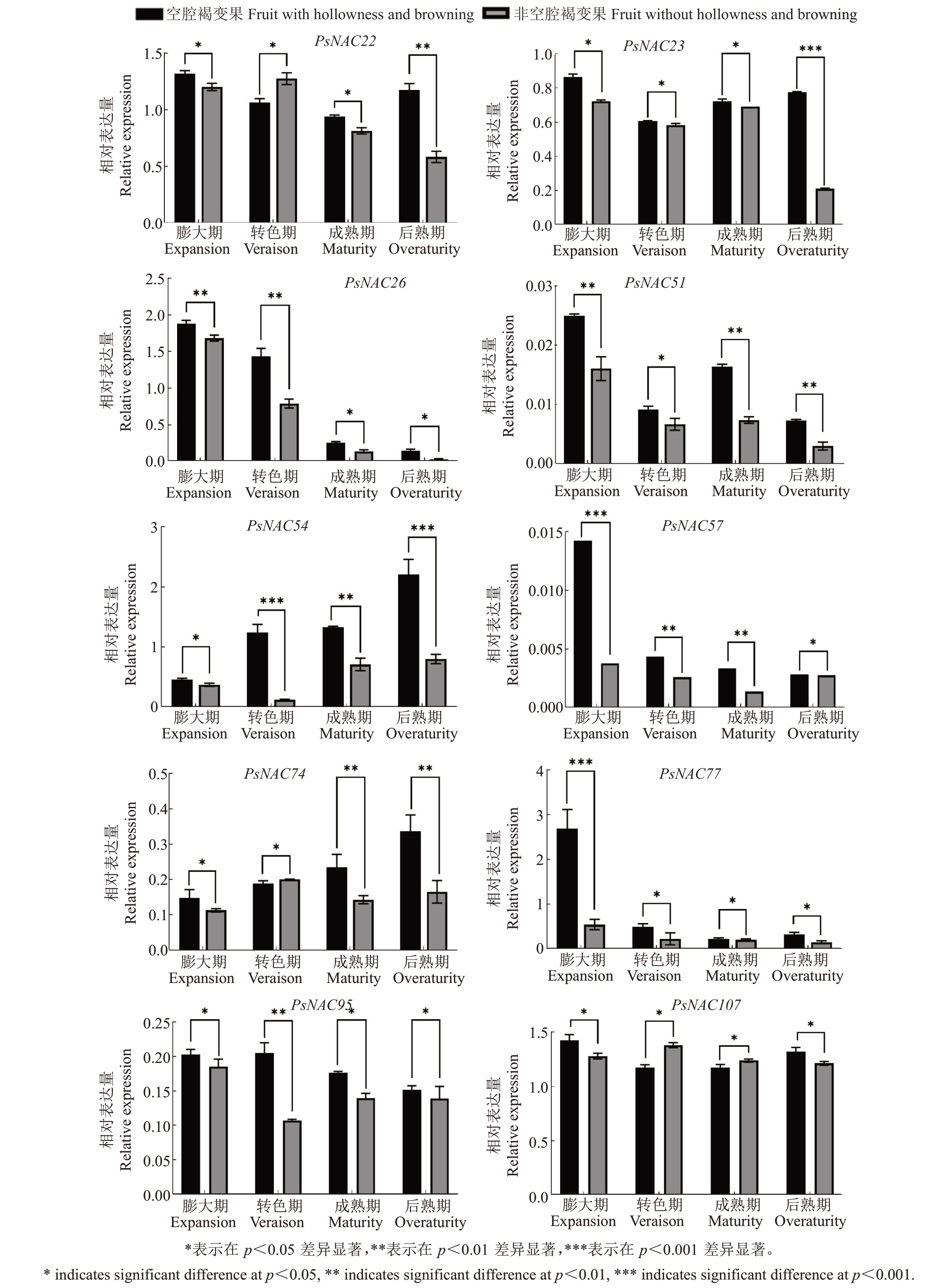
图6 PsNAC 基因在皇冠李空腔褐变和非空腔褐变果中不同发育时期的表达水平
Fig.6 Expression analysis of PsNAC genes in Huangguan fruit with and without hollowness and browning during different development stages
3 讨 论
NAC 转录因子家族是植物中最大的转录因子家族之一,在调节植物的生长发育、抵御病原菌、响应非生物胁迫(如干旱、高盐和低温等)以及调控果实品质等方面发挥着重要作用。尽管对NAC 基因家族的鉴定和功能分类在多种植物中已有广泛的研究,但NAC转录因子在李的系统性研究方面尚不充分。木质素与果实品质和风味密切相关,其过度积累可能导致园艺植物的果实口感变差、果肉变硬、风味变淡、颜色变褐加深等,致使感官特性劣变、商品价值降低[38-39]。笔者课题组前期观察发现,皇冠李果实空腔褐变这一产业问题与木质素密切相关。作为木质素生物合成途径的上游调控因子[35-37],NAC转录因子在木质素生物合成的转录调控方面发挥关键作用。因此,对PsNAC转录因子进行全面生物信息学鉴定和分析,有望为未来制定防控柰李果实空腔褐变的技术方案、品种改良和种质创新提供重要参考。
笔者在本研究中鉴定到115个PsNAC转录因子成员,该数量多于番茄(93个)[9]、辣椒(104个)[10]、火龙果(64个)[14]、菠萝(73个)[15]和欧李(76个)[16],但低于拟南芥(117 个)[5]、毛果杨(170 个)[6]、水稻(151个)[5]、玉米(148 个)[7]、大豆(152 个)[8]、大白菜(188个)[11]、苹果(180个)[12]和梨(185个)[13]。与其他物种相比,李PsNAC 基因家族成员数量存在一定的差异,显示出明显的物种间的差异性。
笔者在本研究中的蛋白理化性质分析结果显示,李PsNAC 基因家族成员在氨基酸序列长度、相对分子质量、等电点、CDS长度等存在明显差异,表明了PsNAC 基因家族成员在结构和功能上的多样性。尽管PsNAC蛋白呈现亲水性特征,但其稳定性存在较大变异,反映了它们在不同生物学环境中的适应性和功能多样性。亚细胞定位预测结果显示,PsNAC家族成员主要定位于细胞核中,也有少数位于细胞质、叶绿体、线粒体等中,说明李PsNAC转录因子可能在不同细胞部位发挥着多样性的功能,从而为植物体的正常生命活动提供保障。
以拟南芥NAC 基因序列作为对照,将PsNACs基因分为17个亚族。其中,6个PsNAC基因聚集在OsNAC003 亚族,10 个PsNACs 聚集在OsNAC7 亚族。前人研究表明,OsNAC003和OsNAC7亚族的大部分基因参与植物的次生细胞壁合成[40]。因此,推测聚类在这2 个亚族的PsNACs 可能具有相似的功能。OsNAC7 亚族在NAC 家族中被广泛研究,包括SND1、NST1、URP7、BRN1/2、VND1-7 基因,其主要功能集中在调控茎、根和花药次生壁的形成,在木质素调控植物生长发育和逆境胁迫中起重要作用[41-42]。
启动子顺式作用元件的预测可以为进一步研究基因的转录调控机制和潜在功能提供理论依据[43]。本研究中启动子顺式作用元件分析结果表明,PsNACs含大量与激素相关元件的成员,推测这些成员在激素调控木质素生物合成的转录调控网络中发挥重要作用。研究表明,NAC转录因子在植物次生细胞壁合成的转录调控网络起关键作用,是该复杂调控网络的主要开关。枇杷EjNAC1 通过反式激活木质素合成相关基因EjPAL1 和Ej4CL1 的启动子,使其表达上调,从而导致果实木质素积累,呈现出明显的木质化特征[33]。Ge 等[34]发现,EjNAC3 蛋白能直接结合并激活木质素合成结构基因EjCAD-like的启动子,参与调控枇杷果实木质化过程。三红蜜柚CgNAC043 能激活下游转录因子CgMYB46 和木质素合成基因CgC3H 和CgCCoAOMT1 的启动子,共同参与蜜柚汁胞木质素合成的转录调控[44]。冬枣MYB 激 活 子(LOC107425254)和 抑 制 子(LOC107415078)通过调节F5H和CCR参木质素生物合成,而NAC 转录因子(LOC107435239)则促进F5H 的表达,从而正向调控木质素的积累[45]。石榴NAC类蛋白PgSND1-like能结合木质素合成酶基因启动子特异元件,提高PAL、4CL、F4H、CCR 和CAD表达水平,从而调控石榴籽粒的硬度[46]。
笔者在本研究中结合PsNAC 基因家族成员顺式作用元件预测,共鉴定到10个PsNAC基因家族成员,其中PsNAC57、PsNAC54 和PsNAC51 既是Os-NAC7和OsNAC003的亚族成员,又同时具有生长素和赤霉素响应元件。因此,这3 个基因在激素参与木质素生物合成的转录调控网络中至关重要。PsNAC26、PsNAC57、PsNAC77 和PsNAC95 在果实膨大期表达量最高,随果实发育逐渐降低,在空腔褐变果中的表达量高于非空腔褐变果,推测这些家族成员可能负向调控木质素生物合成。PsNAC54 和PsNAC74 在果实膨大期的表达量最低,随果实发育逐渐上升,在空腔褐变果中的表达量高于非空腔褐变果,PsNAC54和PsNAC74可能正向调控木质素生物合成。本研究为后续研究皇冠李空腔褐变的分子机制提供了重要靶标。
4 结 论
首次鉴定并分析了115个李PsNAC基因家族成员,分析了PsNAC家族基因的理化性质、基因结构、染色体定位、系统进化、亚细胞定位等特征,采用qRT-PCR 技术分析PsNAC 家族基因在皇冠李不同发育时期的空腔褐变果和非空腔褐变果的基因表达模式。研究结果将为进一步研究PsNAC 家族基因的生物学功能、PsNAC家族成员与皇冠李果实空腔褐变的具体关联性奠定了重要基础。
[1] LIU G S,LI H L,GRIERSON D,FU D Q.NAC transcription factor family regulation of fruit ripening and quality:A review[J].Cells,2022,11(3):525.
[2] SINGH S,KOYAMA H,BHATI K K,ALOK A.The biotechnological importance of the plant-specific NAC transcription factor family in crop improvement[J]. Journal of Plant Research,2021,134(3):475-495.
[3] AIDA M,ISHIDA T,FUKAKI H,FUJISAWA H,TASAKA M.Genes involved in organ separation in Arabidopsis:An analysis of the cup-shaped Cotyledon mutant[J]. The Plant Cell,1997,9(6):841-857.
[4] PURANIK S,SAHU P P,SRIVASTAVA P S,PRASAD M.NAC proteins:Regulation and role in stress tolerance[J].Trends in Plant Science,2012,17(6):369-381.
[5] NURUZZAMAN M,MANIMEKALAI R,SHARONI A M,SATOH K,KONDOH H,OOKA H,KIKUCHI S. Genome-wide analysis of NAC transcription factor family in rice[J]. Gene,2010,465(1/2):30-44.
[6] MENG L,CHEN S Y,LI D W,HUANG M R,ZHU S.Genomewide characterization and evolutionary expansion of poplar NAC transcription factors and their tissue-specific expression profiles under drought[J].International Journal of Molecular Sciences,2022,24(1):253.
[7] PENG X J,ZHAO Y,LI X M,WU M,CHAI W B,SHENG L,WANG Y,DONG Q,JIANG H Y,CHENG B J. Genomewide identification,classification and analysis of NAC type gene family in maize[J].Journal of Genetics,2015,94(3):377-390.
[8] LE D T,NISHIYAMA R,WATANABE Y,MOCHIDA K,YAMAGUCHI-SHINOZAKI K,SHINOZAKI K,TRAN L S P.Genome-wide survey and expression analysis of the plant-specific NAC transcription factor family in soybean during development and dehydration stress[J]. DNA Research,2011,18(4):263-276.
[9] JIN J F,WANG Z Q,HE Q Y,WANG J Y,LI P F,XU J M,ZHENG S J,FAN W,YANG J L. Genome-wide identification and expression analysis of the NAC transcription factor family in tomato (Solanum lycopersicum) during aluminum stress[J].BMC Genomics,2020,21(1):288.
[10] DIAO W P,SNYDER J C,WANG S B,LIU J B,PAN B G,GUO G J,GE W,DAWOOD M H S A. Genome-wide analyses of the NAC transcription factor gene family in pepper (Capsicum annuum L.):Chromosome location,phylogeny,structure,expression patterns,Cis-elements in the promoter,and interaction network[J]. International Journal of Molecular Sciences,2018,19(4):1028.
[11] MA J,WANG F,LI M Y,JIANG Q,TAN G F,XIONG A S.Genome wide analysis of the NAC transcription factor family in Chinese cabbage to elucidate responses to temperature stress[J].Scientia Horticulturae,2014,165:82-90.
[12] SU H Y,ZHANG S Z,YUAN X W,CHEN C T,WANG X F,HAO Y J. Genome-wide analysis and identification of stress-responsive genes of the NAM-ATAF1 2-CUC2 transcription factor family in apple[J]. Plant Physiology and Biochemistry,2013,71:11-21.
[13] AHMAD M,YAN X H,LI J Z,YANG Q S,JAMIL W,TENG Y W,BAI S L. Genome wide identification and predicted functional analyses of NAC transcription factors in Asian pears[J].BMC Plant Biology,2018,18(1):214.
[14] HU X L,XIE F F,LIANG W W,LIANG Y H,ZHANG Z K,ZHAO J T,HU G B,QIN Y H. HuNAC20 and HuNAC25,two novel NAC genes from pitaya,confer cold tolerance in transgenic Arabidopsis[J]. International Journal of Molecular Sciences,2022,23(4):2189.
[15] HE Q,LIU Y H,ZHANG M,BAI M Y,PRIYADARSHANI S V G N,CHAI M N,CHEN F Q,HUANG Y M,LIU L P,CAI H Y,QIN Y. Genome-wide identification and expression analysis of the NAC transcription factor family in pineapple[J]. Tropical Plant Biology,2019,12(4):255-267.
[16] 张忠鑫,郭夕雯,汪泽文,王鹏飞,张建成,杜俊杰,穆霄鹏.欧李NAC 基因家族的鉴定及表达分析[J]. 果树学报,2023,40(2):206-222.ZHANG Zhongxin,GUO Xiwen,WANG Zewen,WANG Pengfei,ZHANG Jiancheng,DU Junjie,MU Xiaopeng. Identification and expression analysis of NAC gene family in Cerasus humilis[J].Journal of Fruit Science,2023,40(2):206-222.
[17] NURUZZAMAN M,SHARONI A M,KIKUCHI S. Roles of NAC transcription factors in the regulation of biotic and abiotic stress responses in plants[J].Frontiers in Microbiology,2013,4:248.
[18] HOU X M,ZHANG H F,LIU S Y,WANG X K,ZHANG Y M,MENG Y C,LUO D,CHEN R G.The NAC transcription factor CaNAC064 is a regulator of cold stress tolerance in peppers[J].Plant Science,2020,291:110346.
[19] XU Y,LI P F,MA F N,HUANG D M,XING W T,WU B,SUN P G,XU B Q,SONG S.Characterization of the NAC transcription factor in passion fruit (Passiflora edulis) and functional identification of PeNAC-19 in cold stress[J]. Plants,2023,12(6):1393.
[20] HE X J,MU R L,CAO W H,ZHANG Z G,ZHANG J S,CHEN S Y.AtNAC2,a transcription factor downstream of ethylene and auxin signaling pathways,is involved in salt stress response and lateral root development[J]. Plant Journal,2005,44(6):903-916.
[21] SABLOWSKI R W M,MEYEROWITZ E M.A homolog of NO APICAL MERISTEM is an immediate target of the floral homeotic genes APETALA3/PISTILLATA[J].Cell,1998,92(1):93-103.
[22] BERGER Y,HARPAZ-SAAD S,BRAND A,MELNIK H,SIRDING N,ALVAREZ J P,ZINDER M,SAMACH A,ESHED Y,ORI N. The NAC-domain transcription factor GOBLET specifies leaflet boundaries in compound tomato leaves[J].Development,2009,136(5):823-832.
[23] KATO H,MOTOMURA T,KOMEDA Y,SAITO T,KATO A. Overexpression of the NAC transcription factor family gene ANAC036 results in a dwarf phenotype in Arabidopsis thaliana[J].Journal of Plant Physiology,2010,167(7):571-577.
[24] KO J H,YANG S H,PARK A H,LEROUXEL O,HAN K H.ANAC012,a member of the plant-specific NAC transcription factor family,negatively regulates xylary fiber development in Arabidopsis thaliana[J].Plant Journal,2007,50(6):1035-1048.
[25] NAKANO Y,YAMAGUCHI M,ENDO H,REJAB N A,OHTANI M.NAC-MYB-based transcriptional regulation of secondary cell wall biosynthesis in land plants[J].Frontiers in Plant Science,2015,6:288.
[26] SOUER E,VAN HOUWELINGEN A,KLOOS D,MOL J,KOES R. The No apical meristem gene of Petunia is required for pattern formation in embryos and flowers and is expressed at meristem and primordia boundaries[J]. Cell,1996,85(2):159-170.
[27] XIE Q,FRUGIS G,COLGAN D,CHUA N H. Arabidopsis NAC1 transduces auxin signal downstream of TIR1 to promote lateral root development[J]. Genes & Development,2000,14(23):3024-3036.
[28] PODZIMSKA-SROKA D,O’SHEA C,GREGERSEN P L,SKRIVER K. NAC transcription factors in senescence:From molecular structure to function in crops[J]. Plants,2015,4(3):412-448.
[29] FORLANI S,MIZZOTTI C,MASIERO S.The NAC side of the fruit:Tuning of fruit development and maturation[J]. BMC Plant Biology,2021,21(1):238.
[30] CAO X M,WEI C Y,DUAN W Y,GAO Y,KUANG J F,LIU M C,CHEN K S,KLEE H,ZHANG B.Transcriptional and epigenetic analysis reveals that NAC transcription factors regulate fruit flavor ester biosynthesis[J]. Plant Journal,2021,106(3):785-800.
[31] FU B L,WANG W Q,LI X,QI T H,SHEN Q F,LI K F,LIU X F,LI S J,ALLAN A C,YIN X R.A dramatic decline in fruit citrate induced by mutagenesis of a NAC transcription factor,AcNAC1[J]. Plant Biotechnology Journal,2023,21(8):1695-1706.
[32] 卓茂根,王惠聪.NAC 转录因子在果实成熟中的调控作用[J].果树学报,2023,40(7):1455-1470.ZHUO Maogen,WANG Huicong. The roles of NAC transcription factors in regulating fruit ripening[J]. Journal of Fruit Science,2023,40(7):1455-1470.
[33] XU Q,WANG W Q,ZENG J K,ZHANG J,GRIERSON D,LI X,YIN X R,CHEN K S.A NAC transcription factor,EjNAC1,affects lignification of loquat fruit by regulating lignin[J]. Postharvest Biology and Technology,2015,102:25-31.
[34] GE H,ZHANG J,ZHANG Y J,LI X,YIN X R,GRIERSON D,CHEN K S. EjNAC3 transcriptionally regulates chilling- induced lignification of loquat fruit via physical interaction with an atypical CAD-like gene[J]. Journal of Experimental Botany,2017,68(18):5129-5136.
[35] 郭光艳,柏峰,刘伟,秘彩莉.转录因子对木质素生物合成调控的研究进展[J].中国农业科学,2015,48(7):1277-1287.GUO Guangyan,BAI Feng,LIU Wei,BI Caili.Advances in research of the regulation of transcription factors of lignin biosynthesis[J].Scientia Agricultura Sinica,2015,48(7):1277-1287.
[36] 张雨,赵明洁,张蔚.植物次生细胞壁生物合成的转录调控网络[J].植物学报,2020,55(3):351-368.ZHANG Yu,ZHAO Mingjie,ZHANG Wei.Transcriptional regulatory network of secondary cell wall biosynthesis in plants[J].Chinese Bulletin of Botany,2020,55(3):351-368.
[37] 陈倩,游双梅,邢乐华,徐凡,罗明,郭启高.果树NAC 转录因子的研究进展[J].分子植物育种,2021,19(19):6396-6405.CHEN Qian,YOU Shuangmei,XING Lehua,XU Fan,LUO Ming,GUO Qigao.Research progress of NAC transcription factors in fruit trees[J]. Molecular Plant Breeding,2021,19(19):6396-6405.
[38] 薛维文,周显芳,张昭其,方方.果蔬采后木质素积累及其调控对品质的影响研究进展[J].园艺学报,2022,49(9):2023-2036.XUE Weiwen,ZHOU Xianfang,ZHANG Zhaoqi,FANG Fang.Advances in lignin accumulation and its regulation on the quality of postharvest fruit and vegetables[J].Acta Horticulturae Sinica,2022,49(9):2023-2036.
[39] SHI Y N,LI B J,SU G Q,ZHANG M X,GRIERSON D,CHEN K S.Transcriptional regulation of fleshy fruit texture[J]. Journal of Integrative Plant Biology,2022,64(9):1649-1672.
[40] HUSSEY S G,MIZRACHI E,SPOKEVICIUS A V,BOSSINGER G,BERGER D K,MYBURG A A. SND2 a NAC transcription factor gene,regulates genes involved in secondary cell wall development in Arabidopsis fibres and increases fibre cell area in Eucalyptus[J].BMC Plant Biology,2011,11:173.
[41] MITSUDA N,SEKI M,SHINOZAKI K,OHME-TAKAGI M.The NAC transcription factors NST1 and NST2 of Arabidopsis regulate secondary wall thickenings and are required for anther dehiscence[J].The Plant Cell,2005,17(11):2993-3006.
[42] ZHONG R Q,DEMURA T,YE Z H. SND1,a NAC domain transcription factor,is a key regulator of secondary wall synthesis in fibers of Arabidopsis[J]. The Plant Cell,2006,18(11):3158-3170.
[43] HERNANDEZ-GARCIA C M,FINER J J. Identification and validation of promoters and Cis-acting regulatory elements[J].Plant Science,2014,217:109-119.
[44] LI X T,WANG N Y,SHE W Q,GUO Z X,PAN H L,YU Y,YE J W,PAN D M,PAN T F.Identification and functional analysis of the CgNAC043 gene involved in lignin synthesis from Citrus grandis‘San Hong’[J].Plants,2022,11(3):403.
[45] ZHANG Q,WANG L H,WANG Z T,ZHANG R T,LIU P,LIU M J,LIU Z G,ZHAO Z H,WANG L L,CHEN X,XU H F.The regulation of cell wall lignification and lignin biosynthesis during pigmentation of winter jujube[J]. Horticulture Research,2021,8(1):238.
[46] XIA X C,LI H X,CAO D,LUO X,YANG X W,CHEN L N,LIU B B,WANG Q,JING D,CAO S Y. Characterization of a NAC transcription factor involved in the regulation of pomegranate seed hardness (Punica granatum L.)[J]. Plant Physiology and Biochemistry,2019,139:379-388.