同源盒基因家族(HB 基因家族)广泛存在于植物和动物中。HB 基因家族的成员通常都包含一个高度保守的同源异型框结构域(homeodomain,HD),能够与特定的DNA 序列结合,调控下游基因的表达[1-3]。Mukherjee 等[4]根据蛋白质序列进化分析,将植物中HB基因家族分为14类:HD-ZIPⅠ、HDZIP Ⅱ、HD-ZIP Ⅲ、HD-ZIP Ⅳ、PLINC、WOX、NDX、DDT、PHD、LD、SAWADEE、PINTOX、BEL和KNOX[5]。其中HD-ZIP Ⅰ~Ⅳ亚家族共同含有HD和亮氨酸拉链(leucine zipper,LZ)结构域[6],HD-ZIP Ⅲ亚族蛋白包含START 结构域和MEKHLA 结构域,HD-ZIP Ⅳ相比于HD-ZIP Ⅲ亚族不含有MEKHLA结构域[7-8]。PLINC 亚族特有的是PLINC 结构域;WOX家族特有的是WUS Box结构域;NDX家族具有NDX A 和NDX B 结构域;DDT 亚族特有的是DDT 和WSD 结构域;PHD 亚族特有的是PHD 结构域;LD亚族特有的是LD 1~5结构域;SAWADEE亚族特有的是SAWADEE 结构域;PINTOX 亚族特有的是ACID PINT 和PINTOX 结构域;BEL 亚族特有的是ZIBEL 结构域;KONX 亚族含有KNOX 结构域[4]。
HB基因家族编码的转录因子在植物生长发育、形态建成以及逆境胁迫响应中发挥着关键作用[6]。KNOX 基因可以参与细胞分生组织能力的调控,从而促进芽的形成[9]。PtrWOX4 能够促进形成层分化[10],WOX11/12 可以调控叶子边缘形状、大小和长宽[11-12]。通过RNA 干扰技术沉默SlBL4 基因的番茄突变体表现出脱落区表皮细胞的显著增大,进而影响了果柄的正常形成,并导致了果实的过早脱落[13]。苹果中MdHB7和MdHB7-like基因在ABA和干旱胁迫下被上调,其过表达显著提高了ABA水平进而增强了植物的抗逆性[14]。CaHB12 转录因子的过表达提高了棉花的抗旱性,并通过抑制茉莉酸响应基因负调控对大丽弧菌的抗性[15]。CaATHB-12沉默提高了寒冷胁迫下辣椒果实中的抗氧化酶活性,而过表达降低了低温胁迫下转基因拟南芥品系中抗氧化酶活性,表明CaATHB-12参与辣椒果实中寒冷胁迫的调节[16]。
库尔勒香梨(Pyrus sinkiangensis Yü)是蔷薇科梨属中的一个优良新疆地方梨品种,属于多年生木本植物,主要种植在中国新疆的库尔勒和阿克苏地区。该品种因独特的香气和高品质深受消费者青睐,并且已经有1400 多年的栽培历史,成为当地的重要经济作物[17]。然而,随着气候变化的加剧,新疆地区频繁出现极端低温天气,导致库尔勒香梨树体受到严重的冻害[18-19],进而引发腐烂病等病害,对梨树的健康和产量产生严重影响。HB 基因家族作为一类调控植物发育和逆境胁迫应答的转录因子,在库尔勒香梨的抗寒性研究中具有重要意义。目前关于HB 家族的研究主要集中在模式植物中,且绝大部分为草本植物,例如拟南芥[20]、水稻[21]和马铃薯[22]等,但是对于多年生木本植物的研究相对较为匮乏。笔者在本研究中对库尔勒香梨基因组中HB基因进行了比较全面的分析,为后续研究库尔勒香梨HB基因功能研究提供一定的基础,也为库尔勒香梨优良抗性品种的进一步选育提供理论指导,对库尔勒香梨产业的可持续发展具有重要的经济价值和指导意义。
1 材料和方法
1.1 试验材料
基因组测序所用的库尔勒香梨组培苗试验材料由石河子大学生命科学学院保存。基因组数据的测定由百迈克生物有限公司(http://www.biomarker.com.cn/)完成。
1.2 试验方法
1.2.1 库尔勒香梨HB家族成员全基因组的鉴定 为鉴定库尔勒香梨基因组中全部的HB 家族成员,从拟南芥(Arabidopsis thaliana)数据库(https://www.arabidopsis.org/)和苹果(Malus domestica)数据库(https://iris.angers.inra.fr/gddh13/)中下载全部的HB家族成员的蛋白序列,使用本团队前期构建的库尔勒香梨全基因组数据[23](https://ngdc.cncb.ac.cn/,数据编号为PRJCA007928)进行比对。以拟南芥和苹果HB 基因序列为靶序列,参考Li 等[24]的方法筛选库尔勒香梨的HB 序列,利用BLASTp 检索出库尔勒香梨全基因组中的HB 候选序列,用隐马尔可夫模型(HMM)进一步检索库尔勒香梨HB 基因结构域(PF00046、PF02183),E值为e-5。检索后的序列利用NCBI网站中的Batch CD-Search工具对笔者上一步筛选出的库尔勒香梨HB基因家族蛋白结构域进行预测,剔除不含有HB 相关结构域的序列。将获得的PsHB 基因家族蛋白序列的长度、相对分子质量、等电点等信息,由在线网站ExPASY(http://www.expasy.org)分析。库尔勒香梨HB 基因家族的亚细胞定位预测利用SignaIP4.1(http://www.cbs.dtu.dk/services/SignalP-4.1/)完成。
1.2.2 库尔勒香梨HB 基因在染色体上的分布 库尔勒香梨HB基因家族的染色体定位分析参照林艺灵等[5]的方法。
1.2.3 库尔勒香梨HB 基因家族系统进化树构建库尔勒香梨HB基因家族系统进化树构建参照林艺灵等[5]的方法。利用MEGA11 软件[25]构建进化树。进化树美化使用tvBOT 在线网站(https://www.chiplot.online/tvbot.html)完成[26]。
1.2.4 库尔勒香梨HB基因结构和所编码蛋白的保守结构域分析 基因结构的注释和MEME 保守基序分析参考叶明辉等[22]的方法。使用NCBI 中蛋白结构保守结构域预测网站CDD(https://www.ncbi.nlm.nih.gov/cdd/)来搜索库尔勒香梨HB蛋白包含的结构域,可视化图形利用TBtools软件[27]完成。
1.2.5 库尔勒香梨HB基因家族共线性和选择压力分析 库尔勒香梨HB基因家族共线性和选择压力分析参考林艺灵等[5]的方法。
1.2.6 库尔勒香梨HB基因家族启动子顺式作用元件分析 利用PlantCARE[28]在线网站对库尔勒香梨HB 基因家族ATG 上游2000 bp 的启动子序列进行预测分析。使用基因结构可视化网站GSDS v2.0(http://gsds.gao-lab.org/)进行可视化。
1.2.7 越冬过程中库尔勒香梨HB基因的表达特性分析 根据本课题组前期库尔勒香梨1年生枝条韧皮部的转录组序列数据[23]进行库尔勒香梨HB 基因家族的表达分析,库尔勒香梨越冬转录组测定的取样时间分别是2019 年10 月中旬的越冬初期(TB)、2020 年1 月中旬的越冬最冷期(TM)和2020 年3 月中旬的越冬末期(TF)。根据基因组ID 号从转录组数据中提取HB基因在越冬过程中的基因表达量信息,以|log2foldchange|≥1为条件筛选差异表达基因,使用TBtools绘制热图。
笔者选取3 个HB 基因(PsHB3、PsHB23 和PsHB66)用于qRT-PCR 验证。使用Primer Premier软件设计用于实时荧光定量PCR 的引物,PsHB3 使用的引物序列(5'→3')上游:GTGGGTCTGTGTCTAATCTTGG,下游:GCATCAGGGTTCAAGGTCTAG;PsHB23 使用的引物序列(5'→3')上游:GGTCTCTGAAGGCGAAGTATC,下游:AAGAATCACCAGGCTCCAAG;PsHB66 使用的引物序列(5'→3')上游:TCAAAGTCCCACAAGTTCTCC,下游 :AGGTGAATGATCCGAAGCTATG。 采 用ROCHE LightCycler® 480 system 仪器检测各基因的表达水平,以SYBR Green(KAPA Biosystems,Wilmington,MA,USA)作为荧光染料。PCR总体系为10 μL,每管分别加入2 ng RNA模板、上下游引物各为0.5 μL 和5 μL 荧光染料,最后用去离子水补足至10 μL。以PsGADPH作为内参基因,采用2-ΔΔCt法计算基因的相对表达水平。
1.3 数据处理
数据统计分析采用Excel 和SPSS v23.0 统计软件(SPSS Inc.;Chicago,IL,USA)。作图与相关性分析采用Origin 2020 软件(OriginLab; Northampton,MA,USA)和TBtools软件。
2 结果与分析
2.1 库尔勒香梨HB家族成员鉴定及理化性质分析
以拟南芥和苹果HB 基因家族的蛋白序列作为参考,最终在库尔勒香梨基因组中鉴定出93 个HB基因。根据其在库尔勒香梨染色体上的分布位置情况,将这些基因命名为库尔勒香梨HB(PsHB)1~93(表1)。基因编码的蛋白长度、相对分子质量、等电点、信号肽的预测和亚细胞定位的预测如表1 所示。HB 蛋白的长度范围在176~1196 个氨基酸,分子质量范围在20.18~135.08 kDa 之间,最大的是PsHB89,最小的是PsHB4。由此可见,库尔勒香梨HB 基因家族的氨基酸和相对分子质量的差异是比较大的。等电点(pI)的范围为4.69~9.32(表1),其中超过75%的蛋白等电点在7.0以下,表明库尔勒香梨的HB基因编码的可能是一类酸性蛋白。所有的成员中仅PsHB5 具有信号肽,剩下的PsHB 成员没有信号肽。将库尔勒香梨PsHB 蛋白序列上传至SignaIP4.1网站,发现有6个成员(PsHB90、PsHB54、PsHB15、PsHB48、PsHB36 和PsHB12)定位在叶绿体上,其余全部定位于细胞核。
表1 库尔勒香梨HB 基因家特征
Table 1 HB gene family characteristics of Pyrus sinkiangensis
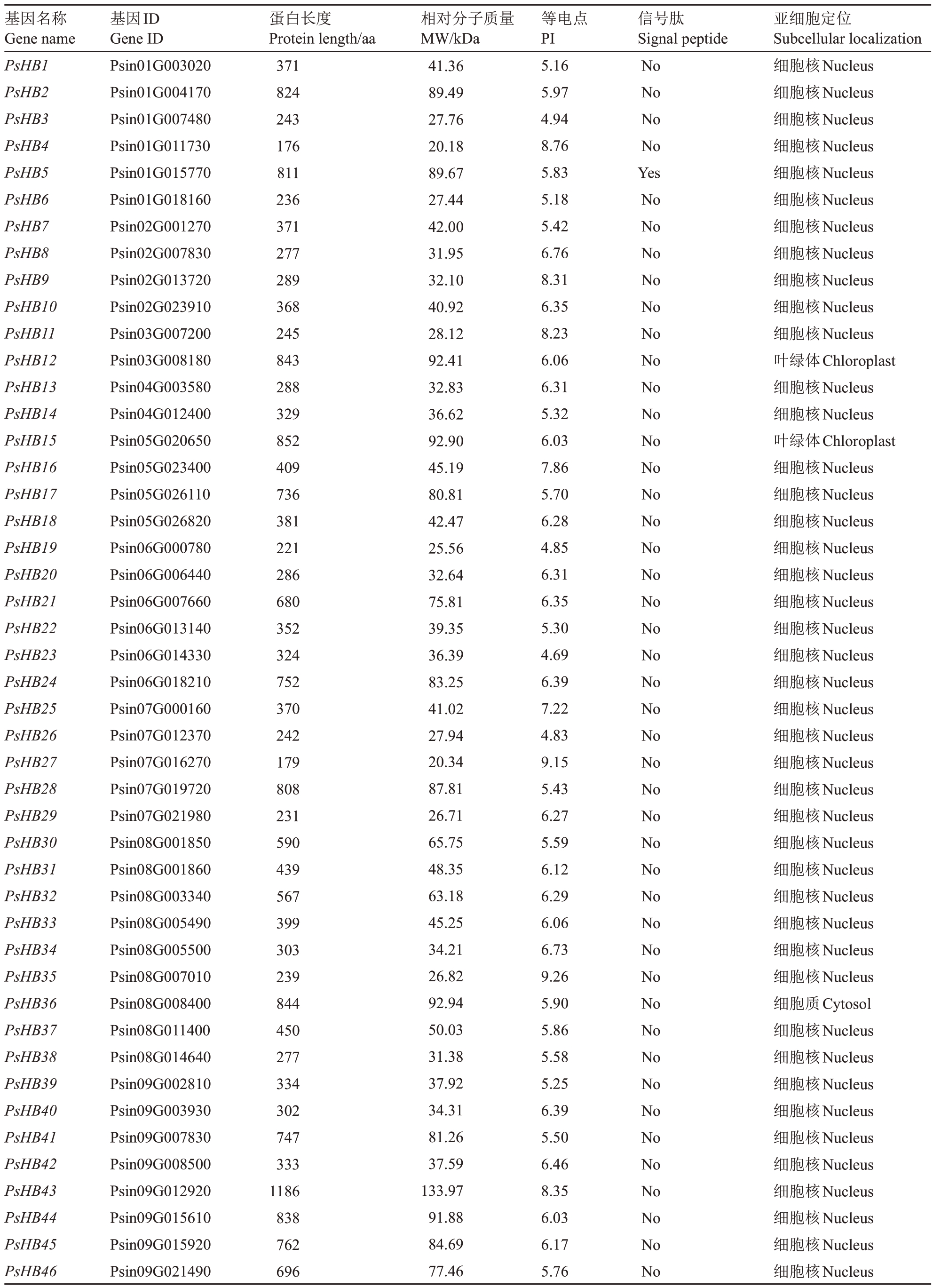
基因名称Gene name基因ID Gene ID蛋白长度Protein length/aa相对分子质量MW/kDa等电点PI信号肽Signal peptide亚细胞定位Subcellular localization PsHB1 PsHB2 PsHB3 PsHB4 PsHB5 PsHB6 PsHB7 PsHB8 PsHB9 PsHB10 PsHB11 PsHB12 PsHB13 PsHB14 PsHB15 PsHB16 PsHB17 PsHB18 PsHB19 PsHB20 PsHB21 PsHB22 PsHB23 PsHB24 PsHB25 PsHB26 PsHB27 PsHB28 PsHB29 PsHB30 PsHB31 PsHB32 PsHB33 PsHB34 PsHB35 PsHB36 PsHB37 PsHB38 PsHB39 PsHB40 PsHB41 PsHB42 PsHB43 PsHB44 PsHB45 PsHB46 Psin01G003020 Psin01G004170 Psin01G007480 Psin01G011730 Psin01G015770 Psin01G018160 Psin02G001270 Psin02G007830 Psin02G013720 Psin02G023910 Psin03G007200 Psin03G008180 Psin04G003580 Psin04G012400 Psin05G020650 Psin05G023400 Psin05G026110 Psin05G026820 Psin06G000780 Psin06G006440 Psin06G007660 Psin06G013140 Psin06G014330 Psin06G018210 Psin07G000160 Psin07G012370 Psin07G016270 Psin07G019720 Psin07G021980 Psin08G001850 Psin08G001860 Psin08G003340 Psin08G005490 Psin08G005500 Psin08G007010 Psin08G008400 Psin08G011400 Psin08G014640 Psin09G002810 Psin09G003930 Psin09G007830 Psin09G008500 Psin09G012920 Psin09G015610 Psin09G015920 Psin09G021490 371 824 243 176 811 236 371 277 289 368 245 843 288 329 852 409 736 381 221 286 680 352 324 752 370 242 179 808 231 590 439 567 399 303 239 844 450 277 334 302 747 333 1186 838 762 696 41.36 89.49 27.76 20.18 89.67 27.44 42.00 31.95 32.10 40.92 28.12 92.41 32.83 36.62 92.90 45.19 80.81 42.47 25.56 32.64 75.81 39.35 36.39 83.25 41.02 27.94 20.34 87.81 26.71 65.75 48.35 63.18 45.25 34.21 26.82 92.94 50.03 31.38 37.92 34.31 81.26 37.59 133.97 91.88 84.69 77.46 5.16 5.97 4.94 8.76 5.83 5.18 5.42 6.76 8.31 6.35 8.23 6.06 6.31 5.32 6.03 7.86 5.70 6.28 4.85 6.31 6.35 5.30 4.69 6.39 7.22 4.83 9.15 5.43 6.27 5.59 6.12 6.29 6.06 6.73 9.26 5.90 5.86 5.58 5.25 6.39 5.50 6.46 8.35 6.03 6.17 5.76 No No No No Yes No No No No No No No No No No No No No No No No No No No No No No No No No No No No No No No No No No No No No No No No No细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus叶绿体Chloroplast细胞核Nucleus细胞核Nucleus叶绿体Chloroplast细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞质Cytosol细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus
表1 (续) Table 1 (Continued)
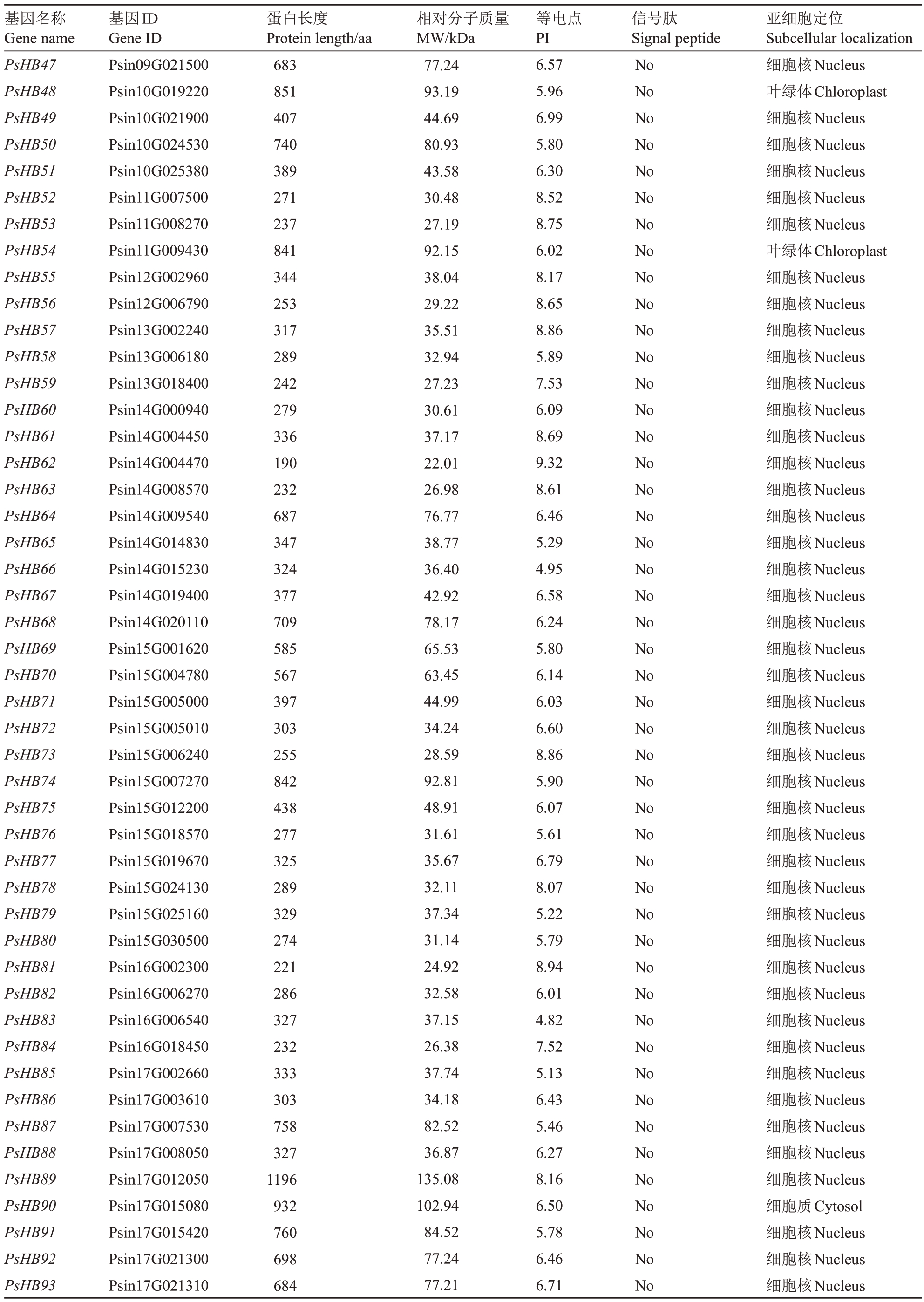
基因名称Gene name基因ID Gene ID蛋白长度Protein length/aa相对分子质量MW/kDa等电点PI信号肽Signal peptide亚细胞定位Subcellular localization PsHB47 PsHB48 PsHB49 PsHB50 PsHB51 PsHB52 PsHB53 PsHB54 PsHB55 PsHB56 PsHB57 PsHB58 PsHB59 PsHB60 PsHB61 PsHB62 PsHB63 PsHB64 PsHB65 PsHB66 PsHB67 PsHB68 PsHB69 PsHB70 PsHB71 PsHB72 PsHB73 PsHB74 PsHB75 PsHB76 PsHB77 PsHB78 PsHB79 PsHB80 PsHB81 PsHB82 PsHB83 PsHB84 PsHB85 PsHB86 PsHB87 PsHB88 PsHB89 PsHB90 PsHB91 PsHB92 PsHB93 Psin09G021500 Psin10G019220 Psin10G021900 Psin10G024530 Psin10G025380 Psin11G007500 Psin11G008270 Psin11G009430 Psin12G002960 Psin12G006790 Psin13G002240 Psin13G006180 Psin13G018400 Psin14G000940 Psin14G004450 Psin14G004470 Psin14G008570 Psin14G009540 Psin14G014830 Psin14G015230 Psin14G019400 Psin14G020110 Psin15G001620 Psin15G004780 Psin15G005000 Psin15G005010 Psin15G006240 Psin15G007270 Psin15G012200 Psin15G018570 Psin15G019670 Psin15G024130 Psin15G025160 Psin15G030500 Psin16G002300 Psin16G006270 Psin16G006540 Psin16G018450 Psin17G002660 Psin17G003610 Psin17G007530 Psin17G008050 Psin17G012050 Psin17G015080 Psin17G015420 Psin17G021300 Psin17G021310 683 851 407 740 389 271 237 841 344 253 317 289 242 279 336 190 232 687 347 324 377 709 585 567 397 303 255 842 438 277 325 289 329 274 221 286 327 232 333 303 758 327 1196 932 760 698 684 77.24 93.19 44.69 80.93 43.58 30.48 27.19 92.15 38.04 29.22 35.51 32.94 27.23 30.61 37.17 22.01 26.98 76.77 38.77 36.40 42.92 78.17 65.53 63.45 44.99 34.24 28.59 92.81 48.91 31.61 35.67 32.11 37.34 31.14 24.92 32.58 37.15 26.38 37.74 34.18 82.52 36.87 135.08 102.94 84.52 77.24 77.21 6.57 5.96 6.99 5.80 6.30 8.52 8.75 6.02 8.17 8.65 8.86 5.89 7.53 6.09 8.69 9.32 8.61 6.46 5.29 4.95 6.58 6.24 5.80 6.14 6.03 6.60 8.86 5.90 6.07 5.61 6.79 8.07 5.22 5.79 8.94 6.01 4.82 7.52 5.13 6.43 5.46 6.27 8.16 6.50 5.78 6.46 6.71 No No No No No No No No No No No No No No No No No No No No No No No No No No No No No No No No No No No No No No No No No No No No No No No细胞核Nucleus叶绿体Chloroplast细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus叶绿体Chloroplast细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞质Cytosol细胞核Nucleus细胞核Nucleus细胞核Nucleus
2.2 库尔勒香梨HB基因家族染色体定位分析
93个库尔勒香梨HB基因家族成员分别定位在17 条染色体上(图1)。其中在库尔勒香梨Chr15 上HB基因家族的成员数量最多,为12个,其余染色体上的成员个数较少(2~9个)。多数成员存在染色体的上端和下端,在染色体中部分布较少。有1/3以上的成员集中分布在Chr14~17这4条染色体上面。另外,在9 号染色体和17 号染色体上的PsHB46、PsHB47和PsHB92、PsHB93两对成员位置相近且序列具有高度一致性。同时,它们4个均属于HD-ZIPⅣ家族的成员。
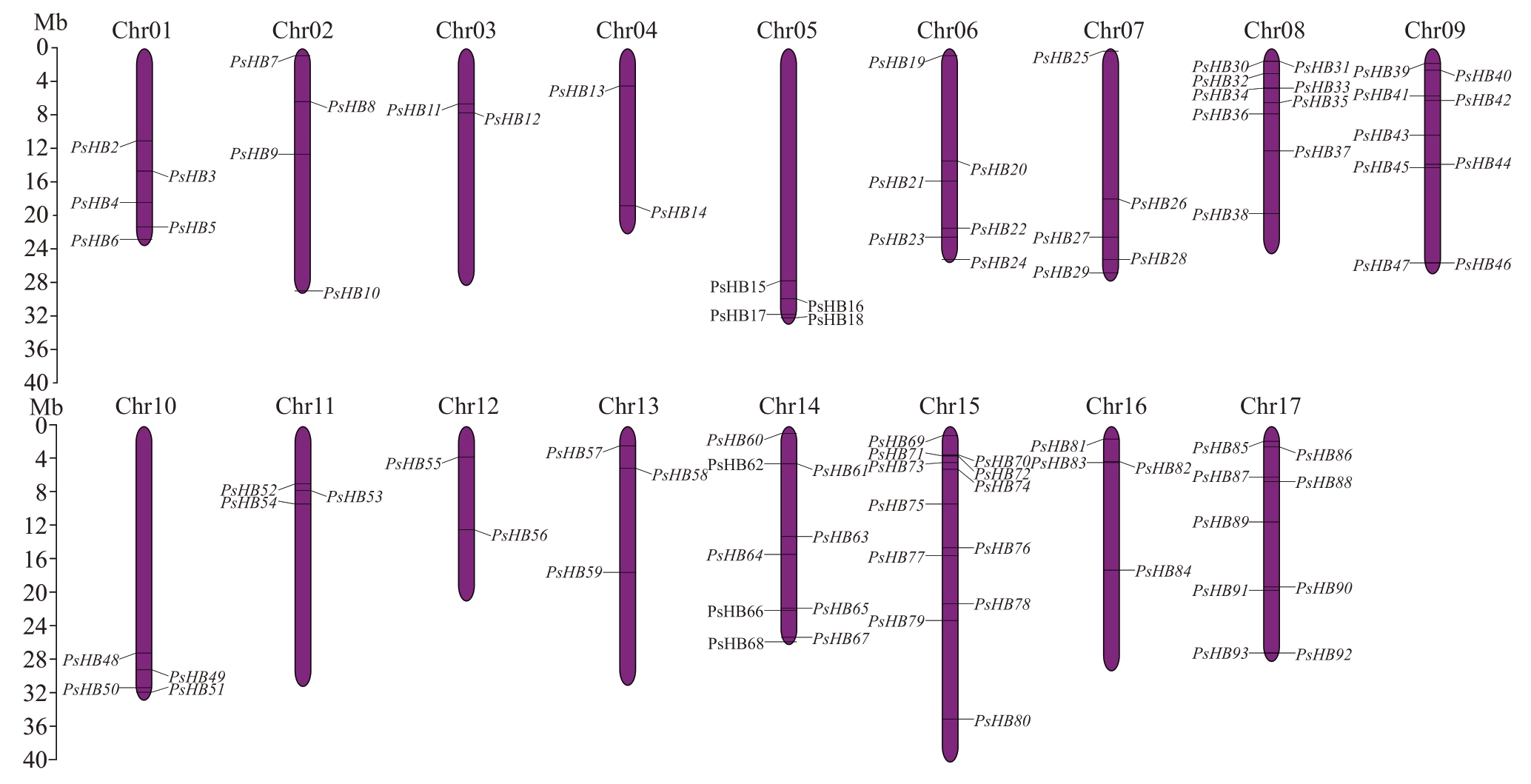
图1 库尔勒香梨HB 基因家族染色体定位分析
Fig.1 Chromosome localization analysis of HB gene family in P.sinkiangensis
2.3 库尔勒香梨HB基因家族系统进化树分析
为了解库尔勒香梨HB 基因家族间的进化关系,构建了库尔勒香梨、拟南芥和苹果HB 基因家族系统进化树(图2)。参考拟南芥HB 基因家族与苹果HD-ZIPⅠ~Ⅳ基因家族分类,使用ClustalW 多重序列比对方法将笔者获得的库尔勒香梨HB蛋白序列进行对比,最终根据进化关系将其分为8 个亚家族。其中HD-ZIPⅠ和Ⅳ家族的成员在基因组中包含HB 的成员数量较多,两者共有34 个,占所有成员的1/3 以上。最少的亚家族是DDT,有2 个成员。

图2 拟南芥、苹果和库尔勒香梨HB 基因家族系统进化树
Fig.2 Phylogenetic tree of HB gene families in Arabidopsis thaliana,Malus domestica and P.sinkiangensis
2.4 库尔勒香梨HB基因结构和蛋白保守结构域分析
库尔勒香梨中HB 成员均包含CDS(Coding DNA Sequence),数目在2~20个(图3),34个成员有非翻译区(Untranslated Region,UTR)。库尔勒香梨HD-ZIPⅠ亚家族的CDS数量为2~4个,且CDS和内含子(Intron)排列的分布规律一致,为CDS-Intron-CDS。由于一些基因在长度上差异较大,不同亚家族的内含子和外显子的分布位置和数目也存在很大差异。
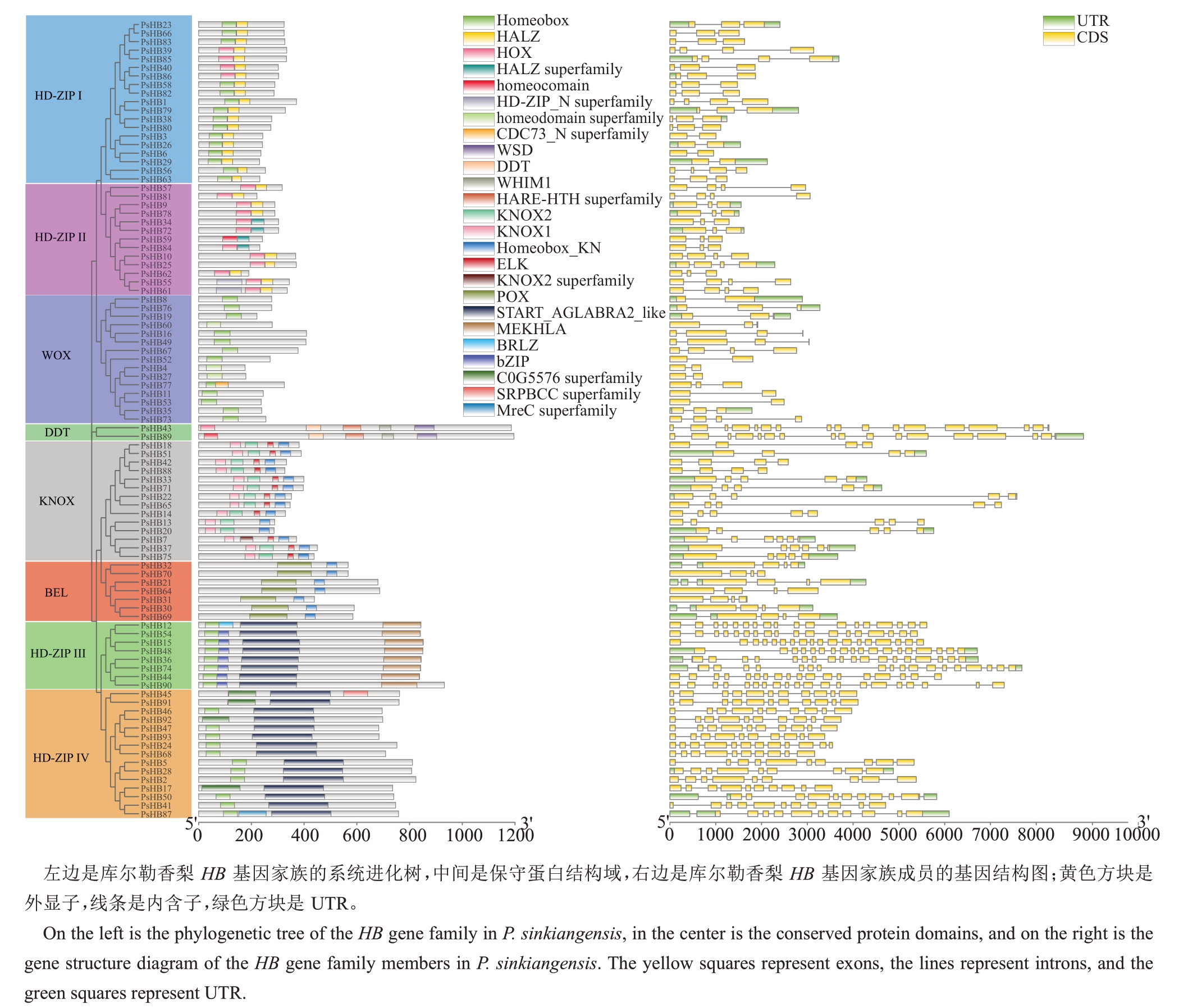
图3 库尔勒香梨HB 基因结构及其所编码蛋白的保守结构域
Fig.3 HB gene structure and the conserved domains of the encoded proteins in P.sinkiangensis
使用NCBI网站中提供的Batch CD-Search工具对库尔勒香梨HB 基因家族蛋白结构域进行预测,结果显示均含有HD 结构域(图3)。所有库尔勒香梨PsHB 成员共有25 种结构域,除HD 结构域外,同源异型盒相关亮氨酸(HALZ)结构域的数量最多。
2.5 库尔勒香梨HB基因家族共线性和选择压力分析
通过TBtools中的MCScanX插件分析库尔勒香梨HB基因家族的共线性关系,结果(表2和图4)表明库尔勒香梨HB基因家族有70对共线性基因。共鉴定出了6 个串联复制事件(PsHB30/PsHB31、PsHB33/PsHB34、PsHB46/PsHB47、PsHB61/PsHB62、PsHB70/PsHB71/PsHB72、PsHB92/PsHB93),分别位于第8、8、9、14、15 和17 号染色体上。笔者发现,在第8 号染色体和15号染色体上PsHB基因以串联复制的形式出现,但属于不同的亚家族,这表明在串联复制之后,这些基因的序列发生了很大程度的改变,可以通过结构域的重组获得蛋白质的多样化。通过鉴定库尔勒香梨HB家族复制基因的Ka/Ks值发现,Ka/Ks值的取值范围在0.06~0.49之间。库尔勒香梨70 对共线性基因中67 对复制基因鉴定出Ka/Ks 值小于1。表明整体序列偏向于保守型,进化时受纯化选择影响。其中3 对复制基因鉴定产生异常值,异常值产生的原因可能是序列分歧度太大,进化距离太远。
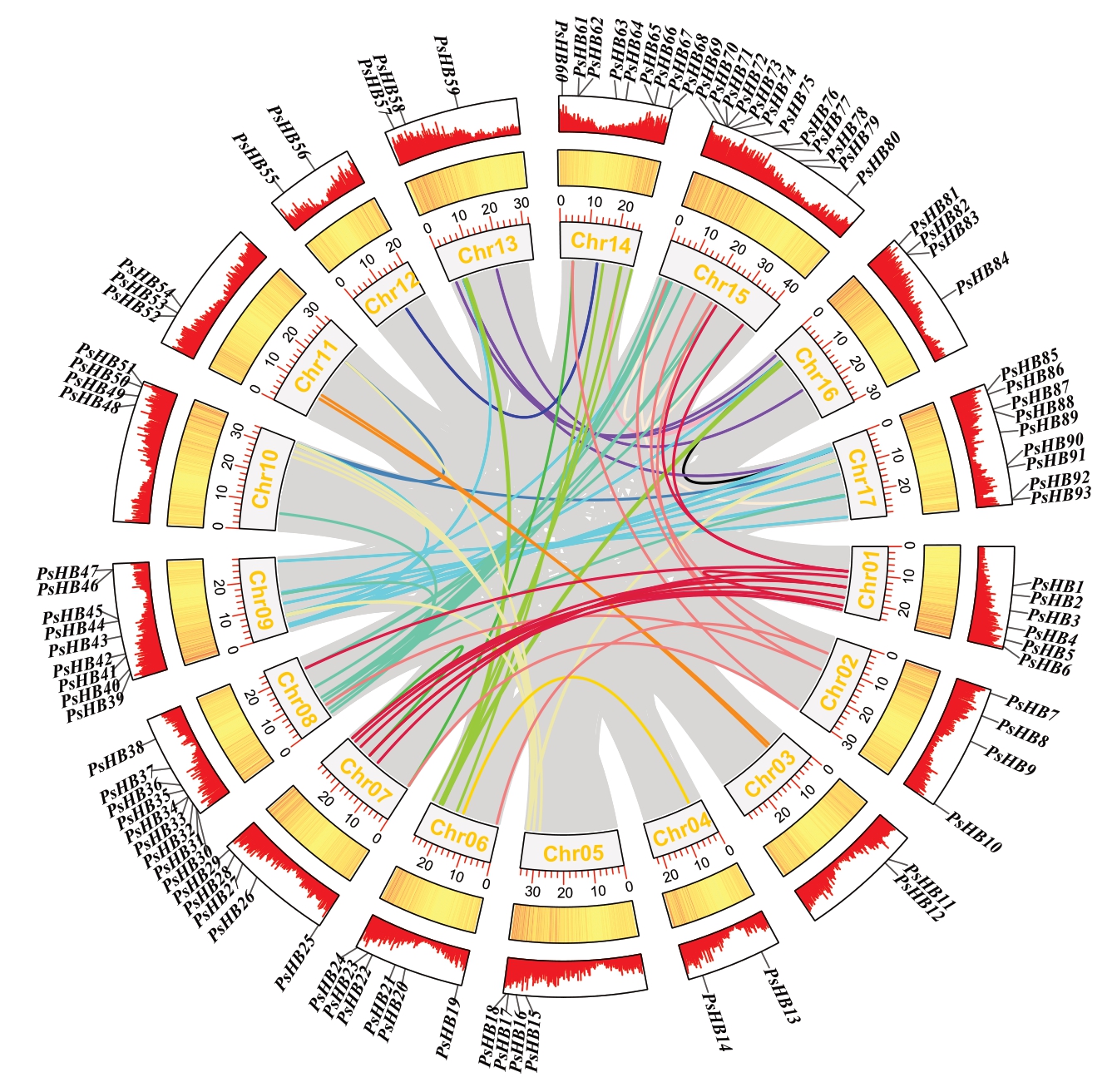
图4 库尔勒香梨HB 基因共线性分析
Fig.4 Collinearity analysis of HB gene in P.sinkiangensis
表2 库尔勒香梨HB 基因家族共线性关系及非同义替换率(Ka)和同义替换率(Ks)
Table 2 Collinearity of HB gene family and non-synonymous replacement rate(Ka)and synonymous replacement rate(Ks)in P.sinkiangensis
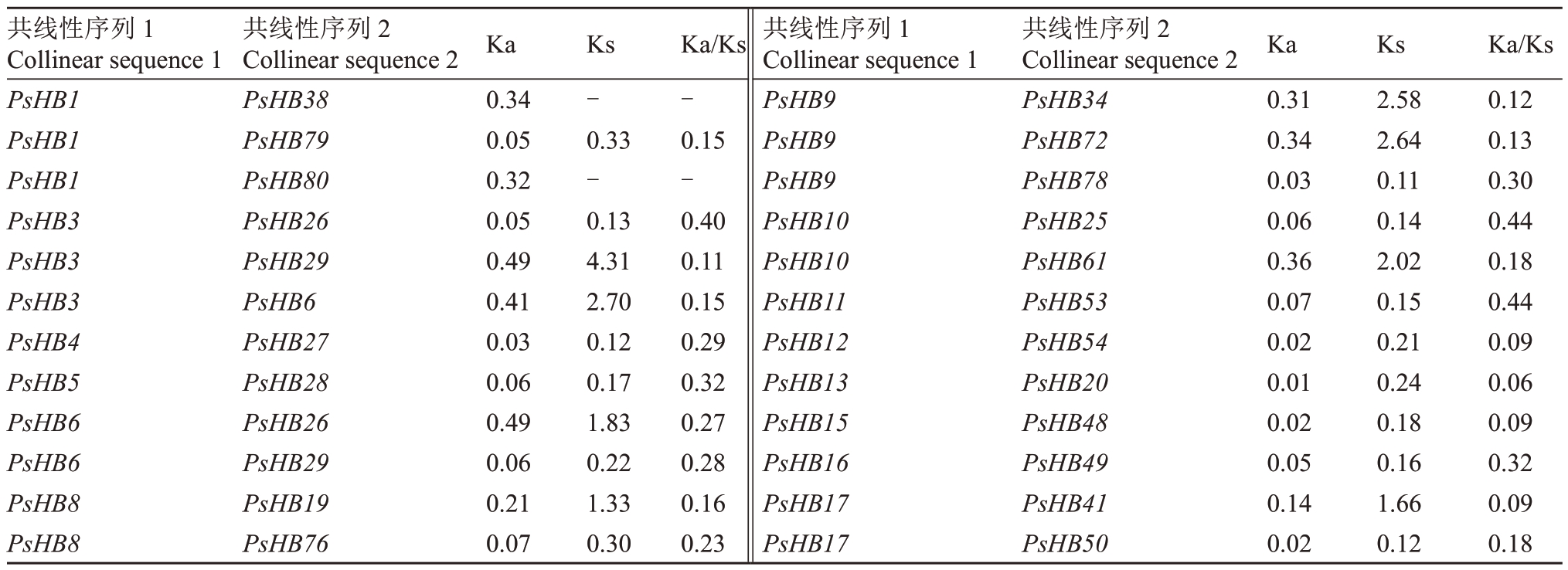
共线性序列1 Collinear sequence 1 Collinear sequence 2 KaKsKa/Ks 共线性序列1 Collinear sequence 1共线性序列2共线性序列2 Collinear sequence 2 KaKsKa/Ks PsHB1 PsHB1 PsHB1 PsHB3 PsHB3 PsHB3 PsHB4 PsHB5 PsHB6 PsHB6 PsHB8 PsHB8 PsHB38 PsHB79 PsHB80 PsHB26 PsHB29 PsHB6 PsHB27 PsHB28 PsHB26 PsHB29 PsHB19 PsHB76 0.34 0.05 0.32 0.05 0.49 0.41 0.03 0.06 0.49 0.06 0.21 0.07--0.33-0.13 4.31 2.70 0.12 0.17 1.83 0.22 1.33 0.30 0.15-0.40 0.11 0.15 0.29 0.32 0.27 0.28 0.16 0.23 PsHB9 PsHB9 PsHB9 PsHB10 PsHB10 PsHB11 PsHB12 PsHB13 PsHB15 PsHB16 PsHB17 PsHB17 PsHB34 PsHB72 PsHB78 PsHB25 PsHB61 PsHB53 PsHB54 PsHB20 PsHB48 PsHB49 PsHB41 PsHB50 0.31 0.34 0.03 0.06 0.36 0.07 0.02 0.01 0.02 0.05 0.14 0.02 2.58 2.64 0.11 0.14 2.02 0.15 0.21 0.24 0.18 0.16 1.66 0.12 0.12 0.13 0.30 0.44 0.18 0.44 0.09 0.06 0.09 0.32 0.09 0.18
表2 (续) Table 2 (Continued)
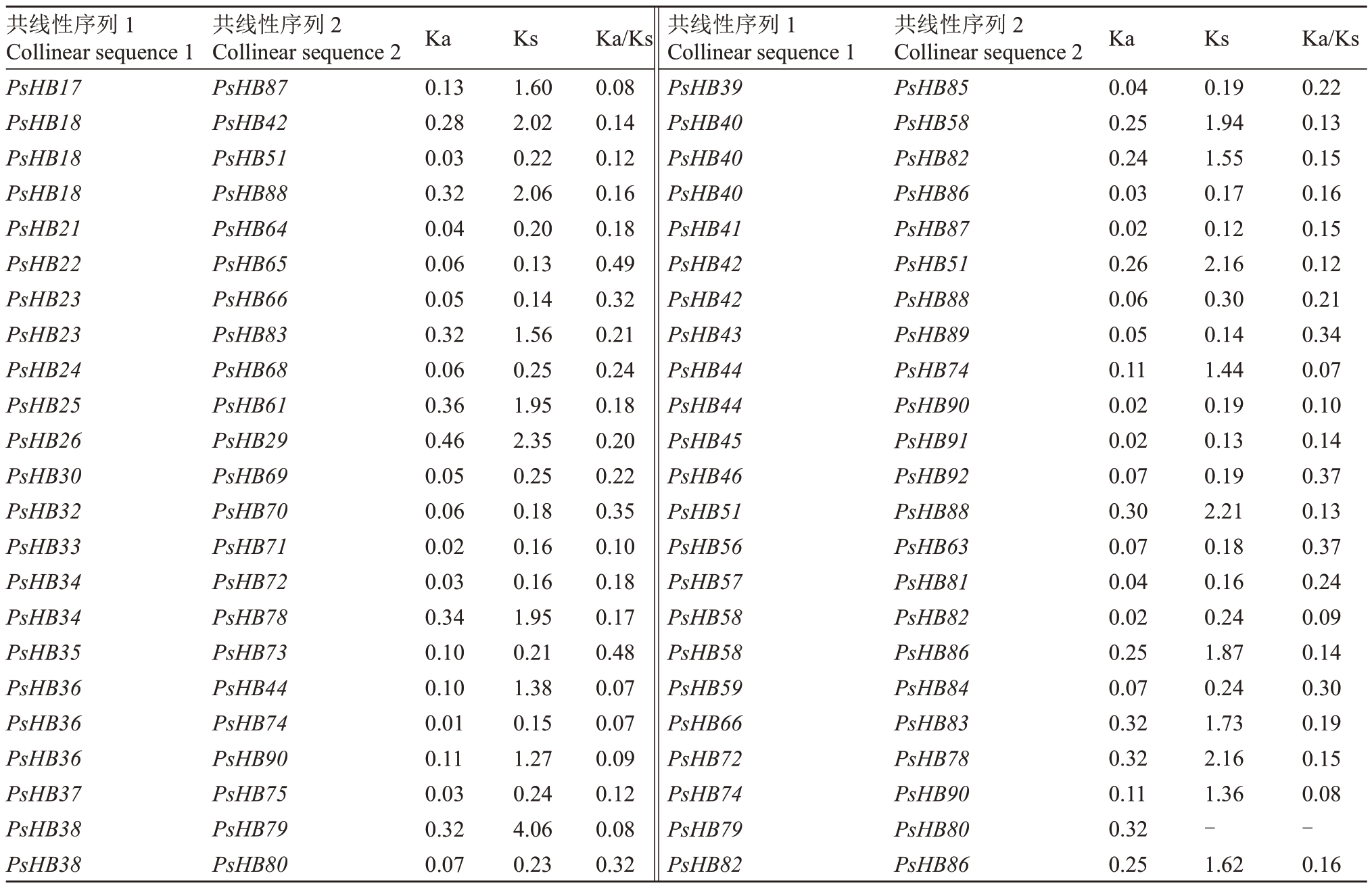
共线性序列1 Collinear sequence 1 Collinear sequence 2 KaKsKa/Ks 共线性序列1 Collinear sequence 1共线性序列2共线性序列2 Collinear sequence 2 KaKsKa/Ks PsHB17 PsHB18 PsHB18 PsHB18 PsHB21 PsHB22 PsHB23 PsHB23 PsHB24 PsHB25 PsHB26 PsHB30 PsHB32 PsHB33 PsHB34 PsHB34 PsHB35 PsHB36 PsHB36 PsHB36 PsHB37 PsHB38 PsHB38 PsHB87 PsHB42 PsHB51 PsHB88 PsHB64 PsHB65 PsHB66 PsHB83 PsHB68 PsHB61 PsHB29 PsHB69 PsHB70 PsHB71 PsHB72 PsHB78 PsHB73 PsHB44 PsHB74 PsHB90 PsHB75 PsHB79 PsHB80 0.13 0.28 0.03 0.32 0.04 0.06 0.05 0.32 0.06 0.36 0.46 0.05 0.06 0.02 0.03 0.34 0.10 0.10 0.01 0.11 0.03 0.32 0.07 1.60 2.02 0.22 2.06 0.20 0.13 0.14 1.56 0.25 1.95 2.35 0.25 0.18 0.16 0.16 1.95 0.21 1.38 0.15 1.27 0.24 4.06 0.23 0.08 0.14 0.12 0.16 0.18 0.49 0.32 0.21 0.24 0.18 0.20 0.22 0.35 0.10 0.18 0.17 0.48 0.07 0.07 0.09 0.12 0.08 0.32 PsHB39 PsHB40 PsHB40 PsHB40 PsHB41 PsHB42 PsHB42 PsHB43 PsHB44 PsHB44 PsHB45 PsHB46 PsHB51 PsHB56 PsHB57 PsHB58 PsHB58 PsHB59 PsHB66 PsHB72 PsHB74 PsHB79 PsHB82 PsHB85 PsHB58 PsHB82 PsHB86 PsHB87 PsHB51 PsHB88 PsHB89 PsHB74 PsHB90 PsHB91 PsHB92 PsHB88 PsHB63 PsHB81 PsHB82 PsHB86 PsHB84 PsHB83 PsHB78 PsHB90 PsHB80 PsHB86 0.04 0.25 0.24 0.03 0.02 0.26 0.06 0.05 0.11 0.02 0.02 0.07 0.30 0.07 0.04 0.02 0.25 0.07 0.32 0.32 0.11 0.32 0.25 0.19 1.94 1.55 0.17 0.12 2.16 0.30 0.14 1.44 0.19 0.13 0.19 2.21 0.18 0.16 0.24 1.87 0.24 1.73 2.16 1.36-1.62 0.22 0.13 0.15 0.16 0.15 0.12 0.21 0.34 0.07 0.10 0.14 0.37 0.13 0.37 0.24 0.09 0.14 0.30 0.19 0.15 0.08-0.16
2.6 库尔勒香梨HB基因顺式作用元件分析
对PsHB 基因家族成员上游2000 bp 的启动子进行顺式作用元件分析,鉴定出与植物激素和胁迫响应相关的12 种启动子顺式作用元件(图5)。其中,激素类相关的响应元件包括茉莉酸甲酯响应元件(CGTCA-motif、TGACG-motif)、水杨酸响应元件(TCA-element、SARE)、赤霉素响应元件(P-box、GARE-motif)、脱落酸响应元件(ABRE)和生长素响应元件(TGA-element、AuxRR-core)等5 类。PsHB基因中,78.49%含有ABRE,50.54%含有P-box、GARE-motif。与胁迫响应相关的元件有7 类,包括无防御和应激反应元件(TC-rich repeats、TATCbox)、伤口响应元件(WUN-motif)、光响应元件(ACE、GT1-motif、3-AF1 binding site、AAAC-motif、MRE、Sp1、G-Box/box)、氧诱导响应元件(ARE)、低温响应诱导元件(LTR)、缺氧特异性诱导元件(GCmotif)和干旱响应诱导元件(MBS)。其中,含有MBS 和LTR 元件的PsHB 基因分别占41.94%和54.84%。而PsHB25、PsHB55、PsHB61、PsHB84 和PsHB91 这4 个基因含有与创伤修复相关的元件。这些结果表明PsHB基因广泛参与库尔勒香梨的胁迫响应及激素信号转导过程。

图5 PsHB 基因家族中启动子的顺式元件
Fig.5 The cis-element of promoter in PsHB gene family
2.7 库尔勒香梨HB基因家族在越冬过程中的表达分析
对库尔勒香梨越冬过程中3个时期转录组数据分析发现,有39个基因在越冬过程中差异表达。图6-A 显示,在鉴定出来的差异表达基因中有超过半数的基因(21 个)在3 月份越冬末期(TF)高表达,11个基因在12月份的越冬最冷期(TM)高表达,其余7个基因在10月份越冬初期(TB)高表达。其中在12月份的越冬最冷期,PsHB11 和PsHB78 相比于越冬初期分别上调10.5和7.0倍,表明这两个基因可能在芽休眠期发挥一定的作用。大约42%的PsHB 基因在越冬过程中显著差异表达,并且大部分基因在越冬最冷时期12月份时表达量达到最高,提示库尔勒香梨中的HB基因家族在越冬时期低温胁迫中发挥作用。荧光定量PCR 的结果显示,PsHB3、PsHB23和PsHB66 基因在越冬过程中的表达趋势与转录组测序一致(图6-B)。
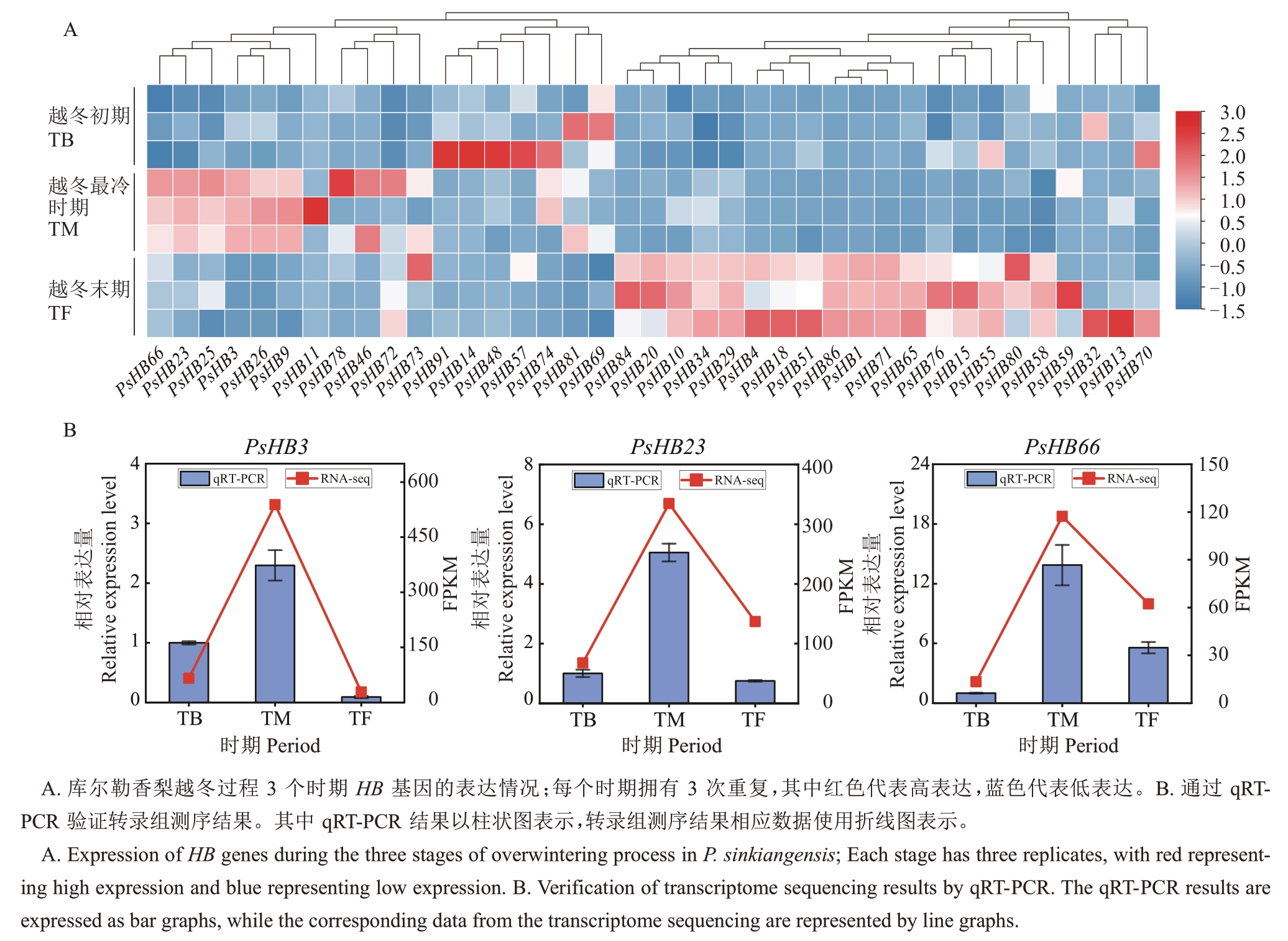
图6 PsHB 基因在越冬过程中的表达情况
Fig.6 Expression of PsHB gene family in P.sinkiangensis during overwintering
3 讨 论
前人的研究已经对多个物种中的HD-Zip 转录因子进行了鉴定。例如,拟南芥中发现了110个HB基因[20],胡萝卜(Daucus carota)中鉴定出了140 个HB 基因[29],水稻(Oryza sativa)中有107 个HB 基因[21],油菜(Brassica rapa)中有113 个HB 基因[30],毛杨果(Populus trichocarpa)中有156 个HB 基因[31]。而在笔者的研究中,库尔勒香梨的基因组中共鉴定出了93 个HB 基因。与其他物种相比,库尔勒香梨中HB基因的数目相对来说比较少,库尔勒香梨HB基因可能在进化过程中发生了部分基因缺失。根据库尔勒香梨基因组基因结构的注释,PsHBs 基因在所有17 条染色体上分布不均匀,染色体编号为14、15 和17 这几条染色体上的PsHBs 基因约占总数的三分之一。因此,笔者推测这3 条染色体上存在PsHBs基因的进化热点。类似的结果也在其他物种中得到了验证。例如,在毛杨果中Chrs 1、2 和5 同样是约占PtrHBs 基因总数的三分之一[31]。葡萄中的VvHBs 基因在Chrs 4、8 和18 上占基因总数的三分之一[5]。
根据拟南芥和苹果HB 基因家族的分类和系统发育树,笔者将库尔勒香梨的HB基因划分为8个亚家族,包括HD-ZipⅠ~Ⅳ、WOX、BEL、KNOX 和DDT。与拟南芥相比,库尔勒香梨的HB 基因家族缺少了PINTOX、NDX、LD、PLINC、PHD 和SAWADEE 类成员[20]。而在其他物种中对HB 基因家族的鉴定也发现存在差异,如在水稻、油菜中也仅分别鉴定出10个和9个HB基因亚家族[21,30]。将与其同为蔷薇科的苹果HB基因家族成员数量进行比较分析,发现HD-Zip Ⅱ家族成员的数量是一致的,均为13 个[32]。而且库尔勒香梨HD-Zip Ⅰ、HD-Zip Ⅲ和HD-Zip Ⅳ亚家族成员的数量与苹果中亚家族的数量相似,分别为19、8和15个。表明库尔勒香梨和苹果作为蔷薇科植物,在进化过程中表现出一定程度的保守性,并且在形态发育、生长和逆境响应等方面可能存在类似的基因调控机制。这种相似性可能源于他们的共同祖先或者遗传机制的保守性,这对笔者理解这些植物的遗传调控网络以及他们之间的关系具有重要意义。库尔勒香梨HB基因中存在70对片段重复基因,表明在库尔勒香梨进化过程中可能促进了基因组的重组和重排,进而增加了遗传多样性,在进化过程中发挥了重要作用。此外,还存在6 对串联重复基因,这将可能导致基因副本间的相互作用,可能影响基因的表达和功能,有助于库尔勒香梨在逆境胁迫下进行基因组调节和进化,表明HB基因家族成员的扩增以片段重复为主。在9号染色体和17 号染色体上的PsHB46、PsHB47 和PsHB92、PsHB93 两对成员位置相近并且序列具有高度的一致性,它们4 个都属于HD-ZIP Ⅳ家族的成员,推测可能是基因间的串联重复造成的。8 号染色体第2个位置和15 号染色体上PsHBs 基因以串联复制的形式出现,但属于不同的亚家族。这表明在串联复制之后这些基因的序列发生了很大程度的改变,可以通过结构域的重组获得蛋白质的多样化,推测这些基因可能在串联复制后失去了原有的功能或获得了新的功能。
转录因子与基因启动子或增强子上的顺式作用元件结合以在各种生物过程中调节基因表达[33-35]。笔者分析了库尔勒香梨所有PsHBs 基因上游2000 bp 启动子区的顺式作用元件,发现这些启动子含有多种参与激素和非生物胁迫反应的顺式作用元件,包括茉莉酸甲酯(MeJA)反应元件、光反应元件、ABA反应元件和厌氧诱导反应元件。这些结果在油菜中也存在[30],大多数HB基因启动子含有与光反应、激素反应和应激反应相关的顺式作用元件。其中只有PsHB25、PsHB55、PsHB61、PsHB84和PsHB91这5个基因含有创伤修复相关的元件,暗示其可能在库尔勒香梨受到创伤时发挥一定作用。表明PsHBs基因广泛参与库尔勒香梨的胁迫响应和激素信号转导。
植物在越冬过程中,ABA是最主要的诱导剂和维持者[36]。大量的HD-Zip I 蛋白在受到ABA 信号诱导时参与植物应对胁迫的过程[37]。PsHBs基因上游2 kb 的启动子中包含大量的ABA 响应元件。此外,笔者前期的研究中发现,库尔勒香梨越冬过程中PsHBs基因随着越冬过程中环境温度的波动,其表达水平也随之发生动态变化。PsHB基因的上调表达可能与越冬过程中ABA信号的调控相关联。油菜型甘蓝HB7/12提高了拟南芥种子在萌发时对ABA的敏感性,并且过表达株系具有更高的抗旱性[38]。油菜型甘蓝HB7/12 与越冬最冷时期高表达的PsHB3 和PsHB23同属HD-Zip Ⅰ亚家族,具有大量的ABA响应元件并且在越冬过程中具有较高的表达量。因此,推测PsHB3 和PsHB23 基因在库尔勒香梨越冬过程中扮演着参与ABA信号调控的重要角色,并响应越冬时期低温胁迫。这一结果深化了对库尔勒香梨越冬适应性的理解,为未来探究其越冬机制提供了重要线索。然而,需进一步的研究来明确HB 基因在ABA信号调控中的具体调控机制,以及他们在库尔勒香梨越冬过程中的具体作用机制。
综上所述,本研究全面鉴定并分析了库尔勒香梨HB 基因家族,揭示了其在环境响应特别是越冬适应性中的重要潜在作用。这些结果不仅丰富了对库尔勒香梨基因组资源的了解,还为深入探究HB基因家族在其他植物物种中的功能提供了参考。
4 结 论
通过对库尔勒香梨全基因组的系统分析,成功鉴定出93 个HB 基因家族成员。这些基因在17 条染色体上不均匀分布,且编码的蛋白质在大小、相对分子质量和等电点等参数表现出显著的差异。系统进化分析将这些基因归为BEL、DDT、HD-ZIPⅠ~Ⅳ、KNOX 和WOX 等8 个亚家族。顺式作用元件分析显示,大部分HB 基因含有响应脱落酸、赤霉素、低温、干旱和无氧等环境应激的元件,暗示这些基因可能在库尔勒香梨的环境适应性中发挥重要作用。越冬适应性分析进一步发现,在越冬过程中有39 个HB 基因表现出显著的差异表达,还有11 个基因在最冷的12月(TM时期)表达量达到最高。这些结果表明,HB基因家族可能在库尔勒香梨的越冬适应性中扮演关键角色。综上所述,本研究不仅为库尔勒香梨的基因组研究提供了新的资源,也为探讨HB基因在其他植物中的功能及其在环境适应性中的调控机制提供了重要参考。
[1] WANG H L,CHENG X,YIN D M,CHEN D L,LUO C,LIU H,HUANG C L.Advances in the research on plant WRKY transcription factors responsive to external stresses[J]. Current Issues in Molecular Biology,2023,45(4):2861-2880.
[2] RADANI Y,LI R X,KORBOE H M,MA H Y,YANG L M.Transcriptional and post-translational regulation of plant bHLH transcription factors during the response to environmental stresses[J].Plants,2023,12(11):2113.
[3] LIU H T,TANG X,ZHANG N,LI S G,SI H J. Role of bZIP transcription factors in plant salt stress[J]. International Journal of Molecular Sciences,2023,24(9):7893.
[4] MUKHERJEE K,BROCCHIERI L,BÜRGLIN T R.A comprehensive classification and evolutionary analysis of plant homeobox genes[J]. Molecular Biology and Evolution,2009,26(12):2775-2794.
[5] 林艺灵,刘众杰,王子诚,杨毓贤,王令宇,张川,王晨,贾海锋,卢素文,房经贵,上官凌飞.葡萄HB 基因家族全基因组鉴定及表达分析[J].南京农业大学学报,2022,45(6):1126-1139.LIN Yiling,LIU Zhongjie,WANG Zicheng,YANG Yuxian,WANG Lingyu,ZHANG Chuan,WANG Chen,JIA Haifeng,LU Suwen,FANG Jinggui,SHANGGUAN Lingfei. Genomewide identification and expression analysis of HB gene family in Vitis vinifera[J]. Journal of Nanjing Agricultural University,2022,45(6):1126-1139.
[6] SESSA G,CARABELLI M,SASSI M. The ins and outs of homeodomain-leucine zipper/hormone networks in the regulation of plant development[J]. International Journal of Molecular Sciences,2024,25(11):5657.
[7] ŻYŁA N,BABULA-SKOWROŃSKA D. Evolutionary consequences of functional and regulatory divergence of HD-zip I transcription factors as a source of diversity in protein interaction networks in plants[J]. Journal of Molecular Evolution,2023,91(5):581-597.
[8] SCHRICK K,AHMAD B,NGUYEN H V.HD-zip Ⅳtranscription factors:Drivers of epidermal cell fate integrate metabolic signals[J].Current Opinion in Plant Biology,2023,75:102417.
[9] YANG Q Q,CONG T C,YAO Y C,CHENG T R,YUAN C Q,ZHANG Q X.KNOX genes were involved in regulating axillary bud formation of Chrysanthemum × morifolium[J]. International Journal of Molecular Sciences,2023,24(8):7081.
[10] DAI X F,ZHAI R,LIN J J,WANG Z F,MENG D K,LI M,MAO Y L,GAO B Y,MA H Y,ZHANG B F,SUN Y,LI S,ZHOU C G,LIN Y C J,WANG J P,CHIANG V L,LI W.Celltype-specific PtrWOX4a and PtrVCS2 form a regulatory nexus with a histone modification system for stem cambium development in Populus trichocarpa[J]. Nature Plants,2023,9(1):96-111.
[11] 李真,王留强,卢孟柱.毛白杨PtoWOX11/12a 对杨树扦插苗生长发育的影响[J].林业科学,2017,53(11):69-76.LI Zhen,WANG Liuqiang,LU Mengzhu.Effects of PtoWOX11/12a gene from Populus tomentosa on the growth and development of cutting seedlings in poplar[J]. Scientia Silvae Sinicae,2017,53(11):69-76.
[12] 文爽爽,王留强,卢孟柱.银腺杨PagWOX11/12a 基因对茎生长发育的影响[J].林业科学研究,2023,36(1):39-46.WEN Shuangshuang,WANG Liuqiang,LU Mengzhu. Effects of PagWOX11/12a gene on stem growth and development of Populus alba × P. Glandulosa[J]. Forest Research,2023,36(1):39-46.
[13] YAN F,GONG Z H,HU G J,MA X S,BAI R Y,YU R N,ZHANG Q,DENG W,LI Z G,WURIYANGHAN H. Tomato SlBL4 plays an important role in fruit pedicel organogenesis and abscission[J].Horticulture Research,2021,8(1):78.
[14] ZHAO S,GAO H B,JIA X M,WEI J T,MAO K,MA F W.Md-HB-7 regulates water use efficiency in transgenic apple (Malus domestica) under long-term moderate water deficit[J]. Frontiers in Plant Science,2021,12:740492.
[15] BASSO M F,COSTA J A,RIBEIRO T P,ARRAES F B M,LOURENÇO-TESSUTTI I T,MACEDO A F,DAS NEVES M R,NARDELI S M,ARGE L W,PEREZ C E A,SILVA P L R,DE MACEDO L L P,LISEI-DE-SA M E,AMORIM R M S,DE CAMPOS P E R,SILVA M C M,MORGANTE C V,FLOH E I S,ALVES-FERREIRA M,GROSSI-DE-SA M F. Overexpression of the CaHB12 transcription factor in cotton (Gossypium hirsutum) improves drought tolerance[J]. Plant Physiology and Biochemistry,2021,165:80-93.
[16] ZHANG R X,ZHU W C,CHENG G X,YU Y N,LI Q H,HAQ S U,SAID F,GONG Z H.A novel gene,CaATHB-12,negatively regulates fruit carotenoid content under cold stress in Capsicum annuum[J].Food&Nutrition Research,2020,64:64.
[17] 饶媛媛,王晗瑶.库尔勒市香梨产业发展现状、问题及对策[J].山西农经,2024(14):192-194.RAO Yuanyuan,WANG Hanyao. The current status,problems,and strategies of the fragrant pear industry development in Korla city[J].Shanxi Agricultural Economy,2024(14):192-194.
[18] 马建江,陈久红,黄国辉.库尔勒香梨冻害发生原因及预防措施[J].果树实用技术与信息,2021(6):28-29.MA Jianjiang,CHEN Jiuhong,HUANG Guohui. The causes of freeze damage to Pyrus sinkiangensis and preventive measures[J].Practical Fruit Tree Technology and Information,2021(6):28-29.
[19] 林彩霞,吴运建,鲁晓燕,刘艳,王刚.不同冻害程度对香梨生理指标的影响[J].安徽农业科学,2015,43(10):121-123.LIN Caixia,WU Yunjian,LU Xiaoyan,LIU Yan,WANG Gang.Effect of different freezing injury degree on physiological indexes in fragrant pear[J]. Journal of Anhui Agricultural Sciences,2015,43(10):121-123.
[20] CHAN R L,GAGO G M,PALENA C M,GONZALEZ D H.Homeoboxes in plant development[J]. Biochimica Et Biophysica Acta,1998,1442(1):1-19.
[21] JAIN M,TYAGI A K,KHURANA J P.Genome-wide identification,classification,evolutionary expansion and expression analyses of homeobox genes in rice[J]. FEBS Journal,2008,275(11):2845-2861.
[22] 叶明辉,赵朋,牛洋,王冬冬,陈勤.马铃薯同源异形框基因家族的鉴定和表达分析[J].农业生物技术学报,2021,29(2):224-239.YE Minghui,ZHAO Peng,NIU Yang,WANG Dongdong,CHEN Qin. Identification and expression analysis of homeobox gene family in potato (Solanum tuberosum)[J]. Journal of Agricultural Biotechnology,2021,29(2):224-239.
[23] XIA W W,WANG S S,LIU X Y,CHEN Y F,LIN C X,LIU R N,LIU H L,LI J,ZHU J B.Chromosome-level genome provides new insight into the overwintering process of Korla pear (Pyrus sinkiangensis Yu)[J].BMC Plant Biology,2024,24(1):773.
[24] LI Y D,ZHU Y X,YAO J,ZHANG S L,WANG L,GUO C L,VAN NOCKER S,WANG X P.Genome-wide identification and expression analyses of the homeobox transcription factor family during ovule development in seedless and seeded grapes[J].Scientific Reports,2017,7(1):12638.
[25] TAMURA K,STECHER G,KUMAR S. MEGA11:Molecular evolutionary genetics analysis version 11[J]. Molecular Biology and Evolution,2021,38(7):3022-3027.
[26] XIE J M,CHEN Y R,CAI G J,CAI R L,HU Z,WANG H.Tree visualization by one table(tvBOT):A web application for visualizing,modifying and annotating phylogenetic trees[J]. Nucleic Acids Research,2023,51(W1):W587-W592.
[27] CHEN C J,WU Y,LI J W,WANG X,ZENG Z H,XU J,LIU Y L,FENG J T,CHEN H,HE Y H,XIA R.TBtools-II:A“one for all,all for one”bioinformatics platform for biological big-data mining[J].Molecular Plant,2023,16(11):1733-1742.
[28] LESCOT M,DÉHAIS P,THIJS G,MARCHAL K,MOREAU Y,VAN DE PEER Y,ROUZÉ P,ROMBAUTS S.PlantCARE,a database of plant Cis-acting regulatory elements and a portal to tools for in silico analysis of promoter sequences[J]. Nucleic Acids Research,2002,30(1):325-327.
[29] QUE F,WANG G L,LI T,WANG Y H,XU Z S,XIONG A S.Genome-wide identification,expansion,and evolution analysis of homeobox genes and their expression profiles during root development in carrot[J]. Functional & Integrative Genomics,2018,18(6):685-700.
[30] KHAN N,HU C M,KHAN W A,WANG W L,KE H,DONG H J,ZHANG Z S,HOU X L.Genome-wide identification,classification,and expression pattern of homeobox gene family in Brassica rapa under various stresses[J]. Scientific Reports,2018,8(1):16265.
[31] HOU J,SUN Y,WANG L,JIANG Y Z,CHEN N N,TONG S F. Genome-wide analysis of the homeobox gene family and identification of drought-responsive members in Populus trichocarpa[J].Plants,2021,10(11):2284.
[32] 赵双.苹果MdHB-7 和MdHB7-like 在抗旱耐盐中的功能及其机制研究[D].杨凌:西北农林科技大学,2021.ZHAO Shuang.Study on the function and mechanism of MdHB-7 and MdHB7-like in apple on drought and salt tolerance[D].Yangling:Northwest A&F University,2021.
[33] GAO Y L,MA X L,ZHANG Z X,WANG Y X. Transcription factors and plant hormones mediate wax metabolism in response to drought stress[J]. Physiologia Plantarum,2024,176(4):e14478.
[34] WEI H,WANG X,WANG K T,TANG X,ZHANG N,SI H J.Transcription factors as molecular switches regulating plant responses to drought stress[J]. Physiologia Plantarum,2024,176(3):e14366.
[35] DHATTERWAL P,SHARMA N,PRASAD M. Decoding the functionality of plant transcription factors[J]. Journal of Experimental Botany,2024,75(16):4745-4759.
[36] SINGH R K,MISKOLCZI P,MAURYA J P,BHALERAO R P.A tree ortholog of SHORT VEGETATIVE PHASE floral repressor mediates photoperiodic control of bud dormancy[J]. Current Biology,2019,29(1):128-133.
[37] ZHU Y Y,SONG D L,XU P,SUN J Y,LI L G.A HD-ZIP III gene,PtrHB4,is required for interfascicular cambium development in Populus[J]. Plant Biotechnology Journal,2018,16(3):808-817.
[38] 张馨.过表达甘蓝型油菜HB7 和HB12 基因对拟南芥生长发育的影响[D].曲阜:曲阜师范大学,2024.ZHANG Xin. The impact of overexpressing Brassica napus HB7 and HB12 genes on the growth and development of Arabidopsis thaliana[D].Qufu:Qufu Normal University,2024.