干旱胁迫是最严重的环境限制因素之一,影响植物的地理分布和生长,极大地限制了作物的产量[1]。转录因子(TF)在植物响应逆境胁迫过程中发挥着重要的调控作用,被认为是作物耐旱性遗传改良的靶标[2-3]。同源结构域-亮氨酸拉链(HD-Zip)转录因子是植物中所特有的,在植物逆境信号转导及适应中发挥着重要作用[4-6]。所有HD-Zip 转录因子都含有2个结构域,HD同源结构域和Zip结构域,根据其基因结构和功能可将其分为4 个亚家族[5]。很多证据表明,HD-Zip I 转录因子广泛参与植物对干旱胁迫的响应[6-8],在番茄中过表达HD-Zip I 转录因子ATHB7 可提高转基因番茄的耐旱性[9];过表达玉米HD-Zip I 基因ZmHDZ4 和ZmHDZ10 可降低转基因水稻的相对电导率(REL)和丙二醛(MDA)含量,从而提高转基因水稻的抗旱性[10-11];此外,HD-Zip I转录因子ZmHDZ9 通过调控脱落酸和木质素积累来提高玉米的抗旱性[12]。
枇杷(Eriobotrya japonica)是多年生常绿小乔木,秋冬开花,初夏成熟,果实酸甜适度,风味独特,是中国重要的亚热带水果之一[13]。但枇杷根系分布浅,须根稀少,对水分要求较高,而中国的枇杷园大多建在灌溉条件差的山坡上,特别容易受干旱危害,因此,季节性干旱严重影响了枇杷的生长发育、果实产量和品质[14-16]。目前有关枇杷耐旱性的研究相对较少,且针对枇杷HD-Zip I 转录因子的系统研究更是鲜见报道。鉴于HD-Zip I转录因子在植物中的重要意义,笔者在本研究中利用Jiang等[17]发布的大七星枇杷参考基因组,对枇杷HD-Zip I 转录因子家族的基因结构、蛋白结构域、共线性关系及系统发育进行分析,并通过qPCR 检测该家族成员在不同组织中及干旱处理下的表达模式,为进一步研究其在干旱下的生理功能与作用机制奠定基础,从而为开展枇杷抗旱基因工程育种提供参考。
1 材料和方法
1.1 植物材料及试验处理
试验所用植物材料的生长地点位于江苏省苏州市苏州农业职业技术学院东山校区(120°40′E,31°08′N)。干旱处理所用材料来源于江苏省珍稀树种白沙枇杷种质资源保护与培育长期科研基地的2年生白玉枇杷嫁接苗。将长势一致且种植在具有相同质量营养基质的2 年生白玉枇杷苗,置于苏州农业职业技术学院东山校区的玻璃温室中进行干旱处理。干旱处理开始前,对枇杷植株充分灌溉。浇水后计为干旱处理第0天,并收集枇杷叶片样品,然后中止浇水,于处理第3、6、9 和12 天收集枇杷叶片样品,并用于枇杷HD-Zip I 基因干旱处理下的表达分析。正常浇水的组培生根多年生冠玉枇杷的侧根、茎、成熟叶片、花和成熟的果实样品用于枇杷HDZip I 基因组织特异性表达分析。采收的样品用液氮速冻,储存在-80 ℃的冰箱,然后用于提取RNA。
1.2 RNA提取和cDNA合成
根据制造商的试剂盒说明书,使用RNAprep Pure多糖多酚植物总RNA提取试剂盒(TIANGEN)提取枇杷叶片的总RNA。根据制造商的试剂盒说明书,使用PrimeScript 第一链cDNA 合成试剂盒(TaKaRa,日本)反转录合成单链cDNA。
1.3 枇杷HD-Zip Ⅰ家族成员的序列筛选
从TAIR 网站(http://www.arabidopsis.org/)下载拟南芥中HD-Zip I基因序列。拟南芥中的HD-Zip I蛋白被用作大七星枇杷基因组数据库进行BLASTP检索的查询对象。将属于枇杷中HD-Zip I基因家族的最佳检索结果提交到保守结构域数据库(http://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi)和SMART(http://smart. emblheidelberg.de/),以检查HD-Zip结构域的存在和完整性。
1.4 枇杷HD-Zip I基因序列比对、基因结构和保守基序分析
利用DNAMAN软件对枇杷HD-Zip I家族成员的氨基酸序列进行多序列比对。利用在线Gene Structure Display Server(GSDS,http://gsds.cbi.pku.edu.ch)程序构建HD-Zip I 基因家族的外显子-内含子的结构。利用MEME 程序(http://meme-suite.org/tools/meme)预测出枇杷HD-Zip I 蛋白的保守基序(Motif),最优基序宽度设置为6~50 个氨基酸,程序设置为搜索10个Motif。
1.5 共线性和系统发育分析
使用MCScanX 对HD-Zip I 家族成员进行共线性分析,并使用TBtools对HD-Zip I基因的共线性关系进行可视化[18-19]。利用TBtools软件计算Ks(同义替换率)和Ka(非同义替代率)。使用邻接法在MEGA 10版本中构建系统发育树,bootstrap值1000。
1.6 启动子中的顺式作用元件分析
为了检测枇杷HD-Zip I基因启动子中可能存在的顺式作用元件,笔者根据枇杷基因组序列下载基因起始密码子上游2000 bp 的序列。利用Plant CARE (http://bioinformatics.psb.ugent.be/beg/tools/plantcare)预测枇杷HD-Zip I 基因启动子中潜在的顺式作用元件。
1.7 基因的表达分析
根据枇杷HD-Zip I基因的预测序列设计了特定引物(表1),用于表达分析。通过荧光实时定量PCR(qRT-PCR)方法检测枇杷HD-Zip I基因在不同组织中和干旱处理下的表达情况,所用仪器为:Bio-Rad CFX Opus 96(Bio-Rad),qRT-PCR 的反应体系和反应程序根据荧光实时定量染料说明书进行设置。EjActin 被用作内源对照基因来计算目的基因的△Ct值[20]。使用2-ΔΔCT方法计算相对表达量[21]。使用熔解曲线确定扩增片段的特异性。
表1 枇杷HD-Zip I 相对表达实时荧光定量PCR 引物
Table 1 Real-time fluorescence quantitative PCR primers for relative expression of HD-Zip I genes in loquat
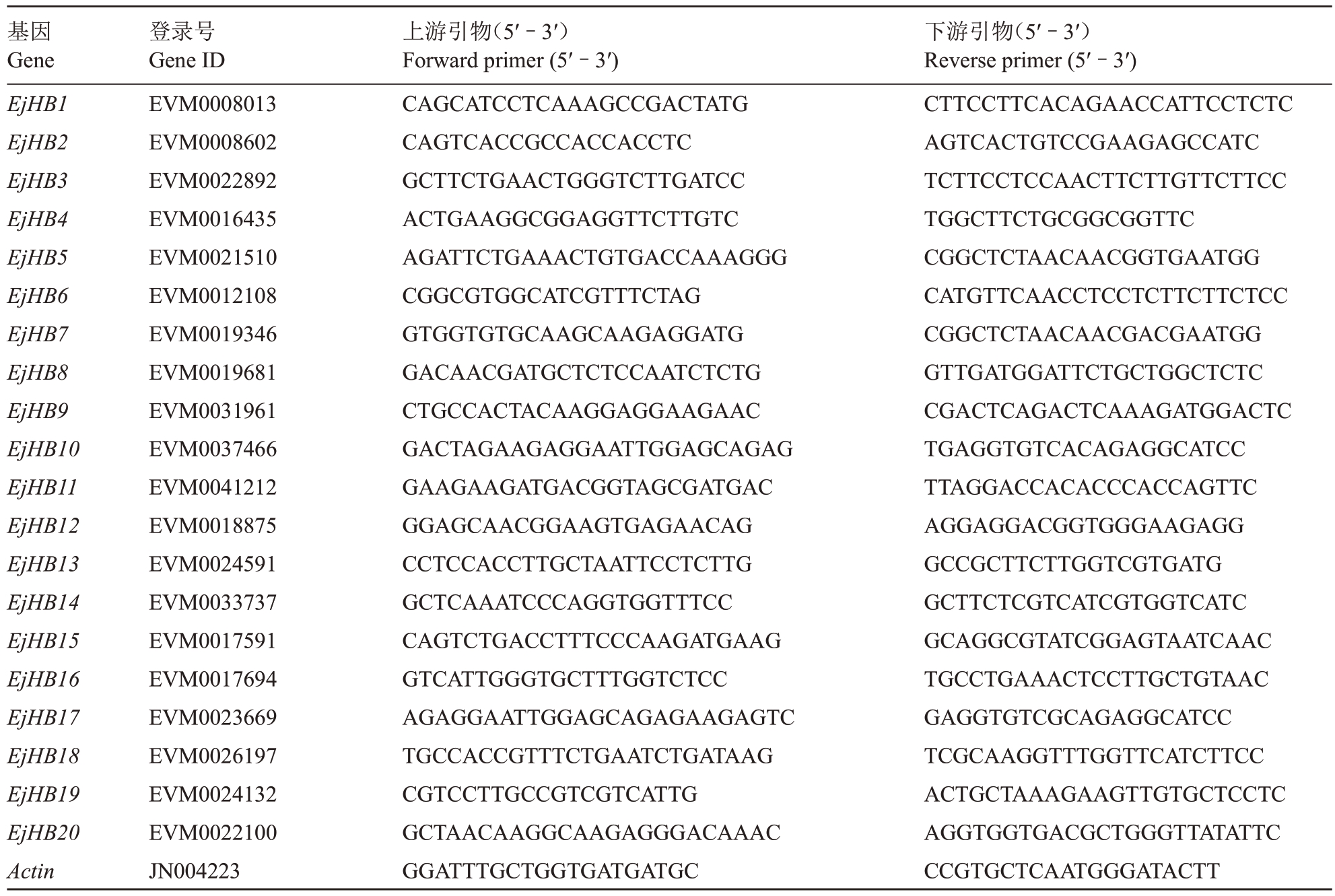
基因Gene EjHB1 EjHB2 EjHB3 EjHB4 EjHB5 EjHB6 EjHB7 EjHB8 EjHB9 EjHB10 EjHB11 EjHB12 EjHB13 EjHB14 EjHB15 EjHB16 EjHB17 EjHB18 EjHB19 EjHB20 Actin登录号Gene ID EVM0008013 EVM0008602 EVM0022892 EVM0016435 EVM0021510 EVM0012108 EVM0019346 EVM0019681 EVM0031961 EVM0037466 EVM0041212 EVM0018875 EVM0024591 EVM0033737 EVM0017591 EVM0017694 EVM0023669 EVM0026197 EVM0024132 EVM0022100 JN004223上游引物(5′-3′)Forward primer(5′-3′)CAGCATCCTCAAAGCCGACTATG CAGTCACCGCCACCACCTC GCTTCTGAACTGGGTCTTGATCC ACTGAAGGCGGAGGTTCTTGTC AGATTCTGAAACTGTGACCAAAGGG CGGCGTGGCATCGTTTCTAG GTGGTGTGCAAGCAAGAGGATG GACAACGATGCTCTCCAATCTCTG CTGCCACTACAAGGAGGAAGAAC GACTAGAAGAGGAATTGGAGCAGAG GAAGAAGATGACGGTAGCGATGAC GGAGCAACGGAAGTGAGAACAG CCTCCACCTTGCTAATTCCTCTTG GCTCAAATCCCAGGTGGTTTCC CAGTCTGACCTTTCCCAAGATGAAG GTCATTGGGTGCTTTGGTCTCC AGAGGAATTGGAGCAGAGAAGAGTC TGCCACCGTTTCTGAATCTGATAAG CGTCCTTGCCGTCGTCATTG GCTAACAAGGCAAGAGGGACAAAC GGATTTGCTGGTGATGATGC下游引物(5′-3′)Reverse primer(5′-3′)CTTCCTTCACAGAACCATTCCTCTC AGTCACTGTCCGAAGAGCCATC TCTTCCTCCAACTTCTTGTTCTTCC TGGCTTCTGCGGCGGTTC CGGCTCTAACAACGGTGAATGG CATGTTCAACCTCCTCTTCTTCTCC CGGCTCTAACAACGACGAATGG GTTGATGGATTCTGCTGGCTCTC CGACTCAGACTCAAAGATGGACTC TGAGGTGTCACAGAGGCATCC TTAGGACCACACCCACCAGTTC AGGAGGACGGTGGGAAGAGG GCCGCTTCTTGGTCGTGATG GCTTCTCGTCATCGTGGTCATC GCAGGCGTATCGGAGTAATCAAC TGCCTGAAACTCCTTGCTGTAAC GAGGTGTCGCAGAGGCATCC TCGCAAGGTTTGGTTCATCTTCC ACTGCTAAAGAAGTTGTGCTCCTC AGGTGGTGACGCTGGGTTATATTC CCGTGCTCAATGGGATACTT
1.8 数据分析
使用IBM SPSS Statistics 17.0 软件中的单因素方差分析(one-way analysis of variance,ANOVA)进行试验数据的统计分析。枇杷HD-Zip I在不同组织中的相对表达量采用Tukey 多重比较分析法分析(p<0.05)。
2 结果与分析
2.1 枇杷HD-Zip I家族成员鉴定
通过对大七星枇杷基因组数据库的搜索,共鉴定到20个假定枇杷HD-Zip I家族成员,检测了这些候选蛋白序列中HD 和Zip 结合结构域的存在和完整性,共有20 个基因被确认为HD-Zip I 家族成员,并根据HD-Zip I成员在染色体上的位置对其进行命名(表2)。HD-Zip I 蛋白的长度在215~333 个氨基酸(aa)之间,分子质量在24.82~37.50 kDa 之间,理论等电点为4.51~7.93。
表2 枇杷HD-Zip I 家族成员特征
Table 2 Characteristics of the HD-Zip I members in loquat

基因Gene EjHB1 EjHB2 EjHB3 EjHB4 EjHB5 EjHB6 EjHB7 EjHB8 EjHB9 EjHB10 EjHB11 EjHB12 EjHB13 EjHB14 EjHB15 EjHB16 EjHB17 EjHB18 EjHB19 EjHB20登录号Gene ID EVM0008013 EVM0008602 EVM0022892 EVM0016435 EVM0021510 EVM0012108 EVM0019346 EVM0019681 EVM0031961 EVM0037466 EVM0041212 EVM0018875 EVM0024591 EVM0033737 EVM0017591 EVM0017694 EVM0023669 EVM0026197 EVM0024132 EVM0022100染色体定位Chromosome location LG03:10186645-10187781 LG03:23204370-23206151 LG03:32463730-32465702 LG03:40454690-40456201 LG07:38102798-38104488 LG07:38355270-38356769 LG08:35330188-35331862 LG08:35618390-35619865 LG10:23076229-23077228 LG10:36015001-36015943 LG11:2228092-2231357 LG11:3041533-3043439 LG13:32607861-32609606 LG13:35699824-35702856 LG14:32656733-32658402 LG15:7582830-7583956 LG16:1018672-1019642 LG16:12437868-12438873 LG16:21367039-21368861 LG17:16254559-16255822氨基酸数量Number of amino acids/aa 274 329 215 324 327 281 333 286 242 233 332 303 304 332 324 275 236 243 329 231分子质量MW/kDa 31.11 37.18 24.82 36.54 37.07 32.07 37.46 32.53 27.90 27.00 37.50 34.43 34.41 37.34 36.34 31.02 27.39 27.76 37.19 27.03等电点pI 6.21 5.28 6.37 4.69 4.80 6.05 4.51 6.19 4.69 5.79 4.91 6.80 6.71 4.96 4.63 5.40 5.32 4.90 4.93 7.93内含子/外显子Intron/Exon 2/3 2/3 2/3 2/3 2/3 2/3 2/3 2/3 1/2 1/2 3/4 2/3 2/3 3/4 2/3 2/3 1/2 1/2 2/3 2/3
2.2 枇杷HD-Zip I基因的共线性和系统发育分析
如图1 所示,HD-Zip I 家族基因在枇杷10 条染色体上随机分布。4 个HD-Zip I家族成员位于第3条染色体上,3 个HD-Zip I 家族成员位于第16 条染色体上,2个HD-Zip I家族成员位于第7、8、10、11和13 染色体上,1 个HD-Zip I 家族成员位于第14、15、和17 染色体上。笔者在枇杷基因组内进行了共线性分析,发现HD-Zip I 家族中存在25 个重复基因对,其中3对串联复制基因和22对片段复制基因(图1)。HD-Zip I重复对的核苷酸序列一致性在38.11%~93.71%之间,Ka/Ks 比值在0.08~0.43 之间(表3)。这些结果表明,不同基因之间的差异主要是由纯化选择驱动的。重复对的Ks 值变化范围为0.13~2.30(表3),表明基因对两两之间的进化速率不同。

图1 染色体定位及同线性分析
Fig.1 Chromosomal distribution and synteny analyses
表3 枇杷HD-Zip I 家族中鉴定的基因对
Table 3 HD-Zip Ⅰduplicates identified in loquat
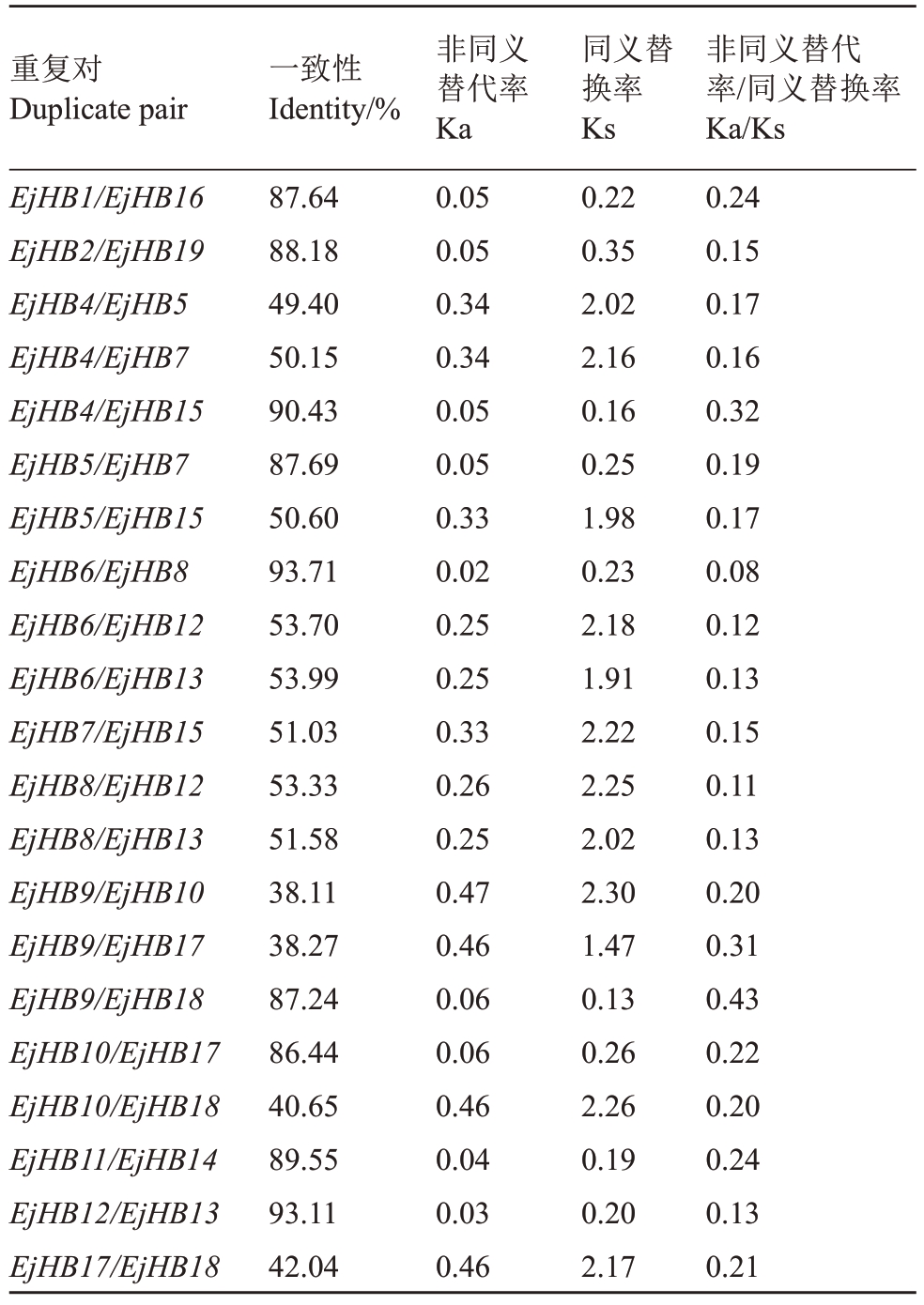
重复对Duplicate pair EjHB1/EjHB16 EjHB2/EjHB19 EjHB4/EjHB5 EjHB4/EjHB7 EjHB4/EjHB15 EjHB5/EjHB7 EjHB5/EjHB15 EjHB6/EjHB8 EjHB6/EjHB12 EjHB6/EjHB13 EjHB7/EjHB15 EjHB8/EjHB12 EjHB8/EjHB13 EjHB9/EjHB10 EjHB9/EjHB17 EjHB9/EjHB18 EjHB10/EjHB17 EjHB10/EjHB18 EjHB11/EjHB14 EjHB12/EjHB13 EjHB17/EjHB18一致性Identity/%87.64 88.18 49.40 50.15 90.43 87.69 50.60 93.71 53.70 53.99 51.03 53.33 51.58 38.11 38.27 87.24 86.44 40.65 89.55 93.11 42.04非同义替代率Ka 0.05 0.05 0.34 0.34 0.05 0.05 0.33 0.02 0.25 0.25 0.33 0.26 0.25 0.47 0.46 0.06 0.06 0.46 0.04 0.03 0.46同义替换率Ks 0.22 0.35 2.02 2.16 0.16 0.25 1.98 0.23 2.18 1.91 2.22 2.25 2.02 2.30 1.47 0.13 0.26 2.26 0.19 0.20 2.17非同义替代率/同义替换率Ka/Ks 0.24 0.15 0.17 0.16 0.32 0.19 0.17 0.08 0.12 0.13 0.15 0.11 0.13 0.20 0.31 0.43 0.22 0.20 0.24 0.13 0.21
为了进一步研究不同植物物种中HD-Zip I家族成员之间的系统发育关系,对枇杷、苹果、拟南芥和水稻中的HD-Zip I 蛋白序列构建了系统发育树(图2)。HD-Zip I 蛋白被分为9 个分支,分别是α,β1,β 2,γ,δ,ε,φ1,φ2 和ζ,其中φ1 分支和ζ 分支分别只含有拟南芥和水稻的家族成员,与苹果、枇杷的HDZip I 基因不聚在一起。枇杷HD-Zip I 各分支成员与苹果的同系物聚集更近,与水稻的同系物相距较远。另外,枇杷、苹果、拟南芥和水稻在α 分支的成员最多,其次是γ分支。
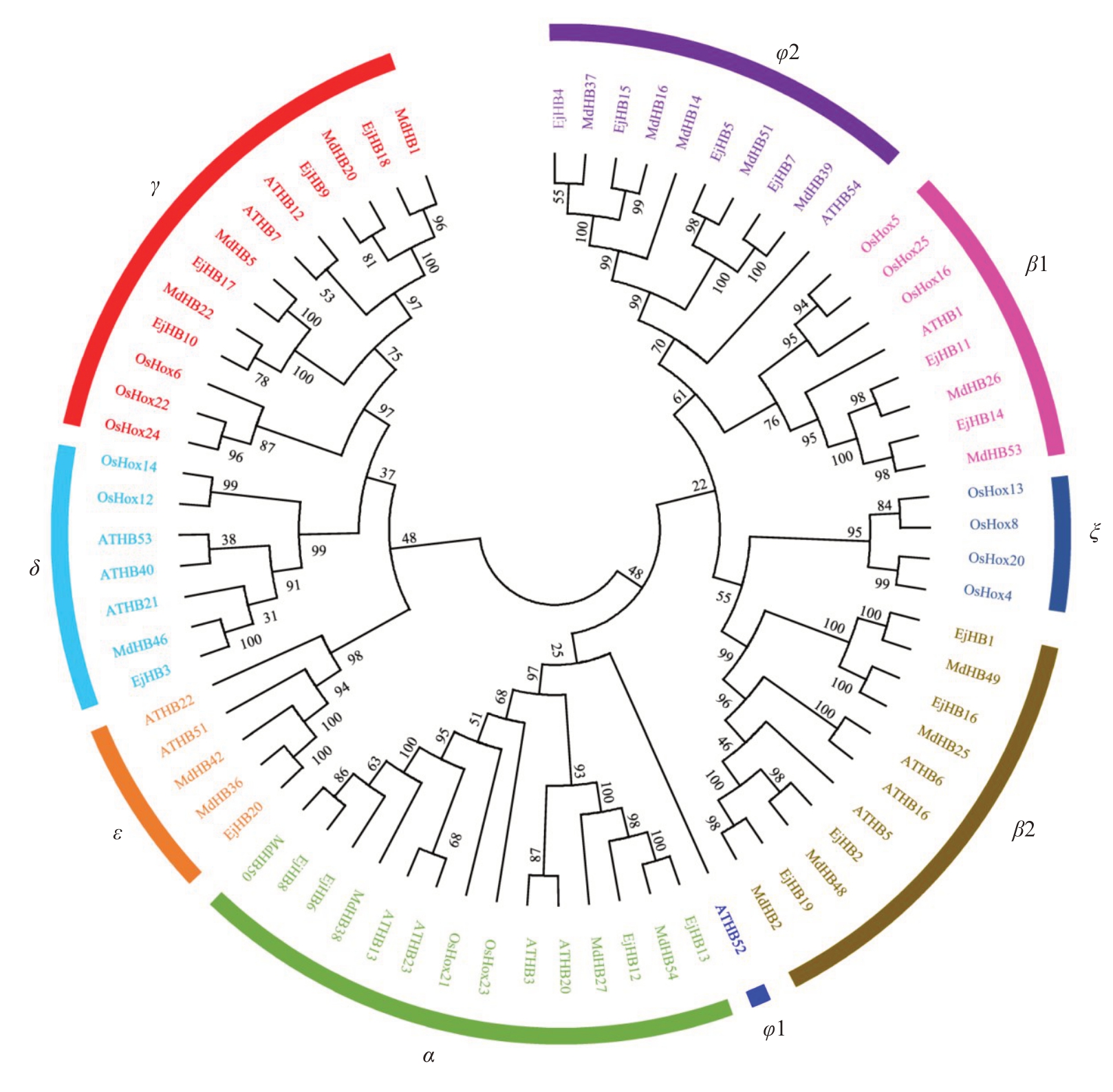
图2 枇杷、苹果、拟南芥和水稻中HD-Zip Ⅰ蛋白系统发育分析
Fig.2 Phylogenetic analysis of HD-Zip Ⅰprotein in loquat,apple,Arabidopsis and rice
2.3 枇杷HD-Zip I蛋白的多重序列比对、基因结构和Motif分析
如图3 所示,使用DNAMAN 软件对枇杷20 个HD-Zip I蛋白进行多重序列比对,发现所有的HDZip I 蛋白都具有HD 和Zip 保守结构域。为了进一步研究枇杷HD-Zip I 蛋白之间的关系,构建了所有枇杷HD-Zip I蛋白序列的系统发育树,并分析了他们的基因结构和Motif(图4)。与上述系统发育分析的结果相似,枇杷HD-Zip I 基因家族被分为7 个分支:α,β1,β2,γ,δ,ε和φ2(图4-A)。由于基因的内含子-外显子结构在多基因家族的进化中起至关重要的作用[22],笔者研究了20 个枇杷HD-Zip I 基因的内含子-外显子结构,以更好地了解其结构进化。γ 分支成员有1个内含子,β1分支成员有3个内含子,其他分支成员含有2 个内含子(图4-B)。结合系统进化分析,笔者发现同一分支的基因具有相似的内含子-外显子结构,而不同分支的内含子-外显子结构存在差异。这一结果表明,基因结构是枇杷HD-Zip I家族基因进化的重要组成部分。

图3 HD-Zip Ⅰ蛋白序列比对
Fig.3 Multiple sequence alignment of the HD-Zip Ⅰprotein
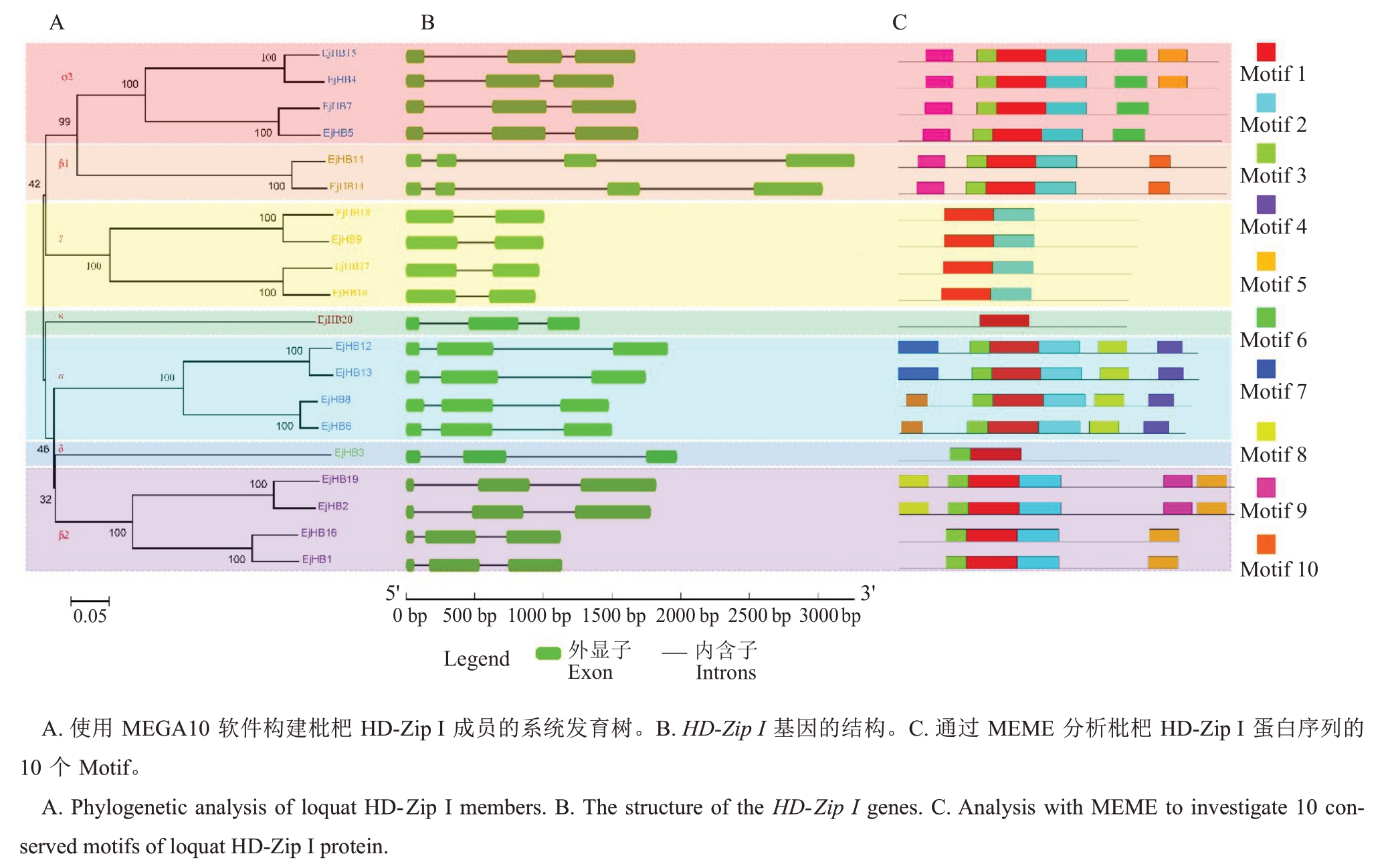
图4 HD-Zip Ⅰ成员的系统发育树、基因结构和蛋白保守序列元件
Fig.4 Phylogenetic tree,gene structure and protein motif analysis of HD-Zip Ⅰmembers
为了深入了解枇杷HD-Zip I 蛋白的差异和功能,笔者对其Motif 进行了鉴定。笔者鉴定了10 个Motif,宽度从20到50个残基不等(表4)。所有预测的Motif 在每个HD-Zip I 蛋白中仅识别一次(图4-C)。除Motif 1在所有HD-Zip I蛋白中都存在外,其余9个Motif只存在于特定的分支中。例如,Motif 4只存在于α 分支中,Motif 10 只存在于β1 分支中。这与枇杷HD-Zip I 蛋白的系统发育一致,表明来自同一分支的HD-Zip I具有相似的氨基酸序列和相似的Motif,暗示他们也具有相似的功能。
表4 HD-Zip I 蛋白保守基序及其氨基酸序列
Table 4 Conserved motifs and amino acid sequences in HD-Zip Ⅰproteins

保守基序Motif 123456789 10宽度Width 50 41 20 25 29 32 41 28 21 29氨基酸序列The amino acid sequences LEKSFEVENKLEPERKVQLAKELGLQPRQVAIWFQNRRARWKTKQLERDY DVLKANYDALKABYDSLQKENQKLKAZVQKLKDKLGSEEEG DEGEQVPEKKRRLTAEQVKA DLQCQKIDQMIKDEGLCNMFNGIDD LVKVEEQSFFSTDEACNFFSDEQAPSLHW VSKVAAVVSKQEDASSGKSDIFDSDSPHYTDG MMAFPPCHSFMFQNHEDPDLHLANSSCPPQHFNGGGMPFMM SDALESLFLNNPSSSFLGSRSMVSFEDV FFPTNFHLQYPHDHDDHQPPT SDRHENNDDLGLEISRTPATDSPPPSNNH
2.4 枇杷HD-Zip I家族成员在不同组织中的表达
为了研究HD-Zip I家族成员在枇杷不同组织中的表达模式,对HD-Zip I在枇杷根、茎、叶、花和果实中的表达进行分析。结果表明,EjHB3、EjHB6、EjHB8、EjHB15 和EjHB20 主要在叶中表达,EjHB9、EjHB16和EjHB18主要在根中表达。多数成员在茎中具有较高的表达水平,在果实中具有较低的表达水平。此外,EjHB10、EjHB11、EjHB12、EjHB13、EjHB14 和EjHB17 在花中的表达量高于其他组织,而其他成员在花中的表达量也普遍较低(图5)。
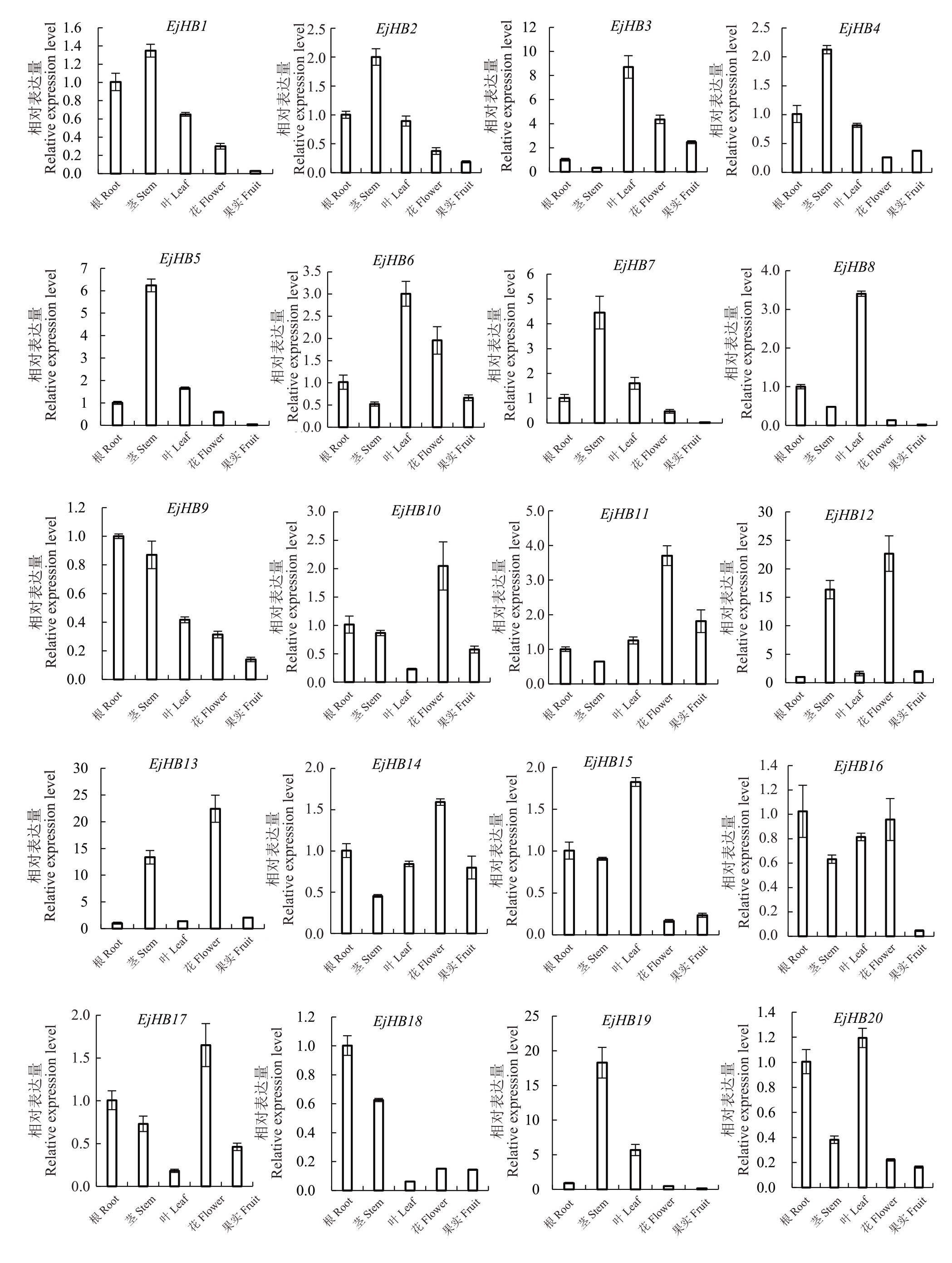
图5 枇杷HD-Zip Ⅰ基因在不同组织中的表达分析
Fig.5 Expression analysis of different tissues of HD-Zip Ⅰgenes in loquat
2.5 枇杷HD-Zip I 家族成员启动子分析和在干旱处理下的表达分析
启动子顺式调控元件对调控基因表达具有重要意义。通过PlantCARE 数据库(图6)鉴定了HDZip I启动子中推定的顺式元件(从假定翻译起始位点上游<2 kb),并鉴定了与胁迫和激素反应相关的顺式作用元件。大多数HD-Zip I启动子含有ABRE元件,该元件通常参与ABA相关的反应。启动子中还含有干旱诱导元件(MBS)、防御和胁迫响应元件(TC-rich repeats)。此外,还有许多顺式元件与胁迫和胁迫相关激素信号的响应有关,如MYB、MYC、SA、MeJA。MBS 和ABRE 等干旱响应相关顺式元件的存在,表明HD-Zip I 可能在枇杷干旱胁迫响应中发挥重要作用。

图6 枇杷HD-Zip Ⅰ基因启动子的顺式作用元件预测
Fig.6 Prediction of cis-acting elements of HD-Zip Ⅰgenes promoter in loquat
HD-Zip I家族包含与干旱胁迫相关的顺式作用元件。为了鉴定HD-Zip I在枇杷抗旱性调控中发挥的作用,笔者分析了干旱胁迫下20个HD-Zip I基因的表达情况(图7)。研究表明,干旱处理后,γ 分支的EjHB9、EjHB10、EjHB17、EjHB18 和ε 分支的EjHB20的表达水平大幅度升高,β2分支的EjHB2和EjHB19受干旱影响显著下调。
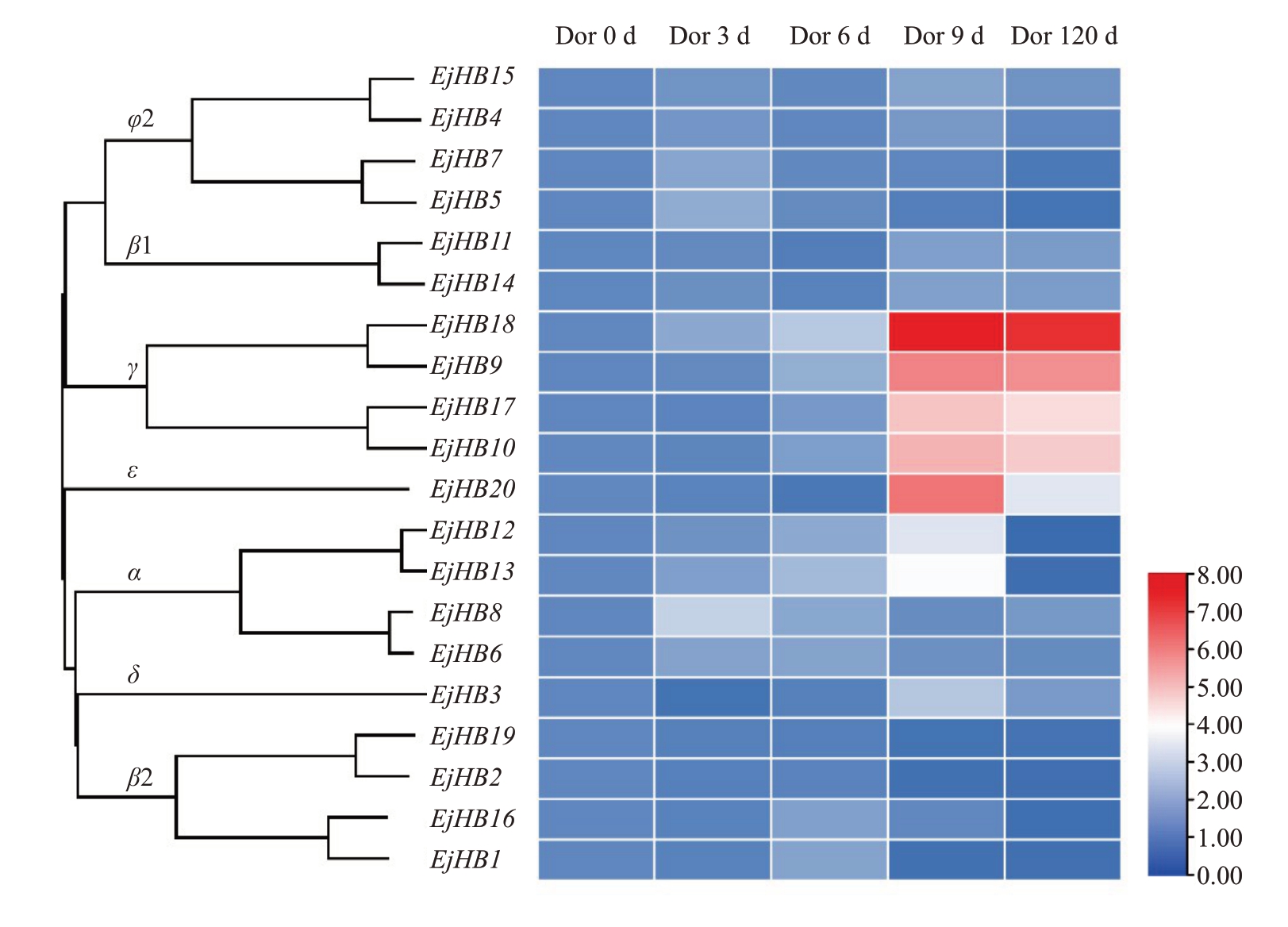
图7 枇杷HD-Zip Ⅰ基因对干旱胁迫响应的表达分析
Fig.7 Expression analysis of drought stress response of HD-Zip Ⅰgenes in loquat
3 讨 论
HD-Zip I家族的鉴定和全基因组分析已经在各种物种中进行,并且HD-Zip I 基因的数量在物种间差异显著。在番茄[23]、梨[24]、葡萄[25]、毛竹[26]、木薯[27]、水稻[28]和小麦[29]中分别鉴定出22、18、11、17、23、14和20 个HD-Zip I 基因。笔者在本研究中对枇杷中的HD-Zip I基因进行了全基因组普查,共鉴定出20个HD-Zip I 家族基因,包括7 个分支。与拟南芥等其他植物相比,HD-Zip I 家族成员在枇杷中较为丰富。
串联复制和片段复制是基因家族扩张的主要机制。基因复制事件在不同物种中产生了许多旁系同源基因对,因此在基因家族的快速扩张和进化中发挥了重要作用[30]。本研究结果表明,片段重复是枇杷HD-Zip I家族扩张的主要方式。此外,22对HDZip I 重复对的Ka/Ks 比值表明,枇杷HD-Zip I 基因家族在进化过程中主要经历了纯化选择。
研究表明,拟南芥中的HD-Zip I 基因被划分为8 个分支(α,β1,β2,γ,δ,ε,φ1 和φ2)[28],在水稻、玉米、大豆和小麦中还含有ζ 分支[28-29]。在本研究中,系统发育分析表明,枇杷的20个HD-Zip I蛋白被分为7个分支,不含有φ1和ζ分支,表明枇杷在进化过程中失去了这两个分支的成员。相同的分支拥有相似的内含子-外显子组织结构。结合系统发育树,内含子-外显子结构分析使笔者能够进一步区分属于不同分支的基因。但保守基序分析发现,β2和φ2分支中的成员的保守基序存在一定差异。
植物通过诱导胁迫相关基因表达来适应环境胁迫,如干旱,许多HD-Zip I家族基因的表达已被证明受干旱胁迫调控,并在干旱胁迫响应中扮演重要的角色[31]。笔者对枇杷HD-Zip I家族基因的启动子顺式作用元件的分析发现,枇杷HD-Zip I 基因的启动子中含有多个与干旱胁迫相关的顺式作用元件,因此笔者研究了枇杷HD-Zip I基因在干旱胁迫响应中的表达模式。结果表明,大多数枇杷HD-Zip I 基因响应干旱胁迫,且γ 分支成员在干旱胁迫下被强烈诱导,这一结果与拟南芥、小麦、桑树和苹果中该家族基因的研究结果相同[32-34]。研究发现,HD-Zip I转录因子γ 分支成员AtHB12 受干旱胁迫诱导表达[32],且AtHB12 过表达增强了转基因拟南芥对干旱胁迫的耐受性[35]。HD-Zip I转录因子γ分支成员MdHB-7和MdHB7-like 通过调节苹果的气孔密度提高了转基因苹果植株对干旱的适应性和水分利用效率[36-37]。这表明该分支的成员可能在干旱胁迫响应中发挥重要作用。
4 结 论
首次从枇杷全基因组中鉴定了HD-Zip I转录因子,启动子预测分析表明HD-Zip I 转录因子响应干旱胁迫。对HD-Zip I家族基因在干旱处理后的表达进行了分析,结果表明,EjHB9、EjHB10、EjHB17、EjHB18 和EjHB20 在干旱胁迫下表达量均显著上调,因此推测这些基因可能在干旱胁迫响应中发挥重要作用。
[1] QIN T,TIAN Q Z,WANG G F,XIONG L M. LOWER TEMPERATURE 1 enhances ABA responses and plant drought tolerance by modulating the stability and localization of C2-domain ABA-related proteins in Arabidopsis[J]. Molecular Plant,2019,12(9):1243-1258.
[2] CENTURY K,REUBER T L,RATCLIFFE O J. Regulating the regulators:The future prospects for transcription-factor-based agricultural biotechnology products[J]. Plant Physiology,2008,147(1):20-29.
[3] MAO H D,LI S M,WANG Z X,CHENG X X,LI F F,MEI F M,CHEN N,KANG Z S. Regulatory changes in TaSNAC8-6A are associated with drought tolerance in wheat seedlings[J].Plant Biotechnology Journal,2020,18(4):1078-1092.
[4] SCHENA M,DAVIS R W.HD-Zip proteins:Members of an Arabidopsis homeodomain protein superfamily[J]. Proceedings of the National Academy of Sciences of the United States of America,1992,89(9):3894-3898.
[5] ARIEL F D,MANAVELLA P A,DEZAR C A,CHAN R L.The true story of the HD-Zip family[J]. Trends in Plant Science,2007,12(9):419-426.
[6] GONG S H,DING Y F,HU S S,DING L H,CHEN Z X,ZHU C. The role of HD-Zip class I transcription factors in plant response to abiotic stresses[J]. Physiologia Plantarum,2019,167(4):516-525.
[7] 郝陆洋,张晓静,高晨曦,张登峰,李永祥,李春辉,宋燕春,石云素,王天宇,刘旭洋,黎裕.玉米HD-Zip 转录因子基因Zmhdz6 的克隆与功能分析[J].植物遗传资源学报,2022,23(3):823-831.HAO Luyang,ZHANG Xiaojing,GAO Chenxi,ZHANG Dengfeng,LI Yongxiang,LI Chunhui,SONG Yanchun,SHI Yunsu,WANG Tianyu,LIU Xuyang,LI Yu. Cloning and functional analysis of HD-Zip transcription factor gene Zmhdz6 in maize[J].Journal of Plant Genetic Resources,2022,23(3):823-831.
[8] 胡景涛,李彦杰,段艳艳,阮宇,顾欣,肖国生.桑树HD-Zip Ⅰ亚家族基因的鉴定及表达分析[J].林业科学研究,2022,35(4):130-142.HU Jingtao,LI Yanjie,DUAN Yanyan,RUAN Yu,GU Xin,XIAO Guosheng. Identification and expression analysis of the HD-Zip Ⅰsubfamily genes in mulberry[J]. Forest Research,2022,35(4):130-142.
[9] MISHRA K B,IANNACONE R,PETROZZA A,MISHRA A,ARMENTANO N,LA VECCHIA G,TRTÍLEK M,CELLINI F,NEDBAL L. Engineered drought tolerance in tomato plants is reflected in chlorophyll fluorescence emission[J].Plant Science,2012,182:79-86.
[10] ZHAO Y,MA Q,JIN X L,PENG X J,LIU J Y,DENG L,YAN H W,SHENG L,JIANG H Y,CHENG B J.A novel maize homeodomain-leucine zipper (HD-Zip) I gene,Zmhdz10,positively regulates drought and salt tolerance in both rice and Arabidopsis[J].Plant Cell Physiology,2014,55(6):1142-1156.
Cell Physiology,2014,55(6):1142-1156.
[11] WU J D,ZHOU W,GONG X F,CHENG B J. Expression of ZmHDZ4,a maize homeodomain-leucine zipper I gene,confers tolerance to drought stress in transgenic rice[J]. Plant Molecular Biology Reporter,2016,34(4):845-853.
[12] JIAO P,JIANG Z Z,MIAO M,WEI X T,WANG C L,LIU S Y,GUAN S Y,MA Y Y. Zmhdz9,an HD-Zip transcription factor,promotes drought stress resistance in maize by modulating ABA and lignin accumulation[J]. International Journal of Biological Macromolecules,2024,258:128849.
[13] 赵双,尤伟忠,黄颖宏,郄红丽.基于主成分分析综合评价23个白沙枇杷品种果实品质[J].中国南方果树,2023,52(6):114-118.ZHAO Shuang,YOU Weizhong,HUANG Yinghong,QIE Hongli. Comprehensive evaluation of fruit quality of 23 white flesh loquats based on principal component analysis[J]. South China Fruits,2023,52(6):114-118.
[14] 罗华建,刘星辉.干旱对枇杷生长的影响[J].中国南方果树,2004,33(3):26-27.LUO Huajian,LIU Xinghui. The impact of drought on loquat growth[J].South China Fruits,2004,33(3):26-27.
[15] 王剑毛,郭亚端,刘进平,王一迪,张燕.干旱胁迫对“早钟6号”枇杷幼苗生长及生理特性的影响[J]. 中国南方果树,2018,47(6):34-38.WANG Jianmao,GUO Yaduan,LIU Jinping,WANG Yidi,ZHANG Yan. The effects of drought stress on the growth and physiological characteristics of“Zaozhong 6”loquat seedlings[J].South China Fruits,2018,47(6):34-38.
[16] WANG D,CHEN Q Y,CHEN W W,GUO Q G,XIA Y,WANG S M,JING D L,LIANG G L. Physiological and transcription analyses reveal the regulatory mechanism of melatonin in inducing drought resistance in loquat (Eriobotrya japonica Lindl.)seedlings[J]. Environmental and Experimental Botany,2021,181:104291.
[17] JIANG S,AN H S,XU F J,ZHANG X Y.Chromosome-level genome assembly and annotation of the loquat (Eriobotrya japonica)genome[J].GigaScience,2020,9(3):giaa015.
[18] WANG Y P,TANG H B,DEBARRY J D,TAN X,LI J P,WANG X Y,LEE T H,JIN H Z,MARLER B,GUO H,KISSINGER J C,PATERSON A H.MCScanX:A toolkit for detection and evolutionary analysis of gene synteny and collinearity[J].Nucleic Acids Research,2012,40(7):e49.
[19] ZHAO S,GAO H B,LUO J W,WANG H B,DONG Q L,WANG Y P,YANG K Y,MAO K,MA F W.Genome-wide analysis of the light-harvesting chlorophyll a/b-binding gene family in apple (Malus domestica) and functional characterization of MdLhcb4.3,which confers tolerance to drought and osmotic stress[J]. Plant Physiology and Biochemistry,2020,154:517-529.
[20] 李晓颖,徐红霞,陈俊伟.枇杷WRKY 转录因子鉴定与表达分析[J].园艺学报,2019,46(5):939-954.LI Xiaoying,XU Hongxia,CHEN Junwei.Identification and expression analysis of WRKY transcription factors in Eriobotrya japonica[J].Acta Horticulturae Sinica,2019,46(5):939-954.
[21] LIVAK K J,SCHMITTGEN T D.Analysis of relative gene expression data using real-time quantitative PCR and the 2- Δ Δ CT method[J].Methods,2001,25(4):402-408.
[22] XU G X,GUO C C,SHAN H Y,KONG H Z.Divergence of duplicate genes in exon-intron structure[J]. Proceedings of the National Academy of Sciences of the United States of America,2012,109(4):1187-1192.
[23] ZHANG Z Z,CHEN X L,GUAN X,LIU Y,CHEN H Y,WANG T T,MOUEKOUBA L D O,LI J F,WANG A X.A genome-wide survey of homeodomain-leucine zipper genes and analysis of cold-responsive HD-Zip I members'expression in tomato[J].Bioscience,Biotechnology,and Biochemistry,2014,78(8):1337-1349.
[24] WANG H,LIN J,LI X G,CHANG Y H.Genome-wide identification of pear HD-Zip gene family and expression patterns under stress induced by drought,salinity,and pathogen[J]. Acta Physiologiae Plantarum,2015,37(9):189.
[25] JIANG H Y,JIN J,LIU H,DONG Q,YAN H W,GAN D F,ZHANG W,ZHU S W.Genome-wide analysis of HD-Zip genes in grape (Vitis vinifera)[J]. Tree Genetics  Genomes,2014,11(1):827.
Genomes,2014,11(1):827.
[26] CHEN D M,CHEN Z,WU M,WANG Y,WANG Y J,YAN H W,XIANG Y.Genome-wide identification and expression analysis of the HD-zip gene family in moso bamboo (Phyllostachys edulis)[J].Journal of Plant Growth Regulation,2017,36(2):323-337.
[27] DING Z H,FU L L,YAN Y,TIE W W,XIA Z Q,WANG W Q,PENG M,HU W,ZHANG J M. Genome-wide characterization and expression profiling of HD-Zip gene family related to abiotic stress in cassava[J].PLoS One,2017,12(3):e0173043.
[28] AGALOU A,PURWANTOMO S,OVERNÄS E,JOHANNESSON H,ZHU X Y,ESTIATI A,DE KAM R J,ENGSTRÖM P,SLAMET-LOEDIN I H,ZHU Z,WANG M,XIONG L Z,MEIJER A H,OUWERKERK P B F.A genome-wide survey of HDZip genes in rice and analysis of drought-responsive family members[J].Plant Molecular Biology,2008,66(1/2):87-103.
[29] YUE H,SHU D T,WANG M,XING G W,ZHAN H S,DU X H,SONG W N,NIE X J. Genome-wide identification and expression analysis of the HD-Zip gene family in wheat (Triticum aestivum L.)[J].Genes,2018,9(2):70.
[30] CANNON S B,MITRA A,BAUMGARTEN A,YOUNG N D,MAY G.The roles of segmental and tandem gene duplication in the evolution of large gene families in Arabidopsis thaliana[J].BMC Plant Biology,2004,4:10.
[31] 关淑艳,魏小童,焦鹏,蒋振忠,曲静,刘思言,马义勇.HD-Zip I转录因子在植物非生物胁迫中的研究进展[J].吉林农业大学学报,2022,44(2):127-134.GUAN Shuyan,WEI Xiaotong,JIAO Peng,JIANG Zhenzhong,QU Jing,LIU Siyan,MA Yiyong.HD-Zip I transcription factors in plant abiotic stress[J].Journal of Jilin Agricultural University,2022,44(2):127-134.
[32] RÉ D A,CAPELLA M,BONAVENTURE G,CHAN R L.Arabidopsis AtHB7 and AtHB12 evolved divergently to fine tune processes associated with growth and responses to water stress[J].BMC Plant Biology,2014,14:150.
[33] HARRIS J C,SORNARAJ P,TAYLOR M,BAZANOVA N,BAUMANN U,LOVELL B,LANGRIDGE P,LOPATO S,HRMOVA M. Molecular interactions of the γ-clade homeodomainleucine zipper class I transcription factors during the wheat response to water deficit[J]. Plant Molecular Biology,2016,90(4/5):435-452.
[34] ZHAO S,GAO H B,JIA X M,WANG H B,KE M,MA F W.The HD-Zip I transcription factor MdHB-7 regulates drought tolerance in transgenic apple (Malus domestica)[J]. Environmental and Experimental Botany,2020,180:104246.
[35] ROMANI F,RIBONE P A,CAPELLA M,MIGUEL V N,CHAN R L. A matter of quantity:Common features in the drought response of transgenic plants overexpressing HD-Zip I transcription factors[J].Plant Science,2016,251:139-154.
[36] ZHAO S,GAO H B,JIA X M,WEI J T,MAO K,MA F W.Md-HB-7 regulates water use efficiency in transgenic apple (Malus domestica) under long-term moderate water deficit[J]. Frontiers in Plant Science,2021,12:740492.
[37] ZHAO S,GAO H B,JIA X M,ZHOU K,WANG H B,MAO K,MA F W. MdHB7-like confers drought tolerance and improves water-use efficiency through modulating stomatal density in apple (Malus domestica)[J]. Scientia Horticulturae,2022,294:110758.