AP2/ERF(APETALA2/Ethylene responsive fac‐tor)家族转录因子是植物特有的一类转录因子,以AP2保守结构域为特征。AP2/ERF家族转录因子主要分为AP2(APETALA2)、ERF(Ethylene-respon‐sive-element-bindingprotein)、CBF/DREB(Dehydra‐tion responsive element binding)、RAV(Related to ABI3/VP)和Soloist 5 个亚家族[1]。其中AP2 亚家族转录因子蛋白序列含有2 个AP2 结构域;ERF 和CBF/DREB亚家族均只含有1个AP2结构域;RAV亚家族转录因子含有1个AP2结构域和1个B3结构域;Soloist 亚家族仅含有单一AP2 结构域,但其序列和结构与其他四个亚家族成员不同,因此独立分为1个亚家族[2]。AP2 亚家族成员根据核定位和氨基酸序列以及其他保守基序进一步细分为euAP2、basalANT和euANT 三个分支[3]。AP2 亚家族转录因子可以与下游基因启动子上富含T/A的元件结合,并通过第二个AP2 结构域直接激活下游基因表达[4]。据报道,AP2亚家族基因主要参与响应非生物胁迫和调节植物各器官生长发育[5]。例如:杜丹(Paeonia×suffruti‐cosa Andr.)PoBBM在体细胞胚胎发育中具有重要作用,在种子、愈伤组织和叶中的表达量最高[6];大豆(Glycine max)GmBBM7调控体细胞胚胎发生和根系生长,并可以通过激素途径增加愈伤组织的形成率和形成密度[7];小麦(Triticum aestivum L.)TaAP2-10正向调节小麦条锈病的抗性[8];辣椒(Capsicum annuum L.)CaAIL1通过与下游基因启动子中的不同顺式元件结合来抑制负免疫调节因子,从而正向调节辣椒对青枯雷尔氏菌(Ralstonia solanacearum infection,RSI)的免疫力[9];杂交枫香(Liquidambar styraciflua\times Liquidambar formosana)AIL1、AIL5 在从愈伤组织到体细胞胚胎发生过程中起着重要作用[10];拟南芥(Ara‐bidopsis thaliana)AtANT和AtAIL6调节发育中的花器官的生长和形态建成[11];AtAIL5、AtAIL6 和AtAIL7 在拟南芥ant 突变体中具有改变花器官定位和生长的功能,从而导致萼片融合和花瓣数量减少[12];盐穗木(Halostachys caspica)HcTOE3正向调节植物冷冻胁迫的耐受性,且通过上调冷响应基因和脱落酸信号转导通路基因的转录水平来提高植物冷冻耐受性[13];牡丹(Paeonia lactiflora Pall.)PlTOE3 直接调控色氨酸脱羧酶基因表达,增强牡丹耐高温性[14]。
甜橙是中国乃至全球第一大柑橘种类,探究甜橙非生物胁迫的响应机制,对促进甜橙的经济效益提升具有重要意义。在本研究中采用生物信息学的方法鉴定和分析了甜橙AP2亚家族的14个基因,并采用荧光定量PCR(quantitative real-time PCR,qRT-PCR)分析甜橙AP2亚家族基因在不同组织部位和非生物胁迫下的基因表达差异。研究为后续验证甜橙AP2亚家族基因响应非生物胁迫的功能奠定了基础。
1 材料和方法
1.1 甜橙AP2亚家族基因鉴定与分析
甜橙(Citrus sinensis)基因组数据来源于柑橘基因组数据库(http://citrus.hzau.edu.cn/index.php)[15]。拟南芥AP2 亚家族序列来源于拟南芥(Arabidopsis thaliana)数据库(https://www.arabidopsis.org/)。利用以下方法鉴定AP2亚家族基因。首先,从Pfam 数据库(https://pfam- docs.readthedocs.io/en/latest/search‐ing-pfam.html)下载AP2 结构域序列(PF00847)[16]。其次,使用TBtools(v1.09867+),以PF00847 为模型从甜橙基因组搜索出AP2 亚家族候选基因。利用CDD 数据库(https://www.ncbi.nlm.nih.gov/cdd)验证序列中双AP2 结构域的存在,确定甜橙AP2 亚家族基因。利用ExPASy 网站(https://www.expasy.org/)对甜橙AP2 亚家族蛋白进行二级结构预测和理化性质分析[17]。利用WOLF-PSORT(https://wolfpsort.hgc.jp/)对该家族蛋白进行亚细胞定位分析[18]。
1.2 甜橙AP2亚家族蛋白系统进化分析
水稻(Oryza sativa)、克里曼丁橘(C. clementi‐na)和苹果(Malus pumila)AP2亚家族蛋白序列来源于基因组数据库(https://asia.ensembl.org/index.ht‐ml)[19]。通过MEGA X 软件中的邻接法(Neighborjoining)进行系统进化树的构建,参数设置默认[20]。并通过Evolview(https://www.evolgenius.info/help/)进行美化。
1.3 甜橙AP2亚家族蛋白结构与基序分析
利用Jalview 软件对甜橙AP2 亚家族蛋白的双AP2 域进行可视化。使用MEME(https://memesuite.org/meme/)对甜橙AP2亚家族蛋白的基序进行分析,其中基序最大值为10[21]。
1.4 甜橙AP2 亚家族基因启动子顺式作用元件分析与miRNA预测
利用Tbtools 提取甜橙AP2 亚家族基因起始密码子上游2 kb序列,具体参照马青龄等[16]的方法。
1.5 甜橙AP2亚家族基因染色体定位和共线性分析
利用TBtools进行甜橙内,拟南芥、水稻和甜橙;克里曼丁橘、苹果和甜橙的AP2 亚家族基因之间的共线性可视化分析。
1.6 甜橙AP2亚家族基因表达分析
将甜橙幼苗置于霍格兰(Hoagland)营养液中,在光照培养箱培养一周。再将植株分别置于含有20% PEG6000、250 mmol·L-1 NaCl、100 µmol·L-1 ABA和4 ℃的霍格兰(Hoagland)营养液中进行模拟干旱、高盐、ABA 和低温处理,在0、1、3、6、12和24 h后分别采集叶片。样品RNA提取采用试剂盒法(普洛麦格,中国),利用FastKing gDNA Dispelling RT SuperMix 试剂盒(天根,中国)合成cDNA 第一链。利用Primer 软件设计实时荧光PCR 定量引物(表1)。反应体系20 μL:2×FastReal qPCR PreMix(SYBR GREEN)(天根,中国)10 μL,模板1 μL,上、下游引物各0.5 μL,ddH2O 8 μL。反应程序为:95 ℃2 min;95 ℃5 s,60 ℃15 s,循环40 次。用2-△△CT方法计算相对表达量。利用SPSS 软件进行差异显著性分析。
表1 实时荧光定量PCR 引物
Table 1 qRT-PCR primers

基因名称Gene name CsRAP2-1 CsRAP2-2 CsRAP2-3 CsRAP2-4 CsRAP2-5 CsRAP2-6 CsRAP2-7 CsRAP2-8 CsRAP2-9 CsRAP2-10 CsRAP2-11 CsRAP2-12 CsRAP2-13 CsRAP2-14上游引物(5'-3')Forward primer(5'-3')TCAGGAGCAGCAAGCCACATC ATGGCAAGCAAGGATTGGCAGAG TGACCACCAACAGGATCTCAAAGC GACTCTGCTCCGCTTGTTCTCG AAACCTAGCAACCCTCAACAAAACC GCAAACGCAGCCAGCATTATTACC CTGCTGCTGATGATGTTGACGATG AGCAGCAGCAGCAGCAACAG TGGTTTGACTGGCATCGTTGGAC GAATGGCAGTGGTGGCAGTAGTG TCCATTCCTCCGTCGTCACCTC TGTGACTTCACTTGGCTGCGATG TTAATAAGAGAAGGCGGCGTGAGC CCTGCGAGTGGTGGAGGAGAG下游引物(5'-3')Forward primer(5'-3')ACTTGACACCAACCCAGTTAGCC CTTCAGCAGCTTCCTCCTGTGTG GCGAACTCAGCGGCAGGTAAC GGCACTCCACACCATGTCAACC CGATGACGCCGAGCCACTAC GTCAGTAGCCGCCATAGCACAAC TCTCACTTGCTGCGGCTTCTG TGGCGGCGGTGGTGAAGG TGTGCTGCCGCTTCTTCATCG TTGAGAAGCGGCAAGGTTGAGC GGTTGCTGCCACTGTTGTTGTTAG TCCACCGATGCCTGCTGACTC GCAGCAGTAGTAGCAGCAGCAG GCTGGGGCTGTTGGTTCTGATAC
2 结果与分析
2.1 甜橙AP2亚家族基因鉴定与分析
鉴定出甜橙AP2 亚家族成员14 个。根据染色体位置与拟南芥同源基因的相似性进行命名(Cs‐RAP2-1~CsRAP2-6、CsRAP2-7、CsRAP2-8~CsRAP2-14)。甜橙AP2 亚家族基因的CDS 序列长度为915(CsRAP2-9)~2181(CsRAP2-10)bp。氨基酸数目为316(CsRAP2-5)~691(CsRAP2-10)aa;蛋白分子质量为36.830(CsRAP2-1)~75.314(CsRAP2-10)kDa ;等电点为5.33(CsRAP2-6)~9.06(CsRAP2-13);不稳定系数为40.42(CsRAP2-10)~63.45(CsRAP2-13),亲水性为-0.954(CsRAP2-6)~-585.000(CsRAP2-8),表现为不稳定的亲水蛋白;该家族的亚细胞定位大多位于细胞核,这与AP2 亚家族作为转录因子的功能和特性一致(表2)。
表2 甜橙AP2 亚家族基因信息
Table 2 Information of AP2 subfamily genes in C.sinensis

注:nucl.细胞核;chlo.叶绿体;cyto.细胞骨架;extr.细胞外基质;mito.线粒体;plas.质膜;pero.过氧物酶体;vacu.液泡。
Note:nucl. Nucleus; chlo. Chloroplast; cyto. Cytoskeleton; extr. Extracell; plas. Plasma membrane; mito. Mitochondrion; pero. Peroxisome; vacu.Vacuole.
基因名称Gene name CsRAP2-1 CsRAP2-2 CsRAP2-3 CsRAP2-4 CsRAP2-5 CsRAP2-6 CsRAP2-7 CsRAP2-8 CsRAP2-9 CsRAP2-10 CsRAP2-11 CsRAP2-12 CsRAP2-13 CsRAP2-14编码区长度CDS Length/bp 1560 2043 1608 1863 1047 1362 1512 1728 915 2181 1467 1182 1203 1533登录号Accession Number Cs1g_pb017560 Cs1g_pb018830 Cs1g_pb019650 Cs1g_pb016600 Cs1g_pb009560 Cs3g_pb008940 Cs7g_pb020540 Cs4g_pb002610 Cs4g_pb001200 Cs6g_pb002300 Cs6g_pb004660 Cs6g_pb019310 Cs7g_pb003570 Cs8g_pb019330氨基酸数Number of amino acids 330 651 532 583 316 416 471 543 339 691 456 361 328 469分子质量Molecular weight/Da 36 830.98 72 100.17 58 446.33 64 616.54 35 544.91 47 000.55 51 525.09 58 938.78 38 460.04 75 314.18 50 786.81 40 592.88 36 862.84 50 593.56等电点PI 6.49 6.30 6.17 6.46 6.03 5.33 8.65 8.20 5.70 5.97 6.80 7.80 9.06 6.90不稳定系数Instability index(II)60.46 57.95 50.39 40.82 51.65 61.95 49.90 46.45 49.23 40.42 44.38 58.39 63.45 49.49亚细胞定位Subcellular location nucl:14 nucl:14 nucl:9,chlo:3,cyto:1,extr:1 nucl:14 chlo:8,nucl:5,mito:1 nucl:11,chlo:2,nucl:13,plas:1 nucl:11,cyto:2,pero:5,chlo:4,nucl:3 nucl:14 nucl:13,vacu:1 nucl:14 nucl:11,chlo:1,cyto:1,pero:1 nucl:14亲水性Gravy-0.693-0.712-0.605-0.842-0.919-0.954-0.632-0.585-0.847-0.703-0.795-0.764-0.778-0.661
2.2 甜橙AP2 亚家族蛋白的系统发育树构建及多序列比对
为了分析甜橙AP2 亚家族蛋白的系统发育关系,构建了拟南芥、水稻、克里曼丁橘和甜橙的AP2亚家族蛋白的系统发育树(图1)。结果显示,甜橙AP2 亚家族蛋白分为古代进化支(CsRAP2-5、Cs‐RAP2-6、CsRAP2-9、CsRAP2-12 和CsRAP2-13)、中间进化支(CsRAP2-1、CsRAP2-7、CsRAP2-11 和Cs‐RAP2-14)和现代进化支(CsRAP2-2~CsRAP2-4、Cs‐RAP2-8和CsRAP2-10)。根据进化树的拓扑结构分类,14 个甜橙AP2 亚家族蛋白可以分为10 组,用不同的颜色绘制。同一组的蛋白结构同源性较高,可能有相似功能。聚在同一组中的拟南芥AP2、水稻AP2、克里曼丁橘AP2 和甜橙AP2 蛋白成员数量的差异表明4 个物种之间AP2 亚家族蛋白存在明显的种间差异。甜橙AP2亚家族蛋白与克里曼丁橘的亲缘关系较近,与拟南芥和水稻的关系较远。
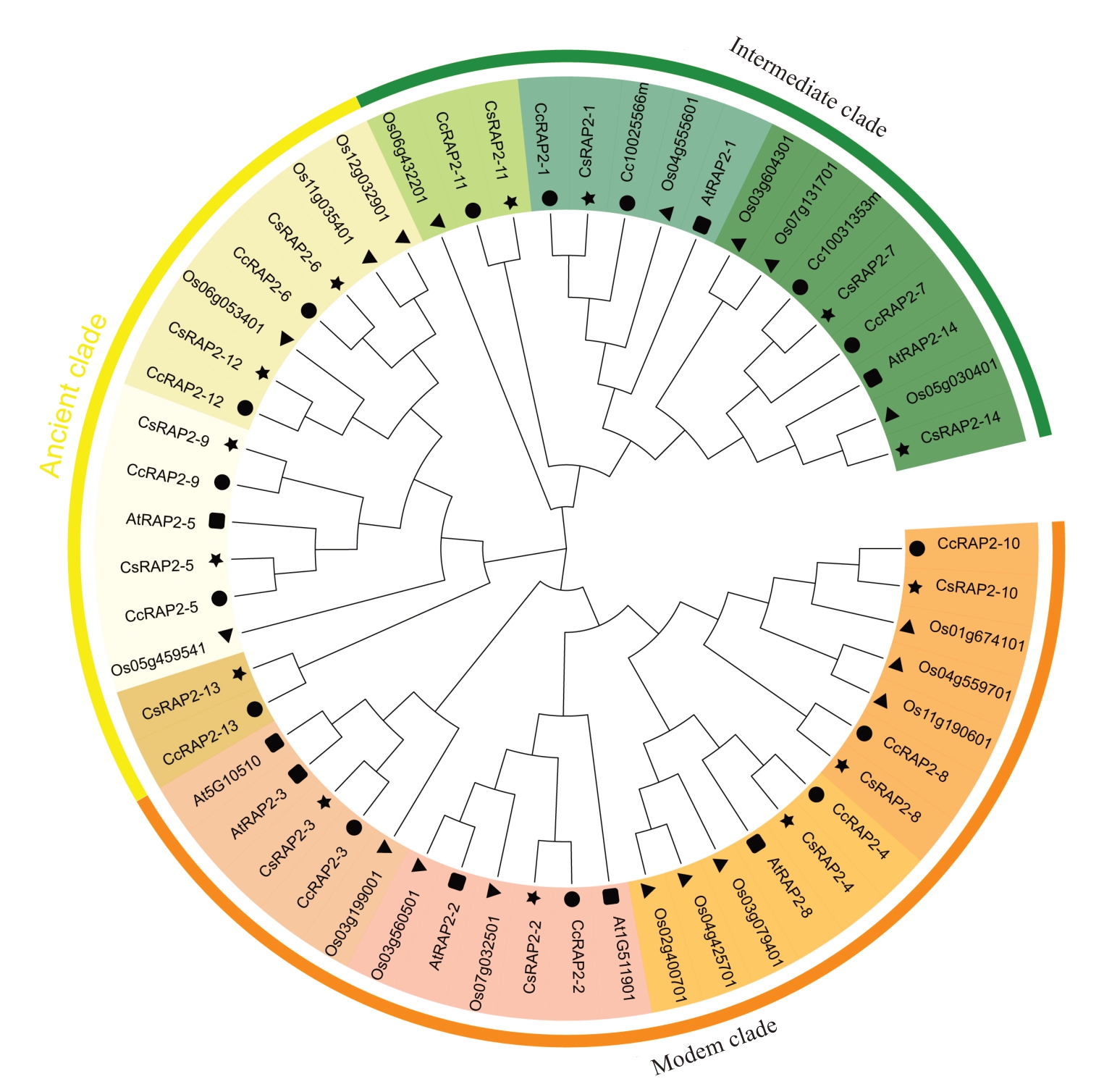
图1 4 个物种AP2 蛋白的系统进化树
Fig.1 Phylogenetic tree of AP2 proteins from four plant species
为了进一步分析和鉴定甜橙AP2亚家族蛋白中双AP2 结构域重复序列的特征,进行了多序列比对(图2)。比对结果揭示了氨基酸残基S-3、R-6、G-7、V-8、R-12、T-14、R-16、E-18、H-20、W-22、D-23、G-35、Q-37、G-41、D-44、A-49、A-50、Y-53、D-54、A-56、A-57、K-59、G-62、N-68、F-69、Y-74、M-81、E-88、L-93、R-94、R-95、R-103、G-104、S-106、Y-108、R-109、G-110、V-111、H-115、R-119、W-120、A-122、R-123、G-125、G-129、K-131、Y-134、L-135、G-136、E-143、A-144、A-145、A-147、Y-148、D-149、A-151、G-157、A-160、V-161、T-162、N-163、F-164、Y-168 在甜橙AP2亚家族基因蛋白中完全保守。在两个AP2结构域中皆含有3 个精氨酸(R)、4 个甘氨酸(G)、1 个缬氨酸(V)、1 个谷氨酸(E)、1 个色氨酸(W)、1 个天冬氨酸(D)、4 个丙氨酸(A)、1 个赖氨酸(K)、1 个天冬酰胺(N)、1个苯丙氨酸(F)和1个酪氨酸(Y)。

图2 甜橙 AP2 家族蛋白结构域分析
Fig. 2 Domain analysis of AP2 subfamily proteins in C. sinensis
2.3 甜橙AP2 亚家族蛋白保守结构域、基因结构和保守基序分析
为了分析甜橙AP2 亚家族基因结构和遗传性,揭示该基因家族的进化规律,使用TBtools 软件对该家族基因结构进行可视化。结果表明,甜橙AP2 亚家族基因包含5~9个内含子和6~10个外显子。甜橙AP2 亚家族蛋白可分为10 个亚组,且同一亚组的内含子与外显子的数量基本相等。
使用MEME预测14个甜橙AP2亚家族蛋白的10个保守基序(图3)。结果表明,甜橙AP2亚家族都含有Motif 3(VPKKSRDTPGQRSSQYRGVTRHRWT‐GRYEAHLWD)、Motif 4(KQVYLGGYDKEEAAA)、Motif 2(RAYDLAALKYWGPDTTINFPVSDYEKEL‐EEMKNLTKZEYVASLRRKSSGF)、Motif 5(RGAS‐KYRGVTR)和Motif 1(HQNGRWEARIGRVFGNKY‐LYLGTFSTZEEAAEAYDIAAIKYRGLNAVTNF) 。甜橙AP2 亚家族蛋白的古代进化支(CsRAP2-5、Cs‐RAP2-6、CsRAP2-9、CsRAP2-2和CsRAP2-13)都含有Motif 7,Motif 6只存在于古代进化支成员(CsRAP2-5、CsRAP2-9、CsRAP2-2 和CsRAP2-13),现代进化支(CsRAP2-2~CsRAP2-4、CsRAP2-8和CsRAP2-10)都有Motif 9和Motif 8。研究结果表明,甜橙AP2亚家族蛋白同一亚组的成员高度保守,这也反映出构建的系统进化分析结果可靠。
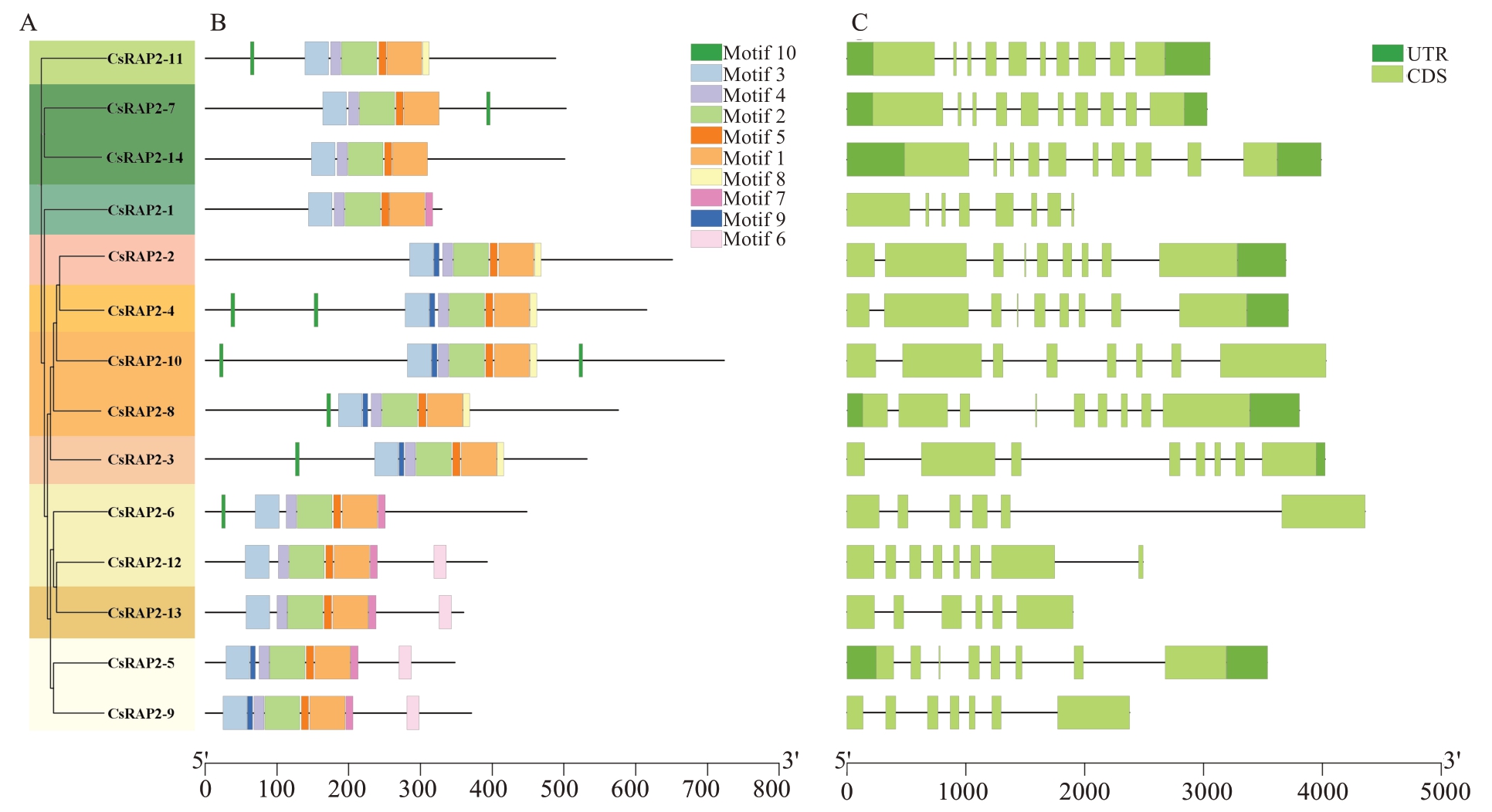
图3 甜橙AP2 家族蛋白系统发育树(A)、蛋白基序(B)和基因结构(C)
Fig.3 Phylogenetic tree(A),conserved domains(B)and gene structure(C)of AP2 subfamily proteins in C.sinensis
2.4 甜橙AP2 亚家族启动子顺式作用元件分析与miRNA的预测
通过PlantCARE 网站对甜橙AP2 亚家族启动子的顺式作用元件进行预测。结果显示(图4),甜橙AP2 亚家族基因启动子上存在激素响应、防御与应激响应、脱落酸响应、厌氧诱导、昼夜节律调控、分生组织表达相关、干旱响应等元件。例如,植物激素调控元件中的生长素响应元件Ⅰ(TGA-element)和脱落酸响应元件Ⅲ(ABRE);逆境防御响应元件Ⅱ(TC-rich repeats)、干旱诱导元件Ⅷ(MBS)、损伤响应元件Ⅹ(WUN-motif);植物生长发育相关元件包括参与昼夜节律调控的顺式调控元件Ⅴ(circadian)和分生组织表达相关的元件Ⅶ(CAT-box);光响应元件Ⅸ(G-box、MRE、Box4、GATA-motif、G-Box、GT1-motif、TCT-motif 和AE-box)。该家族基因的启动子中都至少含有6 个光响应元件,其中Cs‐RAP2-5 和CsRAP2-14 最多,含有18 个光响应元件。综上所述,该亚家族基因可能与胁迫响应和激素应答有关,并参与植物生长发育过程。
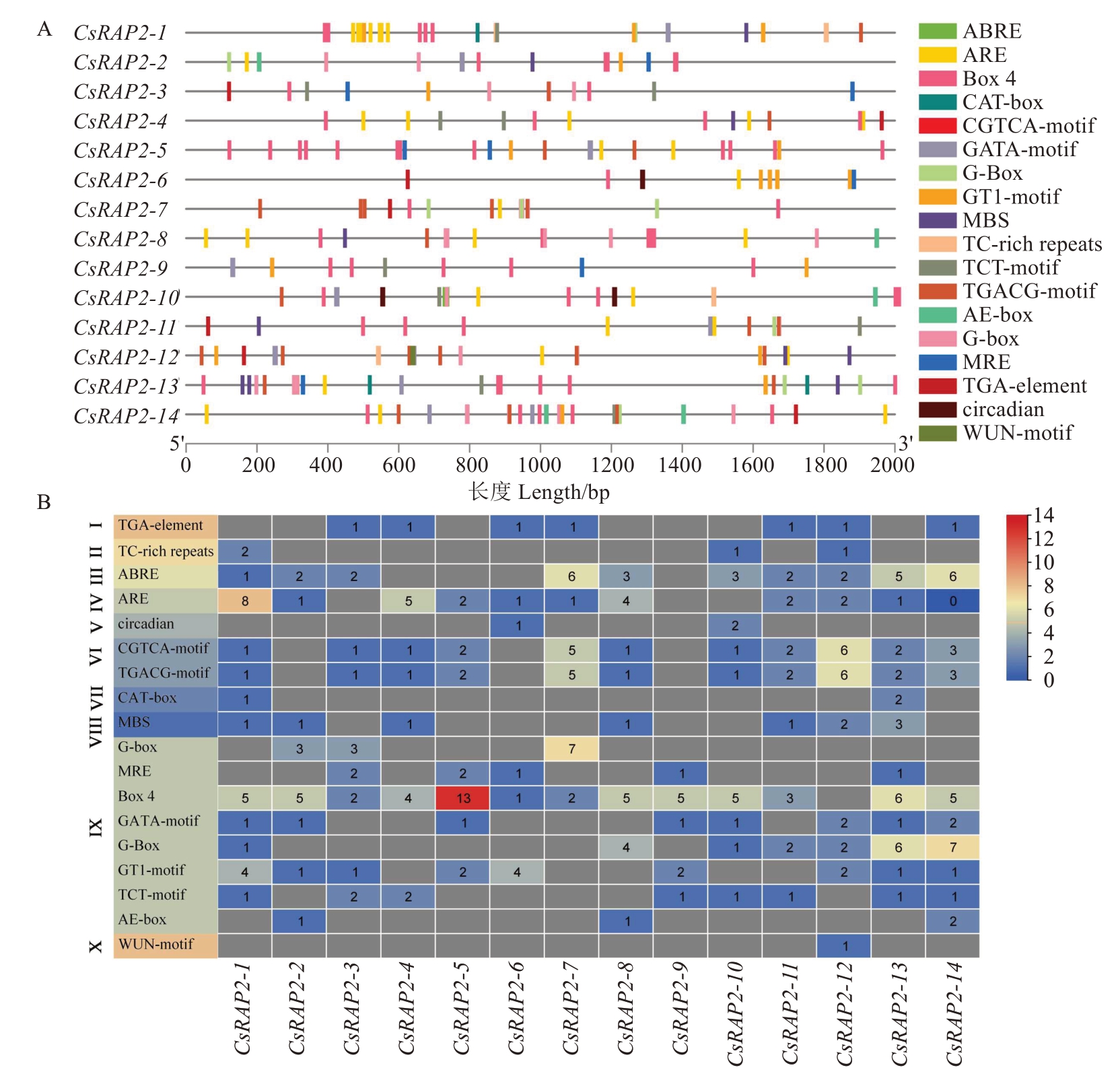
图4 甜橙AP2 家族基因启动子的顺式作用元件预测
Fig.4 Prediction of cis-acting elements on the promoter of AP2 subfamily proteins in C.sinensis
microRNA(miRNA)可能在植物中参与转录基因表达调控。通过在线网站分析了甜橙AP2 亚家族基因可能被靶向的miRNA,包括对果实成熟和变色、开花时间和植物耐盐性有调控作用的miR172[22],参与调控植物根系生长发育和抗逆(干旱、盐碱和高温)的miR164[23],以及参与调节幼苗生长、气孔密度和耐旱性的miR393[24](表3)。以上结果表明这几个miRNA 可能靶向甜橙AP2 亚家族基因表达来调控植物生长发育和响应激素、非生物胁迫。
表3 与甜橙AP2 亚家族基因相关的miRNA
Table 3 The prediction of miRNA related to AP2 subfamily genes in C.sinensis

靶基因Target gene CsRAP2-1(Cs1g_pb017560.1)CsRAP2-1(Cs1g_pb017560.1)CsRAP2-1(Cs1g_pb017560.1)CsRAP2-2(Cs1g_pb018830.1)CsRAP2-4(Cs1g_pb016600.1)CsRAP2-6(Cs3g_pb008940.1)CsRAP2-7(Cs7g_pb020540.1)CsRAP2-7(Cs7g_pb020540.1)CsRAP2-7(Cs7g_pb020540.1)CsRAP2-9(Cs4g_pb001200.1)CsRAP2-10(Cs6g_pb002300.1)CsRAP2-11(Cs6g_pb004660.1)CsRAP2-11(Cs6g_pb004660.1)CsRAP2-11(Cs6g_pb004660.1)CsRAP2-12(Cs6g_pb019310.1)CsRAP2-14(Cs8g_pb019330.1)CsRAP2-14(Cs8g_pb019330.1)CsRAP2-14(Cs8g_pb019330.1)miRNA名称miRNA name csi-miR172a-3p csi-miR172c csi-miR172b csi-miR164 csi-miR393 csi-miR164 csi-miR172a-3p csi-miR172b csi-miR172c csi-miR156 csi-miR3946 csi-miR172a-3p csi-miR172c csi-miR172b csi-miR396c csi-miR172a-3p csi-miR172c csi-miR172b抑制类型Inhibition Cleavage Cleavage Cleavage Cleavage Cleavage Cleavage Cleavage Cleavage Cleavage Cleavage Cleavage Cleavage Cleavage Cleavage Cleavage Cleavage Cleavage Cleavage
2.5 甜橙AP2亚家族基因染色体定位和共线性分析
染色体定位分析表明,甜橙AP2 亚家族基因分别定位于1 号(CsRAP2-1~CsRAP2-5)、3 号(Cs‐RAP2-6)、4 号(CsRAP2-8、CsRAP2-9)、6 号(Cs‐RAP2-10~CsRAP2-12)、7号(CsRAP2-7、CsRAP2-13)和8号染色体(CsRAP2-14)。共线性分析结果显示,一共有4 个片段存在重复(CsRAP2-7 和CsRAP2-11、CsRAP2-7 和CsRAP2-14、CsRAP2-11 和CsRAP2-14、CsRAP2-12 和CsRAP2-13),说明在甜橙AP2 亚家族基因的进化过程中存在基因复制(图5)。
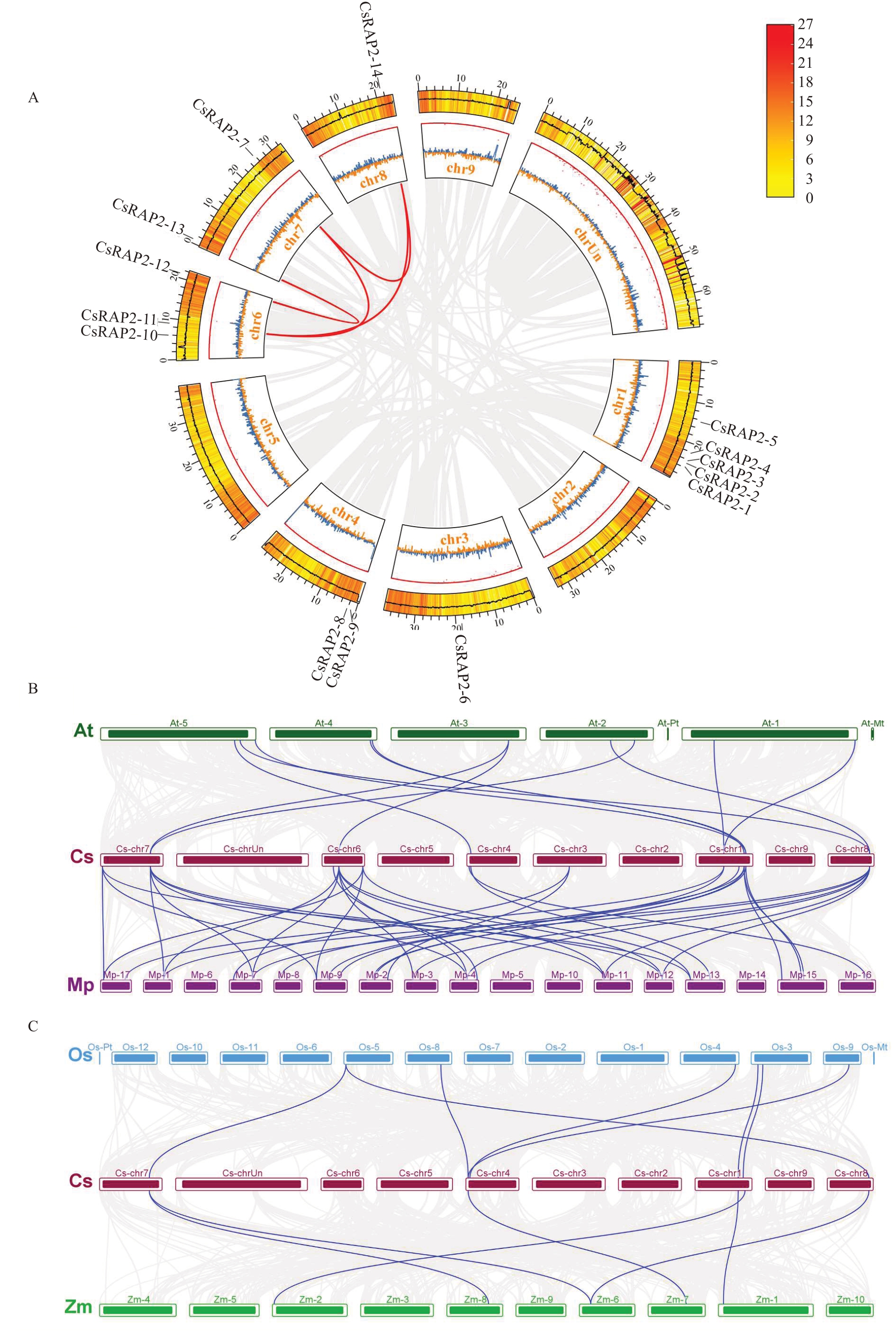
图5 甜橙AP2 亚家族成员染色体定位(A)和共线性关系(B、C)
Fig.5 Chromosomal location(A)and syntenic relationship(B,C)of AP2 subfamily proteins in C.sinensis
物种间共线性分析表明,甜橙与拟南芥之间有10个同源基因对,分布在1、4、6、7、8号染色体上;甜橙与苹果之间有37个同源基因对,分布在1、3、4、6、7、8 号染色体上;甜橙与水稻之间有7 个同源基因对,分布在1、4、7、8 号染色体上;甜橙与玉米之间有6个同源基因对,分布在1、4、7、8号染色体上。甜橙AP2 亚家族基因中有4 个基因(CsRAP2-7、CsRAP2-9、CsRAP2-1 和CsRAP2-12)能在其他物种中都找到同源基因(图5),可能在植物进化过程中有重要作用。与水稻和玉米相比,甜橙与拟南芥、苹果的AP2亚家族同源基因对更多,说明AP2 亚家族基因在物种间分化时较保守。
2.6 甜橙AP2亚家族蛋白二级结构预测
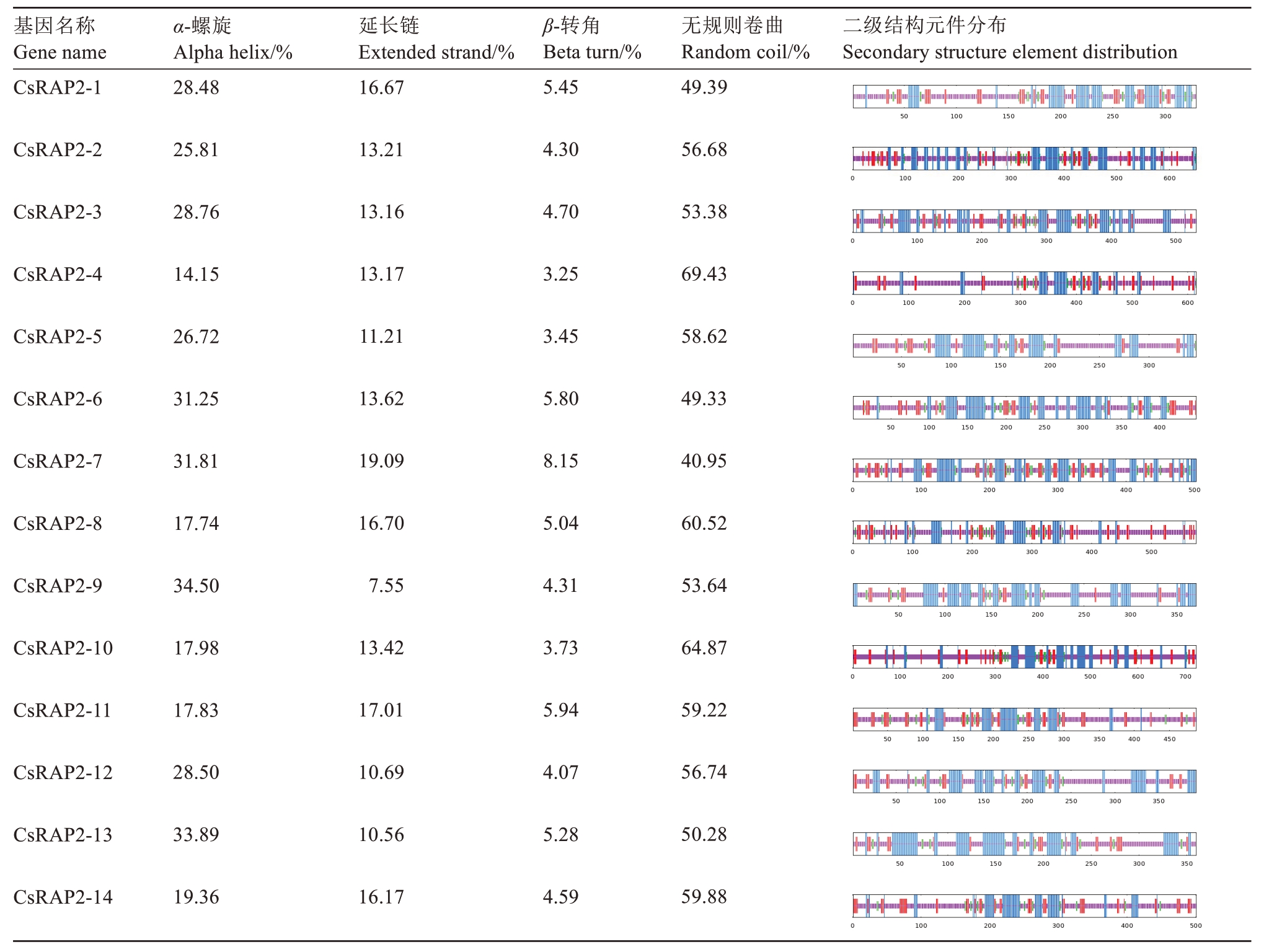
通过ProtParam 在线分析工具预测甜橙AP2 亚家族成员的二级结构(表4)。结果表明,甜橙AP2亚家族基因均含有α-螺旋、β-转角、无规则卷曲和延伸链4 种构型,其中α-螺旋占比在14.15%(CsRAP2-5)~34.50%(CsRAP2-9)之间;延伸链占比在7.55%(CsRAP2-9)~19.09%(CsRAP2-7)之间;β-转角占比在3.25%(CsRAP2-4)~8.15%(CsRAP2-7)之间;无规则卷曲占比在40.95%(CsRAP2-7)~69.43%(Cs‐RAP2-4)之间。α-螺旋和无规则卷曲比例最高。
表4 甜橙AP2 亚家族蛋白二级结构
Table 4 The protein secondary structure of AP2 subfamily genes in C.sinensis
?
2.7 甜橙AP2亚家族基因的组织特异性表达分析
对甜橙AP2 亚家族在甜橙根、茎和叶中的表达进行分析,结果显示,CsRAP2-1、CsRAP2-2、Cs‐RAP2-5、CsRAP2-7、CsRAP2-9、CsRAP2-11、CsRAP2-12 和CsRAP2-14 在茎中表达量较高。CsRAP2-4、CsRAP2-10 在茎中表达量最低。CsRAP2-3 和Cs‐RAP2-6 在根中表达量较高。CsRAP2-2 主要在根和茎中表达(图6)。CsRAP2-3、CsRAP2-6 和CsRAP2-8 主要在根中表达。综上,甜橙AP2 亚家族基因在不同组织部位中的表达存在差异。
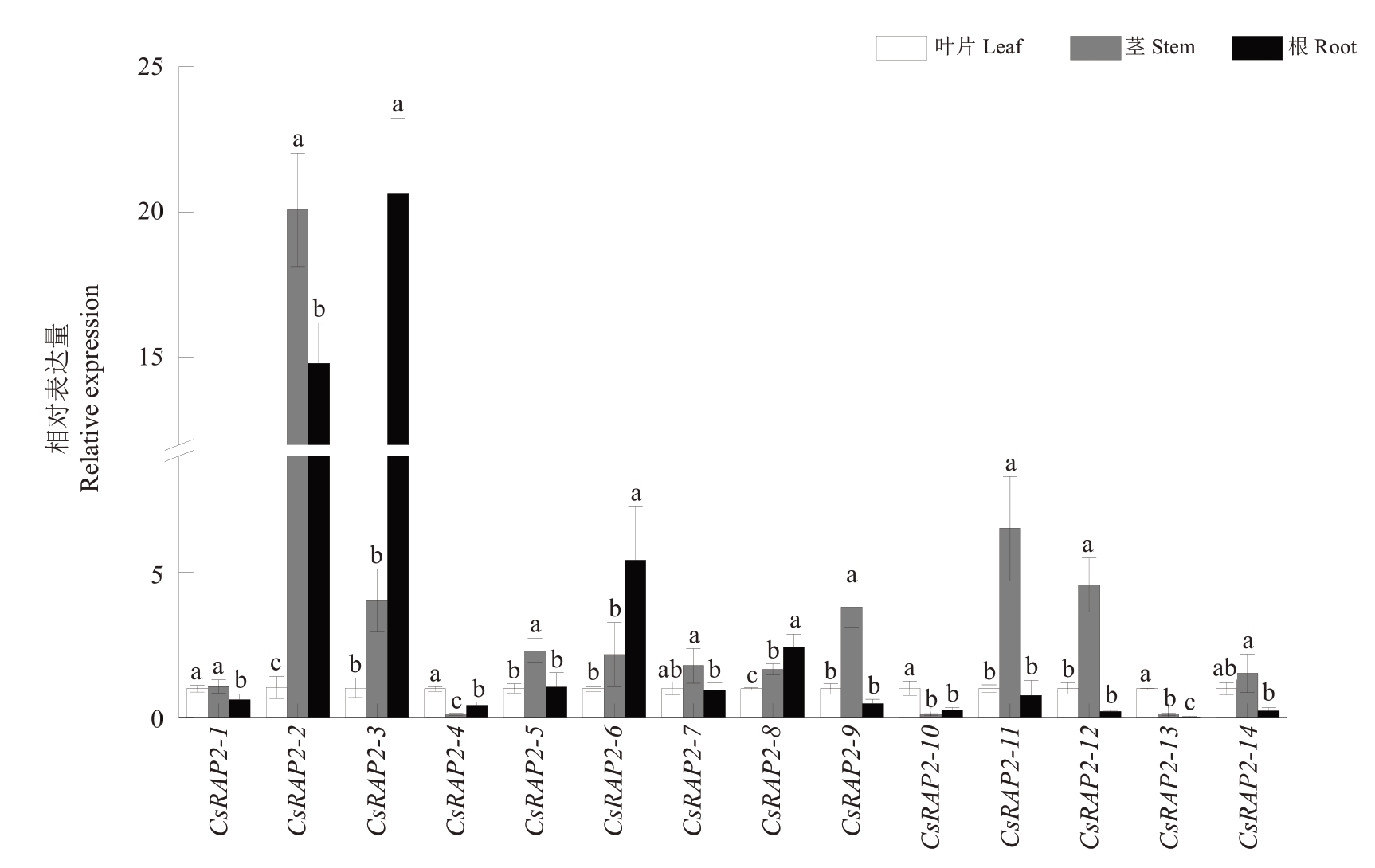
图6 甜橙AP2 亚家族基因不同组织中表达量
Fig.6 The expression levels in different tissues of AP2 subfamily proteins in C.sinensis
2.8 甜橙AP2 亚家族基因在非生物胁迫下的表达分析
14 个甜橙AP2 亚家族基因在不同非生物胁迫处理下的表达量差异较大(图7)。模拟干旱处理后,CsRAP2-1、CsRAP2-2、CsRAP2-8 和CsRAP2-10在叶中的表达总体呈下调趋势。处理6 h 时,Cs‐RAP2-3、CsRAP2-5、CsRAP2-6、CsRAP2-9、CsRAP2-11 和CsRAP2-14 在叶中的表达量显著上升。Cs‐RAP2-7 在处理3 h 时表达量显著上升,随后陡然下降。

图7 甜橙AP2 亚家族基因在非生物胁迫下的表达分析
Fig.7 Expression analysis of AP2 subfamily genes in C.sinensis under abiotic stress
在盐处理下,CsRAP2-5 和CsRAP2-14 的表达量与处理前差异不显著。在处理6 h 时,CsRAP2-4、CsRAP2-6、CsRAP2-7、CsRAP2-11 和CsRAP2-13 在叶中的表达量显著上升并达到峰值。CsRAP2-1、CsRAP2-2、CsRAP2-8 和CsRAP2-12 在叶中的表达量在处理后总体呈下调趋势。CsRAP2-10 在叶中的表达量先下调,在12 h上升达到峰值。
ABA 处理后,CsRAP2-1、CsRAP2-2、CsRAP2-4、CsRAP2-6、CsRAP2-8、CsRAP2-10~CsRAP2-14 在叶中的表达总体呈下调趋势,表达量低于处理前。Cs‐RAP2-3 和CsRAP2-5 在叶中的表达量先显著下调,然后在6 h 时上升且表达量接近处理前。CsRAP2-9的表达量与处理前差异不显著。在12 h 时,Cs‐RAP2-7在叶中的表达量显著上升并达到峰值。
在低温处理后,CsRAP2-13 的表达量先下调,在处理3 h 时上调后又下调,并在12 h 上升达到峰值,表达量总体呈现“M”形双峰变化趋势。除CsRAP2-13外,其他13个基因的表达量基本呈下调趋势。
3 讨 论
甜橙AP2 亚家族转录因子在植物体内广泛存在,目前已在拟南芥[1]、山核桃[25]、马尾松[26]、菠萝[27]、铁皮石斛[28]和油棕[29]等植物中分别鉴定到18、30、7、24、14、34 个成员。笔者在本研究中共鉴定出14 个甜橙AP2亚家族基因,相较于Ito等[30]提出的存在13个甜橙AP2 亚家族基因,基于柑橘基因组数据库的搜索还包括了一个新基因CsRAP2-1。蛋白理化性质分析和亚细胞定位预测表明,甜橙AP2 亚家族均为不稳定的亲水性蛋白,且主要定位于细胞核,这与该家族作为转录因子的特征相符,且与栀子AP2 亚家族的分析一致[31]。系统进化树显示,甜橙AP2 亚家族可分为古代进化支(CsRAP2-5、CsRAP2-6、Cs‐RAP2-9、CsRAP2-12 和CsRAP2-13)、中间进化支(CsRAP2-1、CsRAP2-7、CsRAP2-11 和CsRAP2-14)和现代进化支(CsRAP2-2~CsRAP2-4、CsRAP2-8 和CsRAP2-10),这与蜡梅AP2 亚家族分类相近[32]。在植物AP2 亚家族蛋白的系统进化树中,聚在同一组中的拟南芥AP2、水稻AP2、克里曼丁橘AP2 和甜橙AP2 蛋白成员数量存在明显的种间差异。甜橙AP2亚家族蛋白与克里曼丁橘的亲缘关系较近,与拟南芥和水稻的关系较远。蛋白基序预测表明,甜橙AP2 亚家族皆包含Motif 3、Motif 4、Motif 2、Motif 5和Motif 1。另外,甜橙AP2 亚家族蛋白进化树中每个组都有特异性的保守元件,证明了系统进化树分析的可信度。甜橙AP2亚家族基因包含5~9个内含子和6~10个外显子,这与拟南芥中AP2亚家族基因的外显子-内含子结构相似[1]。甜橙AP2亚家族基因启动子序列含有内源激素响应、生长发育和非生物胁迫响应等顺式作用元件,这与山核桃中AP2 亚家族基因启动子的预测分析类似[25]。蛋白二级结构预测显示,甜橙AP2 亚家族蛋白的α-螺旋和无规则卷曲比例最高,α-螺旋平均比例为25.48%,无规则卷曲比例为55.92%,这个比例与栀子中AP2 亚家族的比例相近[31]。
组织特异性表达分析表明,CsRAP2-6主要在根中表达。CsRAP2-6 同源基因拟南芥AtWRI1 主要在根中表达,与本研究结果一致。而AtWRI1在拟南芥中通过调控生长素响应基因表达维持根系生长素含量,最终调控拟南芥根系发育[33]。因此,CsRAP2-6基因也可能在甜橙根系发育中起重要作用。Cs‐RAP2-7 主要在茎中表达。这与CsRAP2-7 同源基因烟草NtTOE3 在茎中表达量较高的结果一致[34]。但是,CsRAP2-7 在玉米中的同源基因ZmRAP2-7 主要在根中表达,刺激根发育[35],这与本研究结果不同。CsRAP2-3 在甜橙根中表达量最高,但CsRAP2-3 在拟南芥中的同源基因AtAIL6 却在花中的表达量最高,并且调节发育中的花器官生长和形态建成[11]。这些结果说明AP2 亚家族基因在不同物种中的组织部位表达存在差异。
前人研究表明,AP2 亚家族的基因主要参与响应非生物胁迫和调节植物的生长发育,如花器官形成、胚胎发生和根系发育[5]。笔者在本研究中从甜橙AP2 亚家族成员启动子区域中鉴定出内源激素响应、生长发育和非生物胁迫响应等顺式作用元件,表明甜橙AP2 亚家族可能在甜橙响应非生物胁迫过程中起重要作用。同时研究分析了这些基因在非生物胁迫下叶中的表达模式。结果表明,CsRAP2-1、CsRAP2-2、CsRAP2-8 和CsRAP2-10 在模拟干旱、高盐、ABA 和低温处理下,表达量总体上都呈下调趋势。CsRAP2-12 的表达量在高盐、ABA 和低温处理后下调表达,在模拟干旱处理1~12 h 下调表达,但在24 h 时表达量高于0 h。除这5 个基因外,其余9 个甜橙AP2 亚家族基因的表达量在干旱处理后均呈上调趋势。CsRAP2-3、CsRAP2-4、CsRAP2-6、Cs‐RAP2-7、CsRAP2-9、CsRAP2-11 和CsRAP2-13 均被高盐胁迫诱导表达。ABA 处理后,只有CsRAP2-7的表达量呈上调趋势。除CsRAP2-13 外,其他13 个基因的表达量在低温处理后都呈下调趋势。这些结果说明,多数甜橙AP2 亚家族基因在干旱和高盐胁迫下表达量上升,在ABA 和低温处理下表达量下降。CsRAP2-2 同源基因拟南芥AtANT 在高盐胁迫下表达量显著降低[36],与本研究结果一致。且AtANT 与SCABP8/CBL10(SOS3- LIKE CALCIUM BINDING PROTEIN 8)启动子结合,负调节拟南芥耐盐性,因此CsRAP2-2 也可能在甜橙中负调节耐盐性。CsRAP2-7 同源基因烟草NtTOE3 也可被高盐、干旱和ABA 诱导表达。不同的是,NtTOE3 对低温敏感,这种现象在CsRAP2-7 同源基因盐穗木HcTOE3 同样存在,且相较4 ℃低温处理,冰冻胁迫(-2 ℃)更能强烈诱导HcTOE3[13],而CsRAP2-7 在低温处理下的表达量与处理前没有明显变化[34]。CsRAP2-2 同源基因拟南芥AtANT 在干旱和ABA 处理后表达量都显著上升[37],而CsRAP2-2 在这两个处理后表达量都显著降低。这些结果说明不同植物的AP2 亚家族基因对非生物胁迫的响应存在差异。
目前有关甜橙AP2亚家族基因的研究还没有较多报道,笔者在本研究中鉴定和分析甜橙AP2 亚家族基因,对该亚家族基因在不同组织部位和非生物胁迫下的表达进行分析。研究结果为后续研究这些基因调控甜橙非生物胁迫的功能提供了依据。
4 结 论
研究鉴定和分析了甜橙14 个AP2 亚家族基因,均编码不稳定的亲水性蛋白。甜橙AP2亚家族基因在不同组织部位中的表达存在差异。14 个成员中,8 个基因在模拟干旱胁迫下表达上升,7 个基因在高盐胁迫下表达上升,大多数基因在ABA 和低温胁迫下表达下调,推测该家族基因可能在甜橙响应非生物胁迫过程中起重要作用。
[1] NAKANO T,SUZUKI K,FUJIMURA T,SHINSHI H.Genomewide analysis of the ERF gene family in Arabidopsis and rice[J].Plant Physiology,2006,140(2):411-432.
[2] LICAUSI F,OHME-TAKAGI M,PERATA P.APETALA2/Eth‐ylene Responsive Factor (AP2/ERF) transcription factors:Medi‐ators of stress responses and developmental programs[J]. New Phytologist,2013,199(3):639-649.
[3] KIM S,SOLTIS P S,WALL K,SOLTIS D E.Phylogeny and do‐main evolution in the APETALA2-like gene family[J]. Molecu‐lar Biology and Evolution,2006,23(1):107-120.
[4] DINH T T,GIRKE T,LIU X G,YANT L,SCHMID M,CHEN X M. The floral homeotic protein APETALA2 recognizes and acts through an AT- rich sequence element[J]. Development,2012,139(11):1978-1986.
[5] JOFUKU K D,DEN BOER B G,VAN MONTAGU M,OKA‐MURO J K. Control of Arabidopsis flower and seed develop‐ment by the homeotic gene APETALA2[J].The Plant Cell,1994,6(9):1211-1225.
[6] ZHANG X,ZHANG W B,CHANG Y T,MA Y J,DENG Y Y,ZHANG N,BAI Y W,JIANG Z H,HU T.Cloning,characteriza‐tion,and expression pattern analysis of the BBM gene in tree pe‐ony(Paeonia ostii)[J].Forests,2023,15(1):36.
[7] ZHOU R N,ZHAO Y J,CHENG P,ZHANG B S,LIU Z,WANG S H,LI H B,CHEN Q S,ZHAO Y,LI S N,WU X X.GmBBM7 promotes callus and root growth during somatic em‐bryogenesis of soybean (Glycine max)[J].Biotechnology & Bio‐technological Equipment,2023,37(1):2238833.
[8] HU Z Y,WANG X J,WEI L,WANSEE S,NASAB H R,CHEN L,KANG Z S,WANG J F.TaAP2-10,an AP2/ERF tran‐scription factor,contributes to wheat resistance against stripe rust[J].Journal of Plant Physiology,2023,288:154078.
[9] ZHENG Y T,HE S C,CAI W W,SHEN L,HUANG X Y,YANG S,HUANG Y,LU Q L,WANG H,GUAN D Y,HE S L.CaAIL1 acts positively in pepper immunity against Ralstonia so‐lanacearum by repressing negative regulators[J]. Plant & Cell Physiology,2021,62(11):1702-1717.
[10] QI S Z,ZHAO R R,YAN J C,FAN Y M,HUANG C,LI H X,CHEN S Y,ZHANG T,KONG L S,ZHAO J,ZHANG J F.Global transcriptome and coexpression network analyses reveal new insights into somatic embryogenesis in hybrid sweetgum(Liquidambar styraciflua × Liquidambar formosana)[J]. Fron‐tiers in Plant Science,2021,12:751866.
[11] KRIZEK B A,BANTLE A T,HEFLIN J M,HAN H,FREESE N H,LORAINE A E.AINTEGUMENTA and AINTEGUMEN‐TA-LIKE6 directly regulate floral homeotic,growth,and vascu‐lar development genes in young Arabidopsis flowers[J]. Journal of Experimental Botany,2021,72(15):5478-5493.
[12] KRIZEK B A.AINTEGUMENTA-LIKE genes have partly over‐lapping functions with AINTEGUMENTA but make distinct contributions to Arabidopsis thaliana flower development[J].Journal of Experimental Botany,2015,66(15):4537-4549.
[13] YIN F L,ZENG Y L,JI J Y,WANG P J,ZHANG Y F,LI W H.The halophyte Halostachys caspica AP2/ERF transcription fac‐tor HcTOE3 positively regulates freezing tolerance in Arabidop‐sis[J].Frontiers in Plant Science,2021,12:638788.
[14] ZHANG T T,TANG Y H,LUAN Y T,CHENG Z Y,WANG X X,TAO J,ZHAO D Q. Herbaceous peony AP2/ERF transcrip‐tion factor binds the promoter of the tryptophan decarboxylase gene to enhance high-temperature stress tolerance[J]. Plant,Cell&Environment,2022,45(9):2729-2743.
[15] XU Q,CHEN L L,RUAN X A,CHEN D J,ZHU A D,CHEN C L,BERTRAND D,JIAO W B,HAO B H,LYON M P,CHEN J J,GAO S,XING F,LAN H,CHANG J W,GE X H,LEI Y,HU Q,MIAO Y,WANG L,XIAO S X,BISWAS M K,ZENG W F,GUO F,CAO H B,YANG X M,XU X W,CHENG Y J,XU J,LIU J H,LUO O J,TANG Z H,GUO W W,KUANG H H,ZHANG H Y,ROOSE M L,NAGARAJAN N,DENG X X,RU‐AN Y J. The draft genome of sweet orange (Citrus sinensis)[J].Nature Genetics,2013,45(1):59-66.
[16] 马青龄,梁贝贝,杨莉,胡威,刘勇,刘德春.枳WOX 基因家族全基因组鉴定及表达分析[J].果树学报,2022,39(5):712-729.MA Qingling,LIANG Beibei,YANG Li,HU Wei,LIU Yong,LIU Dechun. Genome-wide identification and expression analy‐sis of WOX gene family in Trifoliate orange[J]. Journal of Fruit Science,2022,39(5):712-729
[17] 张丹,郑平,邓彪,柴改凤,安昌,邓考,张文斌,陆宇明,卢翔宇,王小媚,秦源. 百香果(Passiflora edulis)R2R3-MYB 基因家族的结构和功能分析[J].果树学报,2024,41(1):12-29.ZHANG Dan,ZHENG Ping,DENG Biao,CHAI Gaifeng,AN Chang,DENG Kao,ZHANG Wenbin,LU Yuming,LU Xiangyu,WANG Xiaomei,QIN Yuan. Structure and function analysis of R2R3-MYB gene family from passion fruit (Passiflo‐ra edulis)[J].Journal of Fruit Science,2024,41(1):12-29.
[18] HORTON P,PARK K J,OBAYASHI T,FUJITA N,HARADA H,ADAMS-COLLIER C J,NAKAI K T. WoLF PSORT:Pro‐tein localization predictor[J]. Nucleic Acids Research,2007,35(Web Server issue):W585-W587.
[19] 方森,宋雪莲,韩轩轩,宋春晖,焦健,王苗苗,宋尚伟,郑先波,白团辉.苹果PIN 家族基因鉴定及在不定根形成中的表达分析[J].果树学报,2023,40(9):1789-1799.FANG Sen,SONG Xuelian,HAN Xuanxuan,SONG Chunhui,JIAO Jian,WANG Miaomiao,SONG Shangwei,ZHENG Xian‐bo,BAI Tuanhui. Identification and expression analysis of PIN gene family during adventi-tious root formation in apple[J].Jour‐nal of Fruit Science,2023,40(9):1789-1799.
[20] KUMAR S,STECHER G,TAMURA K. MEGA7:Molecular evolutionary genetics analysis version 7.0 for bigger datasets[J].Molecular Biology and Evolution,2016,33(7):1870-1874.
[21] BAILEY T L,WILLIAMS N,MISLEH C,LI W W.MEME:Dis‐covering and analyzing DNA and protein sequence motifs[J].Nu‐cleic Acids Research,2006,34(Web Server issue):W369-W373.
[22] CHENG X L,HE Q,TANG S,WANG H R,ZHANG X X,LV M J,LIU H F,GAO Q,ZHOU Y,WANG Q,MAN X Y,LIU J,HUANG R F,WANG H,CHEN T,LIU J. The miR172/IDS1 signaling module confers salt tolerance through maintaining ROS homeostasis in cereal crops[J]. The New Phytologist,2021,230(3):1017-1033.
[23] CHI Q,DU LY,MA W,NIU R Y,WU B W,GUO L J,MA M,LIU X L,ZHAO H X.The miR164-TaNAC14 module regulates root development and abiotic-stress tolerance in wheat seedlings[J].Journal of Integrative Agriculture,2023,22(4):981-998.
[24] YUAN W Y,SUO J Q,SHI B,ZHOU C L,BAI B,BIAN H W,ZHU M Y,HAN N. The barley miR393 has multiple roles in regulation of seedling growth,stomatal density,and drought stress tolerance[J]. Plant Physiology and Biochemistry,2019,142:303-311.
[25] YANG Z F,JIN H M,CHEN J H,LI C Y,WANG J N,LUO J,WANG Z J. Identification and analysis of the AP2 subfamily transcription factors in the pecan (Carya illinoinensis)[J]. Inter‐national Journal of Molecular Sciences,2021,22(24):13568.
[26] ZHU P H,CHEN Y,ZHANG J F,WU F,WANG X F,PAN T,WEI Q,HAO Y P,CHEN X L,JIANG C W,JI K S. Identifica‐tion,classification,and characterization of AP2/ERF superfami‐ly genes in Masson pine (Pinus massoniana Lamb.)[J]. Scientif‐ic Reports,2021,11(1):5441.
[27] ZHANG H N,PAN X L,LIU S H,LIN W Q,LI Y H,ZHANG X M. Genome-wide analysis of AP2/ERF transcription factors in pineapple reveals functional divergence during flowering in‐duction mediated by ethylene and floral organ development[J].Genomics,2021,113(2):474-489.
[28] ZENG D Q,TEIXEIRA DA SILVA J A,ZHANG M Z,YU Z M,SI C,ZHAO C H,DAI G Y,HE C M,DUAN J. Genomewide identification and analysis of the APETALA2 (AP2) tran‐scription factor in Dendrobium officinale[J]. International Jour‐nal of Molecular Sciences,2021,22(10):5221.
[29] ZHOU L X,YARRA R.Genome-wide identification and charac‐terization of AP2/ERF transcription factor family genes in oil palm under abiotic stress conditions[J]. International Journal of Molecular Sciences,2021,22(6):2821.
[30] ITO T M,POLIDO P B,RAMPIM M C,KASCHUK G,SOU‐ZA S G H. Genome-wide identification and phylogenetic analy‐sis of the AP2/ERF gene superfamily in sweet orange (Citrus si‐nensis)[J]. Genetics and Molecular Research,2014,13(3):7839-7851.
[31] 张麒功,林协全,叶泽霖,徐易溱,邹双全,邹小兴. 栀子AP2基因家族成员鉴定及其在盐胁迫下的表达模式[J].福建农林大学学报(自然科学版),2023,52(4):457-465.ZHANG Qigong,LIN Xiequan,YE Zelin,XU Yizhen,ZOU Shuangquan,ZOU Xiaoxing. Identification of AP2 gene family in Gardenia jasminoides and its expression analysis under salt stress[J]. Journal of Fujian Agriculture and Forestry University(Natural Science Edition),2023,52(4):457-465.
[32] 田明康,徐智祥,刘秀群,眭顺照,李名扬,李志能. 蜡梅AP2亚家族转录因子鉴定及CpAP2-L11 功能研究[J].园艺学报,2023,50(2):382-396.TIAN Mingkang,XU Zhixiang,LIU Xiuqun,SUI Shunzhao,LI Mingyang,LI Zhineng.Identification of the AP2 subfamily tran‐scription factors in Chimonanthus praecox and the functional study of CpAP2-L11[J].Acta Horticulturae Sinica,2023,50(2):382-396.
[33] KONG Q,LOW P M,LIM A R Q,YANG Y Z,YUAN L,MA W. Functional antagonism of WRI1 and TCP20 modulates GH3.3 expression to maintain auxin homeostasis in roots[J].Plants,2022,11(3):454.
[34] 卓维,陈倩,罗银,杨尚谕,鲁黎明,李立芹.烟草乙烯转录因子ERF 基因NtTOE3 的克隆、载体构建及表达分析[J]. 植物研究,2018,38(5):733-740.ZHUO Wei,CHEN Qian,LUO Yin,YANG Shangyu,LU Liming,LI Liqin. Cloning,construction of expression vector and expression analysis of the Ethylene-responsive factor ERF gene NtTOE3 in Nicotiana tabacum[J]. Bulletin of Botanical Re‐search,2018,38(5):733-740.
[35] LI J P,CHEN F J,LI Y Q,LI P C,WANG Y Q,MI G H,YUAN L X. ZmRAP2.7,an AP2 transcription factor,is involved in maize brace roots development[J]. Frontiers in Plant Science,2019,10:820.
[36] MENG L S,WANG Y B,YAO S Q,LIU A Z. Arabidopsis AINTEGUMENTA mediates salt tolerance by trans-repressing SCABP8[J].Journal of Cell Science,2015,128(15):2919-2927.
[37] MENG L S,WANG Z B,YAO S Q,LIU A Z. The ARF2-ANTCOR15A gene cascade regulates ABA-signaling-mediated resis‐tance of large seeds to drought in Arabidopsis[J]. Journal of Cell Science,2015,128(21):3922-3932.