胡柚(Citrus changshanensis K.S.Chen et C.X.Fu)又名金柚,是一种主产于浙江省衢州市的地方特色柑橘[1-3]。胡柚花型较大、果皮较厚、囊衣稍厚、果肉微苦、部分种子具单胚性,具有柚的特征;同时,与柚相比,胡柚翼叶小、果型小、果肉软而多汁、部分种子具有多胚性,又不同于柚[1-3]。早在1987 年,吴耕民[3]通过形态特征比较,提出胡柚可能是由柚与甜橙自然杂交而来。随着分子标记技术的发展,学者们先后应用随机扩增多态性DNA(RAPD)和简单序列重复区间扩增多态性(ISSR)等分子标记进行分析,认为胡柚起源与柚和甜橙密切相关[4-5]。在柑橘类中,起源于柚和甜橙的杂种只有葡萄柚。但胡柚抗寒性明显强于葡萄柚,这表明胡柚可能并不起源于柚和甜橙的杂交。先前笔者基于内转录间隔区(ITS)序列分析,发现柚和酸橙最有可能是胡柚的亲本[6],这与新近基于基因组分析提出的胡柚具有酸橙和柚的遗传背景[7]这一结论一致。柑橘易发生种间杂交[8],但目前为止,源于柚与酸橙杂交的就只有胡柚一种,使得胡柚成为一种独特的杂柑。胡柚耐寒、耐贮、具独特微苦风味,鲜食、加工(如用于双柚汁)和入药(作为衢枳壳[9])兼用,深受消费者欢迎,给当地带来了可观的经济收益,成为中国国家地理标志产品和浙江省重点开发与推广应用的名特优产品。
胡柚栽培历史悠久,位于常山县青石镇澄潭村的祖宗树是现存最古老的实生胡柚树,树龄接近120 a(年)。胡柚在良种选育过程中经历了较长的零星种植和实生繁殖时期[10],从普通胡柚中实生选育了脆红、红肉胡柚、夏红橘柚(也称红皮胡柚)、胡柚优株a、胡柚优株b和01-7等品种/优系/优株,其中01-7因风味浓、耐贮藏、抗性强等特点成为目前胡柚产业的主导推广品种。然而,不同胡柚间在苗木形态上具有高度相似性,肉眼难以区分。
相较于形态学、细胞学和同工酶等鉴别技术,分子标记具有数量丰富、多态性高、不受环境因素的影响等优点,因而被广泛用于果树品种鉴别,包括随机扩增多态性DNA(RAPD)、扩增片段长度多态性(AFLP)、简单序列重复(SSR)、序列相关扩增多态性(SRAP)、单核苷酸多态性(SNP)等在内的各种分子标记得到发展和应用[10-13]。近十余年来随着测序技术的发展,SNP 因具有高通量、分布广、二等位性等特点,成为最具应用潜力的第三代分子标记,被广泛应用于果树分子标记辅助育种[14-18]。在品种鉴定方面,SNP 已同SSR 一起作为国内《植物品种鉴定DNA 指纹方法总则》(NY/T 2594—2016)推荐的分子标记方法。迄今,出现了基于各种原理的SNP 分型方法,如等位基因特异PCR(AS-PCR)、酶切扩增多态性序列(CAPS)、衍生酶切扩增多态性(dCAPS)、高分辨熔解曲线分析(HRMA)、竞争性等位基因特异PCR(KASP)等[19]。在这些SNP 分型方法中,最便利和普适的当属AS-PCR和dCAPS。
AS-PCR 是根据SNPs 位点的野生型和突变型等位基因序列设计引物,通过等位基因特异引物和模板DNA 的结合情况对SNP 进行基因分型。相较于其他分型方法,AS-PCR 具有对设备要求不高、成本低、操作易等优点。dCAPS 标记是一种CAPS 标记变型,通过人为引入错配碱基制造酶切位点,从而突破SNP 需出现在限制性内切酶识别位点这一限制,且具有共显性、操作简单、适用性强等特点。因此,开始应用AS-PCR 和dCAPS 标记鉴别果树品种。如Heo 等[20]以宫川温州蜜柑及其新突变体Araunshiu 为试材,将存在于两者间的纯合SNP 开发成AS-PCR 标记,成功地将突变体、宫川温州蜜柑和其他柑橘进行区分。又如Peng 等[21]基于桃液泡膜糖转运载体基因(PpTST1)第三外显子的一个SNP 开发了dCAPS 标记,成功地对18 份桃材料进行了分型。虽然基于SNP 的分子标记在果树品种鉴别中的应用已经开始普及,但在胡柚上仍是空白。因此,急需开发基于DNA 水平的分子标记,建立品种/优系/优株鉴定体系,以保障苗木的准确性和纯度,促进产业发展。
笔者在本研究中旨在利用全基因组重测序技术对01-7a 胡柚和普通胡柚ZZ(祖宗树)进行重测序,挖掘出两个材料之间的差异SNP,通过将SNP 转化成AS-PCR 和dCAPS 标记,以期区分01-7 与其他胡柚,填补胡柚分子鉴别研究上的空白,解决胡柚苗木混杂问题,为产业上实施新品种种苗鉴别提供技术保障。
1 材料和方法
1.1 试验材料
用于重测序的普通胡柚ZZ(祖宗树)幼嫩叶片采摘于浙江省衢州市常山县青石镇澄潭村,用于重测序的01-7a 幼嫩叶片采摘于浙江大学(常山)现代发展研究中心试验示范基地。用于标记应用检验的12 份胡柚材料覆盖主产区主要基地和主要类型,具体包括取自4个基地的01-7(01-7a、01-7b、01-7c、01-7d)、3 个基地的普通胡柚(HYa、HYb、HYc)、2 个胡柚优株(HYYa、HYYb)、脆红(CH)、红肉胡柚(HR)和夏红橘柚(又名红皮胡柚,XH)(表1)。
表1 用于标记应用检验的12 份胡柚资源信息表
Table 1 Information of 12 huyou accessions involved in marker application evaluation

编号No.1 2 3 4 5 6 7 8 9材料名称Name 01-7a缩写Abbreviation 01-7a 01-7b 01-7c 01-7d普通胡柚a Huyou a 01-7b 01-7c 01-7d HYa普通胡柚b Huyou b普通胡柚c Huyou c胡柚优株a Huyou elite plant a HYb HYc HYYa胡柚优株b Huyou elite plant b HYYb 10脆红Cuihong CH 11红肉胡柚Hongrou huyou HR 12夏红橘柚Xiahong XH来源Source浙江大学(常山)现代发展研究中心试验示范基地Experimental Station of Zhejiang University (Changshan) Modern Agricultural Research &Development Center常山县球川镇馒头山村Mantoushan Village,Qiuchuan Town,Changshan County常山县同弓乡同心村Tongxin Village,Tonggong Town,Changshan County常山县白石镇新安村Xinan Village,Baishi Town,Changshan County浙江大学(常山)现代发展研究中心试验示范基地Experimental Station of Zhejiang University (Changshan) Modern Agricultural Research &Development Center常山县同弓乡中和村Zhonghe Village,Tonggong Town,Changshan County常山县大桥镇袁青口村Yuanqingkou Village,Daqiao Town,Changshan County浙江大学(常山)现代发展研究中心试验示范基地Experimental Station of Zhejiang University (Changshan) Modern Agricultural Research &Development Center浙江大学(常山)现代发展研究中心试验示范基地Experimental Station of Zhejiang University (Changshan) Modern Agricultural Research &Development Center浙江大学(常山)现代发展研究中心试验示范基地Experimental Station of Zhejiang University (Changshan) Modern Agricultural Research &Development Center浙江大学(常山)现代发展研究中心试验示范基地Experimental Station of Zhejiang University (Changshan) Modern Agricultural Research &Development Center衢州市龙游县团石村Tuanshi Village,Longyou County,Quzhou City
1.2 DNA提取
用十六烷基三甲基溴化铵(CTAB)法[22]提取胡柚基因组DNA,将DNA稀释至100 ng·μL-1备用。
1.3 全基因组重测序分析
采集01-7a 和普通胡柚ZZ 幼嫩叶片,委托深圳华大基因科技有限公司进行全基因组重测序。使用DNBSEQ-T7 测序仪,采用PCR-free 文库构建方法,测序读长为PE150。利用fastqc 对原始数据进行过滤,用trimmomatics v0.39 去除接头和低质量数据。以晚白柚基因组(http://citrus.hzau.edu.cn/data/Genome_info/HWB.v1.0/HWB.v1.0.genome.fa)为参考基因组建立索引,利用BWA-MEM v0.7.17[23]将测序片段比对到参考基因组上,利用shell脚本去掉mapping 质量值小于30 的片段。使用PICARD(http://broadinstitute.github.io/picard/)去除重复和冗余片段。使用BCFtools v1.9[24]和GATK v3.6[25]进行SNPcalling,取交集位点做后续分析。
1.4 纯合SNP挖掘
从检测到的全基因组变异中挖掘纯合SNP,方法和参数如下:(1)使用vcftools软件初次过滤,去除碱基质量值小于30、测序深度大于60 或小于10 的位点,筛选出二等位位点;(2)使用GATK 软件的VariantFiltration 模块进行二次过滤,过滤条件为“QUAL<30.0 || QD<2.0 || MQ<40.0 || FS>60.0 ||SOR>3.0”。在以上过滤条件下得到高质量的SNP集合,筛出集合中01-7a 和普通胡柚ZZ 之间的纯合差异SNP,最后结合IGV 基因组浏览器检查这些位点在bam 文件中的信息,筛选等位基因频率大于90%的SNP,选择其中一个SNP 用于序列验证及后续分子标记开发。
1.5 含SNP片段的克隆与测序验证
采用基因克隆和Sanger 测序进行SNP 验证。根据vcf 文件中的定位信息从参考基因组提取SNP上下游各600 bp序列,使用Primer3web version 4.1.0(https://primer3.ut.ee/)在SNP两侧设计引物,上游引物 Chr1_7111834_G/A- F 序列为:5'- TGTCTGGGTTCGTTTGTAATCA- 3',下游引物 Chr1_7111834_G/A-R 序列为:5'-CTCCAAAAGTCCAGCACAGG-3'。以01-7a 和普通胡柚ZZ 基因组DNA为模板,使用诺唯赞高保真DNA 聚合酶产品Phanta Max Super-Fidelity DNA Polymerase(P505)进行PCR 扩增。PCR 反应体系为:1 μL Phanta Max Super-Fidelity DNA Polymerase,25 μL 2 × Phanta Max Buffer,1 μL dNTP Mix,2 μL 10 μmol·L-1引物(上游引物和下游引物各2 μL),0.1 μg 模板DNA,补足ddH2O 至50 μL。PCR 循环程序为:95 ℃10 min;95 ℃10 s,57 ℃15 s,72 ℃30 s,35 个循环;72 ℃10 min,4 ℃保存。部分PCR 产物直接进行Sanger测序,另外部分PCR 产物进行常规基因克隆操作,选择阳性克隆进行Sanger 测序。采用相同的方法对用于标记应用的12 份胡柚材料的SNP 基因型进行验证。
1.6 AS-PCR反应
应用BatchPrimer3 v1.0(https://wheat.pw.usda.gov/demos/BatchPrimer3/)网站上的工具设计ASPCR 共用引物(下游引物)和2条等位基因特异引物(上游引物),2条等位基因特异引物的差异在于3'末端最后一位碱基,它们分别与SNP 的两个等位碱基互补配对。上游等位基因特异引物AS-PCR-F1 和AS-PCR-F2 的序列分别为:5'-GCAGAGAAATCGCCTTTGTCA-3'和5'-GCAGAGAAATCGCCTTTGTCG-3',下游共用引物AS-PCR-R 的序列为:5'-CGTGTGGTCTCCAAAAGTCC-3'。试验确定的最佳反应体系为:3 μL 2 × Taq Master Mix,0.8 μL 10 μmol·L-1引物(上游引物和下游引物各0.8 μL),30 ng 模板DNA,补足ddH2O 至10 μL。PCR 反应程序为:95 ℃10 min;95 ℃10 s,60 ℃15 s,72 ℃5 s,30 个循环;72 ℃10 min,4 ℃保存。采用1.5%琼脂糖凝胶,在110 V 稳压电泳25 min 后对凝胶进行GelRed染色,置于凝胶成像系统中成像。
1.7 dCAPS扩增与酶切
利用dCAPS Finder 2.0(http://helix.wustl.edu/dcaps/dcaps.html)进行dCAPS 内切酶选择和上游引物设计,引物dCAPS-F 的序列为:5'-GAGTCGCAGAGAAATCGCCTTTGCT- 3'。 应用 Primer3 4.1.0 设计下游引物dCAPS-R,序列为:5'-CTGTGAAGTTTCAATTGGCCCATTA-3'。PCR 反应体系为:25 μL 2×Taq Master Mix,2 μL 10 μmol·L-1引物(上游引物和下游引物各2 μL),160 ng模板DNA,补足ddH2O 至50 μL。PCR 反应程序为95 ℃10 min;95 ℃10 s,58 ℃15 s,72 ℃20 s,35 个循环;72 ℃10 min,4 ℃保存。扩增产物经纯化回收后用于酶切,DNA 纯化使用杭州新景生物试剂开发有限公司的凝胶DNA 回收试剂盒&DNA 纯化试剂盒。酶切反应体系为:2 μL 10×FastDigest Green Buffer,2.6 μL FspBI,0.5 μg PCR 纯化产物,补足ddH2O 至20 μL。酶切反应条件为:37 ℃,4 h。采用3%琼脂糖凝胶,在110 V 稳压电泳30 min,之后凝胶经Gel-Red染色后成像。
2 结果与分析
2.1 全基因组重测序分析
通过对01-7a胡柚和普通胡柚ZZ原始数据进行去污染、去接头及去低质量数据后,共获得高质量片段数120.21 M,高质量碱基数36.06 Gb。Q20和Q30的平均得分分别为96.55%和89.97%,可知数据质量较高。将测序片段与晚白柚参考基因组进行比对,平均碱基比对率达98.54%,1×基因组覆盖率达95%,表明可以以晚白柚作为参考基因组进行胡柚分析。平均测序深度达到46.75×,可以用于亲缘关系相近样本的重测序分析。测序数据质量评估统计结果如表2所示。
表2 测序数据质量评估统计
Table 2 Statistics of quality evaluation of sequencing data

样品Sample 01-7a普通胡柚ZZ Ordinary huyou ZZ高质量片段Clean reads 60 151 675 60 055 719高质量碱基Clean base/bp 18 045 502 500 18 016 715 700 Q20/%96.79 96.31 Q30/%90.60 89.34 GC/%37 37平均测序深度Average depth/(×)47.30 46.20 1×覆盖率1×Cover ratio/%95.10 95.20 10×覆盖率10×Cover ratio/%87.70 87.80碱基比对率Mapping rate/%98.48 98.60
2.2 SNP检测和验证
比较01-7a 和普通胡柚ZZ 的基因型,得到530 286个候选SNP。筛选质量值大于30、测序深度在10 到60 之间等的高置信度SNP,得到96 114 个,在此基础上筛选01-7a 和普通胡柚ZZ 之间的纯合SNP,得到23 个。利用IGV 基因组浏览器检查bam文件中23个SNP位点测序片段的碱基分布情况,筛选出9 个等位基因频率大于90%的纯合SNP,选择其中一个位于1号染色体上第7 111 834个碱基位上的SNP(命名为Chr1_7111834_G/A)用于序列验证。检查该SNP 所在位点的注释信息,发现其位于基因间区域。
在该SNP 两侧设计了一对PCR 引物用于测序验证,PCR扩增得到预期长度为498 bp的条带,且无论是PCR 产物直接Sanger 测序还是经连接转化后的质粒Sanger测序,都表明01-7a在这个位点的基因型是A/A,而普通胡柚ZZ 是G/G(图1),与生信挖掘出的基因型一致,表明该纯合SNP真实存在。
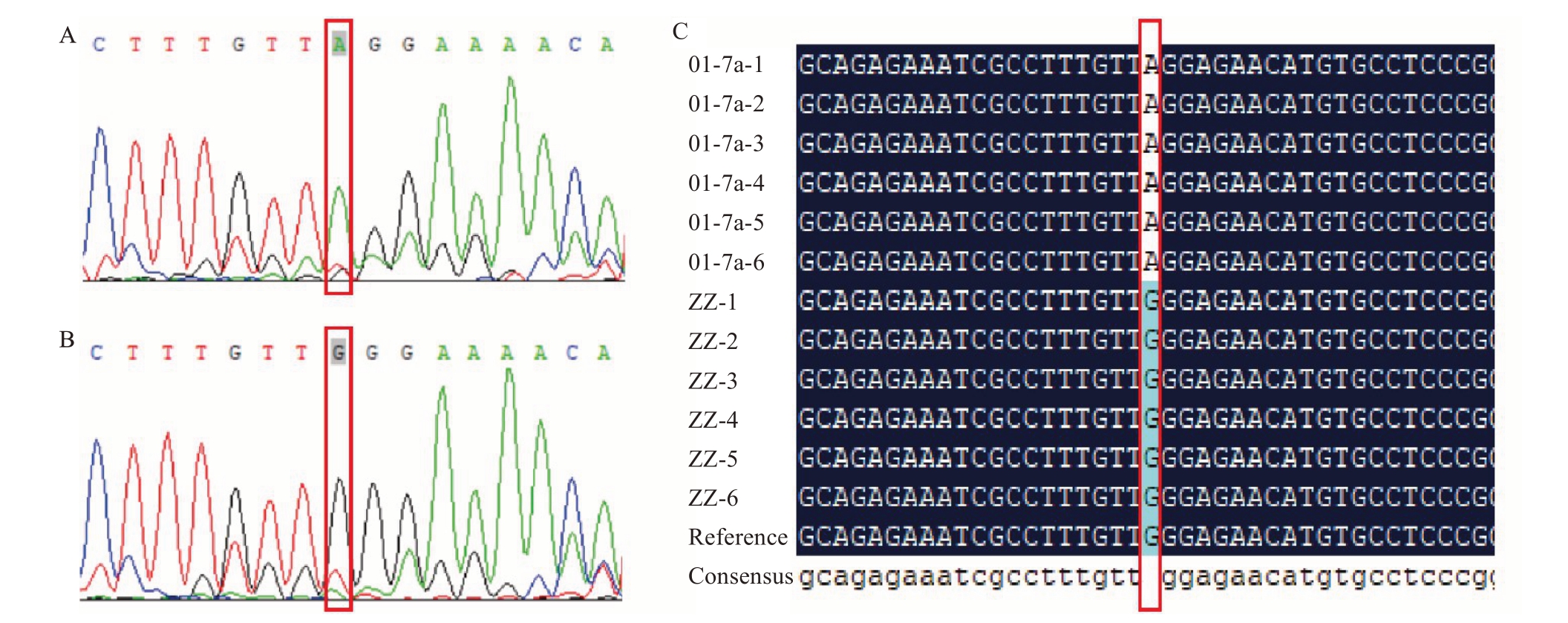
图1 01-7a 和普通胡柚ZZ 间SNP 的Sanger 测序验证
Fig.1 Sanger sequencing validation of the SNP between 01-7a huyou and ordinary huyou ZZ
A.01-7a 胡柚的PCR 产物测序图谱;B.普通胡柚ZZ 的PCR 产物测序图谱;C.01-7a 和普通胡柚ZZ 各6 个重组质粒测序结果比对图,竖框所示的是SNP(Chr1_7111834_G/A)位点。
A.Sequencing electropherogram of the PCR product of 01-7a; B.Sequencing electropherogram of the PCR product of the ordinary huyou ZZ;C.Comparison of the sequencing results of the recombinant plasmids harboring the PCR product from either 01-7a or the ordinary huyou ZZ; The boxed area indicates the SNP(Chr1_7111834_G/A)locus.
2.3 AS-PCR和dCAPS引物设计
鉴于01-7a 和普通胡柚ZZ 在Chr1_7111834_G/A 位点处具有一个纯合SNP,笔者在本研究中旨在基于此SNP 开发能够鉴别两个胡柚材料的AS-PCR和dCAPS引物。
由于AS-PCR 引物3'末端碱基的不配对并不能完全抑制引物和模板的结合延伸,因此在等位基因特异引物的3'末端倒数第二位引入1 个碱基的额外错配(图2)。研究设计了两对AS-PCR引物,其中下游引物共用,而上游引物具有等位基因特异性:ASPCR-F1 和AS-PCR-F2 引物的3'末端碱基分别与01-7a 和普通胡柚ZZ 相同(图2)。预期使用AS-PCRF1只能从01-7a扩增出的条带,同理AS-PCR-F2只能从普通胡柚ZZ中扩增出条带,条带长度为205 bp。
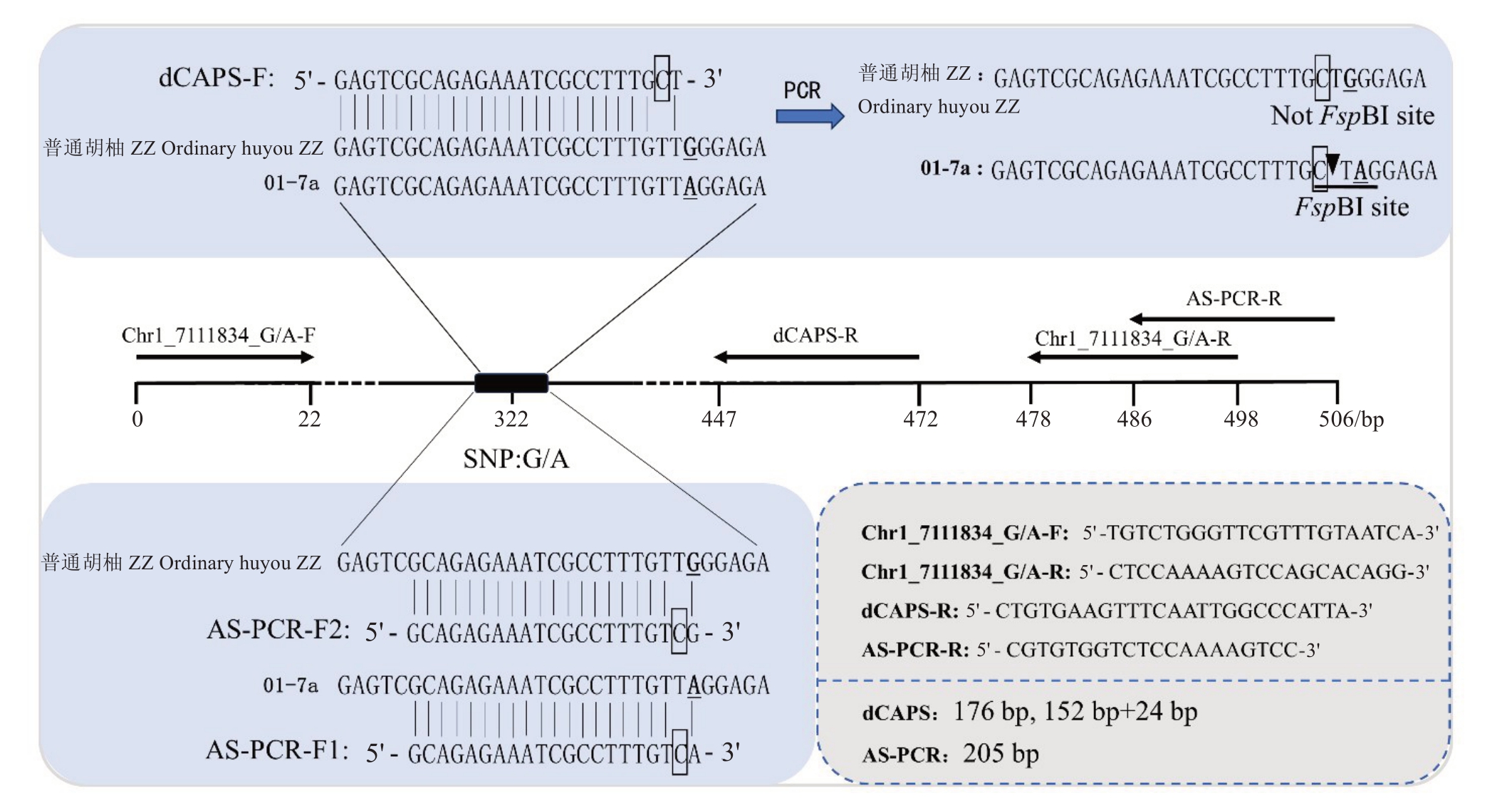
图2 AS-PCR 和dCAPS 分子标记引物
Fig.2 Schematic diagram representing the primers for AS-PCR and dCAPS molecular markers
带框的碱基C 是人为引入的错配碱基,加下划线的是SNP(Chr1_7111834_G/A)位点组成碱基。
The artificial mismatch base C was boxed and the SNP(Chr1_7111834_G/A)comprising bases are underlined.
因SNP 位点不是限制性内切酶的识别位点,不能直接开发酶切扩增多态性(CAPS)标记。基于SNP 侧翼序列,通过人为引入如图2 所示的错配碱基C,其中一个等位基因的PCR 扩增产物就拥有FspBI酶切位点,因而开发了一对dCAPS标记引物(图2)。该标记的预期结果是:以两个胡柚材料DNA 为模板时均可扩增出长为176 bp的条带,但只有当以01-7a 胡柚DNA 为模板时,所扩增的条带可以被FspBI酶切成长度分别为152 bp和24 bp的条带(图2)。
2.4 AS-PCR和dCAPS标记开发
应用所设计的两对AS-PCR 引物,以01-7a和普通胡柚ZZ 的DNA 为模板,PCR 扩增结果如图3-A所示。对01-7a 使用AS-PCR-F1 为上游引物时可扩增出与预期长度205 bp 相符的条带,而以AS-PCRF2 为上游引物时则没有扩增,由此判断01-7a 在Chr1_7111834_G/A 位点的基因型为A/A;以普通胡柚ZZ DNA 为模板时,扩增结果则和01-7a 正好相反,即以AS-PCR-F2 为上游引物时可扩增得到与预期长度205 bp 相符的条带而以AS-PCR-F1 为上游引物时则没有扩增,判断普通胡柚ZZ 的基因型是G/G。AS-PCR 扩增结果与预期结果一致,表明该AS-PCR标记正确有效。
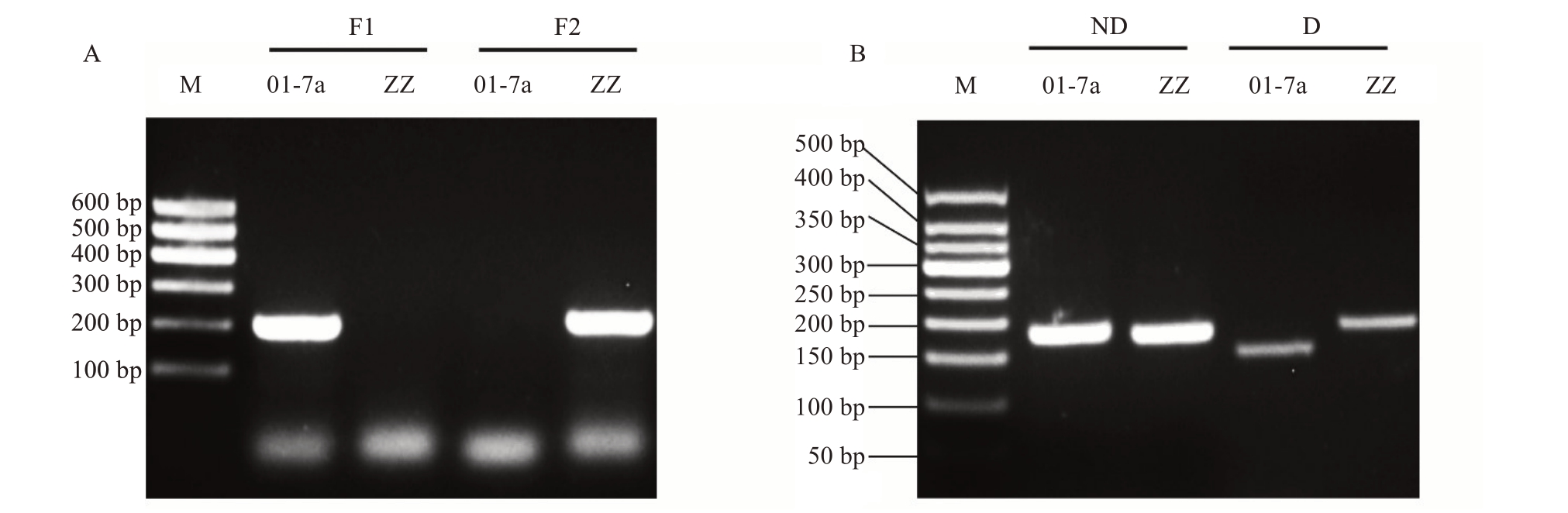
图3 01-7a 和普通胡柚ZZ 的AS-PCR 和dCAPS 反应
Fig.3 AS-PCR and dCAPS reactions for 01-7a huyou and ordinary huyou ZZ
A.AS-PCR 反应,F1 和F2 分别为AS-PCR-F1 和AS-PCR-F2 引物对;B.dCAPS 反应,ND 和D 分别指不酶切对照和酶切反应;ZZ 指代普通胡柚ZZ。M.DNA 长度标记。
A.AS-PCR reaction, F1 and F2 represent for the AS-PCR-F1 and AS-PCR-F2 primer pairs, respectively; B.dCAPS reaction, ND and D refer to the non-digested control and digestion reaction,respectively;ZZ stands for ordinary huyou ZZ.M.DNA Marker.
应用所设计的一对dCAPS引物,以01-7a胡柚和普通胡柚ZZ DNA为模板,PCR扩增结果如图3-B所示,均获得与预期长度176 bp 相符的条带。但只有以01-7a 胡柚DNA 为模板时,PCR 产物可被酶切成两条长度分别为152 bp和24 bp的条带,由此判断01-7a 在Chr1_7111834_G/A 位点的基因型为A/A;以普通胡柚ZZ DNA 为模板时,PCR产物不会被酶切,表明普通胡柚ZZ 的基因型不是A/A。dCAPS 分析结果与预期结果一致,表明该dCAPS标记正确有效。
2.5 AS-PCR 和dCAPS 标记在12 份胡柚材料鉴别中的应用
为进一步验证AS-PCR标记和dCAPS标记的有效性,将标记应用于12份胡柚材料的鉴别。这12份胡柚材料覆盖主产区的主要基地和主要品种,包括01-7 胡柚4 份、其他胡柚8 份(表1)。结果表明,ASPCR-F1 特异引物在所有4 份01-7 胡柚材料中均扩增出预期长度的条带,而在其他胡柚材料中均未得到扩增(图4-A);AS-PCR-F2则在所有4份01-7材料和脆红中不出现扩增条带,而在其余胡柚中均出现条带(图4-B)。dCAPS 标记分析表明,所有胡柚材料均扩增出176 bp 的条带(图5-A)没有差异,但在使用限制性内切酶酶切后,4 份01-7 胡柚材料均出现152 bp的酶切产物,而3份普通胡柚、胡柚优株a、胡柚优株b、红肉胡柚、夏红中条带未被酶切(图5-B)。除脆红外,以上结果均符合预期。脆红在两种标记分析时均表现出非预期的情况,表现为在两个AS-PCR 反应中均未能扩增出条带,在dCAPS 分析时经酶切产生长度既非152 bp 也非176 bp 的片段。笔者在后文中通过分析Sanger 测序结果对此现象作出了解释。
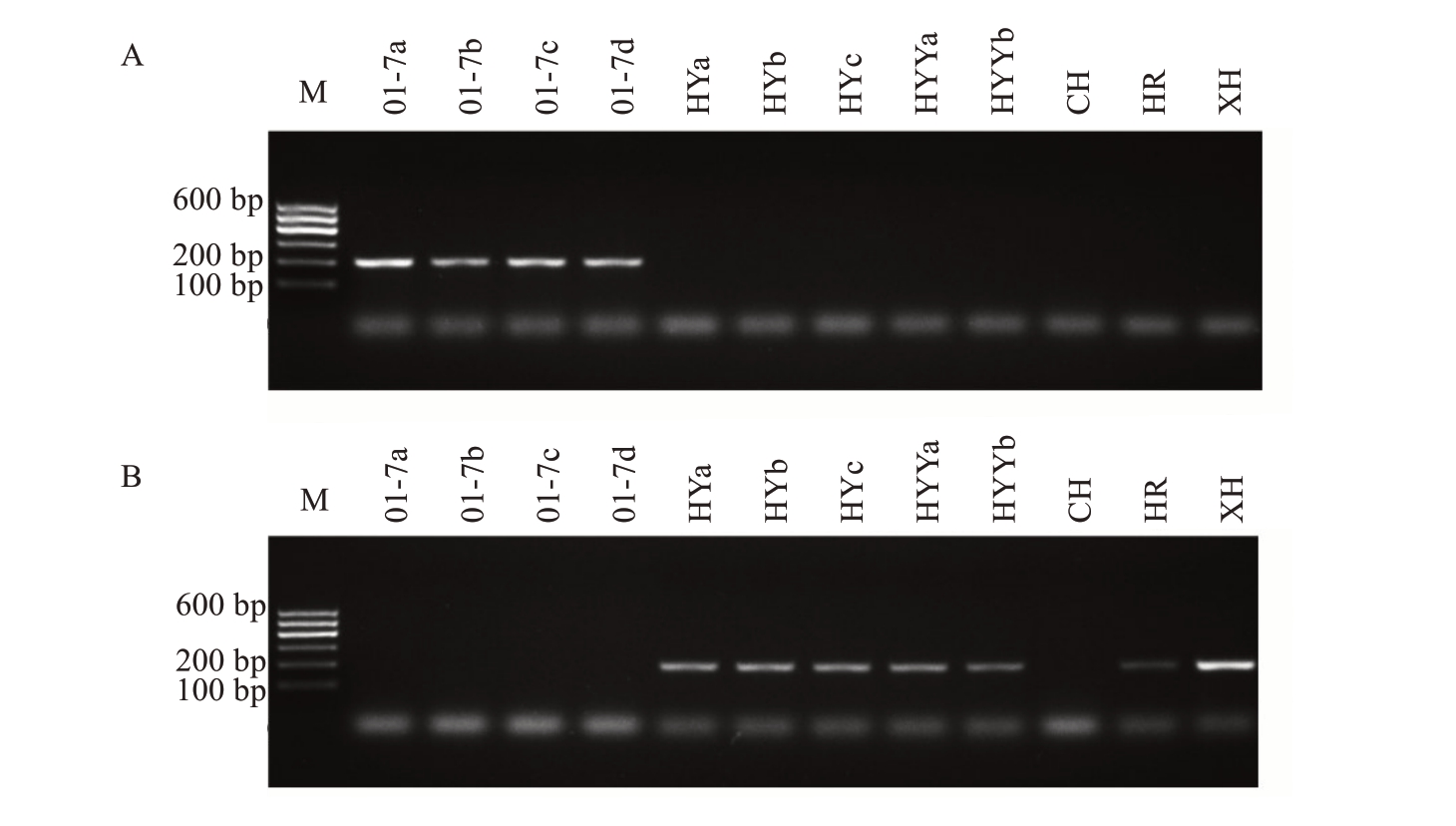
图4 AS-PCR 分子标记在12 份胡柚材料鉴别中的应用
Fig.4 Application of AS-PCR molecular markers in discrimination of 12 huyou accessions
A.AS-PCR-F1 引物对;B.AS-PCR-F2 引物对。12 份材料信息见表1。M.DNA 长度标记。
A.AS-PCR-F1 primer pairs;B.AS-PCR-F2 primer pairs.Refer to Table 1 for the information on 12 accessions.M.DNA Marker.
图5 dCAPS 分子标记在12 份胡柚材料鉴别中的应用
Fig.5 Application of dCAPS molecular marker in discrimination of 12 huyou accessions
A.酶切前;B.酶切后。12 份材料信息见表1。M.DNA 长度标记。
A.Before digestion;B.After digestion.Refer to Table 1 for the information on 12 accessions.M.DNA Marker.
为确认两种标记在这些胡柚材料中对Chr1_7111834_G/A 位点基因分型表现的可靠性,对12 份材料的这一SNP 位点侧翼序列进行了PCR 扩增、克隆和Sanger 测序验证。测序比对结果表明,所有4份01-7 材料在这个SNP 位点的基因型都是A/A,而其他胡柚基因型都是G/G(图6 竖框所示),与ASPCR 和dCAPS 分析结果一致。同时,测序结果解释了为何脆红在两个AS-PCR 反应中均不能扩增出条带,是因为脆红在等位基因特异引物处有额外的3个碱基突变(图6 左横框所示),导致引物结合模板DNA 的能力被大大削弱。而在dCAPS 分析时脆红出现非预期酶切片段则是因为在这段序列中脆红存在天然的FspBI识别位点CTAG(图6 右横框所示)。值得一提的是,脆红中存在的天然FspBI识别位点与人为设计dCAPS 标记时针对01-7 引入的FspBI识别位点并不重合,而是相距38 bp,因而,脆红PCR产物被FspBI酶切成62 bp和117 bp这两条片段,与01-7 不同。因此,综合运用这两种标记不仅可以鉴别出01-7胡柚,还可以鉴别出脆红。
图6 12 份胡柚材料SNP(Chr1_7111834_G/A)位点测序结果
Fig.6 Sequencing results of 12 huyou accessions at the SNP(Chr1_7111834_G/A)site
竖框所示的是SNP 位点;左边横框所示的是脆红在AS-PCR 引物处的3 个额外突变,右边横框所示的是脆红上的一个碱基突变形成的天然的FspBI识别位点。
The vertical box outlines the SNP site;the left horizontal box outlines the three extra mutations in Cuihong at the AS-PCR primer location,and the right horizontal box outlines the natural FspBIrestriction site formed by one base mutation.
3 讨 论
品种分子鉴别通常更倾向于使用基于PCR 的分子标记,但这些方法中有些复杂耗时,有些则可靠度不高,如RAPD 标记鉴定的特异性不强且重复性较差[26],SSR 标记很难区分遗传十分相似的品种[27]。随着下一代测序技术的兴起,SNPs 因数量丰富、可遗传、突变频率较低、二等位性等特点已成为应用最广泛的基因分型标记[28]。全基因组重测序作为一种高通量测序技术,拥有成熟的测序平台、多样的测序策略、丰富的生信分析工具和数据处理流程,并且因其具有较高的全基因组分辨率,使其非常适合用于基因分型[29]。随着测序成本日益降低,全基因组重测序技术越来越成为挖掘SNP 的重要手段。Pei 等[30]对巨峰、乍娜及其早熟芽变丰早和90-1分别进行了全基因组重测序分析,发现早熟芽变品种中存在丰富的遗传变异,通过Sanger 测序验证了巨峰和丰早中的5个SNP;Guan等[31]利用360份桃种质的全基因组重测序数据筛选并构建了由775 个SNP 组成的微阵列芯片。Serra 等[32]对Rocha 梨的8个无性系进行了基因组重测序,经过变异检测和注释,挖掘了位于编码序列上的216个SNP。笔者在本研究中对01-7a 和代表普通胡柚的祖宗树开展了全基因组重测序,对SNP 进行了挖掘。为便于后续分子标记开发和应用,试验出挖掘纯合SNP 的生物信息学分析参数,并成功获得一个纯合SNP,开发了相应的AS-PCR 和dCAPS 标记,填补了胡柚上的研究空白,也可为其他果树开展类似工作提供参考。
AS-PCR 标记和dCAPS 标记都是基于PCR 的SNP 分型方法,有着操作简单、效率高等优点。同时,两者也存在各自缺点。AS-PCR 标记相较于dCAPS 标记而言更加经济,但其对PCR 反应体系和程序更加敏感,并且较容易出现假阳性[33],因而对PCR 体系中各组分比例、Tm 值、PCR 循环数等都有更加严格的要求,需要通过设置梯度试验解决[34]。dCAPS 标记相较于AS-PCR 标记更稳定,但由于有些内切酶较昂贵,在一定程度上增加了dCAPS 分析的成本。同时,dCAPS 需要试验适宜的酶切时间,既要保障充分酶切又要避免“星活性”[35],以免影响基因型判断。此外,也并非每个SNP 位点都能通过改变一个碱基序列引入酶切识别位点,即有些SNP无法用于dCAPS 标记开发。因此,两种标记方法各有所长,可结合使用。
笔者在本研究中开发的AS-PCR 和dCAPS 标记单独应用即可区分01-7 胡柚、脆红与其他胡柚。分子标记分析以及对SNP 侧翼序列测序结果均表明脆红与其他胡柚存在较大差异,推测其在起源上略有不同,这值得后续进一步研究。夏红橘柚一直存在同物异名现象,它又被称为红皮胡柚。根据SNP 侧翼序列,夏红橘柚与01-7 胡柚、普通胡柚、胡柚优株a、胡柚优株b、红肉胡柚基本一致(同源率高达99.6%),因此,宜称其为红皮胡柚。笔者在本研究中开发的标记尚不能区分除01-7 胡柚和脆红之外的其他胡柚材料,后续可验证更多的纯合SNP 位点并开发相应的标记。
4 结 论
利用01-7a和普通胡柚ZZ(祖宗树)的全基因组重测序数据,找到了一个纯合SNP(Chr1_7111834_G/A),01-7a 和普通胡柚ZZ 的基因型分别为A/A 和G/G。进一步将该SNP 开发成AS-PCR 和dCAPS 标记,在12 份胡柚材料中的应用表明,两个标记单独应用即可将01-7 和其他胡柚区分。由于脆红在SNP 侧翼序列中存在额外变异,导致这两种分子标记还可以将脆红从其他胡柚中鉴别出来。笔者在本研究中开发了能鉴别胡柚品种的分子标记,所开发的分子标记可应用于01-7 胡柚、脆红的苗木真伪鉴别,利于苗木纯度保障,从而助力新优品种的推广种植。基于SNP 侧翼序列,发现脆红与其他胡柚的亲缘关系相对较远,夏红橘柚与除脆红外的10份胡柚材料的序列同源率高达99.6%,表明属于胡柚而非橘柚,应称为红皮胡柚。
[1] 张韵冰.中国柑桔属植物一新种[J].植物研究,1991,11(2):5-7.ZHANG Yunbing.A new species of genus citrus from China[J].Bulletin of Botanical Research,1991,11(2):5-7.
[2] 贝增明,叶杏元.常山胡柚特性及栽培技术[M].北京:中国科学技术出版社,2003:15-17.BEIZengming,YE Xingyuan.Characteristics and cultivation techniques of Citrus changshan-Huyou[M].Beijing:China Science and Technology Press,2003:15-17.
[3] 吴耕民.金柚特性及其栽培技术要点[J].中国柑桔,1987,16(1):16-18.WU Gengmin.The characteristics and cultivation technology points of golden pomelo[J].South China Fruits,1987,16(1):16-18.
[4] 陈力耕,胡西琴,赵四清.关于胡柚起源的分子研究[J].园艺学报,2002,29(3):276-277.CHEN Ligeng,HU Xiqin,ZHAO Siqing.Molecular research on Huyou origin[J].Acta Horticulturae Sinica,2002,29(3):276-277.
[5] 陈士超,杨红,郑勇平,陈跃磊,邱英雄.分子标记鉴定常山胡柚优良基因型的初步研究[J].分子细胞生物学报,2006,39(6):502-508.CHEN Shichao,YANG Hong,ZHENG Yongping,CHEN Yuelei,QIU Yingxiong.Preliminary identification of Citrus changshan- Huyou elite genotypes by molecular markers[J].Journal of Molecular Cell Biology,2006,39(6):502-508.
[6] XU C J,BAO L,ZHANG B,BEIZ M,YE X Y,ZHANG S L,CHEN K S.Parentage analysis of Huyou (Citrus changshanen‐sis) based on internal transcribed spacer sequences[J].Plant Breeding,2006,125(5):519-522.
[7] 毛桑隐,汪丽霞,宋剑锋,刘胜军,王沦,徐强.基于全基因组信息的常山胡柚遗传鉴定[J].果树学报,2023,40(1):25-34.MAO Sangyin,WANG Lixia,SONG Jianfeng,LIU Shengjun,WANG Lun,XU Qiang.Genetic identification of Changshan Huyou based on whole genome information[J].Journal of Fruit Science,2023,40(1):25-34.
[8] VELASCO R,LICCIARDELLO C.A genealogy of the citrus family[J].Nature Biotechnology,2014,32(7):640-642.
[9] 赵维良,黄琴伟,张文婷,岳超,宋剑锋.中药材衢枳壳的基源植物研究[J].中国现代应用药学,2019,36(13):1652-1655.ZHAO Weiliang,HUANG Qinwei,ZHANG Wenting,YUE Chao,SONG Jianfeng.Research for the original plant of Chinese medicinal materials Qu Aurantii fructus[J].Chinese Journal of Modern Applied Pharmacy,2019,36(13):1652-1655.
[10] 俞日梁,陈新建,叶杏元,杨兴良,贝增明,郑国民.常山胡柚品种起源及栽培研究进展[J].浙江林业科技,2006,26(3):83-86.YU Riliang,CHEN Xinjian,YE Xingyuan,YANG Xingliang,BEIZengming,ZHENG Guomin.Study on origin and cultivation of Citrus Changshan-Huyou[J].Journal of Zhejiang Forestry Science and Technology,2006,26(3):83-86.
[11] 林晓洁,丁弘扬,叶镔姬,王超,廖汝玉,李永裕.果树分子标记技术研究进展及在 育种上的应用展望[J].东南园艺,2022,10(3):220-227.LIN Xiaojie,DING Hongyang,YE Binji,WANG Chao,LIAO Ruyu,LIYongyu.Research progress of molecular marker technology in fruit trees and its application prospect in Nai plum(Prunus salicina) breeding[J].Southeast Horticulture,2022,10(3):220-227.
[12] 吴仕蔓,娄兵海,陈传武,唐艳,邓崇岭,武晓晓.应用SSR 荧光标记法构建22 个柚类品种的分子身份证[J].果树学报,2023,40(4):605-614.WU Shiman,LOU Binghai,CHEN Chuanwu,TANG Yan,DENG Chongling,WU Xiaoxiao.Establishment of molecular identity of 22 pomelo varieties using fluorescent labeled SSR markers[J].Journal of Fruit Science,2023,40(4):605-614.
[13] 吉爽秋,王力荣,李勇,朱更瑞,曹珂,方伟超,陈昌文,王新卫,张琦,吴金龙.桃花花型(铃形/蔷薇形)基因型鉴定、分子标记开发与利用[J].果树学报,2023,40(3):422-431.JIShuangqiu,WANG Lirong,LIYong,ZHU Gengrui,CAO Ke,FANG Weichao,CHEN Changwen,WANG Xinwei,ZHANG Qi,WU Jinlong.Identification of peach flower genotype (nonshowy/showy),development of flower-type related molecular markers[J].Journal of Fruit Science,2023,40(3):422-431.
[14] 唐海霞,高瑞,王中堂,张琼.基于SNP 标记的枣高密度遗传连锁图谱重新构建[J].园艺学报,2021,48(11):2275-2285.TANG Haixia,GAO Rui,WANG Zhongtang,ZHANG Qiong.High-density genetic linkage map reconstruction in jujube using SNP markers[J].Acta Horticulturae Sinica,2021,48(11):2275-2285.
[15] FONG S K,KAWASH J,WANG Y F,JOHNSON-CICALESE J,POLASHOCK J,VORSA N.A low malic acid trait in cranberry fruit:Genetics,molecular mapping,and interaction with a citric acid locus[J].Tree Genetics&Genomes,2021,17(1):4.
[16] WANG L,ZHANG S L,JIAO C,LIZ,LIU C H,WANG X P.QTL-seq analysis of the seed size trait in grape provides new molecular insights on seedlessness[J].Journal of Integrative Agriculture,2022,21(10):2910-2925.
[17] DE MORIG,CIPRIANIG.Marker-assisted selection in breeding for fruit trait improvement:A review[J].International Journal of Molecular Sciences,2023,24(10):8984.
[18] HIRAOKA Y,FERRANTE S P,WU G A,FEDERICIC T,ROOSE M L.Development and assessment of SNP genotyping arrays for Citrus and its close relatives[J].Plants,2024,13(5):691.
[19] YOU Q,YANG X P,PENG Z,XU L P,WANG J P.Development and applications of a high throughput genotyping tool for polyploid crops:Single nucleotide polymorphism(SNP)array[J].Frontiers in Plant Science,2018,9:104.
[20] HEO J M,EUN C H,KIM IJ.Identification of late ripening citrus mutant,Ara-unshiu (Citrus unshiu),and its selectable marker[J].Plants,2023,12(19):3355.
[21] PENG Q,WANG L,OGUTU C,LIU J J,LIU L,MOLLAH M D A,HAN Y P.Functional analysis reveals the regulatory role of PpTST1 encoding tonoplast sugar transporter in sugar accumulation of peach fruit[J].International Journal of Molecular Sciences,2020,21(3):1112.
[22] 陈昆松,李方,徐昌杰,张上隆,傅承新.改良CTAB 法用于多年生植物组织基因组DNA 的大量提取[J].遗传,2004,26(4):529-531.CHEN Kunsong,LIFang,XU Changjie,ZHANG Shanglong,FU Chengxin.An efficient macro-method of genomic DNA isolation from Actinidia chinensis leaves[J].Hereditas (Beijing),2004,26(4):529-531.
[23] LIH,DURBIN R.Fast and accurate short read alignment with Burrows-Wheeler transform[J].Bioinformatics,2009,25(14):1754-1760.
[24] LIH,HANDSAKER B,WYSOKER A,FENNELL T,RUAN J,HOMER N,MARTH G,ABECASIS G,DURBIN R,1000 Genome Project Data Processing Subgroup.The sequence alignment/map format and SAMtools[J].Bioinformatics,2009,25(16):2078-2079.
[25] MCKENNA A,HANNA M,BANKS E,SIVACHENKO A,CIBULSKIS K,KERNYTSKY A,GARIMELLA K,ALTSHULER D,GABRIEL S,DALY M,DEPRISTO M A.The Genome analysis toolkit:A MapReduce framework for analyzing nextgeneration DNA sequencing data[J].Genome Research,2010,20(9):1297-1303.
[26] 焦锋,楼程富.RAPD 技术应用中的一些问题及对策[J].西北农业学报,2000,9(4):98-102.JIAO Feng,LOU Chengfu.Problems and its counter measure in application of RAPD[J].Acta Agriculturae Boreali-occidentalis Sinica,2000,9(4):98-102.
[27] HONG J H,CHAE C W,CHOIK J,KWON Y S.A database of simple sequence repeat(SSR)marker-based DNA profiles of citrus and related cultivars and germplasm[J].Horticultural Science and Technology,2016,34(1):142-153.
[28] FLANAGAN S P,JONES A G.The future of parentage analysis:From microsatellites to SNPs and beyond[J].Molecular Ecology,2019,28(3):544-567.
[29] SCHEBEN A,BATLEY J,EDWARDS D.Genotyping-by-sequencing approaches to characterize crop genomes:Choosing the right tool for the right application[J].Plant Biotechnology Journal,2017,15(2):149-161.
[30] PEIM S,LIU H N,WEIT L,YU Y H,GUO D L.Detection and characterization of genome-wide genetic variation associated with the early-ripening phenotype of grape mutants[J].Scientia Horticulturae,2021,285:110195.
[31] GUAN L P,XU Q,CAO K,LIY,ZHU G R,FANG W C,WANG X W,CHEN C W,GUO J,WANG Q,ZHAO Y L,WANG L R.Development of a 775 SNP array for peach based on whole-genome resequencing data,and assessment of the potential of its application[J].Scientia Horticulturae,2021,276:109760.
[32] SERRA O,DE SOUSA R M,GUIMARÃES J B,MATOS J,VI-CENTE P,DE SOUSA M L,SIMÕES F.Genome-wide clonal variability in European pear‘Rocha’using high-throughput sequencing[J].Horticulture Research,2022,9:uhac111.
[33] 罗梅,阴伟晓,罗朝喜.桃褐腐病菌对多菌灵抗性的AS-PCR检测技术[J].植物保护,2020,46(6):136-143.LUO Mei,YIN Weixiao,LUO Chaoxi.An allele-specific PCR for detecting the carbendazim resistance in Monilinia fructicola[J].Plant Protection,2020,46(6):136-143.
[34] 杨芩,付燕,王永清,陶炼,邓群仙,范建新,邓仁菊.枇杷ASPCR 反应体系的建立和优化[J].果树学报,2013,30(1):62-68.YANG Qin,FU Yan,WANG Yongqing,TAO Lian,DENG Qunxian,FAN Jianxin,DENG Renju.Establishment and optimization of AS-PCR reaction system for Eriobotrya[J].Journal of Fruit Science,2013,30(1):62-68.
[35] CONLAN L H,JOSÉ T J,THORNTON K C,DUPUREUR C M.Modulating restriction endonuclease activities and specificities using neutral detergents[J].BioTechniques,1999,27(5):955-960.