光合作用是影响产量和品质形成的重要生理过程,光合作用相关性状基因的定位对提高杏优质高产分子育种效率具有重要的理论意义和应用价值。果实中90%以上的干物质来自叶片的光合同化产物,因此,要提高果树产量与品质首要的问题就是增强叶片的光合作用[1-3]。杏树大多数品种自交不亲和,导致其遗传背景复杂并且杂合度高,致使光合作用相关性状遗传规律的研究相对滞后[4-5]。大量研究表明,植物的光合作用是一个复杂的数量性状,传统果树育种方法在增强光合作用方面具有一定的盲目性,并且选育周期较长,一般在10~15 a(年)[6-7]。另外,杏树为多年生木本植物,杂交群体构建难度比农作物大,也制约了基因QTL 定位研究工作的开展[8]。近年来,随着测序技术与现代分子生物技术的发展,数量性状基因座(QTL)定位分析可以把数量性状变异与基因组上单个座位联系起来,在新基因发掘以及育种实践等方面具有重要的利用价值[9-11]。目前,已在农作物上开展了光合相关性状的QTL 定位研究的工作,并定位到一些主效的QTLs位点[12-16],但是,在果树上的研究相对较少。目前,在其他作物上已经定位和克隆的光合作用相关基因并不能完全解释杏树光合作用的遗传及变异规律,其分子机制有待深入研究。笔者在本项目中以杏树为试验材料进行光合作用QTLs定位的研究,利用光合速率存在显著差异的串枝红(母本)和骆驼黄(父本)的F1群体为作图群体构建遗传连锁图谱,并结合分离群体分组分析(BSA),进一步挖掘控制杏光合作用相关的主效QTLs位点,旨在为杏高光效育种中亲本科学选配及高光效杂交后代筛选提供理论依据。
1 材料和方法
1.1 试验材料
以高光合速率品种串枝红为母本,以低光合速率品种骆驼黄为父本,在2007 年春进行套袋杂交,获得的种子经层积处理萌发后移至温室栽培。2008年春将150 株杂交苗定植在北京平谷马坊杏育种基地,杂交后代株行距1.0 m×2.0 m,树形为开心形。为尽量保证取样的一致性,该工作由同一人完成。样品采集工作于09:00—11:00 进行,每株树从东、南、西、北四个方向选取新梢的嫩芽,将混合后的叶片组织放入液氮中速冻后带回实验室保存,用于后续的DNA提取和建库测序工作。
1.2 杏树遗传群体光合相关性状的测定
杏光合性状的测定参照姜凤超等[17]的方法,并进行了部分改进。于杏树开花后60 d开始测定净光合速率,使用Li-6400XT便携式光合测定仪于北京时间08:30—11:00测定树冠中部外侧向阳1年生枝条功能叶的净光合速率(Pn,photosynthesis rate,μmol·m-2·s-1),蒸腾速率(Tr,transpiration rate,μmol·m-2·s-1)、气孔导度(Gs,stomatal conductance,μmol·m-2·s-1)、胞间CO2 浓度(Ci,internal CO2 concentration,μmol·mol-1)。为保证杏杂交后代光合性状测定的一致性与稳定性,所有操作由同一人完成,并且严格控制测定条件,设定光照度1500 μmol·m-2·s-1(红蓝光源)、叶室温度为25 ℃、气体流速为500 mmol·s-1。根据测定结果的稳定性确定测定的叶片数,一般情况下每株杏树测定3~5枚叶片,每枚叶片均在中部测量1次光合数据。
1.3 光合相关性状的遗传变异分析
数据均采用Excel 2013 进行整理,使用R 语言对群体的表型数据进行统计分析。广义遗传力(H2)及变异系数(CV)的计算参照姜凤超等[17]的方法。
1.4 杏遗传连锁图谱构建与QTLs定位
分别提取亲本和F1杂交群体的DNA构建300 bp插入片段文库进行二代测序,测序数据经fastp 进行质控后,利用bwa 比对至串枝红参考基因组v2.0(https://www.rosaceae.org/species/prunus_armeniaca/genome_v1.0),使用GATK 进行Indel 变异的分析。使用“QD<2.0||FS>200.0||SOR>10.0||MQRankSum<-12.5 || ReadPosRankSum<-8.0”作为过滤条件对获得的变异进行质控。在遗传图谱的构建过程中多个标记对应1个图距按1个标记计算,然后利用Lep-MAP3 构建遗传连锁图谱。结合R 包(qtl2)进行净光合相关性状的QTL 定位,根据定位结果选取适当的LOD值提取主效QTLs位点。
1.5 采用混合分离分析法鉴定杏光合特性的主效位点
杂交后代群体根据测定的光合速率分为高光合/低光合2 组,分别从2 组中随机抽取30 株,构建高光合、低光合2个混池。将每组中30份DNA样品等量混合后进行二代测序。以亲本的SNP 作为参考,筛选纯合且不一致、测序深度均>20 的位点;2 个混池过滤SNP index<0.3、SNP index>0.7 且SNP 深度>10 的位点。利用滑窗分析法计算子代样本中SNP的频率分布,并统计各个窗口内SNP数量,剔除SNP数量<10 的窗口以减少其对后续分析的干扰,以400 kb作为窗口统计SNP密度。以窗口大小400 kb,步长20 kb 计算子代样本中SNP-index,使用曼哈顿图对结果进行展示,并绘制拟合曲线,定位光合作用在染色体上的位置区间。
2 结果与分析
2.1 光合作用相关性状的表型统计分析
杏F1群体的光合速率、蒸腾速率、胞间CO2浓度和气孔导度的变化规律和分布范围如图1所示,2016与2017年具有相似的变化趋势。光合速率变化范围在2016、2017 年分别为67.60~109.10 μmol·m-2·s-1、19.50~92.60 μmol·m-2·s-1,并且不同年份间杂交后代光合速率的趋向性不同,2016 年趋向高光合速率区域,而2017年则趋向低光合速率区域。2016年杂交后代蒸腾速率的变化幅度低于2017 年,不同年份间的蒸腾速率均趋向于高蒸腾的区域。2017年杂交后代胞间CO2浓度的变化幅度高于2017 年,且不同年份间均趋向于高CO2浓度的区域。F1群体气孔导度的变化规律与胞间CO2浓度类似。光合作用相关的4个性状中,只有光合速率和气孔导度有超出本体值的极值存在,表明杂交后代中有极端超亲单株出现,为选育高光合速率与高气孔导度的品种提供了资源。
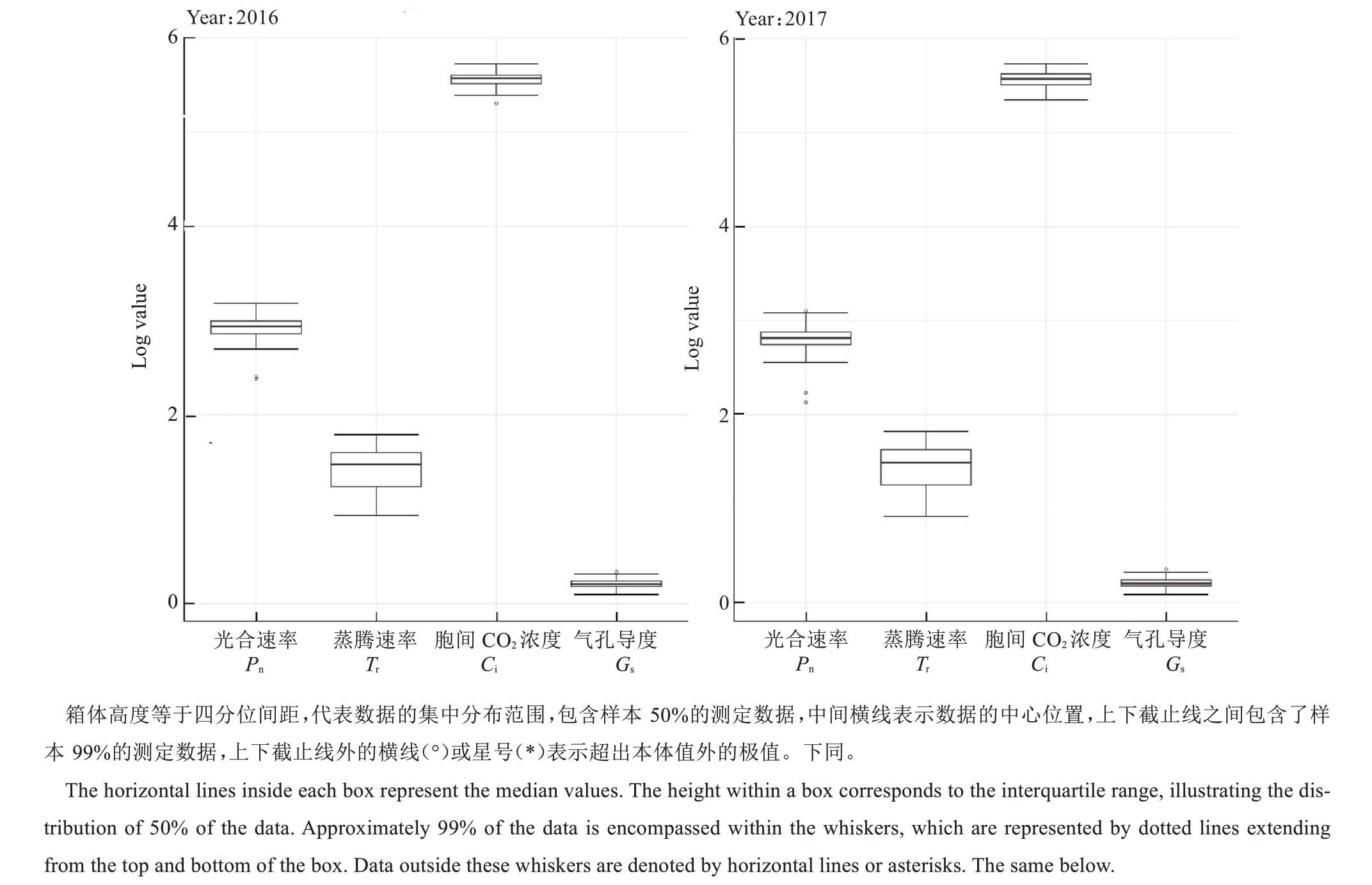
图1 杏F1群体光合相关性状的分布规律
Fig.1 Distribution patterns of photosynthetic traits in the apricot F1 population
2.2 光合作用相关性状的频次分析
杏F1群体光合作用相关性状的频次分布见图2,不同年份间的光合速率、气孔导度、胞间CO2浓度和蒸腾速率均为正态分布,并且不同年份的变化规律相似,表明杏光合作用相关性状是多基因控制的数量性状。2016 年光合相关性状的频次最大值一般高于2017 年,其中,光合速率和胞间CO2浓度的正态分布趋势优于气孔导度和蒸腾速率,更适合于进行QTLs定位研究。

图2 杏F1群体光合相关性状的频次分布图
Fig.2 Frequency distribution of photosynthetic traits in the apricot F1 population
2.3 光合作用相关性状的遗传变异分析
不同年份间(2016与2017年)群体的光合速率、气孔导度、胞间CO2浓度和蒸腾速率的平均值均高于亲中值,表现为超显性遗传(表1)。不同年份间(2016 与2017 年)气孔导度和蒸腾速率在F1群体中均出现广泛分离,它们的变异系数均在20%以上,并且这二者的差异显著高于光合速率和胞间CO2浓度,表明气孔导度和蒸腾速率的遗传选择潜力更大。不同年份间气孔导度和蒸腾速率的广义遗传力(H2)高于光合速率和胞间CO2浓度,均在0.9 以上,光合速率的广义遗传力在2017 年超过0.8,表明这些性状(光合速率、蒸腾速率和胞间CO2浓度)的变异主要受遗传效应的影响,并不易受环境因素影响。
表1 杏F1群体光合作用的遗传变异
Table 1 The genetic variation in photosynthesis of apricot F1 population

年份Year 2016性状Trait Pn Gs Ci 2017 Tr Pn Gs Ci Tr母本Female 19.30±0.38 0.34±0.02 259.90±8.08 2.52±0.09 17.56±1.55 0.31±0.05 257.20±9.97 2.32±0.29父本Male 10.05±0.70 0.10±0.01 200.10±6.38 1.83±0.11 9.35±1.25 0.09±0.01 220.10±9.58 1.81±0.03亲中值Mid-parent 14.67 0.22 229.97 2.18 13.45 0.20 238.68 2.07子代平均值Average 17.68±0.90 0.24±0.02 259.10±8.93 3.25±0.35 17.73±0.43 0.24±0.01 256.90±8.98 3.25±0.26变异系数CV/%12.43 25.27 7.55 28.08 14.88 25.94 8.97 21.30广义遗传传递力H2/%61.94 91.75 60.44 96.24 82.69 85.95 57.16 97.18
2.4 遗传图谱构建与光合作用相关性状的QTLs定位
以串枝红杏基因组为参考,对串枝红×骆驼黄F1群体重测序数据进行indel变异分析,将获得的indel标记经分型与质控后构建高密度遗传图谱,共定位标记1356 个(多个标记对应1 个图距按1 个标记计算),标记平均间距为0.44 cM(图3-A)。基于该图谱对控制光合作用相关性状的基因进行QTLs 定位分析。

图3 标记在连锁群上的分布及光合相关性状的QTLs 定位
Fig.3 Distribution of markers on linkage groups and mapping of QTLs for photosynthesis-related traits
采用qtl2对光合速率、蒸腾速率、气孔导度、胞间CO2浓度等性状进行定位分析,共检测到7个QTLs,分布在1、4、6连锁群上(图3-B,表2),其中,控制光合速率的2个QTL位点均位于连锁群1上,不同年份间定位区间基本一致(2016年62.25~69.00 cM,LOD 值在2.54~2.79 之间;2017 年63.28~69.00 cM,LOD 值在3.96~4.14之间);仅在2016年检测到2个与气孔导度相关的QTLs 位点(61.00~62.25 cM,LOD 值1.68~1.83;104.42~106.45 cM,LOD 值在1.54~1.89 之间),均位于1号连锁群上;仅在2017年检测到1个控制胞间CO2浓度的QTLs位点(65.40~68.68 cM,LOD值在3.03~3.25 之间)。分别在2016 与2017 年检测到1 个控制蒸腾速率的QTLs 位点,分别为13.50~14.85 cM和52.60~53.95 cM,LOD值分别为2.01~2.17与2.20~2.21。光合速率、蒸腾速率和气孔导度在1号连锁群中均定位在62.27~69.00间获得1个显著的QTLs,表明光合作用主效基因可能位于该连锁群上。
表2 杏光合作用相关性状的QTL 分布
Table 2 The distribution of QTLs related to photosynthetic traits in apricot
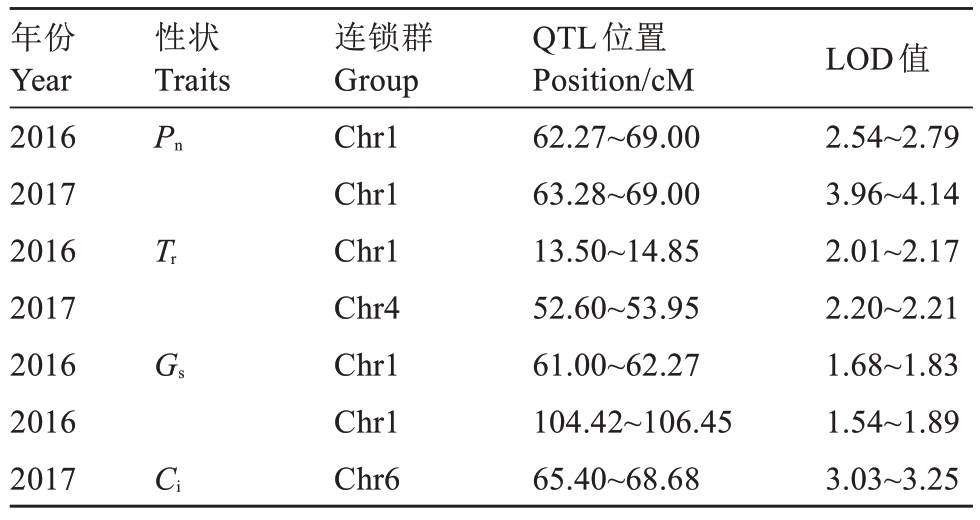
年份Year 2016 2017 2016 2017 2016 2016 2017性状Traits Pn Tr Gs Ci连锁群Group Chr1 Chr1 Chr1 Chr4 Chr1 Chr1 Chr6 QTL位置Position/cM 62.27~69.00 63.28~69.00 13.50~14.85 52.60~53.95 61.00~62.27 104.42~106.45 65.40~68.68 LOD值2.54~2.79 3.96~4.14 2.01~2.17 2.20~2.21 1.68~1.83 1.54~1.89 3.03~3.25
2.5 光合速率BSA定位分析
基于BSA 对控制光合速率的基因进行定位分析,根据SNP密度以窗口大小为400 kb、滑动步长为20 kb,计算子代样本中SNP 的频率分布,分别计算每个窗口SNP 标记位点的SNP index 的均值,并绘制拟合曲线图(图4),其中,计算过程进行1000次置换检验,获得各个位点以及窗口的△(SNP index)95%置信区间(蓝色阈值线,即相当于P-value=5%),以及99%置信区间(红色阈值线,即相当于P-value=1%)。Chr7 染色体负阈值区域:9 800 001~10 600 001 bp,(负阈值为△SNP-index<0,箭头所指处)存在最为显著的候选区域。

图4 基于BSA 的光合速率定位结果
Fig.4 Localization results of photosynthetic rate based on BSA
2.6 光合作用主效基因的筛选
将遗传图谱中的定位信息转换为对应基因组中的物理位置信息,利用基因组的注释文件提取QTLs区间中的基因信息,QTLs区间与物理位置的对应关系及基因数量见表3。光合速率的QTLs 区间内共获得217个候选基因,蒸腾速率共获得186个候选基因,气孔导度共获得111 个候选基因,胞间CO2浓度共得114 个候选基因。光合速率的候选基因富集分析结果表明,候选基因在光合生物的固碳作用(carbon fixation in photosynthetic organisms)通路富集(图5),它与光合速率的关系密切,其中涉及的基因为PA01G03444(phosphoenolpyruvate carboxylase),并且该基因也定位在蒸腾速率和气孔导度的QTLs中,因此,推测该基因可能是控制光合作用的1个主效基因。
表3 杏光合作用性状QTLs 区间的基因信息统计
Table 3 The statistics of gene information in QTL regions for apricot photosynthetic traits
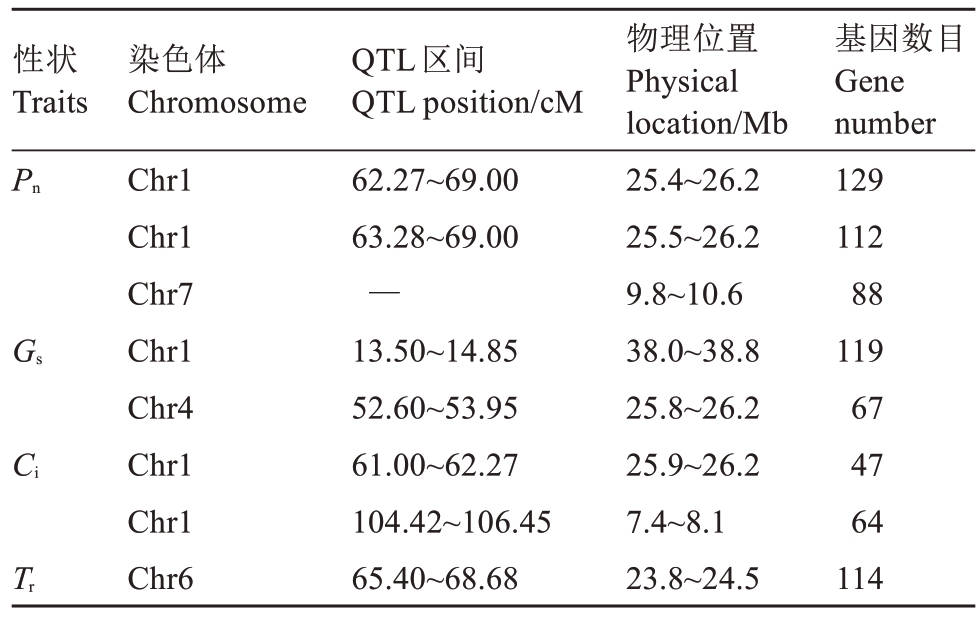
性状Traits Pn Gs Ci Tr染色体Chromosome Chr1 Chr1 Chr7 Chr1 Chr4 Chr1 Chr1 Chr6 QTL区间QTL position/cM 62.27~69.00 63.28~69.00—13.50~14.85 52.60~53.95 61.00~62.27 104.42~106.45 65.40~68.68物理位置Physical location/Mb 25.4~26.2 25.5~26.2 9.8~10.6 38.0~38.8 25.8~26.2 25.9~26.2 7.4~8.1 23.8~24.5基因数目Gene number 129 112 88 119 67 47 64 114
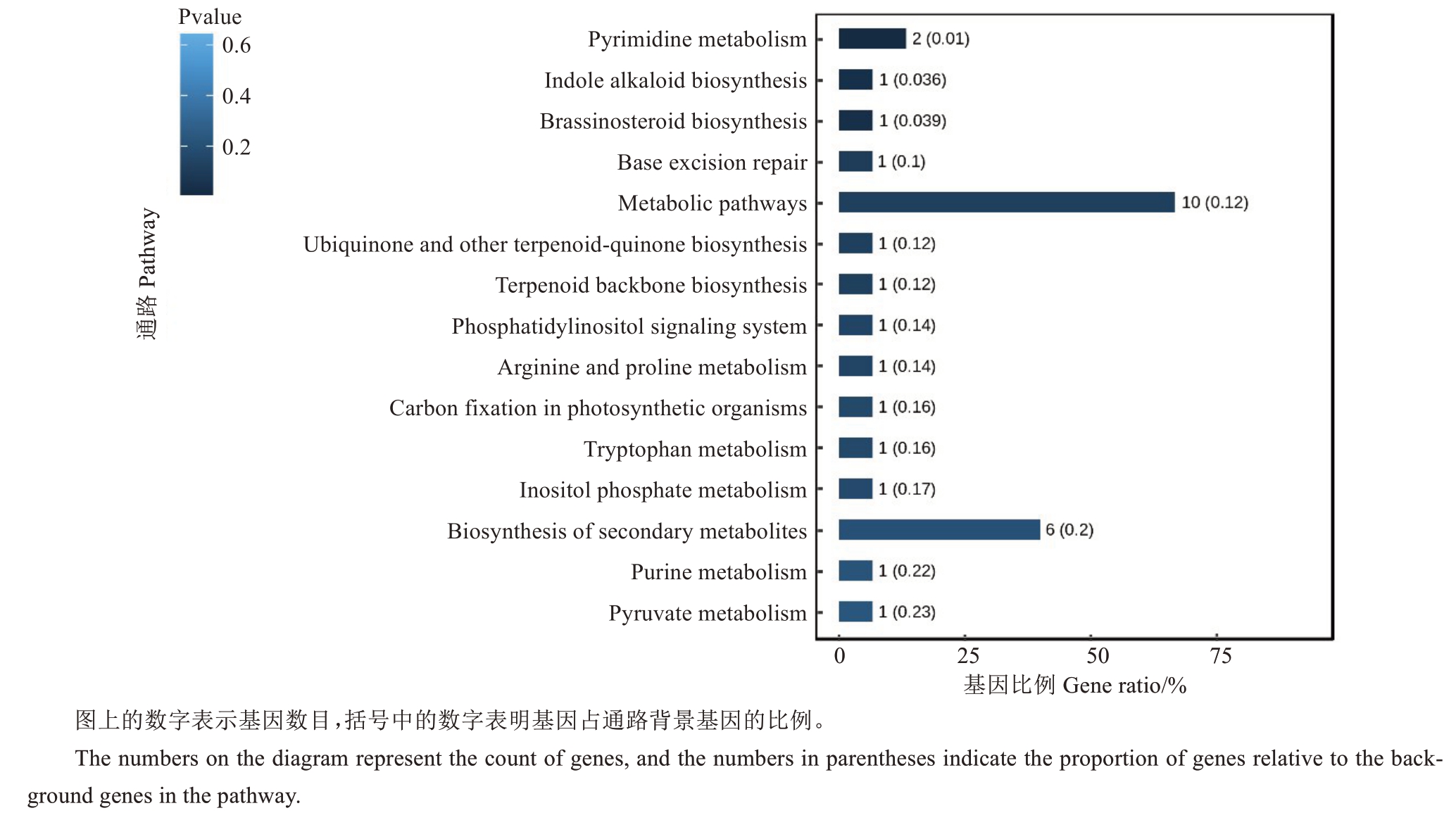
图5 杏光合速率QTLs 区间基因富集分析
Fig.5 Enrichment analysis of genes within QTL intervals of photosynthesis rate for apricot
3 讨 论
光合作用是果树产量和品质形成的基础,是评价品种优劣的一个重要指标,传统果树育种在增强果树光合作用方面具有一定的盲目性,并且选育周期较长,在针对一些复杂性状改良及多性状联合改良的育种过程中更是显得无能为力,因此,急需借助现代分子技术推动育种进程[18-20]。笔者在本研究中,利用QTL 定位将光合性状的变异与基因组上单个座位联系起来,在新基因发掘以及育种实践等方面具有重要的应用价值。基于杏F1群体重测序数据获得了高密度的indel 遗传图谱,共定位到7 个与光合性状相关的QTLs,这为进一步筛选控制光合作用的候选基因奠定了基础。
关于光合作用的遗传变异规律的研究有利于在育种过程中科学选配亲本。笔者在本研究中,发现光合速率、气孔导度、胞间CO2浓度和蒸腾速率均呈正态分布,在杂交后代中呈连续变异,具有典型的数量性状遗传特征,这与对其他物种研究的报道一致[14,21-22]。在本研究中,杏杂交群体中蒸腾速率和胞间CO2浓度的广义遗传力(H2)均在0.80 左右,说明这些性状的变异主要来自遗传效应,受环境影响较小,遗传潜能较大。光合H2在不同年份间存在差异,可能是环境条件(温度、降水、土壤肥力)等对杏的光合作用产生了显著影响,而对其他性状影响较小,从而导致遗传传递力的变化[23-26]。光合相关性状的子代平均值均高于亲中值,呈现超显性遗传,这与相关的报道结果不完全一致,这种现象的原因可能与物种有关。杂交后代中光合速率和气孔导度存在极端超亲单株,这为杏杂交育种提供了亲本资源,使在后代中选育高光合速率和气孔导度的品种成为可能。
遗传图谱是定位目标性状基因并进行分子标记辅助选择的有效工具,在基因定位方面具有显著优势。遗传图谱中标记间的平均距离与密度是衡量遗传图谱质量高低的重要指标,与传统低密度的遗传图谱相比,基于高通量测序技术构建的杏indel遗传图谱,精度达到0.44 cM,表明遗传图谱的质量较高,显著提高了定位的精度。张俊环等[27]采用SALF 标记构建了杏遗传连锁图谱,分别得到母本、父本和中性3 个遗传连锁图谱,最终绘制到遗传图谱上的标记数量2003个,中性连锁图的图距为1 296.885 cM,平均每cM 相当于200 kb 的物理距离。Zhang 等[28]利用对双亲和169 个F1子代进行了SLAF 简化基因组测序,共获得6006个高质量的SNP标记,并进一步构建了包含8 条染色体的高密度遗传连锁图谱,父本和母本连锁群长度分别为809.6 cM和1 076.4 cM,平均遗传距离分别为0.62 cM 和0.95 cM。笔者在本研究中利用全基因组测序技术构建了包含1356个indel共定位标记的高密度遗传连锁图谱,且标记顺序与基因组保持一致,表明该遗传图谱共线性较好,为下一步挖掘控制光合速率的基因奠定了基础。今后,通过分析不同个体的标记的排列顺序,并结合杏基因组和注释信息探索杏杂交育种中重组的热点区域,将为指导杏树育种提供理论指导。
光合作用相关性状的QTLs 为进一步筛选调控光合作用的关键基因奠定了基础。通过对QTLs 区间的基因进行富集分析,结果表明,光合速率与碳固定通路有关,光合速率、蒸腾速率和气孔导度共同涉及的基因为PA01G03444,表明它可能是调控光合作用的主效基因,研究结果为进一步探索杏树光合作用的分子机制提供了基因资源。
4 结 论
基于杏F1群体重测序数据构建了高密度的indel 遗传连锁图谱,共包含1356 个标记,平均间距为0.44 cM。利用复合区间作图法检测到7 个控制杏树光合性状的QTLs,这些位点大部分位于1 号连锁群。分别获得与光合速率、气孔导度、胞间CO2浓度和蒸腾速率相关基因217、186、111 和114 个。经分析发现PA01G03444 是1 个调控光合作用的主效基因,这为筛选控制光合作用的候选基因奠定了基础。
[1] 许大全.光合作用效率[M].上海:上海科学技术出版社,2002.XU Daquan. The efficiency of photosynthesis[M]. Shanghai:Shanghai Scientific&Technical Publishers,2002.
[2] BLANKE M M,LENZ F. Fruit photosynthesis[J]. Plant,Cell &Environment,1989,12(1):31-46.
[3] SALEM A,KHANDAKER M M,MAHMUD K,ALSUFYANI S J,MAJRASHI A A,RASHID Z M,ALENAZI M M,OSMAN N,BADALUDDIN N A.Enhancing photosynthesis and root development for better fruit quality,aroma,and lessening of radioactive materials in key lime (Citrus aurantifolia) using Trichoderma harzianum and Bacillus thuringiensis[J]. Plant Physiology and Biochemistry,2024,206:108295.
[4] 王玉柱.几个优良杏品种授粉组合亲和性试验[J].中国果树,1989(2):33-35.WANG Yuzhu. Compatibility testing of several excellent apricot varieties for pollination combination[J]. China Fruits,1989(2):33-35.
[5] 孙浩元,张俊环,杨丽,姜凤超,张美玲,王玉柱.新中国果树科学研究70 年:杏[J].果树学报,2019,36(10):1302-1319.SUN Haoyuan,ZHANG Junhuan,YANG Li,JIANG Fengchao,ZHANG Meiling,WANG Yuzhu. Fruit scientific research in New China in the past 70 years:Apricot[J]. Journal of Fruit Science,2019,36(10):1302-1319.
[6]FLORE J A,LAKSO A N,JANICK J.Environmental and physiological regulation of photosynthesis in fruit crops[M]//JANICK J.Horticultural Reviews.Timber Press,1989:111-157.
[7] GARRIDO A,CONDE A,SERÔDIO J,DE VOS R C H,CUNHA A N. Fruit photosynthesis:More to know about where,how and why[J].Plants,2023,12(13):2393.
[8] 孙猛,吕德国,刘威生. 杏属植物光合作用研究进展[J]. 果树学报,2009,26(6):878-885.SUN Meng,LÜ Deguo,LIU Weisheng.Advances in research on photosynthesis of apricot[J]. Journal of Fruit Science,2009,26(6):878-885.
[9] TEVFIK H,KARCI H,WAI J,KAFKAS S. Construction of high density linkage maps in almond and synteny analysis with peach,Japanese apricot and sweet cherry genomes[J]. Euphytica,2023,219(8):82.
[10] SONG B,NING W D,WEI D,JIANG M Y,ZHU K,WANG X W,EDWARDS D,ODENY D A,CHENG S F.Plant genome resequencing and population genomics:Current status and future prospects[J].Molecular Plant,2023,16(8):1252-1268.
[11] LALLI D A,ABBOTT A G,ZHEBENTYAYEVA T N,BADENES M L,DAMSTEEGT V,POLÁK J,KRŠKA B,SALAVA J. A genetic linkage map for an apricot (Prunus armeniaca L.)BC1 population mapping plum pox virus resistance[J]. Tree Genetics&Genomes,2008,4(3):481-493.
[12] WANG L,YANG Y M,YANG Z Y,LI W L,HU D Z,YU H L,LI X,CHENG H,KAN G Z,CHE Z J,ZHANG D,ZHANG H Y,WANG H,HUANG F,YU D Y. GmFtsH25 overexpression increases soybean seed yield by enhancing photosynthesis and photosynthates[J].Journal of Integrative Plant Biology,2023,65(4):1026-1040.
[13] YI Q,LÓPEZ-MALVAR A,ÁLVAREZ-IGLESIAS L,ROMAY M C,REVILLA P. Genome-wide association analysis identified newly natural variation for photosynthesis-related traits in a large maize panel[J].Agronomy,2023,13(3):801.
[14] HONDA S,IMAMURA A,SEKI Y,CHIGIRA K,IWASA M,HAYAMI K,NOMURA T,OHKUBO S,OOKAWA T,NAGANO A J,MATSUOKA M,TANAKA Y,ADACHI S. Genomewide association study of leaf photosynthesis using a highthroughput gas exchange system in rice[J]. Photosynthesis Research,2024,159(1):17-28.
[15] THEEUWEN T P J M,LOGIE L L,HARBINSON J,AARTS M G M. Genetics as a key to improving crop photosynthesis[J].Journal of Experimental Botany,2022,73(10):3122-3137.
[16] SUNDARAM R M,BADRI J,ABDUL FIYAZ R,SENGUTTUVEL P,MANGRAUTHIA S K,CHAITHANYA U,NEERAJA C N,SUBBA RAO L V,HARIPRASAD A S. Conventional and new breeding approaches to enhance grain yield in rice[J]. Oryza-an International Journal on Rice,2023,60:1-20.
[17] 姜凤超,孙浩元,杨丽,张俊环,王玉柱.不同生态群杏种质资源的光合特性研究[J].果树学报,2018,35(10):1216-1224.JIANG Fengchao,SUN Haoyuan,YANG Li,ZHANG Junhuan,WANG Yuzhu. Study on the photosynthetic characteristics of apricots in different ecological groups[J]. Journal of Fruit Science,2018,35(10):1216-1224.
[18] EVANS J R. Improving photosynthesis[J]. Plant Physiology,2013,162(4):1780-1793.
[19] SUN C W,HU H T,CHENG Y Z,YANG X,QIAO Q,WANG C G,ZHANG L L,CHEN D Y,ZHAO S M,DONG Z D,CHEN F. Genomics-assisted breeding:The next-generation wheat breeding era[J].Plant Breeding,2023,142(3):259-268.
[20] VARGHESE R,CHERUKURI A K,DODDRELL N H,DOSS C G P,SIMKIN A J,RAMAMOORTHY S.Machine learning in photosynthesis:Prospects on sustainable crop development[J].Plant Science,2023,335:111795.
[21] TAKAI T,OHSUMI A,ARAI Y,IWASAWA N,YANO M,YAMAMOTO T,YOSHINAGA S,KONDO M. QTL analysis of leaf photosynthesis in rice[J]. Japan Agricultural Research Quarterly,2013,47(3):227-235.
[22] YIN X Y,GU J F,DINGKUHN M,STRUIK P C. A modelguided holistic review of exploiting natural variation of photosynthesis traits in crop improvement[J]. Journal of Experimental Botany,2022,73(10):3173-3188.
[23] KADIUM V R,SVYANTEK A.Broad-sense heritability for horticultural production traits in eggplant[J]. Crop Breeding,Genetics and Genomics,2023,5(2):e230003.
[24] ARDIE S W.Gene action and heritability estimates in F2 populations of foxtail millet (Setaria italica L.)[J]. SABRAO Journal of Breeding and Genetics,2024,56(1):65-75.
[25] FARID N,RIYANTO A,WAHYUDININGSIH T S,RAHMIYAH M,HABIBULLAH M,ULINNUHA Z. Performance,genetic variability and heritability of M1 generation mandarin citrus(Citrus reticulata)mutants[J].2023,30(3):169-176.
[26] TITIRICĂ I,ROMAN I A,NICOLA C,STURZEANU M,IUREA E,BOTU M H,SESTRAS R E,POP R,MILITARU M,ERCISLI S,SESTRAS A F. The main morphological characteristics and chemical components of fruits and the possibilities of their improvement in raspberry breeding[J].Horticulturae,2023,9(1):50.
[27] ZHANG J H,SUN H Y,YANG L,JIANG F C,ZHANG M L,WANG Y Z. Construction of a high-density linkage map and QTL analysis for pistil abortion in apricot (Prunus armeniaca L.)[J].Canadian Journal of Plant Science,2019,99(5):599-610.
[28] ZHANG Q P,LIU J C,LIU W S,LIU N,ZHANG Y P,XU M,LIU S,MA X X,ZHANG Y J. Construction of a high-density genetic map and identification of quantitative trait loci linked to fruit quality traits in apricots using specific-locus amplified fragment sequencing[J]. Frontiers in Plant Science,2022,13:798700.