APETALA2/ethylene response factor(AP2/ERF)转录因子处于乙烯信号通路下游,广泛参与果实生长、成熟软化、叶绿素降解、类黄酮和芳香物质的合成,对植物生长发育、逆境响应和其他生物学过程具有重要调控作用[1-6]。香蕉MaERFs通过调控与乙烯生物合成相关的基因表达来调控果实成熟过程[7],进一步研究发现MaDof23 能与MaERF9 结合作为抑制子调控果实成熟[8]。
AP2/ERF 转录因子家族因含有1~2 个约由60个氨基酸组成的AP2/ERF 结构域而得名,根据保守结构域特征和数目分为AP2、ERF、RAV 和Soloist四个亚家族[9]。自Jofuku 等[10]首次从拟南芥中分离出APETALA2(AP2)转录因子后,经过近30 年的发展,已有20种植物中的AP2/ERF家族成员被分离鉴定。其中数目最多的油菜含531 个[11],其次是烟草375 个[12],玉米214 个[13],水稻170 个[14],拟南芥147个[15],番茄134个[16],最少的菠萝也有97个[17]。
在全球大食物观的背景下,香蕉是世界重要的果粮兼用作物,是全球近20 亿人碳水化合物的主要来源。现有的香蕉栽培品种均来源于两个原始祖先种,即尖叶蕉A 基因组(Musa acuminata,A genome)和长梗蕉B 基因组(M.balbisiana,B genome)[18]。然而到目前为止,除了Jourda 等[19]和侯晓婉等[20]在第一版香蕉A基因组测序背景下报道了香蕉A基因组和栽培品种巴西蕉中AP2/ERFs 家族分别有122 和117 个成员外,还没有在全基因组水平上对香蕉AP2/ERF 基因家族进行系统分离鉴定的更新报道。随着测序技术的飞速发展和大数据分析技术的逐步完善,原有的分析结果已适应不了新的发展需求,需要及时更新。笔者在本研究中对香蕉A基因组中的AP2/ERF 家族成员进行全基因组分析,包括系统进化、在染色体上的分布、基因结构、理化特性,分析他们在两个主栽品种巴西蕉(AAA)和粉蕉(ABB)果实采后成熟过程中的差异表达特性,筛选出可能参与香蕉果实成熟和品质调控的关键基因。研究结果为深入解析AP2/ERF 家族成员在香蕉果实成熟过程中的作用奠定基础,为香蕉品质改良和生物育种提供基因资源。
1 材料和方法
1.1 试验材料和处理
盛花期后80 d(80 DAF),即采收后0 d(0 DPH)的香蕉(M.spp.)果实来自中国热带农业科学院热带生物技术研究所香蕉种植园(海南澄迈)。选择处于相似发育阶段的香蕉果实并使其自然成熟。由于果实采后粉蕉果实的乙烯释放量迅速上升使其比巴西蕉更快地达到全黄色,因此巴西蕉(BX)的果实分别在采后第8天和第14天达到乙烯生物合成启动期和高峰期,而粉蕉(FJ)果实分别在采后第3天和第6天达到乙烯生物合成启动期和高峰期。将果实样品在液氮中迅速冷冻并储存在-80 ℃,用于总RNA 提取和转录组分析。
1.2 基因鉴定和进化分析
香蕉MaAP2/ERFs蛋白序列来自香蕉全基因组测序数据库(http://banana-genome-hub.Southgreen.fr/download)[21],番茄的AP2/ERFs 序列来自Yang等[16]的报道。为了鉴定MaAP2/ERFs 超家族成员基因,以已知的MaAP2/ERFs 去搜索香蕉全基因组数据库[21]。随后,将从香蕉全基因组数据库中得到的所有MaAP2/ERFs 与番茄的AP2/ERF 进行比对。所有候选蛋白序列通过CDD(http://www.ncbi.nlm.nih.gov/cdd/)和PFAM(http://pfam.sanger.ac.ac.uk/)数据库进行检验。最后,使用多个序列比对确认预测的MaAP2/ERF 蛋白的保守域。使用Clustal X V.2.0 对香蕉与番茄的AP2/ERFs 全长序列进行多序列比对。采用MEGA 5.0软件构建系统进化树[22]。
1.3 基因结构特征分析
利用香蕉基因组数据库(https://banana-genomehub.southgreen.fr/)获取香蕉A 基因组的gff 功能注释文件,而后利用TBtools 软件中的Visualize Gene Structure 功能对MaAP2/ERFs 家族成员的基因结构进行可视化分析:首先在set input.gff3 界面导入香蕉A 基因组.gff 注释文件,而后在set input ID list 界面导入MaAP2/ERF 家族成员基因编号,即可得到MaERF家族成员基因结构。
1.4 蛋白质特性和保守结构域分析
采用ExPASy 软件(http://expasy.org/)检测预测的MaAP2/ERF 蛋白的分子质量和等电点。利用NCBI在线网站的CD-Search Tool功能(https://www.ncbi.nlm.nih.gov/Structure/bwrpsb/bwrpsb.cgi)对目标蛋白序列进行保守结构域分析,并下载在线分析结果。而后将在线分析结果导入Tbtools,利用Visualize NCBI CDD Domain Pattern 功能对目标蛋白保守结构域进行可视化分析。
1.5 转录组分析
选取80 DAF(0 DPH)、BX 8 DPH 和14 DPH、FJ 3 DPH和6 DPH的香蕉果实,用植物RNeasy提取试剂盒(天根,北京,中国)提取总RNA,进行转录组分析。按照说明书,用Illumina GAⅡ进行测序。每个样本2 次重复。测序深度平均为5.34X。使用FASTX 软件去除原始序列中的接头序列。用FastQC 去除低质量序列。用TopHat v.2.0.10软件将高质量的序列与参考基因组DH-Pahang(M.acumi‐nata,A-Genome,2n=22)进行比对[20]。转录组使用Cufflinks[23]进行组装。基因表达水平计算为每百万映射读取的外显子模型每千碱基读取数(FPKM)。DEGseq 用于鉴定差异表达的基因。RNAseq 数据登陆在NCBI- SRA 数据库中(登录号:PRJNA343716)。
2 结果与分析
2.1 香蕉AP2/ERFs 超家族的鉴定和系统进化分析
为了准确地鉴定出香蕉AP2/ERFs 超家族成员,采用BLAST 和Hidden Markov Mode 两个软件将从香蕉A 基因组数据库(http://banana-genome.cirad.fr/)(2019 年发布版)中得到的AP2/ERFs 超家族成员进行比对和鉴定,共得到317 个家族成员,分为AP2(49 个),ERF(253 个)和RAV(15 个)三个亚家族。他们编码蛋白的氨基酸数量为68~716,分子质量为7.50~76.67 kDa,等电点为4.60~10.34,不稳定系数为31.63~108.56,脂溶指数为44.03~83.47,亲水系数为-1.29~-0.18。与番茄中的AP2/ERFs 家族构建系统进化树,发现ERF 亚家族又分为a、b、c、d、e、f、h、i、j 和k,10 个亚类,其中h 亚类所含的基因数目最多,为67 个;其次是c 亚类,为44 个;再次是i亚类,为37 个,所含基因数目最少的是j 亚类,仅含有1个基因。AP2亚家族被分为3个亚类,所含基因数目最多的有28 个,其次是含有20 个,所含基因数目最少的亚类只有1个基因。RAV 亚家族也被分为3个亚类,所含基因数目最多的有8个,其次是含有6个,所含基因数目最少的亚类也只有1 个基因(图1)。

图1 香蕉A 基因组与番茄AP2/ERFs 家族成员的系统进化分析
Fig.1 Phylogenetic analysis of the AP2/ERFs from tomato and banana
A.AP2 亚家族;B.RAV 亚家族;C.ERF 亚家族。
A.AP2 group;B.RAV group;C.ERF group.
2.2 香蕉AP2/ERFs超家族成员在染色体上的分布特征
香蕉AP2/ERFs 的317 个家族成员不均匀地分布在11条染色体上,其中数量最多的是4号染色体,41个,占12.93%,其中AP2家族3个,RAV家族3个,ERF 家族35 个;其次是3 号染色体,37 个,占11.67%,其中AP2 家族10 个,RAV 家族2 个,ERF 家族25 个;再次是6 号和10 号染色体,均为33 个,占10.41%;6 号染色体上分布5 个AP2,2 个RAV,26 个ERF;10 号染色体上分布3 个AP2,1 个RAV,29 个ERF;数量最少的是1号染色体,仅12个,占3.8%,其中AP2 家族5 个,RAV 家族2 个,ERF 家族5 个(图2)。从253 个ERF 亚家族成员在染色体上的分布规律来看,除了1 号和random 染色体外,其余的10 条染色体上都存在一个或多个由串联重复引起的基因簇,这些基因簇的出现为香蕉更好地适应多变的环境奠定了基础。

图2 香蕉AP2/ERFs 超家族成员在染色体上的分布特征
Fig.2 Distribution of MaAP2/ERFs subfamilies on chromosomes
A、B、C 分别代表ERF、AP2 和RAV 亚家族。
A,B,C represent ERF,AP2 and RAV subfamily,respectively.
2.3 香蕉AP2/ERFs超家族成员的基因结构特征
AP2 亚家族具有7~10 个外显子,6~9 个内含子。RAV 亚家族15 个成员中14 个都是单外显子,2个具有单内含子。ERF亚家族a、b、c、j亚类中只有2个具有单内含子,其余的都是单外显子无内含子结构;d、f、i、h 亚类中大多数也是单外显子,少数有单内含子;k 亚类全都具有2 个外显子和1 个内含子;e亚类中绝大多数具有6 个内含子(图3)。以上结果表明同一亚族不同成员之间具有相似的基因结构,决定其在香蕉生长发育、成熟和对环境适应性等方面可能具有类似作用。
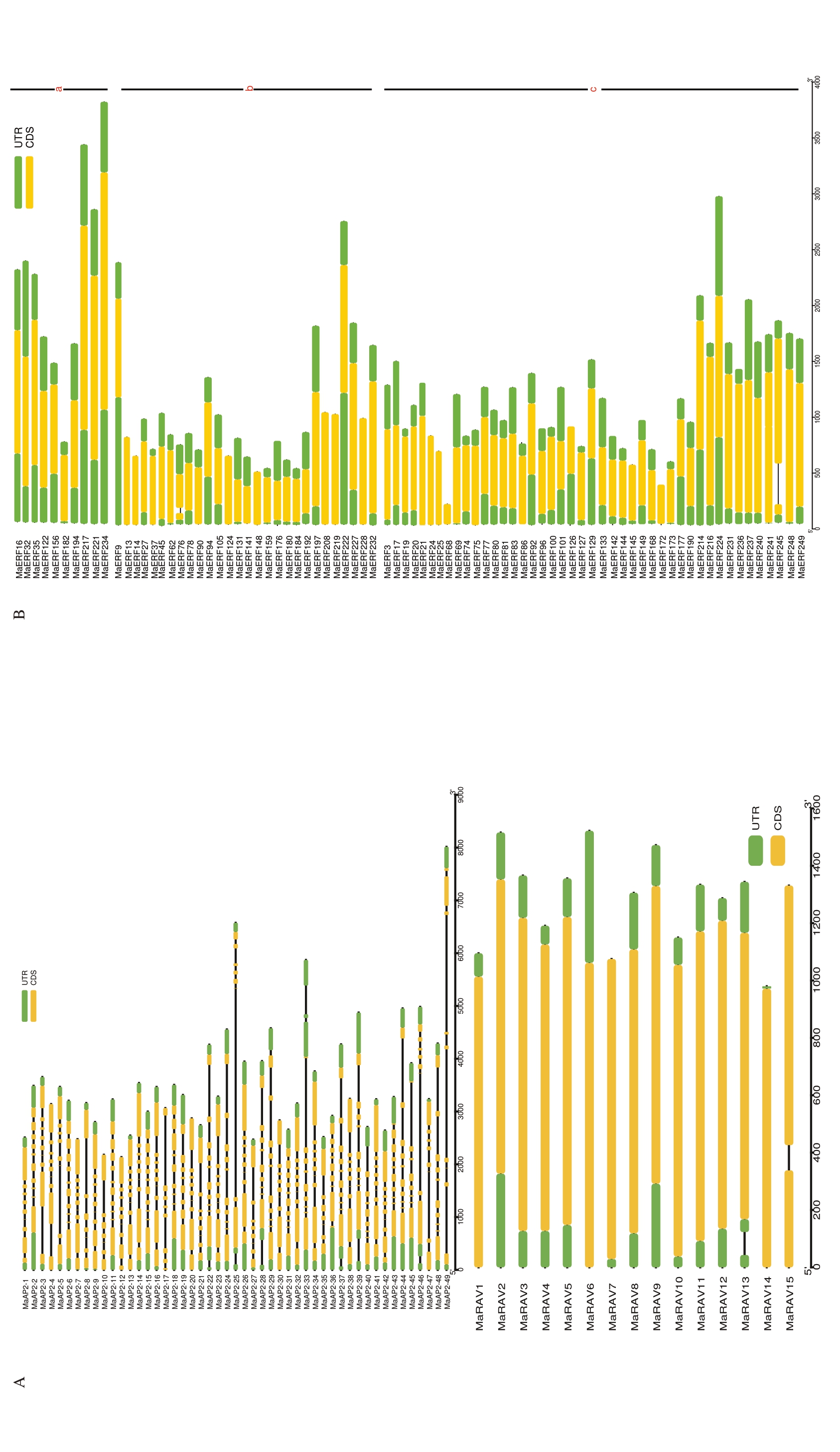
图3 MaAP2/ERFs 的基因结构分析
Fig.3 Gene structure analysis of MaAP2/ERFs
外显子-内含子结构采用GSDS 软件在线分析。A. 上为AP2,下为RAV;B. ERF 亚家族的a、b、c 3 个亚类;C. d、e、f 和i 4 个亚类;D. h、k 和j 3 个亚类。绿色盒代表上/下游结构域,黄色盒代表外显子,黑线代表内含子。
Exon-intron structure analyses were performed by GSDS database. A. the up represents AP2, the down represents RAV; B. a, b, c subgroup of ERF subfamily;C. d, e, f and i subgroup of ERF subfamily; D. h, k and j subgroup of ERF subfamily. The blue boxes indicate upstream/downstream, the yellow boxes indicate exons, and the black lines indicate introns.
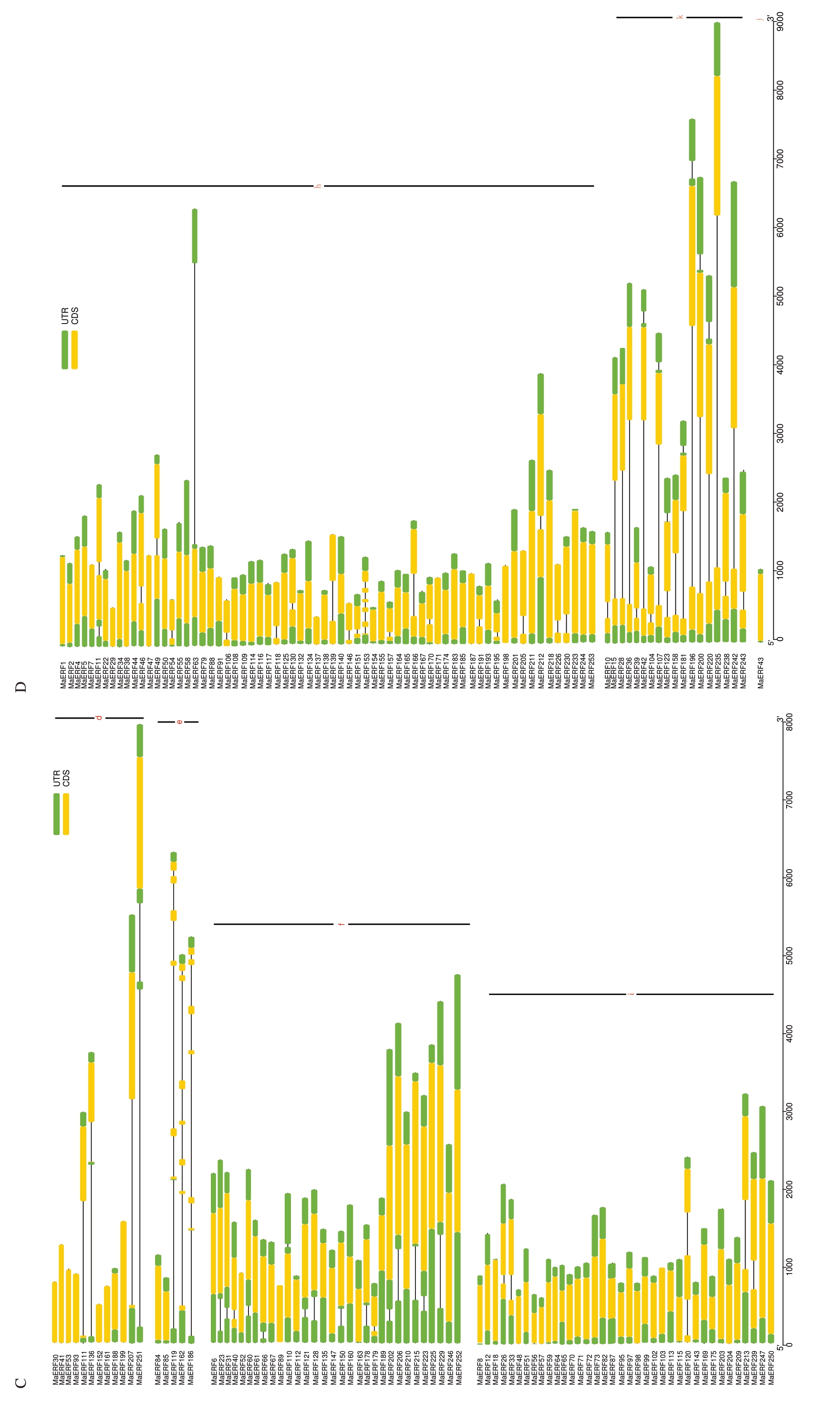
图3 (续) Fig.3 (Continued)
2.4 香蕉AP2/ERFs超家族成员的保守结构域分析
所有AP2/ERFs 超家族成员都具有AP2 和AP2 superfamily 两个保守结构域,因此,AP2结构域是该家族成员执行生物学功能的结构基础。除此之外,AP2 亚家族还具有PHA03247 superfamily,RAV 亚家族还具有B3 superfamily,ERF 亚家族的a、b、c、d、e、f、i 具有DNA-bind superfamily(图4)。这些亚家族成员具有的特异性保守结构域为香蕉AP2/ERFs超家族成员的功能分化奠定了结构基础。
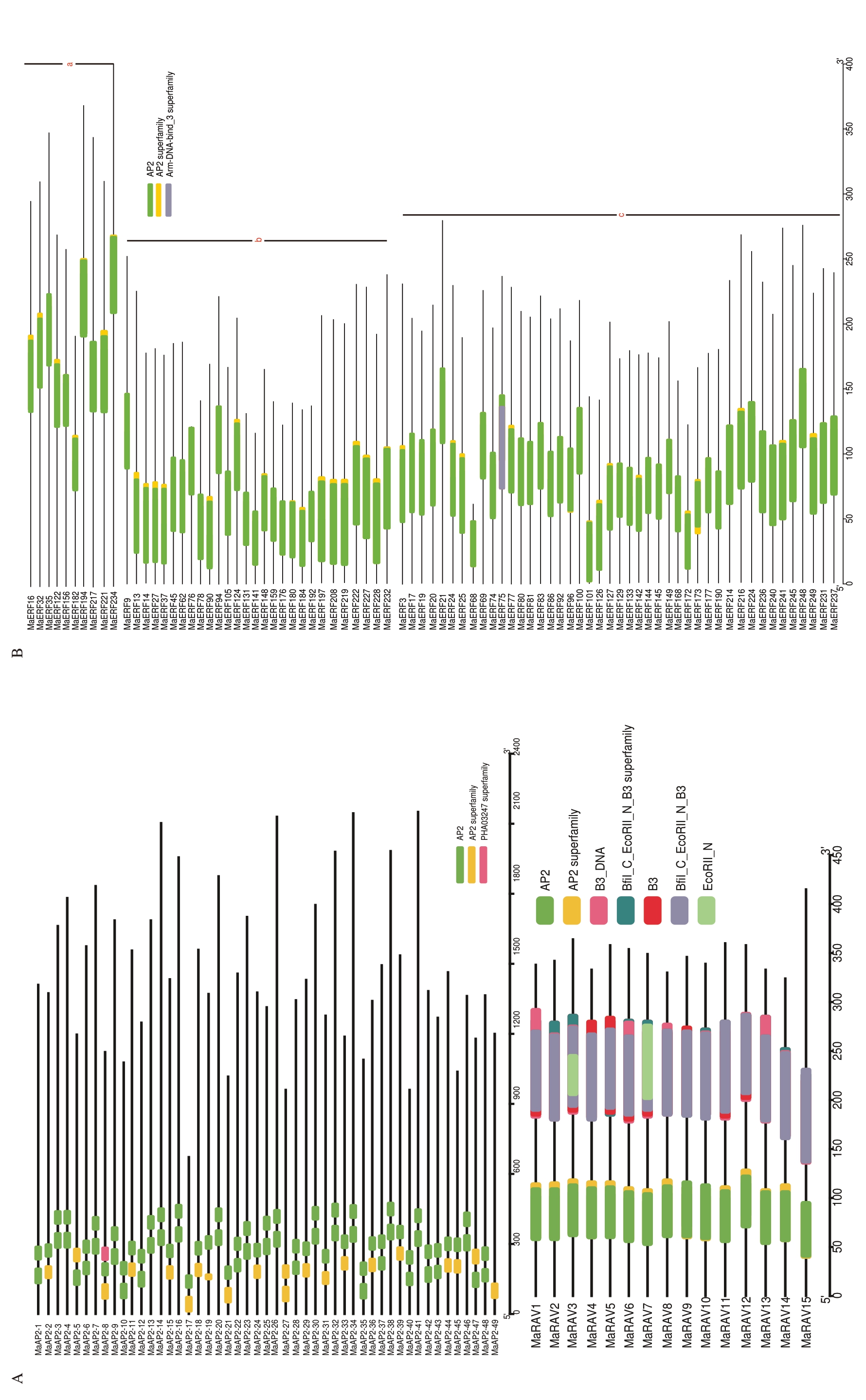
图4 香蕉MaAP2/ERFs 超家族保守结构域分析
Fig.4 Conserved motifs analysis of banana MaAP2/ERFs superfamilies
A.上为AP2,下为RAV;B.ERF 亚家族的a、b、c 3 个亚类;C.d、e、f 和i 4 个亚类;D.h,k 和j 3 个亚类。
A.The up was AP2, the down was RAV; B.a, b, c subgroup of ERF subfamily; C.d, e, f and i subgroup of ERF subfamily; D.h, k and j subgroup of ERF subfamily.
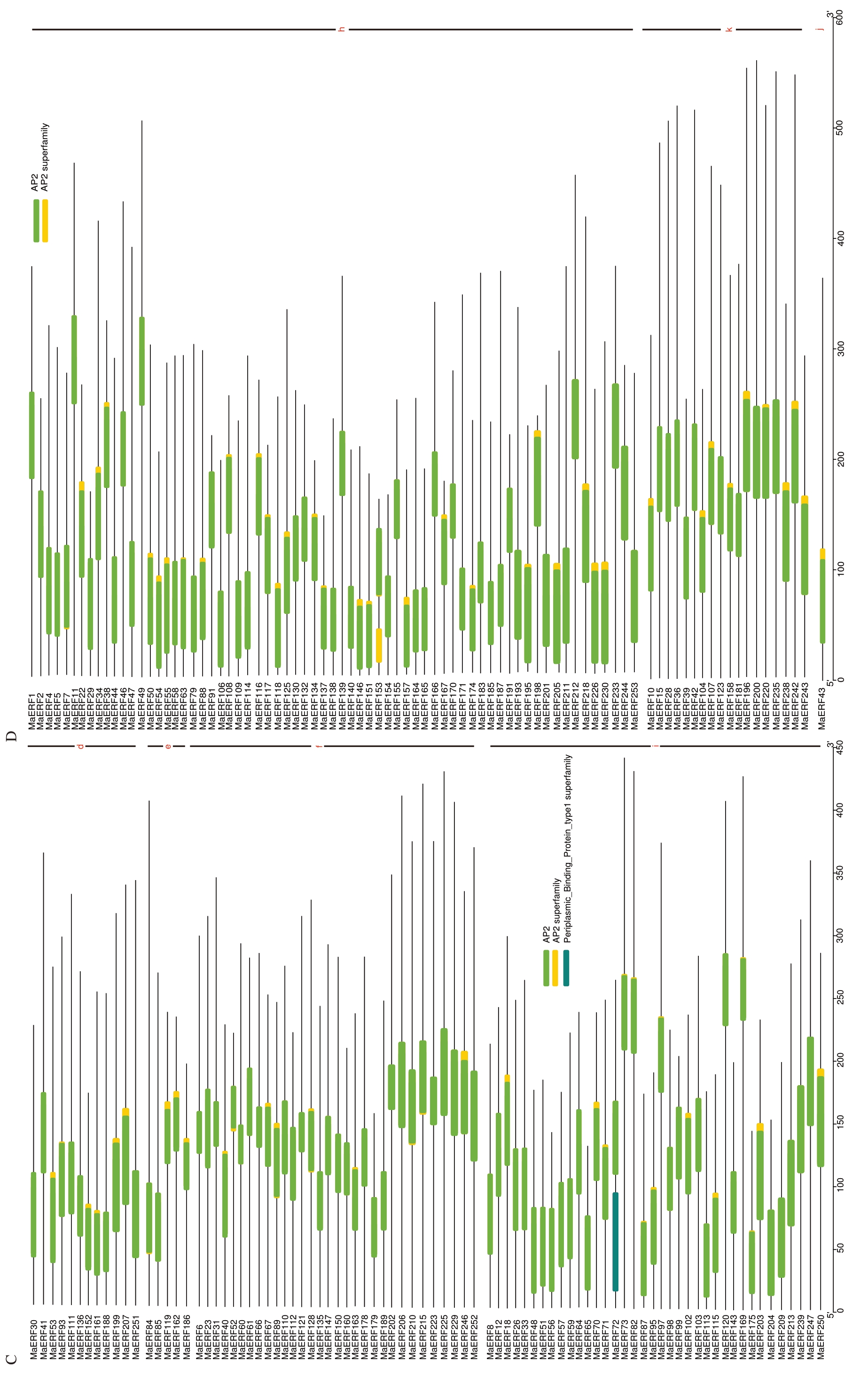
图4 (续) Fig.4 (Continued)
2.5 香蕉AP2/ERFs超家族成员在香蕉果实采后成熟过程中的差异表达分析
为了解AP2/ERFs 超家族成员在不同香蕉果实采后成熟过程中的作用,采用转录组学研究他们在巴西蕉和粉蕉果实采后成熟过程中的差异表达特性(图5-A)。结果表明,AP2/ERFs 超家族的317 个成员中,在巴西蕉果实采后成熟过程中差异表达的有77个,占24.3%,其中FPKM 值大于10的有45个,大于50 的有14 个,分别为MaERF15、36、42、44、103、115、132、156、180、181、222、242,AP2- 28 和MaRAV2;FPKM 值大于100 的有4 个,分别是MaE‐RF15,36,42 和AP2-28。在粉蕉果实采后成熟过程中差异表达的有74个,占23.3%,其中FPKM 值大于10 的有32 个,大于50 的有4 个,分别为MaERF15、42,AP2-28 和MaRAV2;FPKM 值大于100 的有2 个,分别是MaERF42 和AP2-28。同时在巴西蕉和粉蕉果实成熟过程中差异表达的有57 个。只在巴西蕉果实中特异表达的有20 个,只在粉蕉中特异表达的有17 个。在上述差异表达的基因中,与巴西蕉果实成熟过程密切相关的有34 个,分别是MaERF5、15、22、32、42、49、63、72、77、103、109、111、131、139、140、141、142、143、165、167、174、179、180、185、193、207、213、222、228、234、242、250、MaRAV2 和MaRAV4,他们在巴西蕉果实采后乙烯生物合成启动期(8 DPH)表达量迅速上升,为采收时(0 DPH)的2 倍以上,推测他们在巴西蕉果实采后成熟过程中具有重要调控作用。在上述差异表达的基因中,与粉蕉果实成熟过程密切相关的有29 个,分别是MaERF11、32、36、45、49、50、70、72、73、97、103、111、120、122、140、142、143、164、165、174、193、204、240、242、247、250、252、AP2-44 和MaRAV4,他们在粉蕉果实采后乙烯生物合成启动期(3 DPH)表达量迅速上升,为采收时(0 DPH)的2 倍以上,推测他们在粉蕉果实采后成熟过程中具有重要调控作用。
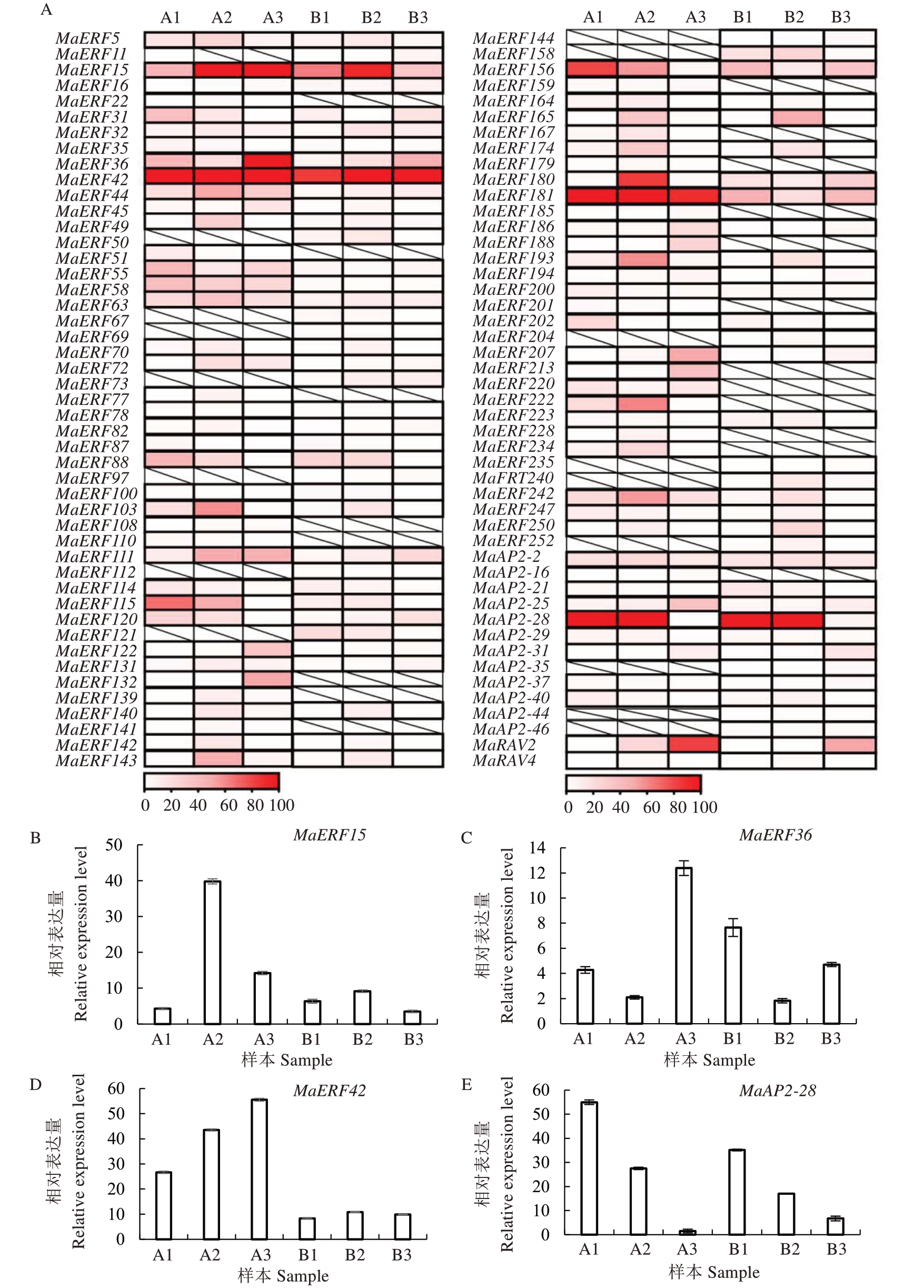
图5 巴西蕉和粉蕉果实采后成熟过程中MaAP2/ERFs 的差异表达特性
Fig.5 Expression patterns of MaAP2/ERFs at different ripening stages of BX and FJ bananas
A 基于MaAP2/ERFs 的FPKM 值绘制热图,颜色深浅代表基因表达水平高低,红色越深代表基因表达水平越高;B、C、D、E 分别为MaE‐RF15,MaERF36,MaERF42 和MaAP2-28 的qRT-PCR 验证。A1、A2、A3 分别为巴西蕉果实采后0 DPH、8 DPH、14 DPH;B1、B2、B3 分别为粉蕉果实采后0 DPH、3 DPH、6 DPH。
A The heat map was created based on the FPKM value of MaAP2/ERFs.The darker the red color,the higher the gene expression level.B,C,D,E represent qRT-PCR identification of MaERF15,MaERF36,MaERF42 and MaAP2-28,respectively.A1,A2 and A3 represent postharvest BX fruits at 0 DPH,8 DPH,14 DPH,respectively;B1,B2 and B3 represent postharvest FJ fruits at 0 DPH,3 DPH,6 DPH,respectively.
采用qRT-PCR 技术对MaERF15、36、42 和AP2-28 在巴西蕉和粉蕉果实采后成熟过程中的差异表达特性进行验证,结果表明,这4 个基因在果实采后成熟过程中的表达趋势一致,且MaAP2-28 的表达与果实采后成熟呈负相关,MaERF15、36 和42 等3个基因在乙烯生物合成启动期和高峰期的表达水平,巴西蕉高于粉蕉(图5-B~E)。这些结果与转录组的结果相符,表明转录组的结果是较为准确的。
3 讨 论
3.1 AP2/ERFs 是植物中超大基因家族,不同植物中的成员数量各不相同
通过全基因组分析,发现香蕉中有317 个AP2/ERFs 成员,比Jourda 等[19]报道的香蕉A 基因组中AP2/ERFs 多195 个,比侯晓婉等[20]报道的巴西蕉中AP2/ERFs多200个。更丰富的MaAP2/ERFs超家族成员的获得离不开全基因组测序及分析技术的高质量发展,笔者在本研究中分离的MaAP2/ERFs 是建立在第二版分析结果的基础上的,与第一版相比质量更高。在已报道的植物中,其数目仅次于油菜[11]和烟草[12],排第三位。香蕉AP2/ERFs 分为AP2、RAV、ERF三个亚家族,这与大多数植物AP2/ERFs家族的分类是一致的,然而香蕉中并未见到Soloist 这一个亚类,与悦曼芳等[9]的报道不一致,这可能是因为香蕉AP2/ERFs 家族基因的结构一致性较高。ERF 亚家族又细分为a、b、c、d、e、f、h、i、j 和k 共10 个亚类,这是与番茄中的ERF 亚家族构建系统进化树得到的,选择番茄AP2/ERFs全基因家族进行比较的原因是香蕉与番茄同属典型的呼吸跃变型果实,乙烯在果实成熟过程中具有非常重要的作用,而AP2/ERFs又是乙烯信号途径下游重要的乙烯响应因子。
3.2 MaAP2/ERFs 在染色体上的不均匀分布及基因结构的差异性
317 个MaAP2/ERFs 超家族成员不均匀地分布在11 条染色体和random 染色体上,特别是253 个MaERFs,从其在染色体上的分布规律来看,除了1号和random 染色体外,其余的10 条染色体上都存在一个或多个由于串联重复事件引起的基因簇,说明香蕉在漫长的进化过程中,MaERFs 家族在数量上显著扩张,这与Wang等[18]的研究结果类似。这些基因簇的出现为香蕉更好地适应多变的环境奠定了基础。
3.3 MaAP2/ERFs 在不同品种果实采后成熟过程中的差异表达
巴西蕉(基因型AAA)和粉蕉(基因型ABB)是目前生产上两大类主栽品种,均是由两个野生的二倍体祖先种通过种内或种间杂交后,经过漫长的进化和选择而形成的[24-25]。从转录组分析MaAP2/ERFs 超家族成员在巴西蕉和粉蕉果实采后成熟过程的差异表达特性来看,有24.3%和23.3%的AP2/ERFs 超家族成员分别在巴西蕉和粉蕉果实采后成熟过程中差异表达。有趣的是,他们中有57个同时在巴西蕉和粉蕉中差异表达,有20 个只在巴西蕉中差异表达,有17 个只在粉蕉中差异表达,表明他们在巴西蕉和粉蕉果实采后成熟过程中虽然都具有重要作用,但扮演的角色随着品种基因型不同而有差异。特别是与巴西蕉和粉蕉果实成熟过程密切相关的34 个和29 个MaAP2/ERFs 以及在巴西蕉和粉蕉果实成熟过程中超高水平表达的MaERF15、36、42和AP2-28 等基因,利用qRT-PCR 对这4 个基因的验证结果也证明了这一点,对其功能及作用机制的深入研究,将为香蕉果实成熟品质的形成调控提供重要的基因资源。
4 结 论
从香蕉A 基因组中共鉴定到317 个AP2/ERFs家族成员,归为AP2(49 个)、ERF(253 个)和RAV(15 个)三个亚家族。根据保守结构域和基因结构特征,ERF 又分为a、b、c、d、e、f、h、i、j 和k 共10 个亚类。转录组分析结果表明,在巴西蕉果实采后成熟过程中高水平表达的有MaERF15、36、42 和AP2-28。在粉蕉果实采后成熟过程中高水平表达的有MaERF42 和AP2-28。同时在巴西蕉和粉蕉果实成熟过程中高水平表达的基因有MaERF15、42 和AP2-28,只在巴西蕉果实中特异表达的有20 个,只在粉蕉中特异表达的有17个。
[1] LIU M C,GOMES B L,MILA I,PURGATTO E,PERES L E P,FRASSE P,MAZA E,ZOUINE M,ROUSTAN J P,BOUZAYEN M,PIRRELLO J.Comprehensive profiling of ethylene response factor expression identifies ripening- associated ERF genes and their link to key regulators of fruit ripening in tomato[J].Plant Physiology,2016,170(3):1732-1744.
[2] GU C,GUO Z H,HAO P P,WANG G M,JIN Z M,ZHANG S L.Multiple regulatory roles of AP2/ERF transcription factor in angiosperm[J].Botanical Studies,2017,58(1):6.
[3] CHEN T,QIN G Z,TIAN S P.Regulatory network of fruit ripening:Current understanding and future challenges[J].The New Phytologist,2020,228(4):1219-1226.
[4] ZHOU H,ZHAO L,YANG Q R,AMAR M H,OGUTU C,PENG Q,LIAO L,ZHANG J Y,HAN Y P.Identification of EIL and ERF genes related to fruit ripening in peach[J].International Journal of Molecular Sciences,2020,21(8):2846.
[5] DANG Q Y,SHA H Y,NIE J Y,WANG Y Z,YUAN Y B,JIA D J.An apple (Malus domestica) AP2/ERF transcription factor modulates carotenoid accumulation[J].Horticulture Research,2021,8:223.
[6] ZHAI Y L,FAN Z Y,CUI Y Y,GU X J,CHEN S W,MA H Q.APETALA2/ethylene responsive factor in fruit ripening:Roles,interactions and expression regulation[J].Frontiers in Plant Science,2022,13:979348.
[7] XIAO Y Y,CHEN J Y,KUANG J F,SHAN W,XIE H,JIANG Y M,LU W J.Banana ethylene response factors are involved in fruit ripening through their interactions with ethylene biosynthesis genes[J].Journal of Experimental Botany,2013,64(8):2499-2510.
[8] FENG B H,HAN Y C,XIAO Y Y,KUANG J F,FAN Z Q,CHEN J Y,LU W J.The banana fruit Dof transcription factor MaDof23 acts as a repressor and interacts with MaERF9 in regulating ripening-related genes[J].Journal of Experimental Botany,2016,67(8):2263-2275.
[9] 悦曼芳,张春,吴忠义.植物转录因子AP2/ERF 家族蛋白结构和功能的研究进展[J].生物技术通报,2022,38(12):11-26.YUE Manfang,ZHANG Chun,WU Zhongyi.Research progress in the structural and functional analysis of plant transcription factor AP2/ERF protein family[J].Biotechnology Bulletin,2022,38(12):11-26.
[10] JOFUKU K D,DEN BOER B G,VAN MONTAGU M,OKAMURO J K.Control of Arabidopsis flower and seed development by the homeotic gene APETALA2[J].The Plant Cell,1994,6(9):1211-1225.
[11] GHORBANI R,ZAKIPOUR Z,ALEMZADEH A,RAZI H.Genome-wide analysis of AP2/ERF transcription factors family in Brassica napus[J].Physiology and Molecular Biology of Plants,2020,26(7):1463-1476.
[12] GAO Y,HAN D,JIA W,MA X H,YANG Y X,XU Z C.Molecular characterization and systematic analysis of NtAP2/ERF in tobacco and functional determination of NtRAV-4 under drought stress[J].Plant Physiology and Biochemistry,2020,156:420-435.
[13] ZHANG J,LIAO J Y,LING Q Q,XI Y,QIAN Y X.Genomewide identification and expression profiling analysis of maize AP2/ERF superfamily genes reveal essential roles in abiotic stress tolerance[J].BMC Genomics,2022,23(1):125.
[14] RASHID M,HE G Y,YANG G X,HUSSAIN J,YAN X.AP2/ERF transcription factor in rice:Genome-wide canvas and syntenic relationships between monocots and eudicots[J].Evolutionary Bioinformatics Online,2012,8:321-355.
[15] NAKANO T,SUZUKI K,FUJIMURA T,SHINSHI H.Genome-wide analysis of the ERF gene family in Arabidopsis and rice[J].Plant Physiology,2006,140(2):411-432.
[16] YANG H H,SUN Y G,WANG H X,ZHAO T T,XU X Y,JIANG J B,LI J F.Genome-wide identification and functional analysis of the ERF2 gene family in response to disease resistance against Stemphylium lycopersici in tomato[J].BMC Plant Biology,2021,21(1):72.
[17] ZHANG H N,PAN X L,LIU S H,LIN W Q,LI Y H,ZHANG X M.Genome-wide analysis of AP2/ERF transcription factors in pineapple reveals functional divergence during flowering induction mediated by ethylene and floral organ development[J].Genomics,2021,113(2):474-489.
[18] WANG Z,MIAO H X,LIU J H,XU B Y,YAO X M,XU C Y,ZHAO S C,FANG X D,JIA C H,WANG J Y,ZHANG J B,LI J Y,XU Y,WANG J S,MA W H,WU Z Y,YU L L,YANG Y L,LIU C,GUO Y,SUN S L,BAURENS F C,MARTIN G,SALMON F,GARSMEUR O,YAHIAOUI N,HERVOUET C,ROUARD M,LABOUREAU N,HABAS R,RICCI S,PENG M,GUO A P,XIE J H,LI Y,DING Z H,YAN Y,TIE W W,D’HONT A,HU W,JIN Z Q. Musa balbisiana genome reveals subgenome evolution and functional divergence[J].Nature Plants,2019,5:810-821.
[19] JOURDA C,CARDI C,MBÉGUIÉ-A-MBÉGUIÉ D,BOCS S,GARSMEUR O,D’HONT A,YAHIAOUI N.Expansion of banana (Musa acuminata) gene families involved in ethylene biosynthesis and signalling after lineage-specific whole-genome duplications[J].The New Phytologist,2014,202(3):986-1000.
[20] 侯晓婉,胡伟,徐碧玉,金志强,张鲁斌,鹿志伟.巴西蕉AP2/ERF 超家族全基因组分析[J].热带作物学报,2017,38(2):302-312.HOU Xiaowan,HU Wei,XU Biyu,JIN Zhiqiang,ZHANG Lubin,LU Zhiwei.Genome-wide analysis of AP2/ERF super family in Musaceae,Musa[J].Chinese Journal of Tropical Crops,2017,38(2):302-312.
[21] D’HONT A,DENOEUD F,AURY J M,BAURENS F C,CARREEL F,GARSMEUR O,NOEL B,BOCS S,DROC G,ROUARD M,DA SILVA C,JABBARI K,CARDI C,POULAIN J,SOUQUET M,LABADIE K,JOURDA C,LENGELLÉ J,RODIER-GOUD M,ALBERTI A,BERNARD M,CORREA M,AYYAMPALAYAM S,MCKAIN M R,LEEBENS-MACK J,BURGESS D,FREELING M,MBÉGUIÉ-AMBÉGUIÉ D,CHABANNES M,WICKER T,PANAUD O,BARBOSA J,HRIBOVA E,HESLOP-HARRISON P,HABAS R,RIVALLAN R,FRANCOIS P,POIRON C,KILIAN A,BURTHIA D,JENNY C,BAKRY F,BROWN S,GUIGNON V,KEMA G,DITA M,WAALWIJK C,JOSEPH S,DIEVART A,JAILLON O,LECLERCQ J,ARGOUT X,LYONS E,ALMEIDA A,JERIDI M,DOLEZEL J,ROUX N,RISTERUCCI A M,WEISSENBACH J,RUIZ M,GLASZMANN J C,QUÉTIER F,YAHIAOUI N,WINCKER P.The banana (Musa acuminata) genome and the evolution of monocotyledonous plants[J].Nature,2012,488:213-217.
[22] TAMURA K,PETERSON D,PETERSON N,STECHER G,NEI M,KUMAR S.MEGA5:Molecular evolutionary genetics analysis using maximum likelihood,evolutionary distance,and maximum parsimony methods[J].Molecular Biology and Evolution,2011,28(10):2731-2739.
[23] TRAPNELL C,ROBERTS A,GOFF L,PERTEA G,KIM D,KELLEY D R,PIMENTEL H,SALZBERG S L,RINN J L,PACHTER L.Differential gene and transcript expression analysis of RNA-seq experiments with TopHat and Cufflinks[J].Nature Protocols,2012,7:562-578.
[24] HESLOP-HARRISON J S,SCHWARZACHER T.Domestication,genomics and the future for banana[J].Annals of Botany,2007,100(5):1073-1084.
[25] WU W,YANG Y L,HE W M,ROUARD M,LI W M,XU M,ROUX N,GE X J.Whole genome sequencing of a banana wild relative Musa itinerans provides insights into lineage-specific diversification of the Musa genus[J].Scientific Reports,2016,6:31586.