马家柚[Citrus maxima(L.)Osbeck‘Majiayou’]为芸香科(Rutaceae)柑橘亚科(Aurantioideae)柑橘属(Citrus)亚热带常绿果树,为江西省地方特色红心柚品种,原产于江西省上饶市广丰区大南镇古村马家村[1],是由当地农业部门通过对诸多柑橘资源进行多年筛选和普查而获得的优良柚种质,2009 年由江西省农作物品种审定委员会审定并命名,2010 年被农业部核准为国家地理标志农产品[2]。马家柚药食兼用。食用,马家柚果大皮黄,肉粉多汁,甜酸清爽,口味独特,营养丰富[3];药用,马家柚的柚皮、柚肉、柚花和柚核含有多种活性成分如β-柠檬烯、黄酮、柠檬苦素等,具有理气和胃、消食化痰、镇痛消炎、抗癌抑瘤、抗病毒、抗氧化、解酒毒等功效[4]。目前对马家柚的研究主要集中于套袋处理[3,5-6]、营养成分[7]、光合特性[4]、花粉直感[2]、授粉昆虫[1]、发酵工艺[8]、解剖观察[9]、土壤养分[10]等方面,但马家柚叶绿体基因组特征及其密码子偏好性分析的研究尚未见报道,马家柚的进化起源及其系统发育的亲缘关系尚未得到明确的鉴定。目前,上饶市各县市均在大力发展马家柚产业,如何保证广丰马家柚的道地性,急需对广丰马家柚正本溯源。
通过对同属植物叶绿体基因组序列的比较和分析,构建同属植物系统发育树,可综合评估该品种的系统发育位置和演化关系[11]。叶绿体是绿色植物细胞内可将光能转化为化学能的半自主性细胞器[12],其基因组为四分体双链环状结构,一般由1个大单拷贝(Large single copy,LSC)区、1个小单拷贝(Small single copy,SSC)区和2 个反向重复(Inverted repeats,IRs,包括IRa 和IRb)区组成[13]。叶绿体编码区的核酸替代速率相对较低的特点为植物深层次系统进化研究提供了必要条件。越来越多的叶绿体编码基因被广泛应用于不同科、目乃至整个被子植物的系统学研究,对植物间的系统发育研究和进化关系分析做出了重大贡献[14]。在植物系统学研究中较为常用的叶绿体编码基因有rbc、matK、atpB 和ndhF 等[15]。叶绿体基因组序列揭示了植物物种内部和物种之间的序列差异和结构变异较大,这些信息对了解重要作物的适应能力,促进密切相关物种的育种以及识别和保护有价值的性状具有重大的意义[16]。通过完整叶绿体基因组的多态性和高通量基因组比较,对复杂的遗传关系进行探究,目前已从属传递到科,并达到目水平[17]。叶绿体的完整序列也为分子育种和DNA 条形码标记的开发提供了有用的信息,并已在植物种质资源的保护方面得到了有效的应用。叶绿体基因组包含的简单重复序列(Simple sequence repeat,SSR)和长重复序列(Long repeat sequence,Longrepeat)均可以作为有效的DNA 分子标记用于物种遗传多样性和遗传稳定性的检测,有利于植物的分子辅助育种和种质资源保存[18-20]。徐世荣等[21]对六月早蜜柚(C.maxima‘Liuyuezao’)的叶绿体基因组及其特征进行了分析,发现六月早蜜柚与甜橙(C.sinensis)、柠檬(C.limon)和C.platymamma的亲缘关系较近;Xu等[22]对福建琯溪蜜柚[C.maxima(Burm.)Merr.‘Guanximiyou’]的叶绿体基因组序列进行了分析,发现福建琯溪蜜柚与柚(C.maxima)、甜橙(C.sinensis)、C.platy‐moma 和柠檬(C.limon)亲缘关系较近;Zhang 等[23]对云南红河柑橘(C.hongheensis)的叶绿体基因组特征进行了分析,发现云南红河柑橘与C.maxima 亲缘关系较近;Cai 等[24]对手指柠檬(C.australasic)栽培种的叶绿体基因组序列进行了分析,发现手指柠檬栽培种与C.medica 亲缘关系较近;Su 等[25]对手指柠檬(C.australasic)栽培种的叶绿体基因组序列进行了分析,确定了阿曼酸橙(Omani lime,C.aurantiifolia)3 个基因间区域和94 个简单序列重复(SSR),是具有种间关系分辨率的潜在信息标记,可以利用这些标记更好地了解栽培柑橘的起源。Ishikawa等[26]利用叶绿体全基因组序列及其生物多样性对扁实柠檬(Shiikuwasha,C.depressa Hayata)多个谱系进行评价,发现与野生种群相比,栽培种群已失去基因的多样性。Bausher等[27]通过组织与系统发育以及与其他被子植物的关系对C.sinensis(L.)Osbeck‘Ridge Pineapple’的叶绿体基因组序列进行了分析,发现反向重复区的扩展包括rps19 和部分rpl22 以及rpl22 的两个截短拷贝的存在是不寻常的,完整的柑橘叶绿体基因组序列的可用性可为叶绿体基因工程提供关于基因间隔区和内源性调控序列有价值的信息。目前,有关广丰马家柚的叶绿体基因组组装、注释、基因组特征及系统发育方面的研究尚未见报道。笔者在本研究中对广丰马家柚叶绿体基因组序列组装注释,明确其叶绿体基因组特征、密码子偏好性及系统进化等相关问题,为广丰马家柚种质资源的鉴定、开发和利用提供参考。研究首次对广丰马家柚的叶绿体基因组进行测序、组装和注释,进一步分析其叶绿体基因组特征和密码子偏好性等,筛选有效的最优密码子,并将其与已公布叶绿体基因组的柑橘属同属种构建系统发育树,阐明广丰马家柚与其他柑橘属同属种的进化关系及其在系统发育中的地位,可为柑橘属植物的遗传进化研究提供思路,也为种质资源开发利用和叶绿体基因工程研究提供参考。
1 材料和方法
1.1 材料
广丰马家柚(代号:MJY)盆栽苗由上饶师范学院马家柚产业研究院提供。
1.2 方法
1.2.1 DNA提取和测序 马家柚叶片的总DNA提取采用改良的CTAB 法[28];叶片DNA 纯度的检测采用NanoDrop 2000 分光光度计(美国,Thermo Scientific公司)检测;马家柚叶片DNA 文库采用美国Invitrogen Qubit® 2.0 荧光定量仪初步定量;马家柚叶片DNA 文库插入片段采用Agilent 2100 生物分析仪系统检测;马家柚叶片DNA文库有效浓度采用实时荧光定量PCR(Real-time quantitative PCR,RT-qPCR)准确定量;马家柚叶片DNA文库采用广州佰数生物科技有限公司(Bio&Data Biotechnologies)DNBSEQ-T7测序仪平台(华大智造)测序。
1.2.2 叶绿体全基因组的组装与注释 采用fastp V0.23.2 软件(默认参数)[29]过滤马家柚叶片DNA 文库Raw Data 原始数据,去除马家柚叶片DNA 文库低质量Reads,获得马家柚叶片DNA 文库高质量Clean Data。马家柚叶绿体基因组的组装采用Noveplastys[30]和CAP3[31]软件,Noveplastys 和CAP3软件适用于组装,其中Noveplastys 是主程序,在Noveplastys 未环化情况下,CAP3 软件参与序列环化处理;马家柚叶绿体基因组的注释采用GeSeq[32]和tRNAscan-SE[33]软件,GeSeq 和tRNAscan-SE 软件用于基因组注释,tRNA-scan-SE 用于补充GeSeq 对tRNA 注释的不足;马家柚叶绿体基因组图谱的制作采用OGDRAW[34]软件。注释完成后,将马家柚叶绿体基因组序列提交至NCBI(https://www.ncbi.nlm.nih.gov/),获得登录号(PP024602)。
1.2.3 叶绿体基因组特征分析 马家柚叶绿体基因组的大单拷贝区(LSC)、小单拷贝区(SSC)和反向重复区(IR)GC 含量的分析和统计采用CGViewServer[35]软件;马家柚叶绿体基因组的SSR分析采用MISA(MIcroSAtellite identification tool)[36]软件;马家柚叶绿体基因组的Longrepeat 分析采用REPuter[37]软件;马家柚叶绿体基因组同义密码子相对使用度(relative synonymous codon usage,RSCU)的计算和分析采用CodonW 软件[38];马家柚及其18个同属种(表1序号1~19)叶绿体基因组变异圈图的绘制和序列相似性的计算均采用Gview[39]软件;马家柚及其18 个同属种(表1 序号1~19)叶绿体基因组IR结构变异的绘图采用IRscope[40]软件;马家柚及其18 个同属种(表1 序号1~19)叶绿体基因组核苷酸多态性(Nucleotide polymorphism,Pi)的计算采用NADnaSP6.0[41]软件;马家柚及其85 个同科种(芸香科柑橘亚科、芸香亚科和飞龙掌血亚科)和3个外群种(橄榄NC048982、苦树MW801117、海人树MK830069)(表1)叶绿体基因组的序列比对和建树分别采用MAFFT 7.0[42]软件和FastTree 2.1.10[43]软件。
表1 马家柚和85 个同科种以及3 个外群种的叶绿体基因组ID
Table 1 Chloroplast genome IDs of C.maxima (L.) Osbeck ‘Majiayou’, 85 same family, and 3 outgroup species
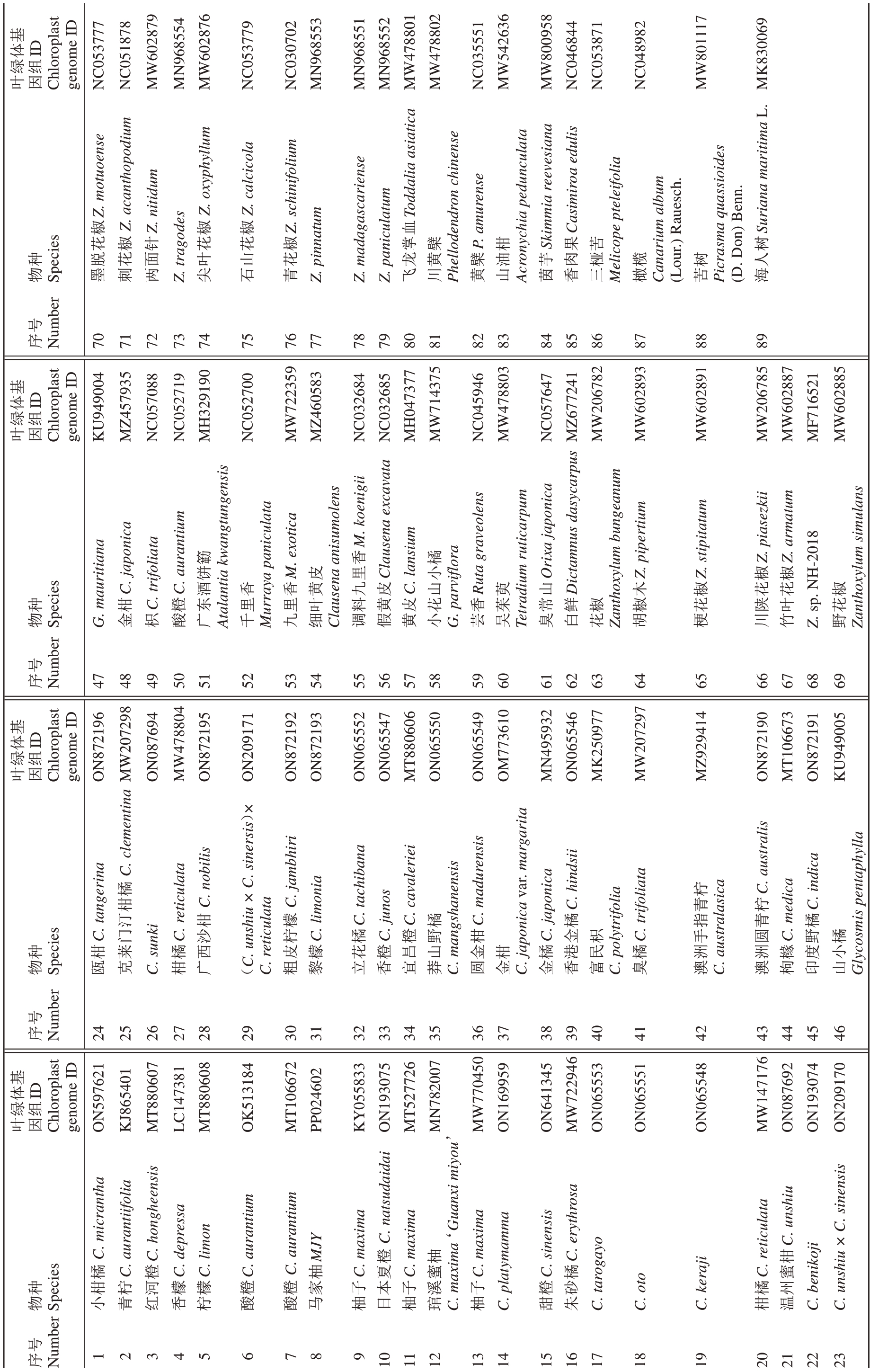
绿组ID Chloroplast genome ID 基体叶因NC053777 NC051878 MW602879 MN968554 MW602876 NC053779 NC030702 MN968553 MN968551 MN968552 MW478801 MW478802 NC035551 MW542636 MW800958 NC046844 NC053871 NC048982 MW801117 MK830069 Z.motuoense Z.oxyphyllum Z.calcicola Z.acanthopodium Toddalia asiatica种Species 椒Z.nitidum椒椒Z.schinifolium血Casimiroa edulis Suriana maritima L.针花花花椒椒掌檗P.amurense柑Skimmia reevesiana果苦树脱花Z.tragodes山面叶花龙黄檗油芋肉桠榄树人物墨两尖刺石青Z.pinnatum Z.madagascariense Z.paniculatum飞川Phellodendron chinense黄山Acronychia pedunculata茵香三Melicope pteleifolia橄Canarium album(Lour.)Rauesch.苦Picrasma quassioides(D.Don)Benn.海号Number序7071727374757677787980818283848586878889体叶因绿组ID Chloroplast genome ID KU949004基MZ457935 NC057088 NC052719 MH329190 NC052700 MW722359 MZ460583 NC032684 NC032685 MH047377 MW714375 NC045946 MW478803 NC057647 MZ677241 MW206782 MW602893 MW602891 MW206785 MW602887 MF716521 MW602885 M.koenigii簕香橘Z.piasezkii Z.armatum饼M.exotica皮里Clausena excavata小Orixa japonica Z.pipertium Z.stipitatum椒椒C.japonica C.aurantium酒香香黄九皮C.lansium山Ruta graveolens萸山Dictamnus dasycarpus木椒花花椒柑C.trifoliata橙东里里叶料黄皮花香茱常鲜椒椒花陕叶花种Species物G.mauritiana金枳酸广Atalantia kwangtungensis千Murraya paniculata九细Clausena anisumolens调假黄小G.parviflora芸吴Tetradium ruticarpum臭白花Zanthoxylum bungeanum胡梗川竹Z.sp.NH-2018野Zanthoxylum simulans号Number序4748495051525354555657585960616263646566676869绿组ID Chloroplast genome ID 基体叶因ON872196 MW207298 ON087694 MW478804 ON872195 ON209171 ON872192 ON872193 ON065552 ON065547 MT880606 ON065550 ON065549 OM773610 MN495932 ON065546 MK250977 MW207297 MZ929414 ON872190 MT106673 ON872191 KU949005 C.clementina橘C.nobilis C.jambhiri C.hindsii柠C.australis C.indica柑青柠种Species C.tangerina汀柑檬C.tachibana C.cavaleriei橘C.madurensis橘指青橘门C.reticulata沙柠C.limonia橘C.junos橙野柑C.japonica金枳C.trifoliata手圆C.medica野橘柑莱橘西皮檬花橙昌山金柑橘港民橘洲洲橼度小物瓯克C.sunki柑广C.reticulata粗黎立香宜莽C.mangshanensis圆金C.japonica var.margarita金香富C.polytrifolia臭澳C.australasica澳枸印山Glycosmis pentaphylla号Number (C.unshiu×C.sinersis)×序2425262728293031323334353637383940414243444546绿组ID Chloroplast genome ID 基体叶因ON597621 KJ865401 MT880607 LC147381 MT880608 OK513184 MT106672 PP024602 KY055833 ON193075 MT527726 MN782007 MW770450 ON169959 ON641345 MW722946 ON065553 ON065551 ON065548 MW147176 ON087692 ON193074 ON209170 C.micrantha种Species C.hongheensis C.aurantiifolia C.depressa C.limon C.aurantium C.aurantium MJY C.maxima C.natsudaidai C.maxima琯C.maxima‘Guanxi miyou’C.maxima C.platymamma C.sinensis C.erythrosa C.tarogayo C.keraji C.reticulata C.unshiu橙柚柑橘橙柚夏蜜橘蜜柑柠河檬檬橙橙家子本子溪子橙砂橘州物号Number 小青红香柠酸酸马柚日柚柚甜朱C.oto柑温C.benikoji C.unshiu×C.sinensis序1234567891011121314151617181920212223
1.2.4 叶绿体基因组密码子使用偏好性分析GC3-GC12(Neutrality-plot)、ENC-plot、PR2-plot 和最优密码子分别采用参考Liu 等[19]的方法进行分析。其中,GC3-GC12 分析用R 语言做GC3 和GC12的线性回归分析;ENC-plot 用EMBOSS(6.6.0.0)计算ENC,用R 语言绘制ENC-plot;PR2-plotPR2 通过A3、T3、G3、C3(分别表示密码子第三位碱基的A、T、G、C 含量)分别计算AT-bias[A3/(A3+T3)]与GCbias[G3/(G3+C3)],利用二者的对应关系来分析选择和突变对密码子使用模式的影响,所用数据由字写python脚本计算。
1.2.5 叶绿体基因组特异性标记筛选 通过分析,马家柚叶绿体基因组与KJ865401、KY055833、LC147381、MK250977、MN495932、MT106672、MT106673、MT880606、MT880607、MT880608、MW147176、MW207297、MW207298、MW478804、MW722946、MW770450、MZ929414、OK513184、OM773610、ON065546、ON065547、ON065548、ON065549、ON065550、ON065551、ON065552、ON065553、ON087692、ON087694、ON169959、ON193074、ON209170、ON209171、ON597621、ON641345、ON872190、ON872191、ON872192、ON872193、ON872195、ON872196存在变异的序列,通过这些变异的序列筛选出马家柚叶绿体基因组的特异性标记。
2 结果与分析
2.1 马家柚叶绿体基因组的基本结构
马家柚叶绿体基因组全长160 186 bp,包括1个LSC区(87 791 bp)、1个SSC区(18 395 bp)和2个IR区(包括IRa 和IRb,均为27 000 bp),其结构呈典型闭合环状双链结构(图1)。马家柚叶绿体基因组平均GC 含量为38.74% ,其中IR 区的GC 含量(42.95%)高于LSC区(36.8%)和SSC区(33.34%)。
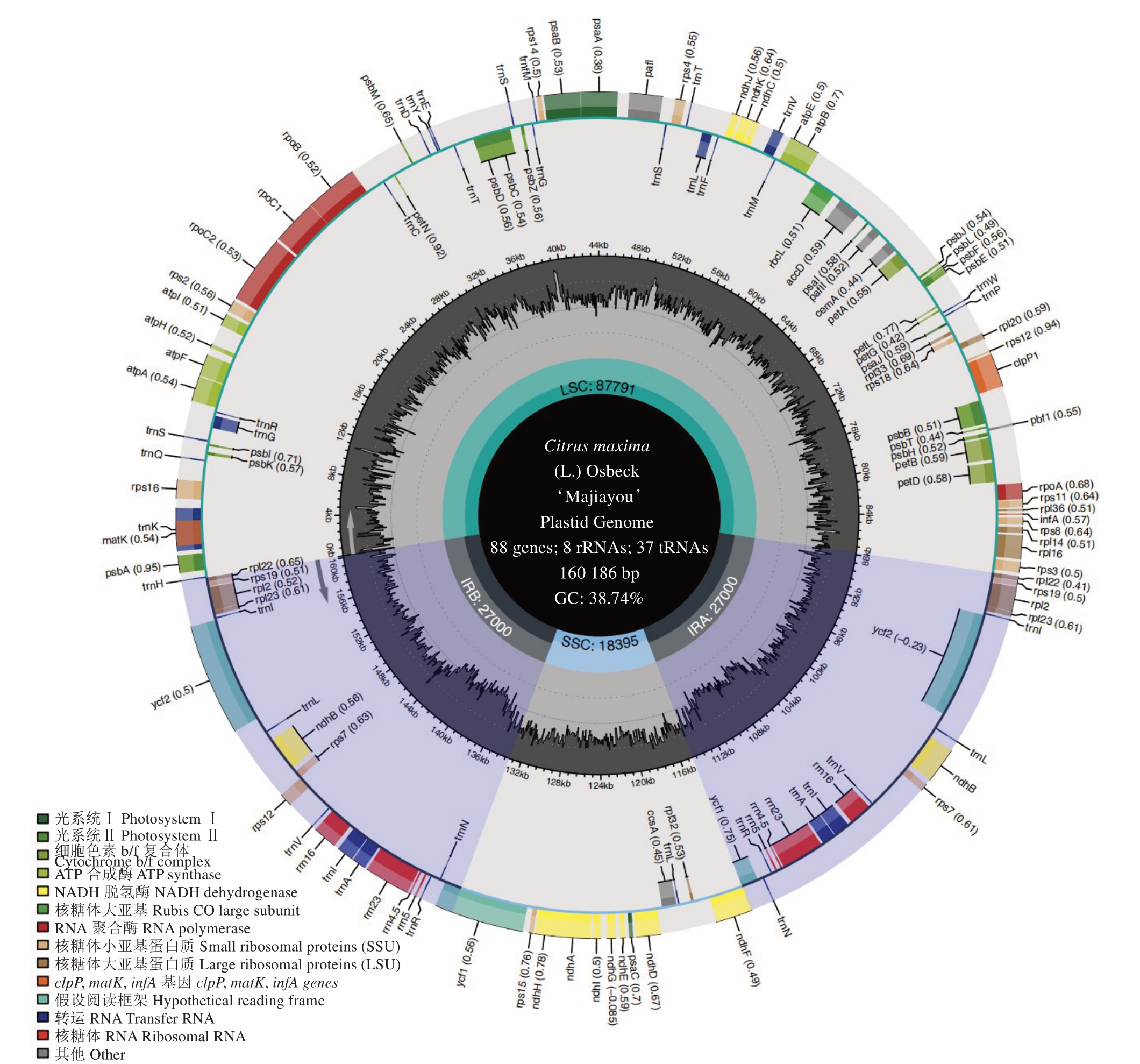
图1 马家柚叶绿体基因组图谱
Fig.1 Chloroplast genome map of C.maxima(L.)Osbeck‘Majiayou’
图形中的环为GC 含量。
The ring in the graph represents the GC content.
2.2 马家柚叶绿体基因组的基因组成
马家柚叶绿体基因组共注释到133 个功能基因(表2),包括88 个编码蛋白(Coding sequence,CDS)基因、8 个核糖体RNA(rRNA)基因和37 个转运RNA(tRNA)基因,其中假基因为0 个。按照基因功能,马家柚叶绿体基因组的基因可分为四大类:第一类是与光合作用有关的44 种基因;第二类是自我复制的77种基因;第三类是其他功能的6种基因;第四类是未知功能的6 种基因。其中,trnH-GUG、trnKUUU、trnI-GAU、trnA-UGC、trnG-UCC、trnV-UAC、trnL-UAA、rpoC1、ndhB、ndhA、rpl2、rpl16、petB、at‐pF、petD、rps16、rps12 基因具有2 个外显子,rps12、clpP1、pafI 基因具有3 个外显子(rps12 有2 个拷贝,每个拷贝具有3 个外显子,且两个拷贝共享第一个外显子,第一个外显子位于LSC 区域,另外两个外显子位于IR 区域);完全在LSC 区的基因有80 个(21 个tRNA 基因和59 个CDS 基因),完全在SSC 区的基因有11 个(1 个tRNA 基因和10 个CDS 基因),完全在IRB 和IRA 区的基因有18 个(4 个rRNA 基因、7 个tRNA 基因和7 个CDS 基因),在SSC-IRB 连接处的基因有2 个CDS 基因(ndhF 和ycf1),在LSCIRB 连接处的基因有1 个tRNA 基因(trnH-GUG),在SSC-IRA连接处的基因有1个CDS基因(ycf1)。
表2 马家柚叶绿体基因组的基因组成
Table 2 Gene composition of chloroplast genome of C.maxima(L.)Osbeck‘Majiayou’

注:*表示为2 个拷贝。
Note:*represents 2 copies.
基因功能Gene function光合作用Photosynthesis基因类型Gene type光系统ⅠPhotosystem Ⅰ光系统ⅡPhotosystem Ⅱ基因名Gene name psaA、psaB、psaC、psaI、psaJ psbA、psbB、psbC、psbD、psbE、psbF、psbH、psbI、ps‐bJ、psbK、psbL、psbM、psbT、psbZ、psbN(pbf1)ndhA、ndhB*、ndhC、ndhD、ndhE、ndhF、ndhG、ndhH、ndhI、ndhJ、ndhK petA、petB、petD、petG、petL、petN基因数量Number of genes 5 15 12 atpA、atpB、atpE、atpF、atpH、atpI 6 6自我复制Self-replication NADH 脱氢酶NADH dehydrogenase细胞色素b/f 复合体cytochrome b/f complex ATP 合成酶ATP synthase核糖体大亚基蛋白质Proteins of large ribosomal subunit核糖体小亚基蛋白质Proteins of small ribosomal subunit核糖体大亚基Large subunit of rubisco RNA 聚合酶RNA polymerase核糖体RNA Ribosomal RNAs转运RNA Transfer RNAs rpl14、rpl16、rpl2*、rpl20、rpl22*、rpl23*、rpl32、rpl33、rpl36 rps11、rps12*、rps14、rps15、rps16、rps18、rps19*、rps2、rps3、rps4、rps7*、rps8 rbcL 12 15 rpoA、rpoB、rpoC1、rpoC2 1 4 8 37其他基因Other genes rrn16*、rrn23*、rrn4.5*、rrn5*trnA-UGC*、trnC-GCA、trnD-GUC、trnE-UUC、trnFGAA、trnfM-CAU、trnG-GCC、trnG-UCC、trnH-GUG、trnI-CAU*、trnI-GAU*、trnK-UUU、trnL-CAA*、trnLUAA、trnL-UAG、trnM-CAU、trnN-GUU*、trnP-UGG、trnQ-UUG、trnR-ACG*、trnR-UCU、trnS-GCU、trnSGGA、trnS-UGA、trnT-GGU、trnT-UGU、trnV-GAC*、trnV-UAC、trnW-CCA、trnY-GUA matK clpP1 cemA accD ccsA infA未知功能基因Unknown function gene合计Total成熟酶Maturase蛋白酶Protease囊膜蛋白Envelope membrane protein乙酰辅酶A 羧化酶Acetyl-CoA carboxylase c-型细胞色素合成基因c-type cytochrome synthesis gene翻译起始因子Translation initiation factor保守假设叶绿体阅读框架Conserved hypothetical chloroplast Reading Frames ycf1*、ycf2*、ycf3(pafI)、ycf4(pafII)1 1 1 1 1 1 6 133
2.3 马家柚叶绿体基因组SSR和Longrepeat分析
马家柚叶绿体基因组共检测到79 个SSR(表3),只包括单核苷酸重复序列和三核苷酸重复序列,单核苷酸重复序列有78个(97.44%),其中77个为A和T 重复(98.72%),1 个为G 和C 重复(1.28%);三核苷酸重复序列1 个(1.27%)。马家柚18 个同属种叶绿体基因组(KJ865401、KY055833、LC147381、MN782007、MT106672、MT527726、MT880607、MT880608、MW722946、MW770450、OK513184、ON065548、ON065551、ON065553、ON169959、ON193075、ON597621、ON641345)分别具有77、76、84、79、80、79、76、79、76、76、80、76、76、76、76、79、77和76 个SSR,且均以A 和T 的单核苷酸重复序列为主,说明马家柚及其18 个同属种叶绿体基因组SSR偏好使用A 和T碱基。马家柚叶绿体基因组共检测到34 个Longrepeat(表4),包括分散重复(Dispersed repeats,D)和回文重复(palindromic repeats,P),其中分散重复D 分为正向重复(forward repeats,F)、反向重复R(reverse repeats,R)和互补重复C(complement repeats,C)3 种。马家柚叶绿体基因组具有13个分散重复D(30~50 bp)和21 个回文重复P(30~27 000 bp)。
表3 马家柚叶绿体基因组中SSR 的类型及分布
Table 3 Type and distribution of SSR in chloroplast genome of C.maxima(L.)Osbeck‘Majiayou’
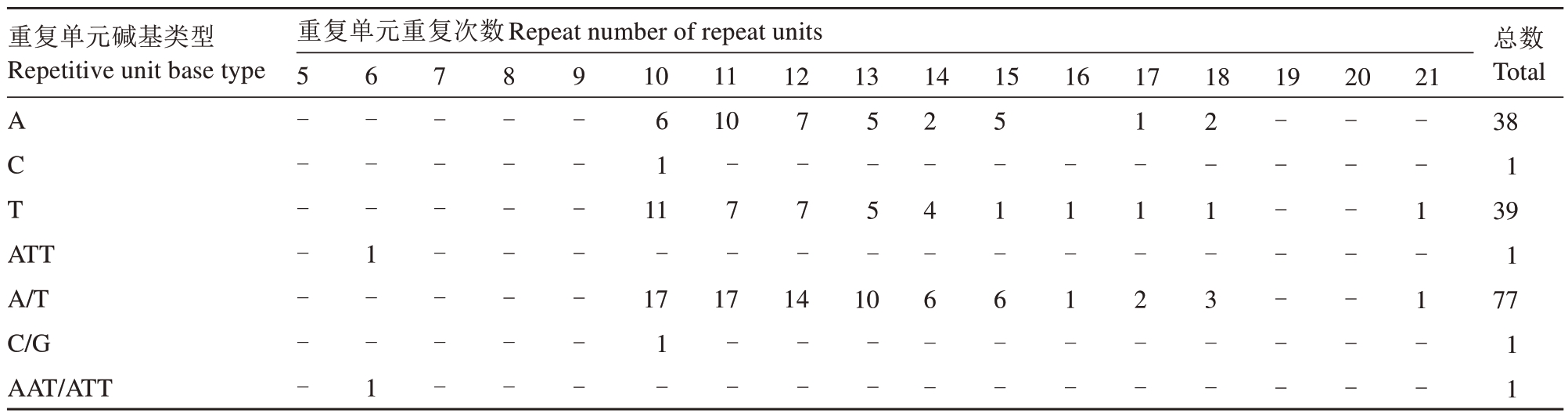
重复单元碱基类型Repetitive unit base type重复单元重复次数Repeat number of repeat units 10 12 13 14 15 16 17 18 19 20 21 A C T 6 1 1 11 10 ATT A/T C/G AAT/ATT 5-------6---1--1 7-------8-------9-------1--7-7-7-5-5-17 17 14 10总数Total 38 1 39 1 77 1-------2-4-6--5-1-6---1-1--1-1-2--2-1-3------------------1-1--1 1
表4 马家柚叶绿体基因组Longrepeat 分析
Table 4 Longrepeat analysis in chloroplast genome of C.maxima(L.)Osbeck‘Majiayou’
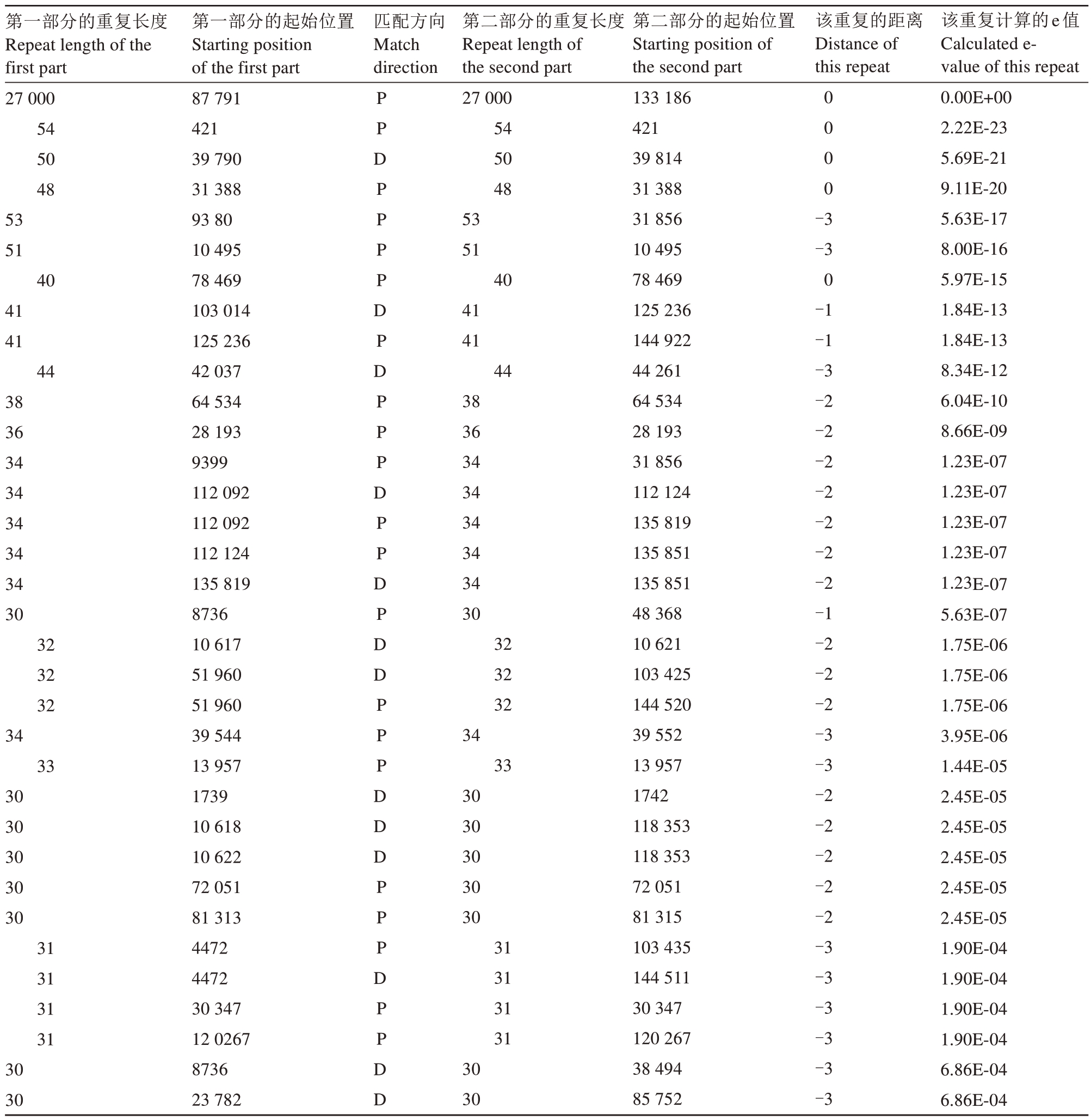
注:D.分散重复;P.回文重复。
Note:D.Dispersed repeats;P.Palindromic repeats.
第一部分的重复长度Repeat length of the first part 27 000 54 50 48 53 51 40 41 41 44 38 36 34 34 34 34 34 30 32 32 32 34 33 30 30 30 30 30 31 31 31 31 30 30该重复计算的e值Calculated evalue of this repeat 0.00E+00 2.22E-23 5.69E-21 9.11E-20 5.63E-17 8.00E-16 5.97E-15 1.84E-13 1.84E-13 8.34E-12 6.04E-10 8.66E-09 1.23E-07 1.23E-07 1.23E-07 1.23E-07 1.23E-07 5.63E-07 1.75E-06 1.75E-06 1.75E-06 3.95E-06 1.44E-05 2.45E-05 2.45E-05 2.45E-05 2.45E-05 2.45E-05 1.90E-04 1.90E-04 1.90E-04 1.90E-04 6.86E-04 6.86E-04第一部分的起始位置Starting position of the first part 87 791 421 39 790 31 388 93 80 10 495 78 469 103 014 125 236 42 037 64 534 28 193 9399 112 092 112 092 112 124 135 819 8736 10 617 51 960 51 960 39 544 13 957 1739 10 618 10 622 72 051 81 313 4472 4472 30 347 12 0267 8736 23 782匹配方向Match direction该重复的距离Distance of this repeat P P D P P P P D P D P P P D P P D P D D P P P D D D P P P D P P D D第二部分的重复长度Repeat length of the second part 27 000 54 50 48 53 51 40 41 41 44 38 36 34 34 34 34 34 30 32 32 32 34 33 30 30 30 30 30 31 31 31 31 30 30第二部分的起始位置Starting position of the second part 133 186 421 39 814 31 388 31 856 10 495 78 469 125 236 144 922 44 261 64 534 28 193 31 856 112 124 135 819 135 851 135 851 48 368 10 621 103 425 144 520 39 552 13 957 1742 118 353 118 353 72 051 81 315 103 435 144 511 30 347 120 267 38 494 85 752 0 0 0 0-3-3 0-1-1-3-2-2-2-2-2-2-2-1-2-2-2-3-3-2-2-2-2-2-3-3-3-3-3-3
2.4 叶绿体基因组特异性标记筛选
与KJ865401、 KY055833、 LC147381、MK250977、MN495932、MT106672、MT106673、MT880606、MT880607、MT880608、MW147176、MW207297、MW207298、MW478804、MW722946、MW770450、MZ929414、OK513184、OM773610、ON065546、ON065547、ON065548、ON065549、ON065550、ON065551、ON065552、ON065553、ON087692、ON087694、ON169959、ON193074、ON209170、ON209171、ON597621、ON641345、ON872190、ON872191、ON872192、ON872193、ON872195、ON872196 比较,马家柚叶绿体基因组的部分特异性标记见表5。马家柚叶绿体基因组的rps18、rpl36、psbZ、psbJ、psbF 基因有1 个变异位点,rps7、rpl23、rpl14、psbK、psbH、psaC、psaJ、atpE、ndhB、petL、pafI、petN、ndhE 基因有2 个变异位点,atpH 基因有3 个变异位点,rpl32、rpl33、psbL、ndhC、petA、ndhK、petD 基因有4 个变异位点,rps11、rpl16、clpP1 基因有5 个变异位点,rps15、rps12、ndhI、atpB基因有6 个变异位点,ndhJ、rps4、rps2、rps16、infA 基因有7 个变异位点,ndhG、cemA 基因有8 个变异位点,rps8、psbD、atpI、petB 基因有9 个变异位点,rpl20、pafII基因有10个变异位点,ndhA基因有11个变异位点,atpF、psaB、psbA 基因有12 个变异位点,rps19、rps14、rpoA、psbB 基因有14 个变异位点,rps3、psbC、atpA基因有15个变异位点,ndhH 基因有16 个变异位点,psaA 基因有19 个变异位点,accD 基因有23 个变异位点,rpoC1 基因有25 个变异位点,rbcL基因有28个变异位点,ccsA基因有29个变异位点,rpl22 基因有34 个变异位点,ndhD 基因有36 个变异位点,matK 基因有42 个变异位点,rpoB 基因有44个变异位点,ycf2基因有52 个变异位点,rpoC2基因有69 个变异位点,ndhF 基因有71 个变异位点,ycf1基因有254个变异位点。
表5 马家柚叶绿体基因组的部分特异性标记
Table 5 Part specific markers of chloroplast genome in C.maxima(L.)Osbeck‘Majiayou’
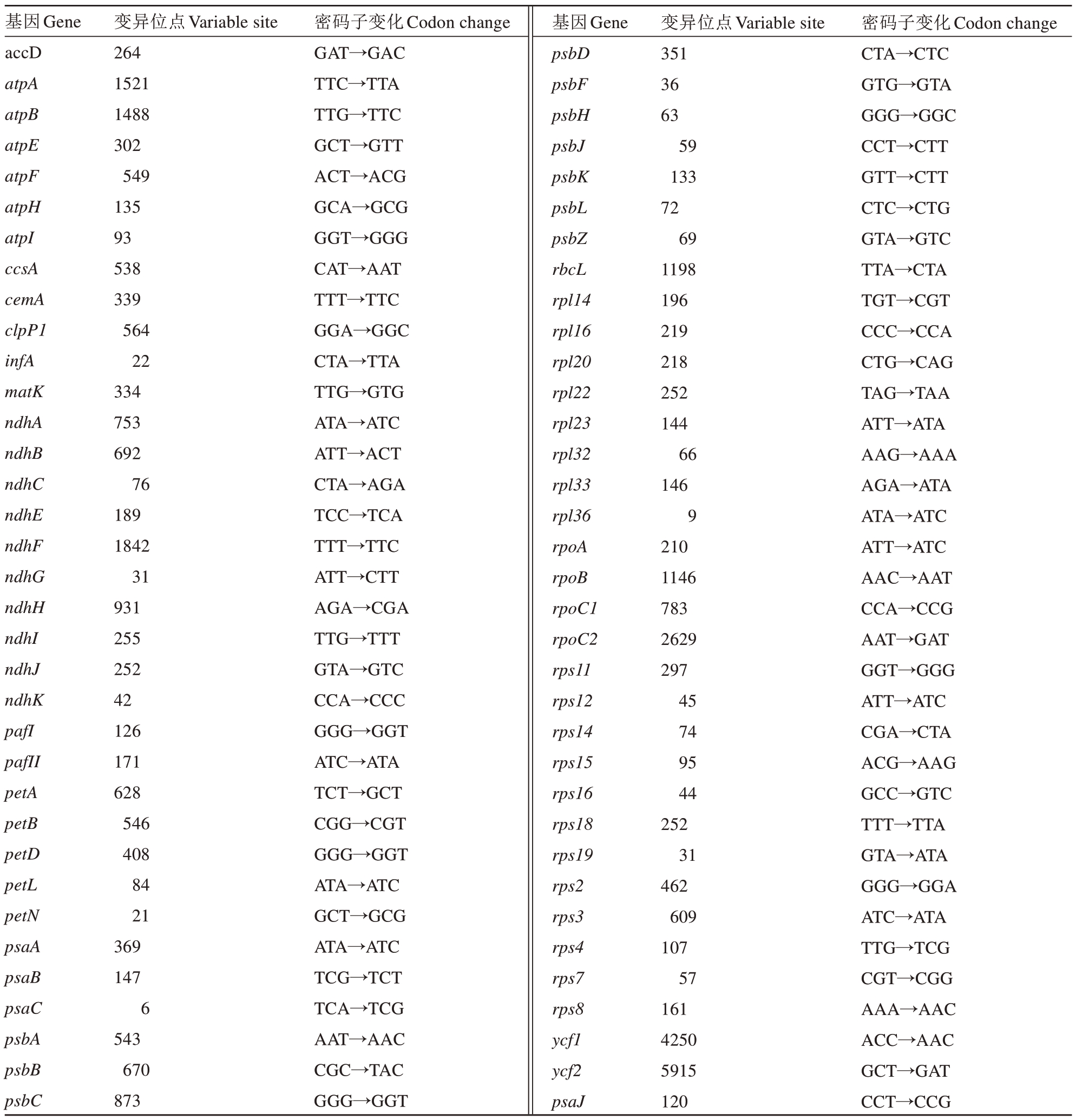
基因Gene accD atpA atpB atpE atpF atpH atpI ccsA cemA clpP1 infA matK ndhA ndhB ndhC ndhE ndhF ndhG ndhH ndhI ndhJ ndhK pafI pafII petA petB petD petL petN psaA psaB psaC psbA psbB psbC变异位点Variable site 264 1521 1488 302 549 135 93 538 339 564 22 334 753 692 76 189 1842 31 931 255 252 42 126 171 628 546 408 84 21 369 147 6 543 670 873密码子变化Codon change GAT→GAC TTC→TTA TTG→TTC GCT→GTT ACT→ACG GCA→GCG GGT→GGG CAT→AAT TTT→TTC GGA→GGC CTA→TTA TTG→GTG ATA→ATC ATT→ACT CTA→AGA TCC→TCA TTT→TTC ATT→CTT AGA→CGA TTG→TTT GTA→GTC CCA→CCC GGG→GGT ATC→ATA TCT→GCT CGG→CGT GGG→GGT ATA→ATC GCT→GCG ATA→ATC TCG→TCT TCA→TCG AAT→AAC CGC→TAC GGG→GGT基因Gene psbD psbF psbH psbJ psbK psbL psbZ rbcL rpl14 rpl16 rpl20 rpl22 rpl23 rpl32 rpl33 rpl36 rpoA rpoB rpoC1 rpoC2 rps11 rps12 rps14 rps15 rps16 rps18 rps19 rps2 rps3 rps4 rps7 rps8 ycf1 ycf2 psaJ变异位点Variable site 351 36 63 59 133 72 69 1198 196 219 218 252 144 66 146 9 210 1146 783 2629 297 45 74 95 44 252 31 462 609 107 57 161 4250 5915 120密码子变化Codon change CTA→CTC GTG→GTA GGG→GGC CCT→CTT GTT→CTT CTC→CTG GTA→GTC TTA→CTA TGT→CGT CCC→CCA CTG→CAG TAG→TAA ATT→ATA AAG→AAA AGA→ATA ATA→ATC ATT→ATC AAC→AAT CCA→CCG AAT→GAT GGT→GGG ATT→ATC CGA→CTA ACG→AAG GCC→GTC TTT→TTA GTA→ATA GGG→GGA ATC→ATA TTG→TCG CGT→CGG AAA→AAC ACC→AAC GCT→GAT CCT→CCG
2.5 叶绿体基因组比对分析
马家柚及其18 个同属种叶绿体基因组的变异圈图(图2)、mVIST结构变异图(图3)和Pi多样性指数分析图(图4)表明,马家柚及其18 个同属种的叶绿体基因组序列高度保守,LSC 和SSC 区中petN、petL、psbI、psbK、psaI、pafII、trnT-GGU、trnR-UCU、trns-GGA、trnL-UAA 等基因之间存在较大的序列差异。由图4 可知,马家柚叶绿体基因组核苷酸多样性的变化范围为0~0.006 29;马家柚叶绿体基因组非编码区的变异程度高于基因编码区,LSC 区的变异性整体较高,其次是SSC 区,IR 区变异性最低,为最为保守的区域;通过Pi(≥0.003 6)筛选出10 个高变异区域,均位于LSC 和SSC 区,LSC 区有8 个高变异区域:Inter、trnS-GCU_trnG-UCC、trnT-UGU_trnLUAA、accD-psaI、psbE-petL、rps18、rps3-rpl22、rpl22;SSC区有2个高变异区域:rpl32_trnL-UAG、ycf1-2。
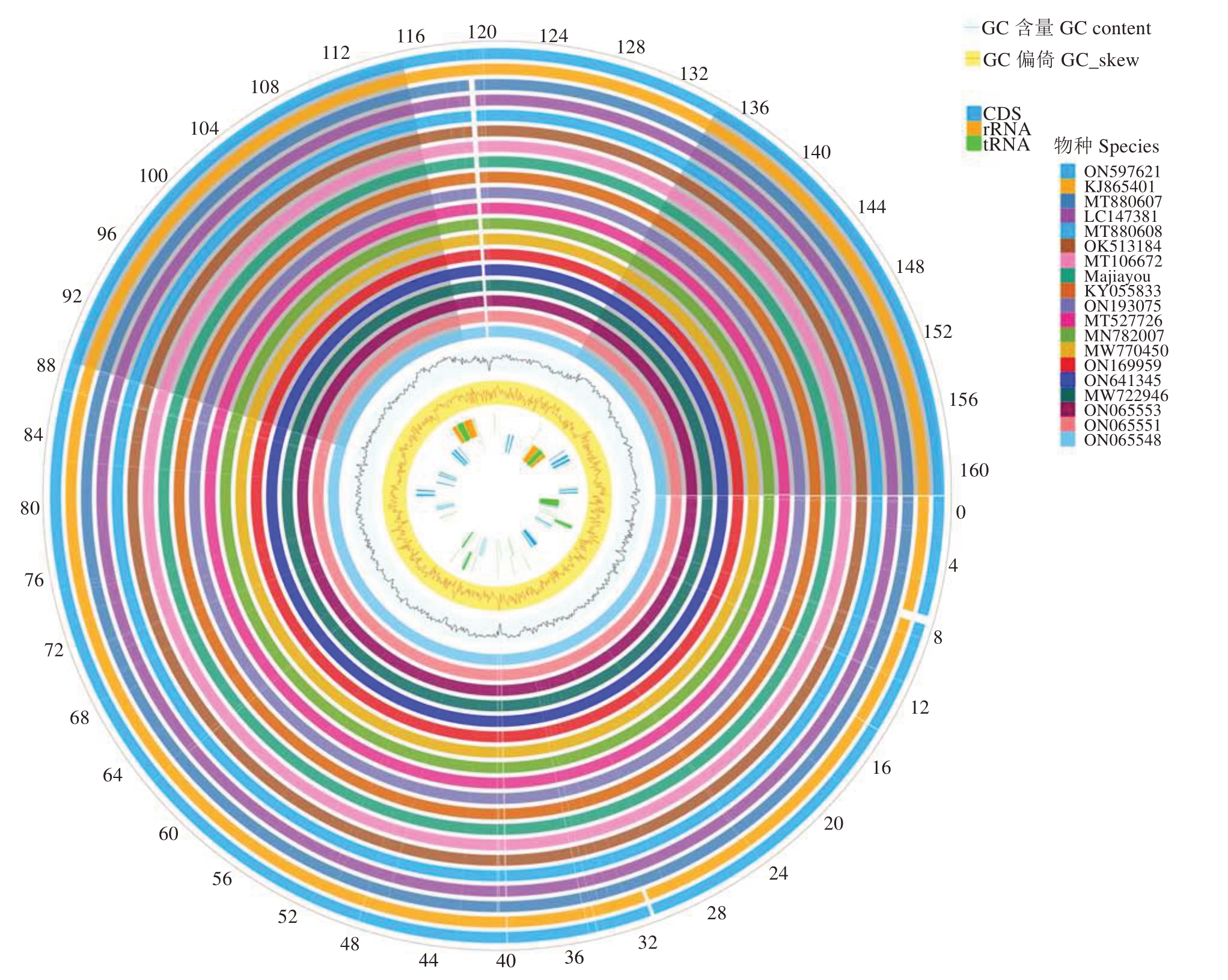
图2 马家柚及其18 个同属种叶绿体基因组Gview 变异圈
Fig.2 Gview variation circle of chloroplast genomes of C.maxima(L.)Osbeck‘Majiayou’and its 18 congeneric species
图中从内到外,第一环表示CDS(蓝色)、rRNA(橙色)和tRNA(绿色)在正链和反链上的位置;然后依次为参考基因组GC 偏斜(黄色,红色线条),GC 含量(青色,黑色线条);最外圈的数字表示基因组大小(kbp);其余的圆圈显示了质体基因组序列的BLAST 比较,不同颜色代表不同物种;叶绿体基因组中相似和不同的位置分别用连续和断开的轨迹线表示;两段阴影部分为叶绿体基因组IR 区。
In the figure,from inside out,the first ring represents the positions of CDS(blue),rRNA(orange),and tRNA(green)on the positive and negative chains;Then,the reference genome GC skewed(yellow,red lines)and GC content(cyan,black lines)were sequentially identified;The outermost circle digit represents genome size (kbp);The remaining circles display BLAST comparisons of plastid genome sequences, with different colors representing different species; Similar and different positions in the chloroplast genome are represented by continuous and broken trajectory lines, respectively;The two shaded sections represent the IR region of the chloroplast genome.
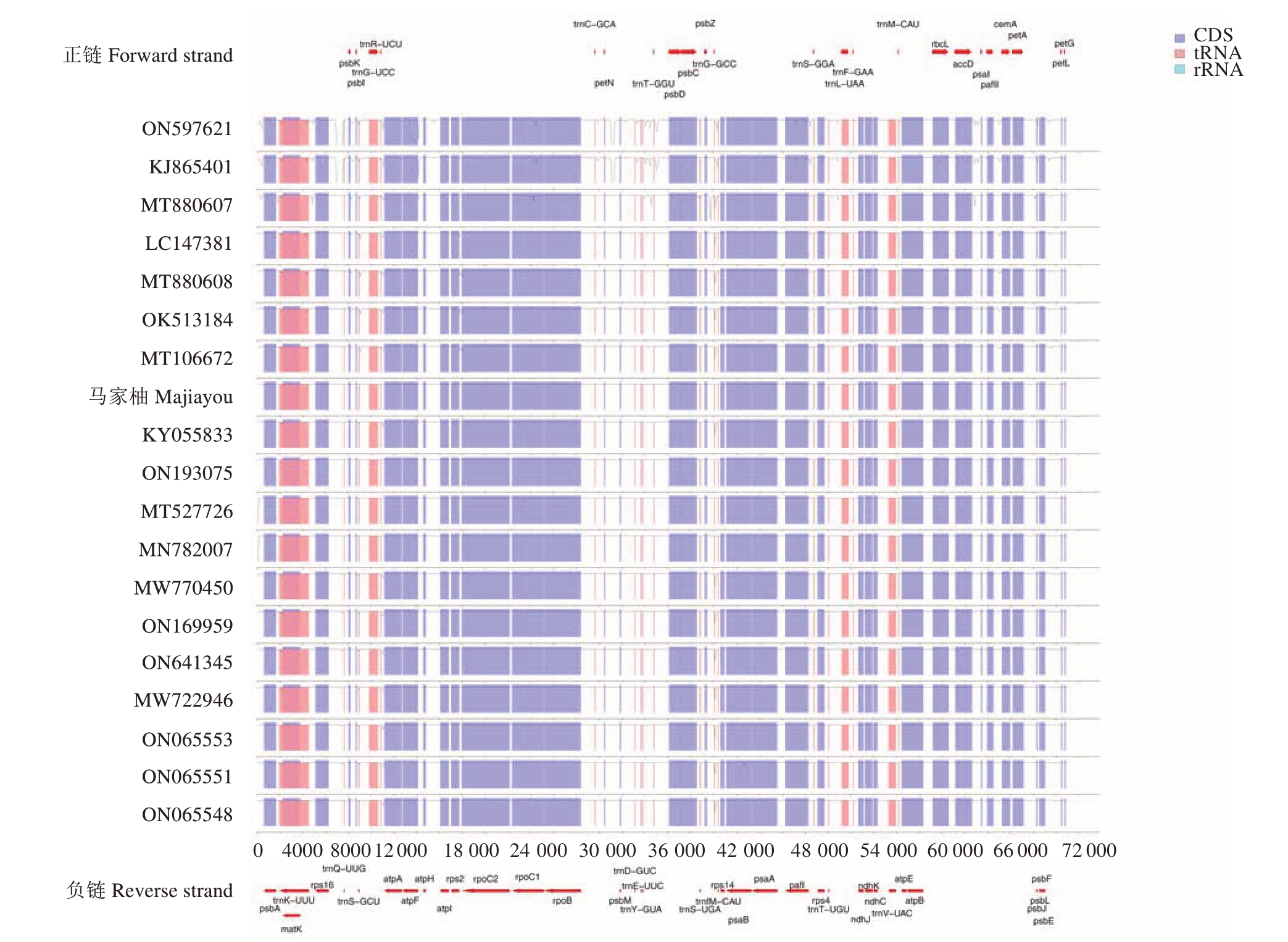
图3 马家柚及其18 个同属种叶绿体基因组mVISTA 分析
Fig.3 mVISTA analysis of chloroplast genomes of C.maxima(L.)Osbeck‘Majiayou’and its 18 congeneric species
横坐标为序列的位置,纵坐标为相似度;图中蓝色为外显子,青色为内含子,红色的为基因间隔区。
The horizontal axis represents the position of the sequence,and the vertical axis represents similarity;The blue ones represent exons,the blue ones represent introns,and the red ones represent gene spacer regions.
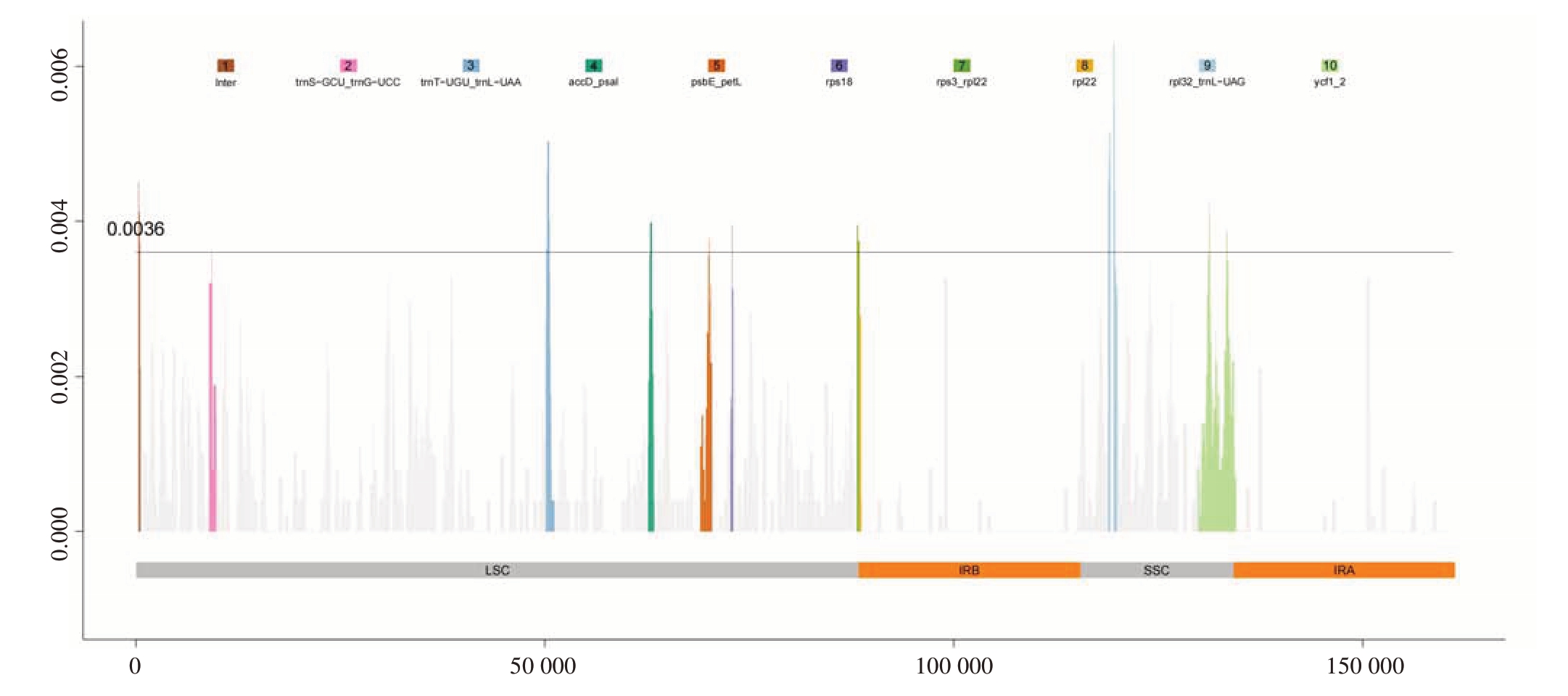
图4 马家柚及其18 个同属种叶绿体基因组Pi 多样性指数分析
Fig.4 Pi diversity index analysis of chloroplast genomes of C.maxima(L.)Osbeck‘Majiayou’and its 18 congeneric species
基于18 个近缘物种的叶绿体基因组滑动窗口分析。x 轴显示基因组区域;y 轴表示Pi 值。颜色高亮的区域为Pi 值最高的10 个区段。
Sliding window analysis of chloroplast genomes based on 18 closely related species.The X-axis displays genomic regions; The Y-axis represents the Pi value.The highlighted areas are the 10 segments with the highest Pi value.
2.6 叶绿体基因组的SC/IR/边界分析
马家柚及其18 个同属种叶绿体基因组四分体结构的SC/IR 边界收缩扩张情况(图5)表明:马家柚及其18 个同属种叶绿体基因组的4 个边界较为保守。LSC/IRb[LSC 和IRb 之间的结合点(junction sites),JLB]边界均位于rps19 基因内,rps19 基因在IRb 区内长为46 bp,在LSC 区内长为233 bp;IRb/SSC(SSC 和IRb 之间的结合点,JSB)边界均位于IRb 区的ycf1 基因和SSC 区的ndhF 基因之间,ycf1基因距离IRb/SSC 边界均为2 bp。SSC/IR(SSC 和IRa 之间的结合点,JSA)边界均位于ycf1 基因内,ycf1 基因在SSC 区长18 264~18 283 bp,ycf1 基因在IRa 区长5607~5622 bp。IRa/LSC(LSC 和IRa 之间的结合点,JLA)边界均位于IRa 区的rpl2 基因和LSC 区的trnH 基因之间,trnH 基因距离IRb/SSC 边界均为1 bp。

图5 马家柚及其18 个同属种叶绿体基因组的IR 分析
Fig.5 IR analysis of chloroplast genomes of C.maxima(L.)Osbeck‘Majiayou’and its 18 congeneric species
每一行为一个物种叶绿体基因组,每行的左侧为每个物种及其相应的叶绿体基因组序列长度。在正链和负链上转录的基因,分别在其相应的基因组上方从右到左和从左到右的方向显示。箭头表示给定基因的起始或结束位置与相应连接位点的距离。对于从一区域延伸到另一区域的基因,顶部或下方的T 条显示其扩张范围及其相应的值。在连接点附近的基因是每个位点的bp 距离的逼真缩放投影。JLB(IRb/LSC),JSB(IRb/SSC),JSA(SSC/IRa)和JLA(IRa/LSC)表示每个相应区域之间的JSs。
Each species and their corresponding chloroplast genome sequence length are depicted to the left of each track.Genes transcribed in positive and negative strands are presented above and below of their corresponding tracks with from right-to-left and left-to-right directions, respectively.The arrows are showing the bp distance of the start or end coordinate of a given gene from the corresponding junction site.For the genes extending from a region to another,the T bar on the top or below shows the extent of their parts with their corresponding values.The genes in the vicinity of the junctions are the realistically scaled projections of the bp distances for each site.JLB(IRb/LSC),JSB(IRb/SSC),JSA(SSC/IRa)and JLA(IRa/LSC)denote the JSs between each corresponding region in the genome.
2.7 马家柚叶绿体基因组密码子使用偏好性分析
2.7.1 同义密码子的偏好性分析 马家柚及其18个同属种叶绿体基因组CDS 基因密码子3 个位置GC含量的平均值为39.06%,GC1、GC2、GC3含量分别为46.37%、40.02%、30.79%,马家柚及其18 个同属种叶绿体基因组密码子3 个位置GC 含量的变化趋势为:GC3<GC2<GC1(图6);马家柚叶绿体基因组88 个CDS 基因密码子的ENC 值介于28.311~61.000 之间,平均值为48.02,84 个基因的ENC 值>35,4 个基因的ENC 值<35,密码子偏好性较弱;马家柚18 个同属种叶绿体基因组CDS 基因密码子的ENC 值介于26.309~61.000 之间,平均值为48.04,1511 个基因的ENC 值>35,71 个基因的ENC 值<35,密码子偏好性同样较弱。马家柚叶绿体基因组88 个CDS 基因序列共有32 个RSCU>1 的密码子,在这32个密码子中,除UGG、UUG、AUG外,其余都以A、U结尾;马家柚18个同属种叶绿体基因组CDS基因序列共有576 个RSCU>1 的密码子,在这576个密码子中,除UGG、UUG、AUG 外,其余也均以A、U结尾,密码子同样偏好以A、U结尾(图7)。
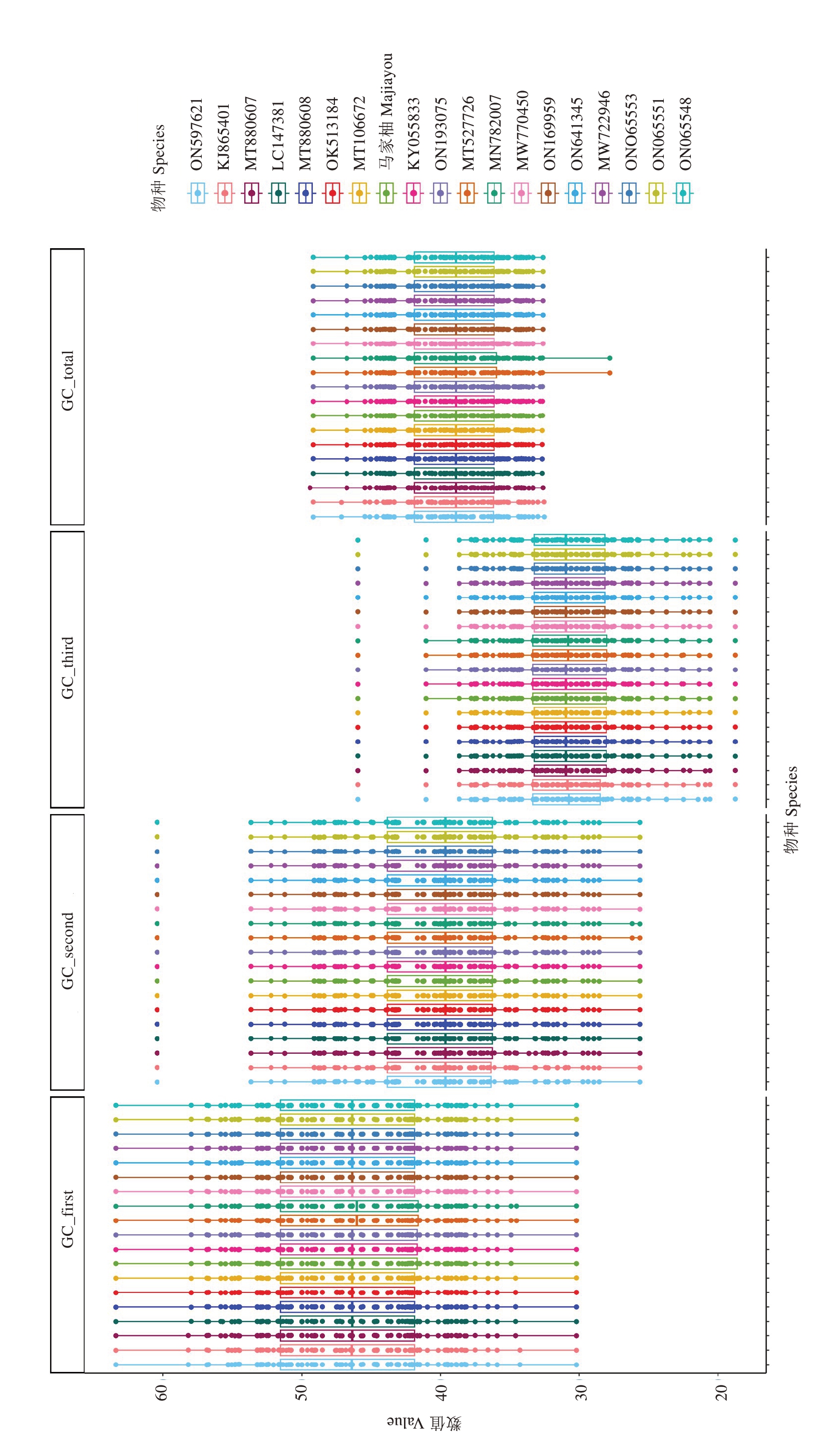
图6 马家柚及其18 个同属种叶绿体基因组密码子组成成分分析
Fig.6 Composition analysis of chloroplast genome codons of C.maxima (L.) Osbeck ‘Majiayou’ and its 18 congeneric species
GC_first、GC_second、GC_third 分别表示密码子第1、2、3 位的GC 含量;GC_total 表示密码子总GC 含量。
GC_first, GC_second and GC_third represent the GC content of codon 1, 2 and 3 respectively; GC_total represents the total GC content of the codons.
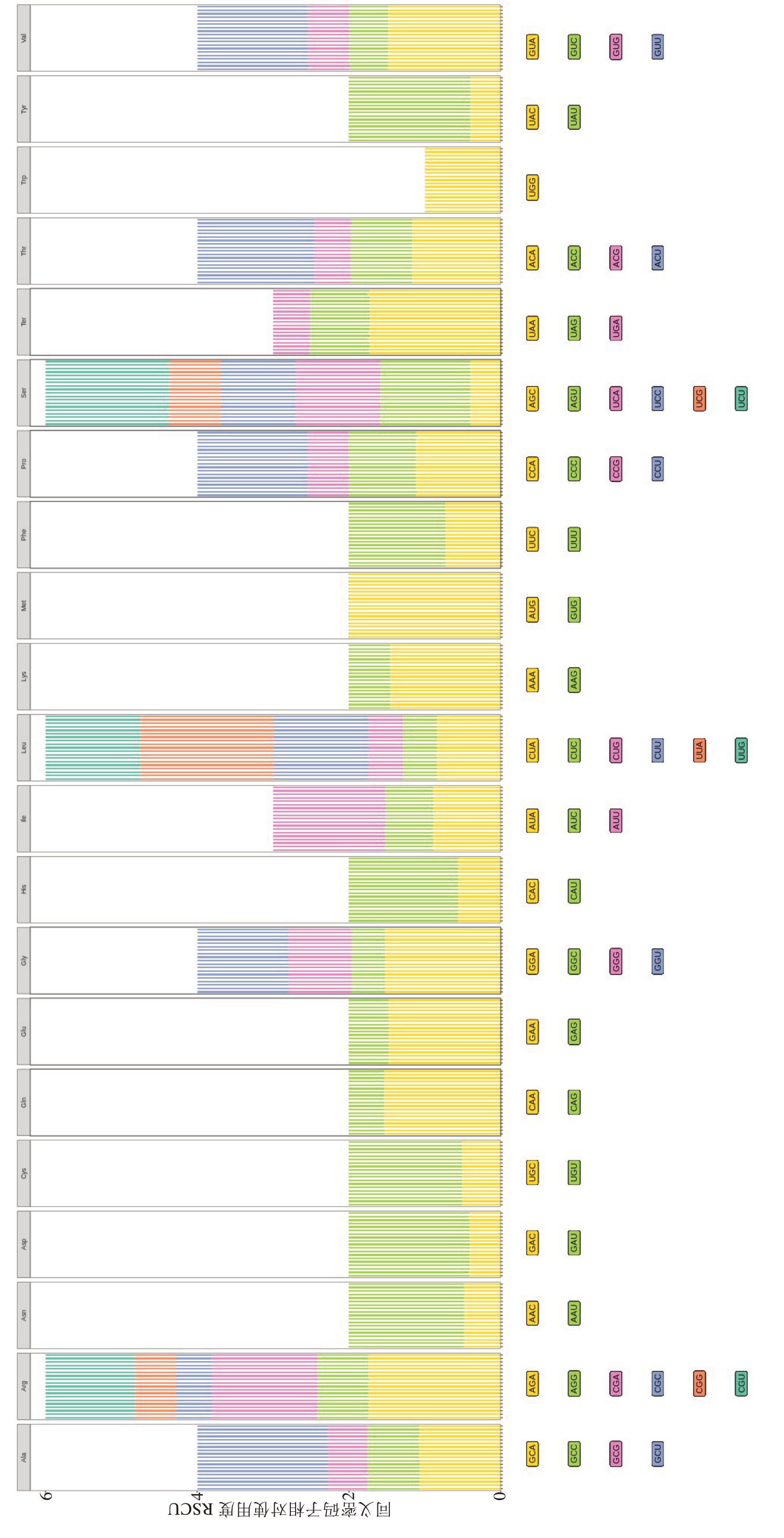
图7 马家柚及其18 个同属种叶绿体基因组密码子RSCU 分析
Fig.7 RSCU analysis of chloroplast genome codons of C.maxima (L.) Osbeck ‘Majiayou’ and its 18 congeneric species
2.7.2 中性绘图分析(GC3-GC12 分析)、ENC-plot分析和PR2-plot 分析 马家柚及其18 个同属叶绿体基因的GC3 含量分布在0.187 5~0.459 5 之间,GC12 含量分布在0.256 4~0.634 1 之间,二者绝大多数沿对角线上方分布(图8)。两者的线性相关系数r=0.031 6(R2=0.001),相关显著(p<0.05),说明GC12 与GC3 相关性不显著。回归系数为0.025,内部突变贡献率仅2.5%,自然选择贡献率为97.5%,表明马家柚及其18 个同属种叶绿体基因组密码子使用偏好性主要受自然选择的影响,而受内部突变压力的影响小。马家柚及其18 个同属种叶绿体基因组大部分基因位于标准曲线的下方(图8),大部分基因ENC 的实际值与预期值存在较大差异,且GC3值分布较为集中,可见自然选择是影响马家柚及其18 个同属种叶绿体基因组密码子使用偏好性的重要因子。从G3/GC3 轴看,马家柚及其18 个同属种叶绿体基因组中较多的基因分布于PR2-plot图的下部分(图8),说明4 种碱基在同义密码子第3 位上存在C>G 现象。从A3/AU3 轴看,马家柚及其18 个同属种叶绿体基因组中较多的基因分布于PR2-plot图的左部(图8),说明4 种碱基在同义密码子第3 位上存在T>A 现象。如密码子使用存在偏好性只受内部突变压力影响时,C 和G 以及A 和T 在第3位上的分布应相等,说明马家柚及其18 个同属种叶绿体基因组密码子使用偏好性在受内部突变影响的同时也会受到自然选择的影响。
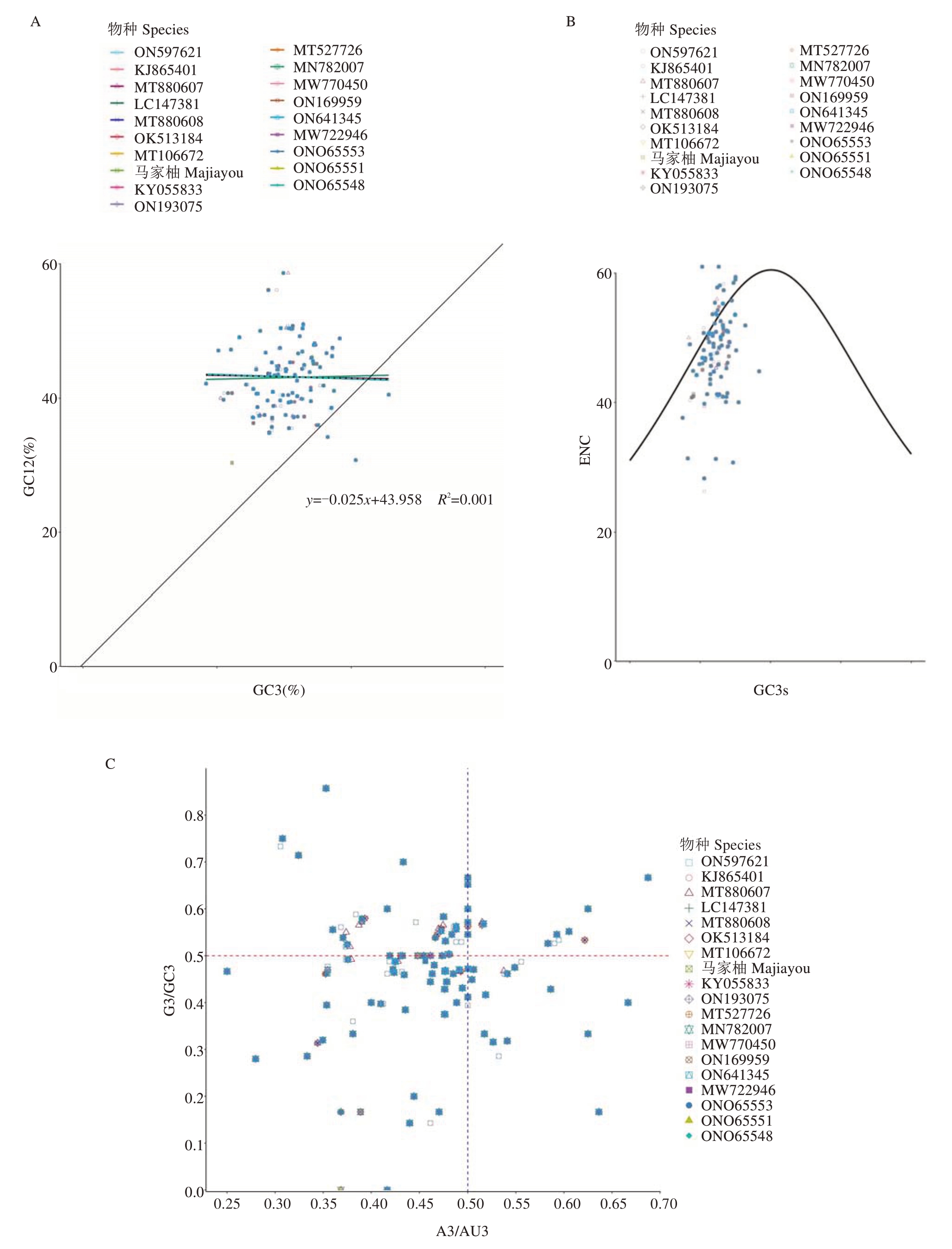
图8 马家柚及其18 个同属种叶绿体基因组密码子GC3-GC12 分析(A)、ENC-plot 分析(B)和PR2-plot 分析(C)
Fig.8 GC3-GC12 analysis(A),ENC-plot analysis(B)and PR2-plot analysis(C)of chloroplast genome codons of C.maxima(L.)Osbeck‘Majiayou’and its 18 congeneric species
2.7.3 最优密码子确定 马家柚叶绿体基因组满足相对同义密码子使用度(Relative synonymous codon usage,RSCU)>1(高频率密码子)且ΔRSCU(=RSCU 高表达-RSCU 低表达)≥0.08 的最优密码子有AAU、UGU、AAA、UUU、GCU、GGA、CCA、ACU、CGU、AGU(表6),共10 个,均以A、U 结尾。说明马家柚叶绿体基因组密码子偏好性是以A 和U结尾。
表6 马家柚叶绿体基因最优密码子
Table 6 Optimal codon of chloroplast genome of C.maxima(L.)Osbeck‘Majiayou’
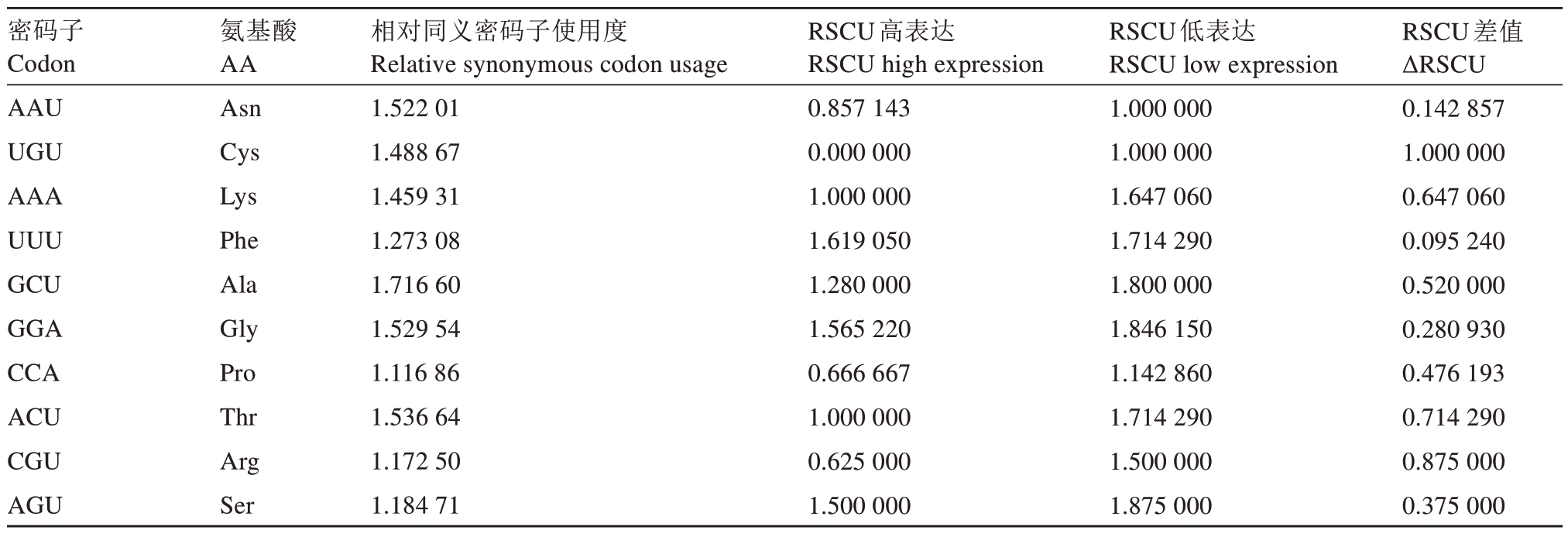
密码子Codon AAU UGU AAA UUU GCU GGA CCA ACU CGU AGU氨基酸AA Asn Cys Lys Phe Ala Gly Pro Thr Arg Ser相对同义密码子使用度Relative synonymous codon usage 1.522 01 1.488 67 1.459 31 1.273 08 1.716 60 1.529 54 1.116 86 1.536 64 1.172 50 1.184 71 RSCU高表达RSCU high expression 0.857 143 0.000 000 1.000 000 1.619 050 1.280 000 1.565 220 0.666 667 1.000 000 0.625 000 1.500 000 RSCU低表达RSCU low expression 1.000 000 1.000 000 1.647 060 1.714 290 1.800 000 1.846 150 1.142 860 1.714 290 1.500 000 1.875 000 RSCU差值ΔRSCU 0.142 857 1.000 000 0.647 060 0.095 240 0.520 000 0.280 930 0.476 193 0.714 290 0.875 000 0.375 000
2.8 系统发育分析
基于马家柚及其85 个同科种(芸香科柑橘亚科、芸香亚科和飞龙掌血亚科)和3 个外群种(橄榄NC048982、苦树MW801117、海人树MK830069)叶绿体基因组构建的系统发育树(图9)表明,马家柚[MJY,C.maxima(L.)Osbeck‘Majiayou’]与柚子(C.maxima,KY055833)、日本夏橙(C.natsudaidai,ON193075)、柚子(C.maxima,MT527726)、柚子(C.maxima,MN782007)单独聚为一分支。在这个分支中,马家柚[MJY,C.maxima(L.)Osbeck‘Majiayou’]又单独成一分支。说明马家柚[MJY,C.maxima(L.)Osbeck‘Majiayou’]与西双版纳东试早柚(C.maxima,KY055833,来源地:云南)、日本夏橙(C.natsudaidai,ON193075,来源地:韩国)、福建六月早蜜柚(C.maxima‘Liuyuezao’,MT527726,来源地:福建)、福建琯溪蜜柚[C.maxima(Burm.)Merr.‘Guanximiyou’,MN782007,来源地:福建]有亲缘关系,是一个柑橘属中较为独特的品种。

图9 基于叶绿体基因组的马家柚及其85 个同科种和3 个外群种的系统发育树
Fig.9 Phylogenetic tree of C.maxima(L.)Osbeck‘Majiayou’,85 species in the same family and 3 outgroup species(Glycosmis)based on chloroplast genome
3 讨 论
马家柚叶绿体基因组全长160 186 bp,与其他柑橘属植物叶绿体基因组大小差别不大,伍泓昆[44]的研究表明,红河大翼橙、枳、金柑、莽山野橘、柚和枸橼叶绿体基因组全长分别为161 419 bp、160 589 bp、160 184 bp、160 152 bp、160 097 bp 和159 952 bp;徐世荣等[21]和Xu 等[22]的研究也表明,福建六月早蜜柚(C.maxima‘Liuyuezao’)和福建琯溪蜜柚[C.maxi‐ma(Burm.)Merr.‘Guanximiyou’]的叶绿体基因组全长均为160 186 bp,与马家柚叶绿体基因组全长相同,说明在柑橘属内各个种的叶绿体基因组相对保守。福建六月早蜜柚[21]、福建琯溪蜜柚[22]、云南红河柑橘(C.hongheensis)[23]、阿曼酸橙(Omani lime,C.aurantiifolia)[25]、扁实柠檬(Shiikuwasha,C.de‐pressa Hayata)[26]、C.sinensis(L.)Osbeck‘Ridge Pineapple’[27]、手指柠檬(C.australasic)栽培种[24]和马家柚(MJY)叶绿体基因组平均GC 含量为38%~39%,其中IR 区的GC 含量高于LSC 区和SSC 区,SSC 区的GC 含量最低,究其原因,IR 区的rRNA 基因的GC 含量高,而SSC 区的GC 含量低,与位于SSC区的NADH基因相关[17]。
SSR又称微卫星,一般是1~6 bp的重复序列,具有高多态性和广泛分布的特点,是高等真核生物叶绿体基因组的重要组成部分,成为研究植物遗传多样性、植物品种鉴定和植物遗传稳定性的重要工具[17]。在本试验中,马家柚叶绿体基因组共检测到79 个SSR,包括单核苷酸重复序列和三核苷酸重复序列2 种不同类型,其中A 和T 重复占98.72%,表明马家柚叶绿体基因组SSR 偏好使用碱基A 和T,这与大多数柑橘属植物叶绿体SSR 序列的组成相似。例如,六月早蜜柚叶绿体基因组的SSR 为101 个,其中68 个A 和T 单碱基重复[21];阿曼酸橙叶绿体基因组的SSR 为109 个(多为A 和T 单碱基重复),大多数SSR 位于基因间区,少数位于CDS 基因(如matK和ycf1)[25]。本研究从马家柚叶绿体基因组中检测到的SSR 可说明马家柚的种间多态性,并获知ycf1基因有254个变异位点,变异位点最多。ycf1序列是叶绿体基因组内的片段,由于其进化速率快,序列变异较大,近年来已被陆续应用于兰科[45]等植物的分子鉴定中;且陆地植物物种在PCR 成功率、序列位点变异率及物种鉴别率等方面优于matK、rbcL 及trnH-psbA 等常用叶绿体DNA 条形码,因此被认为是最具潜力的陆地植物DNA 条形码序列[46]。可为马家柚品种的道地性的分子鉴定提供可靠的遗传标记,为后续对马家柚种质资源的化学成分、抗逆性和其他品质性状进行深入研究,为选育产量高、抗逆性强、品质优的新品种奠定了分子基础。
植物的叶绿体基因组的IR/SC 边界时常会发生收缩和扩张,这些收缩和扩张会导致假基因的产生、基因的重复以及基因的缺失[17]。马家柚及其18 个同属种叶绿体基因组的变异圈图、mVIST 结构变异图和Pi 多样性指数分析图显示,LSC 区的变异性整体较高,其次是SSC 区,IR 区变异性最低,10个高变异区域均位于LSC和SSC区,这可能与IR区进化的保守性有关[17]。IR/SC 边界的收缩和扩张决定在植物叶绿体基因组的演化进程[17]。徐世荣等[21]的研究表明,芸香科植物的基因结构和基因顺序较为相似,只有JLB(LSC与IRb边界)和JSB(IRb与SSC边界)2 个边界存在差异。JLB 边界包括2 种类型(第1 种类型:无基因横跨边界;第2 种类型:rpl22 基因横跨JLB 边界),JSB 边界包括3 种类型(第1 种类型:无基因横跨边界;第2种类型:ycf1基因横跨JSB边界;第3种类型:ndhF基因横跨JSB边界)。Su等[25]的研究表明,阿曼酸橙的IR、LSC 和SSC 区域之间的连接与血橙(C.sinensis)相似,rpl22 基因横跨JLB 边界。本试验结果也证实了这种观点,在本试验中,马家柚及其18 个同属种叶绿体基因组各个结构极为稳定,未发现明显的IR 扩张和收缩,马家柚及其18个同属种叶绿体基因组的JLB 边界有rps19 基因横跨边界,属于JLB 边界的第2种类型;马家柚及其18个同属种叶绿体基因组JSB 边界无基因横跨边界,均位于IRb 区的ycf1 基因和SSC 区的ndhF 基因之间,属于JSB边界的第1种类型。
植物叶绿体基因组第3 位碱基的突变为同义突变。叶绿体基因组密码子的偏好性是指植物使用同义密码子的频率存在差异,这种差异多是由碱基组成所造成的[17]。中性进化理论认为,氨基酸的改变取决于第1位和第2位碱基的非同义突变,不取决于第3 位碱基的同义突变,因此,GC3 一般作为衡量密码子偏好性的重要指标[17]。本研究中马家柚及其18个同属种叶绿体基因组密码子3 个位置GC 含量的平均值为39.06%,表明马家柚及其18个同属种叶绿体更倾向于使用A/U 密码子,这与徐世荣等[21]对六月早蜜柚叶绿体基因组的研究结果一致。马家柚及其18个同属种叶绿体基因组密码子GC3含量较低,这一特征与六月早蜜柚[21]相同,符合Campbll等[47]提出的假设:高等植物的密码子一般偏好以A/U 结尾。ENC 表示密码子偏离随机选择的程度,是衡量密码子使用偏好性的重要指标[44]。ENC 值越大,密码子使用偏好性越弱,ENC 值≤35,密码子偏好性较强[45]。马家柚叶绿体基因组88 个CDS 基因密码子的ENC 值介于28.311~61.000 之间,平均值为48.02,84 个基因的ENC 值>35,4 个基因的ENC 值<35,密码子偏好性较弱。马家柚叶绿体基因组psbM 和petG 基因ENC 值最高(61),说明这两个基因具有保守的DNA 序列,在进化过程中受自然选择影响小;马家柚叶绿体基因组中rpl32 基因的ENC 值最低(28.311),受突变的影响较小,密码子使用偏好性较强。
植物叶绿体基因组密码子偏好性的影响因子一般有自然选择和内部突变两种。在本试验中,中性绘图分析(GC3-GC12 分析)、ENC-plot 分析和PR2-plot分析表明在马家柚及其18个同属种在进化过程中,其密码子偏好性主要受自然选择而非受内部突变的影响,究其原因,可能是叶绿体作为进行光合作用的重要细胞器,其基因演化必然要受到自然选择的影响[17]。RSCU 分析是一种根据相对密码子偏好性来分析基因表达水平的方法。本研究采用高表达优越密码子方法确定了马家柚叶绿体基因组10 个最优密码子,3 个以A 结尾,7 个以U 结尾,由此可见马家柚叶绿体基因组中密码子偏好NNA、NNU型。这与徐世荣等[21]对六月早蜜柚叶绿体基因组的研究结果一致,说明柑橘属植物的密码子偏好性存在着一定的相似性,这种密码子使用模式可能是由于柑橘属植物叶绿体基因组密码子的使用偏好性在进化关系上较为保守。一般来说,在正向选择和突变压力较强的情况下,最优密码子的数量较多;而在纯化选择的情况下,最优密码子的数量较少。不同物种的最优密码子及数量不同,说明物种受到的进化压力存在差异[17]。本研究中共确定了10 个马家柚叶绿体基因组的最优密码子,数量偏少,因此推测马家柚叶绿体基因组可能处于纯化选择之下。
叶绿体基因组DNA 能有效进行物种鉴定和系统亲缘关系分析[17]。徐世荣等[21]和Xu等[22]的研究表明,六月早蜜柚与C.platymamma、柠檬和甜橙聚为一小分支,福建琯溪蜜柚与Low acid pummelo(C.maxima,NC034290.1)聚为一小分支;Zhang 等[23]的研究表明,云南红河柑橘与柠檬(C.limon,KY085897.1)、C.platymamma(NC030194.1)、甜橙(C.sinensis,DQ864733.1)和Low acid pummelo(C.maxima,NC034290.1)聚为一分支,但云南红河柑橘单独成一小分支,表明福建琯溪蜜柚与云南红河柑橘存在一定的亲缘关系,但也是一个柑橘属中较为独特的品种;Cai 等[24]的研究表明,手指柠檬栽培种与枸橼(C.medica,NC050939.1)聚为一分支,表明两者亲缘关系较近。目前,马家柚的种植区主要集中在江西上饶,现有研究的取样范围和分子标记选择受限,使得马家柚的属内进化关系仍模糊。刘勇[48]通过SSR 和AFLP 分子鉴定,认为马家柚与广丰周边地区的信木柚亲缘关系较近;徐宸宇等[49]对江西46份柚资源进行了SSR 分子鉴定,认为马家柚可能是由广丰本地土柚衍变而成的变异株系。笔者在本试验中,为了进一步揭示马家柚在柑橘属种间的亲缘关系,选取了马家柚及其85 个同科种(芸香科柑橘亚科、芸香亚科和飞龙掌血亚科)和3个外群种( 橄榄NC048982、苦树MW801117、海人树MK830069),利用FastTree 软件GTR 模型(Generalized time-reversible model)构建ML系统发育树。结果表明,马家柚(MJY,C.maxima(L.)Osbeck‘Majiayou’)与西双版纳东试早柚(C.maxima,KY055833,来源地:云南)、日本夏橙(C.natsudaid‐ai,ON193075,来源地:韩国)、福建六月早蜜柚(C.maxima‘Liuyuezao’,MT527726,来源地:福建)、福建琯溪蜜柚[C.maxima(Burm.)Merr.‘Guanximiyou’,MN782007,来源地:福建]有亲缘关系,是一个柑橘属中较为独特的品种。
4 结 论
马家柚叶绿体基因组全长160 186 bp,共注释到133 个功能基因,共检测到79个SSR 和34 个Longrepeat。马家柚叶绿体基因组SC/IR 边界较为保守,密码子偏好性较弱(主要受自然选择的影响),最优密码子有10 个(AAU、UGU、AAA、UUU、GCU、GGA、CCA、ACU、CGU、AGU)。马家柚与西双版纳东试早柚、日本夏橙、福建六月早蜜柚、福建琯溪蜜柚有亲缘关系,是柑橘属中一个较为独特的品种。
[1] 林涛,何丽云,王帆,李炳根,阿提克穆·麦麦提,周仁辉,程薪宇.马家柚花部特征及其访花昆虫种类调查[J].环境昆虫学报,2023,45(1):101-113.LIN Tao,HE Liyun,WANG Fan,LI Binggen,Atikemu·Maimaiti,ZHOU Renhui,CHENG Xinyu.The floral characteristics and its flower-visiting insects’species investigation of Citrus maxi‐ma(L.)Osbeck cv.‘Majiayou’[J].Journal of Environmental Entomology,2023,45(1):101-113.
[2] 毛桑隐,路志浩,张祥,叶俊丽,伊华林,柴利军,邓秀新,吴方方,徐强.花粉直感对马家柚果实品质的影响[J].果树学报,2023,40(11):2391-2402.MAO Sangyin,LU Zhihao,ZHANG Xiang,YE Junli,YI Hualin,CHAI Lijun,DENG Xiuxin,WU Fangfang,XU Qiang.Effect of xenia on fruit quality of Majiayou[J].Journal of Fruit Science,2023,40(11):2391-2402.
[3] 谢婧蘅,杨莉,旦世浩,邱丽,张王妮,刘德春,胡威,刘勇.套袋对马家柚果实外观及内在品质的影响[J].核农学报,2021,35(1):229-237.XIE Jingheng,YANG Li,DAN Shihao,QIU Li,ZHANG Wangni,LIU Dechun,HU Wei,LIU Yong.Effect of bagging on fruit appearance and inner quality of Majia pomelo[J].Journal of Nuclear Agricultural Sciences,2021,35(1):229-237.
[4] 毛小涛,陈凯,孙志锋,樊海敏,张处平.马家柚光合特性研究[J].上饶师范学院学报,2023,43(3):73-78.MAO Xiaotao,CHEN Kai,SUN Zhifeng,FAN Haimin,ZHANG Chuping.Study on photosynthetic characteristics of Citrus grandis (L.)Osbeck[J].Journal of Shangrao Normal University,2023,43(3):73-78.
[5] 姜启航,朱凯杰,吴方方,徐娟,徐强,柴利军,邓秀新,叶俊丽.套袋处理对‘马家柚’果实挥发性物质积累的影响[J].果树学报,2020,37(11):1701-1710.JIANG Qihang,ZHU Kaijie,WU Fangfang,XU Juan,XU Qiang,CHAI Lijun,DENG Xiuxin,YE Junli.Effects of fruit bagging on the accumulation of volatile compounds in‘Majiayou’pumelo[J].Journal of Fruit Science,2020,37(11):1701-1710.
[6] 邱丽,杨莉,旦世浩,刘德春,胡威,张王妮,刘勇.套袋对‘马家柚’果肉主要类胡萝卜素积累及相关基因表达的影响[J].果树学报,2020,37(2):153-163.QIU Li,YANG Li,DAN Shihao,LIU Dechun,HU Wei,ZHANG Wangni,LIU Yong.Effects of bagging on the accumulation of main carotenoids and the related gene expression in‘Majiayou’pomelo[J].Journal of Fruit Science,2020,37(2):153-163.
[7]易明亮,张王妮,杨莉,匡柳青,刘德春,刘勇,胡威.‘马家柚’果实发育期有机酸含量变化及柠檬酸代谢相关基因的表达分析[J].江西农业大学学报,2022,44(4):841-851.YI Mingliang,ZHANG Wangni,YANG Li,KUANG Liuqing,LIU Dechun,LIU Yong,HU Wei.Analysis of organic acid content and expression of citric acid metabolism related genes during fruit development of‘Majia’pomelo[J].Acta agriculturae universitatis Jiangxiensis,2022,44(4):841-851.
[8] 周丽明,韩金多.马家柚果酒发酵工艺及其抗氧化作用分析[J].南方农业学报,2018,49(2):348-353.ZHOU Liming,HAN Jinduo.Fermentation process and antioxidant effects of Majia pummelo wine[J].Journal of Southern Agriculture,2018,49(2):348-353.
[9] 杨莉,张涓涓,刘德春,刘山蓓,徐炳星,周施清,毛卫平,刘勇.马家柚粗皮果形成过程的解剖学观察[J].经济林研究,2017,35(3):152-155.YANG Li,ZHANG Juanjuan,LIU Dechun,LIU Shanbei,XU Bingxing,ZHOU Shiqing,MAO Weiping,LIU Yong.Anatomically observation on forming process of rough fruits in Citrus grandis[J].Nonwood Forest Research,2017,35(3):152-155.
[10] 张涓涓,杨莉,刘德春,刘山蓓,徐炳星,周施清,毛卫平,刘勇.土壤养分状况与马家柚果实品质相关性的多元分析[J].经济林研究,2015,33(4):25-31.ZHANG Juanjuan,YANG Li,LIU Dechun,LIU Shanbei,XU Bingxing,ZHOU Shiqing,MAO Weiping,LIU Yong.Multivariate analysis of relationship between soil nutrient status and fruit quality of Majia shaddock[J].Nonwood Forest Research,2015,33(4):25-31.
[11] SLOAN D B,TRIANT D A,FORRESTER N J,BERGNER L M,WU M,TAYLOR D R.A recurring syndrome of accelerated plastid genome evolution in the angiosperm tribe Sileneae(Caryophyllaceae)[J].Molecular Phylogenetics and Evolution,2014,72:82-89.
[12] NAZARENO A G,CARLSEN M,LOHMANN L G.Complete chloroplast genome of Tanaecium tetragonolobum:The first Bignoniaceae plastome[J].PLoS One,2015,10(6):e0129930.
[13] LI D M,LI J,WANG D R,XU Y C,ZHU G F.Molecular evolution of chloroplast genomes in subfamily Zingiberoideae(Zingiberaceae)[J].BMC Plant Biology,2021,21(1):558.
[14] LIANG H,ZHANG Y,DENG J B,GAO G,DING C B,ZHANG L,YANG R W.The complete chloroplast genome sequences of 14 Curcuma species:Insights into genome evolution and phylogenetic relationships within Zingiberales[J].Frontiers in Genetics,2020,11:802.
[15] KATHRIARACHCHI H,HOFFMANN P,SAMUEL R,WURDACK K J,CHASE M W.Molecular phylogenetics of Phyllanthaceae inferred from five genes(plastid atpB,matK,3'ndhF,rb‐cL,and nuclear PHYC))[J].Molecular Phylogenetics and Evolution,2005,36(1):112-134.
[16] DANIELL H,LIN C S,YU M,CHANG W J.Chloroplast genomes:Diversity,evolution,and applications in genetic engineering[J].Genome Biology,2016,17(1):134.
[17] 朱强龙,朱子成,王鹏飞,吕慧玲,崔浩楠,栾非时.葫芦科作物线粒体和叶绿体基因组研究进展[J].中国瓜菜,2016,29(8):1-8.ZHU Qianglong,ZHU Zicheng,WANG Pengfei,LÜ Huiling,CUI Haonan,LUAN Feishi.Advances on mitochondria and chloroplast genomes of cucurbit crop[J].China Cucurbits and Vegetables,2016,29(8):1-8.
[18] ABDULLAH,MEHMOOD F,RAHIM A,HEIDARI P,AHMED I,POCZAI P.Comparative plastome analysis of Blumea,with implications for genome evolution and phylogeny of Asteroideae[J].Ecology and Evolution,2021,11(12):7810-7826.
[19] LIU H,HUANG Y,DU X,CHEN Z,ZENG X,CHEN Y,ZHANG H.Patterns of synonymous codon usage bias in the model grass Brachypodium distachyon[J].Genetics and Molecular Research,2012,11(4):4695-4706.
[20] QI Y Y,XU W J,XING T,ZHAO M M,LI N N,YAN L,XIA G M,WANG M C.Synonymous Codon usage bias in the plastid genome is unrelated to gene structure and shows evolutionary heterogeneity[J].Evolutionary Bioinformatics Online,2015,11:65-77.
[21] 徐世荣,陈燕琼,潘东明,潘鹤立.六月早蜜柚叶绿体基因组及其特征分析[J].热带作物学报,2021,42(5):1223-1230.XU Shirong,CHEN Yanqiong,PAN Dongming,PAN Heli.Chloroplast genome sequence and characteristics analysis of Citrus maxima‘Liuyuezao’[J].Chinese Journal of Tropical Crops,2021,42(5):1223-1230.
[22] XU S R,HUANG C Y,DENG Y T,ZHOU L L,PAN D M,PAN H L.The complete chloroplast genome sequence of Citrus maxima (Burm.) Merr.‘Guanximiyou’[J].Mitochondrial DNA.Part B,Resources,2020,5(1):482-483.
[23] ZHANG Z H,LONG C R,JIANG Y,BEI X J,WANG S H.Characterization of the complete chloroplast genome of Citrus hongheensis,a key protected wild plant in Yunnan Province of China[J].Mitochondrial DNA.Part B,Resources,2020,5(3):3514-3515.
[24] CAI Q N,WANG H X,CHEN D J,KE X R,ZHU Z X,WANG H F.The complete chloroplast genome sequence of a Citrus aus‐tralasica cultivar(Rutaceae)[J].Mitochondrial DNA.Part B,Resources,2021,7(1):54-55.
[25] SU H J,HOGENHOUT S A,AL-SADI A M,KUO C H.Complete chloroplast genome sequence of Omani lime(Citrus auran‐tiifolia) and comparative analysis within the rosids[J].PLoS One,2014,9(11):e113049.
[26] ISHIKAWA R,BADENOCH N,MIYAGI K,MEDORUMA K,OSADA T,ONISHI M.Multi-lineages of Shiikuwasha (Citrus depressa Hayata) evaluated by using whole chloroplast genome sequences and its bio-diversity in Okinawa,Japan[J].Breeding Science,2016,66(4):490-498.
[27] BAUSHER M G,SINGH N D,LEE S B,JANSEN R K,DANIELL H.The complete chloroplast genome sequence of Citrus si‐nensis (L.) Osbeck‘Ridge Pineapple’:Organization and phylogenetic relationships to other angiosperms[J].BMC Plant Biology,2006,6:21.
[28] DOYLE J J.A rapid DNA isolation procedure for small quantities of fresh leaf tissue[J].Phytochemical Bulletin,1987,19(1):11-15.
[29] CHEN S F,ZHOU Y Q,CHEN Y R,GU J.Fastp:An ultra-fast all-in-one FASTQ preprocessor[J].Bioinformatics,2018,34(17):i884-i890.
[30] DIERCKXSENS N,MARDULYN P,SMITS G.NOVOPlasty:De novo assembly of organelle genomes from whole genome data[J].Nucleic Acids Research,2017,45(4):e18.
[31] HUANG X,MADAN A.CAP3:A DNA sequence assembly program[J].Genome Research,1999,9(9):868-877.
[32] TILLICH M,LEHWARK P,PELLIZZER T,ULBRICHTJONES E S,FISCHER A,BOCK R,GREINER S.GeSeq-versatile and accurate annotation of organelle genomes[J].Nucleic Acids Research,2017,45(W1):W6-W11.
[33] LOWE T M,EDDY S R.tRNAscan-SE:A program for improved detection of transfer RNA genes in genomic sequence[J].Nucleic Acids Research,1997,25(5):955-964.
[34] LOHSE M,DRECHSEL O,BOCK R.OrganellarGenome-DRAW (OGDRAW):A tool for the easy generation of highquality custom graphical maps of plastid and mitochondrial genomes[J].Current Genetics,2007,52(5/6):267-274.
[35] GRANT J R,STOTHARD P.The CGView Server:A comparative genomics tool for circular genomes[J].Nucleic Acids Research,2008,36(Web Server issue):W181-W184.
[36] THIEL T,MICHALEK W,VARSHNEY R,GRANER A.Exploiting EST databases for the development and characterization of gene-derived SSR-markers in barley(Hordeum vulgare L.)[J].Theoretical and Applied Genetics,2003,106(3):411-422.
[37] KURTZ S,CHOUDHURI J V,OHLEBUSCH E,SCHLEIERMACHER C,STOYE J,GIEGERICH R.REPuter:The manifold applications of repeat analysis on a genomic scale[J].Nucleic Acids Research,2001,29(22):4633-4642.
[38] SHARP P M,LI W H.Codon usage in regulatory genes in Esche‐richia coli does not reflect selection for‘Rare’codons[J].Nucleic Acids Research,1986,14(19):7737-7749.
[39] PETKAU A,STUART-EDWARDS M,STOTHARD P,VAN DOMSELAAR G.Interactive microbial genome visualization with GView[J].Bioinformatics,2010,26(24):3125-3126.
[40] AMIRYOUSEFI A,HYVÖNEN J,POCZAI P.IRscope:An online program to visualize the junction sites of chloroplast genomes[J].Bioinformatics,2018,34(17):3030-3031.
[41] ROZAS J,ROZAS R.DnaSP,DNA sequence polymorphism:An interactive program for estimating population genetics parameters from DNA sequence data[J].Bioinformatics,1995,11(6):621-625.
[42] KATOH K,STANDLEY D M.MAFFT multiple sequence alignment software version 7:Improvements in performance and usability[J].Molecular Biology and Evolution,2013,30(4):772-780.
[43] PRICE M N,DEHAL P S,ARKIN A P.FastTree 2:Approximately maximum-likelihood trees for large alignments[J].PLoS One,2010,5(3):e9490.
[44] 伍泓昆.基于DArT 芯片及叶绿体基因组分析的柑橘属植物进化与分类研究[D].重庆:西南大学,2016.WU Hongkun.Evaluation and phylogenetic relationship of Citrus L.based on DArT markers and chloroplast genomes analyses[D].Chongqing:Southwest University,2016.
[45] NEUBIG K M,WHITTEN W M,CARLSWARD B S,BLANCO M A,ENDARA L,WILLIAMS N H,MOORE M.Phylogenetic utility of ycf1 in orchids:A plastid gene more variable than matK[J].Plant Systematics and Evolution,2009,277(1/2):75-84.
[46] DONG W P,XU C,LI C H,SUN J H,ZUO Y J,SHI S,CHENG T,GUO J J,ZHOU S L.ycf1,the most promising plastid DNA barcode of land plants[J].Scientific Reports,2015,5:8348.
[47] CAMPBELL W H,GOWRI G. Codon usage in higher plants,green algae,and cyanobacteria[J].Plant Physiology,1990,92(1):1-11.
[48] 刘勇.柚类资源分子系统学及其核心种质构建研究[D].武汉:华中农业大学,2005.LIU Yong.Molecular phylogenetic analysis and core collection construction using SSR and AFLP markers in pummelo[D].Wuhan:Huazhong Agricultural University,2005.
[49] 徐宸宇,曹立新,唐启正,吴巨勋,伊华林.马家柚遗传来源鉴定与适宜授粉品种筛选[J].华中农业大学学报(自然科学版),2022,41(2):124-135.XU Chenyu,CAO Lixin,TANG Qizheng,WU Juxun,YI Hualin.Identification of Majia pomelo germplasm and screening of varieties with suitable pollination[J].Journal of Huazhong Agricultural University (Natural Science Edition),2022,41(2):124-135.