在自然生长条件下,植物经常会遭受非生物胁迫的影响。由水、盐、光、温度和营养等环境因素引起的非生物胁迫会抑制植物生长,甚至降低植物存活率,因此,提高植物对非生物胁迫的抗性对农业生产至关重要[1]。在长期进化过程中,为了更好地生存,植物进化出了一系列生理生化机制来适应和抵御不良环境[2-4]。非生物胁迫[5]可激活植物钙离子通路诱导植物产生抗性反应。植物遭受环境刺激后,可通过Ca2+通道等转运系统使细胞胞质中游离Ca2+浓度产生变化,引发相应的保护性生理反应[6],这是植物应对不良环境的一个重要途径。
高渗性门控非选择性钙渗透阳离子通道[Reduced hyperosmolality-induced(Ca2+)increase channel,OSCA]是一种钙渗透性阳离子通道,在感受外源和内源的渗透变化以及调节植物生长发育中起着关键作用[7]。真核细胞通过Ca2+传导通道能够产生胞质Ca2+信号,Ca2+是动植物涉及应激响应和发育的一种多功能信使[8]。钙离子(Ca2+)浓度的空间和时间变化对真核生物信号调节具有重要作用[9-10]。近年来,对许多植物进行的研究表明,OSCA基因在响应渗透相关的非生物胁迫中发挥重要作用[11-14]。2014 年,OSCA 基因家族在拟南芥(Arabidopsis thaliana)中首次被鉴定,其包含15 个家族成员,且均含有与植物响应逆境胁迫相关的DUF221结构域,其中AtOSCA1.1、AtOSCA3.1、AtOSCA4.1 和AtOSCA1.2/AtCSC1均编码OSCA通道蛋白并参与高渗应激响应[13,15]。番茄(Solanum lycopersicum)中鉴定出11 个OSCA 基因家族成员,其中SlOSCA3 在低温、干旱胁迫和脱落酸(ABA)处理下表达量发生显著变化[12]。大豆(Glycine max)中包含20个OSCA家族成员,其中GmOSCA1.1、GmOSCA1.2、GmOSCA1.3、GmOSCA1.4和GmOSCA1.5可能在渗透胁迫响应中发挥作用,且可能增强大豆耐旱性[16]。水稻(Oryza sativa)中鉴定到11 个OSCA 基因,其中10 个OSCA基因受渗透相关的处理(PEG、NaCl、ABA)诱导并产生差异表达[11]。梨(Pyrus bretschneideri)中鉴定到16 个OSCA 基因,其中PbrOSCA2.6 和PbrOSCA3.2是感受渗透胁迫的重要基因,抑制PbrOSCA2.6 和PbrOSCA3.2表达后,花粉管对渗透胁迫的敏感性下降[17]。此外,在小麦[14]、玉米[18]等植物中也鉴定出OSCA基因家族成员,而有关猕猴桃OSCA基因家族及其在非生物胁迫下的表达情况尚未进行研究。
猕猴桃是猕猴桃科猕猴桃属的重要果树,由于其果实风味独特、营养丰富、抗坏血酸含量高而备受关注[19]。然而,在自然条件下,猕猴桃树体经常遭遇各种非生物胁迫,严重影响果实产量和品质。为此,笔者在本研究中对猕猴桃OSCA家族成员进行了全基因组鉴定,分析了其理化性质、序列特征和启动子区域的顺式作用元件。此外,利用实时定量PCR(quantitative real-time PCR,qRT-PCR)方法检测了它们在干旱、盐、低温和高温等非生物胁迫下的表达特征。为猕猴桃OSCA基因功能验证奠定了重要基础,并为利用关键OSCA 基因提高猕猴桃植株对非生物胁迫的抗性提供了理论依据。
1 材料和方法
1.1 材料及处理
试验材料为江西农业大学猕猴桃研究所保存的美味猕猴桃海沃德(Actinidia deliciosa)组培苗(由西北农林科技大学李明军教授惠赠)。在相对湿度75%、温度25 ℃,光照度4000 lx,光周期14 h(光)/10 h(暗)条件下的组培间,在MS培养基中继代培养30 d后,选取长势一致的组培苗,置于温度25 ℃、相对湿度为80%、光周期14 h(光)/10 h(暗)、光照度10 000 lx的人工气候箱中预培养7 d后,对试验材料进行不同非生物胁迫处理。试验共设置4 个处理,分别为:干旱胁迫(15% PEG6000 溶液)、盐胁迫(150 mmol·L-1 NaCl溶液)、低温胁迫(4 ℃)、高温胁迫(42 ℃)。各处理组分别在0 h(对照)、24 h、48 h进行采样,采集猕猴桃组培苗形态学上端第3、4 片完全展开的叶片,每个采样时间点设置3 个生物学重复,每个重复包括5株植株,叶片采集后立即在液氮中冷冻并将样品置于-80 ℃冰箱保存。
1.2 猕猴桃OSCA 基因家族成员鉴定、染色体定位及理化性质分析
在拟南芥基因组数据库(TAIR,https://www.arabidopsis.org/)下载所有拟南芥OSCA 蛋白序列,从猕猴桃基因组数据库(https://kiwifruitgenome.org/)下载中华猕猴桃(A.chnensis)Red 5基因组全蛋白序列和基因注释信息文件,利用TBtools软件鉴定出所有猕猴桃OSCA 候选蛋白。利用Pfam(https://pfam.xfam.org/search/)平台对候选序列进行保守结构域检索并筛选出包含DUF221,也称RSN1_7TM(PF:02714)结构域的猕猴桃OSCA 蛋白序列。使用Tbtools 对猕猴桃OSCA 基因家族成员进行染色体定位。利用在线网站(https://wolfpsort.Hgc.jp/)对猕猴桃OSCA 蛋白进行亚细胞定位预测。在ExPASy 网站(https://web.Expasy.org/protparam/)分析猕猴桃OSCA 蛋白的氨基酸数目、等电点(pI)、分子质量(MW)、不稳定指数等理化信息。
1.3 猕猴桃OSCA蛋白家族成员系统进化分析
从TAIR 数据库获取拟南芥全基因组和OSCA家族成员的蛋白质序列,从Ensembl Plants 数据库(https://plants.ensembl.org/index.html)获取水稻和番茄的全基因组序列。利用TBtools按1.2所述方法获取水稻和番茄OSCA 基因家族成员及其蛋白质序列。使用MEGA 11 软件中的Neighbor-Joining 法对拟南芥、水稻、番茄和猕猴桃OSCA蛋白家族成员序列进行比对并构建系统进化树,设置bootstrap 重复次数为1000次。
1.4 猕猴桃OSCA基因家族成员共线性分析
为了分析猕猴桃与拟南芥OSCA基因的同源关系,利用MCScanX(多重共线扫描工具包),期望值设置为1e-10,分析其共线块。利用TBtools 分析共线性文件,并绘制共线性图像。使用相同方法对猕猴桃基因组内的OSCA基因共线性关系进行分析。
1.5 猕猴桃OSCA家族基因结构、蛋白基序与保守结构域分析
利用TBtools 的MEME 模块分析猕猴桃OSCA基因家族的蛋白质序列,将生成的文件利用TBtools可视化模块对蛋白家族进行motif 分析并保存最终结果。利用NCBI 的Batch-CD-Search 功能(https://www.ncbi.nlm.nih.gov/Structure/bwrpsb/bwrpsb.cgi),导出猕猴桃OSCA 蛋白家族成员数据,并使用TBtools 进行可视化分析。使用TBtools 生成猕猴桃OSCA基因家族成员的基因结构分析图。
1.6 猕猴桃OSCA 基因家族成员启动子区域顺式作用元件分析
利用TBtools筛选出猕猴桃OSCA基因家族成员基因上游2000 bp 的启动子序列,使用PlantCare 系统(https://bioinformatics.psb.ugent.be/webtools/plantcare/html/)检测启动子区域顺式作用元件。利用TBtools对获得的顺式作用元件进行可视化绘图。
1.7 猕猴桃OSCA 基因家族成员在非生物胁迫下的表达分析
利用与中华猕猴桃亲缘关系相近的美味猕猴桃品种海沃德组培苗,使用qRT-PCR 方法对猕猴桃OSCA家族所有基因在非生物胁迫下的相对表达量进行分析。用北京擎科生物科技有限公司的RNAprep Pure 多糖多酚植物RNA 提取试剂盒提取组培苗叶片RNA。用北京聚合美生物科技有限公司M5 Super plus qPCR RT kit with gDNA remover 试剂盒进行cDNA第一链合成。使用Primer Premier 5软件设计实时荧光定量引物(表1)并进行引物合成。以猕猴桃Actin基因[20]作为内参基因。设置3次生物学重复。PCR 仪型号为CFX96TM Real-Time System。qRT-PCR反应程序为:95 ℃1 min,95 ℃10 s,57 ℃5 s,72 ℃12 s,40 个循环。使用2-∆∆CT方法计算基因的相对表达量[21]。使用SPSS 软件的独立样本T 检测方法,对3 次重复结果数据进行差异显著性分析。
表1 实时荧光定量PCR 引物
Table 1 Primers for quantitative real-time PCR analysis

?
2 结果与分析
2.1 猕猴桃OSCA基因家族成员及其理化性质
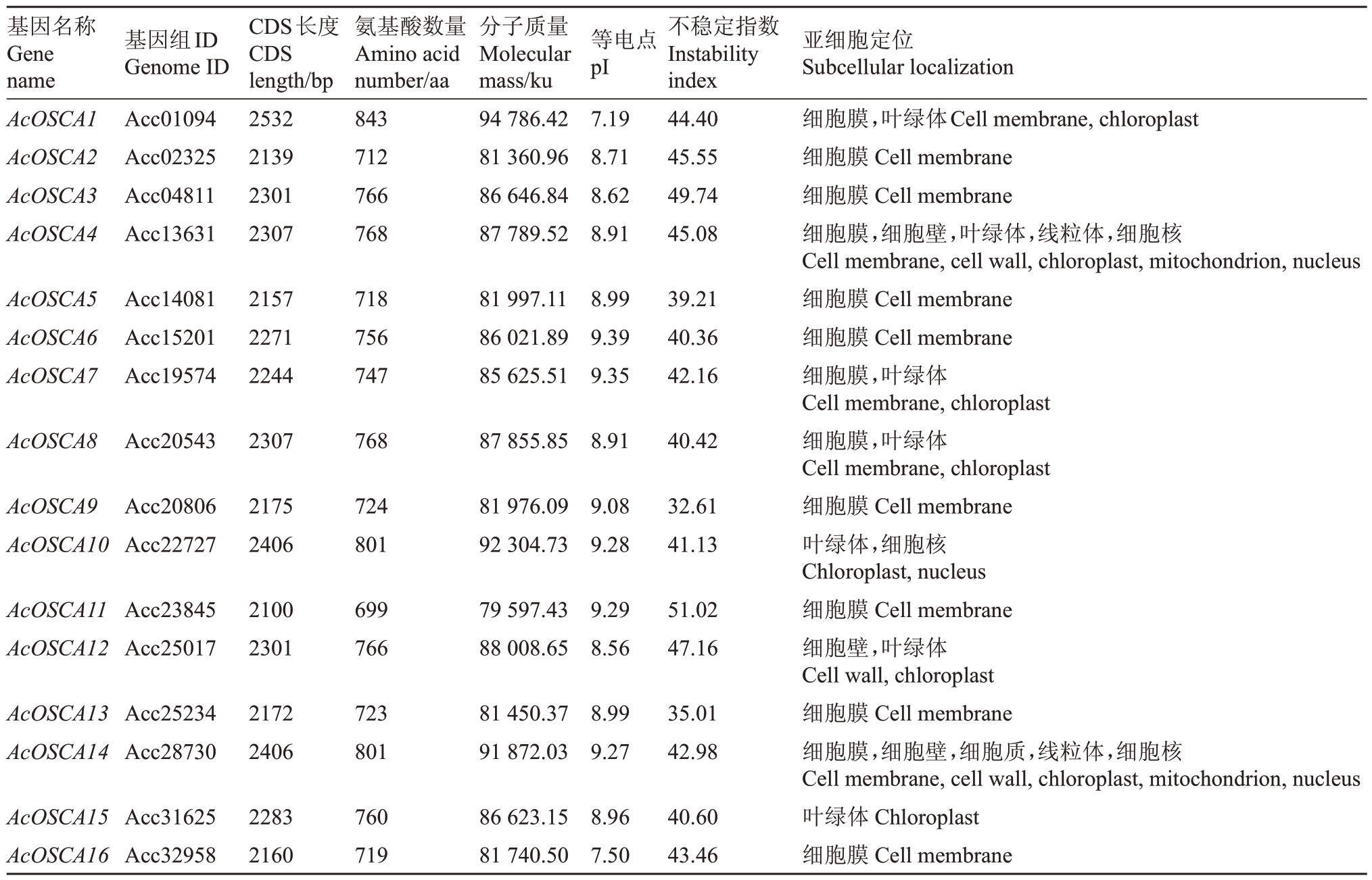
从中华猕猴桃Red5 全基因组中鉴定到16 个OSCA基因家族成员(表2),它们的编码序列(CDS)长度差异较小,范围为2100~2532 bp,其中AcOSCA11 最短,编码699 个氨基酸残基;最长的AcOSCA1 编码843 个氨基酸残基。基因CDS 平均值为2266 bp。等电点最小的是AcOSCA1的编码产物,为7.19,最大的是AcOSCA6编码产物,为9.39。等电点平均值为8.81。猕猴桃OSCA 蛋白均为碱性蛋白,分子质量范围为79 597.43~94 786.42 ku,分子质量平均值为85 978.57 ku。AcOSCA5、AcOSCA9、AcOSCA13 的不稳定指数小于40,编码产物稳定性较高,其余13个AcOSCA基因编码产物稳定性较低。亚细胞定位预测结果表明,AcOSCA4 和AcOSCA14 定位于细胞膜、细胞壁、叶绿体、线粒体和细胞核中,而其他14个成员定位于细胞膜和(或)叶绿体中。
表2 猕猴桃OSCA 基因家族信息
Table 2 Information of OSCA gene family members in kiwifruit
?
2.2 猕猴桃OSCA基因家族成员染色体定位
猕猴桃16 个OSCA 基因分别定位于29 条染色体中的13条,分别为第1、2、4、12、13、17、18、20、21、22、25、28、29 号染色体。依据染色体定位信息,将它们分别命名为AcOSCA1 至AcOSCA16,其中,13、18、22 号染色体上各有2 个OSCA 基因,1、2、4、12、17、20、21、25、28和29号染色体上各包含1个OSCA基因(图1)。
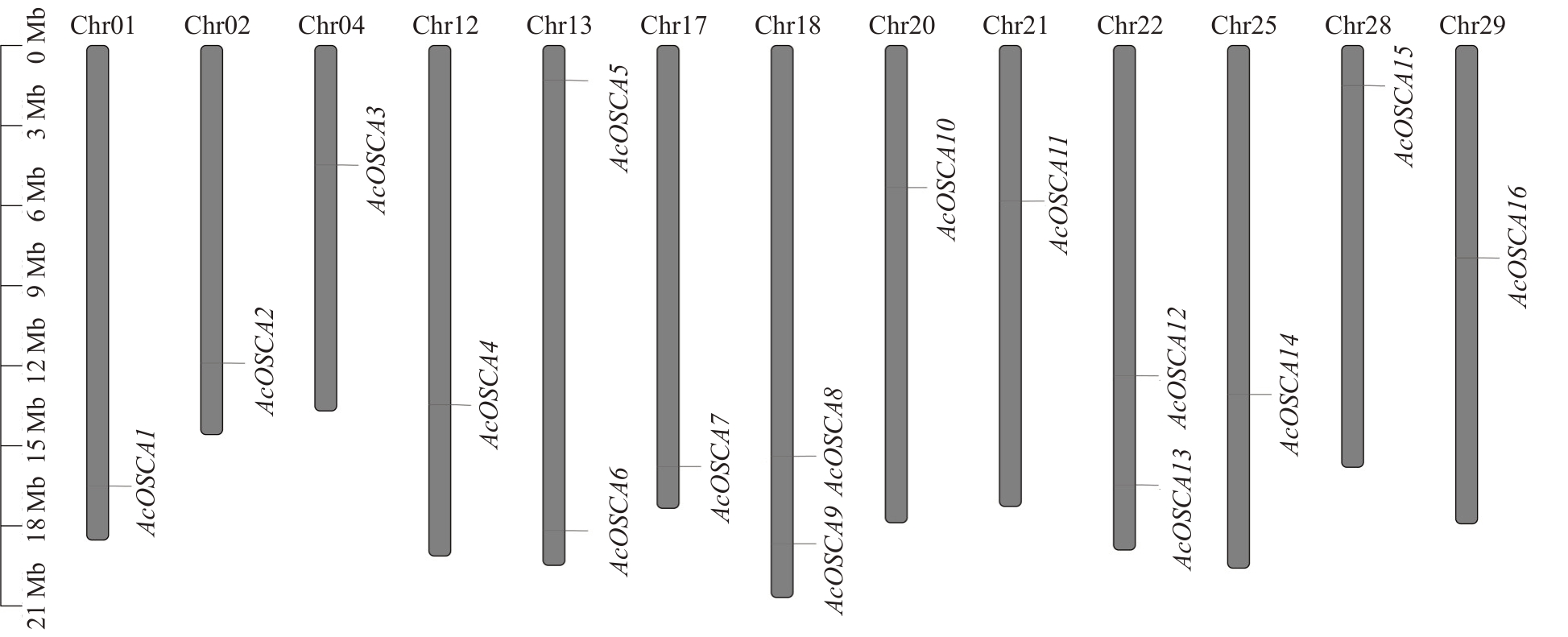
图1 猕猴桃OSCA 基因家族成员在染色体上的分布
Fig.1 Chromosomal location of kiwifruit OSCA gene family members
2.3 猕猴桃OSCA蛋白家族成员聚类
为明确OSCA蛋白家族成员与其他植物中同源蛋白的进化及亲缘关系,利用11 个水稻、15 个拟南芥、12个番茄和16个猕猴桃OSCA蛋白序列构建系统进化树。结果表明,OSCA 进化树可分为4 个亚族,即GroupⅠ、Group Ⅱ、Group Ⅲ、Group Ⅳ。猕猴桃与番茄OSCA蛋白家族成员在4个亚族中均有分布(图2)。AcOSCA 成员在Group Ⅲ和Group Ⅳ中分布较多,分别有5个和8个成员。

图2 4 个物种OSCA 蛋白的系统进化树
Fig.2 Phylogenetic tree of OSCA proteins from four plant species
2.4 猕猴桃与拟南芥OSCA基因成员间的共线性关系
共线性分析结果表明,15 个猕猴桃OSCA 基因与13 个拟南芥OSCA 基因之间存在24 对共线性关系(图3)。其中,AcOSCA4 在拟南芥中存在4 个共线性基因,分别是AT1G11960.3、AT1G62320.5、AT3G21620.1 和AT4G22120.3;AcOSCA8 在拟南芥中存在4 个共线性基因,分别是AT1G11960.3、AT1G62320.5、AT4G04340.3 和AT4G22120.3;AcOSCA2、AcOSCA6、AcOSCA12 在拟南芥中均存在2 个共线性基因,分别是AT1G69450.3 和AT3G01100.4,AT4G02900.1 和 AT4G15430.1,AT4G04340.3 和AT4G22120.3。其他10个猕猴桃OSCA基因成员分别与拟南芥中的1个OSCA基因存在共线性关系,这些基因对分别是AcOSCA1 与AT4G35870.1、AcOSCA3 与AT1G10090.1、AcOSCA5与AT1G69450.3、AcOSCA7与AT4G02900.1、AcOSCA9与AT1G30360.1、AcOSCA10与AT1G32090.1、AcOSCA11 与AT1G10090.1、AcOSCA13 与AT1G30360.1、AcOSCA15 与AT4G02900.1、AcOSCA16与AT1G69450.3。
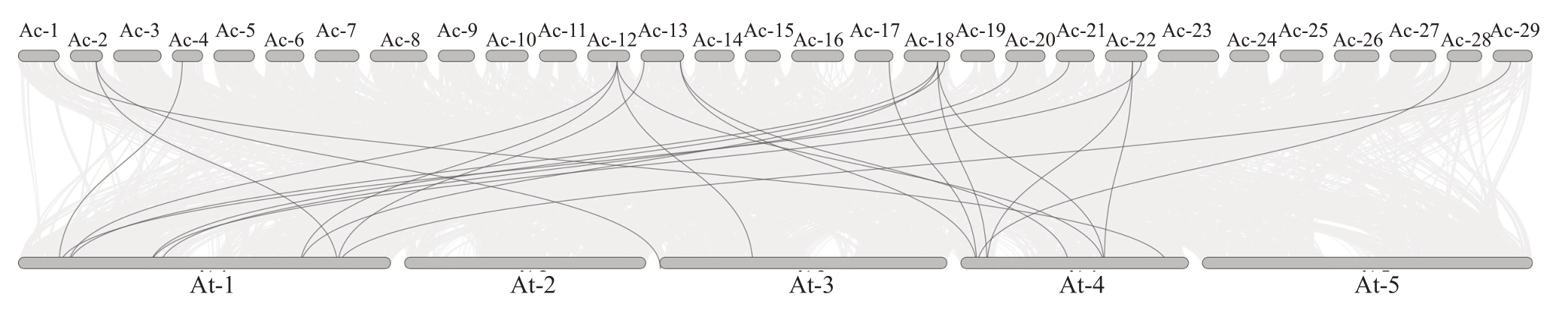
图3 猕猴桃与拟南芥OSCA 基因成员间的共线性关系
Fig.3 The syntenic relationship of OSCA genes between kiwifruit and Arabidopsis
2.5 猕猴桃基因组内OSCA基因间的共线性关系
猕猴桃OSCA基因间存在8对共线性基因对(图4)。分别为AcOSCA3 与AcOSCA11、AcOSCA4 与AcOSCA12、AcOSCA5 与AcOSCA16、AcOSCA6 与AcOSCA7 及AcOSCA15、AcOSCA7 与AcOSCA15、AcOSCA8 与AcOSCA12、AcOSCA9 与AcOSCA13。其中,AcOSCA6、AcOSCA7 和AcOSCA15 分别存在于13、17、28 号染色体上,且三者间均为共线性关系。
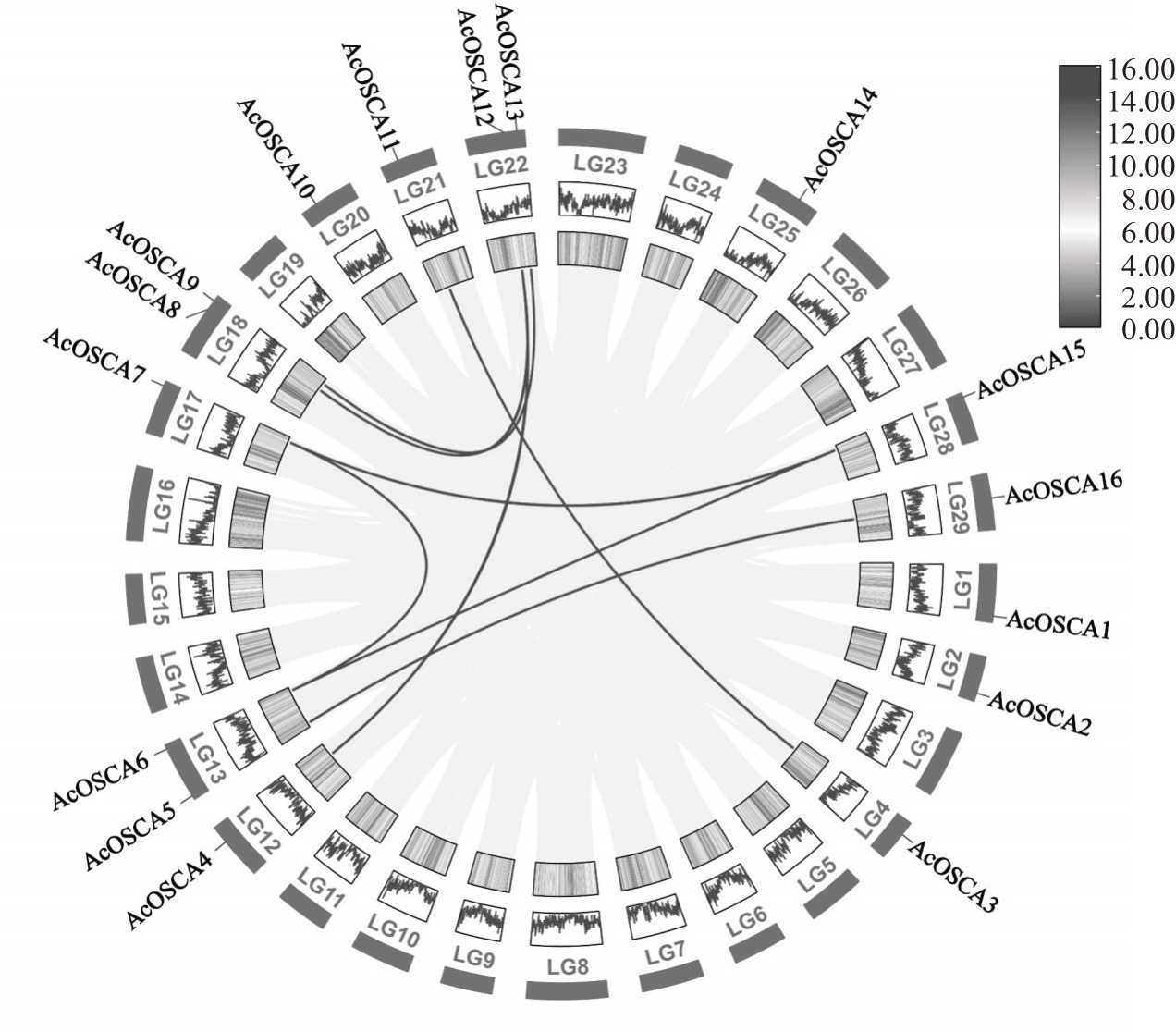
图4 猕猴桃OSCA 基因家族成员共线性关系
Fig.4 The syntenic relationship of OSCA gene family in kiwifruit
2.6 猕猴桃OSCA 家族成员基因结构、蛋白基序与保守结构域
通过可视化AcOSCA基因CDS和相应的基因组DNA 序列可知,其CDS 序列内含子分布较保守,在系统进化树同一发育枝中,大多数成员具有相同数量的内含子,例如,Group Ⅱ、Group Ⅲ、Group Ⅳ中基因内含子数目分别为5、10~11以及9~11个,Group Ⅰ中的基因成员无内含子。通过MEME软件对AcOSCA家族成员编码的蛋白序列进保守基序分析,结果发现,该家族蛋白序列非常保守,每个成员均包含3个相同的Motif基序,且它们的排列顺序相同(图5)。

图5 猕猴桃OSCA 家族成员蛋白基序(A)、基因结构(B)与Motif 基序(C)
Fig.5 Conserved domains(A),gene structure(B)and Motif domains(C)of OSCA family members in kiwifruit
2.7 猕猴桃OSCA 基因家族成员启动子区域顺式作用元件
对AcOSCA 家族基因起始密码子上游2000 bp启动子区域序列进行顺式作用元件进行分析,结果发现,AcOSCA 基因启动子区包含多种与逆境和激素应答相关的作用元件(表3)。主要包括6 种胁迫相关顺式作用元件,即:干旱诱导性的MYB 结合位点(MBS)、CAAT-box、厌氧诱导相关的顺式作用调节元件(ARE)、防御和应激反应相关的顺式作用元件(TC-rich)、低温诱导的顺式元件(LTR)、厌氧诱导的增强子类顺式作用元件(GC-motif);以及4 种激素相关顺式作用元件:脱落酸响应顺式作用元件(ABRE)、赤霉素反应元件(GARE-motif)、茉莉酸甲酯响应元件(TGACG motif/CGTCA-motif)、赤霉素应答元件(P-box)。11 个AcOSCA 基因启动子区域含有涉及干旱诱导性的MBS 元件和厌氧诱导相关ARE 元件,推测这些基因与干旱诱导应答有关。14 个AcOSCA 基因含有脱落酸响应顺式作用元件ABRE,5 个AcOSCA 基因含有低温诱导LTR元件和涉及防御与应激反应的TC-rich repeats 元件,推测这些基因与胁迫应答和应激反应有关。此外,每个AcOSCA 都含有丰富的CAAT-box 和茉莉酸甲酯响应元件TGACG-motif或CGTCA-motif。
表3 猕猴桃OSCA 基因顺式作用元件及数量
Table 3 Cis-elements and number of OSCA genes in kiwifruit
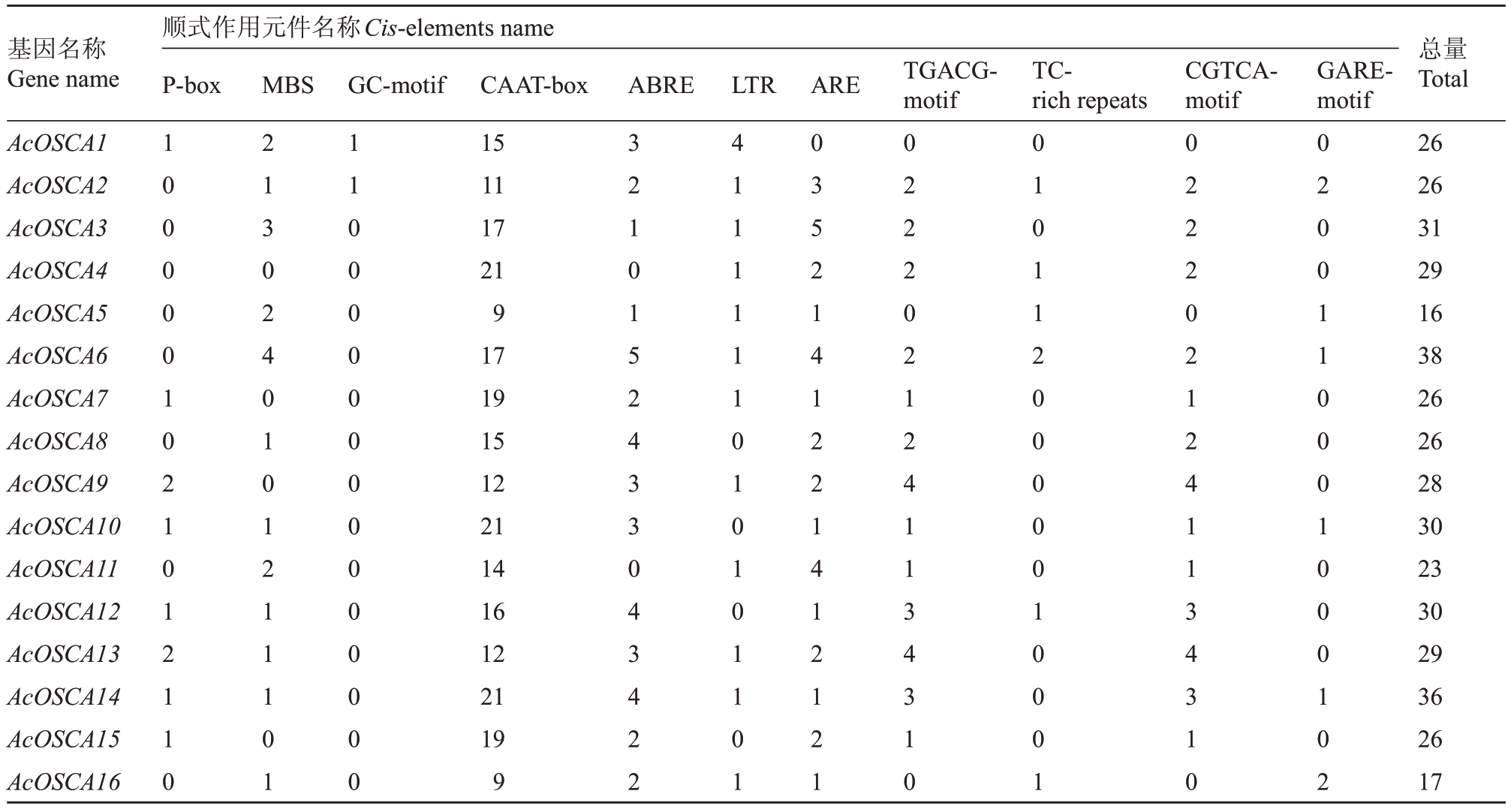
?
2.8 猕猴桃OSCA 家族基因成员在不同非生物胁迫下的表达特征
为了解猕猴桃OSCA家族基因在不同非生物胁迫下的表达情况,对它们在干旱、盐、低温和高温等非生物胁迫下的相对表达量进行了分析。结果发现,大多数OSCA 基因均能响应干旱、盐、高温和低温胁迫,不同AdOSCA 成员在这些非生物胁迫下具有不同的表达模式(图6)。在15%PEG6000处理的干旱胁迫下,与对照(0 h)相比,AdOSCA9 和AdOS-CA13在处理48 h时表达量显著下调,其他AdOSCA基因在遭受干旱胁迫后表达量均有不同程度上调。其中AdOSCA3、AdOSCA4、AdOSCA7、AdOSCA8 和AdOSCA15 在24 h 时相对表达量分别显著上调至对照的3.0、2.8、3.2、3.8 和3.1 倍,AdOSCA8 相对表达量最高且在48 h 时表达量是对照的2.5 倍。在150 mmol·L-1 NaCl 溶液处理的盐胁迫下,除AdOSCA4 表达下调外,其他AdOSCA 基因在24 h 和48 h表达量均上调,其中AdOSCA3、AdOSCA8 和AdOSCA12 在24 h 时表达量分别显著上调至对照的3.2、5.3 和2.8 倍,且在48 h 时显著上调至对照的3.4、10.0 和4.0 倍。在42 ℃处理的高温胁迫条件下,AdOSCA3 在24 h 时上调至对照的2.9 倍,在48 h 时表达量为对照的2.6 倍。在4 ℃处理的低温胁迫下,AdOSCA3 与AdOSCA14 在24 h 时分别上调至对照的2.9 和2.4 倍;AdOSCA8 在24 h 时表达量上调至对照的2.1 倍且在48 h 时表达量为对照的2.9 倍;AdOSCA1 在48 h 时相对表达量最高,为对照3.4 倍(图6)。
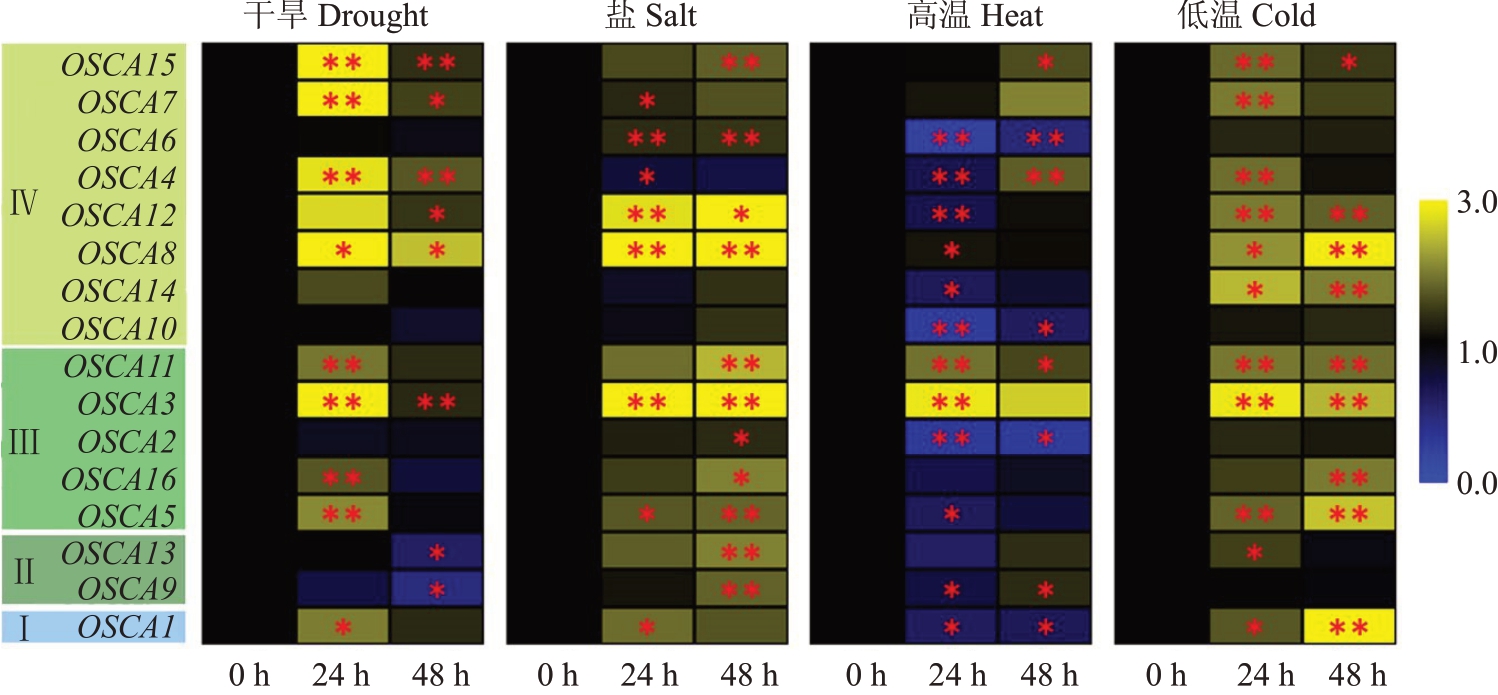
图6 猕猴桃OSCA 基因非生物胁迫条件下表达
Fig.6 Expression of OSCA genes under abiotic stress in kiwifruit
“*”和“**”分别代表差异达到显著(p ≤0.05)和极显著(p ≤0.01)水平。
“*”and“**”represent significant differences at p ≤0.05 and at p ≤0.01 levels,respectively.
在所有猕猴桃OSCA 基因中,与对照相比,AdOSCA3 在干旱、盐、高温和低温胁迫处理下表达上调均非常明显;AdOSCA8 在干旱、盐和低温胁迫下表达上调较明显;AdOSCA1 和AdOSCA14 在低温胁迫处理下表达上调较明显;AdOSCA7 和AdOS-CA15 在干旱胁迫处理下表达上调较明显(图6)。因此,这些基因的表达与相关非生物胁迫密切相关,可能在猕猴桃对干旱、盐、高温或低温胁迫的应答过程中发挥重要作用。
3 讨 论
3.1 AcOSCA基因家族结构与系统进化
OSCA 基因家族在不同植物中广泛存在,系统发育分析表明,植物OSCA 家族蛋白在进化中分为4个主要分支[7],目前已在拟南芥、番茄、大豆、水稻、小麦、黄瓜和辣椒等植物中分别鉴定到多个OSCA基因成员[11-13,16,22-25]。笔者在本研究中首次在猕猴桃基因组中鉴定出16 个OSCA 家族成员,其与拟南芥OSCA基因家族成员数量相近,均含有DUF221结构域,其蛋白成员同样可分为4 个分支。对16 个AcOSCA 基因进行编码蛋白亚细胞定位、基因结构分析、启动子区域顺式作用元件和系统进化关系进行的分析表明,AcOSCA 家族蛋白主要定位于细胞膜和(或)叶绿体,说明该家族主要在细胞膜或叶绿体中发挥作用。顺式作用元件分析发现,多数AcOSCA基因启动子区域含有干旱、激素、防御和应激反应等相关元件,这与番茄中的研究结果相似[26]。与单子叶植物水稻相比,猕猴桃OSCA 蛋白家族成员与双子叶植物番茄和拟南芥进化关系更近,对双子叶植物辣椒的研究也发现类似的结果[25]。四个分支中,Group Ⅲ与Group Ⅳ的基因成员内含子数量相近,Group Ⅱ中的成员内含子数量较少,Group Ⅰ中的成员无内含子。猕猴桃Group Ⅰ中仅含有一个成员,即AcOSCA1,结合顺式作用元件分析发现,启动子区含有较多MBS、ABRE、LTR 元件,它们与干旱、脱落酸和低温诱导相关。因此,推测该基因在猕猴桃逆境胁迫响应中发挥重要作用。猕猴桃OSCA蛋白家族每个分支的成员都包含共有的保守基序,且不同分支间保守基序分布的位置相似,表明其蛋白质结构相对保守。
3.2 OSCA基因家族成员与非生物胁迫的表达关系
近年来,OSCA 基因家族除在梨[17]中有报道外,在猕猴桃等其他果树中尚未进行研究。笔者在本研究中利用qRT-PCR技术检测OSCA家族基因在不同非生物胁迫(干旱、盐、低温、高温)处理下的相对表达量,结果发现,与对照相比,AdOSCA1、AdOSCA3、AdOSCA4、AdOSCA5、AdOSCA7、AdOSCA8、AdOSCA9、AdOSCA11、AdOSCA12、AdOSCA13、AdOSCA14、AdOSCA15 和AdOSCA16 在相应一种或多种非生物胁迫处理下表达量均上调。其中,AdOSCA3和AdOSCA11 在4 种非生物胁迫下均显著上调。基因转录水平受其上游启动子序列中顺式作用元件与相关转录因子相互作用调控[27],结合启动子顺式作用元件分析有助于预测相关基因的功能。本研究发现,MBS、TGACG-motif/CGTCA-motif、ABRE、LTR和ARE这些与干旱、茉莉酸甲酯、脱落酸、低温及厌氧诱导相关的顺式作用元件在OSCA3 基因上游启动子区域均有分布。其中,与其他基因相比,AdOSCA8 基因对干旱和盐两种胁迫处理的响应最强烈,因此,AdOSCA8在响应干旱和盐胁迫过程中可能发挥十分重要的作用,推测AdOSCA8对干旱和盐胁迫的响应通路可能存在一定的交互性。拟南芥OSCA1.1(At4g04340)被鉴定为植株渗透感知通路的重要基因,它编码的蛋白定位于叶和根部细胞的细胞膜上,扮演离子通道角色,可使Ca2+在干旱条件下大量涌入细胞[1]。与OSCA1.1 高度同源的OSCA1.2(At4g22120),是调控Ca2+渗透的阳离子通道,该蛋白可在高渗胁迫下增加细胞内Ca2+浓度,且对K+和Na+具有一定的渗透性[15]。笔者在本研究中发现,猕猴桃OSCA8 与拟南芥OSCA1.1 和OSCA1.2 序列相似度较高,进化上均属于Group Ⅳ分支,且均能响应干旱胁迫,因此,OSCA8 与拟南芥OSCA1.1 和OSCA1.2 可能具有相似的功能。前人研究发现,OSCA1.1 和OSCA1.2 在拟南芥中对渗透压有响应,可被通过膜传递的机械力激活并可作为一种固有的机械敏感性成孔离子通道[15,28-29]。推测猕猴桃OSCA8基因可能对渗透压较敏感。
笔者在本研究中发现,部分OSCA 基因仅响应一种胁迫,例如,OSCA1 与OSCA14、OSCA7 与OSCA15 仅在低温或干旱胁迫处理下表达量发生显著变化,而在其他胁迫处理下表达量未有明显变化。在PEG 模拟的干旱胁迫处理下,番茄SlOSCA7(Solyc06g084330)在4 h上调至对照的80倍[26],与之相似,猕猴桃OSCA7 和OSCA15 与SlOSCA7 在系统进化树中属于同一分支中进化关系相近的基因,且均在干旱胁迫处理下表达量显著上调,说明具有相似结构的OSCA7、OSCA15 和SlOSCA7,可能具有相似的功能,在干旱胁迫的应激响应中发挥重要作用。SlOSCA9(Solyc08g023440)在盐、干旱胁迫下表达差异明显[26],相似地,与SlOSCA9属于同一分支的同源猕猴桃基因OSCA3,在盐、干旱胁迫处理下表达受到显著诱导,推测OSCA3 在猕猴桃应对盐、干旱胁迫响应中发挥重要作用。
4 结 论
笔者在本研究中首次鉴定并分析了猕猴桃OSCA基因家族成员,对OSCA家族基因在不同非生物胁迫处理下的表达特征进行了分析。研究结果表明,猕猴桃AdOSCA1、AdOSCA3、AdOSCA4、AdOSCA5、AdOSCA7、AdOSCA8、AdOSCA9、AdOSCA11、AdOSCA12、AdOSCA13、AdOSCA14、AdOSCA15 和AdOSCA16在相应的一种或多种非生物胁迫处理下表达量显著上调,其中OSCA3和OSCA11在4种不同的非生物胁迫下表达量均显著上调。后期可对相关关键候选基因进行克隆和功能鉴定,深入解析这些OSCA基因在猕猴桃应对非生物胁迫过程中的功能。
[1] ZHANG H,ZHAO Y,ZHU J K. Thriving under stress:How plants balance growth and the stress response[J]. Developmental Cell,2020,55(5):529-543.
[2] VERMA V,RAVINDRAN P,KUMAR P P. Plant hormone-mediated regulation of stress responses[J]. BMC Plant Biology,2016,16:86.
[3] ISAH T.Stress and defense responses in plant secondary metabolites production[J].Biological Research,2019,52(1):39.
[4] WANI S H,ANAND S,SINGH B,BOHRA A,JOSHI R.WRKY transcription factors and plant defense responses:Latest discoveries and future prospects[J]. Plant Cell Reports,2021,40(7):1071-1085.
[5] ZHU J K.Salt and drought stress signal transduction in plants[J].Annual Review of Plant Biology,2002,53:247-273.
[6] BATISTIČ O,KUDLA J. Analysis of calcium signaling pathways in plants[J]. Biochimica et Biophysica Acta-General Subjects,2012,1820(8):1283-1293.
[7] 柴利芳.植物OSCA 家族的起源与进化[D].临汾:山西师范大学,2019.CHAI Lifang.Origin and evolution of OSCA family in plants[D].Linfen:Shanxi Normal University,2019.
[8] PERMYAKOV E A,KRETSINGER R H. Cell signaling,beyond cytosolic calcium in eukaryotes[J]. Journal of Inorganic Biochemistry,2009,103(1):77-86.
[9] KUDLA J,BATISTIČ O,HASHIMOTO K. Calcium signals:the lead currency of plant information processing[J]. The Plant Cell,2010,22(3):541-563.
[10] BERRIDGE M J,LIPP P,BOOTMAN M D.The versatility and universality of calcium signalling[J]. Nature Reviews Molecular Cell Biology,2000,1:11-21.
[11] LI Y S,YUAN F,WEN Z H,LI Y H,WANG F,ZHU T,ZHUO W Q,JIN X,WANG Y D,ZHAO H P,PEI Z M,HAN S C.Genome-wide survey and expression analysis of the OSCA gene family in rice[J].BMC Plant Biology,2015,15:261.
[12] MIAO S,LI F S,HAN Y,YAO Z T,XU Z Q,CHEN X L,LIU J Y,ZHANG Y,WANG A X. Identification of OSCA gene family in Solanum habrochaites and its function analysis under stress[J].BMC Genomics,2022,23(1):547.
[13] YUAN F,YANG H M,XUE Y,KONG D D,YE R,LI C J,ZHANG J Y,THEPRUNGSIRIKUL L,SHRIFT T,KRICHILSKY B,JOHNSON D M,SWIFT G B,HE Y K,SIEDOW J N,PEI Z M. OSCA1 mediates osmotic-stress-evoked Ca2+ increases vital for osmosensing in Arabidopsis[J]. Nature,2014,514:367-371.
[14] 张红娟,朱德鹤,杜琳颖,毛虎德.小麦OSCA 基因家族全基因组鉴定及表达分析[J].西北农林科技大学学报(自然科学版),2022,50(12):25-33.ZHANG Hongjuan,ZHU Dehe,DU Linying,MAO Hude. Genome-wide identification and expression analysis of the OSCA gene family in wheat[J]. Journal of Northwest A& F University(Natural Science Edition),2022,50(12):25-33.
[15] HOU C C,TIAN W,KLEIST T,HE K,GARCIA V,BAI F L,HAO Y L,LUAN S,LI L G. DUF221 proteins are a family of osmosensitive calcium- permeable cation channels conserved across eukaryotes[J].Cell Research,2014,24(5):632-635.
[16] LIU C G,WANG H,ZHANG Y,CHENG H J,HU Z L,PEI Z M,LI Q. Systematic characterization of the OSCA family members in soybean and validation of their functions in osmotic stress[J]. International Journal of Molecular Sciences,2022,23(18):10570.
[17] 顾小雨. 梨OSCA 基因家族分析及PbrOSCA2.6 和PbrOSCA3.2 的功能验证[D].南京:南京农业大学,2017.GU Xiaoyu. OSCA family analysis and functional validation of PbrOSCA2.6 and PbrOSCA3.2 in Pyrus[D]. Nanjing:Nanjing Agricultural University,2017.
[18] LI Y Y,ZHANG Y B,LI B,HOU L Y,YU J N,JIA C G,WANG Z,CHEN S Q,ZHANG M Z,QIN J C,CAO N,CUI J H,SHI W L. Preliminary expression analysis of the OSCA gene family in maize and their involvement in temperature stress[J]. International Journal of Molecular Sciences,2022,23(21):13658.
[19] 徐小彪,张秋明.中国猕猴桃种质资源的研究与利用[J].植物学通报,2003,20(6):648-655.XU Xiaobiao,ZHANG Qiuming. Researches and utilizations of germplasm resource of kiwifruit in China[J]. Chinese Bulletin of Botany,2003,20(6):648-655.
[20] JIA D F,YI S Y,HUANG Q Q,LIU Q,HE Y Q,LIAO G L,HUANG C H,XU X B. Comprehensive analysis of a red-peel kiwi berry mutant reveals key genes are responsible for anthocyanin biosynthesis in fruit[J]. Scientia Horticulturae,2023,309:111682.
[21] VANDESOMPELE J,DE PRETER K,PATTYN F,POPPE B,VAN ROY N,DE PAEPE A,SPELEMAN F.Accurate normalization of real-time quantitative RT-PCR data by geometric averaging of multiple internal control genes[J]. Genome Biology,2002,3(7):RESEARCH0034.
[22] LI M,WU Z Y,GU H,CHENG D W,GUO X Z,LI L,SHI C Y,XU G Y,GU S C,ABID M,ZHONG Y P,QI X J,CHEN J Y.AvNAC030,a NAC domain transcription factor,enhances salt stress tolerance in kiwifruit[J]. International Journal of Molecular Sciences,2021,22(21):11897.
[23] TONG K,WU X Y,HE L,QIU S Y,LIU S,CAI L N,RAO S F,CHEN J P. Genome-wide identification and expression profile of OSCA gene family members in Triticum aestivum L.[J].International Journal of Molecular Sciences,2021,23(1):469.
[24] YANG S T,ZHU C X,CHEN J J,ZHAO J D,HU Z Y,LIU S Q,ZHOU Y.Identification and expression profile analysis of the OSCA gene family related to abiotic and biotic stress response in cucumber[J].Biology,2022,11(8):1134.
[25] 李嘉琪,罗石磊,张帅磊,张文渊,张国斌.辣椒OSCA 基因家族的全基因组鉴定及不同胁迫条件下表达分析[J].植物科学学报,2022,40(2):187-196.LI Jiaqi,LUO Shilei,ZHANG Shuailei,ZHANG Wenyuan,ZHANG Guobin. Genome-wide identification of pepper OSCA gene family and expression analysis under different stress conditions[J].Plant Science Journal,2022,40(2):187-196.
[26] 王傲雪,张可为,张瑶,陈秀玲,刘佳音.番茄OSCA 基因家族鉴定及不同胁迫条件下表达分析[J]. 东北农业大学学报,2019,50(1):19-28.WANG Aoxue,ZHANG Kewei,ZHANG Yao,CHEN Xiuling,LIU Jiayin. Identification of tomato OSCA gene family and expression analysis under different stress conditions[J]. Journal of Northeast Agricultural University,2019,50(1):19-28.
[27] QIU J F,NI L,XIA X,CHEN S H,ZHANG Y,LANG M,LI M Y,LIU B M,PAN Y,LI J H,ZHANG X G.Genome-wide analysis of the protein phosphatase 2C genes in tomato[J]. Genes,2022,13(4):604.
[28] JOJOA-CRUZ S,SAOTOME K,MURTHY S E,TSUI C C A,SANSOM M S,PATAPOUTIAN A,WARD A B. Cryo-EM structure of the mechanically activated ion channel OSCA1.2[J].ELife,2018,7:e41845.
[29] ZHANG M F,WANG D L,KANG Y L,WU J X,YAO F Q,PAN C F,YAN Z Q,SONG C,CHEN L.Structure of the mechanosensitive OSCA channels[J]. Nature Structural & Molecular Biology,2018,25(9):850-858.