梨为蔷薇科(Rosaceae)梨属(Pyrus L.)植物,是我国乃至世界范围内的重要温带果树之一,距今已有3000余年的栽培历史[1]。一般认为梨的原种起源于第三纪甚至更古老的中国西部或者西南部的山区,经过亚欧大陆传播到中亚地区,最后到达亚洲西部和欧洲[2]。根据出身、原产地,梨可分为两大类:东方梨(Oriental pears)和西方梨(Occidental pears)。目前发现的梨属植物至少有30 个种,其中原生种(基本种)大约有20个。在中国,梨产区遍布东西南北中,梨的种质资源极为丰富,可以说,我国是梨属植物的起源中心,也是遗传多样性中心,形成了中国豆梨、柯汉梨或台湾豆梨、川梨、砂梨和秋子梨等基本种[3],还有各种各样的品种资源和地方野生资源,它们形状各异,颜色多样,早、中、晚熟皆有,风味各具特色[4]。中国南疆广西壮族自治区地处亚热带和热带气候区,水、光、热资源充足,生态环境复杂多变,孕育了许多具有特色的梨种质资源,比如砂梨中的优质品种灌阳雪梨,被评为全国农产品地理标志。据统计,2022 年广西梨栽培面积2.1 万hm2,产量50.78万t,是广西重要的水果产业之一。然而,广西梨产业存在品种竞争力不强等突出问题,加快品种改良和种质资源创新利用是有效的解决方法之一。因此,对广西地方梨种质资源进行系统鉴定、遗传多样性和种群结构分析具有重要意义。
种质鉴定和遗传多样性研究是品种改良和种质资源创新的基础。在植物研究中,传统的做法是主要依据形态学、细胞学、酶学等方法来鉴定种质,但实际中仍存在许多困难,而且可靠性不高。20世纪70年代以来,以个体间核苷酸序列变异为基础的分子标记技术得到了快速发展和应用,极大地推动了植物种质鉴定和遗传多样性研究,常见的分子标记有:限制性片段长度多态性标记(RFLP)、随机扩增多态性标记(RAPD)、简单重复序列(SSR)、扩增限制性内切酶片段长度多态性(AFLP)和单核苷酸多态性(SNP)。其中,SSR 分子标记符合孟德尔共显性遗传,具有数量丰富、操作技术简便、可靠性强和重复性好等优点[5],在评价植物遗传多样性、构建遗传连锁图谱、揭示进化历史和分子辅助育种等方面得到了广泛应用。21 世纪以来,SSR 分子标记技术在梨品种鉴定、遗传多样性研究和新品种选育等方面已有许多成功先例。日本学者Yamamoto 等[6]最早借鉴苹果上建立的SSR 分子标记,应用于梨的多态性和遗传多样性鉴定并最终取得成功。在中国,Bao 等[7]利用6 对SSR 引物对东亚主要原生栽培种的遗传多样性和亲缘关系进行研究,结果表明,供试材料分为10个类群。其中,所有中国砂梨被划分到4个类群,具有丰富的遗传多样性,中国白梨被划分到3 个类群,日本梨仅存在于1 个类群中,中国砂梨和中国白梨没有形成离散群甚至亚群,此外,一些日本梨栽培种与中国砂梨栽培种的关系十分相近。Xue 等[8]从筛选出的332 个多态SSR 标记中随机选取18个多态SSR标记,对44个梨品种进行遗传多样性分析,为鉴定梨品种的族谱基因型、系谱推断提供了重要参考。
新中国成立以来,中国学者在梨种质资源、品质发育及遗传育种研究领域取得了丰硕成果,特别是近年来对梨的起源进化、遗传多样性和亲缘关系的研究不断深入,梨的“前世今生”被逐渐揭开神秘面纱。限于多种因素,广西地方梨种质资源的鉴定、遗传多样性和群体结构的研究还比较薄弱,加上各地区之间的苗木繁育和种质交流进程加快,导致品种混杂、关系不清、同物异名等问题突出,极不利于广西梨种质资源的保护和创新利用。笔者在本研究中以119 份梨种质作为试验材料,利用15 个多态性良好的SSR 标记,分析广西地方梨种质资源的遗传多样性和群体结构,及其与外地种质之间的亲缘关系和遗传差异,旨在为广西地方梨种质资源的保护和创新利用以及现代梨育种工作提供依据。
1 材料和方法
1.1 试验材料
119 份供试梨种质采自自治区级农作物种质资源圃—广西梨种质资源圃(桂林),其中原产于广西的种质有71份,外地种质48份。详细信息见表1。
表1 供试梨种质来源
Table 1 Pear germplasm used for analysis

?
表1 (续) Table 1 (Continued)

?
1.2 基因组DNA提取
每份梨种质材料取嫩叶(鲜质量20~50 mg)液氮冷冻后研磨,采用磁珠法基因组提取试剂盒(武汉纳磁生物科技有限公司)提取样本DNA。获得的DNA 经1%琼脂糖凝胶电泳检测合格后,用Nano-DROPTM 8000超微量分光光度计检测纯度和浓度。
1.3 SSR-PCR
根据相关文献中的58对SSR引物信息[9-12],委托武汉天一华煜基因科技有限公司合成,用8 个样本进行引物筛选验证,筛选出15 对扩增成功、峰型良好的引物(表2)。
表2 15 对引物信息
Table 2 The information of 15 pairs of SSR primers

?
PCR反应体系为10 μL,包括:2×Taq Master Mix(Mg2+)5.0 μL,浓度为10 pmol·μL-1 的正反向引物各0.5 μL,20 ng·μL-1 的DNA 模板1 μL,ddH2O 3.0 μL。反应程序为:95 ℃5 min;95 ℃30 s,62~52 ℃30 s,72 ℃30 s,10个循环,每个循环下降1 ℃;95 ℃30 s,52 ℃30 s,72 ℃30 s,25个循环;72 ℃20 min延伸后4 ℃保存。取PCR产物1.0 μL,分子质量内标和甲酰胺混合液(0.5∶8.5,v/v)9.0 μL;95 ℃变性3 min,在ABI 3730XL测序仪上进行分型检测,读取扩增结果。
1.4 数据处理与统计
将从ABI 3730XL测序仪上得到的结果在GeneMarker 软件上进行分析,导出Excel 基因型数据。用GenAlEx version 6.501软件计算观测等位基因数(Na)、有效等位基因数(Ne)、香农指数(I)、多态性信息指数(PIC)、观测杂合度(Ho)、期望杂合度(He)和近交系数(Fis)。用Powermarker软件计算各群体间的遗传距离。用UMPGA 方法进行聚类分析,绘制聚类图。利用STRUCTURE 2.3.4 进行居群遗传结构分析,设置K=1~20,Burn-in周期为10000,MCMC 设为100 000,每个K 值运行20 次,并利用在线工具STRUCTURE HARVESTER 算出最佳△K 值。根据相应的K 值作图。结构分析的结果图用CLUMMP 和DISTRUCT 软件绘制。根据群体遗传结构分析结果,在GenAlEx version 6.501 软件中计算遗传分化系数(Fst)和基因流(Nm)。用GenAIex软件进行主坐标分析。
2 结果与分析
2.1 引物的多态性
利用荧光毛细管电泳技术,从58对引物中筛选出15对峰型良好、特异性较高的引物。由表3可知,15 对引物在119个样本中共检测出190 个等位基因(Na),最少的5 个,最多的19 个,平均为12.667 个。有效等位基因(Ne)数为2.772~9.743,平均为5.454。香农指数(I)为1.277~2.456,平均为1.876。观测杂合度(Ho)为0.437~0.897,平均为0.682。期望杂合度(He)为0.639~0.897,平均为0.762;多态性信息指数(PIC)为0.602~0.889,平均为0.762。这些指标均表明15对SSR引物多态性较高[13]。
表3 15 对SSR 引物的多态性
Table 3 Polymorphism of 15 pairs of SSR primers

注:Na.观测等位基因;Ne.有效等位基因;I.香农指数;Ho.观测杂合度;He.期望杂合度;PIC.多态性信息指数;Fis.近交系数;Fst.遗传分化系数;Nm.基因流。
Note: Na. Observed allele; Ne. Effective allele; 1. Shannon index; Ho. Observed heterozygosity; He. Expected heterozygosity; PIC. Polymorphic information index;Fis.Inbreeding coefficient;Fst.Genetic differentiation coefficient;Nm.Gene flow.
?
2.2 聚类分析
基于遗传距离进行聚类,全部材料明显分为两大类群(图1),第Ⅰ类群包含23 份种质,其中22 份原产于广西,仅1 份为外地种质(横山梨,中国台湾);第Ⅱ类群包含96份种质,其中广西种质49份,外地种质47 份。第Ⅱ类群可进一步划分为两个亚群,亚群1由38份广西种质和1份外地种质(云和粗花雪梨,产地:浙江云和)组成,亚群2由11份广西种质(灌阳2号、龙胜2号、灌阳清香梨、六月梨、富川蜜梨、铜板梨、全州蜜梨、龙胜糖梨、龙胜1 号、灌阳黄蜜、南山梨)和46份外地种质组成,说明这11份广西种质与外地种质的亲缘关系较近。以上结果表明广西种质可大致划分为3种类型;总体上,广西种质和外地种质既形成了相对独立的类群,又存在少量的相互交叉,大部分广西种质与外地种质的亲缘关系较远。

图1 基于Nei 遗传距离的UPGMA 聚类分析图
Fig.1 UPGMA clustering analysis chart based on Nei genetic distance
广西种质用黑色字体标注,外地种质用蓝色字体标注,图中数字为表1 中的材料序号。下同。
Guangxi germplasm are marked in black font and nonlocal germplasm are marked in blue font,figures in the figure represent the material numbers in Table 1.The same below.
2.3 群体遗传结构分析
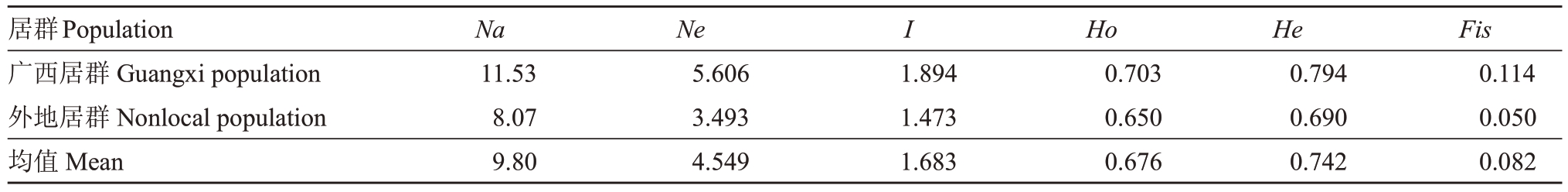
由表4 可知,广西居群的平均等位基因数(Na)为11.53,平均有效等位基因数(Ne)为5.606,平均香农指数(I)为1.894,均高于外地种质,说明广西地方种质的遗传多样性更为丰富;2 个居群的观测杂合度(Ho)均低于期望杂合度(He),Fis>0,说明两个居群内均发生近缘交配。
表4 2 个居群的遗传多样性
Table 4 Genetic diversity of 2 pear populations
?
利用Structure 构建119 份梨种质的群体遗传结构图,结果显示当K=2时,ΔK为最大值,供试材料可分为2个不同群体。由图2可知,群体1由45份外地梨(占比81.81%)和10 份广西梨(占比18.18%)构成,这10 份广西梨为富川蜜梨、南山梨、灌阳黄蜜、六月梨、桂花梨、龙胜糖梨、铜板梨、龙胜1号、灌阳2号、全州蜜梨;群体2 主要由61 份广西梨(占比93.75%)构成,中国台湾的横山梨、浙江的云和粗花雪梨以及湖北选育的鄂梨2号也划归到群体2中,表明广西梨和外地梨在遗传结构上有较大差别,与聚类分析结果对应。

图2 119 份梨种质在 K=2、K=3 和 K=4 的遗传结构
Fig. 2 The genetic structure of 119 accessions with K of 2 to 4
当K=3 时,供试材料被划分为3个群体,原本归属群体2的19份广西梨(灌阳小把子雪梨、灌阳大把子雪梨、米珠山假雪梨、资源1号、黄皮梨、兴安梨、清水梨、早禾梨、龙胜大砂梨、灌阳清香梨、牛卵梨、资源3号、荔浦黄皮糖梨、岑溪墨烟梨、岑溪沙梨、永福青皮梨、永福椭圆形早禾梨、永福圆形早禾梨、平乐糖梨)以及外地的鄂梨2号被重新划分到群体3中。
当K=4时,供试材料被划分为4个群体,原本归属群体3的4份广西梨(资源1号、资源3号、兴安梨、龙胜大砂梨)和原本归属群体1 的4 份外地梨(六月酥、红早酥、新梨7号、中梨4号)被重新划分到群体4 中,说明广西的资源1 号、资源3 号、兴安梨、龙胜大砂梨和外地的六月酥、红早酥、新梨7号、中梨4号有更多相同的遗传组成。有意思的是,六月酥、红早酥、新梨7号、中梨4号都具有早酥的遗传背景,其中六月酥和红早酥为早酥的芽变品种,新梨7 号和中梨4号分别是以早酥为亲本之一的二元交杂品种和三元杂交品种。由此推测广西的资源1 号、资源3号、兴安梨、龙胜大砂梨与早酥有共同的遗传来源。早酥是中国农业科学院果树研究所利用苹果梨和身不知梨育成的早熟新品种,其母本苹果梨,属于白梨系统,是中国优良梨品种之一,主产于吉林省延边朝鲜族自治州,中国各地的苹果梨都发源于此,其父本身不知梨,原产日本,亲本不详,被认为是西洋梨与砂梨的自然杂交后代。
为深入了解广西地方梨种质的遗传结构,对其进行独立的群体遗传结构分析。71 份种质在K=4时ΔK取得最大值,可划分为4个群体(图3),群体1包含37 份材料,其地理来源有桂林、梧州、百色、柳州、贺州、钦州;群体2包含10份材料,其地理来源有桂林、梧州、来宾、河池、南宁;群体3包含10份材料,其地理来源有桂林、梧州;群体4 包含14 份材料,其地理来源有桂林、梧州、柳州。

图3 71 份广西地方梨种质在K=4 的遗传结构
Fig.3 The genetic structure of 71 local pear germplasm at K=4
2.4 主坐标分析
利用GenAIex 软件对供试材料进行主坐标分析(PCoA),绘制出二维主坐标散点图(图4),从图中可以看出,在主坐标1 方向上,整个群体清晰地分为2 个组群,广西材料和外地材料分别聚集成簇,同时也存在少量的相互渗透,说明二者之间亲缘关系较远,但有一定的基因交流。另外,部分材料的坐标位置几乎重叠,比如鄂梨2 号和灌阳清香(广西灌阳);岑溪烟墨梨(广西岑溪)、岑溪沙梨(广西岑溪)、牛卵梨(广西岑溪)和永福椭圆形早禾梨(广西永福);灌阳大把子雪梨(广西灌阳)、灌阳小把子雪梨(广西灌阳)和黄皮梨(广西乐业),与聚类树结果相吻合,证明它们的亲缘关系很近。结合地理位置来看,广西材料在图中的分布未表现明显的地域性特征。

图4 基于15 个SSR 标记的119 份梨材料的主坐标分析
Fig.4 Principal coordinates analysis plots for 119 pear accessions based on 15 SSR markers
2.5 分子方差分析
AMOVA 方差分析(表5)表明,119 份梨种质的遗传变异,8%存在于群体间,10%存在于个体间,82%存在于个体内,个体内差异是指由杂合的等位基因引起的遗传差异,大小与个体杂合位点数相关,即个体的遗传多样性,说明个体内各基因之间存在高度遗传分化,个体的遗传变异是梨变异的主要来源。
表5 梨群体分子方差分析表
Table 5 Molecular variance analysis of pear population

?
3 讨 论
梨属植物间不存在生殖隔离,种间杂交十分普遍,使得梨属种间有时缺乏明显可以区分的形态学性状,导致梨属植物的分类比较混乱[3]。SSR分子标记技术的应用,可以较好地弥补、修正和完善梨传统分类中的一些不足。引物的多态性是评估种质多样性的重要因素,笔者在本研究中使用的15个SSR标记的PIC全部大于0.50,多态性较高,可以较好地反映梨种质资源的遗传多样性和亲缘关系[14]。I和Ho是衡量种质资源遗传多样性的重要指标[15]。在本研究中,广西地方梨种质的I 和Ho 均高于外地种质,反映出广西地方梨种质较高的遗传多样性。相关研究认为梨起源于中国西南部的山区[2],北上向东移动,形成了东亚种群,中国白梨、中国砂梨和日本梨可能起源于共同的野生砂梨祖先[16]。广西可能是梨的起源地之一或早期传播地区,在长期的环境适应下逐渐形成了不同遗传信息的群体。基于Nei’s的遗传距离构建的聚类树中,广西梨被划分为3 个聚群,而外地梨主要归于1个聚群,同时二者又有一定的相互交叉,表明存在基因交流。笔者在本研究中关注的广西梨种质基本为砂梨类型,外地种质主要为经人工驯化选择或杂交获得的栽培梨,与前者相比,后者具有更多地从早期适应发生时积累的多种遗传变异,因此在聚类关系上表现为二者的遗传距离较远。值得注意的是,中国台湾的横山梨以及浙江云和的云和粗花梨并没有与其他外地种质聚在一类,而是与大部分广西梨种质聚在一起,并且在群体结构分析和PCoA 分析中也获得相似的结果,表明横山梨和云和粗花梨与广西梨有较近的亲缘关系,很可能是从广西引种到台湾和浙江的。另据文献记载,中国台湾梨系1890年由我国香港和华南一带引入种植于台湾汉溪、横山一带,主要为横山梨[17],在一定程度上印证了本文的结果。广西的灌阳2 号、龙胜2 号、灌阳清香梨、六月梨、富川蜜梨、铜板梨、全州蜜梨、龙胜糖梨、龙胜1号、灌阳黄蜜、南山梨与外地种质聚在一起,同时群体结构分析和PCoA 分析也呈现相似的分组结果,提示这些材料可能是由外地梨种质杂交获得的或引种到广西而被误认为是广西地方种。
梨是典型的自交不亲和性物种,后代的繁殖依靠异花授粉[18],这种授粉特性增加了居群间的杂交概率,因此具有较高的杂合特性。在本研究中,广西居群和外地居群的观测杂合度(Ho)均低于期望杂合度(He),且Fis>0,居群内发生近缘交配。近缘交配是一种非随机交配,如果一个群体持续进行近缘交配,那么该群体中的杂合子会快速丢失[19]。在主坐标分析图中,广西梨种质没有很明显地被分为多个类群,也反映出广西梨品种在形成过程中,或许杂交较为普遍,导致许多供试地方梨种质资源遗传组成比较复杂。本研究中的119 份梨的遗传变异8%存在于群体间,10%存在于个体间,82%存在于个体内,说明梨居群间的遗传分化程度较低,遗传变异主要来源于居群内的个体。多个内外因素可影响居群遗传结构,其中基因流、突变和演替阶段等会显著影响居群遗传结构[19]。当基因流Nm>1 时,说明居群间的基因流较大,居群间遗传分化程度较低;当Nm<1时,说明居群间的基因流较小,居群间遗传分化程度较高[20]。在本研究中,居群间的Nm=5.717(Nm>1),基因流主要发挥均质化作用,较大的基因流有效地阻止了遗传漂变,降低了梨居群间的遗传分化程度,频繁的基因流使得梨种群间的遗传分化程度低,因此变异主要存在于居群个体内。
梨育种的核心基础就是收集足够多具有遗传多样性的种质资源。种质资源是品种改良的基础,尤其是具有广泛遗传基础的品种资源[21]。胡春云[22]认为我国西南地区拥有最丰富的原始品种资源,是我国砂梨品种的起源中心。笔者在本研究中收集的71份广西地方梨种质遗传多样性丰富,品种间遗传距离大,涵盖了广西主要梨生产区,具有一定代表性,群体遗传结构分析将其分为4个群体,但不同群体未表现明显的区域分化,下一步可采集一些广西邻省如云南、贵州等地原产的梨种质资源,以便加深对广西梨种质的来源及传播的了解。此外,利用分子标记结合数量性状指标可以更全面地反映种质资源间的差异,张莹等[23]对梨种质资源果实若干数量性状评价指标研究的结果表明,果实中可滴定酸含量的变异系数最大,更能体现梨品种间的差异。因此,后续可进一步研究广西梨种质资源的数量性状指标,以便对其遗传多样性进行综合评价。随着日益加快的城市化进程,地方品种会加速流失[24]。而地方品种中可能含有许多有价值的基因,如特殊性状基因、抗病虫基因、抗逆基因等,这些基因在以选择少数理想性状为目标的栽培品种中可能已经丢失[25]。因此,笔者建议各方要重视地方种质资源保护,在有条件的情况下,实行就地保护或建立种质资源圃进行迁地保护,避免一些特殊种质的永久性流失。
4 结 论
广西地方梨种质具有较丰富的遗传多样性,与供试的外地种质形成2 个不同的遗传群体,亲缘关系较远,但二者存在一定的基因交流,此外,两个居群内普遍存在近缘交配,变异的主要来源为居群内个体的遗传变异。群体遗传结构分析表明广西地方种质可划分为4 个群体,但不同群体未呈现明显的区域分化规律,后续可结合不同的个体性状进行深入探究。值得注意的是,中国台湾的横山梨以及浙江的云和粗花梨或许原产于广西,一部分广西本地梨也可能是外地梨种质杂交获得或引种到广西,有待进一步印证。
[1] LI J M,ZHANG M Y,LI X L,KHAN A,KUMAR S,ALLAN A C,WANG K L,ESPLEY R V,WANG C H,WANG R Z,XUE C,YAO G F,QIN M F,SUN M Y,TEGTMEIER R,LIU H N,WEI W L,MING M L,ZHANG S L,ZHAO K J,SONG B B,NI J P,AN J P,KORBAN S S,WU J. Pear genetics:Recent advances,new prospects,and a roadmap for the future[J].Horticulture Research,2022,9:uhab040.
[2] RUBTSOV G A. Geographical distribution of the genus Pyrus and trends and factors in its evolution[J].The American Naturalist,1944,78(777):358-366.
[3] 滕元文.梨属植物系统发育及东方梨品种起源研究进展[J].果树学报,2017,34(3):370-378.TENG Yuanwen.Advances in the research on phylogeny of the genus Pyrus and the origin of pear cultivars native to East Asia[J].Journal of Fruit Science,2017,34(3):370-378.
[4] 陈学森,王楠,张宗营,冯守千,陈晓流,毛志泉.仁果类果树资源育种研究进展Ⅰ:我国梨种质资源、品质发育及遗传育种研究进展[J].植物遗传资源学报,2019,20(4):791-800.CHEN Xuesen,WANG Nan,ZHANG Zongying,FENG Shouqian,CHEN Xiaoliu,MAO Zhiquan. Progress on the resource and breeding of kernel fruits Ⅰ:Progress on the germplasm resources,quality development and genetics and breeding of pear in China[J].Journal of Plant Genetic Resources,2019,20(4):791-800.
[5] GUICHOUX E,LAGACHE L,WAGNER S,CHAUMEIL P,LÉGER P,LEPAIS O,LEPOITTEVIN C,MALAUSA T,REVARDEL E,SALIN F,PETIT R J.Current trends in microsatellite genotyping[J]. Molecular Ecology Resources,2011,11(4):591-611.
[6] YAMAMOTO T,KIMURA T,SAWAMURA Y,KOTOBUKI K,BAN Y,HAYASHI T,MATSUTA N.SSRs isolated from apple can identify polymorphism and genetic diversity in pear[J].Theoretical and Applied Genetics,2001,102(6):865-870.
[7] BAO L,CHEN K S,ZHANG D,CAO Y F,YAMAMOTO T,TENG Y W. Genetic diversity and similarity of pear (Pyrus L.)cultivars native to East Asia revealed by SSR (simple sequence repeat) markers[J]. Genetic Resources and Crop Evolution,2007,54(5):959-971.
[8] XUE H B,ZHANG P J,SHI T,YANG J,WANG L,WANG S K,SU Y L,ZHANG H R,QIAO Y S,LI X G. Genome-wide characterization of simple sequence repeats in Pyrus bretschneideri and their application in an analysis of genetic diversity in pear[J].BMC Genomics,2018,19(1):473.
[9] 蒋爽,骆军,王晓庆,施春晖.基于基因组重测序数据高效筛选梨SSR 标记多态性引物[J].果树学报,2019,36(2):129-136.JIANG Shuang,LUO Jun,WANG Xiaoqing,SHI Chunhui. A study on efficient screening of the primers for selecting polymorphic SSR markers based on the re-sequencing data in Pyrus[J].Journal of Fruit Science,2019,36(2):129-136.
[10] 王斐,张艳杰,欧春青,李佳纯,杨冠宇,马力,姜淑苓.梨品种SSR 分子鉴定体系的建立及应用[J].分子植物育种,2021,19(22):7499-7509.WANG Fei,ZHANG Yanjie,OU Chunqing,LI Jiachun,YANG Guanyu,MA Li,JIANG Shuling. SSR identification system of pear varieties and its application[J]. Molecular Plant Breeding,2021,19(22):7499-7509.
[11] 冉昆,隋静,王宏伟,魏树伟,张勇,董冉,董肖昌,王少敏.利用SSR 荧光标记构建山东地方梨种质资源分子身份证[J].果树学报,2018,35(增刊1):71-78.RAN Kun,SUI Jing,WANG Hongwei,WEI Shuwei,ZHANG Yong,DONG Ran,DONG Xiaochang,WANG Shaomin. Using the fluorescent labeled SSR markers to establish the molecular ID of pear germplasm resources in Shandong[J].Journal of Fruit Science,2018,35(Suppl.1):71-78.
[12] 薛华柏,赵瑞娟,王磊,杨健,王龙,王苏珂,苏艳丽,李秀根.梨品种SSR 特征指纹图谱与分子身份证构建[J]. 中国南方果树,2018,47(S1):42-49.XUE Huabai,ZHAO Ruijuan,WANG Lei,YANG Jian,WANG Long,WANG Suke,SU Yanli,LI Xiugen. Construction of SSR fingerprint and molecular identity card of pear varieties[J].South China Fruits,2018,47(S1):42-49.
[13] BOTSTEIN D,WHITE R L,SKOLNICK M,DAVIS R W.Construction of a genetic linkage map in man using restriction fragment length polymorphisms[J].American Journal of Human Genetics,1980,32(3):314-331.
[14] WANG S Q,LIU Y,MA L Y,LIU H B,TANG Y,WU L P,WANG Z,LI Y Y,WU R L,PANG X M. Isolation and characterization of microsatellite markers and analysis of genetic diversity in Chinese jujube (Ziziphus jujuba Mill.)[J]. PLoS One,2014,9(6):e99842.
[15] WANG H,PAN G,MA Q G,ZHANG J P,PEI D. The genetic diversity and introgression of Juglans regia and Juglans sigillata in Tibet as revealed by SSR markers[J].Tree Genetics & Genomes,2015,11:804.
[16] TENG Y W,TANABE K,TAMURA F,ITAI A. Genetic relationships of Pyrus species and cultivars native to East Asia revealed by randomly amplified polymorphic DNA markers[J].Journal of the American Society for Horticultural Science,2002,127(2):262-270.
[17] 林伯年. 世界及中国的梨生产与贸易[J]. 中国南方果树,2001,30(6):64-67.LIN Bonian.Pear production and trade in the world and China[J].South China Fruits,2001,30(6):64-67.
[18] 刘清文,宋跃,李甲明,张明月,齐开杰,张绍铃,吴俊.利用核心简单重复序列(SSR)标记分析西洋梨品种资源遗传多样性[J].农业生物技术学报,2015,23(5):579-587.LIU Qingwen,SONG Yue,LI Jiaming,ZHANG Mingyue,QI Kaijie,ZHANG Shaoling,WU Jun.Analysis of genetic diversity of European pear (Pyrus communis L.) cultivars using core simple sequence repeat (SSR) markers[J]. Journal of Agricultural Biotechnology,2015,23(5):579-587.
[19] 李胜男.分子标记在梨传播路径及种质鉴定中的应用[D].南京:南京农业大学,2019.LI Shengnan.Application of molecular markers in pear transmission path and germplasm identification[D]. Nanjing:Nanjing Agricultural University,2019.
[20] 陈少瑜,李江,陈伟,冯弦.云南沟谷雨林建群种绒毛番龙眼居群的遗传多样性[J].南方农业学报,2022,53(3):850-858.CHEN Shaoyu,LI Jiang,CHEN Wei,FENG Xian. Genetic diversity of Pometia tomentosa,a major constructive species of valley rainforest in Yunnan[J]. Journal of Southern Agriculture,2022,53(3):850-858.
[21] 邓伟,吕莹,董阳均,徐雨然,杨华涛,张锦文,张建华,奎丽梅,涂建,相罕章,管俊娇,董维,谷安宇,安华,杨丽萍,张笑,李小林.云南水稻种质资源的遗传多样性分析[J].植物遗传资源学报,2023,24(3):624-635.DENG Wei,LÜ Ying,DONG Yangjun,XU Yuran,YANG Huatao,ZHANG Jinwen,ZHANG Jianhua,KUI Limei,TU Jian,XIANG Hanzhang,GUAN Junjiao,DONG Wei,GU Anyu,AN Hua,YANG Liping,ZHANG Xiao,LI Xiaolin. The genetic diversity analysis of rice germplasm resources in Yunnan Province of China[J]. Journal of Plant Genetic Resources,2023,24(3):624-635.
[22] 胡春云.基于叶绿体DNA 非编码区的梨属系统发育关系及东亚栽培梨的演化研究[D].杭州:浙江大学,2012.HU Chunyun. Studies on Phylogenetic analysis of Pyrus L. and evolution of east asian pear cultivar goups based on non-coding cpDNA regions[D].Hangzhou:Zhejiang University,2012.
[23] 张莹,曹玉芬,田路明,董星光,齐丹,霍宏亮,徐家玉,刘超,王立东.梨种质资源果实若干数量性状评价指标研究[J].果树学报,2023,40(6):1053-1063.ZHANG Ying,CAO Yufen,TIAN Luming,DONG Xingguang,QI Dan,HUO Hongliang,XU Jiayu,LIU Chao,WANG Lidong.Evaluating standards of some fruit quantitative traits of pear genetic resources[J]. Journal of Fruit Science,2023,40(6):1053-1063.
[24] FENG S G,HE R F,LU J J,JIANG M Y,SHEN X X,JIANG Y,WANG Z A,WANG H Z. Development of SSR markers and assessment of genetic diversity in medicinal Chrysanthemum morifolium cultivars[J].Frontiers in Genetics,2016,7:113.
[25] ZHOU R,WU Z,CAO X,JIANG F L.Genetic diversity of cultivated and wild tomatoes revealed by morphological traits and SSR markers[J].Genetics and Molecular Research,2015,14(4):13868-13879.