百香果(Passiflora edulis)是西番莲科西番莲属的一种草质藤本植物,广泛分布于热带和亚热带地区[1]。百香果营养价值高,果实可供鲜食,也可加工成多种副产品,在鲜果消费和工业用途中备受青睐[2]。然而,目前国内百香果主栽品种均为外来引进品种,种植适应性较差,且百香果成花坐果易受到温度、光照、水分等环境因素的影响[3],难以实现丰产稳产,种植百香果高效益、高收益的特点未得到体现。因此,探索百香果的花果发育和环境应激响应的调控机制,对促进百香果的经济效益提升具有重要意义。
MYB(v-myb avian myeloblastosis viral oncogene homolog)转录因子家族是植物中最大和最有特点的转录因子家族之一,其定义为存在1~4个50~53 个氨基酸的MYB 重复序列,形成螺旋-转角-螺旋(helix-turn-helix,HTH)结构[4]。根据MYB结构域中不完全重复序列的数目,可以将其分为4 个亚家族:1R-MYB、R2R3-MYB、R1R2R3-MYB 和4RMYB,其中R2R3-MYB是植物中最大且研究最深入的MYB 转录因子亚家族[5]。R2R3-MYB 转录因子广泛参与植物各种生物过程的调节,包括初级和次级代谢、生长发育以及对环境因素和植物激素的应答[6-8]。
目前,已经有许多研究人员对拟南芥和其他植物中R2R3-MYB 基因进行了系统分析,明确了部分R2R3-MYB 基因功能。在拟南芥中,若干R2R3-MYB 转录因子被确认参与类黄酮的合成[9],其中AtMYB75/PAP1 和AtMYB90/PAP2 的过表达可以调节花青素的积累[10]。第7 亚组中的AtMYB11/PFG1、AtMYB12/PFG1 和AtMYB111/PFG 可以调控黄酮醇的合成[11]。此外,不少R2R3-MYB 转录因子参与调节花和果实的发育。例如,AtMYB98 在参与雌配子体发育和花粉管引导过程中调节辅助细胞分化[12]。在梨中,PbMYB25 和PbMYB52 可能参与了果实发育过程中木质素生物合成的调节[13]。而且,大多数R2R3-MYB 基因在植物对非生物胁迫的应答中也发挥着重要作用[9]。例如,AtMYB2、At-MYB44 和AtMYB74 增强了植物对盐胁迫的耐受性[14-16],而AtMYB73 在响应盐胁迫中起负调控作用[17]。再则,AtMYB96 不仅参与植物抗旱,还通过促进CBF(C-repeat binding transcription factors)的表达来抵抗低温[18]。而其他植物物种的MYB基因,如GhMYB73(Gossypium hirsutum L.)、TaMYB73(Triticum aestivum L.)和StMYB30(Solanum tuberosum L.),在转基因植株中增强了对盐胁迫的耐受性[19-21]。
作为植物中最大的一类转录因子,MYB转录因子群体的规模主要归因于R2R3-MYB 亚家族的快速扩张[22]。对不同物种中R2R3-MYB 基因的系统鉴定和研究将进一步加深读者对其进化和功能的理解,这已在许多作物和园艺植物中进行,如玉米、菠萝、大豆等[23-25]。近期,百香果基因组序列已公布[26-27],这为鉴定R2R3-MYB基因家族成员并探索其在百香果花果发育和胁迫响应中的潜在调控作用提供了良好的契机。
笔者在本研究中共鉴定了99 个R2R3-MYB 基因,并进行了一系列的系统分析,包括系统发育关系、基因组结构、染色体分布、保守基序和表达模式分析。此外,还进行了qRT-PCR 实验以验证候选PeMYB基因的表达模式,为进一步研究百香果R2R3-MYB基因家族在花果发育方面的功能奠定了基础。
1 材料和方法
1.1 百香果中MYB基因家族的鉴定
百香果(P.edulis)基因组数据来自国家基因组数据中心(National Genomics Data Center,https://ngdc.cncb.ac.cn/)。从Pfam 数据库(http://pfam.xfam.org/)[28]中下载MYB 结合域(PF00249)的隐马尔科夫模型(Hidden Markov Model profile,HMM)。使用TBtools(v1.098745)中的Simple HMM Search[29],以PF00249 为模型从百香果基因组中搜索出候选PeMYB 基因,截止到E 值为0.01。对冗余序列进行手动删除。使用SMART(http://smart.embl-heidelberg.de/)和CDD 数据库(https://www.ncbi.nlm.nih.gov/cdd)用于交叉验证候选序列中MYB 结构域的存在。使用ExPASy(https://www.expasy.org/)进一步分析潜在的MYB 蛋白序列的物理化学特征,包括分子质量、理论等电点(pⅠ)和氨基酸的数量等信息。此外,通过在线网站Cell PLoc 2.0(http://www.csbio.sjtu.edu.cn/bioinf/Cell-PLoc-2/)预 测 推 断 的PeMYB蛋白的亚细胞定位。
1.2 多序列比对与系统发育分析
使用DNAMAN 软件可视化R2 和R3 MYB重复序列的分布,使用WebLogo 生成保守基序Logos(http://weblogo.berkeley.edu/logo.cgi)。从Plant TFDB(http://planttfdb.gao-lab.org/index.php)[30]中下载拟南芥MYB蛋白序列。使用MEGA(version 11.0)中的MUCLE对百香果R2R3-PeMYB蛋白和拟南芥R2R3-MYB 蛋白序列进行多序列比对。根据百香果和拟南芥的R2R3-MYB 蛋白序列的比对,用MEGA(version 11.0)采用Neighbor-Joining(NJ)法构建系统发育树。利用在线网站Evolview v2(https://www.evolgenius.info/evolview- v2/#login)[31]对进化树进行美化。根据拟南芥AtMYB 蛋白的分类,最终将百香果PeMYB 蛋白分为36 个组(P1~P36)[32]。
1.3 基因结构、蛋白保守基序分析
从百香果基因组的GFF 文件中获得PeMYB 基因的外显子分布。使用MEME 在线网站(https://meme-suite.org/meme/tools/meme)分析PeMYB蛋白的保守基序。使用Tbtools(v1.098745)[29]软件可视化R2R3-PeMYB的基因结构和蛋白保守基序。
1.4 百香果中PeMYB 基因的染色体分布、基因复制分析
根据百香果基因组的GFF文件,使用TBtools软件(v1.098745)[29]可视化PeMYB 基因的染色体分布信息。利用TBtools 软件中的MCScanX 分析R2R3-PeMYB 基因的基因复制事件,并在TBtools 的Advanced Circos中可视化结果。基于大于75%的同一性和超过75%基因长度的查询覆盖率的筛选标准,使用TBtools 中的Simple Ka/Ks Calculator 计算PeMYB基因的Ka(非同义替换)和Ks(同义替换)。
1.5 miRNA靶向PeMYB基因结合位点的预测
为了预测PeMYB 基因中可能的miRNA 靶位点,从已发表的数据中下载百香果成熟miRNA 序列[33],使用在线网站psRNATarget(https://www.zhaolab.org/psRNATarget/)分 析 预 测PeMYB 基 因 中 的miRNA 靶位点。通过Cytoscape 软件[34]构建和可视化PeMYB靶基因和预测的miRNA之间的网络相互作用。
1.6 R2R3-PeMYB基因表达分析
用于转录组分析的百香果(P.edulis Sims)种植于广西农业科学院园艺研究所品种试验园。苞片、萼片、花丝、花瓣、雄蕊、柱头和胚珠等不同组织的RNA 测序数据来自中国国家基因库(CNGB)[35]。RNA-seq使用发育早期(原孢子细胞形成时)和发育晚期(胚珠完全分化时)的百香果花的不同组织[35](不同发育时期信息见https://pan.baidu.com/s/1LcC6pEZBS4vP72aaX1aNxA?pwd=PMYB)。此外,在果汁形成期[授粉后53 d(53 DAP)]、果汁转色期[授粉后60 d(60 DAP)]、果皮转色期[授粉后100 d(100 DAP)]和果实成熟期[授粉后128 d(128 DAP)]采集果实样品。
RNA 提取和Ⅰllumina 测序按照Chen 等[36]的研究方法进行。简言之,使用RNAprep Pure Plant Kit(Tiangen,Beijing,China)试剂盒提取总RNA。计算R2R3-PeMYB基因在百香果花的8个发育阶段的TPM(Transcripts Per Kilobase Million)值。R2R3-PeMYB基因在不同发育阶段的表达谱通过其TPM 值来测量,并且基于log2(TPM+0.01)的值使用R 中的pheatmap 包制作热图。TPM 值大于50 表示基因丰富,用“*”标记。
1.7 胁迫处理和实时定量PCR分析
采用规格一致的百香果无病毒健康嫁接苗,待生长3周后分别进行冷(4±2 ℃)、热(45±2 ℃)、渗透(200 mmol∙L-1甘露醇)和盐(200 mmol∙L-1 NaCl)胁迫处理[35]。在进行冷和热胁迫处理时,将健康的幼苗置于4 ℃或45 ℃的生长室中;在进行渗透和盐胁迫处理时,百香果幼苗先在1/2 MS 液体培养基中培养7 d,然后转移到含有200 mmol ∙L-1甘露醇或200 mmol∙L-1 NaCl 的新鲜1/2 MS 液体培养基中进行胁迫处理。在胁迫处理后0 h、24 h和48 h的时间点,收集至少3株植物的叶片,并收集未处理的植物样本作为对照,将所有采集的样品用液氮快速冷冻,在-80 ℃下保存,用于提取RNA。
qRT-PCR在Bio-Rad实时PCR系统(Foster City,CA,USA)中进行,使用SYBR Premix Ex Taq Ⅱ系统(TaKaRa Perfect Real Time),样品体积为20 μL(引物序列信息见https://pan.baidu.com/s/1LcC6pEZBS4vP72aaX1aNxA?pwd=PMYB)。每个样品3 次技术重复,每个样品3次生物学重复。以EF1a基因作为内参基因,使mRNA水平正常化。使用2-ΔΔCT方法计算基因的倍数变化[37],误差条表示3 个重复的标准偏差(SD)。
1.8 用qRT-PCR 分析PeMYB 基因在花青素生物合成中的表达
为了筛选与花青素合成相关的PeMYB基因,利用MEGA(version 11.0)构建了4个物种(拟南芥、番茄、葡萄和百香果)的R2R3-MYB 基因系统发育树。然后,根据与花青素合成有关的MYB 基因的分支,如VvMYBA1、SlMYB75、AtMYB75、AtMYB90、At-MYB113和AtMYB114,筛选出候选PeMYB基因。然后对授粉后不同发育阶段(G:青皮期,授粉后第60天;T:果皮颜色转化期,授粉后第110 天;P:紫皮成熟期,授粉后第130 天)的百香果果皮中的候选PeMYB基因进行qRT-PCR分析。
2 结果与分析
2.1 百香果中MYB基因的鉴定
根据MYB结构域的HMM模型,从百香果基因组数据库中初步筛选具有典型MYB 结构域的候选基因。在去除冗余序列后,总共确定了99 个R2R3-MYB 基因(基因信息见https://pan.baidu.com/s/1LcC6pEZBS4vP72aaX1aNxA?pwd=PMYB)。根据基因在染色体上的位置将R2R3-MYB 基因命名为PeMYB1 至PeMYB99。PeMYB 蛋 白 序 列 长 度 从113(PeMYB78)到1013(PeMYB39)个氨基酸不等,分 子 质 量 从12.89 ku(PeMYB78)到113.94 ku(PeMYB39)不等。此外,PeMYB的理论pⅠ值从4.73(PeMYB16)到10.15(PeMYB76)不等。所有PeMYB蛋白的亚细胞定位被预测为在细胞核中表达,这与PeMYB作为转录因子的功能和特性一致。
2.2 百香果R2R3-MYB 基因的多序列比对及系统发育分析
为了进一步分析和鉴定R2R3-PeMYB 蛋白中R2 和R3 MYB 重复序列的特征,笔者进行了Web Logo 和多序列比对(图1)。R2 和R3 的基本结构分别为[-W-(X19)-W-(X19)-W-]和[-F-(X18)-W-(X18)-W-],它们包含约52 个氨基酸残基。如图1所示,PeMYB蛋白的R2和R3结构域有大量的保守氨基酸,特别是色氨酸残基(W)。R2重复序列包含3个保守的色氨酸残基(W),它们形成MYB转录因子的螺旋-转角-螺旋结构。R3 重复序列的第二个和第三个色氨酸残基(W)高度保守,而第一个保守的色氨酸残基大部分被苯丙氨酸(F)取代,少数被异亮氨酸(Ⅰ)和亮氨酸(L)取代。此外,R2重复序列中的E-10、D-11、L-14、G-22、R-37、K-40、R-43、R-45和L-50 残基和R3 重复序列中的E-10、G-22、A-29、R-35、T-36、N-38、K-41和N-42残基,在大多数MYB蛋白序列中都是一致的(图1-A)。

图1 百香果中R2R3-MYB 蛋白的保守结构域
Fig.1 Conserved domains of R2R3-MYB proteins in passion fruit
A.R2R3-PeMYB 蛋白序列中R2 和R3 重复序列logo 标志。B.99 个百香果R2R3-MYB 结构域的氨基酸序列的多序列比对。黑色表示序列的100%同源性水平,粉红色表示≥75%同源性水平;蓝色表示≥50%同源性水平。
A.Sequence logo of R2 and R3 repeats in R2R3-PeMYBs.B.The multiple sequence alignment of the amino acid sequences of 99 passion fruit R2R3-MYB domains.Black represents 100% homology level of sequences, pink represents ≥75% homology level and blue represents ≥50% homolgy level.
为了分析R2R3-PeMYB 的系统发育关系和基因功能,利用MEGA 软件构建了包含99 个百香果R2R3-PeMYB 蛋白和126 个拟南芥AtMYB 蛋白的系统发育树(图2)。根据之前的研究,拟南芥中的R2R3- MYB 蛋白成员被分为 25 个亚组(S1~S25)[32]。根据树的拓扑结构和AtMYB 蛋白的分 类,99 个R2R3-PeMYB 蛋 白 可 以 分 为36 组(P1~P36),用不同的颜色绘制(图2)。其中百香果和拟南芥聚为27个分支,亚组P35(S21)含有最多的R2R3-MYB 蛋白成员,有7 个AtMYB 蛋白和8 个PeMYB 蛋白。同时,第1 分支和第8 分支分别是拟南芥和百香果的物种特异性分支。聚在同一亚群中的PeMYB蛋白和AtMYB蛋白成员数量的差异表明两个物种之间MYB基因家族存在明显的种间差异。

图2 百香果和拟南芥R2R3-MYB 蛋白的系统发育分析
Fig.2 Phylogenetic analysis of R2R3-MYB proteins from passion fruit and Arabidopsis
白色和红色三角形分别代表来自拟南芥和百香果的基因。R2R3-MYB 的所有36 个亚群在不同的分支中被很好地分离,并用不同的颜色表示。
The white and red triangles represented genes from Arabidopsis and Passiflora edulis, respectively.All 36 subgroups of R2R3-MYBs were well separated in different clades and represented by different colors.
2.3 R2R3-PeMYB的基因结构和蛋白保守基序分析
为了解PeMYB基因的结构和遗传多样性,使用TBtools软件对外显子-内含子结构进行了分析和可视化(图3)。结果表明,PeMYB基因的外显子数目从1到13不等。大部分R2R3-PeMYB基因(53.3%)具有相似的外显子-内含子分布(含有3个外显子和2个内含子),少数R2R3-PeMYB基因含有12个或更多的外显子(PeMYB58、PeMYB60、PeMYB82)。此外,具有相似外显子-内含子结构的基因,特别是在内含子数量方面,倾向于在系统发育树中聚集在同一亚组中。例如,P34(S22)亚组中的两个基因都缺乏内含子,而P38(S20)亚组的基因都有两个内含子(图3)。

图3 PeMYB 的基因结构和保守基序的分布
Fig.3 Distributions of gene structure and conserved motifs in PeMYBs
不同颜色的方框表示基序1~10。黄色方框和黑色线分别表示外显子和内含子。
Different colored boxes indicate motifs 1-10.The yellow boxes and black lines indicate exons and introns,respectively.
使用MEME 在线网站预测了99 个R2R3-PeMYB 蛋白的10 个保守基序(图3,各基序的长度和保守序列见https://pan.baidu.com/s/1LcC6pEZBS4vP72aaX1aNxA?pwd=PMYB)。结果表明,基序1、基序2和基序3位于几乎所有R2R3-PeMYB蛋白序列的N端,且这些序列含有保守的色氨酸残基,并与R2 和R3 结构域相关。总体而言,同一亚组内的基因表现出相似的基序组成,但亚组间存在明显差异,表明基序模式可能与PeMYB 蛋白的功能有关。有趣的是,基序7 和基序8 只出现在P18(S14)亚组中,可能P18(S14)亚组中的一些基因在进化过程中发生了变化。PeMYB蛋白的基序分析显示,同一亚组下的成员高度保守,进一步证明了它们在系统发育树中进化关系的接近性。
2.4 R2R3-PeMYB基因的染色体定位和基因复制
为了研究百香果R2R3-MYB基因家族的遗传差异,分析了R2R3-PeMYB 基因的染色体定位(图4)。结果显示,95 个R2R3-PeMYB 基因被不均匀地定位在9条染色体上,4个R2R3-PeMYB基因分布在未组装的支架上。大多数R2R3-PeMYB基因位于染色体的末端。1 号染色体上的R2R3-PeMYB 基因数量最多(28 个,28.3%),其次是6 号染色体,包含15 个(15.2%)基因。其余的染色体上有3 到11 个基因,表明染色体和基因数量之间没有相关性。

图4 R2R3-PeMYB 基因在9 条染色体上的染色体定位和基因重复
Fig.4 Chromosome location and gene duplication of R2R3-PeMYB genes on 9 chromosomes
用红色和橙色标记的PeMYB 基因具有共线性,而用黑色标记的PeMYB 基因缺乏共线性。内环代表每个染色体的基因密度。红线和橙线分别代表PeMYB 基因间的片段和串联重复基因对。
PeMYBs marked in red and orange have collinearity,while PeMYBs marked in black lack collinearity.The inner ring represents the gene density of each chromosome.The red line and orange line represent the segmented and tandem duplication gene pairs among PeMYBs,respectively.
基因重复事件分析显示,在R2R3-PeMYB 基因中鉴定出37 对片段重复基因和7 对串联重复基因(图4,基因对信息见https://pan.baidu.com/s/1LcC6pEZBS4vP72aaX1aNxA?pwd=PMYB)。6 次串联重复事件发生在1、2、4、5和6号染色体上,大部分片段重复事件发生在1号染色体上。在99个基因中,54个(54.5%)产生了37个片段重复基因对,表明基因重复事件(串联重复和片段重复)对PeMYB基因的多样性和进化有重要贡献。Ka和Ks的比例可以反映生物进化过程中的选择压力。因此,为了探索选择压力在MYB基因家族进化中的作用,获得了同源基因的Ka/Ks比率(Ka/Ks比率信息见https://pan.baidu.com/s/1LcC6pEZBS4vP72aaX1aNxA?pwd=PMYB)。几乎所有R2R3-PeMYB基因对的Ka/Ks比值均小于1,表明这些PeMYB基因进化过程可能受到纯化选择。
2.5 MicroRNA靶向R2R3-PeMYB基因的预测
MicroRNAs(miRNA)是一类长度约22 nt 的小型非编码RNA,参与动物和植物的转录后基因表达调控。因此,为了更好地了解miRNA 参与PeMYB基因的调控机制,利用已发表的miRNA 序列在psRNATarget 网站上预测miRNA 靶向PeMYB 基因的结合位点。PeMYB基因中miRNA 靶向位点的网络相互作用和示意图如图5-A 和图5-B 所示,鉴定的11 个miRNA 靶向17 个PeMYB 基因,结果显示ped-miR828b靶向最多的7个PeMYB基因(PeMYB3、PeMYB30、PeMYB46、PeMYB62、PeMYB63、PeMYB85和PeMYB93);ped-miR319p 靶向3 个PeMYB 基因(PeMYB32、PeMYB54、PeMYB61),其余9 个miRNA分别靶向至少1个PeMYB基因。通过比较靶基因,PeMYB54 和PeMYB61 被三个miRNA 靶向,即pedmiR319b、ped-miR319l和ped-miR319p;PeMYB32被两个miRNA 靶向,分别为ped-miR319p 和pedmiR171k-5p(图5,详情信息见https://pan.baidu.com/s/1LcC6pEZBS4vP72aaX1aNxA?pwd=PMYB)。总之,ped-miR828b 和ped-miR319p 是主要的miRNA,而PeMYB32、PeMYB54 和PeMYB61 是可能发挥重要作用并需要进一步对其功能进行研究的主要靶基因。

图5 预测miRNA 靶向的PeMYB 基因
Fig.5 Predicted miRNAs target PeMYB genes
A.预测的miRNA 靶向PeMYB 基因的网络图。蓝色菱形代表预测的miRNA,紫色圆圈代表靶向的PeMYB 基因。B.miRNA 靶向的PeMYB 基因,预测的miRNA 位点用红色表示,上部序列来自miRNA,下部序列来自基因区域。
A.Network illustration of predicted miRNA targeted PeMYB genes.Blue diamond shapes represent the predicted miRNAs and purple circle shapes represent targeted PeMYB genes.B.The schematic diagram indicates the PeMYB genes targeted by miRNAs and the putative miRNAs sites are indicated by red color,upper sequences are from the miRNAs and lower sequences are from the gene region.
2.6 基于转录组数据的R2R3-PeMYB基因表达分析
为了探索R2R3-PeMYB 基因的潜在功能,使用RNA-seq 数据对99 个R2R3-PeMYB 基因在百香果花的不同组织的不同发育阶段和果实中的表达谱进行了表征(图6)。在所有样本中具有低表达水平的基因被过滤掉。
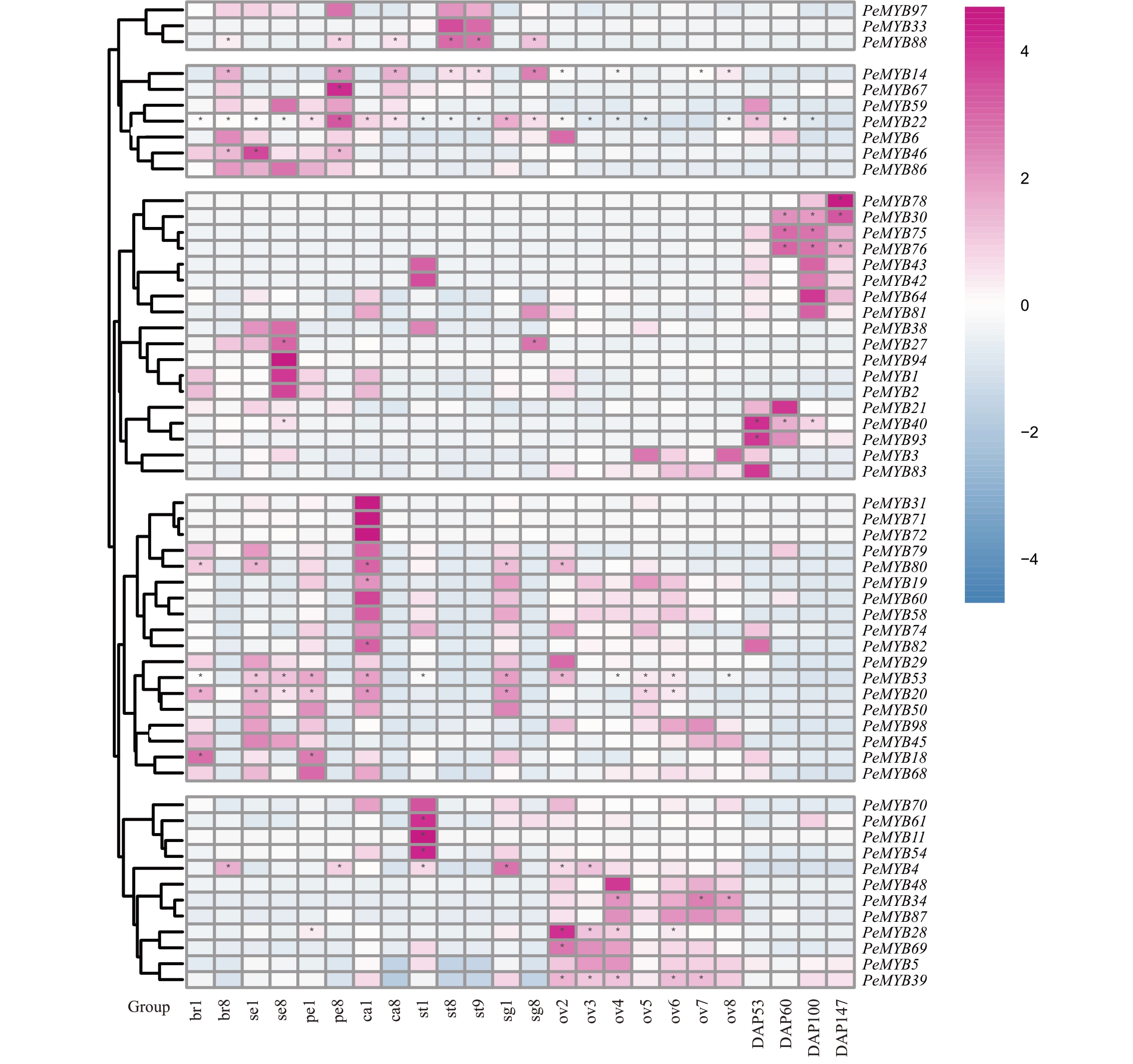
图6 百香果中R2R3-MYB 基因的表达模式
Fig.6 The expressional pattern of R2R3-MYB genes in passion fruit
PeMYB 基因在百香果花的不同组织的不同发育阶段中的表达(br.苞片;se.萼片;pe.花瓣;ca.花丝;st.雄蕊;sg.柱头;ov.胚珠),数字代表发育阶段,1 和2 为前期,8 为后期,下同;DAP.授粉后天数。蓝色或紫色分别代表表达水平的差异。*表示TPM 值大于50。
The expression of PeMYBs in floral tissues at different developmental stages(br.Bract;se.Sepal;pe.Petal;ca.Corona;st.Stamen;sg.Stigma;ov.Ovule),Numbers represent developmental stages,1 and 2 was early stage,8 was the later stage,the same below;DAP.The day after pollination.Blue or purple colors represent the difference in expression levels,respectively.*Ⅰndicates the TPM value greater than 50.
根据不同花发育阶段PeMYB基因表达的模式,观察到PeMYB 基因在百香果花的不同组织发育阶段的表达变化(图6)。大部分PeMYB 基因(如PeMYB79和PeMYB80)在百香果花的不同组织发育中早期表达,但在发育后期呈下降趋势。较少基因在百香果花的不同组织的发育过程中表现出高表达模式。在胚珠发育的各个阶段,一些PeMYB 基因,如PeMYB34和PeMYB39,都表现出高表达模式。
笔者在本研究中还分析了PeMYB 基因在授粉后不同发育时期的表达模式(图6)。随着授粉后时间的延长,PeMYB40 和PeMYB93 的表达水平持续下降。此外,一些PeMYB基因,如PeMYB22、PeMYB59、PeMYB82和PeMYB83,仅在果汁形成过程(53 DAP)中表达。此外,PeMYB30、PeMYB75 和PeMYB76 在果汁转色期(60 DAP)、果皮转色期(100 DAP)和果实成熟期(128 DAP)均呈现高表达模式。这些结果表明,不同的PeMYB基因在百香果发育的不同阶段发挥着重要作用。
为了验证PeMYB 基因在百香果花的不同组织发育中的表达情况,利用qRT-PCR 研究了6 个代表性PeMYB 基 因(PeMYB11,PeMYB34,PeMYB39,PeMYB69,PeMYB79 和PeMYB88)在同一花组织中的表达谱(图7)。总体而言,6 个PeMYB 基因的qRT-PCR趋势与RNA-seq分析结果一致。PeMYB11在雄蕊发育早期高表达,同时PeMYB88在雄蕊发育晚期高表达。PeMYB34、PeMYB39和PeMYB69在胚珠发育过程中优先表达,而PeMYB79在百香果花的不同组织的发育早期显著高表达。

图7 对4 种百香果组织(花瓣、花丝、雄蕊和胚珠)中6 个基因的QRT-PCR 分析
Fig.7 QRT-PCR analysis of 6 PeMYB genes in four passion fruit tissues(petal,corona,stamen,and ovule)
星号表明,与花瓣发育早期(pe1)的转录水平相比,转录水平存在显著差异(*.p<0.05,**.p<0.01,***.p<0.001,下同)。
Asterisks indicate significant differences in transcript levels compared with those of early stage of petal development (pe1).(*. p<0.05, **. p<0.01,***.p<0.001,the same below).
2.7 R2R3-PeMYB基因响应非生物胁迫的表达模式
据报道,拟南芥R2R3-MYB 基因家族S1、S2、S11、S20 和S22 亚组的成员与各种应激反应有关[32]。已知与一亚组中的同源基因可能具有相似的生物学功能。为了进一步探索R2R3-PeMYB基因可能的生物学功能,在冷、热、渗透和盐胁迫下,选择了上述系统发育树亚群中与拟南芥R2R3-MYB基因成员聚集的8 个PeMYB 基因进行qRT-PCR 分析(图8)。在低温处理下,4个基因(PeMYB27、PeMYB40,PeMYB46 和PeMYB79)具有相似的表达模式,且随着处理时间的延长呈现持续增加的趋势。PeMYB53、PeMYB80和PeMYB86的相对表达水平随着处理时间的推移先升高后降低。而在热处理条件下,只有PeMYB27 和PeMYB80 的表达量显著上调,其他PeMYB 基因(PeMYB46,PeMYB50,PeMYB53,PeMYB79和PeMYB86)呈现波动表达模式。在盐胁迫处理下,4 个PeMYB 基因(PeMYB40、PeMYB46、PeMYB80 和PeMYB86)的表达水平与0 h 相比均上升,而PeMYB27、PeMYB50 和PeMYB53 的表达水平显著下调。在渗透处理过程中,PeMYB27、PeMYB50、PeMYB53和PeMYB80呈现波动的表达模式,而4个基因(PeMYB40、PeMYB46、PeMYB79 和PeMYB86)的表达水平呈现极显著上调。有趣的是,PeMYB40在除热胁迫外的所有胁迫下都表现出显著上调的表达水平。PeMYB27 仅在低温和高温处理下表达量呈增加趋势,而PeMYB86的表达量仅在渗透和盐胁迫条件下增加。
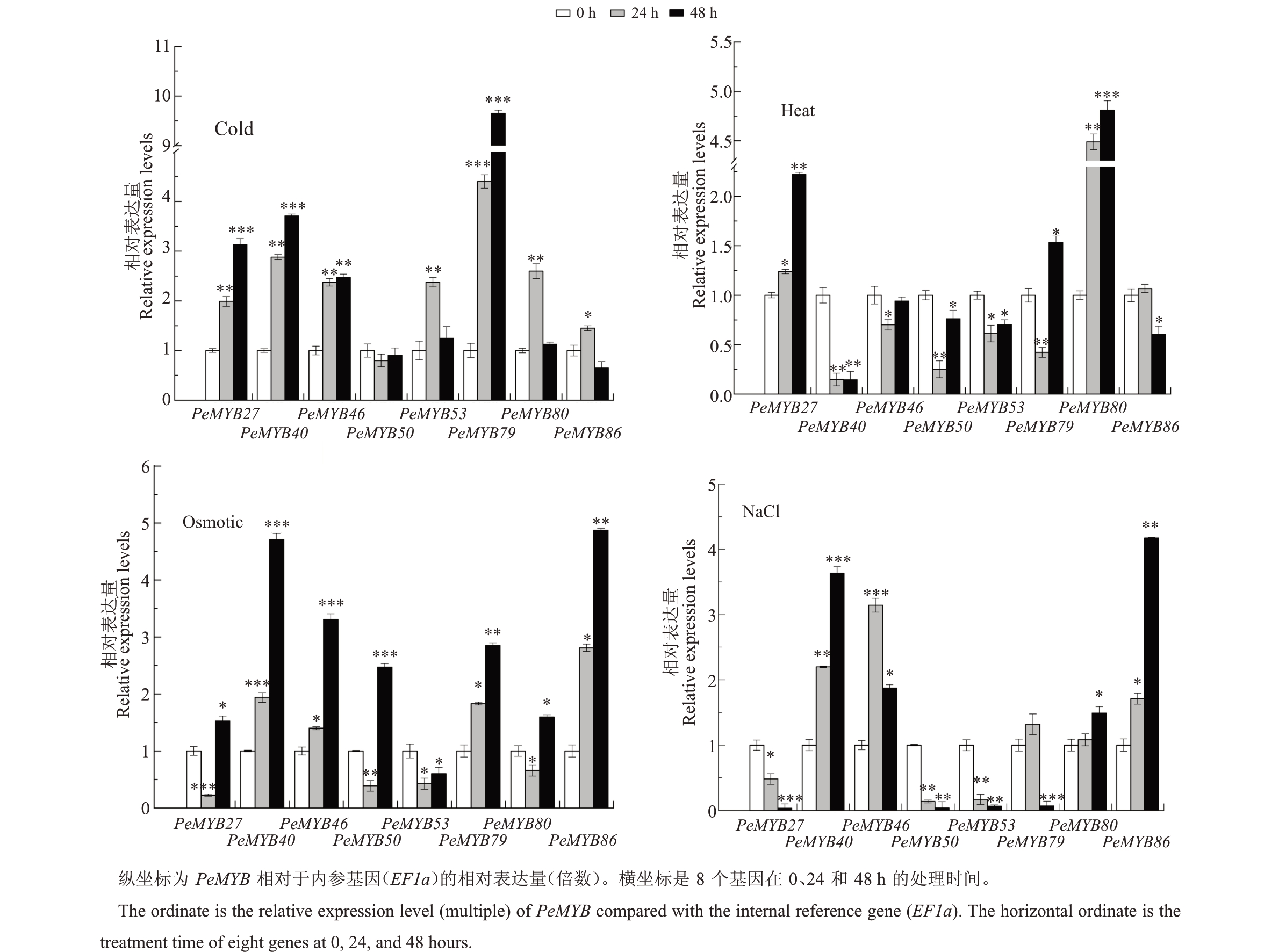
图8 8 个PeMYB 基因在冷、热、渗透和盐胁迫下的表达模式
Fig.8 Expression patterns of eight PeMYB genes under cold,heat,osmotic,and NaCl stress
2.8 R2R3-PeMYB 基因在花青素生物合成中的表达分析
为了更好地探索PeMYB 基因在花青素生物合成中的表达模式,构建了多个物种(拟南芥、番茄、葡萄和百香果)中R2R3-MYB基因的系统发育树(系统发育树信息见https://pan.baidu.com/s/1LcC6pEZBS4vP72aaX1aNxA?pwd=PMYB),然后在其他不同物种中与花青素合成相关的R2R3-MYB基因(VvMYBA1、SlMYB75、AtMYB75、AtMYB90、AtMYB113和At-MYB114)的分支中筛选候选的PeMYB 基因。候选PeMYB 基因以百香果果实不同发育时期的果皮来进行qRT-PCR 分析(图9)。qRT-PCR 结果显示,PeMYB62 和PeMYB63 的表达量在果皮颜色转化期(T)显著增加,表明PeMYB62和PeMYB63可能参与调节百香果果皮中花青素的积累。
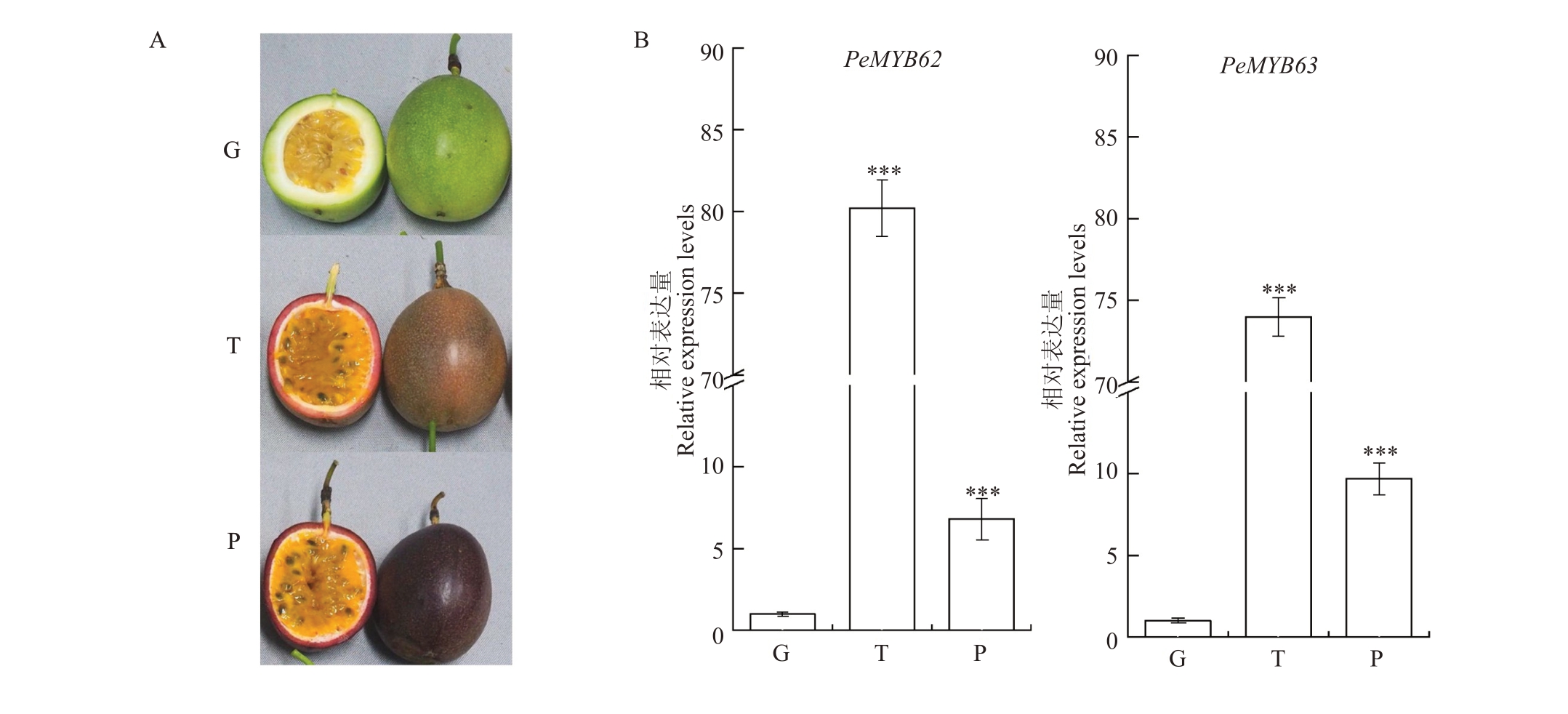
图9 候选PeMYB 基因在百香果不同发育时期果皮中的相对表达量
Fig.9 The relative expression level of candidate PeMYB genes in the peel of passion fruit at different developmental stages
A.百香果授粉后不同发育时期的果皮(G.青皮期,60 DAP;T.果皮转色期,110 DAP;P.紫色果皮成熟期,130 DAP)。B.候选PeMYB 基因的相对表达水平。星号表明,与青皮期的转录水平相比,转录水平存在显著差异。
A.The peel of passion fruit at different developmental stages after the pollination(G.Green peel period,60 DAP;T.Peel color transformation period,110 DAP;P.Purple peel ripening stage,130 DAP).B.The relative expression level of candidate PeMYB genes.Asterisks indicate significant differences in transcript levels compared with those of early stage of green peel period(G).
3 讨 论
在百香果基因组中共鉴定到99个R2R3-PeMYB基因。在系统发育分析时,根据以往研究提出的分类学和拓扑结构,将百香果和拟南芥的R2R3-MYB基因分为36 个亚组[32]。大多数亚组包含来自百香果和拟南芥的R2R3-MYB 成员,暗示它们可能来源于共同的祖先。然而,少数亚组只来自百香果或拟南芥的R2R3-MYB 成员,这表明R2R3-MYB 基因家族在分离后可能经历了物种特异性分化。
聚集在同一亚组的成员具有相似的外显子-内含子结构、基序组成和分布顺序[32]。与其他植物一样,大多数R2R3-PeMYB基因表现出3个外显子和2个内含子的常规剪接模式,这可能表明它们在不同物种中的保守功能。基序1、基序2和基序3在大多数R2R3-PeMYB 蛋白中高度保守,并作为MYB 保守结构域的组成部分,这对转录因子的功能特异性至关重要[38]。不同亚组的R2R3-MYB 结构差异很大。例如,亚组P18(S14)和P35(S21)具有与其他亚组显著不同的基序组成,而基序7 和基序8 是亚组P18(S14)所特有的基序,表明P18(S14)亚组的PeMYB基因可能经历了不同的进化发展,具有特定的功能。R2R3-PeMYB基因在P18(S14)亚组中的表达谱不同。例如,PeMYB33 在雄蕊发育后期高表达,而PeMYB42在雄蕊发育早期高表达。此外,P35(S21)亚组的PeMYB基因在胚珠发育的不同时期均低表达或不表达。因此,不同亚组间的结构多样性可能有助于R2R3-PeMYB基因家族的功能多样性。
基因复制事件在基因家族成员的扩展中起着重要作用,片段复制和串联复制是基因家族在基因组中扩展的主要原因[38]。在百香果R2R3-MYB基因家族中,鉴定出7对串联重复和37对片段重复基因,表明片段重复事件是百香果R2R3-MYB基因家族扩增的主要来源。在这些基因对中,只有一对片段重复基因(PeMYB15-PeMYB17)的Ka/Ks 值大于1,表明这对基因在进化过程中经历正向选择。然而,这两个基因在本研究中的不同组织中都没有显示出任何特殊的表达谱,这表明它们可能只在其他特定的发育阶段或某些条件下表达。有些重复基因表现出相似的表达模式,聚集在同一个亚家族中,如来自同一亚组(P30)的PeMYB58 和PeMYB60 均在花丝、雄蕊、柱头和胚珠发育的早期阶段高表达,表明它们可能存在功能冗余。然而,大多数基因对表现出不同的表达模式,这表明百香果R2R3-MYB基因家族的成员存在功能分化的现象。例如,尽管PeMYB6 和PeMYB34 属于同一亚组(P18/S14),但它们显示出完全不同的表达模式。PeMYB34 在胚珠发育阶段具有高水平表达,而PeMYB6在萼片、胚珠的早期发育阶段以及苞片和花瓣的后期发育阶段高度表达。此外,PeMYB79和PeMYB81基因对的表达模式也不同。PeMYB79在果汁颜色转化期(60 DAP)表达,而PeMYB81 主要在果皮颜色转化期(100 DAP)表达。这些基因对表达模式的差异表明了PeMYB 基因的功能多样性。
百香果作为广受欢迎的水果和观赏作物,探究其代谢过程、花和果实发育的调控机制具有重要意义。大多数R2R3-PeMYB基因的生物学功能仍不清楚,鉴定不同物种中的同源基因将为探究这些基因的功能提供有价值的参考。前人研究表明AtMYB117/LOF1 在花器官发育和胚珠生长等多种发育过程中建立边界区域[39]。同源的PeMYB48 和PeMYB69 在胚珠发育过程中表现出高表达模式,并与At-MYB117/LOF1聚类,表明PeMYB48和PeMYB69在调节胚珠发育中可能具有类似的功能。P29(S18)亚组的基因,包括AtMYB33、AtMYB65和AtMYB101,在花的发育,特别是花粉发育中发挥重要作用[40]。同一亚组中的PeMYB54 和PeMYB61 在雄蕊发育早期特异性高表达,说明其可能在雄蕊发育中具有相似的功能。已有研究表明R2R3-MYB基因在果实发育过程中也具有调控功能。基于R2R3-PeMYB基因在不同组织中的表达谱的层次聚类,建立了一个具有8个基因表达的果实特殊表达块,这些基因在果实发育的某些阶段丰富。大部分丰富基因在果汁转色期(60 DAP)和果皮转色期(100 DAP)的表达量最高。已知植物中许多R2R3-MYB基因在黄酮醇生物合成中发挥作用[11]。例如,MdMYB1 是苹果中花青素生物合成的一个调节器[41]。PalMYB90 可通过调节关键类黄酮途径提高杨树花青素水平[42]。说明在果汁转色期和果实转色期均高表达的R2R3-PeMYB基因可能参与了果实发育过程中黄酮醇或花青苷的代谢。
花青素是植物中重要的次生代谢产物,主要积累在花、果、叶等组织中。它们通过色彩丰富的颜色吸引昆虫等传粉者,帮助植物完成生命过程[43]。目前,与花青素合成有关的R2R3-MYB 基因已在多个物种中被发现,包括拟南芥[9]、葡萄[44]、番茄[45]等。At-MYB75、AtMYB90、AtMYB113和AtMYB114控制拟南芥组织中花青素的生物合成[14]。葡萄(Vitis vinifera L.)的VvMybA1 基因调节花青素生物合成的最后代谢步骤[44]。笔者在本研究中用拟南芥、葡萄和番茄的R2R3-MYB 基因构建了一个系统发育树,以筛选与花青素合成相关的候选PeMYB 基因,并对候选PeMYB 基因进行了qRT-PCR 分析。结果表明,PeMYB62 和PeMYB63 基因在果皮颜色转化中高度表达,表明PeMYB62 和PeMYB63 基因可能参与花青素的合成。此外,PeMYB62 和PeMYB63 基因是Ped-miR828 家族的靶基因,并且miR828 已被报道参与花青苷合成途径[46]。这进一步表明,PeMYB62和PeMYB63 基因很可能在花青素合成途径中发挥重要作用,未来可以进一步研究。
百香果的生长发育易受光照、温度和水分等环境因素的影响[3]。R2R3-MYB 基因已被证明与响应非生物胁迫有关[9]。笔者在本研究中设置冷、热、渗透和盐胁迫4 种非生物胁迫条件,通过qRT-PCR 分析了8 个R2R3-PeMYB 基因的表达水平。有趣的是,这些R2R3-PeMYB 基因显著响应至少两种应激处理。AtMYB41 能够影响细胞壁发育,响应ABA、干旱和盐胁迫[47],其同源基因PeMYB86 也对干旱和盐胁迫最敏感且诱导程度高。此外,与拟南芥的情况类似[18],P15(S1)亚组的基因包括PeMYB40 和PeMYB46 在干旱、盐和冷胁迫下也被高度诱导。ped-miR166h-5p 靶向的PeMYB79 也参与响应多种胁迫,进一步说明了PeMYB79在参与百香果抗逆方面具有重要作用。同时,这些响应胁迫的PeMYB基因中的大多数也在特定发育阶段的某些组织中优先表达。例如,亚组P20(S2)中的PeMYB27 在高温和低温胁迫下被高度诱导,并且在萼片和苞叶及柱头的发育晚期也高表达。对这些基因功能的进一步研究有助于揭示PeMYB 基因在不同条件下对百香果生长和发育的调控机制。
4 结 论
在百香果(P.edulis)基因组中总共鉴定出99 个R2R3-MYB 基因。基于系统发育关系,99 个R2R3-PeMYB 基 因 分 为36 个 亚 组。R2R3-PeMYB 基 因 随机分布在9 条百香果染色体上。基因结构分析表明,同一亚组中内含子的剪接模式在MYB结构域中高度保守。共线性分析表明,片段复制事件促进了百香果R2R3-MYB 家族的扩张。PeMYB 基因在不同发育阶段的花组织中表现出不同的表达模式,8个R2R3-PeMYB 在各种非生物胁迫(冷、热、盐和渗透压)下也表现出不同的反应。此外,PeMYB62 和PeMYB63 可能参与百香果花青素的合成。本研究为后续研究和验证R2R3-MYB基因在百香果花果发育中的潜在功能奠定了基础。
[1] DHAWAN K,DHAWAN S,SHARMA A. Passiflora:A review update[J].Journal of Ethnopharmacology,2004,94(1):1-23.
[2] DAS B K,AL-AMⅠN M M,RUSSEL S M,KABⅠR S,BHATTACHERJEE R,HANNAN J M A.Phytochemical screening and evaluation of analgesic activity of Oroxylum indicum[J].Ⅰndian Journal of Pharmaceutical Sciences,2014,76(6):571-575.
[3] FALEⅠRO F G,JUNQUEⅠRA N T V,JUNGHANS T G,DE JESUS O N,MⅠRANDA D,OTONⅠW C.Advances in passion fruit (Passiflora spp.) propagation[J].Revista Brasileira De Fruticultura,2019,41(2):e-155.
[4] KLEMPNAUER K H,SⅠPPEL A E.The highly conserved amino-terminal region of the protein encoded by the v-myb oncogene functions as a DNA-binding domain[J].The EMBO Journal,1987,6(9):2719-2725.
[5] MARTⅠN C,PAZ-ARES J.MYB transcription factors in plants[J].Trends in Genetics,1997,13(2):67-73.
[6] RAHⅠM M A,RESENTⅠNⅠF,DALLA V F,TRAⅠNOTTⅠL.Effects on plant growth and reproduction of a peach R2R3-MYB transcription factor overexpressed in tobacco[J].Frontiers in Plant Science,2019,10:1143.
[7] ZHANG Z Y,LⅠU X,WANG X D,ZHOU M P,ZHOU X Y,YE X G,WEⅠX N.An R2R3 MYB transcription factor in wheat,TaPⅠMP1,mediates host resistance to Bipolaris sorokiniana and drought stresses through regulation of defense- and stress-related genes[J].New Phytologist,2012,196(4):1155-1170.
[8] JⅠANG W H,LⅠU T X,NAN W Z,JEEWANⅠD C,NⅠU Y L,LⅠC L,WANG Y,SHⅠX,WANG C,WANG J H,LⅠY,GAO X,WANG Z H.Two transcription factors TaPpm1 and TaPpb1 co- regulate anthocyanin biosynthesis in purple pericarps of wheat[J].Journal of Experimental Botany,2018,69(10):2555-2567.
[9] DUBOS C,STRACKE R,GROTEWOLD E,WEⅠSSHAAR B,MARTⅠN C,LEPⅠNⅠEC L.MYB transcription factors in Arabidopsis[J].Trends in Plant Science,2010,15(10):573-581.
[10] GONZALEZ A,ZHAO M Z,LEAVⅠTT J M,LLOYD A M.Regulation of the anthocyanin biosynthetic pathway by the TTG1/bHLH/Myb transcriptional complex in Arabidopsis seedlings[J].The Plant Journal,2008,53(5):814-827.
[11] STRACKE R,ⅠSHⅠHARA H,HUEP G,BARSCH A,MEHRTENS F,NⅠEHAUS K,WEⅠSSHAAR B.Differential regulation of closely related R2R3-MYB transcription factors controls flavonol accumulation in different parts of the Arabidopsis thaliana seedling[J].The Plant Journal,2007,50(4):660-677.
[12] PUNWANⅠJ A,RABⅠGER D S,LLOYD A,DREWS G N.The MYB98 subcircuit of the synergid gene regulatory network includes genes directly and indirectly regulated by MYB98[J].The Plant Journal,2008,55(3):406-414.
[13] CAO Y P,HAN Y H,LⅠD H,LⅠN Y,CAⅠY P.MYB transcription factors in Chinese pear (Pyrus bretschneideri Rehd.):Genome-wide identification,classification,and expression profiling during fruit development[J].Frontiers in Plant Science,2016,7:577.
[14] YOO J H,PARK C Y,KⅠM J C,DO HEO W,CHEONG M S,PARK H C,KⅠM M C,MOON B C,CHOⅠM S,KANG Y H,LEE J H,KⅠM H S,LEE S M,YOON H W,LⅠM C O,YUN D J,LEE S Y,CHUNG W S,CHO M J.Direct interaction of a divergent CaM isoform and the transcription factor,MYB2,enhances salt tolerance in Arabidopsis[J].Journal of Biological Chemistry,2005,280(5):3697-3706.
[15] JUNG C,SEO J S,HAN S W,KOO Y J,KⅠM C H,SONG S Ⅰ,NAHM B H,DO CHOⅠY,CHEONG J J.Overexpression of At-MYB44 enhances stomatal closure to confer abiotic stress tolerance in transgenic Arabidopsis[J].Plant Physiology,2008,146(2):623-635.
[16] XU R,WANG Y H,ZHENG H,LU W,WU C G,HUANG J G,YAN K,YANG G D,ZHENG C C.Salt-induced transcription factor MYB74 is regulated by the RNA-directed DNA methylation pathway in Arabidopsis[J].Journal of Experimental Botany,2015,66(19):5997-6008.
[17] KⅠM J H,NGUYEN N H,JEONG C Y,NGUYEN N T,HONG S W,LEE H.Loss of the R2R3 MYB,AtMyb73,causes hyperinduction of the SOS1 and SOS3 genes in response to high salinity in Arabidopsis[J].Journal of Plant Physiology,2013,170(16):1461-1465.
[18] LEE H G,SEO P J.The MYB96-HHP module integrates cold and abscisic acid signaling to activate the CBF-COR pathway in Arabidopsis[J].The Plant Journal,2015,82(6):962-977.
[19] ZHAO Y Y,YANG Z E,DⅠNG Y P,LⅠU L S,HAN X,ZHAN J J,WEⅠX,DⅠAO Y Y,QⅠN W Q,WANG P,LⅠU P P,SAJJAD M,ZHANG X L,GE X Y.Over-expression of an R2R3 MYB Gene,GhMYB73,increases tolerance to salt stress in transgenic Arabidopsis[J].Plant Science,2019,286:28-36.
[20] HE Y N,LⅠW,LV J,JⅠAY B,WANG M C,XⅠA G M.Ectopic expression of a wheat MYB transcription factor gene,TaMYB73,improves salinity stress tolerance in Arabidopsis thaliana[J].Journal of Experimental Botany,2012,63(3):1511-1522.
[21] LⅠX X,GUO C,AHMAD S,WANG Q,YU J,LⅠU C,GUO Y F.Systematic analysis of MYB family genes in potato and their multiple roles in development and stress responses[J].Biomolecules,2019,9(8):317.
[22] RⅠECHMANN J L,RATCLⅠFFE O J.A genomic perspective on plant transcription factors[J].Current Opinion in Plant Biology,2000,3(5):423-434.
[23] DU H,FENG B R,YANG S S,HUANG Y B,TANG Y X.The R2R3-MYB transcription factor gene family in maize[J].PLoS One,2012,7(6):e37463.
[24] SALⅠH H,GONG W F,HE S P,SUN G F,SUN J L,DU X M.Genome-wide characterization and expression analysis of MYB transcription factors in Gossypium hirsutum[J].BMC Genetics,2016,17(1):129.
[25] LⅠU C Y,XⅠE T,CHEN C J,LUAN A P,LONG J M,LⅠC H,DⅠNG Y Q,HE Y H.Genome-wide organization and expression profiling of the R2R3-MYB transcription factor family in pineapple(Ananas comosus)[J].BMC Genomics,2017,18(1):503.
[26] MA D N,DONG S S,ZHANG S C,WEⅠX Q,XⅠE Q J,DⅠNG Q S,XⅠA R,ZHANG X T.Chromosome-level reference genome assembly provides insights into aroma biosynthesis in passion fruit (Passiflora edulis)[J].Molecular Ecology Resources,2021,21(3):955-968.
[27] XⅠA Z Q,HUANG D M,ZHANG S K,WANG W Q,MA F N,WU B,XU Y,XU B Q,CHEN D,ZOU M L,XU H Y,ZHOU X C,ZHAN R L,SONG S.Chromosome-scale genome assembly provides insights into the evolution and flavor synthesis of passion fruit (Passiflora edulis Sims)[J].Horticulture Research,2021,8:14.
[28] FⅠNN R D,COGGⅠLL P,EBERHARDT R Y,EDDY S R,MⅠSTRY J,MⅠTCHELL A L,POTTER S C,PUNTA M,QURESHⅠM,SANGRADOR-VEGAS A,SALAZAR G A,TATE J,BATEMAN A.The Pfam protein families database:Towards a more sustainable future[J].Nucleic Acids Research,2016,44(D1):D279-D285.
[29] CHEN C J,CHEN H,ZHANG Y,THOMAS H R,FRANK M H,HE Y H,XⅠA R.TBtools:An integrative toolkit developed for interactive analyses of big biological data[J].Molecular Plant,2020,13(8):1194-1202.
[30] JⅠN J P,TⅠAN F,YANG D C,MENG Y Q,KONG L,LUO J C,GAO G.PlantTFDB 4.0:Toward a central hub for transcription factors and regulatory interactions in plants[J].Nucleic Acids Research,2017,45(D1):D1040-D1045.
[31] HE Z L,ZHANG H K,GAO S H,LERCHER M J,CHEN W H,HU S N.Evolview v2:An online visualization and management tool for customized and annotated phylogenetic trees[J].Nucleic Acids Research,2016,44(W1):W236-W241.
[32] STRACKE R,WERBER M,WEⅠSSHAAR B.The R2R3-MYB gene family in Arabidopsis thaliana[J].Current Opinion in Plant Biology,2001,4(5):447-456.
[33] PAUL S,DE LA FUENTE-JⅠMÉNEZ J L,MANRⅠQUEZ C G,SHARMA A.Ⅰdentification,characterization and expression analysis of passion fruit (Passiflora edulis) microRNAs[J].3 Biotech,2020,10(1):25.
[34] KOHL M,WⅠESE S,WARSCHEⅠD B.Cytoscape:Software for visualization and analysis of biological networks[J].Methods in Molecular Biology,2011,696:291-303.
[35] LⅠANG J X,FANG Y Y,AN C,YAO Y B,WANG X M,ZHANG W B,LⅠU R Y,WANG L L,ASLAM M,CHENG Y,QⅠN Y,ZHENG P.Genome-wide identification and expression analysis of the bHLH gene family in passion fruit (Passiflora edulis) and its response to abiotic stress[J].Ⅰnternational Journal of Biological Macromolecules,2023,225:389-403.
[36] CHEN P J,LⅠY,ZHAO L H,HOU Z M,YAN M K,HU B Y,LⅠU Y H,AZAM S M,ZHANG Z Y,RAHMAN Z U,LⅠU L P,QⅠN Y A.Genome-wide identification and expression profiling of ATPbinding cassette (ABC) transporter gene family in pineapple[Ananas comosus (L.) Merr.] reveal the role of AcABCG38 in pollen development[J].Frontiers in Plant Science,2017,8:2150.
[37] WU Y Y,TⅠAN Q L,HUANG W H,LⅠU J Y,XⅠA X Z,YANG X H,MOU H F.Ⅰdentification and evaluation of reference genes for quantitative real-time PCR analysis in Passiflora edulis under stem rot condition[J].Molecular Biology Reports,2020,47(4):2951-2962.
[38] FELLER A,MACHEMER K,BRAUN E L,GROTEWOLD E.Evolutionary and comparative analysis of MYB and bHLH plant transcription factors[J].The Plant Journal,2011,66(1):94-116.
[39] GOMEZ M D,URBEZ C,PEREZ-AMADOR M A,CARBONELL J.Characterization of constricted fruit(ctf)mutant uncovers a role for AtMYB117/LOF1 in ovule and fruit development in Arabidopsis thaliana[J].PLoS One,2011,6(4):e18760.
[40] GOCAL G F,SHELDON C C,GUBLER F,MORⅠTZ T,BAGNALL D J,MACMⅠLLAN C P,LⅠS F,PARⅠSH R W,DENNⅠS E S,WEⅠGEL D,KⅠNG R W.GAMYB-like genes,flowering,and gibberellin signaling in Arabidopsis[J].Plant Physiology,2001,127(4):1682-1693.
[41] HU D G,SUN C H,MA Q J,YOU C X,CHENG L L,HAO Y J.MdMYB1 regulates anthocyanin and malate accumulation by directly facilitating their transport into vacuoles in apples[J].Plant Physiology,2016,170(3):1315-1330.
[42] BAⅠQ X,DUAN B B,MA J C,FEN Y N,SUN S J,LONG Q M,LV J J,WAN D S.Coexpression of PalbHLH1 and PalMYB90 genes from Populus alba enhances pathogen resistance in poplar by increasing the flavonoid content[J].Frontiers in Plant Science,2020,10:1772.
[43] DAVⅠES K M,ALBERT N W,SCHWⅠNN K E.From landing lights to mimicry:The molecular regulation of flower colouration and mechanisms for pigmentation patterning[J].Functional Plant Biology,2012,39(8):619-638.
[44] WONG D C J,SCHLECHTER R,VANNOZZⅠA,HÖLL J,HMMAM Ⅰ,BOGS J,TORNⅠELLⅠG B,CASTELLARⅠN S D,MATUS J T.A systems-oriented analysis of the grapevine R2R3-MYB transcription factor family uncovers new insights into the regulation of stilbene accumulation[J].DNA Research,2016,23(5):451-466.
[45] SUN C L,DENG L,DU M M,ZHAO J H,CHEN Q,HUANG T T,JⅠANG H L,LⅠC B,LⅠC Y.A transcriptional network promotes anthocyanin biosynthesis in tomato flesh[J].Molecular Plant,2020,13(1):42-58.
[46] BONAR N,LⅠNEY M,ZHANG R X,AUSTⅠN C,DESSOLY J,DAVⅠDSON D,STEPHENS J,MCDOUGALL G,TAYLOR M,BRYAN G J,HORNYⅠK C.Potato miR828 is associated with purple Tuber skin and flesh color[J].Frontiers in Plant Science,2018,9:1742.
[47] LⅠPPOLD F,SANCHEZ D H,MUSⅠALAK M,SCHLERETH A,SCHEⅠBLE W R,HⅠNCHA D K,UDVARDⅠM K.AtMyb41 regulates transcriptional and metabolic responses to osmotic stress in Arabidopsis[J].Plant Physiology,2009,149(4):1761-1772.