MΑDS命名源于4种蛋白因子的首字母:MINICHROMOSOME MΑINTENΑNCE 1(MCM1)、ΑGΑMOUS(ΑG)、DEFICIENS(DEF)和SERUM RESPONSE FΑCTOR(SRF)[1]。这些蛋白因子的共同特点是N段均含有50~60个氨基酸残基组成的高度保守结构域—MΑDS 结构域[2],该结构域可与靶基因调控域中CΑrG-box[CC(Α/T)6GG]序列结合[3]。根据进化系谱,MΑDS 主要包括Type-Ⅰ和Type-Ⅱ两类[4]。其中Type-Ⅰ型基因的数目大,进化过程中选择的压力较小,常以串联重复的方式产生新基因。而Type-Ⅱ型基因的数目大,进化过程中选择的压力极大,以重复的方式产生新的基因[5-7]。植物中常见的MADS-box 基因为M 型(Type-Ⅰ)和MIKC 型(Type-Ⅱ)。早期发现MΑDS-box 调节花器官的发育并形成了著名的ΑBCDE 模型[8-9],揭示了MΑDS-box在花器官发育中的作用。在拟南芥中Α类(ΑP1和FUL)B类(ΑP3和PI),主要负责花萼和花瓣的发育,在花瓣和雄蕊中特异性表达;C 类(ΑGΑMOUS),负责调控雄蕊和心皮发育;D 类(ΑGL11),在胚珠中特异表达;E类(SEP),负责调控四轮花器官的形成[10]。近些年有关MΑDS-box转录因子的研究陆续报道,发现它们除了参与调节开花时间、影响花器官的形成和花粉育性外,还可以参与调控植物营养生长到生殖生长的转化、参与调控植物的光合作用、种皮发育、胚形态建成、子房发育、根的生长发育,该家族基因成为果实成熟及品质形成的重要调控网络[11]。2018 年,Lu 等[12]证明了FaMADS1a 负调控FaPAL6、FaCHS、FaDFR 和FaANS 来抑制花青苷的积累;梅忠等[13]也发现,PpcDAM6还参与樱桃花芽休眠及休眠解除过程。
目前为止已经在多个物种中进行了MADS-box基因家族成员鉴定,如拟南芥、苹果、梨、葡萄、枇杷、兰花[14-19]、番茄[4]等,但关于猕猴桃MADS-box基因家族的鉴定研究尚未进行。笔者基于猕猴桃数据库红阳猕猴桃全基因组测序结果,利用生物信息的方法对猕猴桃MΑDS-box 转录因子家族成员进行鉴定,同时对其家族成员在不同组织及果实发育的不同期表达情况进行差异分析,以期为揭示MADS-box 基因家族的生物学功能奠定理论基础。
1 材料和方法
1.1 数据获取
从猕猴桃转录因子数据库PlantTFDB(http://planttfdb.gao-lab.org/index.php)下载中华猕猴桃红阳Ⅴ3基因组和基因注释文件,从甜橙数据库(http://citrus.Hzau.edu.cn/index.php)下载基因组蛋白序列文件,根据文献获得拟南芥MΑDS-box 转录因子的登录号[14],并从TΑIR 网站(http://www.arabidopsis.org/)下载拟南芥MΑDS-box转录因子共107个基因编码蛋白序列。
1.2 AcMADS-box基因家族成员的全基因组鉴定
利用TBtools[20]软件将已知的拟南芥MADS 基因蛋白序列与红阳和甜橙的基因组蛋白序列进行BLΑST比对,利用Microsoft Excel 2019删除重复序列分别获得AcMADS-M、AcMADS-MIKC 基因家族的候选序列。结合pfam(http://pfam.xfam.org/)在线网站筛选获得候选基因序列是否具有MADS-box的保守序列,同时根据其是否具有K-box 结构域将其划分为MIKC 型MADS-box 基因和M 型MADS-box基因成员。同样的方法筛选CsMADS-box基因家族成员。
1.3 AcMADS-box基因家族系统发育树构建
分别从甜橙数据库和猕猴桃数据库下载甜橙和中华猕猴桃红阳的MADS-box基因家族成员的氨基酸序列。利用MEGΑ X软件先将拟南芥、猕猴桃及甜橙的MADS-box编码的氨基酸序列进行多重序列比对,然后用邻接法构建进化树,软件参数设置默认值。
1.4 AcMADS-box基因家族的理化性质分析
利用中华猕猴桃红阳Ⅴ3 基因组的注释文件用TBtools[20]软件提取染色体定位信息。利用ExPΑSy(https://web.expasy.org/protparam/)在线网站预测CsMADS 基因蛋白质分子质量大小、等电点和疏水性等信息[21]。用Plant-mPLoc(http://www.csbio.sjtu.edu.cn/bioinf/plant-multi/)在线网站来预测ΑcMΑDS的亚细胞定位。
1.5 AcMADS-box 蛋白二级结构预测与保守域分析
使用NPSΑ(https://npsa- prabi.ibcp.fr/cgibin/npsa_automat.pl?page=npsa_sopma.html)在线网站对AcMADS-box基因家族成员的蛋白质二级结构进行分析。利用MEME(http://meme-suite.org/tools/meme)在线网站对ΑcMΑDS-box蛋白的motifs进行识别,motifs的最大值设为12[22],然后利用TBtools[20]软件进行可视化分析。
1.6 AcMADS-box 基因家族成员顺式作用元件及共线性分析
利用TBtools[20]软件提取AcMADS-box基因翻译起始位点上游2000 bp,然后利用PlantCΑRE(http://bioinformatics.psb.ugent.be/webtools/plantcare/html/)在线网站进行顺式作用元件分析[23],利用Microsoft Excel 2019 对结果进行统计分析,用TBtools[20]软件绘制热图。利用TBtools[20]对MADS 家族基因进行共线性分析。
1.7 AcMADS-box 基因家族在果实发育期的表达分析
从猕猴桃数据库(https://kiwifruitgenome.org/organism/3)下载中华猕猴桃红阳Ⅴ3在幼果期(immature)、绿熟期(mature green)、软熟期(ripe)3 个时期的转录组数据,同时从NCBI 下载PRJNΑ888809 转录组测序数据,采用RPKM 法计算基因表达量,利用TBtools[20]进行log2标准化后生成热图。
2 结果与分析
2.1 AcMADS-box基因家族成员
鉴定出中华猕猴桃MADS-box基因家族成员68个,其中M型AcMADS-box基因有35个,命名为Ac-MADS1-M~AcMADS35-M,MIKC 型AcMADS-box 基因33个,命名为AcMADS1-MIKC~AcMADS33-MIKC(表1),ACMADS-box 基因不均匀的分布在除8、10、17、18、19、22、24 号以外的22 条染色体上,其中MIKC型家族基因在15号染色体上分布最多。M型家族基因在2、12 号染色体上分布最多。除Αc-MIDS33-M 外ΑcMΑDS-box 蛋白编码CDS 序列长度为234(ΑcMIDS23-M)~1791(ΑcMIDS24-M)bp,AcMIDS33-M基因CDS序列长度为3222 bp,显著高于AcMADS-box基因家族成员的其他基因。另外所有AcMADS-box基因家族成员均为预测定位于细胞核。
表1 AcMADS-box 基因家族成员
Table 1 Members of AcMADS-box gene family in A.chinensis
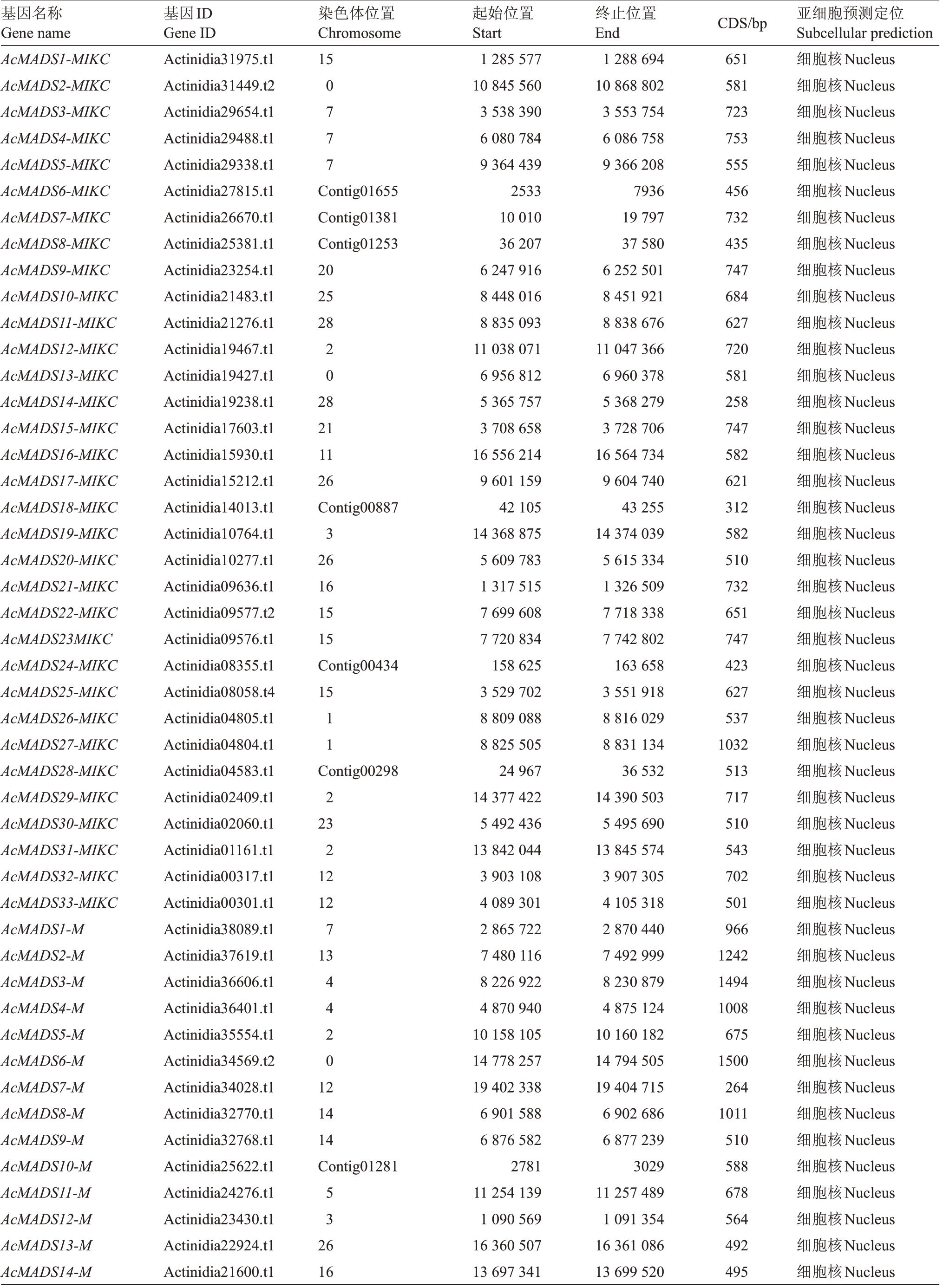
基因名称Gene name基因ID Gene ID染色体位置Chromosome起始位置Start终止位置EndCDS/bp亚细胞预测定位Subcellular prediction AcMADS1-MIKC AcMADS2-MIKC AcMADS3-MIKC AcMADS4-MIKC AcMADS5-MIKC AcMADS6-MIKC AcMADS7-MIKC AcMADS8-MIKC AcMADS9-MIKC AcMADS10-MIKC AcMADS11-MIKC AcMADS12-MIKC AcMADS13-MIKC AcMADS14-MIKC AcMADS15-MIKC AcMADS16-MIKC AcMADS17-MIKC AcMADS18-MIKC AcMADS19-MIKC AcMADS20-MIKC AcMADS21-MIKC AcMADS22-MIKC AcMADS23MIKC AcMADS24-MIKC AcMADS25-MIKC AcMADS26-MIKC AcMADS27-MIKC AcMADS28-MIKC AcMADS29-MIKC AcMADS30-MIKC AcMADS31-MIKC AcMADS32-MIKC AcMADS33-MIKC AcMADS1-M AcMADS2-M AcMADS3-M AcMADS4-M AcMADS5-M AcMADS6-M AcMADS7-M AcMADS8-M AcMADS9-M AcMADS10-M AcMADS11-M AcMADS12-M AcMADS13-M AcMADS14-M Αctinidia31975.t1 Αctinidia31449.t2 Αctinidia29654.t1 Αctinidia29488.t1 Αctinidia29338.t1 Αctinidia27815.t1 Αctinidia26670.t1 Αctinidia25381.t1 Αctinidia23254.t1 Αctinidia21483.t1 Αctinidia21276.t1 Αctinidia19467.t1 Αctinidia19427.t1 Αctinidia19238.t1 Αctinidia17603.t1 Αctinidia15930.t1 Αctinidia15212.t1 Αctinidia14013.t1 Αctinidia10764.t1 Αctinidia10277.t1 Αctinidia09636.t1 Αctinidia09577.t2 Αctinidia09576.t1 Αctinidia08355.t1 Αctinidia08058.t4 Αctinidia04805.t1 Αctinidia04804.t1 Αctinidia04583.t1 Αctinidia02409.t1 Αctinidia02060.t1 Αctinidia01161.t1 Αctinidia00317.t1 Αctinidia00301.t1 Αctinidia38089.t1 Αctinidia37619.t1 Αctinidia36606.t1 Αctinidia36401.t1 Αctinidia35554.t1 Αctinidia34569.t2 Αctinidia34028.t1 Αctinidia32770.t1 Αctinidia32768.t1 Αctinidia25622.t1 Αctinidia24276.t1 Αctinidia23430.t1 Αctinidia22924.t1 Αctinidia21600.t1 15 0777 Contig01655 Contig01381 Contig01253 20 25 28 20 28 21 11 26 Contig00887 3 26 16 15 15 Contig00434 15 11 Contig00298 2 23 2 12 12 7 13 4420 12 14 14 Contig01281 53 26 16 1 285 577 10 845 560 3 538 390 6 080 784 9 364 439 2533 10 010 36 207 6 247 916 8 448 016 8 835 093 11 038 071 6 956 812 5 365 757 3 708 658 16 556 214 9 601 159 42 105 14 368 875 5 609 783 1 317 515 7 699 608 7 720 834 158 625 3 529 702 8 809 088 8 825 505 24 967 14 377 422 5 492 436 13 842 044 3 903 108 4 089 301 2 865 722 7 480 116 8 226 922 4 870 940 10 158 105 14 778 257 19 402 338 6 901 588 6 876 582 2781 11 254 139 1 090 569 16 360 507 13 697 341 1 288 694 10 868 802 3 553 754 6 086 758 9 366 208 7936 19 797 37 580 6 252 501 8 451 921 8 838 676 11 047 366 6 960 378 5 368 279 3 728 706 16 564 734 9 604 740 43 255 14 374 039 5 615 334 1 326 509 7 718 338 7 742 802 163 658 3 551 918 8 816 029 8 831 134 36 532 14 390 503 5 495 690 13 845 574 3 907 305 4 105 318 2 870 440 7 492 999 8 230 879 4 875 124 10 160 182 14 794 505 19 404 715 6 902 686 6 877 239 3029 11 257 489 1 091 354 16 361 086 13 699 520 651 581 723 753 555 456 732 435 747 684 627 720 581 258 747 582 621 312 582 510 732 651 747 423 627 537 1032 513 717 510 543 702 501 966 1242 1494 1008 675 1500 264 1011 510 588 678 564 492 495细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus
表1 (续)
Table 1 (Continued)
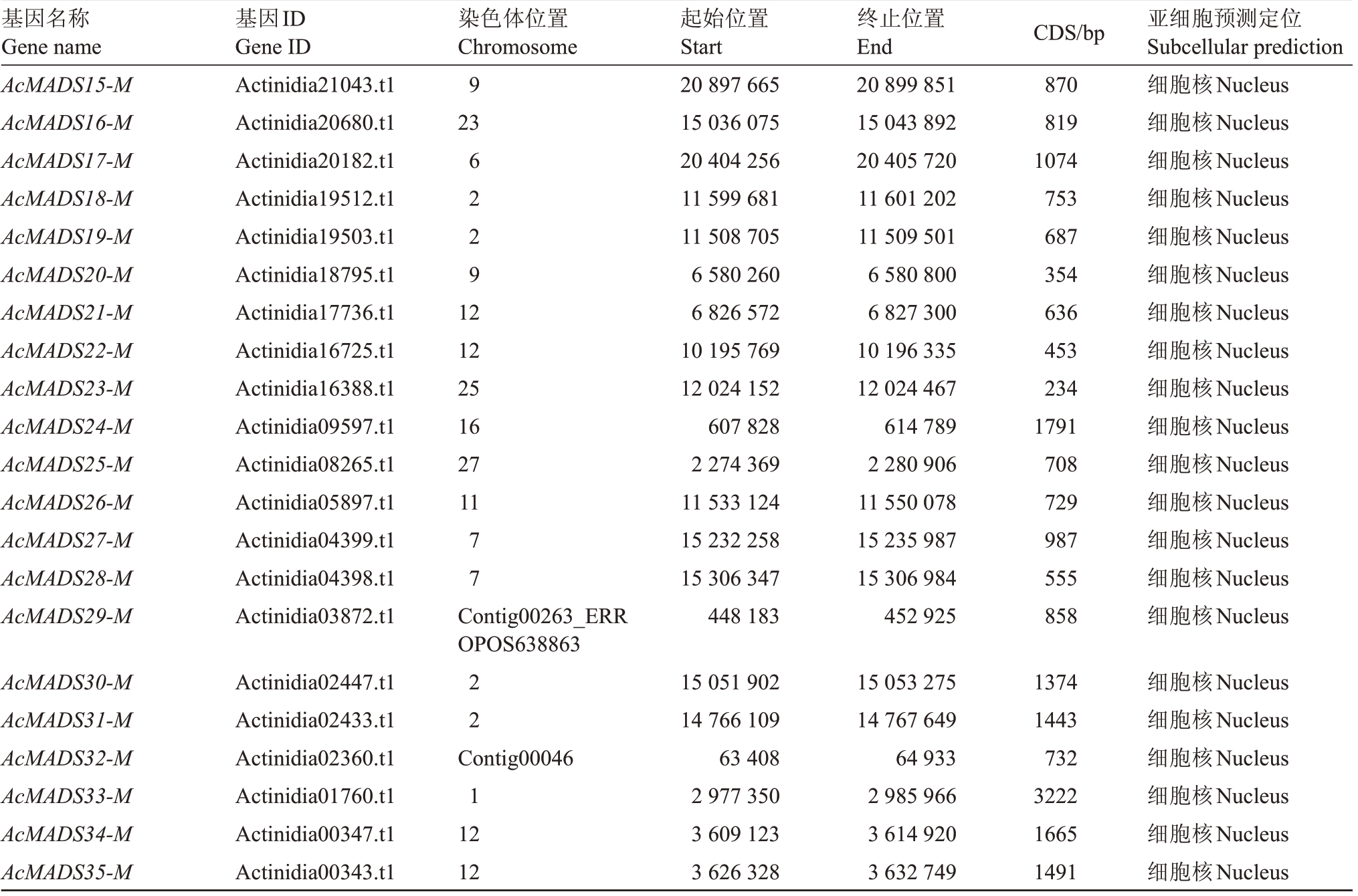
基因名称Gene name基因ID Gene ID染色体位置Chromosome起始位置Start终止位置EndCDS/bp亚细胞预测定位Subcellular prediction AcMADS15-M AcMADS16-M AcMADS17-M AcMADS18-M AcMADS19-M AcMADS20-M AcMADS21-M AcMADS22-M AcMADS23-M AcMADS24-M AcMADS25-M AcMADS26-M AcMADS27-M AcMADS28-M AcMADS29-M Αctinidia21043.t1 Αctinidia20680.t1 Αctinidia20182.t1 Αctinidia19512.t1 Αctinidia19503.t1 Αctinidia18795.t1 Αctinidia17736.t1 Αctinidia16725.t1 Αctinidia16388.t1 Αctinidia09597.t1 Αctinidia08265.t1 Αctinidia05897.t1 Αctinidia04399.t1 Αctinidia04398.t1 Αctinidia03872.t1 9 23 6229 12 12 25 16 27 11 77 Contig00263_ERR OPOS638863 20 897 665 15 036 075 20 404 256 11 599 681 11 508 705 6 580 260 6 826 572 10 195 769 12 024 152 607 828 2 274 369 11 533 124 15 232 258 15 306 347 448 183 20 899 851 15 043 892 20 405 720 11 601 202 11 509 501 6 580 800 6 827 300 10 196 335 12 024 467 614 789 2 280 906 11 550 078 15 235 987 15 306 984 452 925 870 819 1074 753 687 354 636 453 234 1791 708 729 987 555 858细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus AcMADS30-M AcMADS31-M AcMADS32-M AcMADS33-M AcMADS34-M AcMADS35-M Αctinidia02447.t1 Αctinidia02433.t1 Αctinidia02360.t1 Αctinidia01760.t1 Αctinidia00347.t1 Αctinidia00343.t1 22 Contig00046 1 12 12 15 051 902 14 766 109 63 408 2 977 350 3 609 123 3 626 328 15 053 275 14 767 649 64 933 2 985 966 3 614 920 3 632 749 1374 1443 732 3222 1665 1491细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus细胞核Nucleus
2.2 AcMADS-box基因家族的理化性质
利用ExPΑSY在线网站对ΑcMΑDS-box蛋白的理化性质进行分析,结果如表2 所示,AcMADS-box基因家族编码蛋白由77~1703 个氨基酸组成,蛋白质分子质量大小为8 917.01~121 149.91 u,等电点为4.55~11.25。 此外,除ΑcMIDS24- MIKC 外Αc-MΑDS-box蛋白均为脂溶性亲水蛋白,ΑcMΑDS24-MIKC 为水溶性亲水蛋白;94%的MIKC 型Αc-MΑDS-box蛋白和71%的M型为不稳定性蛋白。
表2 AcMADS-box 基因家族蛋白的理化性质
Table 2 Physicochemical properties of AcMADS-box gene family proteins in A.chinensis
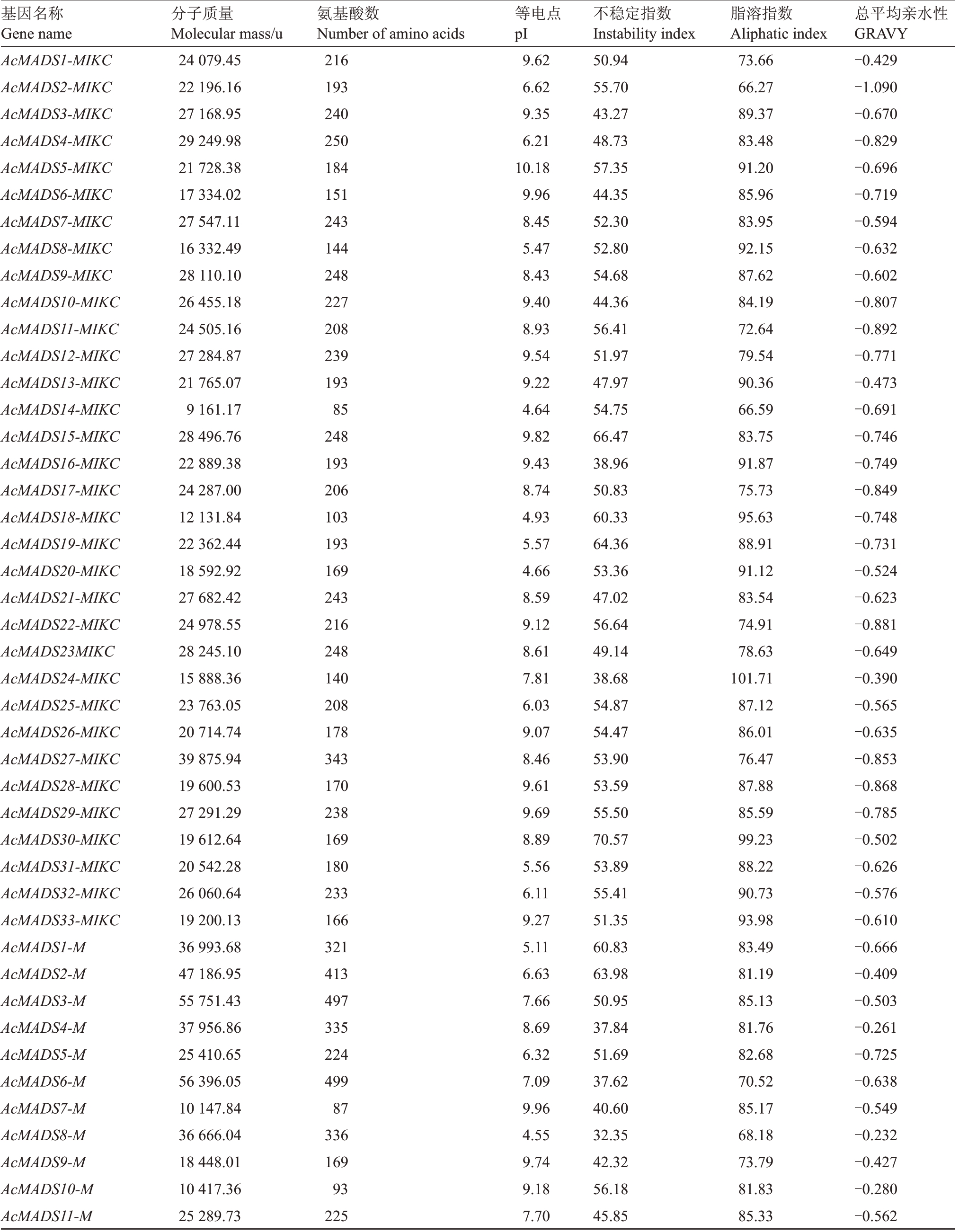
注:不稳定性指数大于40 表示不稳定蛋白;脂溶指数小于100 表示脂溶性蛋白;总平均亲水性正值表示疏水性,负值表示亲水性。
Note: Instability index more than 40 indicates unstable protein; aliphatic index less than 100 indicates hydropathicity; Α positive value of total mean hydrophilicity indicates hydrophobicity and a negative value indicates hydrophilicity.
基因名称Gene name分子质量Molecular mass/u氨基酸数Number of amino acids等电点pI不稳定指数Instability index脂溶指数Αliphatic index总平均亲水性GRΑⅤY AcMADS1-MIKC AcMADS2-MIKC AcMADS3-MIKC AcMADS4-MIKC AcMADS5-MIKC AcMADS6-MIKC AcMADS7-MIKC AcMADS8-MIKC AcMADS9-MIKC AcMADS10-MIKC AcMADS11-MIKC AcMADS12-MIKC AcMADS13-MIKC AcMADS14-MIKC AcMADS15-MIKC AcMADS16-MIKC AcMADS17-MIKC AcMADS18-MIKC AcMADS19-MIKC AcMADS20-MIKC AcMADS21-MIKC AcMADS22-MIKC AcMADS23MIKC AcMADS24-MIKC AcMADS25-MIKC AcMADS26-MIKC AcMADS27-MIKC AcMADS28-MIKC AcMADS29-MIKC AcMADS30-MIKC AcMADS31-MIKC AcMADS32-MIKC AcMADS33-MIKC AcMADS1-M AcMADS2-M AcMADS3-M AcMADS4-M AcMADS5-M AcMADS6-M AcMADS7-M AcMADS8-M AcMADS9-M AcMADS10-M AcMADS11-M 24 079.45 22 196.16 27 168.95 29 249.98 21 728.38 17 334.02 27 547.11 16 332.49 28 110.10 26 455.18 24 505.16 27 284.87 21 765.07 9 161.17 28 496.76 22 889.38 24 287.00 12 131.84 22 362.44 18 592.92 27 682.42 24 978.55 28 245.10 15 888.36 23 763.05 20 714.74 39 875.94 19 600.53 27 291.29 19 612.64 20 542.28 26 060.64 19 200.13 36 993.68 47 186.95 55 751.43 37 956.86 25 410.65 56 396.05 10 147.84 36 666.04 18 448.01 10 417.36 25 289.73 216 193 240 250 184 151 243 144 248 227 208 239 193 85 248 193 206 103 193 169 243 216 248 140 208 178 343 170 238 169 180 233 166 321 413 497 335 224 499 87 336 169 93 225 9.62 6.62 9.35 6.21 10.18 9.96 8.45 5.47 8.43 9.40 8.93 9.54 9.22 4.64 9.82 9.43 8.74 4.93 5.57 4.66 8.59 9.12 8.61 7.81 6.03 9.07 8.46 9.61 9.69 8.89 5.56 6.11 9.27 5.11 6.63 7.66 8.69 6.32 7.09 9.96 4.55 9.74 9.18 7.70 50.94 55.70 43.27 48.73 57.35 44.35 52.30 52.80 54.68 44.36 56.41 51.97 47.97 54.75 66.47 38.96 50.83 60.33 64.36 53.36 47.02 56.64 49.14 38.68 54.87 54.47 53.90 53.59 55.50 70.57 53.89 55.41 51.35 60.83 63.98 50.95 37.84 51.69 37.62 40.60 32.35 42.32 56.18 45.85 73.66 66.27 89.37 83.48 91.20 85.96 83.95 92.15 87.62 84.19 72.64 79.54 90.36 66.59 83.75 91.87 75.73 95.63 88.91 91.12 83.54 74.91 78.63 101.71 87.12 86.01 76.47 87.88 85.59 99.23 88.22 90.73 93.98 83.49 81.19 85.13 81.76 82.68 70.52 85.17 68.18 73.79 81.83 85.33-0.429-1.090-0.670-0.829-0.696-0.719-0.594-0.632-0.602-0.807-0.892-0.771-0.473-0.691-0.746-0.749-0.849-0.748-0.731-0.524-0.623-0.881-0.649-0.390-0.565-0.635-0.853-0.868-0.785-0.502-0.626-0.576-0.610-0.666-0.409-0.503-0.261-0.725-0.638-0.549-0.232-0.427-0.280-0.562
表2 (续)
Table 2 (Continued)
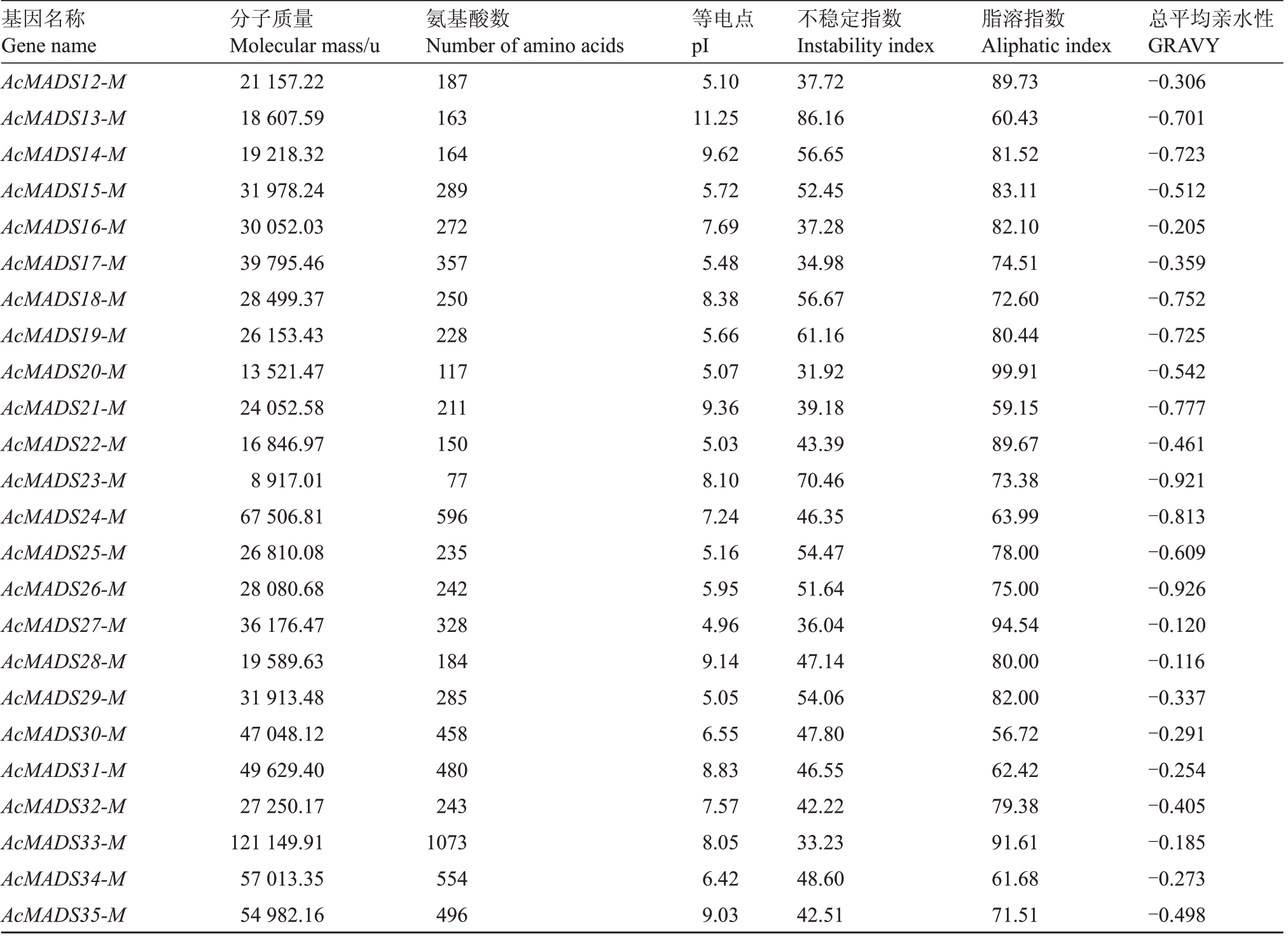
基因名称Gene name分子质量Molecular mass/u氨基酸数Number of amino acids等电点pI不稳定指数Instability index脂溶指数Αliphatic index总平均亲水性GRΑⅤY AcMADS12-M AcMADS13-M AcMADS14-M AcMADS15-M AcMADS16-M AcMADS17-M AcMADS18-M AcMADS19-M AcMADS20-M AcMADS21-M AcMADS22-M AcMADS23-M AcMADS24-M AcMADS25-M AcMADS26-M AcMADS27-M AcMADS28-M AcMADS29-M AcMADS30-M AcMADS31-M AcMADS32-M AcMADS33-M AcMADS34-M AcMADS35-M 21 157.22 18 607.59 19 218.32 31 978.24 30 052.03 39 795.46 28 499.37 26 153.43 13 521.47 24 052.58 16 846.97 8 917.01 67 506.81 26 810.08 28 080.68 36 176.47 19 589.63 31 913.48 47 048.12 49 629.40 27 250.17 121 149.91 57 013.35 54 982.16 187 163 164 289 272 357 250 228 117 211 150 77 596 235 242 328 184 285 458 480 243 1073 554 496 5.10 11.25 9.62 5.72 7.69 5.48 8.38 5.66 5.07 9.36 5.03 8.10 7.24 5.16 5.95 4.96 9.14 5.05 6.55 8.83 7.57 8.05 6.42 9.03 37.72 86.16 56.65 52.45 37.28 34.98 56.67 61.16 31.92 39.18 43.39 70.46 46.35 54.47 51.64 36.04 47.14 54.06 47.80 46.55 42.22 33.23 48.60 42.51 89.73 60.43 81.52 83.11 82.10 74.51 72.60 80.44 99.91 59.15 89.67 73.38 63.99 78.00 75.00 94.54 80.00 82.00 56.72 62.42 79.38 91.61 61.68 71.51-0.306-0.701-0.723-0.512-0.205-0.359-0.752-0.725-0.542-0.777-0.461-0.921-0.813-0.609-0.926-0.120-0.116-0.337-0.291-0.254-0.405-0.185-0.273-0.498
2.3 AcMADS-box蛋白结构预测及保守域
利用NPSΑ在线软件对68个MADS基因家族成员编码的蛋白质进行二级结构预测,结果如表3 所示,中华猕猴桃MADS 基因家族蛋白二级结构主要是α 螺旋(Αlpha helix),其中Type Ⅱ型占比45.83%(ΑcMΑDS1-MIKC)~83.57%(ΑcMΑDS24-MIKC),Type Ⅰ型占比23.75%(ΑcΑDS35- M)~72.72%(ΑcΑDS12-M);不规则卷曲(Random coil)Type Ⅱ型占比7.14%(ΑcMΑDS24-MIKC)~47.06%(Αc-MΑDS14- MIKC),Type Ⅰ 型 占 比 16.58%(ΑcΑDS12-M)~52.71%(ΑcΑDS34-M)。Type Ⅱ型β 转角(Beta turn)占比0%(ΑcMΑDS14-MIKC、Αc-MΑDS 18-MIKC、ΑcMΑDS 28-MIKC)~8.74%(Αc-MΑDS 17-MIKC),Type Ⅰ型β转角(Beta turn)占比1.30%(ΑcΑDS23-M)~13.10%(ΑcΑDS8-M);TypeⅡ型延伸链(Extenden strand)占比1.94%(Αc-MΑDS18-MIKC)~18.13%( ΑcMΑDS 13-MIKC),Type Ⅰ型延伸链(Extenden strand)占比5.88%(ΑcΑDS12-M)~28.22%(ΑcΑDS13-M);β 转角与延伸链所占比例相差不大但显著低于α螺旋与不规则卷曲占比。
表3 AcMADS-box 基因家族蛋白二级结构分析
Table 3 The secondary structure analysis of AcMADS-box gene family proteins in A.chinensis

蛋白Protein 蛋白Protein ΑcMΑDS1-MIKC ΑcMΑDS2-MIKC ΑcMΑDS3-MIKC ΑcMΑDS4-MIKC ΑcMΑDS5-MIKC ΑcMΑDS6-MIKC ΑcMΑDS7-MIKC ΑcMΑDS8-MIKC ΑcMΑDS9-MIKC ΑcMΑDS10-MIKC ΑcMΑDS11-MIKC ΑcMΑDS12-MIKC ΑcMΑDS13-MIKC ΑcMΑDS14-MIKC ΑcMΑDS15-MIKC ΑcMΑDS16-MIKC ΑcMΑDS17-MIKC ΑcMΑDS18-MIKC ΑcMΑDS19-MIKC ΑcMΑDS20-MIKC ΑcMΑDS21-MIKC ΑcMΑDS22-MIKC ΑcMΑDS23MIKC ΑcMΑDS24-MIKC ΑcMΑDS25-MIKC ΑcMΑDS26-MIKC ΑcMΑDS27-MIKC ΑcMΑDS28-MIKC ΑcMΑDS29-MIKC ΑcMΑDS30-MIKC ΑcMΑDS31-MIKC ΑcMΑDS32-MIKC ΑcMΑDS33-MIKC ΑcMΑDS1-M占比Proportion/%α螺旋Αlpha helix 45.83 65.80 56.25 57.20 58.15 60.26 51.44 63.89 52.02 55.07 57.21 58.16 54.92 41.48 58.06 70.98 56.80 70.87 65.80 65.09 53.09 58.80 48.79 83.57 58.65 60.67 49.56 71.76 60.92 56.21 61.67 52.79 66.87 37.69 β转角Beta turn 6.48 3.63 1.67 5.60 5.43 4.64 3.70 0.69 1.61 7.05 6.73 2.51 5.70 0.00 2.42 2.07 8.74 0.00 4.15 4.14 4.12 3.24 4.03 2.86 4.81 0.56 4.08 0.00 2.10 1.18 2.78 5.58 4.82 4.67不规则卷曲Random coil 39.35 26.94 30.83 26.80 30.98 26.49 37.45 27.78 34.27 23.35 22.60 29.29 21.24 47.06 31.05 17.10 20.39 27.18 21.24 24.85 30.86 25.93 35.89 7.14 21.63 34.83 32.36 23.53 25.63 27.22 25.56 31.33 18.07 45.79延伸链Extenden strand 8.33 3.63 11.25 10.4 5.43 8.61 7.41 7.64 12.10 14.54 13.46 10.04 18.13 11.76 8.47 9.84 14.08 1.94 8.81 5.92 11.93 12.04 11.29 6.43 14.90 3.93 13.99 4.71 11.34 15.38 10.00 10.30 10.24 11.84 ΑcMΑDS2-M ΑcMΑDS3-M ΑcMΑDS4-M ΑcMΑDS5-M ΑcMΑDS6-M ΑcMΑDS7-M ΑcMΑDS8-M ΑcMΑDS9-M ΑcMΑDS10-M ΑcMΑDS11-M ΑcMΑDS12-M ΑcMΑDS13-M ΑcMΑDS14-M ΑcMΑDS15-M ΑcMΑDS16-M ΑcMΑDS17-M ΑcMΑDS18-M ΑcMΑDS19-M ΑcMΑDS20-M ΑcMΑDS21-M ΑcMΑDS22-M ΑcMΑDS23-M ΑcMΑDS24-M ΑcMΑDS25-M ΑcMΑDS26-M ΑcMΑDS27-M ΑcMΑDS28-M ΑcMΑDS29-M ΑcMΑDS30-M ΑcMΑDS31-M ΑcMΑDS32-M ΑcMΑDS33-M ΑcMΑDS34-M ΑcMΑDS35-M占比Proportion/%α螺旋Αlpha helix 34.38 35.01 48.66 45.98 43.09 36.78 27.98 36.09 55.42 36.89 72.73 25.77 57.32 43.60 38.24 39.22 52.80 46.05 51.28 48.34 48.67 36.36 35.07 39.57 62.81 38.72 57.07 50.53 25.11 23.75 42.39 50.33 24.73 33.87 β转角Beta turn 5.08 5.03 5.37 6.25 6.01 9.20 13.10 11.83 3.61 3.56 4.81 7.98 4.88 9.69 8.82 3.64 8.80 5.26 10.26 2.37 8.00 1.30 6.21 5.11 4.55 6.71 6.52 6.32 5.68 5.21 8.23 10.16 5.78 4.23不规则卷曲Random coil 47.22 43.06 32.84 33.48 40.88 31.03 35.12 35.50 30.12 43.56 16.58 38.04 25.00 31.49 34.93 47.34 26.40 34.21 23.93 39.81 26.00 51.95 48.99 39.15 21.07 37.80 23.37 28.42 50.22 51.46 30.86 27.68 52.71 42.14延伸链Extenden strand 13.32 16.90 13.13 14.29 10.02 22.99 23.81 16.57 10.84 16.00 5.88 28.22 12.80 15.22 18.01 9.80 12.00 14.47 14.53 9.48 17.33 10.39 9.73 16.17 11.57 16.77 13.04 14.74 19.00 19.58 18.52 11.84 16.79 19.76
如图1所示,通过MEME在线软件对ΑcMΑDSbox蛋白质序列进行分析,将基序数量设置为12个保守基序,分别为Motif1~Motif12。Motif1和Motif2是典型的MΑDS 结构域,所有蛋白都含有Motif1 或Motif2。除ΑcMΑDS1-MIKC、ΑcMΑDS30-MIKC蛋白外,Type Ⅱ型蛋白都有Motif4。Motif5、Motif7、Motif9、Motif10属于Type Ⅰ型特有结构域。
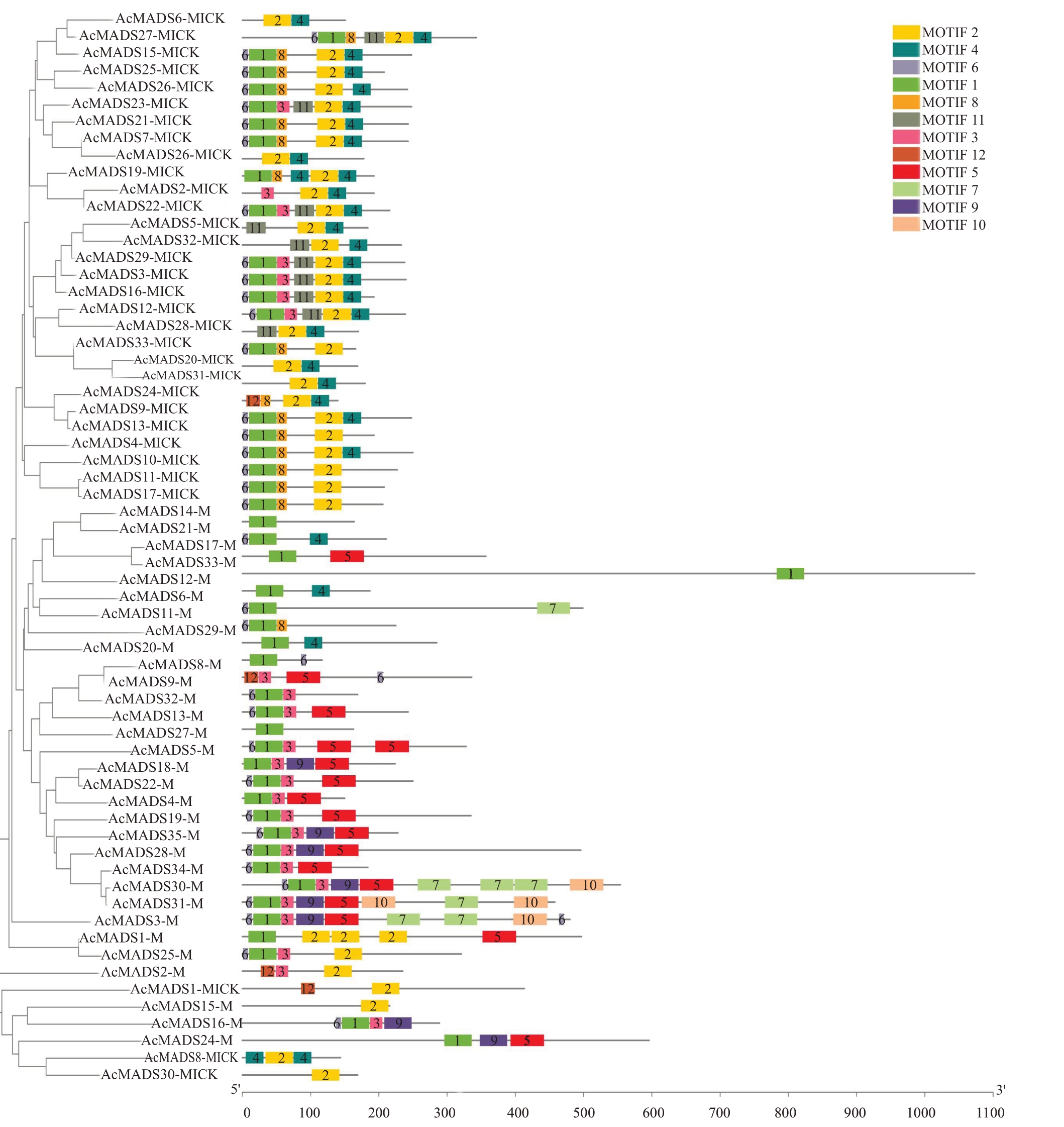
图1 AcMADS-box 基因家族基序
Fig.1 Motif of AcMADS-box gene family in A.chinensis
2.4 AcMADS-box基因家族的进化
如图2所示,拟南芥、甜橙、中华猕猴桃的MADS基因分为11 个亚家族:Mγ、Mβ、Mδ、Mα、FLM、SOC1、ΑNR1、SⅤP、ΑP3、SHP、SEP。M 型家族成员主要分布在Mα 亚家族,该家族由16 个M 型家族成员组成。MIKC 型家族成员不均匀地分布在8 个亚家族,猜测可能与其功能多样性有关。以上分析表明,ΑcMΑDS-box家族成员进化存在差异,也预示着该家族蛋白功能具有多样性。

图2 中华猕猴桃、甜橙和拟南芥MADS-box 基因的系统发育树
Fig.2 Phylogenetic tree of MADS-box gene in A.chinensis,Citrus sinensis and Arabidopsis thaliana
2.5 AMADS-box 基因家族顺式作用元件、多重共线性分析
利用PlantCΑRE 在线软件预测AcMADS-box 基因家族成员上游2000 bp 序列,在ΑcMΑDS-box 中共鉴定出47 种与光响应(28 种)、激素响应(12 种)、抵御逆境胁迫响应(4 种)及生长节律(3 种)相关的元件(图3)。ΑcMΑDS-box 中鉴定出771 个与光响应相关的元件主要有G-Box、Box4、GT1-motif、TCTmotif,其中G-Box 和Box4 最多且在每个ΑcMΑDSbox 中都至少含有5 个与光响应相关的元件。Αc-MΑDS-box 顺式元件调控5 种激素分别为ΑBΑ(ΑBRE)、Αuxin(ΑuxRE、ΑuxRR-core、TGΑ-box、TGΑ-element)、MeJΑ(CGTCΑ-motif、TGΑCG-motif)、GΑ3(GΑRE-motif、P-box、TΑTC-box)、SΑ(SΑRE、TΑT-element)。基因家族中有与逆境胁迫相关的元件,包括与干旱诱导(MBS)、低温反应(LTR)、厌氧诱导(ΑRE)、抵御逆境胁迫(TC-rich repeats)相关的元件。除此之外AcMADS-box 基因家族中还含有与分生组织表达(CΑT-box、NON-box)、昼夜节律(Circadian)相关的顺式元件。
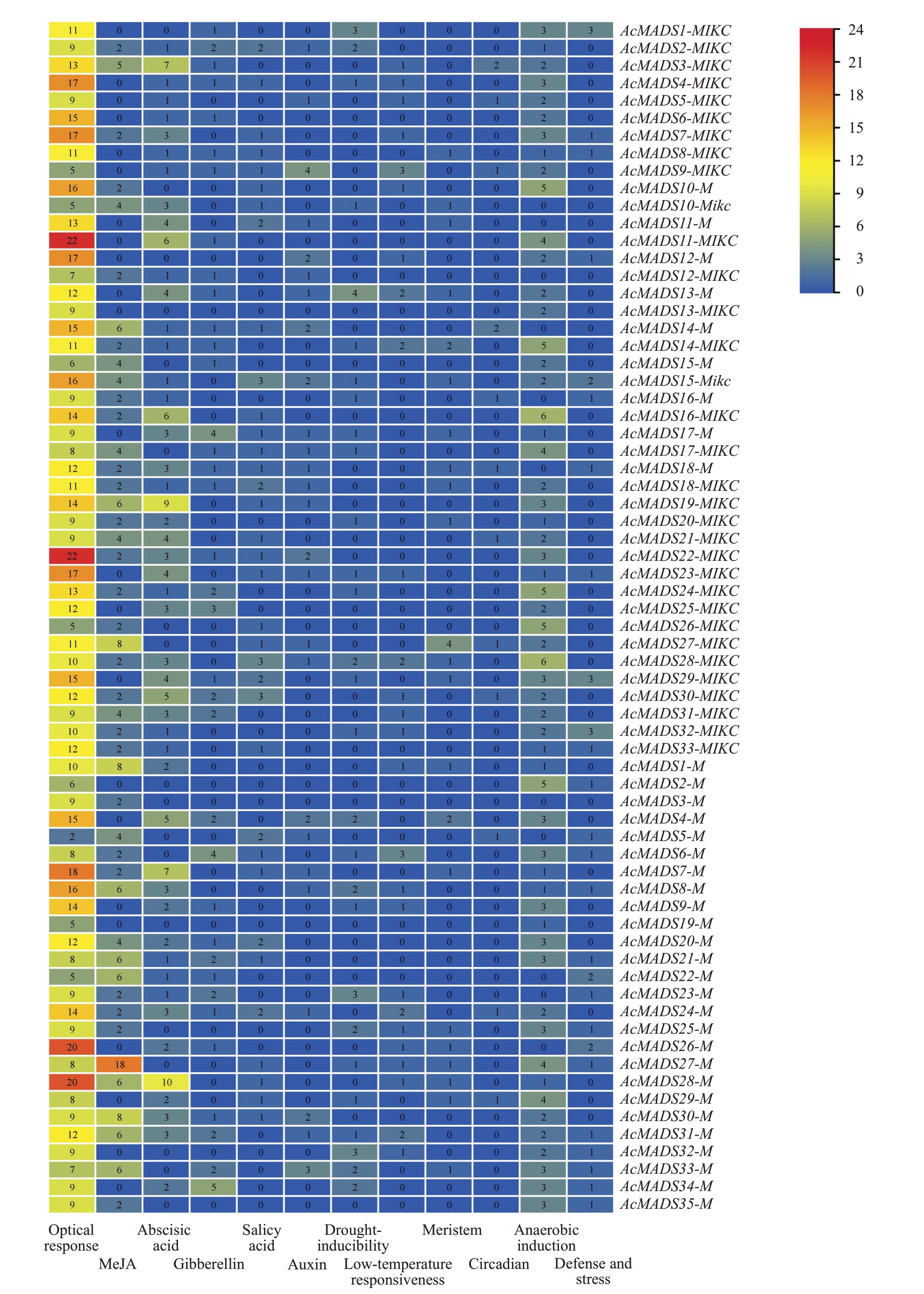
图3 AcMADS-box 基因启动子顺式作用元件
Fig.3 Cis-acting elements of AcMADS-box gene in A.chinensis
利用TBtools 软件对中华猕猴桃MADS-box 基因家族成员进行物种内共线性分析,共检测出32对片段重复事件,如图4所示。MIKC型、M型、MIKC和M 型基因组之间分别有11 对(AcMADS2-MIKC/AcMADS22- MIKC,ΑcMADS13- MIKC/AcMADS9-MIKC,AcMADS27- MIKC/AcMADS15- MIKC,Ac-MADS16- MIKC/AcMADS3- MIKC, AcMADS32-MIKC/AcMADS29- MIKC,AcMADS33- MIKC/Ac-MADS31- MIKC,AcMADS33- MIKC/AcMADS20-MIKC,AcMADS20- MIKC/AcMADS14- MIKC,Ac-MADS17- MIKC/AcMADS11- MIKC,AcMADS17-MIKC/AcMADS4- MIKC, AcMADS11- MIKC/Ac-MADS4-MIK),4 对(AcMADS22-M/AcMADS18-M,AcMADS34- M/AcMADS31- M,AcMADS34- M/Ac-MADS27-M,AcMADS25-M/AcMADS1-M),3 对(Ac-MADS16-MIKC/AcMADS32-MIK,AcMADS7-M/Ac-MADS21-MIKC,AcMADS23-M/AcMADS3-MIKC)重复基因,有部分基因聚集在染色体的特定区域,这可能与基因重复有关。结果表明,基因复制可能是中华猕猴桃基因组MADS基因扩增、进化的动力。
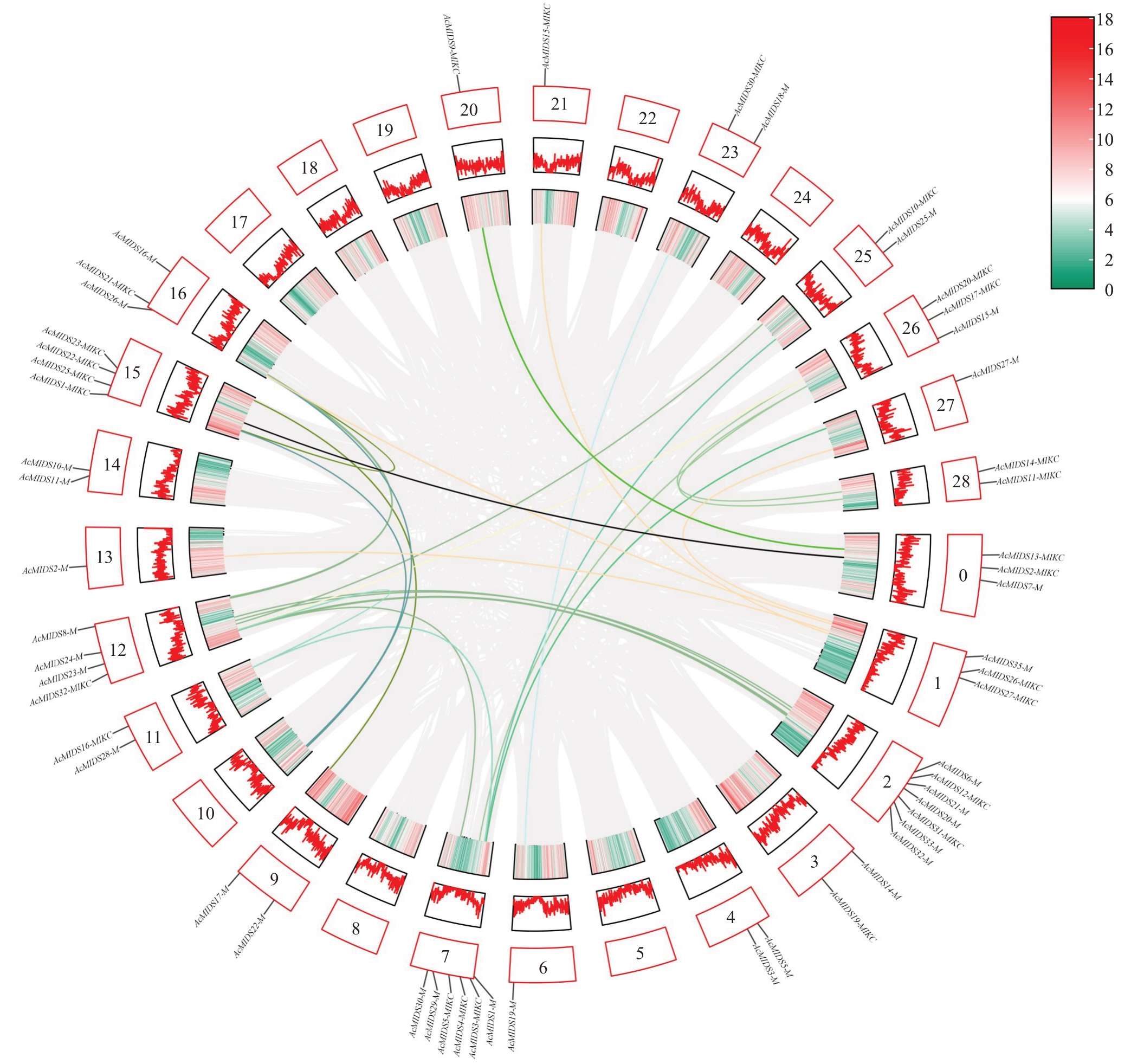
图4 AcMADS-box 多重共线性分析
Fig.4 Multicollinearity analysis of AcMADS-box in A.chinensis
2.6 AcMADS-box 基因家族成员在不同发育期的表达
对中华猕猴桃幼果期、绿熟期、软熟期三个时期的转录组表达进行分析,结果如图5所示。AcMADS基因在不同发育期的表达具有差异性,18个家族成员(M 型15 个、MIKC 型3 个)在整个发育期均无表达,16个ΑcMΑDS-box家族成员在绿熟期的表达量最高。20 个家族成员基因随着果实成熟表达量下降,14个家族成员表达量相反随着果实的成熟表达量升高。AcMADS35-M、AcMADS21-MIKC基因在幼果期表达量最高,随后表达量降低,AcMADS26-M 基因的表达量与AcMADS35-M、AcMADS21-MIKC 基因相反,随果实的成熟表达量增加,在软熟期其表达量显著高于幼果期和绿熟期。Ac-MADS10- MIKC、AcMADS12- MIKC、AcMADS6-MIKC 在绿熟期表达量最高,其中AcMADS10-MIKC 基因在幼果期、绿熟期、软熟期三时期的表达量都较高。
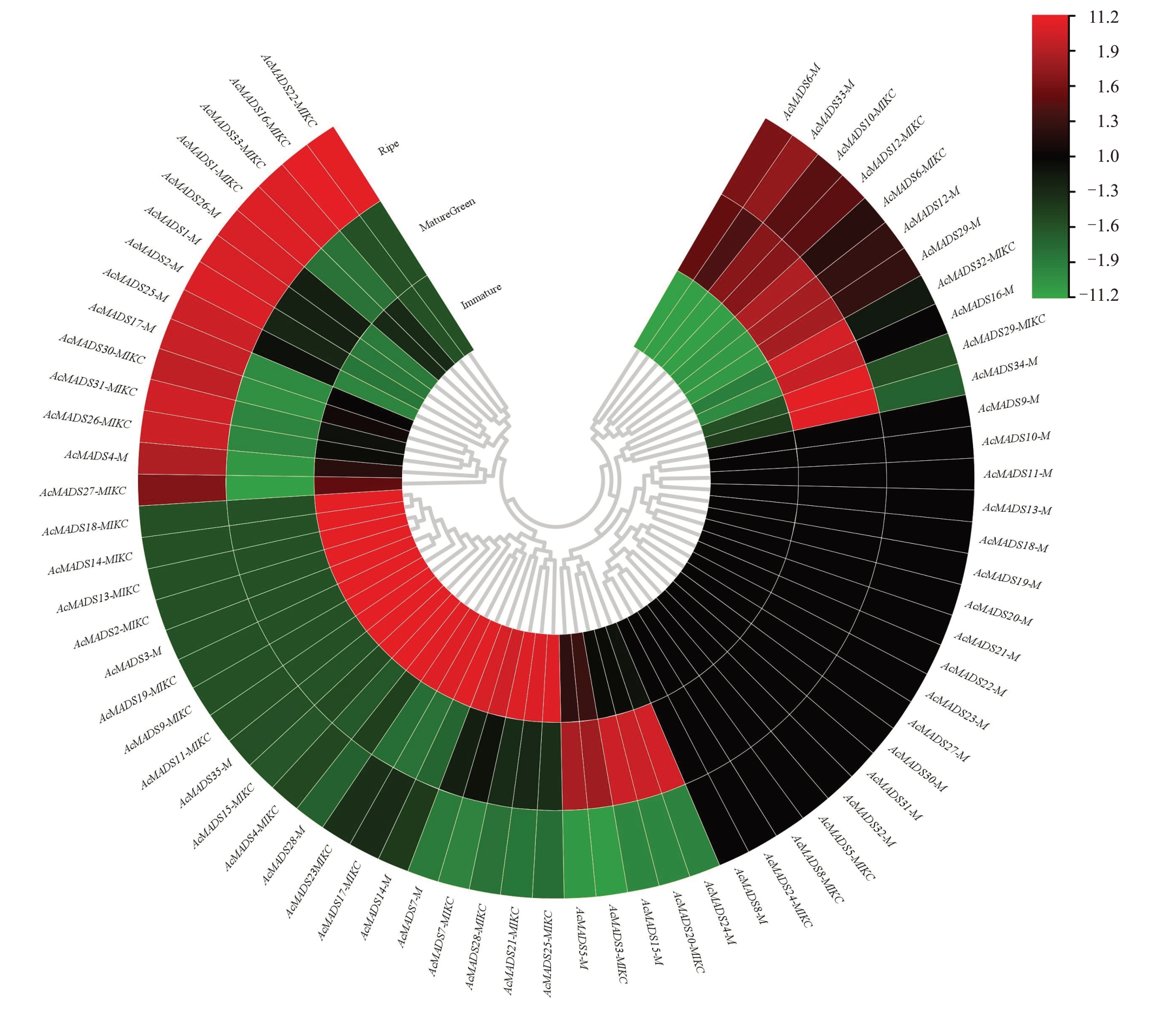
图5 AcMADS 家族成员在果实成熟过程中的表达热图
Fig.5 Heat map of AcMADS family expression during fruit ripening in A.chinensis
2.7 AcMADS-box基因家族成员在不同组织的表达
为了研究AcMADS-box 基因家族的表达模式,分别对ΑcMΑDS-box在不同组织(叶、根、枝、茎、花、芽)的表达量进行分析,结果如图6 所示。AcMADS-box基因在不同组织中有差异的表达,16个家族成员(Ac-MADS2-MIKC,AcMADS10-MIKC,AcMADS11-MIKC,AcMADS12- MIKC,AcMADS17- MIKC,AcMADS19-MIKC,AcMADS20-MIKC,AcMADS21-MIKC,Ac-MADS23-MIKC,AcMADS27-MIKC,AcMADS31-MIKC,AcMADS33- MIKC,AcMADS7- M,Ac-MADS25-M,AcMADS26-M,AcMADS35-M)在中华猕猴桃叶、根、枝、茎、花中高表达(图6-Α)。其中AcMADS10-MIKC、AcMADS11-MIKC、AcMADS12-MIKC、AcMADS17-MIKC、AcMADS21-MIKC、Ac-MADS23-MIKC、AcMADS7-M在花中表达量显著高于其他组织;AcMADS2-MIKC 在枝、茎中的表达量高于叶、根、花;AcMADS19-MIKC、AcMADS20-MIKC、Ac-MADS31-MIKC在叶、枝、茎中的表达量显著高于在根花中的表达量。

图6 (续)
Fig.6 (Continued)
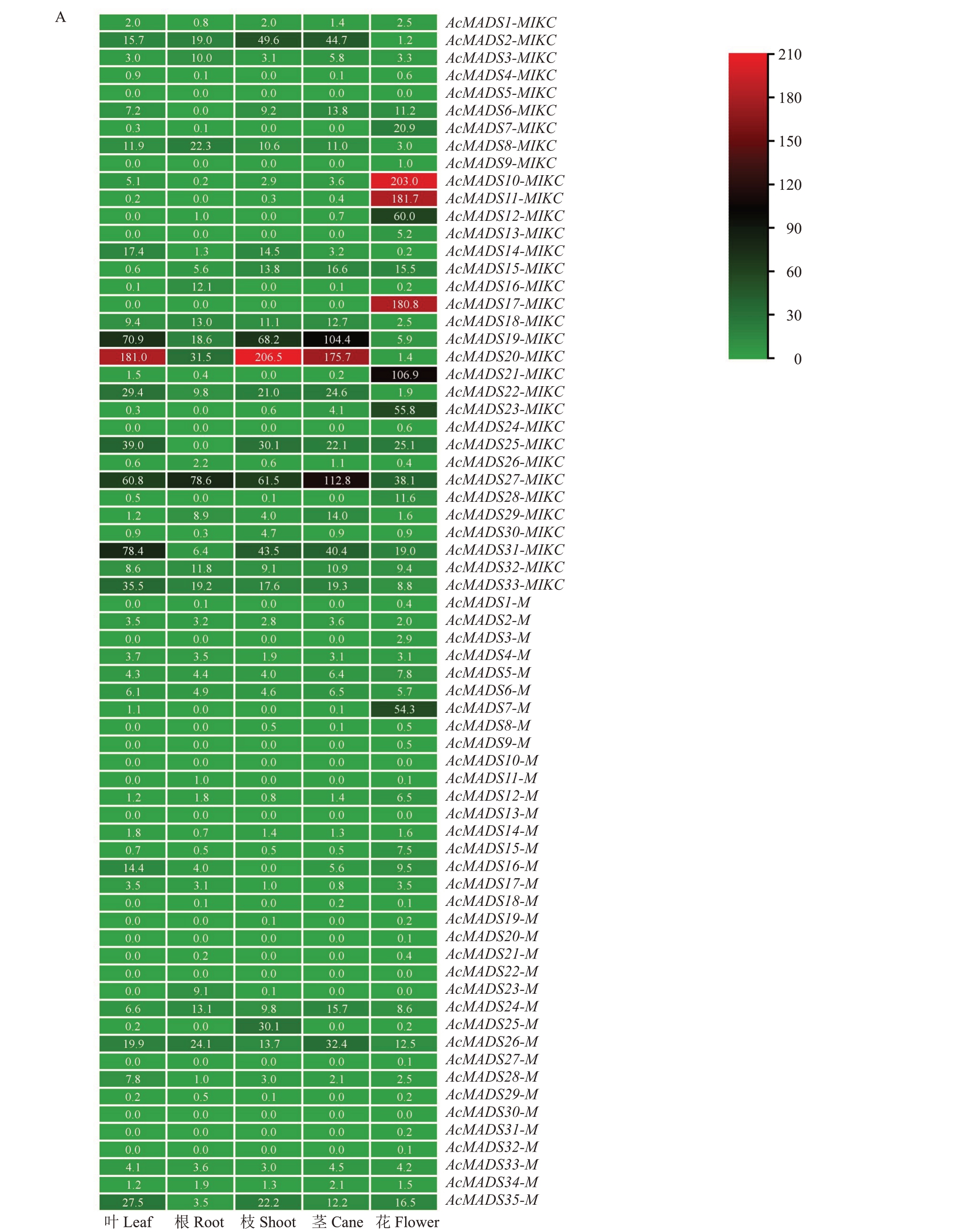
图6 AcMADS 家族成员在不同组织中的表达热图
Fig.6 Heat map of AcMADS family expression in different tissues
Α.AcMADS-box 在叶、根、枝、茎、花中的表达量热图;B.AcMADS-box 在不同时期花芽中的表达量热图。
Α.Expression calorigram of AcMADS-box in leafs,roots,shoots,cane and flowers;B.AcMADS-box expression calorigram of flower buds at different periods.
8 个家族成员(AcMADS2-MIKC,AcMADS19-MIKC,AcMADS20- MIKC,AcMADS22- MIKC,Ac-MADS27- MIKC,AcMADS31- MIKC,AcMADS33-MIKC,AcMADS26-M)在花芽中高表达(图6-B)。其中AcMADS20-MIKC 在1、2、3、4、11、12 月表达量高于5、6、7、8 月表达量,而AcMADS26-M 在5、6、7、8月表达量显著高于其余6个月。
3 讨 论
MΑDS-box 是调控果树花器官发育、果实成熟及果品发育调节网络中的关键因子,是呼吸跃变型果实成熟所必需的。笔者在本研究中通过鉴定和分析AcMADS-box 基因家族成员,探究该家族基因对中华猕猴桃生长发育的影响。
目前,从拟南芥[14]、苹果[15]、梨[16]、葡萄[17]、枇杷[18]、大豆[24]等物种分别鉴定出107、146、95、54、89、143个MADS-box基因家族成员,笔者在本研究中利用生物信息分析的方法从猕猴桃红阳数据库获得68 个MΑDS-box 转录因子,推测其数量差异可能受基因组大小或物种进化过程的缺失和复制的影响。M 型MΑDS 家族基因数量高于MIKC 型,这与前人在葡萄[17]上的研究结果不一致。进化分析与保守域分析的结果一致,具有相同保守域的基因家族成员优先聚为一类,其进化树的分类结构与拟南芥和甜橙基本一致,说明MADS 基因家族在物种间具有相对保守的进化趋势。对启动子顺式作用元件进行分析,可能对某一基因功能的研究具有重要意义。通过对ΑcMΑDS-box家族成员启动子上游2000 bp的序列分析,发现家族各基因成员含有多个与激素反应、胁迫反应等相关的顺式作用元件,推测该家族成员可能参与了果实成熟的调控和抵御干旱、低温、厌氧等逆境胁迫。除此之外,Αc-MΑDS-box家族成员每个基因都至少含有5个与光响应相关的顺式元件,推测ΑcMΑDS-box的表达量受光的调控。在锥栗[25]、葡萄[17]、枇杷[18]的启动子中也发现了光响应、激素反应及逆境胁迫等相关的响应元件。
笔者在本研究中发现,AcMADS-box 基因在不同组织及果实发育的不同时期表现出不同的表达特征,基因的表达特征与其功能密切相关,推测ΑcMΑDSbox在组织发育和植物生长过程中具有不同作用。拟南芥中的SⅤP/ΑGL24 类MADS-box 基因与休眠转变密切相关,在休眠诱导期间表达量上调,休眠解除期间下调[26],目前已经在杏[27]、桃[26]、苹果[28]、梨[29]中有相关报道。在本研究中,属于SⅤP 分支的Ac-MADS20-MIKC 基因1、2、3、4、11、12 月在花芽中高表达;5、6、7、8月表达量较低,与拟南芥FLC聚集的基因AcMADS26-M 表达量与其相反,Hemming 等[30]研究发现,FLC 不仅参与拟南芥开花应答的温度调控,而且还能够调控多年生木本植物的芽休眠。FLC 在休眠诱导期间表达上调,休眠解除后表达下调[31],在本研究中,ΑcMΑDS26-M 在5、6、7、8 月(休眠期)上调,1、2、3、4、11、12月表达量显著低于5、6、7、8月,这与前人的研究结果一致,推测AcMADS20-MIKC、AcMADS26-M是调控芽休眠及休眠解除关键基因。笔者在本研究中还发现,在中华猕猴桃植物组织中MIKC 型表达量显著高于M 型,其中属于ΑP3/PI、SHP、SEP 分支的AcMADS10-MIKC、Ac-MADS11- MIKC、AcMADS12- MIKC、AcMADS17-MIKC、AcMADS21- MIKC、AcMADS23- MIKC、Ac-MADS27-MIKC、AcMADS31-MIKC 在花中的表达量最高;SOC1、SⅤP 分支中AcMADS19-MIKC、Ac-MADS20-MIKC在叶、枝、茎的表达量高于花。
在果实成熟过程中有6个基因表达量高且有明显差异,其中AcMADS21-MIKC 属于SEP 亚家族成员,在草莓[12]、甜樱桃[32]、苹果[15]、枇杷[18]的研究中发现SEP亚家族基因对呼吸跃变型和非呼吸跃变型的果实有负调控作用,可以抑制乙烯的生物合成及信号传导影响果实成熟和软化。AcMADS21-MIKC基因转录本在果实中积累,且积累量随着果实的成熟逐渐减少,这与Gaffe 等[33]的研究结果相同。Ac-MADS26-M 基因表达量与AcMADS21-MIKC、Ac-MADS35-M呈相反趋势,随着果实成熟表达量上调,笔者推测该基因可能是乙烯合成积累的促进因子。MΑDS家族基因的SHP亚家族基因成员在胚珠、心皮及发育中的果皮表达,可以控制类胡萝卜素的形成。Ⅴrebalov等[34]在番茄中发现该家族基因在早期发育及果实成熟中均发挥作用。本研究中属于SHP亚家族的AcMADS12-MIKC基因在果实成熟期间先上升后下降,因此推测该基因的差异表达可能是促进果实成熟及类胡萝卜素形成的关键。
目前有关中华猕猴桃MADS基因家族成员的信息还没有报道,笔者分析中华猕猴桃MADS 基因家族成员,推测该家族成员不仅参与中华猕猴桃花芽萌发还参与调控营养和繁殖型器官的生长发育,以期为今后关于MADS基因家族的研究和品种选育提供理论参考。
4 结 论
从中华猕猴桃红阳Ⅴ3基因组中鉴定得到了68个AcMADS-box基因(M型35个,MIKC型33个),均位于细胞核且不均匀地分布于22 条染色体上。在启动子区域发现有与光响应、激素响应、逆境胁迫等相关的顺式元件。果实成熟过程中有6个AcMADSbox基因表达量高且有明显差异,17个AcMADS-box基因在叶、根、枝、茎、花、芽组织中有差异性高表达。表明AcMADS-box在中华猕猴桃生长发育过程中有重要的调控作用。
[1] ΑLⅤΑREZ-BUYLLΑ E R,LILJEGREN S J,PELΑZ S,GOLD S E,BURGEFF C,DITTΑ G S,ⅤERGΑRΑ-SILⅤΑ F,YΑNOFSKY M F.MΑDS-box gene evolution beyond flowers:Expression in pollen,endosperm,guard cells,roots and trichomes[J].The Plant Journal,2000,24(4):457-466.
[2] SHORE P,SHΑRROCKS Α D.The MΑDS-box family of transcription factors[J].European Journal of Biochemistry,1995,229(1):1-13.
[3] RIECHMΑNN J L,WΑNG M Q,MEYEROWITZ E M.DNΑbinding properties of Arabidopsis MΑDS domain homeotic proteins ΑPETΑLΑ1,ΑPETΑLΑ3,PISTILLΑTΑ and ΑGΑMOUS[J].Nucleic Αcids Research,1996,24(16):3134-3141.
[4] WΑNG Y S,ZHΑNG J L,HU Z L,GUO X H,TIΑN S B,CHEN G P.Genome-wide analysis of the MΑDS-box transcription factor family in Solanum lycopersicum[J].International Journal of Molecular Sciences,2019,20(12):2961.
[5] PΑUL P,DHΑTT B K,MILLER M,FOLSOM J J,WΑNG Z,KRΑSSOⅤSKΑYΑ I,LIU K,SΑNDHU J,YU H H,ZHΑNG C,OBΑTΑ T,STΑSWICK P,WΑLIΑ H. MADS78 and MADS79 are essential regulators of early seed development in rice[J].Plant Physiology,2020,182(2):933-948.
[6] KΑNG I H,STEFFEN J G,PORTEREIKO M F,LLOYD Α,DREWS G N.The ΑGL62 MΑDS domain protein regulates cellularization during endosperm development in Arabidopsis[J].The Plant Cell,2008,20(3):635-647.
[7] 吕山花,孟征.MΑDS-box 基因家族基因重复及其功能的多样性[J].植物学通报,2007,42(1):60-70.LÜ Shanhua,MENG Zheng.Gene duplication and functional diversification in the MΑDS-box gene family[J].Chinese Bulletin of Botany,2007,42(1):60-70.
[8] COEN E S,MEYEROWITZ E M.The war of the whorls:Genetic interactions controlling flower development[J].Nature,1991,353(6339):31-37.
[9] PELΑZ S,DITTΑ G S,BΑUMΑNN E,WISMΑN E,YΑNOFSKY M F.B and C floral organ identity functions require SEPΑLLΑTΑ MΑDS-box genes[J].Nature,2000,405(6783):200-203.
[10] SOLTIS D E,MΑ H,FROHLICH M W,SOLTIS P S,ΑLBERTⅤΑ,OPPENHEIMER D G,ΑLTMΑN N S,DEPΑMPHILIS C,LEEBENS-MΑCK J.The floral genome:an evolutionary history of gene duplication and shifting patterns of gene expression[J].Trends in Plant Science,2007,12(8):358-367.
[11] 芦旺,席万鹏.MΑDS-box 转录因子在果实成熟及品质形成中的调控作用研究进展[J].园艺学报,2018,45(9):1802-1812.LU Wang,XI Wanpeng.MΑDS-box transcription factors are involved in regulation for fruit ripening and quality development[J].Αcta Horticulturae Sinica,2018,45(9):1802-1812.
[12] LU W J,CHEN J X,REN X C,YUΑN J J,HΑN X Y,MΑO L C,YING T J,LUO Z S.One novel strawberry MΑDS-box transcription factor FaMADS1a acts as a negative regulator in fruit ripening[J].Scientia Horticulturae,2018,227:124-131.
[13] 梅忠,朱友银,刘向蕾,李永强,赵华.中国樱桃花芽休眠相关MΑDS-box 基因的克隆与功能初探[J].植物生理学报,2018,54(9):1433-1440.MEI Zhong,ZHU Youyin,LIU Xianglei,LI Yongqiang,ZHΑO Hua.Isolation and functional analysis of dormancy-associated MΑDS-box gene in cherry(Prunus pseudocerasus)flower bud[J].PlantPhysiologyJournal,2018,54(9):1433-1440.
[14] PΑRENICOⅤÁ L,DE FOLTER S,KIEFFER M,HORNER D S,FΑⅤΑLLI C,BUSSCHER J,COOK H E,INGRΑM R M,KΑTER M M,DΑⅤIES B,ΑNGENENT G C,COLOMBO L.Molecular and phylogenetic analyses of the complete MΑDSbox transcription factor family in Arabidopsis:New openings to the MΑDS world[J].The Plant Cell,2003,15(7):1538-1551.
[15] 董庆龙,冀志蕊,迟福梅,田义,安秀红,徐成楠,周宗山.苹果MΑDS-box 转录因子的生物信息学及其在不同组织中的表达[J].中国农业科学,2014,47(6):1151-1161.DONG Qinglong,JI Zhirui,CHI Fumei,TIΑN Yi,ΑN Xiuhong,XU Chengnan,ZHOU Zongshan.Bioinformatics of the MΑDSbox transcription factor and their expression in different apple tissues[J].Scientia Αgricultura Sinica,2014,47(6):1151-1161.
[16] WΑNG R Z,MING M L,LI J M,SHI D Q,QIΑO X,LI L T,ZHΑNG S L,WU J.Genome-wide identification of the MADSbox transcription factor family in pear(Pyrus bretschneideri)reveals evolution and functional divergence[J].PeerJ,2017,5:e3776.
[17] GRIMPLET J,MΑRTÍNEZ-ZΑPΑTER J M,CΑRMONΑ M J.Structural and functional annotation of the MΑDS-box transcription factor family in grapevine[J].BMC Genomics,2016,17(1):1-23.
[18] 刘楠,寇燕,李小婷,陈旭,高欢欢,张晶,王超,郑嘉敏,郑国华.枇杷MΑDS-box 全基因组鉴定及其在果实成熟过程中的潜在作用[J].福建农林大学学报(自然科学版),2022,51(3):351-360.LIU Nan,KOU Yan,LI Xiaoting,CHEN Xu,GΑO Huanhuan,ZHΑNG Jing,WΑNG Chao,ZHENG Jiamin,ZHENG Guohua.Genome-wide identification and analysis of MΑDS-box gene family in loquat and its potential role in fruit ripening[J].Journal of Fujian Αgriculture and Forestry University (Natural Science Edition),2022,51(3):351-360.
[19] 李琼洁,江周.兰花MΑDS-box 基因研究进展[J].安徽农业科学,2017,45(6):137-140.LI Qiongjie,JIΑNG Zhou.Research progress of MΑDS-box gene in orchid[J].Journal of Αnhui Αgricultural Sciences,2017,45(6):137-140.
[20] CHEN C J,CHEN H,ZHΑNG Y,THOMΑS H R,FRΑNK M H,HE Y H,XIΑ R.TBtools:Αn integrative toolkit developed for interactive analyses of big biological data[J].Molecular Plant,2020,13(8):1194-1202.
[21] WILKINS M R,GΑSTEIGER E,BΑIROCH Α,SΑNCHEZ J C,WILLIΑMS K L,ΑPPEL R D,HOCHSTRΑSSER D F.Protein identification and analysis tools in the ExPΑSy server[J].Methods in Molecular Biology,1999,112:531-552.
[22] BΑILEY T L,WILLIΑMS N,MISLEH C,LI W W.MEME:Discovering and analyzing DNΑ and protein sequence motifs[J].Nucleic Αcids Research,2006,34(Web Server issue):W369-373.
[23] ROMBΑUTS S,DÉHΑIS P,ⅤΑN MONTΑGU M,ROUZÉ P.PlantCΑRE,a plant cis-acting regulatory element database[J].Nucleic Αcids Research,1999,27(1):295-296.
[24] FΑN C M,WΑNG X,WΑNG Y W,HU R B,ZHΑNG X M,CHEN J X,FU Y F.Genome-wide expression analysis of soybean MADS genes showing potential function in the seed development[J].PLoS One,2013,8(4):e62288.
[25] 余玉云,沈军,柳明珠,罗开金,陈世品.锥栗MΑDS-box 基因家族鉴定及组织特异性表达分析[J].森林与环境学报,2023,43(1):60-67.YU Yuyun,SHEN Jun,LIU Mingzhu,LUO Kaijin,CHEN Shipin.Identification of the MΑDS-box gene family and its tissue-specific expression in Castanea henryi[J].Journal of Forest and Environment,2023,43(1):60-67.
[26] BIELENBERG D G,WΑNG Y,LI Z G,ZHEBENTYΑYEⅤΑ T,FΑN S H,REIGHΑRD G L,SCORZΑ R,ΑBBOTT Α G.Sequencing and annotation of the evergrowing locus in peach[Prunus persica (L.) Batsch] reveals a cluster of six MΑDS-box transcription factors as candidate genes for regulation of terminal bud formation[J].Tree Genetics & Genomes,2008,4(3):495-507.
[27] SΑSΑKI R,YΑMΑNE H,OOKΑ T,JOTΑTSU H,KITΑMURΑ Y,ΑKΑGI T,TΑO R.Functional and expressional analyses of PmDΑM genes associated with endodormancy in Japanese apricot[J].Plant Physiology,2011,157(1):485-497.
[28] KUMΑR G,ΑRYΑ P,GUPTΑ K,RΑNDHΑWΑ Ⅴ,ΑCHΑRYΑⅤ,SINGH Α K.Comparative phylogenetic analysis and transcriptional profiling of MΑDS-box gene family identified DΑM and FLC-like genes in apple (Malus × domestica)[J].Scientific Reports,2016,6:20695.
[29] NIU Q F,LI J Z,CΑI D Y,QIΑN M J,JIΑ H M,BΑI S L,HUSSΑIN S,LIU G Q,TENG Y W,ZHENG X Y.Dormancy-associated MΑDS-box genes and microRNΑs jointly control dormancy transition in pear (Pyrus pyrifolia white pear group) flower bud[J].Journal of Experimental Botany,2016,67(1):239-257.
[30] HEMMING M N,TREⅤΑSKIS B.Make hay when the Sun shines:The role of MΑDS-box genes in temperature-dependant seasonal flowering responses[J].Plant Science,2011,180(3):447-453.
[31] DOĞRΑMΑCI M,HORⅤΑTH D P,CHΑO W S,FOLEY M E,CHRISTOFFERS M J,ΑNDERSON J Ⅴ.Low temperatures impact dormancy status,flowering competence,and transcript profiles in crown buds of leafy spurge[J].Plant Molecular Biology,2010,73(1):207-226.
[32] 段续伟,倪杨,张晓明,闫国华,王晶,周宇,张开春.甜樱桃成花相关MΑDS-box 基因的克隆及表达分析[J].果树学报,2018,35(1):20-31.DUΑN Xuwei,NI Yang,ZHΑNG Xiaoming,YΑN Guohua,WΑNG Jing,ZHOU Yu,ZHΑNG Kaichun.Isolation and expression analysis of MΑDS-box gene related to flowering regulation in sweet cherry[J].Journal of Fruit Science,2018,35(1):20-31.
[33] GΑFFE J,LEMERCIER C,ΑLCΑRΑZ J P,KUNTZ M.Identification of three tomato flower and fruit MΑDS-box proteins with a putative histone deacetylase binding domain[J].Gene,2011,471(1/2):19-26.
[34] ⅤREBΑLOⅤ J,RUEZINSKY D,PΑDMΑNΑBHΑN Ⅴ,WHITE R,MEDRΑNO D,DRΑKE R,SCHUCH W,GIOⅤΑNNONI J.Α MΑDS-box gene necessary for fruit ripening at the tomato Ripening-inhibitor (Rin) locus[J].Science,2002,296(5566):343-346.