越橘,又名蓝莓(blueberry),果实富含花青苷、超氧化物歧化酶(SOD)、黄酮和其他酚类化合物等功能性营养成分,有助于提高视力、增强抗癌能力和降低心脏病发生等,备受市场关注和消费者青睐[1-2]。种植高效益和巨大的市场潜力使越橘成为目前全球发展最快的果树树种,到2020年,全球越橘栽培总面积20.567万hm2,中国由2008年的0.12万hm2快速增加到2016 年的2.20 万hm2后,又增加到2020 年的6.64 万hm2,总产量34.72 万t,产量和面积跃居全球第一位[3],在中国经济发展中发挥了重要作用。
越橘为杜鹃花科(Ericaceae)越橘属(Vaccinium spp.)植物,全世界越橘属植物约有400 个种[4],染色体倍性多样性丰富。高丛越橘(Vaccinium corymbosum)、兔眼越橘(V. ashei)、矮丛越橘(V. augustifolium)是世界范围内主要的商品化栽培种,其中高丛越橘品种(包括半高丛、北高丛和南高丛类型)为四倍体(2n=4x=48),而兔眼蓝莓是六倍体(2n=6x=72)[5-6],笃斯越橘(V.uliginosum)、红豆越橘(V.vitisidaea)、蔓越橘(V.macrocarpon)等野生种为二倍体(2n=2x=24)[7]类型。最新研究发现,四倍体越橘基因组大小高达1.68 Gb,杂合度高,整个基因组上包含128 559 个基因,每个单倍型染色体上平均有32 140 个蛋白质编码基因[8],是目前组装质量最好的四倍体越橘基因组,为高通量测序获取大规模SNP(single nucleotide polymorphisms)标记深入挖掘遗传变异信息、群体遗传进化和遗传多样性研究奠定了条件基础。
SLAF-seq 技术(特异长度扩增片段测序,Specific-locus amplified fragment sequencing)是一种新的简化基因组测序技术,它具有测序深度高、可避免大量重复序列、能获得大量SNP 标记和In-Del 标记等优点[9],已成功应用在高杂合度木本植物中,而在越橘上鲜见相关报道。
笔者实验室通过前期开发的越橘EST-SSR 标记[7]已初步掌握了我国越橘种质资源存在与抗寒性、低温需冷量等对应的遗传差异[10]。开展科学系统的种质资源评价、特异资源筛选及野生资源利用,离不开遗传多样性及遗传结构的精准鉴定。然而,我国越橘种质资源研究中不同类型的越橘群体间遗传变异、基因交流、群体结构问题仍不明确,导致越橘遗传多样性背景不清晰,无法为越橘育种提供有效参考依据。为此,笔者在本研究中以越橘属多种类型(栽培种和野生种)、不同倍性(二倍体、四倍体和六倍体)越橘为试材,通过SLAF-seq 技术获得大量SNP标记分析遗传变异和群体结构,探讨遗传背景及开发利用途径,为科学开展新品种选育提供参考和依据。
1 材料和方法
1.1 材料
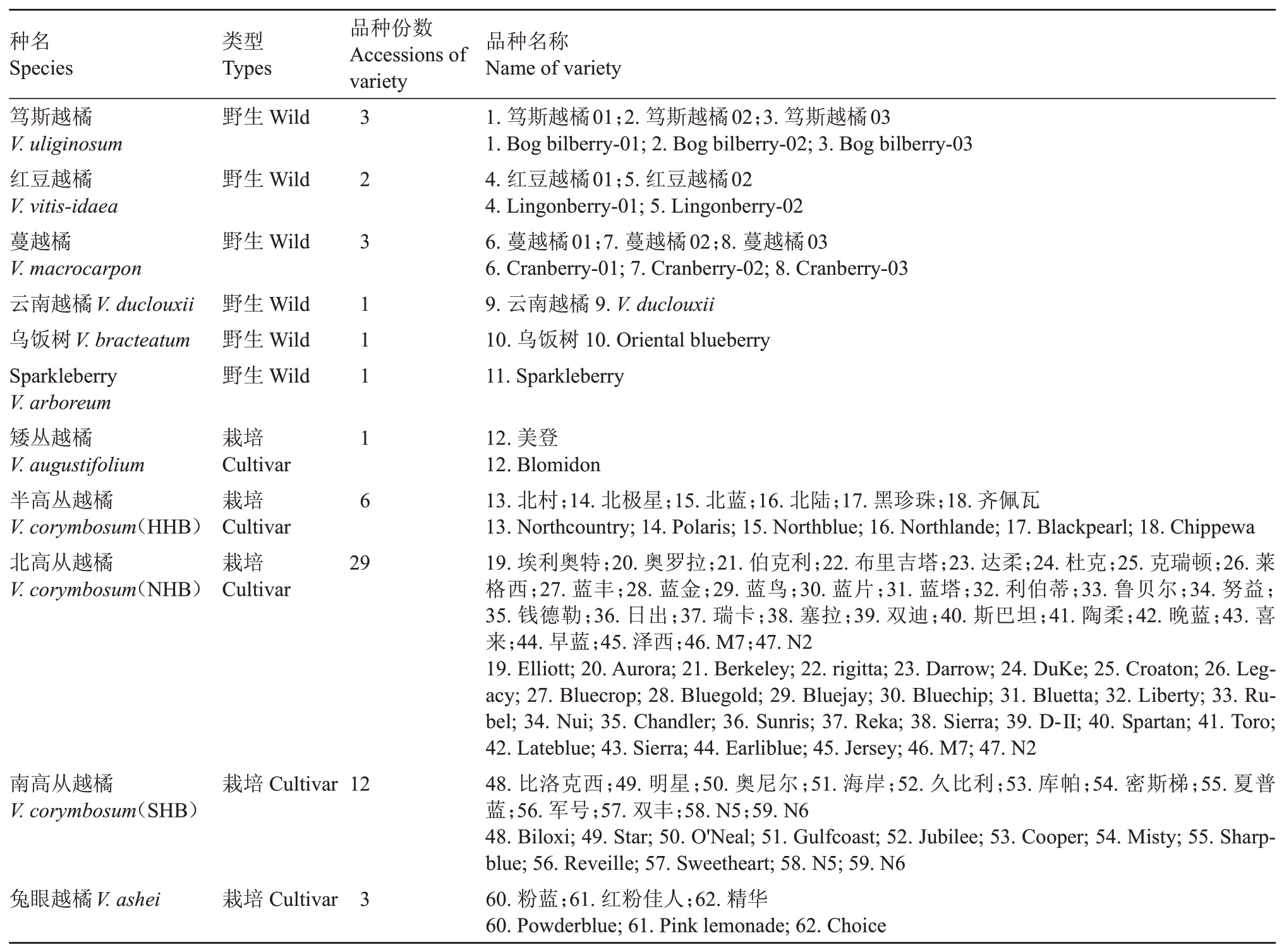
供试越橘种质资源材料取自辽宁省果树科学研究所越橘种质资源圃,其中包括野生类型11 份,从国外引进栽培品种51 份,涉及越橘属9 个种(species)和北高丛、南高丛、半高丛、矮丛、兔眼5个栽培类型(表1)。
表1 越橘采样信息
Table 1 Sampling information of Vaccinium
?
1.2 基因组DNA提取
供试材料幼叶利用液氮速冻后存于-80 ℃冰箱备用。用改良CTAB法[7]提取基因组DNA,用Nanodrop 2000C超微量核酸蛋白测定仪(Thermo Fisher)和Qubit 2.0 荧光计(Thermo Fisher)进行DNA 的质量和浓度检测,所提基因组DNA OD260与OD280的比值分布在1.8~2.0,DNA质量浓度达到30 ng·μL-1,达到测序文库构建要求。
1.3 酶切建库与测序评估
根据酶切片段在基因组上是否均匀分布、大小是否与实验体系相吻合等原则[11],采用SLAF-predict软件利用四倍体越橘品种Draper 作为参考基因组(http://gigadb.org/dataset/100537)[8],以Hae Ⅲ、RsaⅠ、RsaⅠ+Hae Ⅲ和RsaⅠ+RsaⅡ4种酶切方案进行电子酶切预测实验,根据开发的标签数等确定酶切方案。选择水稻品种日本晴(Oryza sativa ssp.japonica)(http://rapdb.dna.affrc.go.jp/)作为对照,同步进行平行试验,利用SOAP[12]软件对日本晴-水稻测序数据和水稻基因组数据进行比对,进行酶切效益评估,确保试验的准确性。
1.4 SLAF标签获得、SNP标记与验证
文库质检合格后用Illumina 测序,供试不同倍性的越橘测序数据依照参考基因组倍性(2n=2x=24)统一标准进行分析。利用BWA软件(v0.7.17)将测序reads 比对到参考基因组上,使用GATK(v4.1.7.0)[13]和SAMtools[14]分别进行数据质控,以两种方法得到的SNP 标记交集作为最终可靠的SNP标记数据集,用于进一步的数据统计和分析。使用vcftools(v0.1.16)软件,剔除缺失率大于20%或MAF小于0.05的标记。
使用SNPhylo 软件(v 20140701)对SNP 进行过滤,过滤标准为:-p 10-c 3-l 0.2-M 0.2(深度低于3的样本比例小于10%,且缺失率小于20%),连锁不平衡R2大于0.2,将vcf文件转置为phy格式后,用Phy-ML3.0软件的最大似然法(maximum likelihood,ML)构建系统发育树,并进行1000 次bootstrap 分析(-m HKY85-b 1000),使用iTOL(v6.6)进行进化树的可视化分析。
1.5 群体遗传结构分析
在进行群体遗传结构分析之前,先使用plink软件(v1.90b)用滑窗法过滤掉连锁不平衡位点,滑窗窗口大小50 kb,滑窗步长10个SNP,连锁不平衡R2阈值0.2(--indep-pairwise 50 10 0.2),根据所筛选出的SNP位点17 747个,占总数0.4%,采用Admixture软件(v1.3.0)进行遗传结构分析(for K in 1 2 3 4 5 6 7;do admixture--cv file.bed $K | tee log${K}.out;done)。利用R(v4.2.1)中的软件包SNP Relate(v1.30.1)进行PCA(principal component analysis)分析,首先使用snpgdsVCF2GDS函数将目标vcf文件转成gds格式,之后用snpgdsPCA函数对文件进行PCA分析,并使用ggplot2(v3.3.6)进行可视化分析。
2 结果与分析
2.1 酶切方案与建库评估
HaeⅢ、RsaⅠ+RsaⅡ、RsaⅠ+HaeⅢ和RsaⅠ不同酶切方案产生的SLAF标签存在差异,其中RsaⅠ酶切产生的SLAF 标签最多,共计148 190 个,片段长度在414~416 bp,在基因组上均匀分布,确定为最终酶切方案。将水稻日本晴的测序reads 与其参考基因组进行比对,结果显示双端比对效率为97.26%,酶切效率为92.76%(表2),SLAF建库正常。
表2 根据酶切方案设计预测信息
Table 2 Information prediction according to the enzyme digestion method

?
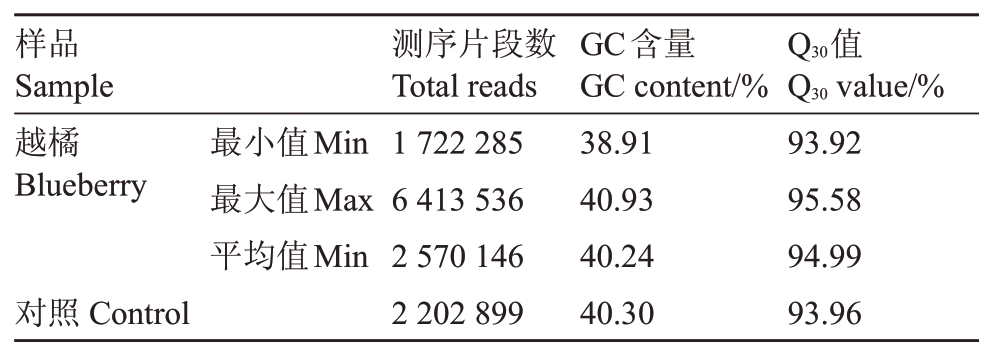
所有供试越橘属植物测序共获得169.87 Mb reads数据,各种质样品获得的读长数目在1 722 285~6 413 536 bp,其中笃斯越橘数据量最小,N6 数据量最大。不同样品测序Q30在93.92%~95.98%,平均为94.99%,GC 含量占38.91%~40.93%,平均为40.24%(表3),表明测序碱基错误率低,获得的数据可靠,可用于后期分析。
表3 越橘及对照测序结果
Table 3 Sequencing result of blueberry and control
?
2.2 SLAF标签、SNP分子标记统计
样品平均测序深度为10.65×,通过对序列进行分析,从62份越橘材料中共检测到319 590个SLAF标签,通过BWA软件将SLAF标签定位到参考基因组上,多态性SLAF 标签172 513 个,利用GATK 和SAMtools 两种方法共得到76 299 105 个群体SNP标记,根据完整度>0.8、次要等位基因频率(MAF)>0.05 过滤,共筛选得到4 133 595 个群体SNP 标记,占SNP 总量的5.08%,各样品完整度为12.92%~44.30%,平均为29.77%,杂合率为3.06%~9.78%,平均为5.44%,说明供试样品基因组杂合度较高(表4)。根据SLAF 标签和SNP 标记在染色体上的分布,绘制染色体分布图,SLAF标签和SNP标记比较均匀的分布在越橘12 条染色体上(图1),其中在第6号染色体上有SNP的集中分布区域。
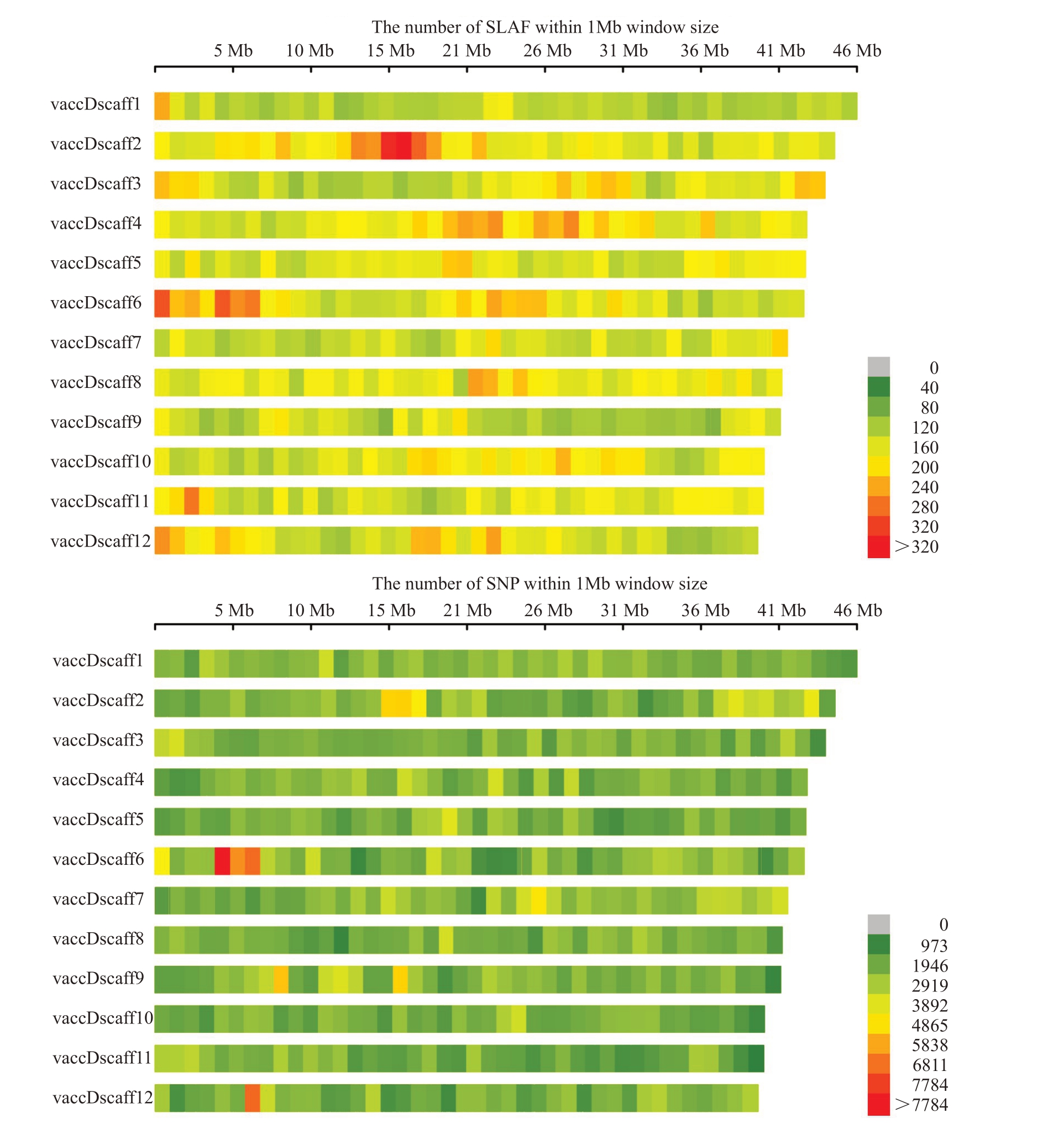
图1 SLAF 标签(上)和SNP 标记(下)在染色体上的分布
Fig.1 Distribution of SLAFs(top)and SNPs(bottom)on chromosomes
上图为SLAF 标签在越橘染色体上的分布情况;下图为SNP 标记在越橘染色体上的分布情况。每一个条带代表一条染色体,图中颜色越红的区域即SLAF 标签和SNP 标记分布越集中的区域。
The top figure represents the distribution of total SLAFs on the reference genome;B:The bottom figure represents the distribution of SNPs on the reference genome.Each strip represents a chromosome,the deeper the red color area is SLAF label and SNPs distribution area.
表4 SLAF 标签和SNP 标记数据统计
Table 4 Data statistics of SLAF and SNP
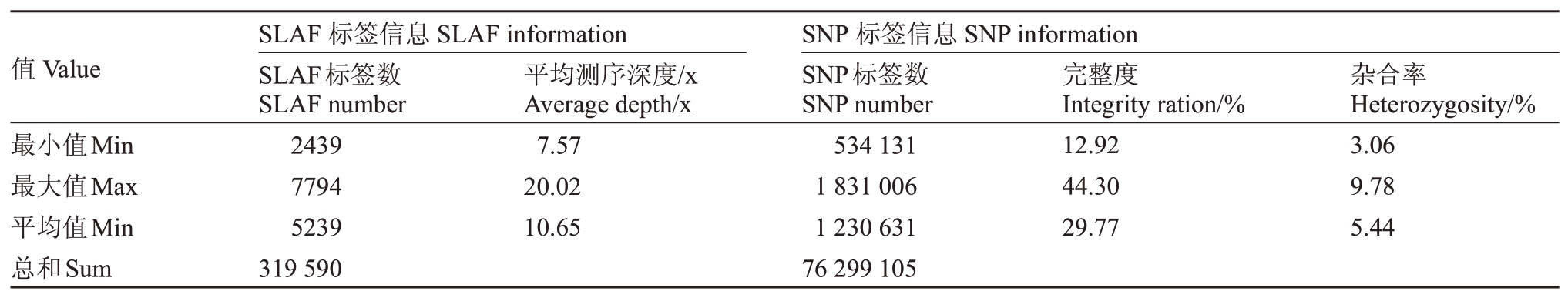
注:x 表示基因组大小倍数;完整度,即每个样品SNP 在全部样品中的信息比重;杂合率,即每个样品含两种或两种以上碱基类型的SNP 所占比重。
Note:x represents the multiple of genome size;Integrity ration,the information proportion of each sample SNP in all samples;Heterozygosity,the proportion of SNPs containing two or more base types in each sample.
?
2.3 SNP标记验证
笔者在本研究中利用基于SNP 标记的系统进化树是否能准确反映供试材料的类群划分和亲缘关系来验证SNP标记的有效性(表5,图2)。如图2(聚类图中心)所示,在系统进化树中,包括野生种和栽培种在内的越橘属9个种62份试材主要形成3个类群。Cluster Ⅰ(绿色):野生种类群,系统进化树的最原始端,包含供试的所有二倍体野生种、六倍体兔眼越橘,矮丛品种美登、半高丛品种北村、黑珍珠等品种在野生种群中形成分支,反映了从野生中驯化而来的进化过程;Cluster Ⅱ(蓝色):北高丛越橘品种为主的栽培类群;Cluster Ⅲ(红色):南高丛越橘品种为主的栽培类群,其中蓝丰、瑞卡、努益3 个北高丛品种分属在该类群上。
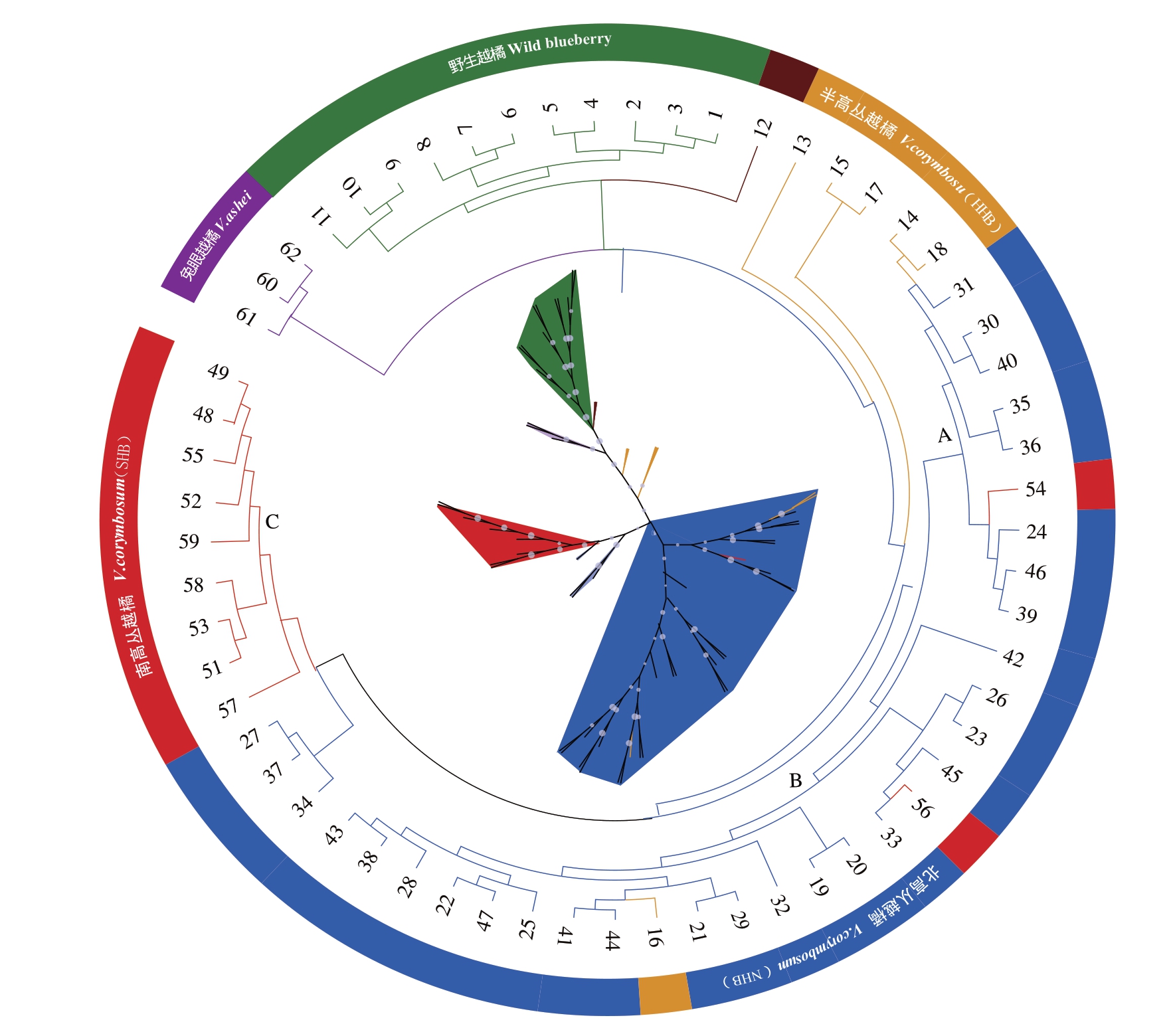
图2 供试蓝莓材料系统进化树
Fig.2 Phylogenetic tree of 62 blueberry accessions
图中编号对应的材料名称和系谱见表5。
The material names and pedigree corresponding to the numbers in the figure are shown in Table 5.
表5 供试材料系谱
Table 5 Pedigree of tested materials

?
为了更清晰、更明确地体现SNP 标记反映的品种间亲缘关系,本研究列出了品种系谱(表5),系谱比对分析发现,在选育过程中拥有相同亲本及互为亲子关系的品种,如库帕与海岸拥有相同父母本,斯巴坦和蓝片拥有相同父本,瑞卡与蓝丰、陶柔与早蓝、蓝鸟与伯克利、奥罗拉与埃利奥特等系亲子关系,均在图2中彼此紧密聚类;骨干亲本的杂交后代聚为一类,如早蓝、达柔、康维尔等为亲本的后代多数聚为类群A,斯坦雷、蓝丰、泽西、先锋、伯克利、维口等为亲本的后代聚为类群B,以美国农业部US系列亲本及佛罗里达大学Florida 系列亲本的杂交后代聚为类群C。综上,基于SNP 的系统进化树和聚类分析能够较准确地反映供试材料的植物学分类和亲缘关系等遗传信息。
2.4 群体遗传结构分析
2.4.1 越橘群体结构分析 基于筛选获得的4 133 595 个SNP 位点,根据K-1 数的Q值分布,设定类群数目(K 值)为1~10 进行聚类,当交叉验证错误率(CV error)最低时为最优分群数,结果显示K=2时为最优结果,选择可以反映供试越橘主要群体结构变化的K值范围,即K=2~7的群体结构结果见图3。K=2时供试材料形成两个清晰完整的类群,类群Ⅰ包含笃斯越橘、蔓越橘、红豆越橘、云南越橘、乌饭树、Sparkleberry,归类为野生群簇,类群Ⅱ由南高丛越橘、北高丛越橘、半高丛越橘、兔眼越橘、矮丛越橘等组成,归类为栽培群簇,表明野生类型与栽培品种在遗传信息组成上存在明显差异,部分栽培品种中嵌合野生资源基因,即存在从野生种到栽培种的单向基因流,但不存在栽培种到野生种的反方向基因流;矮丛品种美登,兔眼品种精华、粉蓝、粉红佳人始终保持着较高比重的野生种遗传背景。随着K值的增大,野生种群簇无亚群划分,说明不同来源的野生资源具有独立的遗传背景,种间不存在基因交流,而栽培种群簇存在明显的亚群结构,说明该群簇内部及与野生种群簇间存在普遍基因交流现象,当K≥5时栽培类群亚群划分趋于平稳,军号、黑珍珠、美登、北村、精华、粉蓝、粉红佳人及N6始终包含大于等于K-2个亚群遗传组分,遗传背景相对复杂,表现为高度杂合,而北蓝、早蓝、北陆、陶柔、布里吉塔、N2、齐佩瓦、北极星、瑞卡、蓝丰、酷帕、海岸、夏普蓝、明星、莱格西等品种亚群间无基因交流或基因交流极少,表现为遗传背景单一。

图3 Admixture 各K 值对应的样品群体结构图
Fig.3 The genetic structure of samples corresponding to each K value of advertisement
A.不同K 值所对应的交叉验证错误率,虚线显示为最佳群体数;B.交叉各个K 值对应的群体结果图,图中编号对应的材料名称见表5。
A.The admixture validation error rate corresponding to the different K values,dashed line represent the optical number of K clustering;B.Admixture the individual cluster values corresponding to each K value,the material names corresponding to the numbers in the figure are shown in Table 5.
2.4.2 主成分分析 为了更好地分析越橘个体间的遗传结构,对样本材料进行主成分分析(图4),栽培品种和野生种在空间上的分布是明显分离的(图4-A),说明栽培种和野生种间亲缘关系远,遗传背景差异大,其中半高丛越橘、北高丛越橘、南高丛越橘、矮高丛越橘及兔眼越橘在PCA图中发生集中重叠;笃斯越橘、蔓越橘、红豆越橘、云南越橘、乌饭树、Sparkleberry等野生种在PCA图上分散分布,其中起源我国东北大兴安岭地区的笃斯越橘、红豆越橘及原产北美地区的蔓越橘相对集中地分布在PCA 的中上部,而起源美国东南部的Sparkleberry越橘及中国西南地区的云南越橘、乌饭树彼此分离,可见野生种在PCA图上的分布与地理起源相关,彼此因地理隔离而遗传背景各异,与遗传结构分析结果一致。
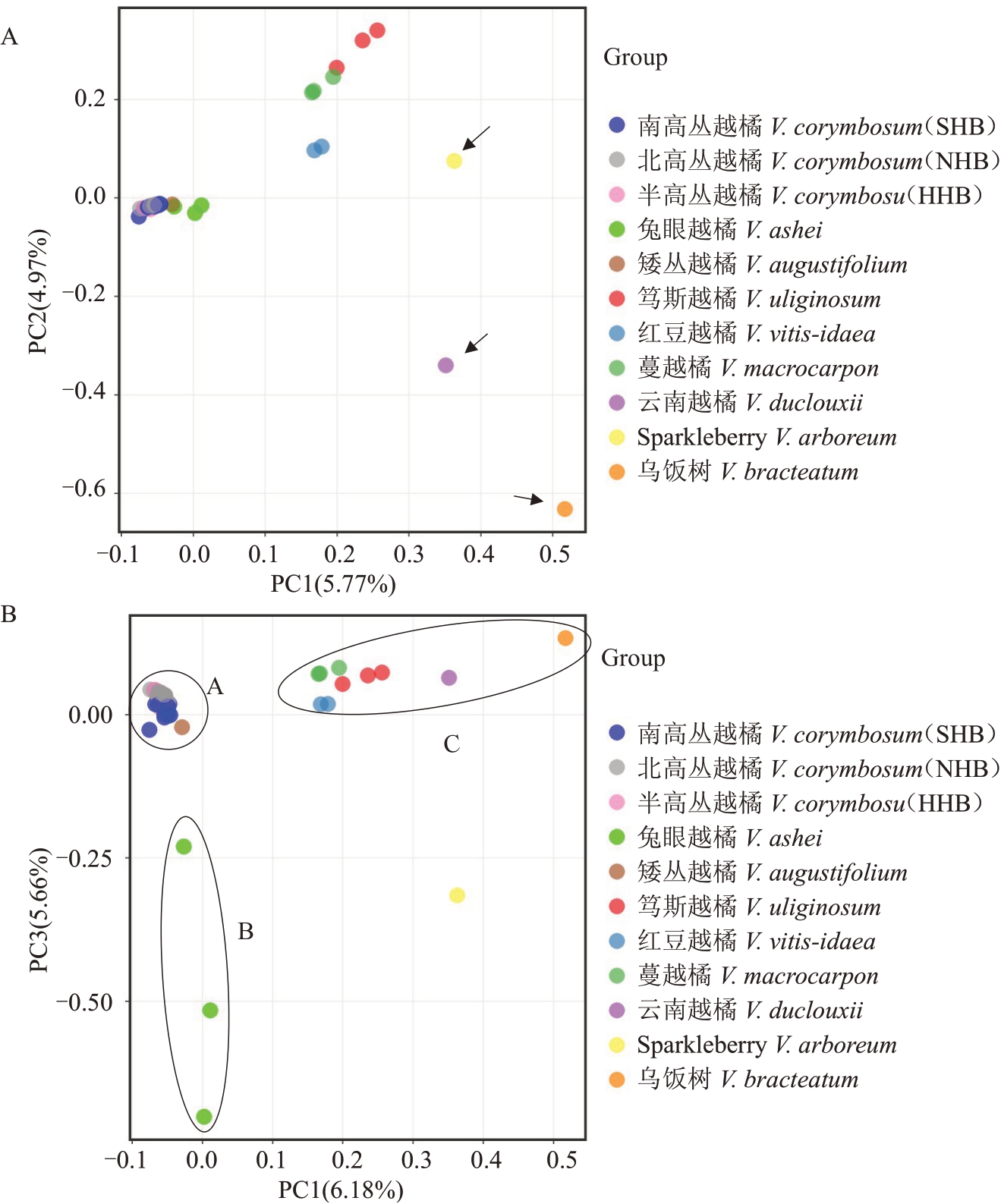
图4 样品PCA 分析
Fig.4 Principal component cluster analysis of samples
为排除云南越橘、乌饭树和Sparkleberry分散分布对其他试材的影响,对除这3份材料以外的其他试材进行PCA分析(图4-B),试材按染色体倍性分别形成四倍体(A)、六倍体(B)和二倍体(C)3个类群,其中四倍体类型仍集中重叠分布,而六倍体类型分布较分散。
3 讨 论
3.1 基于SLAF-seq技术的越橘SNP位点应用
SNP标记是建立在基因组测序基础上的第三代分子标记,具有多态性好、数目多、密度高、遗传稳定、分布广泛及可高通量检测等优点[15-16],是开展群体遗传进化、基因定位、遗传连锁图谱构建及全基因组关联分析等研究的理想标记。SLAF-seq是一种对基因组特定DNA酶切片段进行简化测序的技术[9],非全基因组测序,可避免大量重复序列,大大降低了测序成本,已在多物种SNP位点挖掘中发挥了重要作用,得到的SNP标记五万到几十万个不等[10-13]。本研究利用SLAF-seq技术对越橘属9个种62份试材进行了平均测序深度为10.65×的简化测序,为提高标记质量,采用GATK和SAMtools进行共筛选,得到76 299 105个群体SNP标记,通过筛选、过滤共获得4 133 595个高质量SNP标记,足以支撑供试材料间特异性位点和差异性遗传信息的发掘。
3.2 基因组遗传变异揭示不同类型越橘亲缘关系
笔者在本研究中通过系统进化树和聚类分析结合品种系谱的对应关系判断SNP标记是否有效、可靠。系统进化树显示:供试材料被完整划分为野生种、北高丛品种群和南高丛品种群,与基于植物学特性的类群划分一致;矮丛品种美登、半高丛品种北村、黑珍珠作为分支聚在野生种类群Cluster Ⅰ中,这与3个品种从野生种驯化而来的选育过程一致。此外,基于SNP标记最大似然法的聚类图显示,半高丛品种北极星、齐佩瓦、北陆,南高丛品种军号、密斯梯未按传统分类聚类,剖析系谱发现北极星、齐佩瓦因拥有姊妹系亲本而先一步紧密聚类,其中北极星是北高品种蓝塔的后代,进而与蓝塔聚在一起,蓝塔与斯巴坦拥有同一亲本早蓝而优先聚类,斯巴坦与蓝片为同父(US11-93)异母品种,二者紧密聚类,可见上述因品种间存在共同亲本,彼此亲缘关系近,故聚为一个小群,又如,蓝鸟是伯克利的后代,奥罗拉是埃利奥特的后代,瑞卡是蓝丰的后代等,均在聚类图中彼此紧密聚类,说明SNP标记能有效反映供试材料的真实亲缘关系。北陆原属于半高丛品种,但从系谱可知该品种理论矮丛基因仅占25%,北高丛基因占主导,与崔健民等[17]、孙海悦等[18]的结果相一致,并且是伯克利的后代,因此脱离半高丛品种与北高丛品种聚为一类,其植物学性状也表现出明显的北高丛品种特性。另外,剖析军号系谱发现,该品种是经多代杂交获得的,但亲本中蓝丰、伯克利、维口等北高丛品种占多数,北高丛遗传背景占主导,导致军号脱离南高丛品种与北高丛聚类。所以,本研究结果结合品种系谱,笔者认为北陆和军号划分为北高丛品种更合理。
综上,本研究开发的SNP标记能从基因组水平准确反映品种类群划分和亲缘关系,相比较于ESTSSR 标记的遗传分析[7,10,18],SNP 标记反映的遗传背景信息更全面、更准确,同时也验证了本研究开发的SNP标记的有效性和可靠性,可用于后续分析。
3.3 越橘资源遗传结构分析
聚类分析能反映供试材料的亲缘关系,但无法揭示个体的遗传构成,而遗传结构分析能够从DNA水平上展示试材的遗传结构和个体的遗传构成,明确每个个体的血统来源和不同基因型间遗传信息的交流情况,是分析多类型试材遗传背景的有效手段,是品种遗传多样性、品种进化和性状关联分析等研究的一项基础工作[19]。在本研究的遗传结构分析中,K=2时62份材料被完整划分为野生种群簇和栽培种群簇,且两个类群间有单向的基因流发生,即部分栽培品种中有野生资源基因流进入,如兔眼越橘品种粉蓝、精华、粉红佳人中有≥30%的野生资源遗传背景,矮丛品种美登,南高丛品种明星、比洛克西及优良种质N6、N5 等中有>20%的野生资源遗传背景,而无从栽培种到野生种的反向基因流发生。随着K值的增大,栽培群簇存在明显的亚群结构,说明该类群间及与野生类群间存在不同程度的基因交流,结合PCA分析,栽培品种集中重叠分布,从系谱可知这些品种选育过程中存在近亲杂交和骨干亲本高频使用现象,如供试的29个北高丛品种中有21个品种是泽西、斯坦雷、蓝丰、早蓝、先锋、维口等骨干亲本的一代或多代杂交后代,这使得品种间基因同质性高,导致遗传基础狭窄,遗传多样性水平低;而笃斯越橘、蔓越橘、红豆越橘、云南越橘、乌饭树及Sparkleberry 越橘等野生类群始终无亚群划分,在PCA图上野生种分散分布、种间无重叠,并且起源高纬度地区(我国东北大兴安岭地区、北美地区)和低纬度地区(美国东南部、我国西南地区)的野生资源空间分布距离远,说明不同来源的野生资源具有独立的遗传背景且种间无基因渗透,可能由地理隔离导致生殖隔离,进而保持着更高的遗传多样性所致。
根据本实验室前期开展的越橘性状规律研究[20-22]及育种实践,在排除远缘杂交不亲和情况下,育种双亲遗传背景差异越大,后代性状分离越明显,虽然单果质量、果蒂痕大小等性状存在趋向低值亲本的劣质遗传,但超亲遗传普遍存在,而近亲杂交多数表现为趋中遗传,优选率低。本实验筛选的优质亲本N6在果实等表型性状上与现有品种差异大,从系谱可知该种质为V.corymbosum、V.darrowi、V.ashei、V.angustifolium、V. elliottii、V. constablaei 等多个种的杂交后代,遗传背景复杂,这与本实验群体结构分析结果一致。育种中发现N6作为亲本配置杂交组合后代优选率高,已培育出辽蓝513等[23]优良品种。笔者还发现,除N6外,军号、黑珍珠、美登、北村、精华、粉蓝、粉红佳人始终包含大于等于K-2个亚群遗传组分,表现为高度杂合。综上,建议在越橘育种亲本选择时,除表型性状外,亲本的遗传背景应作为重点考虑的因素。
4 结 论
越橘栽培品种基因交流频繁,导致品种间基因同质性高、遗传基础狭窄,原因是育种中近亲杂交和骨干亲本的高频率使用,而野生种地理生殖隔离种间无基因渗透,具有独立的遗传背景,但存在从野生种到栽培种的基因流,反映出在栽培品种选育中应用了野生资源优良基因,但遗传结构分析显示源自野生资源的基因占比小,应加大野生资源优良基因挖掘及利用的力度,以扩宽越橘遗传研究领域,加快种质创新步伐。
致谢:感谢辽宁省果树科学研究所刘硕副研究员提出的宝贵意见和对英文摘要的修改、校正。
[1] HOU D X. Potential mechanisms of cancer chemoprevention by anthocyanins[J]. Current Molecular Medicine,2003,3(2):149-159.
[2] ROWLAND L J,ALKHAROUF N,DARWISH O,OGDEN E L,POLASHOCK J J,BASSIL N V,MAIN D. Generation and analysis of blueberry transcriptome sequences from leaves,developing fruit,and flower buds from cold acclimation through deacclimation[J].BMC Plant Biology,2012,12:46.
[3] 李亚东,盖禹含,王芳,刘成,刘有春,陈丽.2021 年全球蓝莓产业数据报告[J].吉林农业大学学报,2022,44(1):1-12.LI Yadong,GAI Yuhan,WANG Fang,LIU Cheng,LIU Youchun,CHEN Li. Global blueberry industry report 2021[J]. Journal of Jilin Agricultural University,2022,44(1):1-12.
[4] ROWLAND L J,OGDEN E L,BASSIL N,BUCK E J,MCCALLUM S,GRAHAM J,BROWN A,WIEDOW C,CAMPBELL A M,HAYNES K G,VINYARD B T. Construction of a genetic linkage map of an interspecific diploid blueberry population and identification of QTL for chilling requirement and cold hardiness[J].Molecular Breeding,2014,34(4):2033-2048.
[5] BALLINGTON J R. The role of interspecific hybridization in blueberry improvement[J].Acta Horticulturae,2009(810):49-60.
[6] BREVIS P,HANCOCK J,ROWLAND L J. Development of a genetic linkage map for tetraploid highbush blueberry using SSR and EST-PCR markers[J].HortScience,2007,42(4):963.
[7] LIU Y C,LIU S,LIU D C,WEI Y X,LIU C,YANG Y M,TAO C G,LIU W S. Exploiting EST databases for the development and characterization of EST-SSR markers in blueberry (Vaccinium) and their cross-species transferability in Vaccinium spp.[J].Scientia Horticulturae,2014,176:319-329.
[8] COLLE M,LEISNER C P,WAI C M,OU S J,BIRD K A,WANG J,WISECAVER J H,YOCCA A E,ALGER E I,TANG H B,XIONG Z Y,CALLOW P,BEN-ZVI G,BRODT A,BARUCH K,SWALE T,SHIUE L,SONG G Q,CHILDS K L,SCHILMILLER A,VORSA N,BUELL C R,VANBUREN R,JIANG N,EDGER P P. Haplotype-phased genome and evolution of phytonutrient pathways of tetraploid blueberry[J]. GigaScience,2019,8(3):giz012.
[9] SUN X W,LIU D Y,ZHANG X F,LI W B,LIU H,HONG W G,JIANG C B,GUAN N,MA C X,ZENG H P,XU C H,SONG J,HUANG L,WANG C M,SHI J J,WANG R,ZHENG X H,LU C Y,WANG X W,ZHENG H K. SLAF-seq:An efficient method of large-scale de novo SNP discovery and genotyping using high-throughput sequencing[J].PLoS One,2013,8(3):e58700.
[10] 刘有春,刘成,杨艳敏,孙斌,刘硕,袁兴福,魏永祥.基于ESTSSR 标记的越橘栽培种和几个中国野生种的遗传结构分析[J].果树学报,2017,34(8):956-967.LIU Youchun,LIU Cheng,YANG Yanmin,SUN Bin,LIU Shuo,YUAN Xingfu,WEI Yongxiang. Genetic structure analysis of the cultivated blueberry (Vaccinium spp. ) species and wild species in China based on EST-SSR markers[J]. Journal of Fruit Science,2017,34(8):956-967.
[11] DAVEY J W,CEZARD T,FUENTES-UTRILLA P,ELAND C,GHARBI K,BLAXTER M L. Special features of RAD Sequencing data:Implications for genotyping[J].Molecular Ecology,2013,22(11):3151-3164.
[12] LI R Q,YU C,LI Y R,LAM T W,YIU S M,KRISTIANSEN K,WANG J. SOAP2:An improved ultrafast tool for short read alignment[J].Bioinformatics,2009,25(15):1966-1967.
[13] MCKENNA A,HANNA M,BANKS E,SIVACHENKO A,CIBULSKIS K,KERNYTSKY A,GARIMELLA K,ALTSHULER D,GABRIEL S,DALY M,DEPRISTO M A.The Genome Analysis toolkit:A MapReduce framework for analyzing next-generation DNA sequencing data[J]. Genome Research,2010,20(9):1297-1303.
[14] LI H,HANDSAKER B,WYSOKER A,FENNELL T,RUAN J,HOMER N,MARTH G,ABECASIS G,DURBIN R. 1000 Genome Project Data Processing Subgroup. The sequence alignment/map format and SAMtools[J]. Bioinformatics,2009,25(16):2078-2079.
[15] 张雪霞.基于SCoT 标记的白皮松景观基因组学研究[D].郑州:河南农业大学,2019.ZHANG Xuexia.The landscape genomics studies of Pinus bungeana (Pinaceae) based on SCoT molecular markers[D]. Zhengzhou:Henan Agricultural University,2019.
[16] 周琳,段玉,文博,马媛春,朱旭君,王玉花,房婉萍.SNP 分子标记及其在木本植物遗传育种的应用[J].亚热带植物科学,2018,47(2):187-193.ZHOU Lin,DUAN Yu,WEN Bo,MA Yuanchun,ZHU Xujun,WANG Yuhua,FANG Wanping. Research and application of SNP molecular markers in ligneous plants genetics and breeding[J]. Subtropical Plant Science,2018,47(2):187-193.
[17] 崔建民,刘红霞,邹荣仟,梁英海,李亚东,温景辉.越橘种质资源遗传多样性和亲缘关系研究[J].果树学报,2010,27(3):373-378.CUI Jianmin,LIU Hongxia,ZOU Rongqian,LIANG Yinghai,LI Yadong,WEN Jinghui. Study on genetic diversity and phylogenetic relationship of germplasm resources about Vaccinium spp.[J].Journal of Fruit Science,2010,27(3):373-378.
[18] 孙海悦,郭瑞雪,徐鉴,田友文,董梅,董坤,李亚东.越橘属植物EST-SSR 分子标记开发及遗传多样性分析[J].植物生理学报,2018,54(3):429-440.SUN Haiyue,GUO Ruixue,XU Jian,TIAN Youwen,DONG Mei,DONG Kun,LI Yadong. Development of EST-SSR molecular markers for Vaccinium and genetic diversity analysis[J].Plant Physiology Journal,2018,54(3):429-440.
[19] 魏世平,刘晓芬,杨胜先,吕海燕,牛远,章元明.中国栽培大豆群体结构不同分类方法的比较[J].南京农业大学学报,2011,34(2):13-17.WEI Shiping,LIU Xiaofen,YANG Shengxian,LÜ Haiyan,NIU Yuan,ZHANG Yuanming. Comparison of various clustering methods for population structure in Chinese cultivated soybean[Glycine max(L.)Merr.][J].Journal of Nanjing Agricultural University,2011,34(2):13-17.
[20] 刘有春,魏永祥,王兴东,刘成,蒋明三,张舵,袁兴福,陶承光.南高丛越橘品种‘Sapphire’和北高丛品种‘Berkeley’正反交后代果实糖酸组分含量的遗传倾向[J].中国农业科学,2014,47(24):4878-4885.LIU Youchun,WEI Yongxiang,WANG Xingdong,LIU Cheng,JIANG Mingsan,ZHANG Duo,YUAN Xingfu,TAO Chengguang.Inheritance tendency of sugar and acid contents in the reciprocal cross progenies’fruits of southern×northern high bush blueberry (Vaccinium)[J]. Scientia Agricultura Sinica,2014,47(24):4878-4885.
[21] 刘有春,鄂辉邦,刘成,王兴东,杨艳敏,孙斌,张舵,袁兴福,魏永祥.越橘半同胞系杂交后代果实糖酸性状的变异和遗传倾向[J].果树学报,2016,33(6):664-675.LIU Youchun,E Huibang,LIU Cheng,WANG Xingdong,YANG Yanmin,SUN Bin,ZHANG Duo,YUAN Xingfu,WEI Yongxiang.Variability and inheritance of sugar and acid content in blueberry (Vaccinium) fruit of half-sib crosses[J]. Journal of Fruit Science,2016,33(6):664-675.
[22] 刘有春,刘威生,王兴东,杨艳敏,魏鑫,孙斌,张舵,杨玉春,刘成,李天忠.基于越橘南高丛与北高丛正反交群体的果实贮藏相关性状筛选及其遗传倾向[J]. 中国农业科学,2020,53(19):4045-4056.LIU Youchun,LIU Weisheng,WANG Xingdong,YANG Yanmin,WEI Xin,SUN Bin,ZHANG Duo,YANG Yuchun,LIU Cheng,LI Tianzhong.Screening and inheritance of fruit storagerelated traits based on reciprocal cross of southern × northern high bush blueberry (Vaccinium Linn.)[J]. Scientia Agricultura Sinica,2020,53(19):4045-4056.
[23] 刘有春,王兴东,王宏光,王升,魏鑫,袁兴福,魏永祥,刘成,陶承光. 早熟蓝莓新品种辽蓝513 的选育[J/OL]. 果树学报.[2023-8-1].https://doi.org/10.13925/j.cnki.gsxb.20230289.LIU Youchun,WANG Xingdong,WANG Hongguang,WANG Sheng1,WEI Xin,YUAN Xingfu,WEI Yongxiang,LIU Cheng,TAO Chengguang. Liaolan 513:A new early-ripening blueberry cultivar[J/OL]. Journal of Fruit Science. [2023-8-1]. https://doi.org/10.13925/j.cnki.gsxb.20230289.