葡萄(Vitis spp.)为葡萄科(Vitaceae)葡萄属(Vitis L.)的多年生落叶藤本植物,是世界上广泛栽培的经济果树之一[1]。近年来,我国葡萄优质高效标准化栽培模式及管理技术发展迅猛,栽培面积、产量均居世界前列,葡萄产业已经成为我国果树产业的重要组成部分[2-3]。目前,我国栽培的葡萄多数为欧洲葡萄(V.vinifera L.)品种,具有品质优良但抗寒性差的特点,在越冬和“倒春寒”侵袭过程中,常出现葡萄枝蔓冻伤、花芽冻干、果实冻落,甚至树体死亡等现象,严重影响了葡萄产量、经济效益和产业发展[2,4]。为防止冻害,我国北方葡萄产区多采用葡萄树体埋土的方式,这既增加成本,又对树体产生伤害,影响产量,还不利于环境保护[5-6]。因此,挖掘葡萄中与抗寒相关的基因并研究其功能和作用机制,对解决葡萄冻害问题具有重要的理论意义和实际应用价值。
植物激素在植物生长发育和抵抗逆境胁迫中都有不可或缺的作用。茉莉酸(jasmonic acid,JA)及其衍生物茉莉酸甲酯(methyl jasmonate,MeJA)是主要的茉莉酸类化合物(JAs)[7]。JAs作为一种广泛存在于植物中的内源生长调节物质和信号分子,在调控植物生长发育、次生代谢以及生物和非生物胁迫的防御反应中发挥着重要作用[8-10]。研究发现,在低温胁迫条件下植物内源JAs 含量会增加,而施用外源JAs 则会通过提高抗氧化酶活性、增强抗氧化剂和防御化合物的合成、促进热激蛋白的激活和诱导冷响应基因的表达形式来增强植物的抗寒性,包括番茄(Solanum lycopersicum)[11]、葡萄(V. vinifera)[12]、香蕉(Musa paradisiaca)[13]等。在正常条件下,JA 信号通路中的抑制因子JAZ 蛋白(jasmonate ZIM-domain)通过阻遏ICE1 蛋白(inducer of CBF expression)、MYC2(myelocytomatosis proteins)的激活,来抑制下游冷响应基因的表达;植物受到冷胁迫时内源JAs 含量增加,JA 活性因子JA-Ile(jasmonic acid-isoleucine)大量积累,促使JAZ 蛋白与SCFCOI1(Skp1-Cul1-F-box protein coronatine insensitive 1)的结合使得JAZ蛋白泛素化后被26S蛋白酶降解,解除对ICE1、MYC2 的阻遏,激活下游冷响应基因CBF(C-repeat binding factor)、DREB(dehydration responsive element binding factor)和ICE41的表达[14-15]。
JAZ 蛋白是植物中特有的锌指蛋白,定位于细胞核中,属于TIFY 蛋白家族,由ZIM(zinc-finger protein expressed in inflorescence meristem)、Jas(jasmonates)和NT(N terminal domain)3 个 结 构 域 组成[16-17]。ZIM和Jas是JAZ蛋白主要含有的2个特定保守区,位于N 端的ZIM(又称TIFY)结构域,由包含1 个TIFY 基序(TIF[F/Y]XG)在内的28 个氨基酸组成,该结构域介导JAZ 蛋白同源或异源二聚体的形成以及与NINJA(novel interactor of JAZ)等抑制子的互作[18];位于C 端的Jas(又称CCT_2)结构域,可以与MYC、ICE 蛋白的ACT 和TAD 区域有着广泛互作,也是JA-Ile(jasmonic acid-isoleucine)与COI1(coronatine insensitive 1)相互作用的关键[15,19];NT是JAZ蛋白N端的1个弱保守区,该结构域可以与DELLA 蛋白互作抑制JA 信号[20]。此外,葡萄中有11 个JAZs 蛋白,但目前行使的功能还不明了[21]。将毛葡萄(V.quinquangularis)VqJAZ4、VqJAZ7基因在拟南芥(Arabidopsis thaliana)中异源表达可以减轻白粉病发病症状,增强对白粉病的抗性,但也提高了对灰霉病菌的易感性[22-23]。本研究克隆欧洲葡萄霞多丽中JAZ 家族基因,获得其开放阅读框(open reading frame,ORF)序列,并进行了序列分析,同时对JAZ家族成员基因进行实时荧光定量PCR(quantitative real-time PCR,qRT-PCR)分析,获得葡萄在低温胁迫反应中差异表达的JAZs基因,然后将差异表达的JAZs 基因利用酵母双杂交技术筛选互作蛋白并进行验证,为葡萄抗寒控机制的研究和抗寒葡萄品种的选育提供理论基础与参考。
1 材料和方法
1.1 试验材料及处理
试验于2022 年3 月—9 月在宁夏大学农学院宁夏优势特色作物现代分子育种重点实验室进行。试验材料为2 年生欧洲葡萄霞多丽(V. vinifera L.‘Chardonnay’)盆栽扦插苗,于植物培养间(25 ℃,16 h光照,8 h黑暗)进行培养。选取生长状况良好,长势一致、无病虫害的盆栽葡萄苗为试验植株,试验植株为同一批扦插育苗且萌芽后25 d 的葡萄苗,将选取的试验植株平均分为2 组,一组放置于低温培养箱内(立德泰勀,上海)分别进行4 ℃低温处理,另一组正常培养为对照(CK),光照条件一致,每个处理设置3 次生物学重复。分别在0、3、6、12、24 h 收集2 组供试葡萄苗从顶端往下数的第3 和第4 枚展开的叶片为试验样品,立即放入液氮中冷冻,然后置于-80 ℃冰箱保存,用于后续试验(图1)。

图1 葡萄4 ℃低温处理与样品采集
Fig.1 Grape 4 ℃low temperature treatment and sample collection
1.2 葡萄JAZ家族基因的鉴定
从TAIR 数 据 库(https://www.arabidopsis.org/)中获取拟南芥JAZ家族蛋白序列信息作为搜寻葡萄同源基因的探针序列。利用Pfam 数据库(http://pfam.xfam.org/)下 载TIFY(PF06200)和CCT _2(PF09425)结构域的隐马尔可夫模型文件,使用HMMER 3.0 软件筛选出葡萄全基因组蛋白序列中含有这2 个结构域的候选蛋白。利用NCBI 数据库(https://www.ncbi.nlm.nih.gov/)、Pfam 数 据 库 和SMART(http://smart.embl-heidelberg.de/)进一步验证候选VvJAZ 蛋白的保守结构域,以确定葡萄JAZ家族成员基因。在NCBI 网站和葡萄基因组网站(http://www.genoscope.cns.fr/externe/GenomeBrowser/Vitis/)中获取JAZ家族成员基因的编码序列(coding sequence,CDS)、基因组序列、外显子数量以及在染色体上的位置信息。采用在线软件ExPASy ProtParam(https://web.expasy.org/protparam/)分 析VvJAZs 基因编码的蛋白的理化性质,采用MEGA 7.0软件中的邻接法(neighbor-joining,NJ)构建蛋白系统进化树(bootstrap设为1000)。
1.3 葡萄JAZ家族基因CDS的克隆
使用Plant RNA kit 试剂盒(Omega,美国)分别提取4 ℃低温处理后0、3、6、12和24 h叶片总RNA,然后按照PrimeScript™ⅡStrand cDNA Synthesis Kit剂盒试(TaKaRa,日本)说明书合成第一链cDNA。以混合cDNA 为模板,使用基因特异性引物对葡萄JAZ家族成员基因的CDS序列进行克隆。运用在线网站Oligo Calc: Oligonucleotide Properties Calculator(http://biotools.nubic.northwestern.edu/OligoCalc.html)设计同源克隆所用的全部引物(表1)。参考俞沁含等[24]的反应体系与程序进行PCR 反应。PCR 产物经过凝胶检测、胶回收后获得相应的目的片段,经连接反应与pMD19-T 载体相连接,将重组产物转化大肠杆菌(Escherichia coli)Top10 感受态细胞,经阳性克隆筛选后,送单克隆至北京奥科鼎盛生物科技有限公司测序确认,获得含有目的基因的重组载体。
表1 引物列表
Table 1 List of primers

引物名称Primer name VvJAZ9-F VvJAZ9-R pGBKT7-VvJAZ9-F pGBKT7-VvJAZ9-R pGADT7-VvHAP5A-F pGADT7-VvHAP5A-R pBI221-NE-VvHAP5A-F pBI221-NE-VvHAP5A-R pBI221-CE-VvJAZ9-F pBI221-CE-VvJAZ9-R VvJAZ1-F VvJAZ1-R VvJAZ2-F VvJAZ2-R VvJAZ3-F VvJAZ3-R VvJAZ4-F VvJAZ4-R VvJAZ5/6-F VvJAZ5/6-R VvJAZ7-F VvJAZ7-R VvJAZ8-F VvJAZ8-R VvJAZ9-F VvJAZ9-R VvJAZ10-F VvJAZ10-R VvJAZ11-F VvJAZ11-R bZIP-F bZIP-R HAP5A-F HAP5A-R CONSTANS-LIKE 2-F CONSTANS-LIKE 2-R STOP1-F STOP1-R CSN5b-F CSN5b-R NINJA-F NINJA-R HAP5B-F HAP5B-R GAI2-F GAI2-R GAI1-F GAI1-R VvActin-F VvActin-R引物序列(5’-3’)Primer sequence(5’-3’)ATGTCGAGCTCCTCGGATATTGC CTACTTTGGAGATTTGGCAGCCAAG TATGGCCATGGAGGCCGAATTCATGTCGAGCTCCTCGGATATTGC GCCGCTGCAGGTCGACGGATCCCTACTTTGGAGATTTGGCAGCCAAG GCCATGGAGGCCAGTGAATTCATGGAGAACAACCAGCAGGCCCAAT CAGCTCGAGCTCGATGGATCCTTATACATACCCTTGGCCATCAAGATTCC GGCCCAGGCCTACTAGTGGATCCATGGAGAACAACCAGCAGGCCCAAT TACCCGGGAGCGGTACCCTCGAGTTATACATACCCTTGGCCATCAAGATTCC GAGAACACGGGGGACTCTAGAATGTCGAGCTCCTCGGATATTGC GTACATGGTACCCCGCTCGAGCTTTGGAGATTTGGCAGCCAAG TGGCAGCAGGTGATGGTGTC AGACTGAGCAACAGGATGTGAAGA CACTGGCACGAAGGAAGTCATTG GCGTATTGGATGTGAGGTGGATATG AGACGCTATCTCGCCATCTCTG CTTCCATCCTATTCTTCCGCTTCTG GCTCCTCTTGGCTCTGAACTG TGGAGAAGGTCTGGATGGATTGT TGATGGAGACGGGAAGAGGAAG TGAGGCAGTCGGGGTTCTTGT CTCCACTGCTCACACTTCTTCAAT TGCCGTTGCTGTTGCTCTTG GCCTTCTTCCCTCCACTGCTTA TGCTGCTGCTGCTGCTCTT CGGCTCAACCAGTTGCTTGT GTGCTCTTGCTGTGATCCTATCTT CACCGTCAACAACACTACAATCCTA GAGTAGCCAAGCCACTTATTCCAT CTGCTTCATCTAATGTTGGCTCTCA GGAGGCTCTGGTTGGTTGGTAG ACAGCAGCAGCAGCAACCA GTCACAGCATCAGTGCCAGAAC GGTCCTACCAACGCCAAGAGAT GCACATCCTCATCCGCCTTCA CGCTGACATCCACTCCGCTAAT CTGCCTCGTCTTCGTCTTCCTC GAGGAGCCACTTGAAGCACTGT CTGCTACTGCTGCTGCTGACA GCCTTCGCTTTGCCCGTTGA GTCCTGCCTGCTTGTTGGTCTG CAACCTCAAGGCTGGCTTCACA GAACCTCCTTTGCGACCTCAGA AACTGCTCCAATGGTGGCTTCTG GCTGCTGTTGATGCTGCTGTTG CGTCGTCGTCGTCTTCGTCAT CCTGCGAGTCCACCACTACAAC TCCACACCTGCCTCCAACTGA ACACAGCACATGCCACAAGGA CTATCCTTCGTCTTGACCTTGCTG AGTGGTGAACATGTAACCCCTCTC用途Purpose基因克隆Gene cloning酵母双杂Yeast two-hybrid BiFC验证BiFC verification表达分析Expression analysis
1.4 诱饵表达载体的构建及自激活检测
利用Primer Premier 6.0 软件设计VvJAZ9 基因的全长特异引物(表1),以欧洲葡萄霞多丽叶片的cDNA 为模板进行PCR 反应,胶回收目的片段。同时利用EcoRⅠ和BamHⅠ双酶切诱饵载体pGBKT7后,将回收的PCR产物与酶切后的线性载体进行同源重组,随后转化至E.coli DH5α 感受态细胞,经阳性克隆筛选后,送单克隆至公司测序,测序准确后获得重组质粒pGBKT7-VvJAZ9。以pGBKT7-p53+pGADT7-T 为阳性对照,pGBKT7-Lam+pGADT7-T为阴性对照,pGBKT7 为空载对照,pGBKT7-VvJAZ9+pGADT7 为自激活检测组,采用PEG/LiAc法将其共转化至酵母Y2H Gold感受态细胞中,涂布于SD/-Leu/-Trp/X-α-Gal(DDO/X)培养基上,再将自激活检测组另涂布于SD/-Leu/-Trp/-His/X-α-Gal(TDO/X)和SD/-Leu/-Trp/-His/-Ade/X-α-Gal/AbA(QDO/X/A)培养基上,在恒温培养箱中30 ℃倒置培养3~4 d,观察菌落的生长和颜色变化,确定诱饵蛋白是否具有自激活活性[25]。
1.5 酵母双杂交cDNA文库的构建与VvJAZ9互作蛋白的筛选
运用CloneMiner™ⅡcDNA Library Construction Kit 试剂盒(Invitrogen,美国)构建酵母双杂交cDNA文库,参考韩雪等[25]的方法进行,具体操作步骤如下,选取4 ℃低温处理0、3、6、12和24 h的霞多丽葡萄植株叶片,分别提取总RNA,各取5 μg 等质量均匀混合,根据Oligotex mRNA Midi Kit 试剂盒(Qiagen,德国)说明书对mRNA 进行分离纯化,参考CloneMiner™ⅡcDNA Library Construction Kit试剂盒说明书将分离纯化后的mRNA 反转录合成双链cDNA。将cDNA 与三框attB1 重组接头连接(3种接头分别各连接1份)后,混匀加入TEN Buffer清洗过的分级柱,对cDNA 分级分离及收集。将收集的cDNA与pDONR222载体进行BP重组反应,反应产物通过电转化至E.coli DH10B感受态细胞,加入4 mL SOC 培养基,37 ℃,225 r·min-1培养1 h 后,获得初级文库菌液,并进行库容量、重组率和插入片段长度的鉴定。从检验合格的初级文库(Uncut型)中抽提质粒,将得到的质粒质量浓度调整到300 mg·L-1后与pGADT7-DEST进行LR重组反应,产物经电转化至E.coli DH10B,37 ℃,225 r·min-1培养1 h 后得到次级文库菌液,并进行文库质量鉴定,将次级文库菌液涂布在氨苄平板培养基上,37 ℃过夜培养后,用液体培养基洗脱后抽提质粒,用于后续酵母双杂交共转化。
将10 μg pGADT7 文库质粒与5 μg pGBKT7-VvJAZ9诱饵质粒共转化Y2H Gold感受态细胞。先涂布在SD/-Leu/-Trp/-His/X-α-Gal(TDO/X)固体培养基上,30 ℃倒置培养3~5 d,待单克隆长至1~2 mm时,初筛完成。再挑取TDO/X平板上的蓝色阳性克隆转移到SD/-Leu/-Trp/-His/-Ade/X-α-Gal/AbA(QDO/X/A)固体培养基上进行复筛,然后置于恒温培养箱中30 ℃倒置培养3~4 d 后,挑取阳性克隆并从中提取酵母质粒。然后将>500 bp的PCR产物送至公司进行测序,在NCBI Blastx(https://blast.ncbi.nlm.nih.gov/Blast)进行比对分析候选互作蛋白。挑选含正确ORF的候选互作蛋白,根据其序列设计引物(表1),克隆至pGADT7 载体上,并与pGBKT7-VvJAZ9共转化Y2H Gold感受态细胞中进行回转验证[26]。
1.6 双分子荧光互补试验验证候选蛋白互作关系
设计VvJAZ9和VvHAP5A基因的ORF中不含终止密码子的同源重组引物(表1),通过双分子荧光互补试验(bimolecular fluorescent complimentary,BiFC),将其构建至BiFC 验证载体pB221-cYFP、pB221-nYFP,形 成pB221-JAZ9-cYFP 和pB221-HAP5A-nYFP。然后将构建好的pB221-JAZ9-cYFP和pB221-HAP5A-nYFP及对照载体分别大提质粒之后将所获得的质粒通过乙醇沉淀法浓缩至1500~2000 mg·L-1。采用酶解法分离拟南芥叶肉组织原生质体,通过PEG(polyethylene glycol)介导法分别将质粒两两组合共转至拟南芥原生质体,22 ℃弱光孵育18 h进行瞬时表达,使用激光共聚焦显微镜(Leica TCS SP8 X,德国)观察并拍照。
1.7 实时荧光定量PCR
利用qRT-PCR检测低温胁迫下葡萄JAZ家族成员基因和与VvJAZ9互作候选蛋白的相关基因的表达情况。qRT-PCR 所用引物均使用Primer Premier 6.0 软件设计(表1)。使用Plant RNA kit 试剂盒(Omega,美国)分别提取对照(CK)和4 ℃低温处理0、3、6、12、24 h 的叶片样品总RNA,各取1 μg 通过PrimeScript™RT reagent Kit with gDNA Eraser 试剂盒(TaKaRa,日本)将RNA 反转录为cDNA,稀释10倍后用于后续的qRT-PCR 分析。qRT-PCR 反应用SYBR® Premix Ex TaqTM (Perfect Real Time)试剂盒(TaKaRa,日本)进行;反应体系为:上下游引物各0.8 μL,模板1 μL,SYBR 试剂10 μL,用ddH2O 补齐至20 μL;反应程序为:95 ℃预变性10 min,95 ℃变性15 s,60 ℃退火1 min,72 ℃延伸30 s,循环40次;每次循环第2步进行荧光采集,使用qTOWER 2.0仪器(Analytik Jena,德国)进行qRT-PCR 检测,每个样品设置3 次生物学重复,采用2-ΔΔCT法计算基因的相对表达量。
1.8 数据统计与分析
使用Excel 2019 整理试验数据,利用IBM SPSS 25.0 对试验数据进行统计分析,并采用Student’s t-test 进行显著性分析(p<0.05),然后使用Origin Pro 2021 作图。试验数据以(平均值±标准差)表示。
2 结果与分析
2.1 葡萄JAZ家族成员基因的鉴定与分析
运用生物信息学手段从葡萄基因组中鉴定出11 个JAZ 家族成员基因,根据参考序列在染色体上的分布将所获得的JAZ 家族基因分别命名为VvJAZ1~VvJAZ11。由表2 可知,这11 个VvJAZs 基因不均匀地分布在7条染色体上,其中1号染色体含有2个VvJAZs基因,10号染色体含有4个VvJAZs基因,有5个VvJAZs基因分别位于4、9、11、12、17号染色体上。 这些VvJAZs 基因CDS 长度为297(VvJAZ3)~1167(VvJAZ1)bp,外显子有2~8 个,编码氨基酸序列长度为98(VvJAZ3)~388(VvJAZ1)aa,分子质量最大为40.31 ku,最小为11.26 ku,等电点在9.19~10.46 之间,不稳定系数在46.91~85.17 之间。11个VvJAZ蛋白的GRAVY(grand average of hydropathicity)值为均负值,表明葡萄JAZ 家族蛋白为碱性不稳定亲水蛋白。
表2 葡萄JAZ 家族成员的基因信息
Table 2 Basic information of JAZ gene family in grape

基因名称Gene name VvJAZ1 VvJAZ2 VvJAZ3 VvJAZ4 VvJAZ5 VvJAZ6 VvJAZ7 VvJAZ8 VvJAZ9 VvJAZ10 VvJAZ11基因登录号GenBank No.XM_002284819.2 XM_002262714.3 XM_003634778.3 XM_002272327.4 XM_002277733.4 XM_002277769.4 XM_002277916.4 XM_002271161.4 XM_002277121.3 XM_002263220.4 XM_002282652.4染色体Chromosome Chr1 Chr1 Chr4 Chr9 Chr10 Chr10 Chr10 Chr10 Chr11 Chr12 Chr17外显子Exon 76253323568编码区CDS/bp 1167 639 297 861 384 384 432 366 807 702 1137氨基酸数Amino acid number/aa 388 212 98 286 127 127 143 121 268 233 378等电点pI 9.19 9.24 9.69 9.21 9.77 9.77 10.46 9.55 9.82 9.51 9.24分子质量Molecular mass/ku 40.31 23.75 11.26 30.66 14.73 14.72 16.10 13.74 28.57 26.31 39.36不稳定系数Instability index 46.91 56.84 78.61 55.41 79.09 78.12 84.93 85.17 47.51 68.54 47.82亲水性Hydropathicity-0.226-0.467-0.777-0.707-0.913-0.926-0.785-0.560-0.404-0.735-0.210
2.2 葡萄JAZ家族基因克隆与聚类分析
以4 ℃低温处理后不同时间节点的葡萄叶片的混合cDNA为模板通过同源克隆的方式从欧洲葡萄霞多丽中克隆到11 个JAZ 家族成员基因。结果表明,克隆到葡萄中的JAZs基因与所预测的葡萄基因组中ORF 长度和所编码的氨基酸序列长度基本一致(图2)。
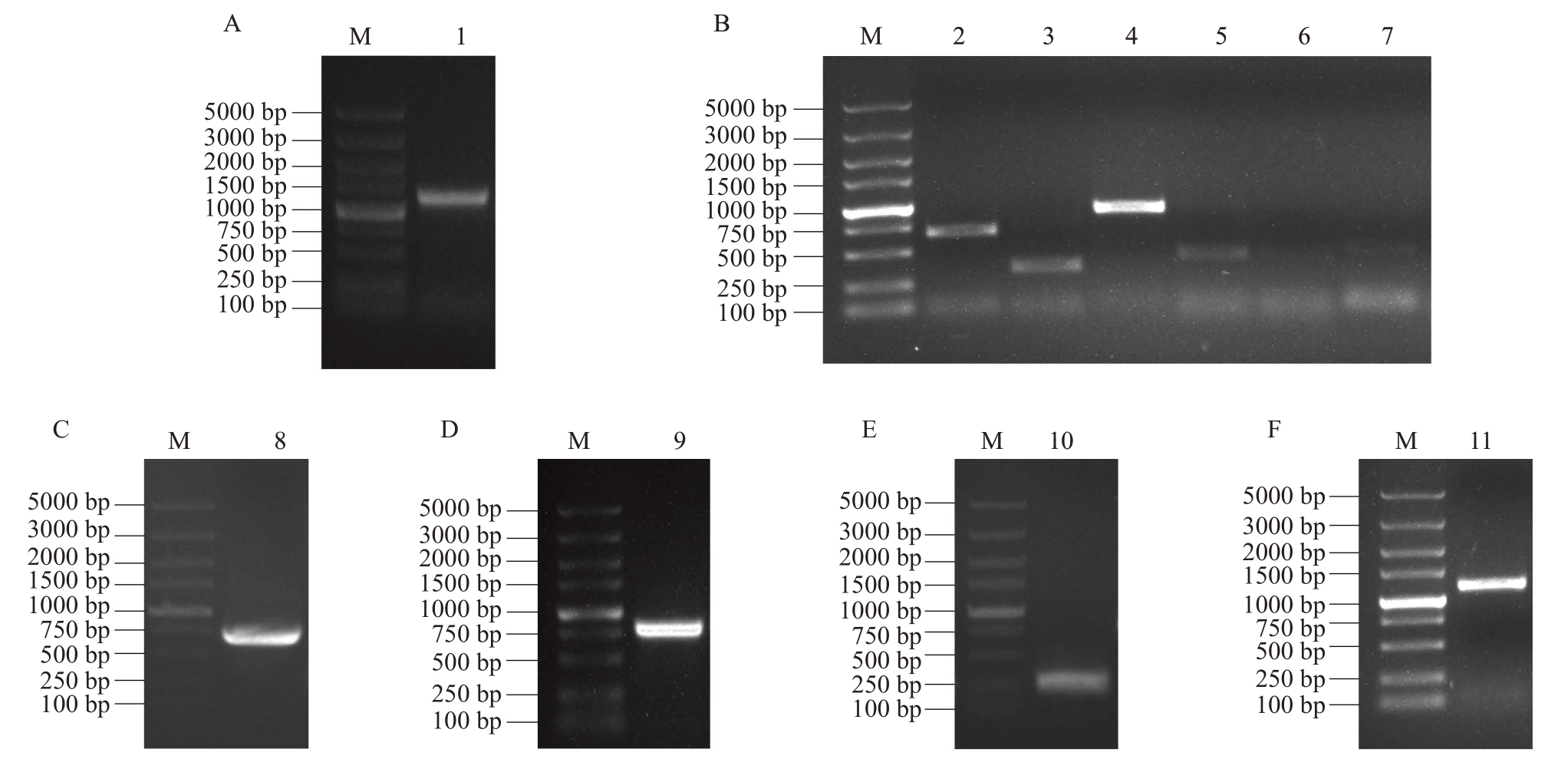
图2 葡萄JAZ 家族基因克隆
Fig.2 Cloning of JAZ family genes in grape
A. VvJAZ1 基因克隆;B. VvJAZ2、VvJAZ3、VvJAZ4、VvJAZ5、VvJAZ6、VvJAZ7 基因克隆;C. VvJAZ8 基因克隆;D. VvJAZ9 基因克隆;E.VvJAZ10 基因克隆;F.VvJAZ11 基因克隆。M.DL5000 bp DNA Marker;1~11.目的基因。
A. Cloning of VvJAZ1 gene;B. Cloning of VvJAZ2,VvJAZ3,VvJAZ4,VvJAZ5,VvJAZ6 and VvJAZ7 genes; C. Cloning of VvJAZ8 gene;D. Cloning of VvJAZ9 gene;E.Cloning of VvJAZ10 gene;F.Cloning of VvJAZ11 gene.M.DL5000 bp DNA Marker;1-11.Target gene.
对获得的葡萄JAZ家族蛋白序列进行多重比对分析,发现11 个VvJAZ 蛋白序列中都含有TIFY 和Jas这2个高度保守的结构域,符合JAZ家族类型特点(图3-A)。葡萄JAZ家族成员的氨基酸序列聚类分 析 显 示,VvJAZ1 与VvJAZ11、VvJAZ4 与VvJAZ9、VvJAZ5 与VvJAZ6 的氨基酸相似度最高(图3-B)。从葡萄JAZs蛋白与拟南芥中的JAZ蛋白的聚类结果可以看出(图3-C),葡萄JAZ 家族蛋白可分为3个亚族;在亚组Ⅰ中的VvJAZ4、VvJAZ9与拟南芥中AtJAZ1 和AtJAZ2 的亲缘关系最近;在亚组Ⅱ中的VvJAZ1、VvJAZ11 与拟南芥中AtJAZ9 的进化距离最近;在亚组Ⅲ中的VvJAZ3、VvJAZ5、VvJAZ6、VvJAZ7、VvJAZ8 与拟南芥中AtJAZ7 和AtJAZ8 的亲缘关系最近;其中,VvJAZ1 与VvJAZ11、VvJAZ5 与VvJAZ61 同源关系最近,推测其在蛋白功能上具有一定的相似性。

图3 葡萄JAZs 蛋白序列比对和聚类分析
Fig.3 Sequence alignment and cluster analysis of grape JAZs protein
A.葡萄JAZ 家族蛋白的多序列比对,图中点表示序列比对空位,黑线区域表示TIFY 和Jas 保守结构域;B.葡萄JAZ 家族氨基酸聚类分析;C.葡萄JAZs 蛋白与拟南芥JAZ 家族成员的系统进化分析。
A.Multiple sequence alignment of grape JAZs protein;The dots represent sequence alignment vacancies,and the black line region represent conserved domains of TIFY and Jas;B.Amino acid cluster analysis of JAZe family in grape;C.Phylogenetic analysis of grape JAZs protein and Arabidopsis JAZ family members.
2.3 葡萄JAZ家族基因在低温胁迫下的表达分析
对所获得的葡萄JAZ家族基因的核苷酸序列聚类分析后发现,VvJAZ5 和VvJAZ6 的核苷酸相似度达到96.88%,因而在设计qRT-PCR 引物时选择VvJAZ5与VvJAZ6的同源片段区域,命名为VvJAZ5/6。qRT-PCR分析结果表明(图4),在欧洲葡萄JAZs基因家族中的VvJAZ9 基因受低温诱导表达最为明显,在0 h 相对表达量最低,在24 h 达到最高;VvJAZ2和VvJAZ10基因均呈现先上升再下降、后期又逐渐上升的表达趋势;VvJAZ3、VvJAZ5/6 及VvJAZ8 基因在12 h 以前相对表达量均处于较低水平,但在24 h 达到顶峰;VvJAZ1 基因在受到低温处理后相对表达量呈现出先降低后又逐渐上升的表达模式,而VvJAZ11 基因在受到低温处理后相对表达量呈现逐渐下降的表达趋势,在24 h相对表达量最低。这11个VvJAZs基因中仅有VvJAZ9基因的相对表达量随着低温时间延长而呈现显著上升趋势,是受低温诱导表达显著的基因,推测该基因可能响应低温调控,参与葡萄抵御冷胁迫的应答过程。

图4 VvJAZ 家族基因在低温条件下qRT-PCR 分析
Fig.4 qRT-PCR analysis of VvJAZ family genes under low temperature
不同小写字母表示在p<0.05 水平差异显著。下同。
Different lowercase letters indicate significant difference at p<0.05 level.The same below.
2.4 诱饵载体自激活检测
在筛库之前,需将构建好的pGBKT7-VvJAZ9诱饵载体进行自激活检测。在SD/-Leu/-Trp/X-α-Gal(DDO/X)培养基上阳性对照(pGBKT7-p53+pGADT7-T)有蓝色酵母菌落生长,而阴性对照(pGBKT7-Lam+pGADT7-T)有菌落生长但未显现蓝色,说明阳性和阴性对照试验成功(图5)。pGBKT7 空载体和pGBKT7-VvJAZ9+pGADT7 在SD/-Leu/-Trp/X-α-Gal 上 生 长,在SD/-Leu/-Trp/-His/X-α-Gal(TDO/X)和SD/-Leu/-Trp/-His/-Ade/X-α-Gal/AbA(QDO/X/A)上不生长,说明pGBKT7-VvJAZ9 质粒成功转入酵母菌株中,且无自激活活性,可以用于后续筛选试验(图6)。
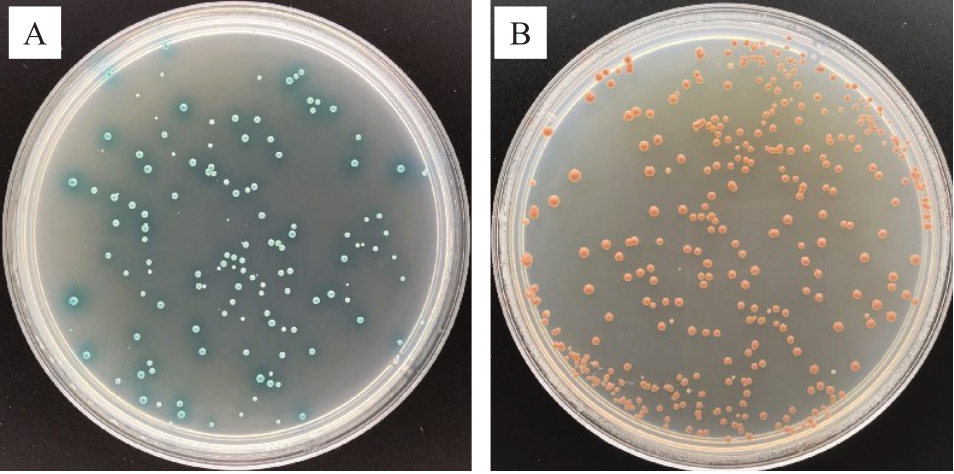
图5 酵母双杂交诱饵载体检测阳性对照与阴性对照
Fig.5 Positive and negative control of yeast two-hybrid bait vector detection
A.阳性对照(pGBKT7-p53+pGADT7-T);B.阴性对照(pGBKT7-Lam+pGADT7-T);A 和B 的培养基为DDO/X。
A.Positive control(pGBKT7-p53+pGADT7-T);B.Negative control(pGBKT7-Lam+pGADT7-T);The medium of A and B is DDO/X.

图6 诱饵蛋白自激活检测
Fig.6 Self-activation detection of the bait protein
A.DDO/X 培养基;B.TDO/X 培养基;C.QDO/X/A 培养基;图示为JAZ9 诱饵蛋白自激活检测,A、B 和C 代表诱饵载体在不同培养基上生长情况。
A.DDO/X medium;B.TDO/X medium;C.QDO/X/A medium;The picture shows is the self-activation detection of JAZ9 bait protein,A,B and C represent the growth of bait vector on different media.
2.5 酵母双杂交筛选VvJAZ9互作蛋白
2.5.1 文库初筛与复筛 将pGBKT7-VvJAZ9 诱饵质粒和构建的酵母双杂交文库质粒共转化Y2H Gold 酵母感受态细胞,转化产物涂布在SD/-Leu/-Trp/-His/X-α-Gal(TDO/X)平板上培养3~5 d,初筛共获得72 个蓝色的酵母克隆(图7-A~B)。在转化过程中检测转化情况,确定转化效率>210 cfu·μg-1,总转化子数>210 cfu。再挑取TDO/X筛选平板上的蓝色阳性克隆转移到SD/-Leu/-Trp/-His/-Ade/X-α-Gal/AbA(QDO/X/A)筛选平板上,共筛选到50个蓝色克隆(图7-C),最终对50个克隆全部测序。
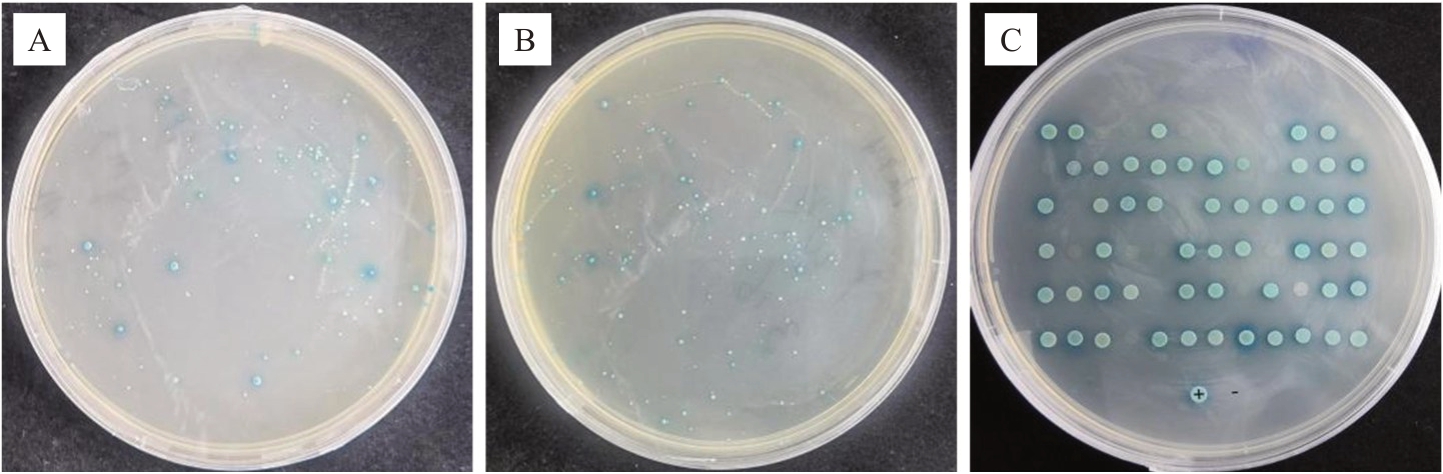
图7 初筛与复筛筛选平板上的阳性克隆
Fig.7 Positive clones on the screening plate of primary and secondary screening
A 和B 是初筛培养基TDO/X,C 是复筛培养基QDO/X/A,“+”和“-”为酵母双杂交阳性对照和阴性对照。
A and B are the primary screening medium TDO/X,C is the secondary screening medium QDO/X/A,“+”and“-”are positive and negative control of yeast two-hybrid.
2.5.2 候选互作蛋白的鉴定 将筛选到的50 个阳性克隆在四缺培养基上扩大培养后,提取酵母质粒,运用pGADT7 载体上通用引物对质粒进行PCR 检测,将所获得的条带中>500 bp 的PCR 产物送公司进行测序,测序结果通过NCBI数据库中进行Blastx比对分析,去除移码的假阳性克隆后,初步获得26个可能与VvJAZ9 互作的候选蛋白,包括核转录因子Y亚基C-9亚型X3、精氨酸脱羧酶、JAZ1蛋白、冠状激素不敏感蛋白、锌指蛋白CONSTANS-like 2、KH结构域蛋白和忍者家族蛋白等(表3)。
表3 VvJAZ9 蛋白酵母双杂交筛选获得的候选互作蛋白
Table 3 Candidate interacting proteins obtained by yeast two-hybrid screening of VvJAZ9

编号Number 1 2 3 45 6 7 8 9登录号Accession No.XP_003633639.1 CAA65585.1 XP_002267588.1 AEP60132.1 XP_002270683.1 RVW44524.1 BAF95867.1 RVW72224.1 RVW95477.1 10 XP_002274576.1 11 XP_002274568.2 12 XP_002283629.2 13 XP_010662714.1 14 CAN73357.1 15 XP_002283561.1 16 XP_002276145.2 17 CAN73357.1 18 CAN59753.1 19 RVX06365.1 20 XP_002267529.1 21 RVW40458.1 22 CAN80969.1 23 XP_003633728.1 24 XP_002283979 25 XP_010664564 26 XP_002266061.1注释Annotation核转录因子Y亚基C-9亚型X3 Nuclear transcription factor Y subunit C-9 isoform X3[V.vinifera]部分精氨酸脱羧酶Arginine decarboxylase,partial[V.vinifera]含有SH3结构域的蛋白2亚型X1 SH3 domain-containing protein 2 isoform X1[V.vinifera]JAZ1[V.rupestris]SNF1相关蛋白激酶调节亚基beta-1 SNF1-related protein kinase regulatory subunit beta-1[V.vinifera]叶绿素a-b结合蛋白CP29.2,叶绿体Chlorophyll a-b binding protein CP29.2,chloroplastic[V.vinifera]假定质体脂相关蛋白,部分Putative plastid lipid-associated protein,partial[Vitis hybrid cultivar]无特征蛋白CK203_059503 Uncharacterized protein CK203_059503[V.vinifera]保守低聚高尔基复合体亚基4 Conserved oligomeric Golgi complex subunit 4[V.vinifera]mec-8和unc-52蛋白同源物2的抑制因子Suppressor of mec-8 and unc-52 protein homolog 2[V.vinifera]蛋白质TIFY 4B亚型X2 Protein TIFY 4B isoform X2[V.vinifera]可能是3-羟基异丁酸脱氢酶样1,线粒体Probable 3-hydroxyisobutyrate dehydrogenase-like 1,mitochondrial[V.vinifera]U-box结构域蛋白32亚型X2 U-box domain-containing protein 32 isoform X2[V.vinifera]假设蛋白VITISV_012625 Hypothetical protein VITISV_012625[V.vinifera]COP9信号体复合体5b亚基COP9 signalosome complex subunit 5b[V.vinifera]冠状激素不敏感蛋白1 Coronatine-insensitive protein 1[V.vinifera]假设蛋白VITISV_012625 Hypothetical protein VITISV_012625[V.vinifera]假设蛋白VITISV_036639 Hypothetical protein VITISV_036639[V.vinifera]锌指蛋白CONSTANS-like 2 Zinc finger protein CONSTANS-like 2[V.vinifera]蛋白质对质子根毒性1敏感Protein sensitive to proton rhizotoxicity 1[V.vinifera]假设异天冬氨酸肽酶/L-天冬酰胺酶2 Putative isoaspartyl peptidase/L-asparaginase 2[V.vinifera]假设蛋白VITISV_032938 Hypothetical protein VITISV_032938[V.vinifera]KH结构域蛋白At4g18375 KH domain-containing protein At4g18375[V.vinifera]忍者家族蛋白mc410亚型X Ninja-family protein mc410 isoform X1[V.vinifera]可能与BOI相关的E3泛素蛋白连接酶2 Probable BOI-related E3 ubiquitin-protein ligase 2[V.vinifera]无特征蛋白At4g06598 bZIP Uncharacterized protein At4g06598 bZIP[V.vinifera]
2.5.3 VvJAZ9 候选互作蛋白的相关基因在低温胁迫下的表达分析 qRT-PCR分析结果如图8所示,在4 ℃低温处理的欧洲葡萄中bZIP(XP_002266061.1)基因表现出先上升后下降的表达趋势,且在3 h相对表达量达到最高;CONSTANS-LIKE 2(RVX06365.1)、STOP1(XP_002267529.1)和GAI2(CAN59753.1)呈现出先下降后逐渐上升的表达趋势,均在6 h相对表达量最低;CSN5B(XP_002283561.1)则呈现出双峰的表达趋势,分别在6 h和24 h相对表达量达到最大;NINJA(XP_002283979)呈现出逐渐下降的表达趋势;HAP5B(XM_002262845.4)和GAI1(XM_002284612.4)表现出先上升后下降的表达趋势,都在3 h相对表达量达到最大;而HAP5A(CAN73357.1)则呈现出逐渐上升的表达趋势,这与已筛选出的VvJAZ9 受低温诱导表达趋势类似。以上结果表明,HAP5A可能是响应低温较为显著的VvJAZ9候选互作蛋白。

图8 VvJAZ9 候选互作蛋白相关基因的qRT-PCR 分析
Fig.8 qRT-PCR analysis of VvJAZ9 candidate interacting protein-related genes
2.5.4 VvJAZ9 与VvHAP5A 回转验证 为进一步验证VvJAZ9与VvHAP5A蛋白互作关系,从霞多丽葡萄cDNA中同源克隆到VvHAP5A的ORF序列,其ORF序列共636 bp,编码211个氨基酸,VvHAP5A基因是1个NF-YC类型的转录因子基因。采用同源重组的方法获得pGADT7-HAP5A 载体,然后将pGADT7-HAP5A 和pGBKT7-JAZ9 共转化酵母 感 受态细胞中进行回转验证。共转化的酵母在SD/-Leu/-Trp培养基上均可正常生长,但在SD/-Leu/-Trp/-His/-Ade/X-α-Gal/AbA(QDO/X/A)培养基上只有pGBKT7-JAZ9+pGADT7-HAP5A 和阳性对照(pGBKT7-p53+pGADT7-T)显现蓝色酵母菌落,并随着稀释倍数的增大,蓝色菌落在减少,说明VvJAZ9与VvHAP5A蛋白具有相互作用(图9)。
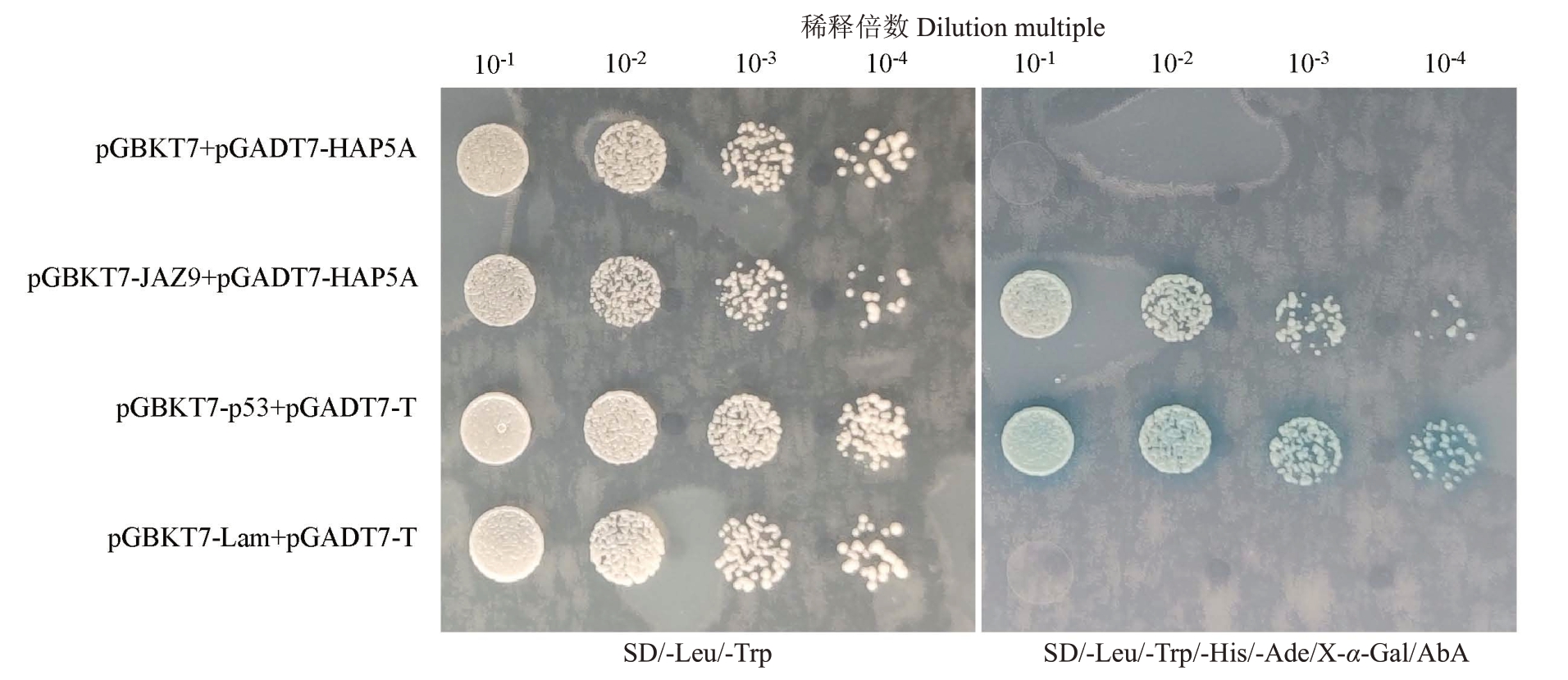
图9 VvJAZ9 与VvHAP5A 候选互作蛋白的回转验证
Fig.9 Rotary verification of VvJAZ9 and VvHAP5A candidate interacting proteins
pGBKT7-p53+pGADT7-T 为阳性对照,pGBKT7-Lam+pGADT7-T 为阴性对照。
pGBKT7-p53+pGADT7-T as positive control,pGBKT7-Lam+pGADT7-T as negative control.
2.6 VvJAZ9与VvHAP5A蛋白互作验证
利用BiFC 试验进一步验证VvJAZ9 与Vv-HAP5A在植物体内的互作关系,结果如图10所示,共转化的cYFPJAZ9+nYFPHAP5A在拟南芥原生质体中可以观察到黄色荧光信号,而cYFPJAZ9+nYFP和cYFP+nYFPHAP5A 均未观察到黄色荧光信号,表明VvJAZ9 和VvHAP5A 蛋白在植物体内存在互作关系。
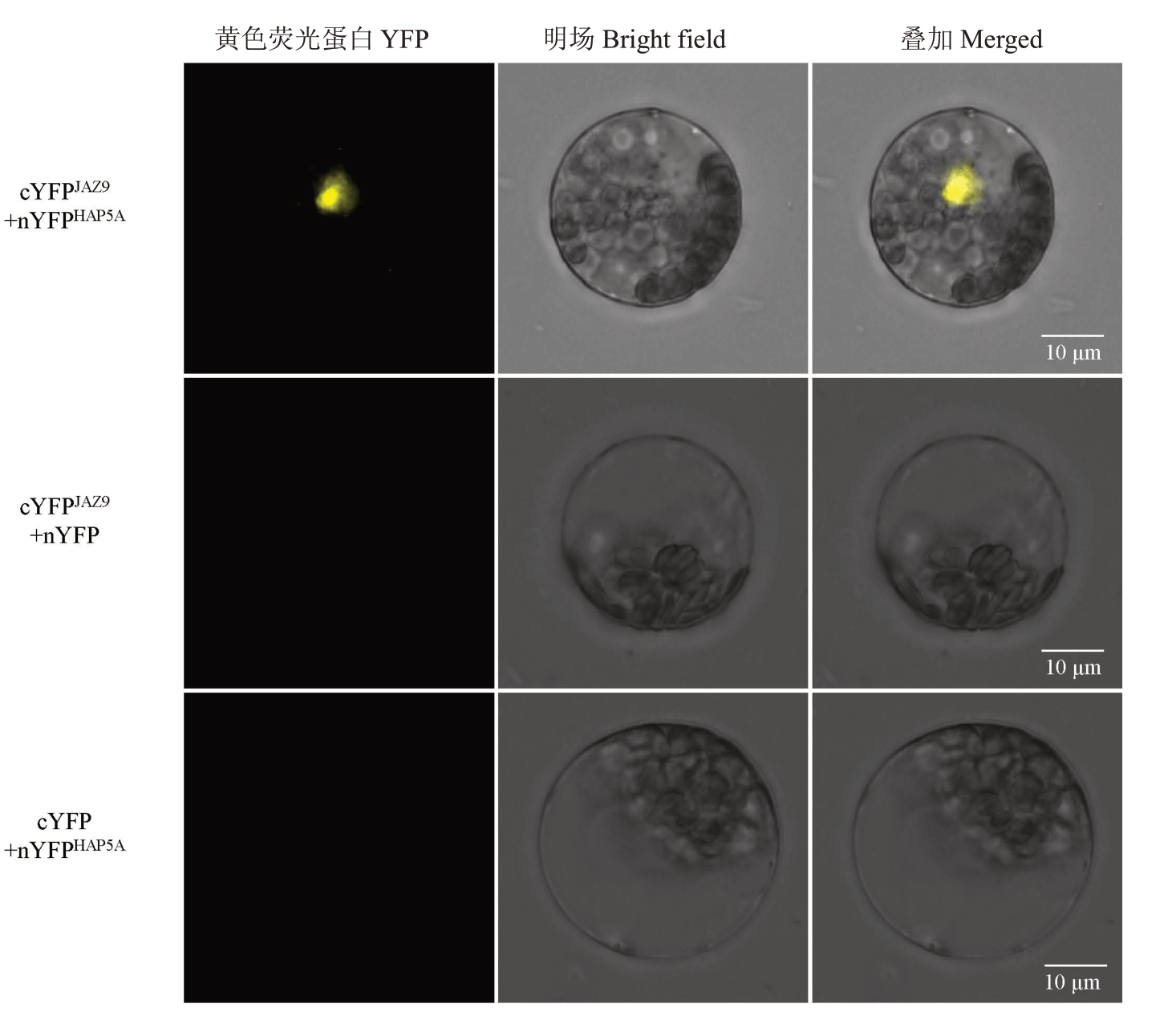
图10 VvJAZ9 与VvHAP5A 蛋白的BiFC 验证
Fig.10 BiFC verification of VvJAZ9 and VvHAP5A proteins
图中白色横线表示10 μm 标尺。
The white horizontal line in the figure indicates 10 μm scale.
3 讨 论
JA 是一种广泛存在于高等植物体内脂肪酸衍生的植物激素,不仅在植物生长发育过程中具有重要的调节作用,而且在植物应对生物和非生物胁迫中也扮演着重要的角色[27]。SCFCOI1泛素蛋白复合体、转录抑制因子JAZ蛋白和转录激活因子MYC蛋白是JA信号途径的3个核心组件,其中JAZ蛋白作为E3泛素连接酶SCFCOI1的靶蛋白和MYC的转录抑制子,是JA信号转导途径的关键组分,在JA调控植物胁迫应答的过程中发挥着重要作用[16]。JAZ 家族成员基因众多,且生物学功能丰富。目前已报道在拟南芥[28]、烟草(Nicotiana tabacum)[29]、小麦(Triticum aestivum)[30]、玉米(Zea mays)[31]、番茄(S. lycopersicum)[32]、黄瓜(Cucumis sativus)[33]、西瓜(Citrul-lus lanatus)[34]等中分别鉴定出12、15、14、38、13、11、8个JAZ成员。
笔者在本研究中利用生物信息学方法,从葡萄全基因组序列中鉴定到11 个葡萄JAZ 家族成员基因,分别命名为VvJAZ1~VvJAZ11,这与前人在葡萄上的筛选鉴定结果一致[21]。这11 个成员不均匀地分布在7条染色体上,但在10号染色体上分布较为密集,这与粗山羊草(Aegilops tauschii)JAZ 家族基因在染色体上的分布研究类似[35]。对这11 个JAZ家族成员的蛋白序列进行比对分析后发现,葡萄中VvJAZ蛋白都具有2个高度保守的TIFY和Jas结构域,属于典型的JAZ 家族蛋白,与甘薯(Ipomoea batatas)中JAZ 蛋白结构研究一致[36]。通过对葡萄JAZ家族成员的理化性质分析发现,11个VvJAZ蛋白的平均亲水性均为负值且等电点均大于7.0,都属于碱性不稳定亲水蛋白,与核桃(Juglans regia)中JAZ 蛋白性质研究一致[37]。根据芥菜(Brassica juncea)[38]与拟南芥JAZ 家族的系统进化树分析结果,将这些JAZ 明显分为5 个亚组(Ⅰ、Ⅱ、Ⅲ、Ⅳ和V),而在本研究中葡萄与拟南芥JAZ家族成员的系统进化树被分为3个亚组;在亚组Ⅰ中的VvJAZ9与拟南芥中AtJAZ1 和AtJAZ2 的亲缘关系最近;在亚组Ⅱ中的VvJAZ1、VvJAZ11 和亚组Ⅲ中的VvJAZ5 与VvJAZ6 同源关系最近,并推测其在蛋白功能上有一定的相似性。
JAZs 基因是茉莉酸信号通路中的关键节点基因,也是植物响应逆境胁迫的关键调控因子[27]。有研究表明,JAZs 基因参与一些植物的低温胁迫,JAZ1 和JAZ4 基因过度表达会使植物对冷胁迫敏感,也会抑制拟南芥中的ICE-CBF信号通路和冷冻反应[14]。JAZ1 基因沉默的拟南芥愈伤组织中钙调素含量显著增加,同时也增强了对冷应激的耐受性[39]。在低温胁迫下,耐低温的棉花(Gossypium hirsutum)品种中GhJAZ1 基因的上调表达更为显著,推测该基因参与棉花低温胁迫防御反应[40]。笔者在本研究中通过对葡萄JAZ 家族成员基因在低温条件下的qRT-PCR 分析发现,这11 个VvJAZs 基因都存在不同程度的受低温诱导表达,但只有VvJAZ9 基因的相对表达量随低温胁迫时间的延长而呈现上升趋势,是受低温诱导表达最为显著的基因。
蛋白之间的相互作用对阐明胞内信号转导有重要意义。JAZ可以与ICE[14]、MYC[41]、bHLH[42]类蛋白互作,在JA 信号通路中发挥作用,参与植物生长发育以及逆境响应。水稻(Oryza sativa)中OsJAZ9蛋白与OsMYB30相互作用负调控BMY基因以调节淀粉分解和麦芽糖积累,从而增强植物的抗寒性[43]。作为抑制子的MdJAZ1/4 可与MdMYC2 结合,促进抗寒相关基因的表达,提高苹果(Malus pumila)愈伤组织对低温的抵抗能力[44]。MdJAZ1 和MdJAZ2 通过与MdABI4 蛋白相互作用负调节MdABI4 基因以促进MdICE2 基因对其下游靶基因MdCBF1 的转录调节,从而提高苹果的耐寒性[45]。甜樱桃(Prunus avium)PavJAZ1/2/3与PavMYC2相互作用共同响应温度胁迫并协同调控开花[46]。本研究通过酵母双杂交试验筛选获得26个可能与VvJAZ9存在互作关系的候选蛋白,进一步运用qRT-PCR技术分析候选互作蛋白的相关基因在低温胁迫下的表达情况后发现,这些相关基因中有1 个HAP5A 基因的相对表达量随低温时间的延长而呈现出显著上升的趋势,这与从葡萄JAZ 家族成员基因中筛选出的VvJAZ9 受低温诱导表达模式相似,推测HAP5A可能是候选互作蛋白中响应低温较为明显的且与JAZ9 有互作关系的蛋白。
NF-Y(nuclear factor-Y)又称为HAP(heme activator protein),是广泛存在于真核生物中的转录因子,以NF-YA(HAP2/CBF-B)、NF-YB(HAP3/ CBFA)和NF-YC(HAP5/CBF-C)3 种亚基构成异源三聚体的形式调控下游基因的表达[47]。研究发现,HAPs 家族参与胚胎合成、种子萌发、开花调控、果实成熟以及响应逆境胁迫等生长发育过程[48]。HAP5 基因通过与GSH2(glutathione synthetase)的启动子结合,诱导GSH2 基因表达,提高细胞中谷胱甘肽含量,减轻活性氧对细胞膜的损伤,从而有效提高假丝酵母(Candida glycerinogenes)对2-苯乙醇的耐受性[49]。将青杄(Picea wilsonii)PwHAP5 基因在拟南芥中过表达可显著提高幼苗对盐分的耐受性[50];AtHAP5A 基因通过与AtXTH2 启动子上的CCAAT 顺式作用元件结合来正向调节拟南芥对冷胁迫的响应,过表达AtHAP5A基因也可增强拟南芥植株对冷冻的耐受性[51-52]。此外,PwHAP5 可以与PwFKBP12 发生互作,参与花粉管发育和花粉管定向控制[53];OsHAP5 和OsHAP2 相互作用,共同参与水稻光周期开花的调控[54]。本研究通过qRT-PCR分析获得1 个响应低温较为显著的JAZ9 候选互作蛋白HAP5A,进一步通过酵母双杂交和双分子荧光互补实验也表明VvJAZ9 与VvHAP5A 蛋白存在互作关系。
4 结 论
从葡萄基因组共鉴定到11 个VvJAZs 基因,编码氨基酸序列长度为98~388 aa,分子质量在11.26~40.31 ku,均为碱性亲水蛋白,分布于3个亚组中,其中VvJAZ9 与拟南芥AtJAZ1 和AtJAZ2 蛋白的亲缘关系最近。qRT-PCR 表明,VvJAZ9 和VvHAP5A 基因均响应低温诱导表达,推测该基因可能在葡萄抵御冷胁迫的应答过程中起重要作用。酵母双杂交和双分子荧光互补试验结果都表明,VvJAZ9 与Vv-HAP5A 蛋白具有互作关系。本研究为进一步开展葡萄JAZ 基因参与低温胁迫的研究奠定理论基础,为探讨葡萄抗逆分子机制研究提供参考。
[1] 陆昱颖,万然,樊秀彩,张颖,孙磊,刘崇怀,姜建福. 中国和UPOV、日本葡萄品种DUS 测试指南比较分析[J].果树学报,2023,40(2):363-375.LU Yuying,WAN Ran,FAN Xiucai,ZHANG Ying,SUN Lei,LIU Chonghuai,JIANG Jianfu. Comparative analysis of test guidelines for distinctness,uniformity and stability of grapevine(Vitis L.) formulated by China,UPOV and Japan[J]. Journal of Fruit Science,2023,40(2):363-375.
[2] 张利鹏,刘怀锋,辛海平.葡萄抗寒机制研究进展[J].果树学报,2023,40(2):350-362.ZHANG Lipeng,LIU Huaifeng,XIN Haiping. Research progress in cold tolerance mechanism of grape [J]. Journal of Fruit Science,2023,40(2):350-362.
[3] 刘砚璞,肖雪,赵建明,朱自果.中国野生燕山葡萄VyCIPK9 基因的克隆与功能分析[J].果树学报,2022,39(10):1748-1758.LIU Yanpu,XIAO Xue,ZHAO Jianming,ZHU Ziguo. Cloning and functional analysis of VyCIPK9 gene in Chinese wild grape(Vitis yeshanensis‘Yanshan’) [J]. Journal of Fruit Science,2022,39(10):1748-1758.
[4] 刘畅,杨兴旺,王小龙,王志强,刘凤之,王海波.植物防冻剂对葡萄抗寒能力的影响研究进展[J].中国果树,2022(3):6-9.LIU Chang,YANG Xingwang,WANG Xiaolong,WANG Zhiqiang,LIU Fengzhi,WANG Haibo. Research progress on the effect of plant antifreeze on grape under low-temperature stress[J].China Fruits,2022(3):6-9.
[5] FENNELL A. Freezing tolerance and injury in grapevines[J].Journal of Crop Improvement,2004,10(1/2):201-235.
[6] 段晓凤,张晓煜,张磊,刘建文,南学军,李楠,李红英.酿酒葡萄越冬防冻技术研究发展概况[J].农业工程,2022,12(6):133-138.DUAN Xiaofeng,ZHANG Xiaoyu,ZHANG Lei,LIU Jianwen,NAN Xuejun,LI Nan,LI Hongying. General situation of research and development on antifreeze technology for wine grape overwintering[J]. Agricultural Engineering,2022,12(6):133-138.
[7] ALI M S,BAEK K H. Jasmonic acid signaling pathway in response to abiotic stresses in plants[J]. International Journal of Molecular Sciences,2020,21(2):621.
[8] 李芳菲,马文瑶,程大伟,黄海娜,顾红,陈锦永,杨英军.植物生长调节物质对葡萄着色影响的研究进展[J]. 果树学报,2019,36(7):928-938.LI Fangfei,MA Wenyao,CHENG Dawei,HUANG Haina,GU Hong,CHEN Jinyong,YANG Yingjun.Advances in grape coloration regulated by plant growth regulators[J]. Journal of Fruit Science,2019,36(7):928-938.
[9] WASTERNACK C,HAUSE B. Jasmonates:biosynthesis,perception,signal transduction and action in plant stress response,growth and development:An update to the 2007 review in annals of botany[J].Annals of Botany,2013,111(6):1021-1058.
[10] GHORBEL M,BRINI F,SHARMA A,LANDI M. Role of jasmonic acid in plants:The molecular point of view[J]. Plant Cell Reports,2021,40(8):1471-1494.
[11] DING F,WANG C,XU N,WANG M L,ZHANG S X.Jasmonic acid- regulated putrescine biosynthesis attenuates cold- induced oxidative stress in tomato plants[J]. Scientia Horticulturae,2021,288:110373.
[12] 解振宇,杨江山.茉莉酸甲酯对高寒地区设施延后栽培葡萄生理的影响[J].中国农学通报,2016,32(16):66-71.XIE Zhenyu,YANG Jiangshan. Effect of methyl jasmonate on physiology of delayed culture grape in alpine greenhouse[J].Chinese Agricultural Science Bulletin,2016,32(16):66-71.
[13] 冯斗,禤维言,黄政树,邓付园,谭湘.茉莉酸甲酯对低温胁迫下香蕉幼苗的生理效应[J].果树学报,2009,26(3):390-393.FENG Dou,XUAN Weiyan,HUANG Zhengshu,DENG Fuyuan,TAN Xiang. Effects of MeJA on cold tolerance of banana seedlings[J].Journal of Fruit Science,2009,26(3):390-393.
[14] HU Y R,JIANG L Q,WANG F,YU D Q. Jasmonate regulates the inducer of cbf expression-c-repeat binding factor/dre binding factor1 cascade and freezing tolerance in Arabidopsis[J]. The Plant Cell,2013,25(8):2907-2924.
[15] 樊晓培,邢津溥,魏铁锁,李欣洋,苍晶,徐庆华,张达. 外源MeJA 对低温胁迫下冬小麦冷响应基因表达的影响[J].麦类作物学报,2020,40(3):292-299.FAN Xiaopei,XING Jinpu,WEI Tiesuo,LI Xinyang,CANG Jing,XU Qinghua,ZHANG Da. Effect of exogenous MeJA on the expression of cold response genes in winter wheat under low temperature stress[J]. Journal of Triticeae Crops,2020,40(3):292-299.
[16] THINES B,KATSIR L,MELOTTO M,NIU Y J,MANDAOKAR A,LIU G H,NOMURA K,HE S Y,HOWE G A,BROWSE J. JAZ repressor proteins are targets of the SCFCOI1 complex during jasmonate signalling[J]. Nature,2007,448(7154):661-665.
[17] THIREAULT C,SHYU C,YOSHIDA Y,ST AUBIN B,CAMPOS M L,HOWE G A.Repression of jasmonate signaling by a non-TIFY JAZ protein in Arabidopsis[J].The Plant Journal,2015,82(4):669-679.
[18] BROWSE J,WALLIS J G.Arabidopsis flowers unlocked the mechanism of jasmonate signaling[J]. Plants,2019,8(8):285.
[19] SHEARD L B,TAN X,MAO H B,WITHERS J,BEN-NISSAN G,HINDS T R,KOBAYASHI Y,HSU F F,SHARON M,BROWSE J,HE S Y,RIZO J,HOWE G A,ZHENG N.Jasmonate perception by inositol-phosphate-potentiated COI1-JAZ co-receptor[J].Nature,2010,468(7322):400-405.
[20] MELOTTO M,MECEY C,NIU Y J,CHUNG H S,KATSIR L,YAO J,ZENG W Q,THINES B,STASWICK P,BROWSE J,HOWE G A,HE S Y.A critical role of two positively charged amino acids in the Jas motif of Arabidopsis JAZ proteins in mediating coronatine- and jasmonoyl isoleucine-dependent interactions with the COI1 F-box protein[J].The Plant Journal,2008,55(6):979-988.
[21] ZHANG Y C,GAO M,SINGER S D,FEI Z J,WANG H,WANG X P. Genome-wide identification and analysis of the TIFY gene family in grape[J].PLoS One,2012,7(9):e44465.
[22] HANIF M,RAHMAN M U,GAO M,YANG J H,AHMAD B,YAN X X,WANG X P. Heterologous expression of the grapevine JAZ7 gene in Arabidopsis confers enhanced resistance to powdery mildew but not to Botrytis cinerea[J]. International Journal of Molecular Sciences,2018,19(12):3889.
[23] ZHANG G F,YAN X X,ZHANG S L,ZHU Y X,ZHANG X M,QIAO H B,VAN NOCKER S,LI Z,WANG X P.The jasmonate-ZIM domain gene VqJAZ4 from the Chinese wild grape Vitis quinquangularis improves resistance to powdery mildew in Arabidopsis thaliana[J]. Plant Physiology and Biochemistry,2019,143:329-339.
[24] 俞沁含,焦淑珍,吴楠,张宁波,徐伟荣. 葡萄E3 泛素酶HOS1 基因克隆、表达及抗血清制备[J].园艺学报,2021,48(6):1173-1182.YU Qinhan,JIAO Shuzhen,WU Nan,ZHANG Ningbo,XU Weirong. Molecular cloning,expression and polyclonal antibody preparation of E3 ubiquitin ligase gene HOS1 from Vitis vinifera[J].Acta Horticulturae Sinica,2021,48(6):1173-1182.
[25] 韩雪,王树芳,林金星.拟南芥酵母双杂交文库构建及ADPG1 互作蛋白筛选[J].中国细胞生物学学报,2022,44(6):1048-1055.HAN Xue,WANG Shufang,LIN Jinxing. Construction of yeast two-hybrid library of Arabidopsis thaliana and screening of ADPG1 interacting proteins[J]. Chinese Journal of Cell Biology,2022,44(6):1048-1055.
[26] 郑巧玲,申威,姚文孔,徐伟荣.山葡萄低温诱导酵母双杂三框cDNA 文库构建和VaCIPK18 互作蛋白筛选鉴定[J].园艺学报,2020,47(12):2301-2316.ZHENG Qiaoling,SHEN Wei,YAO Wenkong,XU Weirong.Construction of yeast two-hybrid three-frame cDNA library of Vitis amurensis and screening of VaCIPK18 interaction protein[J].Acta Horticulturae Sinica,2020,47(12):2301-2316.
[27] WASTERNACK C,STRNAD M. Jasmonates are signals in the biosynthesis of secondary metabolites:Pathways,transcription factors and applied aspects: A brief review[J]. New Biotechnology,2019,48:1-11.
[28] CHICO J M,CHINI A,FONSECA S,SOLANO R.JAZ repressors set the rhythm in jasmonate signaling[J]. Current Opinion in Plant Biology,2008,11(5):486-494.
[29] ZHANG H Y,LI W J,NIU D X,WANG Z J,YAN X X,YANG X L,YANG Y F,CUI H.Tobacco transcription repressors NtJAZ:potential involvement in abiotic stress response and glandular trichome induction[J]. Plant Physiology and Biochemistry,2019,141:388-397.
[30] XIE S F,CUI L C,LEI X L,YANG G,LI J,NIE X J,JI W Q.The TIFY gene family in wheat and its progenitors:genome-wide identification,evolution and expression analysis[J].Current Genomics,2019,20(5):371-388.
[31] SUN P D,SHI Y N,VALERIO A G O,BORREGO E J,LUO Q Y,QIN J,LIU K,YAN Y X. An updated census of the maize TIFY family[J].PLoS One,2021,16(2):e0247271.
[32] CHINI A,BEN-ROMDHANE W,HASSAIRI A,ABOUL-SOUD M A M.Identification of TIFY/JAZ family genes in Solanum lycopersicum and their regulation in response to abiotic stresses[J].PLoS One,2017,12(6):e0177381.
[33] PAN J W,TU J Y,SHARIF R,QI X H,XU X W,CHEN X H.Study of JASMONATE ZIM-Domain gene family to waterlogging stress in Cucumis sativus L.[J]. Vegetable Research,2021,1(1):1-12.
[34] YANG Y X,AHAMMED G J,WAN C P,LIU H J,CHEN R R,ZHOU Y. Comprehensive analysis of TIFY transcription factors and their expression profiles under jasmonic acid and abiotic stresses in watermelon[J]. International Journal of Genomics,2019,2019:6813086.
[35] 王玉昆,苑少华,苑国良,段文静,王鹏,廖祥政,陈兆波,王娜,王拯,张立平,赵昌平.粗山羊草JAZ 基因家族的全基因组鉴定与分析[J].麦类作物学报,2016,36(1):9-17.WANG Yukun,YUAN Shaohua,YUAN Guoliang,DUAN Wenjing,WANG Peng,LIAO Xiangzheng,CHEN Zhaobo,WANG Na,WANG Zheng,ZHANG Liping,ZHAO Changping. Genomewide identification and analysis of JAZ gene family in Aegilops tauschii[J].Journal of Triticeae Crops,2016,36(1):9-17.
[36] 黄小芳,毕楚韵,陈其俊,刘江洪,胡韵卓,黄碧芳,许明,陈选阳,林世强.甘薯基因组JAZ 基因家族的鉴定与分析[J].基因组学与应用生物学,2021,40(s4):3685-3693.HUANG Xiaofang,BI Chuyun,CHEN Qijun,LIU Jianghong,HU Yunzhuo,HUANG Bifang,XU Ming,CHEN Xuanyang,LIN Shiqiang. Identification and analysis of JAZ gene family in Ipomoea batatas genome[J].Genomics and Applied Biology,2021,40(S4):3685-3693.
[37] 刘凯,李津津,王红霞,李林晴,赵书岗,张俊佩,马庆国,张志华.核桃JrJAZ 基因家族鉴定与转录表达分析[J].河北农业大学学报,2021,44(1):41-50.LIU Kai,LI Jinjin,WANG Hongxia,LI Linqing,ZHAO Shugang,ZHANG Junpei,MA Qingguo,ZHANG Zhihua.Identification and transcriptional expression analysis of walnut Jr-JAZ gene family[J]. Journal of Hebei Agricultural University,2021,44(1):41-50.
[38] CAI Z M,CHEN Y Q,LIAO J J,WANG D D. Genome-wide identification and expression analysis of jasmonate ZIM domain gene family in tuber mustard (Brassica juncea var. tumida)[J].PLoS One,2020,15(6):e0234738.
[39] MAKHAZEN D S,VEREMEICHIK G N,SHKRYL Y N,TCHERNODED G K,GRIGORCHUK V P,BULGAKOV V P.Inhibition of the JAZ1 gene causes activation of camalexin biosynthesis in Arabidopsis callus cultures[J]. Journal of Biotechnology,2021,342:102-113.
[40] 蔡肖,甄军波,江振兴,刘琳琳,刘迪,张建宏,田海燕,张香云,迟吉娜.陆地棉低温响应基因GhJAZ1 的克隆及表达分析[J].华北农学报,2018,33(1):7-13.CAI Xiao,ZHEN Junbo,JIANG Zhenxing,LIU Linlin,LIU Di,ZHANG Jianhong,TIAN Haiyan,ZHANG Xiangyun,CHI Jina. Cloning and expression analysis of cold response gene Gh-JAZ1 from Gossypium hirsutum L.[J].Acta Agriculturae Boreali-Sinica,2018,33(1):7-13.
[41] GOOSSENS J,SWINNEN G,VANDEN BOSSCHE R,PAUWELS L,GOOSSENS A. Change of a conserved amino acid in the MYC2 and MYC3 transcription factors leads to release of JAZ repression and increased activity[J]. New Phytologist,2015,206(4):1229-1237.
[42] FONSECA S,FERNÁNDEZ-CALVO P,FERNÁNDEZ G M,DÍEZ-DÍAZ M,GIMENEZ-IBANEZ S,LÓPEZ-VIDRIERO I,GODOY M,FERNÁNDEZ-BARBERO G,VAN LEENE J,DE JAEGER G,FRANCO- ZORRILLA J M,SOLANO R.bHLH003,bHLH013 and bHLH017 are new targets of JAZ repressors negatively regulating JA responses[J]. PLoS One,2014,9(1):e86182.
[43] LV Y,YANG M,HU D,YANG Z Y,MA S Q,LI X H,XIONG L Z. The OsMYB30 transcription factor suppresses cold tolerance by interacting with a JAZ protein and suppressing β-amylase expression[J].Plant Physiology,2017,173(2):1475-1491.
[44] WANG Y C,XU H F,LIU W J,WANG N,QU C Z,JIANG S H,FANG H C,ZHANG Z Y,CHEN X S.Methyl jasmonate enhances apple’cold tolerance through the JAZ-MYC2 pathway[J].Plant Cell,Tissue and Organ Culture (PCTOC),2019,136(1):75-84.
[45] AN J P,XU R R,LIU X,SU L,YANG K,WANG X F,WANG G L,YOU C X.Abscisic acid insensitive 4 interacts with ICE1 and JAZ proteins to regulate ABA signaling-mediated cold tolerance in apple[J]. Journal of Experimental Botany,2022,73(3):980-997.
[46] 王继源,王丽,纠松涛,孙菀霞,徐岩,刘勋菊,张才喜.甜樱桃PavMYC2基因克隆与表达分析[J].果树学报,2022,39(5):701-711.WANG Jiyuan,WANG Li,JIU Songtao,SUN Wanxia,XU Yan,LIU Xunju,ZHANG Caixi. Cloning and expression analysis of PavMYC2 gene in Prunus avium L.[J]. Journal of Fruit Science,2022,39(5):701-711.
[47] 李世贵,马瑞,王芳芳,刘维刚,杨江伟,唐勋,张宁,司怀军.植物NF-Y 转录因子研究进展[J]. 植物生理学报,2021,57(2):248-256.LI Shigui,MA Rui,WANG Fangfang,LIU Weigang,YANG Jiangwei,TANG Xun,ZHANG Ning,SI Huaijun. Research progresses on plant NF-Y transcription factors[J].Plant Physiology Journal,2021,57(2):248-256.
[48] ZANETTI M E,RÍPODAS C,NIEBEL A.Plant NF-Y transcription factors:Key players in plant-microbe interactions,root development and adaptation to stress[J]. Biochimica et Biophysica Acta (BBA)-Gene Regulatory Mechanisms,2017,1860(5):645-654.
[49] WANG Y Q,ZHANG Z Y,LU X Y,ZONG H,ZHUGE B.Transcription factor Hap5 induces gsh2 expression to enhance 2-phenylethanol tolerance and production in an industrial yeast Candida glycerinogenes[J]. Applied Microbiology and Biotechnology,2020,104(9):4093-4107.
[50] LI L L,YU Y L,WEI J,HUANG G X,ZHANG D,LIU Y,ZHANG L Y. Homologous HAP5 subunit from Picea wilsonii improved tolerance to salt and decreased sensitivity to ABA in transformed Arabidopsis[J].Planta,2013,238(2):345-356.
[51] SHI H T,CHAN Z L.AtHAP5A modulates freezing stress resistance in Arabidopsis independent of the CBF pathway[J]. Plant Signaling&Behavior,2014,9(7):e29109.
[52] SHI H T,YE T T,ZHONG B,LIU X,JIN R,CHAN Z L. At-HAP5A modulates freezing stress resistance in Arabidopsis through binding to CCAAT motif of AtXTH21[J].New Phytologist,2014,203(2):554-567.
[53] YU Y L,LI Y Z,HUANG G X,MENG Z D,ZHANG D,WEI J,YAN K,ZHENG C C,ZHANG L Y. PwHAP5,a CCAAT-binding transcription factor,interacts with PwFKBP12 and plays a role in pollen tube growth orientation in Picea wilsonii[J]. Journal of Experimental Botany,2011,62(14):4805-4817.
[54] XING Q K,ZHENG Z M,ZHOU X G,CHEN X J,GUO Z J.Ds9 was isolated encoding as OsHAP3H and its C-terminus was required for interaction with HAP2 and HAP5[J]. Journal of Plant Biology,2015,58(1):26-37.