植物在生长发育过程中会受到高盐、干旱、低温和重金属离子毒害等环境变化引起的非生物胁迫,造成植物生长缓慢或死亡[1-2]。为了适应这些非生物胁迫,植物自身进化出多种保护系统来抵御逆境胁迫所造成的损伤[3]。14-3-3 作为应激诱导蛋白通过参与真核生物转录调控和信号转导来增强植物对逆境的耐受性,它通过激素信号途径和离子通道来调控多种靶标的活性,在非生物胁迫中起关键作用[4-6]。14-3-3蛋白也具有调节植物生长和发育的功能,其与钙调蛋白激酶CDPK(calcium-dependent protein kinase)共同作用调控植物新陈代谢、激素合成和开花等多种生物学途径[7-8]。
目前14-3-3 蛋白已在甜瓜(Cucumis melo)、拟南芥(Arabidopsis thaliana)、番茄(Solanum lycopersicum)、大豆(Glycine max)、苜蓿(Medicago truncatula)、杨树(Populus trichocarpa)、水稻(Oryza sativa)、花生(Arachis hypogaea)、棉花(Gossypium hirsutum)和 杧 果(Mangifera indica)中 完 成 了 鉴定[9-17]。研究发现,根据系统进化和内含子数量,14-3-3基因家族划分为ε类和非ε类,非ε类含有4个外显子和3个内含子,而ε类一般包含6~7个外显子和4~6个内含子[8,16-17]。14-3-3基因在小麦、葡萄和香蕉生殖器官中高表达来调节植物淀粉合成、果实发育和浆果成熟等过程[18-20]。研究发现,4个Md14-3-3蛋白与花整合因子MdTFL1和MdFT相互作用参与植物开花调控[21],而过表达毛竹PvGF14b、PvGF14c和PvGF14e能够延缓拟南芥的开花时间[22]。在番茄根系中,14-3-3 蛋白通过调控脱落酸(abscisic acid,ABA)信号通路来维持植物细胞渗透平衡和气孔开度,进而响应干旱或盐碱逆境胁迫[9]。前人研究发现,小麦TaGF14b 在烟草中过表达增强了植物对非生物胁迫的耐受性[23]。此外,Xia 等[15]通过qPCR 发现杧果植株在干旱处理12 h 后Mi14-3-3-A1 的表达呈现下降趋势。水稻OsCPK21 与OsGF14e 磷酸化作用来调控ABA 信号转导和耐盐性,而拟南芥14-3-3 蛋白质充当盐过度敏感(SOS)信号通路分子开关,响应盐胁迫[24-25]。棉花叶片中的5 个基因(Gh-GRF3、GhGRF4、GhGRF5、GhGRF7和GhGRF16)在盐胁迫下下调表达[26]。同时,过表达MdGRF11可提高转基因苹果愈伤组织和拟南芥对干旱和盐胁迫的耐受性[27]。以上研究表明14-3-3家族蛋白在植物生长、发育和胁迫反应中具有重要作用。
苹果(Malus×domestica Borkh.)是中国广泛种植的经济水果之一,而干旱和盐碱等非生物胁迫仍是限制我国苹果产业发展的重要因素。笔者在本研究中利用拟南芥和水稻蛋白序列在苹果基因组中进行同源比对,鉴定出36 个苹果14-3-3 基因家族成员,对其理化性质、系统进化关系及保守基序进行分析,并探究该家族基因在植物组织和逆境胁迫下的表达情况,以及分析过表达MdGRF13 愈伤组织在PEG和NaCl胁迫下的影响,为该家族基因响应逆境功能研究提供理论依据。
1 材料和方法
1.1 材料与处理
试验于2021 年在甘肃农业大学园艺学院果树生理与生物技术实验室进行。选择继代培养30 d生长良好且一致的苹果品种嘎拉实生苗外植体扩繁的试管苗,转接到含有15% PEG、100 μmol·L-1 ABA 和200 mmol·L-1 NaCl[28]的MS 液体培养基(降低浓度误差)进行处理,以等体积MS 培养基处理为对照,对照与各处理同步时长均为2、12 和24 h,pH 值为5.8~6.0,采用纸桥法进行固定。每组处理设置3 个生物学重复,每个重复5 株试管苗。使用qRT-PCR 对10 年生嘎拉实生苗(甘肃农业大学日光温室)进行组织特异性数据分析。另外,使用本实验保存的苹果王林愈伤组织进行MdGRF13 的过表达分析。
1.2 苹果14-3-3家族基因的鉴定
用拟南芥(https://www.arabidopsis.org/)和水稻(http://rice.plantbiology.msu.edu/index.sh-tml)14-3-3家族的蛋白序列在苹果数据库GDR(https://www.rosaceae.org/)中利用Blast 工具进行比对,然后用SMART(http://smart.emblheidelberg.de/)和 Pfam(http://pfam.sanger.ac.uk/search)在线网站进行保守结构域的检索,去除不含保守结构域的苹果序列,根据苹果14-3-3 家族基因在染色上的位置进行命名。
1.3 苹果14-3-3 家族蛋白理化性质及二级结构分析
对苹果14-3-3 家族蛋白的理论等电点(pI)、分子质量和不稳定性指数等基本信息使用ExPASy(https://web.expasy.org/protparam/)工具进行分析。根据二级结构在线工具SOPMA(https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_sopma.html)对苹果14-3-3家族蛋白进行α螺旋、β转角、延伸链和无规则卷曲比例的预测。
1.4 苹果14-3-3家族蛋白系统进化分析
下载大豆、番茄和二穗短炳草(NCBI)14-3-3家族蛋白序列,与拟南芥、水稻以及苹果蛋白序列用MEGA 7.0软件比对并构建系统进化树,校验参数默认值为1000。
1.5 苹果14-3-3家族蛋白结构和保守基序分析
采用GSDS(http://gsds.cbi.pku.edu.cn/)对苹果14-3-3 家族基因成员进行基因结构分析;利用在线网站MEME(http://meme-suite.org/)对其蛋白保守基序预测。
1.6 苹果14-3-3 家族基因共线性和蛋白互作网络分析
利用软件TBtools 1.6对苹果14-3-3家族基因进行共线性分析,绘制关系图。通过Phytozome v12.1:Home(https://phytozome.jgi.doe.gov/pz/portal.html)下载拟南芥和水稻的CDS序列及基因注释文件,在GDR(https://www.rosaceae.org/)中下载苹果的基因组数据,使用TBtools 1.6软件绘制苹果、水稻和拟南芥之间的关联性图。对36 个苹果14-3-3 家族蛋白通过String 蛋白互作数据库(http://string-db.org/)进行蛋白互作网络分析和参数默认。
1.7 实时荧光定量qRT-PCR分析
36 个苹果14-3-3 家族基因qRT-PCR 引物(表1),由生工生物工程(上海)股份有限公司在线设计,提取RNA,用反转录试剂盒(Prime Script RT reagent Kit,Perfect Real Time,TaKaRa)合成cDNA;用实时荧光定量PCR 仪(LightCycler® 96 Real-Time PCR System,Roche,瑞士)和试剂盒(SYBR GreenⅠ,TaKaRa)进行该家族基因在不同组织、PEG、ABA及NaCl 处理下表达量的分析。反应体系为20 μL,分别加入6 μL ddH2O、2 μL cDNA、上下游引物各1 μL和10 μL SYBR。用SPSS 22.0 和Excel 2010 进行数据处理及作图。
表1 苹果14-3-3 家族基因qRT-PCR 引物
Table 1 qRT-PCR primers of 14-3-3 gene family in apple

基因名称Gene name MdGRF1 MdGRF2 MdGRF3 MdGRF4 MdGRF5 MdGRF6 MdGRF7 MdGRF8 MdGRF9 MdGRF10 MdGRF11 MdGRF12 MdGRF13 MdGRF14 MdGRF15 MdGRF16 MdGRF17 MdGRF18 MdGRF19 MdGRF20 MdGRF21 MdGRF22 MdGRF23 MdGRF24 MdGRF25 MdGRF26 MdGRF27 MdGRF28 MdGRF29 MdGRF30 MdGRF31 MdGRF32 MdGRF33 MdGRF34 MdGRF35 MdGRF36 MdGAPDH上游引物Forward primer 5’-GGAGGAGGCAAAGGGAAGTGAAC-3’5’-CACAACCTGTTGACGGTGGAGAG-3’5’-CAGGCAGAGCGATACGATGAGATG-3’5’-CCAAGCCCCAGCAGTAATCGTATC-3’5’-TGTCCGTTGGGGTCTCACTACTG-3’5’-CCGAGCAAGCCGAGCGTTAC-3’5’-CCGAGCAAGCCGAGCGTTAC-3’5’-TTGACTCCAGGCTCATCCCATCC-3’5’-CGACGGCATTCTCAAGCTCCTC-3’5’-CGACGGCATTCTCAAGCTCCTC-3’5’-CCAACTCAGCAACTCGGACCTTC-3’5’-GCATTGTCCAGCAGCATGTAAACC-3’5’-GCGTTACGAGGAGATGGTGAGTTC-3’5’-TGCCCGCTGGATTCTTCTTCAAAG-3’5’-GCTCGTCTTCCAACACCCATTCC-3’5’-GCGTCGGCCAGTAAGTACAAGG-3’5’-TTGAGCAGAAGGAGGAGAGCAGAG-3’5’-GCGTCATGGCGGATACTGTCTTC-3’5’-CCGAGCAAGCCGAGCGTTAC-3’5’-CGCTCGTCAAGCAGTACAGATCC-3’5’-CCTGACCCTGTGGACCTCTGAC-3’5’-GAGGAGCCCACCGTCGAAGAG-3’5’-GATGAAGGGCACTACCACCGTTAC-3’5’-ACAACCTCACTCTCTGGACCTCTG-3’5’-ACAACCTCACTCTCTGGACCTCTG-3’5’-GGCGTTCGTCAGATCCAAGGTC-3’5’-GGAGTTCAAGTTCGCCGATGAGAG-3’5’-CACAACCTGTTGACGGTGGAGAG-3’5’-GCACCTTGTTCCTTCGGCTACC-3’5’-TTGAGCAGAAGGAGGAGAGCAGAG-3’5’-TTGAGCAGAAGGAGGAGAGCAGAG-3’5’-CAGGCAGAGCGATACGATGAGATG-3’5’-CAGGCAGAGCGATACGATGAGATG-3’5’-GGAGGAGGCAAAGGGAAGTGAAC-3’5’-AGCCGAGCGATACGAGGAGATG-3’5’-ACAACCTCACTCTCTGGACCTCTG-3’5’-GAGCTCGCAGGTATCCTTTCT-3’下游引物Reverse primer 5’-TGTGGACGCTGATGGGAGGAG-3’5’-GAGATTATGCCCAGAGTGCGTAGC-3’5’-CGACGAGCACCAACAACATTCTTG-3’5’-GGAGAGAGGAGGAAGAGGTGAGTG-3’5’-CATCGTATCGCTCTGCCTGCTC-3’5’-TGTACCCAACCGAAAGCAGATTCC-3’5’-TGTACCCAACCGAAAGCAGATTCC-3’5’-TCGGCAAGATAACGGTGGTAATCG-3’5’-TCGGCAAGATAACGGTGGTAATCG-3’5’-TCGGCAAGATAACGGTGGTAATCG-3’5’-CAGCGGATGAGGGTGTGAAAGTC-3’5’-CGACGGCTTTGTTGAAGAGAGGAG-3’5’-TTCTTGTAGGCGACGGAGAGGAG-3’5’-ACTACCTCGTCGTCAACCTCGTC-3’5’-TGAACTGGACCATCTCCTCGTAGC-3’5’-AGTGGAAGAGCAGCAGGTAGGC-3’5’-CGGCAGATGAGGCAGATGGAATG-3’5’-GCAAGCTCGTCTTCAACCCTCTG-3’5’-GTACCCGACCGAAAGCAGATTCC-3’5’-GGTGGTCGCCGAAGGAACAAG-3’5’-TTTGGTTCTTCGGTGGGCTTGG-3’5’-GGAGGAGACGATACGCCAGGAG-3’5’-CTATGTCCTGGGCAGCCTTGTAAG-3’5’-ATTCGGCGGCAGTTGGTTCTTC-3’5’-CAGATTCGGCGGCAGGCTTG-3’5’-CGGTGGTAGTTGCCCTTCATCTTC-3’5’-ATGAGTTGGTGACAGATCCGCAAG-3’5’-GAGATTATGCCCAGAGTGCGTAGC-3’5’-TCGGCGGCGGTCTTTCTCTC-3’-3’5’-GCGGAAGAGGCAGACGGAATG-3’5’-GCGGAAGAGGCAGACGGAATG-3’5’-CGACGAGCACCAACAACATTCTTG-3’5’-CGACGAGCACCAACAACATTCTTG-3’5’-TGTGGACGCTGATGGGAGGAG-3’5’-TTCTTGTAGGCGACGGAGAGGAG-3’5’-ATTCGGCGGCAGTTGGTTCTTC-3’5’-TACCAAGCAATGACCTTGACC-3’
1.8 MdGRF13基因的克隆及亚细胞定位分析
RNA 来自嘎拉苹果试管苗叶片。苹果MdGRF13 基 因 克 隆 引 物 为F:5’-GAGCTCGGTACCCGGGGATCCATGGCGGCAACCACCCCC-3’,R:5’-GGTGTCGACTCTAGAGGATCCGGTCTCGATCTTGGATTGGTAGTC-3’。按照反应程序95 ℃预变性5 min;95 ℃变性30 s,58 ℃退火42 s,72 ℃延伸1 min,40 个循环;72 ℃延伸10 min 进行PCR扩增,使用1%的琼脂糖凝胶进行电泳检测并回收目标条带。连接pCAMBIA1300 载体,转化大肠杆菌DH5α(天根生物科技有限公司),进行菌液PCR鉴定后,送生工生物工程(西安)股份有限公司测序。
将pCAMBIA1300-MdGRF13-GFP 表达载体质粒和pCAMBIA1300-GFP 空载体质粒,分别转入农杆菌GV3101(北京博迈徳基因技术有限公司)。参照刘勇等[29]农杆菌介导烟草叶片的侵染方法,将重悬菌液注射到本氏烟草叶片背面,培养箱培养2 d后,采用激光共聚焦显微镜观察。
1.9 MdGRF13 愈伤组织转化、表型观察及生理指标测定
愈伤组织侵染参照张婷婷等[30]的方法。选取生长状态良好的苹果王林愈伤组织进行农杆菌侵染,并在诱导培养基(含有潮霉素50 mg·L-1和头孢霉素300 mg·L-1)上筛选。使用超光速mix(MF848)试剂盒(北京聚合美生物科技有限公司)鉴定转基因愈伤组织,鉴定所用抗性基因Hyg 引物为,F:5’-ATGAAAAAGCCTGAACTCACCG-3’,R:5’-CTATTTCTTTGCCCTCGGACGA-3’;继代培养。
取野生型和MdGRF13 过表达系OE-4、OE-2 及OE-3 愈伤组织0.05 g,放在分别含有2% PEG、4%PEG、0.06 mol·L-1 NaCl 和0.08 mol·L-1 NaCl 的MS固体培养基[27,31]上处理15 d,拍照并进行鲜质量统计。苹果愈伤组织中丙二醛(MDA)含量及SOD、POD 和CAT 活性分别采用硫代巴比妥酸法[32]、氮蓝四唑(NBT)法、愈创木酚法和紫外吸收法[33]测定。
2 结果与分析
2.1 苹果14-3-3家族基因成员的鉴定
从苹果基因组中共鉴定得到36 个14-3-3 家族基因(表2)。苹果14-3-3家族基因相对分子质量为11 221.52~113 854.16 Da;氨 基 酸 长 度 介 于98~1006 aa 之间;等电点(pI)为4.68~10.55;不稳定系数MdGRF17最小,为36.92,MdGRF8最大,为98.48;大多数苹果14-3-3家族蛋白的pI值小于7,其可能是酸性蛋白。对苹果14-3-3家族36个基因编码蛋白进行亚细胞定位预测,发现其主要在细胞质中进行相关表达,其次是细胞核,其中MdGRF2、MdGRF7、MdGRF8、MdGRF12、MdGRF16、MdGRF28 和MdGRF31 完全定位在细胞核中,预测这7 个蛋白仅在细胞核中发挥相关作用。MdGRF4 和MdGRF11 在叶绿体中有一定量的表达,预测这2 个蛋白可能与植物光合作用相关。
表2 苹果14-3-3 家族基因理化性质分析
Table 2 Physical and chemical properties of 14-3-3 family genes in apple
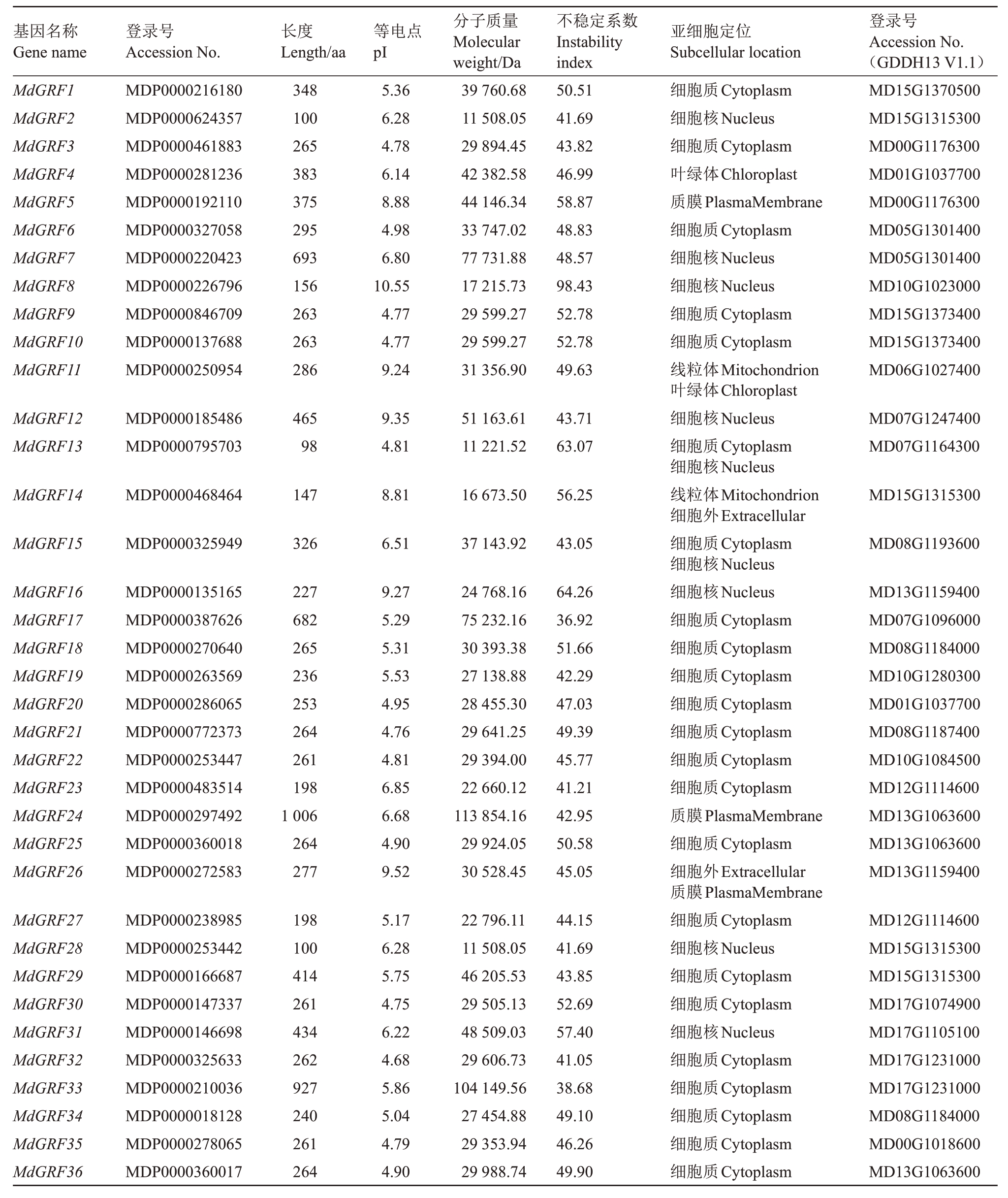
基因名称Gene name MdGRF1 MdGRF2 MdGRF3 MdGRF4 MdGRF5 MdGRF6 MdGRF7 MdGRF8 MdGRF9 MdGRF10 MdGRF11登录号Accession No.MDP0000216180 MDP0000624357 MDP0000461883 MDP0000281236 MDP0000192110 MDP0000327058 MDP0000220423 MDP0000226796 MDP0000846709 MDP0000137688 MDP0000250954长度Length/aa 348 100 265 383 375 295 693 156 263 263 286等电点pI 5.36 6.28 4.78 6.14 8.88 4.98 6.80 10.55 4.77 4.77 9.24分子质量Molecular weight/Da 39 760.68 11 508.05 29 894.45 42 382.58 44 146.34 33 747.02 77 731.88 17 215.73 29 599.27 29 599.27 31 356.90不稳定系数Instability index 50.51 41.69 43.82 46.99 58.87 48.83 48.57 98.43 52.78 52.78 49.63登录号Accession No.(GDDH13 V1.1)MD15G1370500 MD15G1315300 MD00G1176300 MD01G1037700 MD00G1176300 MD05G1301400 MD05G1301400 MD10G1023000 MD15G1373400 MD15G1373400 MD06G1027400 MdGRF12 MdGRF13 MDP0000185486 MDP0000795703 465 98 9.35 4.81 51 163.61 11 221.52 43.71 63.07 MD07G1247400 MD07G1164300 MdGRF14 MDP0000468464 147 8.81 16 673.50 56.25 MD15G1315300 MdGRF15 MDP0000325949 326 6.51 37 143.92 43.05 MD08G1193600 MdGRF16 MdGRF17 MdGRF18 MdGRF19 MdGRF20 MdGRF21 MdGRF22 MdGRF23 MdGRF24 MdGRF25 MdGRF26 MDP0000135165 MDP0000387626 MDP0000270640 MDP0000263569 MDP0000286065 MDP0000772373 MDP0000253447 MDP0000483514 MDP0000297492 MDP0000360018 MDP0000272583 227 682 265 236 253 264 261 198 1 006 264 277 9.27 5.29 5.31 5.53 4.95 4.76 4.81 6.85 6.68 4.90 9.52 24 768.16 75 232.16 30 393.38 27 138.88 28 455.30 29 641.25 29 394.00 22 660.12 113 854.16 29 924.05 30 528.45 64.26 36.92 51.66 42.29 47.03 49.39 45.77 41.21 42.95 50.58 45.05 MD13G1159400 MD07G1096000 MD08G1184000 MD10G1280300 MD01G1037700 MD08G1187400 MD10G1084500 MD12G1114600 MD13G1063600 MD13G1063600 MD13G1159400 MdGRF27 MdGRF28 MdGRF29 MdGRF30 MdGRF31 MdGRF32 MdGRF33 MdGRF34 MdGRF35 MdGRF36 MDP0000238985 MDP0000253442 MDP0000166687 MDP0000147337 MDP0000146698 MDP0000325633 MDP0000210036 MDP0000018128 MDP0000278065 MDP0000360017 198 100 414 261 434 262 927 240 261 264 5.17 6.28 5.75 4.75 6.22 4.68 5.86 5.04 4.79 4.90 22 796.11 11 508.05 46 205.53 29 505.13 48 509.03 29 606.73 104 149.56 27 454.88 29 353.94 29 988.74 44.15 41.69 43.85 52.69 57.40 41.05 38.68 49.10 46.26 49.90亚细胞定位Subcellular location细胞质Cytoplasm细胞核Nucleus细胞质Cytoplasm叶绿体Chloroplast质膜PlasmaMembrane细胞质Cytoplasm细胞核Nucleus细胞核Nucleus细胞质Cytoplasm细胞质Cytoplasm线粒体Mitochondrion叶绿体Chloroplast细胞核Nucleus细胞质Cytoplasm细胞核Nucleus线粒体Mitochondrion细胞外Extracellular细胞质Cytoplasm细胞核Nucleus细胞核Nucleus细胞质Cytoplasm细胞质Cytoplasm细胞质Cytoplasm细胞质Cytoplasm细胞质Cytoplasm细胞质Cytoplasm细胞质Cytoplasm质膜PlasmaMembrane细胞质Cytoplasm细胞外Extracellular质膜PlasmaMembrane细胞质Cytoplasm细胞核Nucleus细胞质Cytoplasm细胞质Cytoplasm细胞核Nucleus细胞质Cytoplasm细胞质Cytoplasm细胞质Cytoplasm细胞质Cytoplasm细胞质Cytoplasm MD12G1114600 MD15G1315300 MD15G1315300 MD17G1074900 MD17G1105100 MD17G1231000 MD17G1231000 MD08G1184000 MD00G1018600 MD13G1063600
2.2 苹果14-3-3家族蛋白二级结构分析
对苹果36 个14-3-3 家族蛋白进行二级结构预测结果表明(图1)。其蛋白二级结构以α 螺旋和无规则卷曲为主,分别占23.08%(MdGRF8)~78.57%(MdGRF13)、15.47%(MdGRF18)~64.10%(MdGRF8)。而β 转角占0%(MdGRF13)~7.93%(MdGRF16),延伸链占2.04%(MdGRF13)~22.65%(MdGRF33),二者所占比例较小。
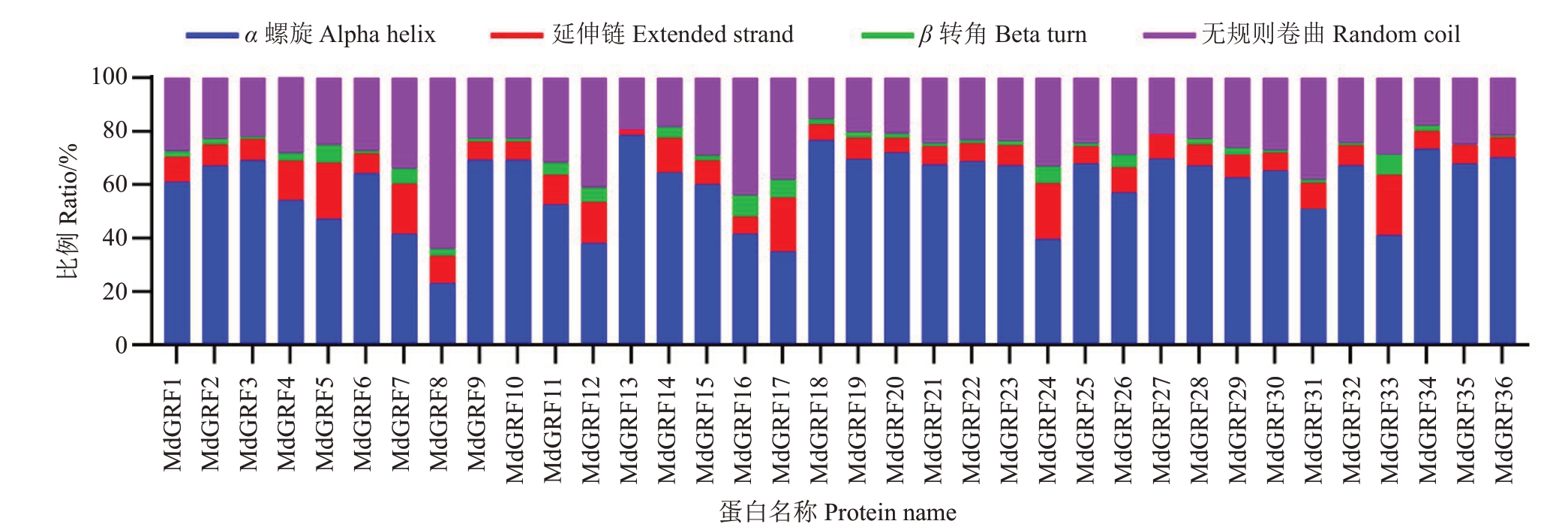
图1 苹果14-3-3 蛋白二级结构分析
Fig.1 The secondary structure analysis of 14-3-3 protein sequence in apple
2.3 苹果14-3-3家族基因进化分析
将苹果、拟南芥、水稻、大豆、二穗短柄草以及番茄的14-3-3 家族蛋白序列构建系统进化树(图2)。结果表明,该基因家族可分为ε类和非ε类,又分为5个亚族。其中,Ⅰ亚族有6个苹果14-3-3家族基因,Ⅱ亚族有10 个苹果14-3-3 家族基因,Ⅲ亚族有3 个苹果14-3-3家族基因,Ⅳ亚族有10个苹果14-3-3家族基因,Ⅴ亚族有7 个苹果14-3-3 家族基因。苹果跟拟南芥14-3-3 家族基因有较高的同源性,进化关系较近。
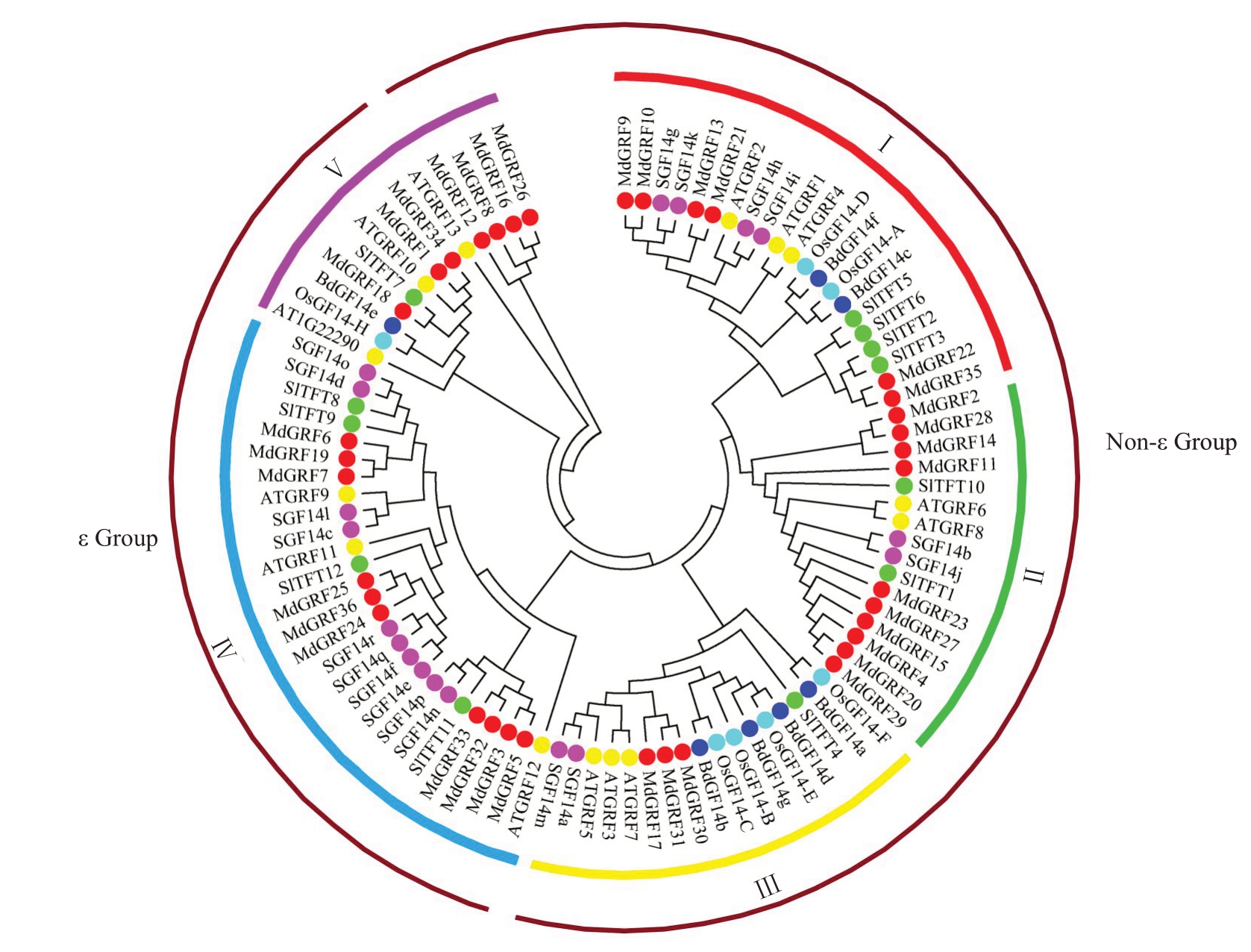
图2 拟南芥(At)、水稻(Os)、大豆(S)、二穗短柄草(Bd)、番茄(Sl)与苹果(Md)14-3-3 蛋白系统进化树
Fig.2 Phylogenetic analysis of 14-3-3 proteins in Arabidopsis thaliana(At),Oryza sativa(Os),Soyabean(S),Brachypodium distachyon(Bd),Solanum lycopersicum(Sl)and Malus domestica(Md)
2.4 苹果14-3-3家族基因结构和motif分析
对36 个苹果14-3-3 基因家族构建进化树以及基因结构分析(图3-A~B),该基因家族可分为ε 类和 非ε 类。 MdGRF11、MdGRF14、MdGRF23 和MdGRF27 仅有一个内含子,MdGRF16 和MdGRF26有2 个内含子,MdGRF7 和MdGRF24 有14 个内含子。MdGRF24 和MdGRF33 明显长于其他基因,序列长度超过8 kb。MdGRF2、MdGRF8、MdGRF13和MdGRF28 属于内含子缺失类,整条序列均为外显子,不同亚族序列外显子分布位置和长度差异较大,推测不同亚族有较大功能差异。对苹果14-3-3家族成员的motif分析显示(图3-C),共鉴定出10个motif,且 都 含 有motif 3。 MdGRF8、MdGRF12 和MdGRF14 蛋白只有一个保守基序,分别为motif 3、motif 2和motif 1。同一亚族成员结构相似,具有较高的保守性,其在苹果生长中可能发挥相似的功能;不同亚族间的motif存在差异,表明其可能具有不同的生物学功能。
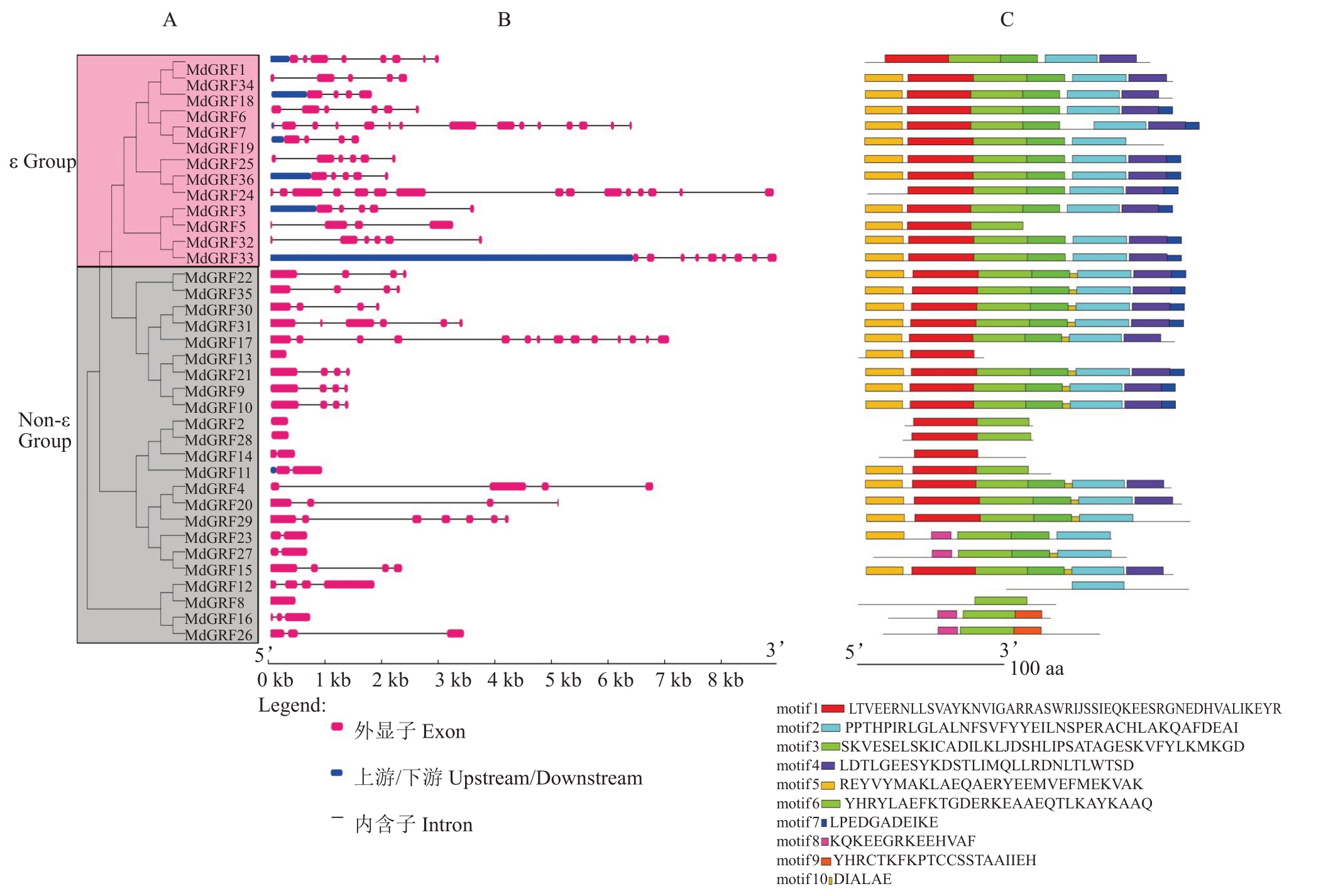
图3 苹果14-3-3 家族基因蛋白系统发育树(A)、基因结构(B)和蛋白保守基序(C)分析
Fig.3 Analysis of phylogenetic tree(A),gene structure(B)and protein conserved motifs(C)of the 14-3-3 family genes in apple
2.5 苹果14-3-3 家族基因的共线性和蛋白互作网络分析
对36 个苹果14-3-3 家族蛋白互作网络分析表明(图4-A)。15 个成员(MdGRF3、MdGRF4、MdGRF6 、MdGRF10 、MdGRF11 、MdGRF13 、MdGRF15 、MdGRF18 、MdGRF22 、MdGRF25 、MdGRF26、MdGRF31、MdGRF32、MdGRF34 和MdGRF36)在 网 络 中 与FT2、MdFT1 和XP—008366662.1蛋白互作,推测苹果14-3-3家族蛋白与植物营养生长、生殖生长和开花调控相关。
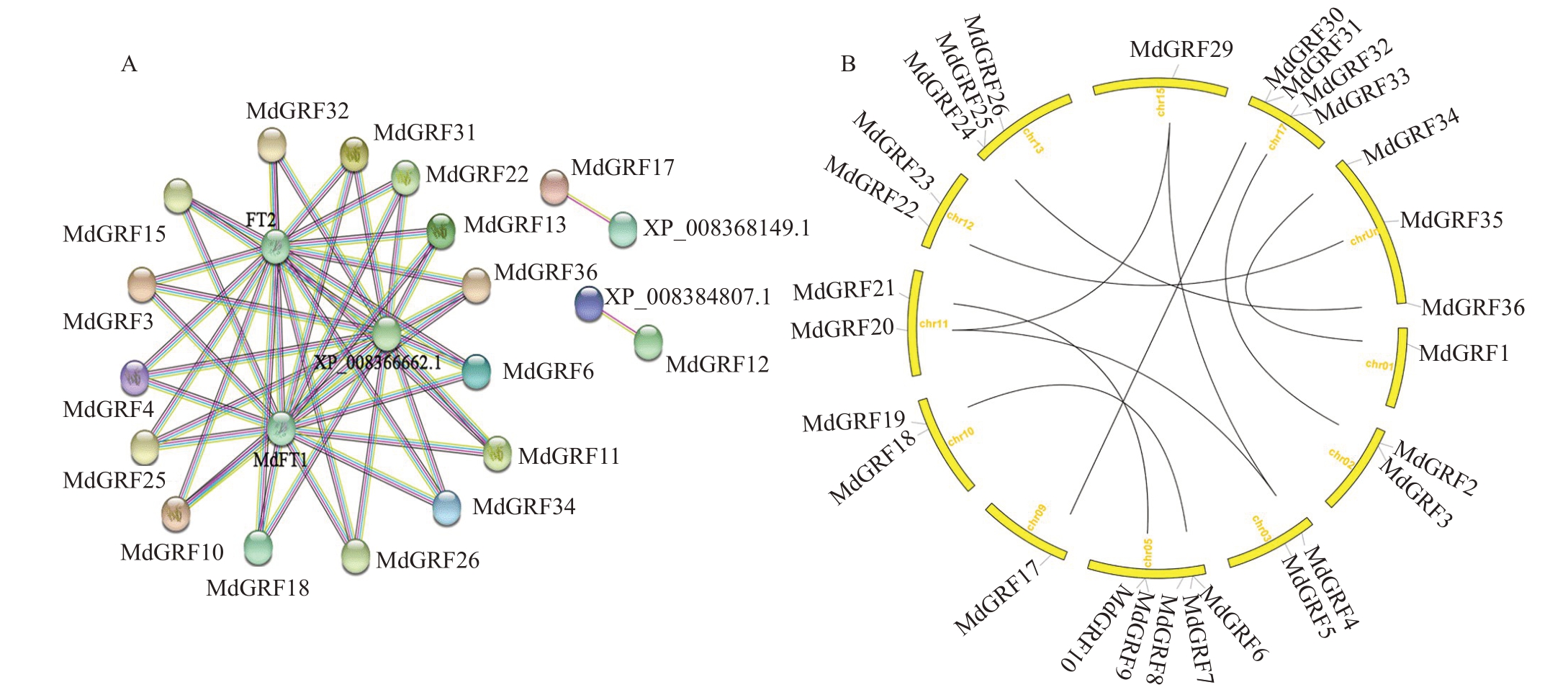
图4 苹果14-3-3 家族成员之间蛋白功能联系网络(A)和共线性(B)
Fig.4 Protein functional network(A)and synteny(B)between members of the apple 14-3-3 family
染色体定位显示,33 个苹果14-3-3 家族基因分布在11 条苹果染色体上,3 个苹果14-3-3 家族基因分布在chrUr 染色体上,而染色体Chr 05 上分布最多,有5个苹果14-3-3家族基因(图4-B)。对苹果的36个14-3-3家族基因进行共线性分析,结果如图4-B 所示:10 对基因存在共线性,分别为MdGRF1/MdGRF34、MdGRF2/MdGRF32、MdGRF4/MdGRF 20、MdGRF4/MdGRF29、MdGRF7/MdGRF18、MdGR F9/MdGRF21、MdGRF17/MdGRF30、MdGRF20/Md GRF29、MdGRF22/MdGRF35 和MdGRF24/MdGRF 36,原因可能与大片段复制有关。存在MdGRF2/MdGRF3、MdGRF6/MdGRF7、MdGRF9/MdGRF10、MdGRF24/MdGRF25、MdGRF30/MdGRF31 和MdG RF32/MdGRF33 6 对串联重复基因,片段重复在苹果14-3-3 基因家族扩展中占主要作用,该家族基因具有较高的同源性。
为了探讨不同物种14-3-3 家族基因的进化关系,分析了苹果、单子叶植物水稻以及模式植物拟南芥之间的同源基因,结果表明(图5),苹果与拟南芥之间的关联性较强,存在20 对共线性基因对,苹果与水稻之间的关联性相对较弱,存在3 对基因对。表明苹果与拟南芥的14-3-3 家族基因亲缘关系较近。
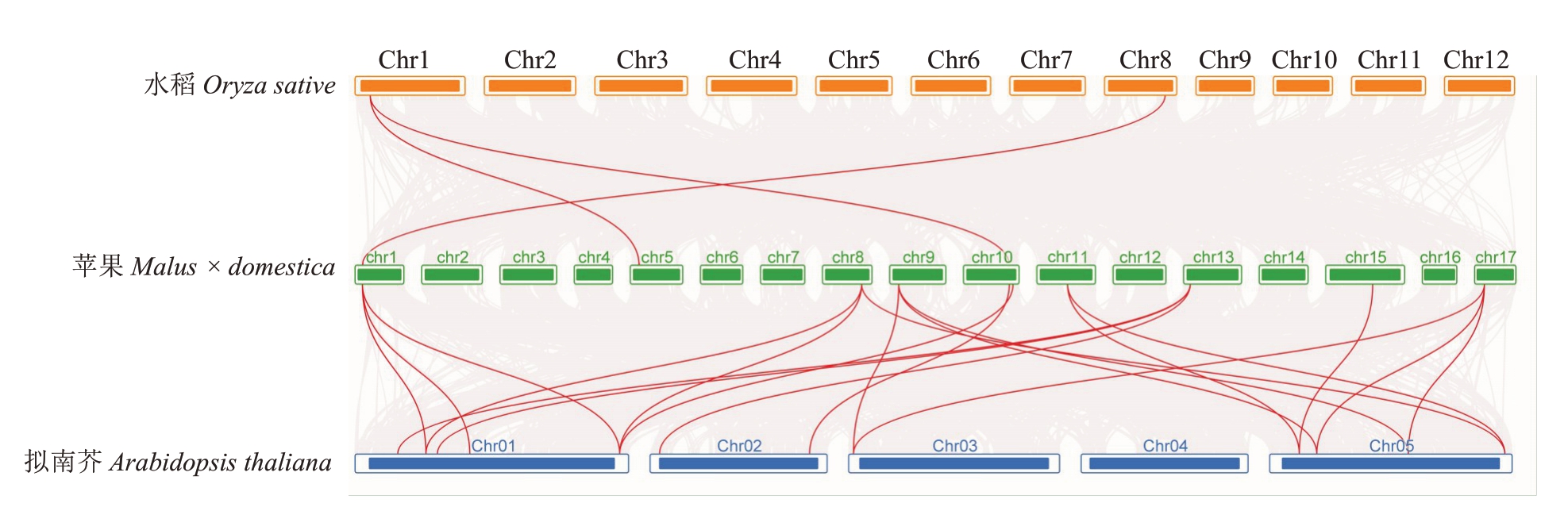
图5 拟南芥(At)、水稻(Os)与苹果(Md)的14-3-3 基因共线性分析
Fig.5 Synteny analysis of 14-3-3 genes in Arabidopsis thaliana(At),Oryza sativa(Os)and Malus domestica(Md)
2.6 苹果14-3-3家族基因的组织特异性表达分析
对苹果14-3-3 家族基因在根、茎和叶中的表达差异分析显示(图6),36个基因在根、茎和叶中都有表达。4 个基因(MdGRF2、MdGRF17、MdGRF25 和MdGRF28)在根中有较高表达,16和12个基因分别在茎和叶中表达量较高。综上所述,苹果14-3-3 家族基因在不同组织中存在表达差异,且在茎中表达量高于叶和根。
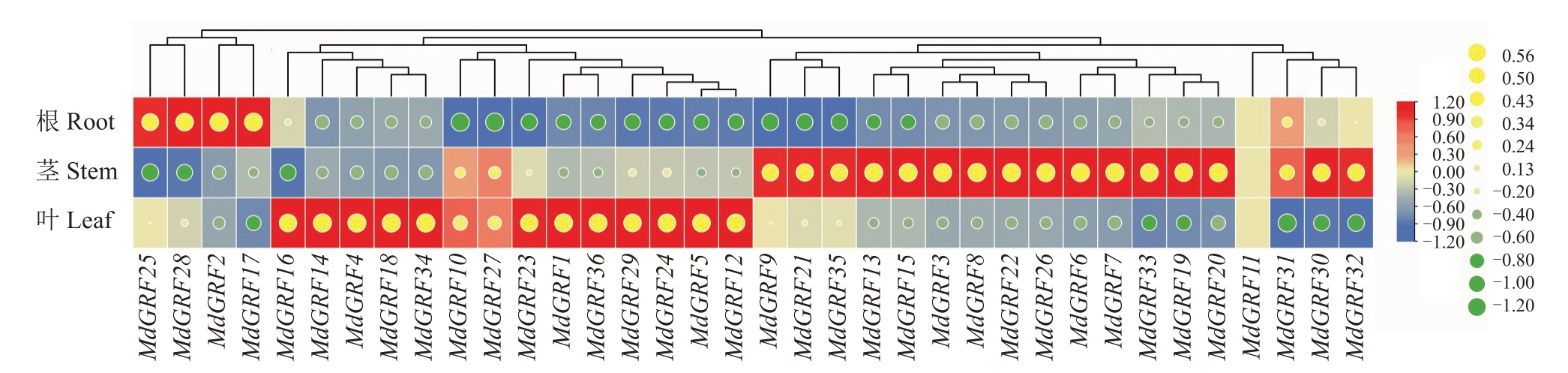
图6 苹果14-3-3 家族基因不同组织中表达量
Fig.6 Heat map of expression levels in different tissues of 14-3-3 family genes from apple
2.7 苹果14-3-3 家族基因对非生物胁迫的表达分析
通过qRT-PCR 分析了36 个苹果14-3-3 家族基因在PEG、ABA 和NaCl 处理下的相对表达量(图7)。结果表明,ABA 处 理2 h 后,MdGRF18 和MdGRF26 较对照显著上调表达,MdGRF4 和MdGRF16 也有上表达,MdGRF24 在ABA 处 理24 h后上调表达,其余基因均下调表达。MdGRF5、MdGRF14、MdGRF24 和MdGRF26 在PEG 处理2 h下与对照相比呈显著上调表达,MdGRF2、MdGRF13和MdGRF26 在12 h 有较高表达,且MdGRF13 在24 h 表达量最高,为对照的1.8 倍,而其余基因表达量较低。NaCl处理下,MdGRF4、MdGRF5、MdGRF12、MdGRF14、MdGRF16、MdGRF19和MdGRF24在2 h较对照上调表达,MdGRF1、MdGRF9、MdGRF10、MdGRF13、MdGRF15 和MdGRF24 在12 h 显 著 上调;MdGRF2 始终处于上调表达,且在2 h 表达量最高,为对照的18.6倍,其可能在盐胁迫中起正调控作用。MdGRF4、MdGRF5、MdGRF14 和MdGRF16 在NaCl处理2 h后表达量分别为对照的7.6、7.5、6.5和15.6 倍,而MdGRF13 在NaCl 处理12 h 及24 h 后显著上调表达,为对照的2.1 倍,推测苹果14-3-3 家族基因可能响应盐胁迫。

图7 (续) Fig.7 (Continued)
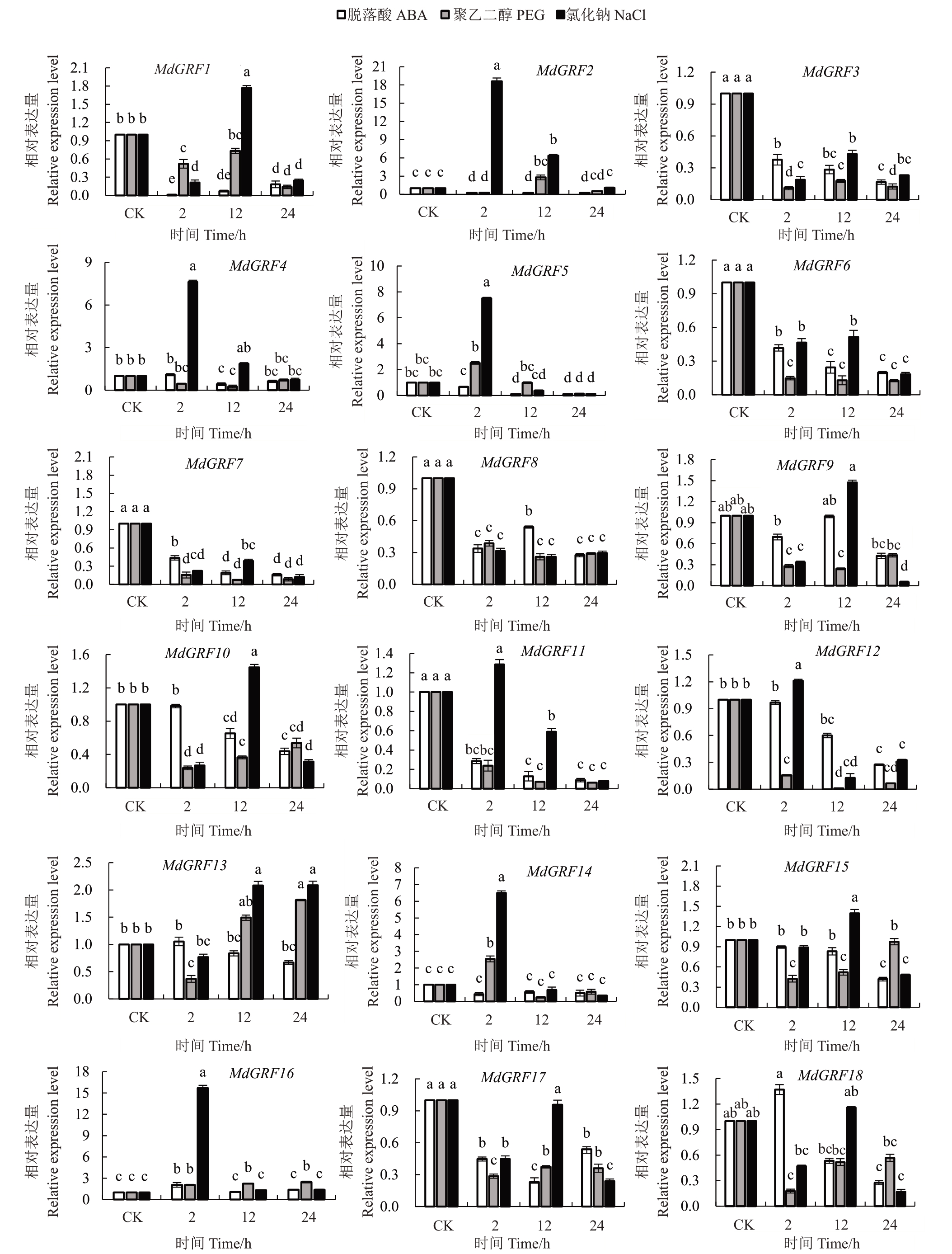
图7 不同处理下苹果14-3-3 家族基因表达分析
Fig.7 Expression analysis of 14-3-3 family genes under different treatments in apple
2.8 苹果MdGRF13基因克隆和亚细胞定位分析
由qRT-PCR 分析(图7)表明MdGRF13 基因在15% PEG 以及200 mmol·L-1 NaCl 处理12 h 和24 h后较对照显著上调表达,说明其在抵御非生物胁迫的过程中发挥重要的作用。因此,选择MdGRF13基因克隆并进行功能验证。
以嘎拉苹果cDNA为模板,进行PCR扩增,得到与预期目标一致的条带(图8-A),将扩增产物回收纯化后连接大肠杆菌DH5α 并通过PCR 阳性检测,所得结果与原序列(297 bp)相吻合(图8-B)。最后转入农杆菌,PCR检测条带合适(图8-C)。
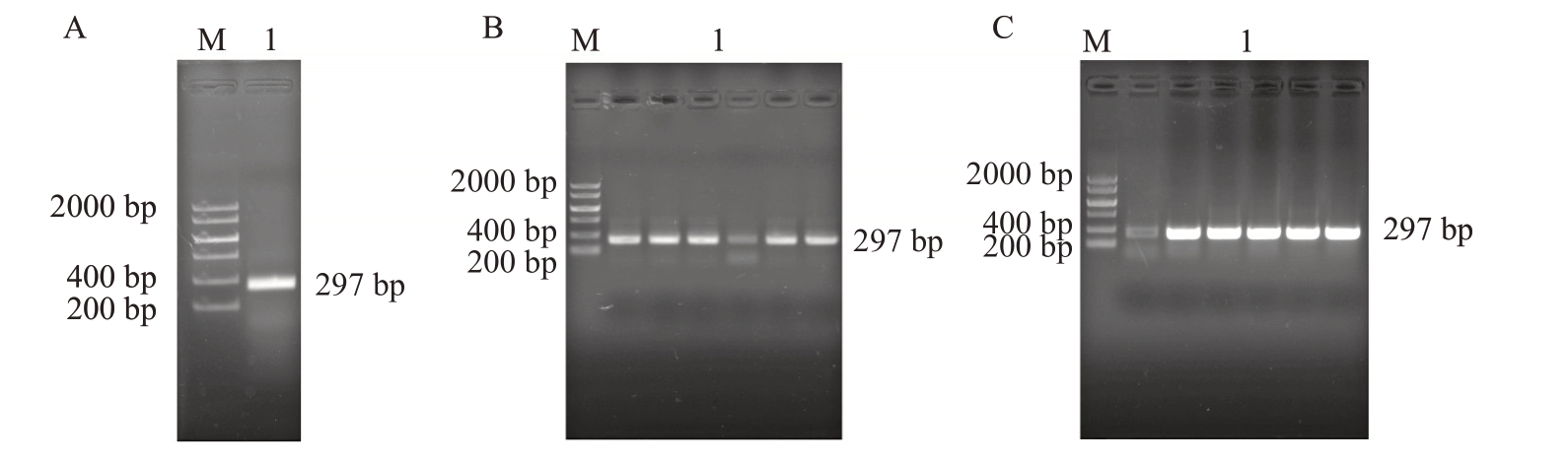
图8 苹果MdGRF13 基因克隆与PCR 检测
Fig.8 MdGRF13 gene cloning and PCR detection
M.Marker,1.苹果MdGRF13 基因PCR 产物。A.苹果MdGRF13 基因扩增电泳图;B.苹果MdGRF13 基因大肠杆菌菌液PCR 检测;C.苹果MdGRF13 基因农杆菌菌液PCR 检测。
M. Marker,1 indicate PCR product of apple MdGRF13 gene.A.Amplification electrophoresis map of MdGRF13 gene; B. Electrophoretogram of MdGRF13 Escherichia coli;C.Electrophoretogram of MdGRF13 Agrobacterium.
以pCAMBIA1300-GFP 空载体为阳性对照,将MdGRF13 重组质粒在烟草叶片中瞬时表达。结果表明(图9),pCAMBIA1300-MdGRF13-GFP 表达载体的荧光定位在细胞核、细胞质和细胞膜上。
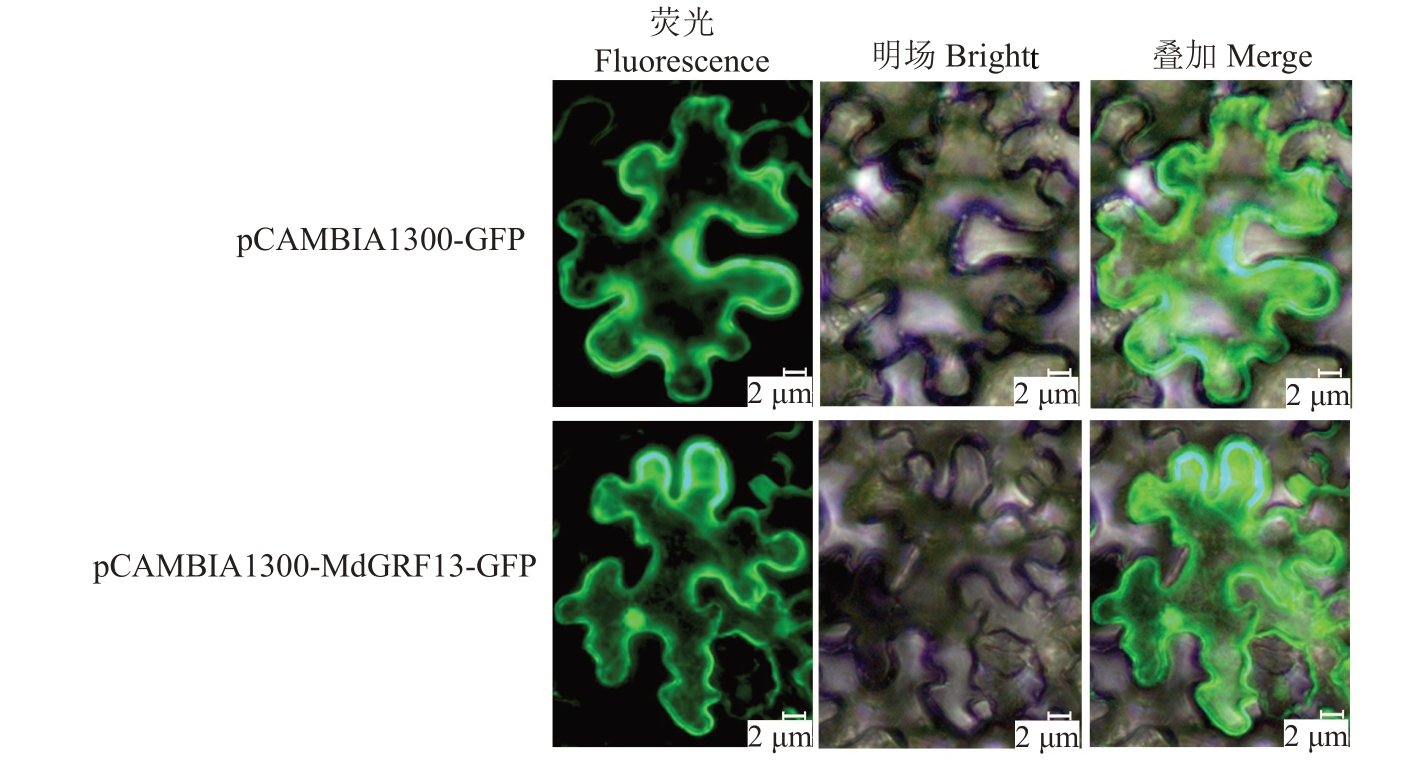
图9 MdGRF13 蛋白的亚细胞定位
Fig.9 Subcellular localization of the MdGRF13 protein
2.9 MdGRF13过表达在苹果愈伤组织的功能鉴定
将过表达载体pCAMBIA1300-MdGRF13-GFP转化王林苹果愈伤组织(图10-A)。对所得转基因愈伤组织通过PCR检测,获得了与阳性对照片段大小一致的条带(图10-B),证明MdGRF13 在苹果愈伤组织中转化成功。选取过表达型OE-4、OE-2 和OE-3进行处理。
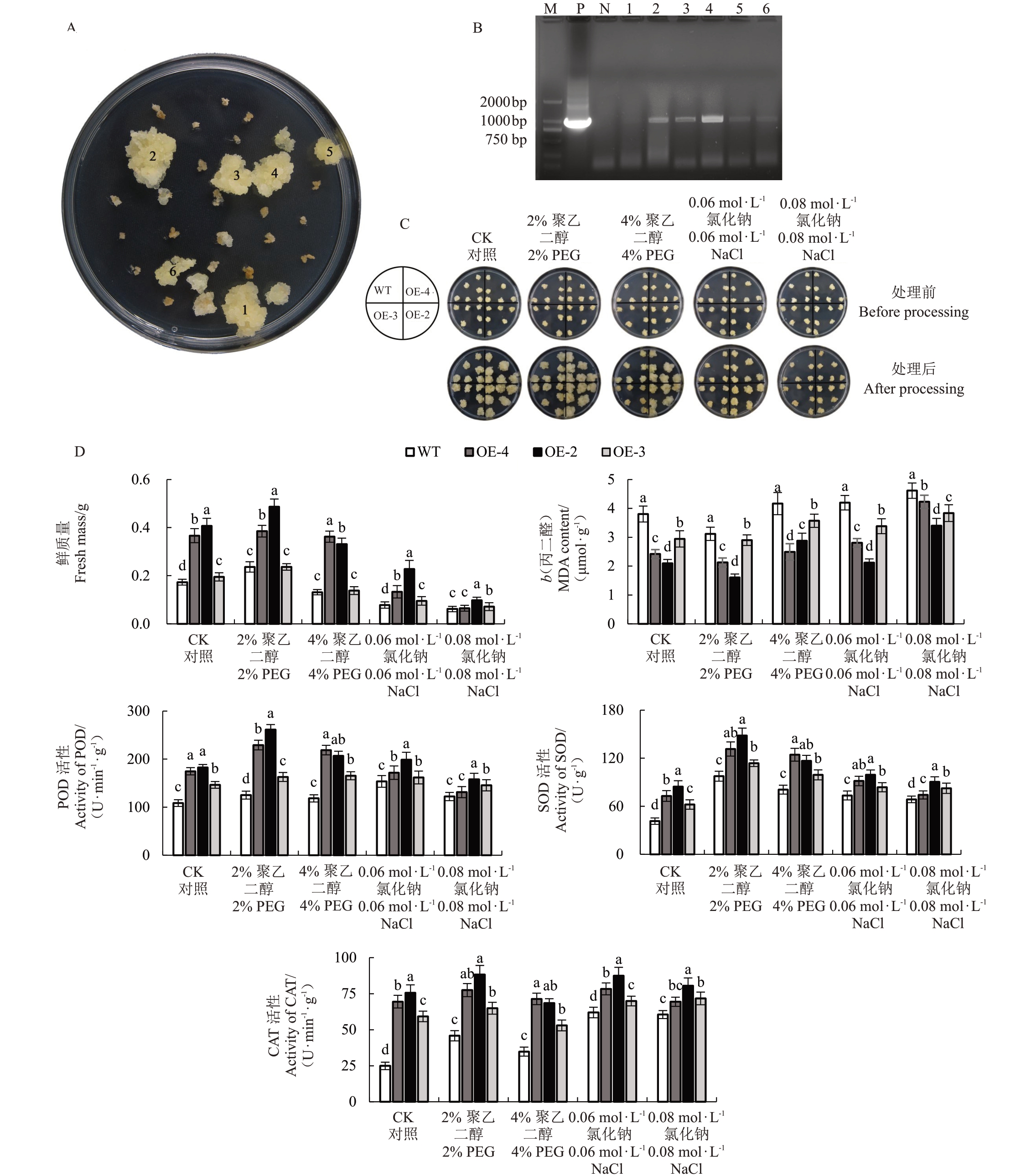
图10 转基因愈伤组织鉴定和酶活性测定
Fig.10 Identification of transgenic callus and assay of enzyme activities
A.转基因愈伤组织;B.愈伤组织阳性检测,M.Marker,P.阳性对照,N.野生型阴性对照,1~6 为转基因愈伤组织PCR 产物;C.不同浓度PEG 及NaCl 处理下愈伤组织表型;D.生理指标测定。
A.Transgenic callus;B.The positive detection of callus,M.Marker,P.positive control,N.Wild type negative control,1~6.Indicate PCR product of transgenic callus,C.Phenotype of callus treated with different concentrations of PEG and NaCl;D.Physiological index determination.
对野生型与过表达型愈伤组织经不同浓度PEG处理下表型观察发现(图10-C),过表达型(OE)愈伤组织长势优于野生型(WT),且差异显著,而2%PEG 处理下愈伤组织整体长势优于4% PEG。0.06 mol·L-1 NaCl 处理下,过表达型(OE)愈伤组织长势优于野生型(WT),而在0.08 mol·L-1 NaCl处理下,过表达型(OE)愈伤组织长势与野生型(WT)无显著差异。
对WT 和OE 愈伤组织进行鲜质量统计,并对MDA(丙二醛)含量以及CAT、POD 和SOD 酶活性进行测定。结果表明(图10-D),0.08 mol·L-1 NaCl处理下,OE 愈伤组织鲜质量、MDA 含量以及CAT、POD和SOD酶活性较WT无显著差异,而其他处理下,OE愈伤组织鲜质量以及CAT、POD和SOD酶活性高于WT,而OE 的MDA 含量低于WT。表明过表达MdGRF13 能够增强苹果愈伤组织对干旱和盐胁迫的抗性。
3 讨 论
14-3-3蛋白是一类调控植物激酶与转录因子信号转导的基础性蛋白,参与调节植物多种生理和发育过程[34-35]。笔者在本研究中通过对苹果基因组数据分析鉴定得到36个苹果14-3-3基因家族成员,与研究鉴定得到的拟南芥[16]、番茄[9]、苜蓿[11]、大豆[12]和茶树[36]14-3-3 家族成员(分别为15、12、18、11 和23个)相比,苹果14-3-3基因家族成员数量大,表明14-3-3基因家族成员在不同物种间有较大差异,而本研究较Ren 等[27]和Zuo 等[21]已发表文章鉴定出更多的苹果14-3-3 家族成员,其中7 个基因(MdGRF8、12、13、16、23、26和27)未被鉴定,且都存在于进化关系的 非 ε 类 ,MdGRF12/MdGRF13、MdGRF16/MdGRF26 及MdGRF23/MdGRF27 相邻(图2),进化关系较近。笔者在本文中通过qPCR 分析发现MdGRF19(MD10G1280300)在茎中表达量最高,而在叶中表达量最低,与Ren等[27]的研究结果相反,与Zuo 等[21] 的研究结果相同。 MdGRF11(MD10G1280300)[27]在ABA 和PEG 处理下表达趋势与本文MdGRF19(MD10G1280300)研究结果基本一致。Zuo 等[21]亚细胞定位发现MdGF14j(MD10G1280300)蛋白定位在细胞质和细胞核中,与Ren等[27]的研究结果相同。推测其原因可能是不同版本的数据库有差异,以苹果不同品种间的数据比对筛选出更多被识别的基因。
理化性质分析表明,苹果14-3-3 蛋白pI 值小于7,这与李菲等[11]14-3-3蛋白是酸性氨基酸的研究结果一致。系统进化分析显示苹果14-3-3基因家族分为ε类和非ε类,且非ε类双子叶植物苹果(52.8%)、拟南芥(57.1%)和番茄(58.3%)比例高于单子叶植物二穗短柄草(85.7%)和番茄(85.7%),在ε 类的结果相同[17]。Zuo 等[21]的研究表明,苹果MdGF14a、MdGF14d、MdGF14i和MdGF14j蛋白与关键的花整合因子MdTFL1和MdFT相互作用,参与开花调控,在本研究中,MdGRF6 在系统发育树与MdGF14i 和MdGF14j 相邻,推测其具有相似的作用。研究发现,14-3-3蛋白以N-端和C-端不同的motif为核心结构发挥不同功能[37],本研究中苹果14-3-3家族蛋白ε和非ε类的N-端与C-端有不同的motif,该家族蛋白可能与靶蛋白相结合发挥重要功能。苹果14-3-3家族基因有10 对片段重复和6 对串联重复基因,表明片段重复在其基因家族的进化过程中起着重要的作用,并且串联和片段重复事件有利于植物进化过程中基因家族成员的扩展[38-39]。烟草叶片瞬时表达亚细胞定位实验表明,MdGRF13 蛋白定位在细胞核、细胞质和细胞膜中,与Ren等[27]和Zuo等[21]所得结果相似,说明14-3-3 家族蛋白在不同植物中有保守功能,其可能在细胞核中调控部分基因的转录。
组织特异性表达有助于了解植物中的基因功能[40-41]。通过qRT-PCR 发现苹果14-3-3 家族基因在根、茎和叶组织中都有表达,而在茎中表达量较高,与木薯14-3-3蛋白在块根不同发育时期有不同组织的表达研究结果相似[42],说明14-3-3 家族基因在苹果中有一定的组织特异性表达。研究发现,水稻OsGF14b 的突变对干旱和渗透胁迫耐受性的提高可能与ABA 信号通路有关[43]。qPCR 结果显示,MdGRF4、MdGRF16、MdGRF18 和MdGRF26 在ABA 处理下上调表达,本文结果与以上研究相似,推测该家族基因功能与ABA相关,表明苹果14-3-3蛋白可能与ABA介导的保卫细胞相结合,进而影响其在激活ABA应答基因中的转录[5]。干旱胁迫会降低植物的光合和碳水化合物的合成,进而减缓植物的生长发育[44]。14-3-3蛋白作为重要的干旱调控因子在胁迫反应中有不同程度的表达,MdGRF26 在PEG处理下高表达,同时,6个基因有一定量的上调表达。本研究在苹果愈伤组织过表达MdGRF13,在不同浓度PEG处理下,转基因愈伤组织长势优于野生型,且以2% PEG 处理下长势最好,与棉花CGF14-4[45]和油菜GF14omeg 基因[46]在干旱胁迫下上调表达研究结果相似,表明过表达MdGRF13 增强了苹果愈伤组织的抗旱性。实时荧光定量PCR发 现,MdGRF2、MdGRF4、MdGRF5、MdGRF13、MdGRF14 和MdGRF16 在NaCl 处理下表达量分别为对照的18.6、7.6、7.5、2.1、6.5 和15.6 倍,且本研究在0.06 mol·L-1 NaCl 处理下,转基因愈伤组织长势优于野生型,与小麦[23]、番茄[9]、水稻[47]及短柄草[48]TaGF14b在烟草中异位表达时增强盐胁迫耐受性的研究结果相似,表明过表达MdGRF13增强了苹果愈伤组织对盐胁迫的抗性。而在0.08 mol·L-1 NaCl处理下,转基因愈伤组织较野生型表型未发生较大变化,可能是较高浓度NaCl 处理抑制了愈伤组织的生长。同时,本文研究结果与Ren 等[27]MdGRF11(MD10G1280300)在愈伤组织中过表达可增强抗旱性和耐盐性研究结果一致。说明苹果14-3-3家族基因在抵御非生物胁迫的过程中发挥了重要的作用。
4 结 论
本研究通过生物信息学分析鉴定出36 个苹果14-3-3 基因家族成员,系统进化分析发现其与拟南芥同源性较高,且各亚族成员结构保守。亚细胞定位显示,MdGRF13 蛋白定位在细胞核、细胞质和细胞膜中。过表达MdGRF13 增强了苹果愈伤组织的抗旱性和耐盐性。对进一步研究苹果14-3-3家族成员的功能具有重要意义。
[1] ANDERAS M,LUCA C.The effect of stress on genome regulation and structure[J].Annals of Botany,2004,94(4):481-495.
[2] BOYKO A,KOVALCHUK I. Epigenetic control of plant stress response[J]. Environmental and Molecular Mutagenesis,2008,49(1):61-72.
[3] SHESHADRI S A,NISHANTH M J,BINDU S. Stress-mediated cis-element transcription factor interactions interconnecting primary and specialized metabolism in planta[J]. Frontiers in Plant Science,2016,7:1725-1748.
[4] YAMAGUCHI-SGINOZAKI K,SHINOZAKI K. Organization of cis-acting regulatory elements in osmotic and cold-stress-responsive promoters[J].Trends in Plant Science,2005,10(2):88-94.
[5] ROBERTS M R,SALINAS J,COLLINGE D B.14-3-3 proteins and the response to abiotic and biotic stress[J]. Plant Molecular Biology,2002,50(6):1031-1039.
[6] OLSSON A,SVENNELID F,SOMMARIN M,LARSSON C.A phosphothreonine residue at the C-terminal end of the plasma membrane H+-ATPase is protected by fusicoccin-induced 14-3-3 binding[J].Plant Physiology,1998,118(2):551-555.
[7] PAUL A L,DENISON F C,SCHULTZ E R,ZUPANSKA A K,FERL R J.14-3-3 phosphoprotein interaction networks-dose isoform diversity present functional interaction specification?[J].Frontiers in Plant Science,2012,3:190-204.
[8] ROSENQUIST M,ALSTERFJORD M,LARSSON C,SOMMARIN M. Data mining the Arabidopsis genome reveals fifteen 14-3-3 genes. Expression is demonstrated for two out of five novel genes[J].Plant Physiology,2001,127(1):142-149.
[9] XU W F,SHI W M.Expression profiling of the 14-3-3 gene family in response to salt stress and potassium and iron deficiencies in young tomato(Solanum lycopersicum)roots:Analysis by realtime RT-PCR[J].Annals of Botany,2006,98(5):965-974.
[10] 马勇,刘嘉荣,巴德仁贵,哈斯阿古拉.甜瓜14-3-3 基因家族全基因组鉴定与进化分析[J]. 广东农业科学,2013,40(24):
104-111.MA Yong,LIU Jiarong,Bade Rengui,Hasi Agula. Genomewide identification and phylogenetic analysis of 14-3-3 gene family in melon[J]. Guangdong Agricultural Sciences,2013,40(24):104-111.
[11] 李菲,何小红,张习敏,龚记熠,乙引.苜蓿14-3-3 基因家族的鉴定与进化和特征分析[J].基因组学与应用生物学,2017,36(12):5238-5243.LI Fei,HE Xiaohong,ZHANG Ximin,GONG Jiyi,YI Yin.Identification,evolution and characteristic analysis of 14-3-3 gene family in medicago[J].Genomics and Applied Biology,2017,36(12):5238-5243.
[12] 李昕文,沙爱华,黄家权.大豆14-3-3 家族基因对低磷胁迫响应及其与磷转运蛋白PHT6 的互作[J]. 中国油料作物学报,2017,39(3):308-314.LI Xinwen,SHA Aihua,HUANG Jiaquan. Response of 14-3-3 family genes to phosphorus starvation in soybean and their interaction with phosphorus transporter PHT6[J]. Chinese Journal of Oil Crop Sciences,2017,39(3):308-314.
[13] 姜焕焕,王通,禹山林,陈明娜,王冕,陈娜,潘丽娟,祁佩时,迟晓元.花生14-3-3 基因家族的生物信息学分析[J].中国油料作物学报,2018,40(4):501-507.JIANG Huanhuan,WANG Tong,YU Shanlin,CHEN Mingna,WANG Mian,CHEN Na,PAN Lijuan,QI Peishi,CHI Xiaoyuan. Bioinformatic analysis of 14-3-3 gene family in peanut[J].Chinese Journal of Oil Crops,2018,40(4):501-507.
[14] 张书芹.陆地棉14-3-3 基因家族的鉴定及逆境胁迫应答[J].分子植物育种,2019,17(11):3468-3476.ZHANG Shuqin. Identification and stress response of 14-3-3 gene family in upland cotton (Gossypium hirsutum)[J]. Molecular Plant Breeding,2019,17(11):3468-3476.
[15] XIA L M,HE X H,HUANG X,YU H X,LU T T,XIE X J,ZENG X M,ZHU J W,LUO C. Genome-wide identification and expression analysis of the 14-3-3 gene family in mango(Mangifera indica L.)[J]. International Journal of Molecular Sciences,2022,23(23):1593-1610.
[16] DELILLE J M,SEHNKE P C,FERL R J.The Arabidopsis 14-3-3 family of signaling regulators[J].Plant Physiology,2001,126(1):35-38.
[17] TIAN F X,WANG T,XIE Y L,ZHANG J,HU J J. Genomewide identification,classification,and expression analysis of 14-3-3 gene family in Populus[J]. PLoS One,2015,10(4):1-22.
[18] GUO J,DAI S,LI H S,LIU A F,LIU C,CHENG D G,CAO X Y,CHU X S,ZHAI S N,LIU J J,ZHAO Z D,SONG J M.Identification and expression analysis of wheat TaGF14 Genes[J].Frontiers in Genetics,2018,9:12-22.
[19] CHENG C,WANG Y,CHAI F M,LI S H,XIN H P,LIANG Z C. Genome-wide identification and characterization of the 14-3-3 family in Vitis vinifera L. during berry development and cold-and heat-stress response[J]. BMC Genomics,2018,19(1):579-593.
[20] LI M Y,REN L C,XU B Y,YANG X L,XIA Q Y,HE P P,XIAO S S,GUO A P,HU W,JIN Z Q.Genome-wide identifcation,phylogeny,and expression analyses of the 14-3-3 family reveal their involvement in the development,ripening,and abiotic stress response in banana[J]. Frontiers in Plant Science,2016,7:1442-1456.
[21] ZUO X Y,WANG S X,XIANG W,YANG H R,TAHIR M M,ZHENG S G,AN N,HAN M Y,ZHAO C P,ZHANG D.Genome-wide identification of the 14-3-3 gene family and its participation in floral transition by interacting with TFL1/FT in apple[J].BMC Genomics,2021,22(1):1-17.
[22] XIAO B J,LUO G H,WANG K S,MAO Z Y,LIN B Z.Overexpression of PvGF14c from phyllostachys violascens delays flowering time in transgenic Arabidopsis[J].Frontiers in Plant Science,2018,9:105-127.
[23] ZHANG Y,ZHAO H Y,ZHOU S Y,HE Y,LUO Q,ZHANG F,QIU D,FENG J,WEI Q,CHRN L. Expression of TaGF14b,a 14-3-3 adaptor protein gene from wheat,enhances drought and salt tolerance in transgenic tobacco[J]. Planta,2018,248(1):117-137.
[24] 李芳,滕建晒,陈宣钦.14-3-3 蛋白参与植物应答非生物胁迫的研究进展[J].植物科学学报,2018,36(3):459-469.LI Fang,TENG Jianshai,CHEN Xuanqin. Research progress on the 14-3-3 protein involved in plant responses to abiotic stress[J].Plant Science Journal,2018,36(3):459-469.
[25] YANG Z J,WANG C W,XUE Y,LIU X,CHEN S,SONG C P,YANG Y Q,GUO Y. Calcium-activated 14-3-3 proteins as a molecular switch in salt stress tolerance[J]. Nature Communications,2019,10(1):1199-1211.
[26] CAO J F,HUANG J Q,LIU X,HUANG C C,ZHENG Z S,ZHANG X F,SHANG G,WANG L J,ZHANG Y G,WENDEL J F,GROVER C E,CHEN Z W. Genome-wide characterization of the GRF family and their roles in response to salt stress in Gossypium[J].BMC Genomics,2020,21(1):1-16.
[27] REN Y R,YANG Y Y,ZHANG R,YOU C X,ZHAO Q.MdGRF11,an apple 14-3-3 protein,acts as a positive regulator of drought and salt tolerance[J]. Plant Science,2019,288:110219-110231.
[28] 马维峰,李艳梅,马宗桓,陈佰鸿,毛娟.苹果POD 家族基因的鉴定与MdPOD15 的功能分析[J].园艺学报,2022,49(6):1181-1199.MA Weifeng,LI Yanmei,MA Zonghuan,CHEN Baihong,MAO Juan.Identification of apple POD gene family and functional analysis of MdPOD15 gene[J].Acta Horticulturae Sinica,2022,49(6):1181-1199.
[29] 刘勇,王泽琼,龚林忠,王富荣,王会良,艾小艳,何华平.桃乙烯应答因子PpERF1a 的克隆与功能分析[J]. 园艺学报,2020,47(6):1165-1171.LIU Yong,WANG Zeqiong,GONG Linzhong,WANG Furong,WANG Huiliang,AI Xiaoyan,HE Huaping.Cloning and functional analysis of ERF transcription factor gene PpERF1a in peach[J].Acta Horticulturae Sinica,2020,47(6):1165-1171.
[30] 张婷婷,康慧,付璐璐,由春香,王小非,郝玉金. 苹果MdCYP707A 家族基因表达分析和MdCYP707A1 的功能鉴定[J].园艺学报,2019,46(8):1429-1444.ZHANG Tingting,KANG Hui,FU Lulu,YOU Chunxiang,WANG Xiaofei,HAO Yujin. Expression analysis of apple MdCYP707A family genes and functional characterization of MdCYP707A1[J].Acta Horticulturae Sinica,2019,46(8):1429-1444.
[31] 杨蕾蕾.苹果质膜内在蛋白基因MdPIP1;1 和MdPIP1;2 在干旱和盐胁迫中的功能分析[D].杨凌:西北农林科技大学,2020.YANG Leilei. Functional analysis of apple plasma membrane intrinsic protein genes MdPIP1;1 and MdPIP1;2 under drought and salt stress[D].Yangling:North West Agriculture and Forestry University,2020.
[32] 赵世杰,许长成,邹琦,孟庆伟.植物组织中丙二醛测定方法的改进[J].植物生理学通讯,1994,30(3):207-210.ZHAO Shijie,XU Changcheng,ZOU Qi,MENG Qingwei. Improvements of method for measurement of malondialdehyde in plant tissues[J]. Plant Physiology Communications,1994,30(3):207-210.
[33] 张志良.植物生理学实验指导[M].北京:高等教育出版社,2008.ZHANG Zhiliang. Guide to plant physiology experiment[M]. Beijing:Higher Education Press,2008.
[34] CAMPO S,PERIS- PERIS C,MONTESINOS L,PENAS G,MESSEGUER J,SAN S B. Expression of the maize ZmGF14-6 gene in rice confers tolerance to drought stress while enhancing susceptibility to pathogen infection[J]. Journal of Experimental Botany,2012,63(2):983-999.
[35] LEE J H,LU H. 14-3-3γ inhibition of MDMX-mediated p21 turnover independent of p53[J]. Journal of Biological Chemistry,2011,286(7):5136-5142.
[36] 安海丽,张宝会,姚新转,吕立堂.茶树14-3-3 基因家族的鉴定和表达模式分析[J/OL].分子植物育种,2021:1-20.[2022-07-07].http://kns.cnki.net/kcms/detail/46.1068.S.20210909.1626.024.html.AN Haili,ZHANG Baohui,YAO Xinzhuan,LÜ Litang.Identification and expression pattern analysis of 14-3-3 gene family in(Camellia sinensis L.)[J/OL]. Molecular Plant Breeding,2021:1- 20. [2022- 07- 07]. http://kns.cnki.net/kcms/detail/46.1068.S.20210909.1626.024.html.
[37] 周颖,李冰樱,李学宝.14-3-3 蛋白对植物发育的调控作用[J].植物学报,2012,47(1):55-64.ZHOU Ying,LI Bingying,LI Xuebao. Roles of 14-3-3 proteins in regulating plant development[J]. Chinese Bulletin of Botany,2012,47(1):55-64.
[38] CANNON S B,MITRA A,BAUMGARTEN A,YOUNG N D,MAY G.The roles of segmental and tandem gene duplication in the evolution of large gene families in Arabidopsis thaliana[J].BMC Plant Biology,2004,4(1):10-30.
[39] SUN H H,WU S,ZHANG G Y,JIAO C,GUO S G.Karyotype stability and unbiased fractionation in the paleo-allotetraploid Cucurbita genomes[J]. Molecular Plant,2017,10(10):1293-1306.
[40] GENG M T,YAO Y,WANG Y L.Structure,expression,and functional analysis of the hexokinase gene family in cassava[J].International Journal of Molecular Sciences,2017,18(5),1041-1059.
[41] MA B Q,LIAO L,FANG T,QIAN P,COLLINS O,HUI Z,MA F W,HAN Y P. A Ma10 gene encoding P-type ATPase is involved in fruit organic acid accumulation in apple[J]. Plant Biotechnology Journal,2019,17(3):674-686.
[42] WANG X C,CHANG L L,TONG Z,WANG D Y,QI Y,WANG D,JIN X,YANG Q,WANG L M,SUN Y,HUANG Q X,GUO A P,PENG M.Proteomics profiling reveals carbohydrate metabolic enzymes and 14-3-3 proteins play important roles for starch accumulation during cassava root tuberization[J]. Scientific Reports,2016,6(1):1-15.
[43] LIU J P,SUN X J,LIAO W C,ZHANG J H,LIANG J S,XU W F. Involvement of OsGF14b adaptation in the drought resistance of rice plants[J].Rice,2019,12(1):1-7.
[44] 吕慧,吉雪花,张中荣,朱冉冉,王世宁,谢雪果,袁雷.制干辣椒果实辣椒素对干旱、盐及其双重胁迫的响应[J].中国瓜菜,2022,35(2):78-84.LÜ Hui,JI Xuehua,ZHANG Zhongrong,ZHU Ranran,WANG Shining,XIE Xueguo,YUAN Lei. Capsaicin of dry pepper fruit grown under drought,salt and combined stress condition[J].China Cucurbits and Vegetables,2022,35(2):78-84.
[45] SUN G L,XIE F L,ZHANG B H.Transcriptome-wide identification and stress properties of the 14-3-3 gene family in cotton(Gossypium hirsutum L.)[J]. Functional & Integrative Genomics,2011,11(4):627-636.
[46] LUO J L,TANG S H,PENG X J,YAN X H,ZENG X H,LI J,LI X F,WU G.Elucidation of cross-talk and specificity of early response mechanisms to salt and PEG-simulated drought stresses in Brassica napus using comparative proteomic analysis[J].PLoS One,2015,10(10):e0138974.
[47] CHEN Y X,ZHOU X J,CHANG S,CHU Z L,WANG H M.Calcium-dependent protein kinase 21 phosphorylates 14-3-3 proteins in response to ABA signaling and salt stress in rice[J].Biochemical and Biophysical Research Communications,2017,493(4):1450-1456.
[48] HE Y,ZHANG Y,CHEN L H,WU C L,LUO Q C,ZHANG F,WEI Q H,LI K X,CHANG J L,YANG G X,HE G Y.A member of the 14-3-3 gene family in Brachypodium distachyon,BdGF14d,confers salt tolerance in transgenic tobacco plants[J].Fronti ers in Plant Science,2017,8:340-353.