葡萄皱木复合征(rugose wood complex disease)是一种普遍发生且危害严重的葡萄病毒病[1],症状包括茎沟、茎痘以及砧木或幼芽木质部的扭曲,葡萄病毒B(Grapevine virus B,GVB)是引起葡萄皱木复合征的病毒之一[2-5]。研究发现,大部分欧洲种葡萄一但嫁接到带有GVB的美洲种砧木上,就会引起植株的早春延迟萌芽,枝蔓树皮粗糙且松散,木质部出现沟或槽,严重影响产量及品质,并易造成藤的早期死亡,对鲜食葡萄和酿酒葡萄产区危害极大。GVB是在1999 年于澳大利亚第一次被检出[6],目前该病毒已在中国、美国、南非、意大利、以色列、法国、希腊和印度等多个国家和地区发生危害[7-10]。在我国的河南、湖北、四川、辽宁、山东、河北、新疆以及宁夏等地也均有该病毒病的发生[11-13]。
GVB 属于乙型线形病毒科(Betaflexiviridae)葡萄病毒属(Vitivirus)成员,该病毒粒子呈线性弯曲状,长725~825 nm,直径12~13 nm[14],基因组为正义单 链RNA 分 子(positive- sense single stranded RNA,+ssRNA),病毒基因组含有核苷酸约为7600个,不包含3′端poly(A)尾巴[15]。基因组编码5个开放阅读框(open reading frame,ORF),ORF1~ORF5分别编码194.7 ku 复制酶蛋白(replication protein,RP)、20.2 ku 的未知功能蛋白、36.5 ku 运动蛋白(movement protein,MP)、21.5 ku外壳蛋白(coat protein,CP)和13.9 ku 核酸结合蛋白(nucleicacid binding protein,NABP)。5’端非编码区(untranslated region,UTR)大小为48 nt,3’-UTR大小为142~148 nt。GVB 主要是通过拟葡萄粉蚧(Pseudococcus affinis)和榕臀纹粉蚧(planococcus ficus)进行传播[16-17],该病毒的草本寄主分别是本氏烟(Nicotiana benthamiana)、西方烟(N. occidentalis)和凯维科拉烟(N.cavicola)[18]。
笔者所在实验室于2018—2020 年之间开展了内蒙古自治区葡萄病毒的检测工作[19],旨在丰富GVB的基因数据库,明确GVB的遗传进化情况,为我国GVB 分类地位、病毒进化规律研究,以及后续开展侵染性克隆奠定一定基础。
1 材料和方法
1.1 供试材料
从内蒙古自治区7 个市区共采集69 份葡萄样品,其中从2 株采自内蒙古自治区呼和浩特市赛罕区的阳光玫瑰葡萄样品中检测到了GVB的侵染,感病葡萄表现为叶片斑驳皱缩并轻微畸形。采样中将2 个样品(GVB 内蒙古分离物,下同)分别命名为22号和28号,GVB的检出频率为2.9%(2/69)。
1.2 主要试剂
总RNA 提取试剂盒SpectrumTM Plant Total RNA Kit 购自西格玛奥德里奇(上海)贸易有限公司;PrimeScript™Ⅱ1st Strand cDNA Synthesis Kit购自普洛麦格(北京)生物技术有限公司;M5 超光速mix 购自北京聚合美生物科技有限公司;琼脂糖凝胶DNA 回收试剂盒、克隆载体CV14-Zero Background pTOPO-TA Cloning Kit购自艾德莱生物技术有限公司;SMARTer®RACE 5’/3’Kit购自宝生物工程(大连)有限公司;KAPA HiFi HotStart Ready Mix PCR Kit 购自北京普凯瑞生物科技有限公司;Marker(DNA Marker Ⅲ、DL2000)购自天根生化科技(北京)有限公司;大肠杆菌感受态Escherichia coli JM109 Competent Cells购自宝日医生物技术(北京)有限公司。
1.3 方法
1.3.1 总RNA提取 总RNA提取具体步骤参考试剂盒SpectrumTM Plant Total RNA Kit 说明书。将最终得到的总RNA经微量分光光度计(杭州奥盛仪器有限公司)测定浓度和纯度,进行电泳检测其完整性,并保存于-20 ℃冰箱备用。
1.3.2 反转录合成cDNA 第一链 使用Prime-Script™Ⅱ1st Strand cDNA Synthesis Kit[普洛麦格(北京)生物技术有限公司]试剂盒将提取的总RNA 反转录成cDNA。步骤如下:总RNA 1.5 μL,Oligo(DT)1 μL,dNTP mixture(2.5 mmol)4 μL,RNase free ddH2O 6.5 μL,溶液混合后瞬离,于PCR仪中65 ℃变性5 min 后迅速冰浴1 min,再加入5×First Strand Buffer 4 μL,0.1 mol·L-1 DTT 1 μL,RNase Inhibitor 1 μL,SuperScipt Ⅲ1 μL,总体系为20 μL。PCR 仪中50 ℃变性1 h,70 ℃反应15 min。反转录产物在-20 ℃冰箱保存。
1.3.3 GVB基因组的克隆 引物设计。根据NCBI中Genbank 数据库中已报道的22 条GVB 全基因组序列的保守区分别设计3 对GVB 基因片段扩增所用引物GVB 1/GVB 2、GVB 3/GVB 4、GVB 5/GVB 6 与3’RACE、5’RACE 引物及其简并引物进行RTPCR扩增,分3段对该基因片段序列进行克隆,最终将获得的所有序列片段拼接整合得到GVB 基因组全序列,引物均由生工生物工程(上海)股份有限公司合成(表1)。
表1 本研究中所用引物
Table 1 Primers used for detection and amplification of full-length genome of GVB
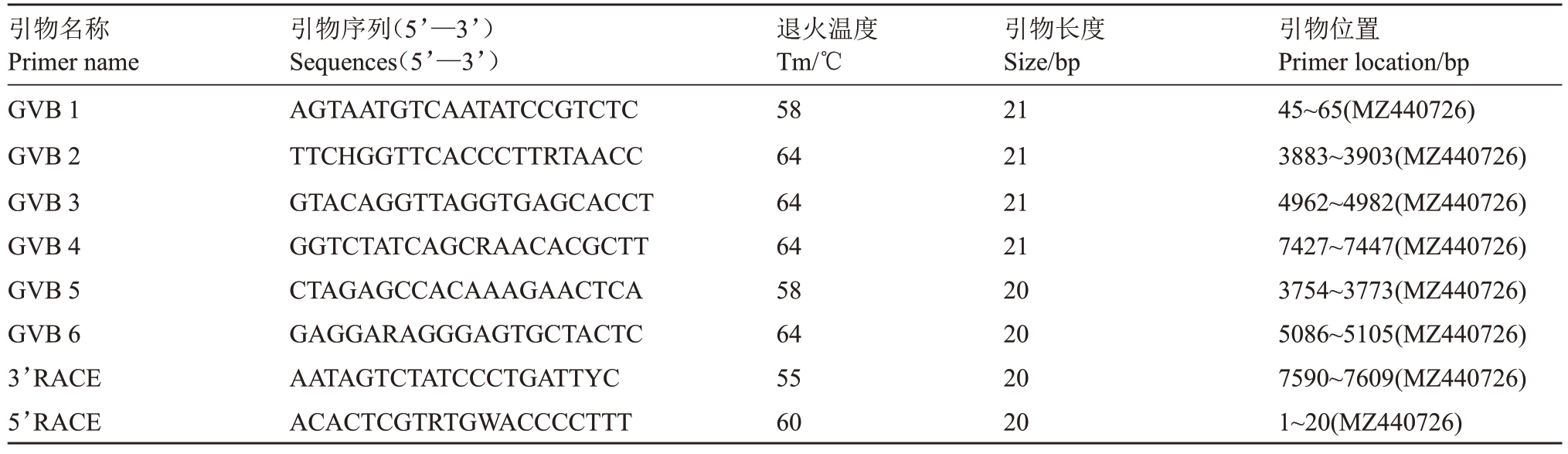
引物名称Primer name GVB 1 GVB 2 GVB 3 GVB 4 GVB 5 GVB 6 3’RACE 5’RACE引物序列(5’—3’)Sequences(5’—3’)AGTAATGTCAATATCCGTCTC TTCHGGTTCACCCTTRTAACC GTACAGGTTAGGTGAGCACCT GGTCTATCAGCRAACACGCTT CTAGAGCCACAAAGAACTCA GAGGARAGGGAGTGCTACTC AATAGTCTATCCCTGATTYC ACACTCGTRTGWACCCCTTT退火温度Tm/℃58 64 64 64 58 64 55 60引物长度Size/bp 21 21 21 21 20 20 20 20引物位置Primer location/bp 45~65(MZ440726)3883~3903(MZ440726)4962~4982(MZ440726)7427~7447(MZ440726)3754~3773(MZ440726)5086~5105(MZ440726)7590~7609(MZ440726)1~20(MZ440726)
GVB 基因组扩增。以反转录合成的cDNA 为模板,使用2×M5 Hiper超光速mix对22号和28号阳光玫瑰葡萄样品分3 段分别扩增GVB 基因组。PCR反应总体系为20 μL,2×M5 Hiper超光速mix 10µL,引物(10µmol·L-1)各1µL,cDNA 2µL,RNase free ddH2O 补平20µL,分别使用引物对GVB 1/GVB 2、GVB 3/GVB 4、GVB 5/GVB 6(表1)。GVB 基因组PCR 扩增程序按下列条件进行35 个循环:95 ℃预变性3 min,95 ℃变性20 s,GVB 1/GVB 2 引物对49 ℃退火20 s,72 ℃延伸210 s;GVB 3/GVB 4引物对56 ℃退火20 s,72℃延伸70 s;GVB 5/GVB 6 引物对53 ℃退火20 s,72 ℃延伸150 s,最后72 ℃补平5 min,12 ℃降温2 min。
使用KAPA HiFi HotStart Ready Mix PCR Kit试剂盒扩增5’和3’端,PCR 反应总体系为25 µL,2× KAPA HiFi HotStart Ready Mix 12.5 µL,引物(10 µmol·L-1)各1 µL,cDNA 2 µL,RNase free ddH2O 补平至25 µL,引物包括SMARTer ®RACE 5’/3’Kit 试剂盒提供的UPM 引物、GVB 3’末端序列特异性引物3’RACE 以及5’末端序列特异性引物5′RACE。5’和3’端PCR 扩增按以下程序进行:98 ℃预变性4 min,30个循环:98 ℃变性30 s,60 ℃退火45 s,72 ℃延伸45 s;72 ℃补平5 min,12 ℃降温1 min。将PCR产物通过1.0%(w)琼脂糖凝胶电泳并在紫外灯下进行检测,判断是否扩增出目标条带。在紫外灯下将目标条带切下,放进2 mL离心管中备用。
1.3.4 PCR产物回收纯化、连接及转化 将PCR扩增产物进行胶回收。具体步骤参考琼脂糖凝胶DNA 回收试剂盒说明书。纯化后的PCR 产物使用零背景pTOPO-TA克隆试剂盒和E.coli JM109 Competent Cells 进行连接转化,挑取清晰饱满的白色单菌落进行扩繁。连接产物用热激法转化E. coli JM109。PCR纯化产物连接反应体系如下:DNA 1~8 μL,pTOPO-TA Vector 1 μL,10× Enhance 1 μL,RNase free ddH2O 0~7 μL,总体系为10 μL。
将体系置于37 ℃连接2 h。经蓝白斑筛选,挑取白色菌落在1 mL LB 液体培养基(含有氨苄青霉素)中37 ℃、180 r·min-1培养,直至菌液混浊。对PCR 鉴定为阳性的菌液进行测序,得到阳性克隆后随机送2个对应的菌液进行测序。
1.3.5 基因组结构与序列一致性 为明确2个GVB内蒙古分离物基因组的分子特征,分别将得到2 个分离物的3 段GVB 序列片段与3′RACE、5′RACE 序列使用Vector NTI 10.1.1 软件进行拼接和校正,获得全长基因组序列。将拼接成功的全长基因组序列通过NCBI 数据库(https://www.ncbi.nlm.nih.gov/)的BLASTn 程序进行同源性搜索,确定其是否为GVB。从Genbank 数据库(https://www.ncbi.nlm.nih.gov/)中下载所有已报道的GVB分离物全基因组序列用于后续一致性分析和系统进化分析。利用DNA MAN 1.2.1软件对本试验获得的2个分离物的基因组全序列进行相似性比对,并分析其与Gen-Bank数据库中其他22个GVB分离物之间的全基因序列以及各个ORF之间的核苷酸和氨基酸相似性。
1.3.6 系统发育分析和重组分析 用MEGA 6.0 软件对本试验获得的2 个GVB 内蒙古分离物以及来自不同国家和寄主的22个GVB分离物基于全长基因组及CP基因核苷酸序列进行系统发育分析。采用邻接法(neighbor-joining method,NJ)构建进化树,自展值设为1000,阈值为50%。用于分析的GVB分离物基因组序列一致性、系统发育分析以及重组分析的具体信息如表2所示。
表2 用于一致性、系统发育和重组分析的GVB 基因组参考序列
Table 2 Reference sequences of GVB genome used for identity,phylogenetic and recombination analyses

注:“-”代表分离物名称不明确。下同。
Note:“-”indicates that the isolate name is not clear.The same below.
分离物Isolate 8415 NCBI登录号NCBI accession No.KY426923长度Length/nt 7614 GVB-QMWH KF700375 7610 ISA-BR KX790785 7605 GVB 248 KX522545 7602 GVB 953-1 KJ524452 7601-NC_003602 7599 94/971 EF583906 7599 GVB-H1 GU733707 7599 VD-102 MF991949 7598 12G455 MZ440726 7609 g2 LC617945 7563 GVB-BIB-BR MW357718 7599-X75448 7599 Cultivated-TN1 20PRT01 MN716773 8012 MZ344593 7605 17VT16 MZ440728 7489 16GVP005 MZ440727 7598 12G435 MZ440725 7583 12G4133 MZ440724 7594 12G413 MZ440723 7592 TS_GVB MT454065 7565国家Country加拿大Canada中国China巴西Brazil南非South Africa南非South Africa意大利Italy南非South Africa南非South Africa克罗地亚Croatia加拿大Canada日本Japan巴西Brazil意大利Italy美国USA加拿大Canada加拿大Canada加拿大Canada加拿大Canada加拿大Canada加拿大Canada印度India GVB 3138-01 JX513897寄主Host雷司令Riesling葡萄Grapevine葡萄Grapevine葡萄Grapevine葡萄Grapevine葡萄Grapevine葡萄Grapevine葡萄Grapevine多布里契奇Dobricic葡萄Grapevine葡萄Vitis sp.杂交葡萄Hybrid grapevine葡萄Grapevine葡萄Vitis vinifera葡萄Grapevine葡萄Grapevine葡萄Grapevine葡萄Grapevine葡萄Grapevine葡萄Grapevine汤普森无核葡萄Thompson Seedless葡萄Vitis vinifera 7601美国USA
应用RDP4 软件对GVB 各分离物进行重组分析,分析过程中采用RDP、GENECO NV、BOOTSCAN、MAXCHI、CHIMAERA、SISCAN 和3SEQ 共7 种方法,且以上7 种方法中应有5 种被验证才能称之为重组。软件运行过程中除了分子类型选择线性外,其他均为默认设置。
2 结果与分析
2.1 GVB分离物基因组的扩增
利用GVB 1/GVB 2、GVB 3/ GVB 4、GVB 5/GVB 6与5′RACE、3′RACE 分别进行RT-PCR扩增,获得的特异性条带大小为3780、2489、1047、310、250 bp,与预期相符(图1~2)。

图1 GVB 基因组片段扩增结果
Fig.1 RT-PCR detection result
M.Marker Ⅲ、DL2000;1.清水对照;2.22 号阳光玫瑰葡萄叶片扩增条带;3.28 号阳光玫瑰葡萄叶片扩增条带。
M.Marker Ⅲ,DL2000;1.Water control;2.Leaf amplified band of No.22 Shine Muscat;3.Leaf amplification band of No.28 Shine Muscat.

图2 GVB 基因组5’RACE 和3’RACE 扩增结果
Fig.2 5’RACE and 3’RACE detection result
2.2 GVB 的2 个分离物基因组结构和序列一致性分析
将测序成功的GVB 3 段基因组片段和5′、3′末端片段进行拼接,获得的2个GVB内蒙古分离物基因组全长均为7609 nt,分别命名为GVB Hohhot-1和GVB Hohhot-2,将其提交至GenbBank,登录号分别为OM103705、OM103706。通过序列比对分析后,结果表明含有5 个ORF,分别编码5 种蛋白。ORF1 编 码1 个 长 度 为5133 nt(49~5181 bp)的RdRp;ORF2 编码1 个长度为510 nt(5174~5683 bp)但功能未知的蛋白;ORF3 编码1 个长度为972 nt(5553~6524 bp)的MP;ORF4编码1个长度为591 nt(6448~7038 bp)的CP;ORF5编码1个长度为372 nt(7083~7454 bp)的NABP。5’非编码区为310 nt,3’非编码区为250 nt。
应用SDTv.1.0 软件分析了本试验获得的2 个GVB内蒙古分离物与已报道的22个GVB分离物间基因组核苷酸一致性以及各个ORF 之间的核苷酸与氨基酸一致性。GVB Hohhot-1和GVB Hohhot-2的核苷酸同源性为99%。GVB Hohhot-1 与22 个GVB 分离物全基因组核苷酸同源性为74.8%~96.5%,GVB Hohhot-1 RdRp 的核苷酸与氨基酸序列与其他22 种分离物的一致性分别为74.3%~96.4%和83.4%~98.0%;其中,核苷酸序列与加拿大分离物(MZ440726)和日本分离物(LC617945)一致性最高,与南非分离物(KJ524452)一致性最低,氨基酸序列与加拿大分离物(MZ440726)一致性最高,与意大利分离物(X75448、NC_003602)一致性最低。ORF2 的核苷酸和氨基酸序列一致性分别为62.5%~94.9%和57.6%~91.8%,核苷酸和氨基酸一致性都与日本分离物(LC617945)最高,核苷酸序列与加拿大分离物(MZ440724)一致性最低,氨基酸序列与加拿大分离物(MZ440727)和印度分离物(MT454065)一致性最低。MP 的核苷酸和氨基酸序列一致性分别为73.7%~95.5%和75.4%~95.1%,核苷酸序列与日本分离物(LC617945)和加拿大分离物(MZ440726)一致性最高,氨基酸序列与日本分离物(LC617945)和加拿大分离物(MZ440728)一致性最高,核苷酸和氨基酸序列都与加拿大分离物(MZ440727)一致性最低。CP 基因相对较保守,不同分离物之间的核苷酸和氨基酸序列一致性分别为78.0%~98.3%和87.3%~99.5%,核苷酸和氨基酸序列与日本分离物(LC617945)一致性均为最高,核苷酸序列与2 个加拿大分离物(MZ440725、MZ440724)和美国分离物(JX513897)一致性最低,氨基酸序列与美国分离物(MN716773)一致性最低。NABP 的核苷酸和氨基酸序列一致性分别为83.1%~97.6%和84.7%~98.4% ,核苷酸序列与加拿大分离物(MZ440726)一致性最高,与加拿大分离物(KY426923)一致性最低,氨基酸序列与加拿大分离物(MZ440726)和日本分离物(LC617945)一致性最高,与美国分离物(MN716773)一致性最低。
GVB Hohhot-2与22个GVB分离物全基因组核苷酸同源性为74.7%~96.4%。RdRp 的核苷酸与氨基酸序列与其他分离物的一致性分别为74.1%~96.4%和83.1%~97.6%。ORF2的核苷酸和氨基酸序列一致性分别为62.9%~94.5%和57.1%~90.6%。MP 的核苷酸和氨基酸序列一致性分别为73.2%~95.3%和74.5%~94.8%。CP的核苷酸和氨基酸序列一致性分别为77.2%~98.1%和86.8%~99.0%。NABP的核苷酸和氨基酸序列一致性分别为82.3%~97.3%和83.9%~96.0%,其中与22 个GVB 分离物的序列一致性最高和最低与GVB Hohhot-1 基本一致(表3~4)。
表3 GVB Hohhot-1 与其他22 个GVB 分离物核苷酸与氨基酸一致性
Table 3 The nucleotide and amino acid identities of GVB Hohhot-1 and 22 other GVB isolates
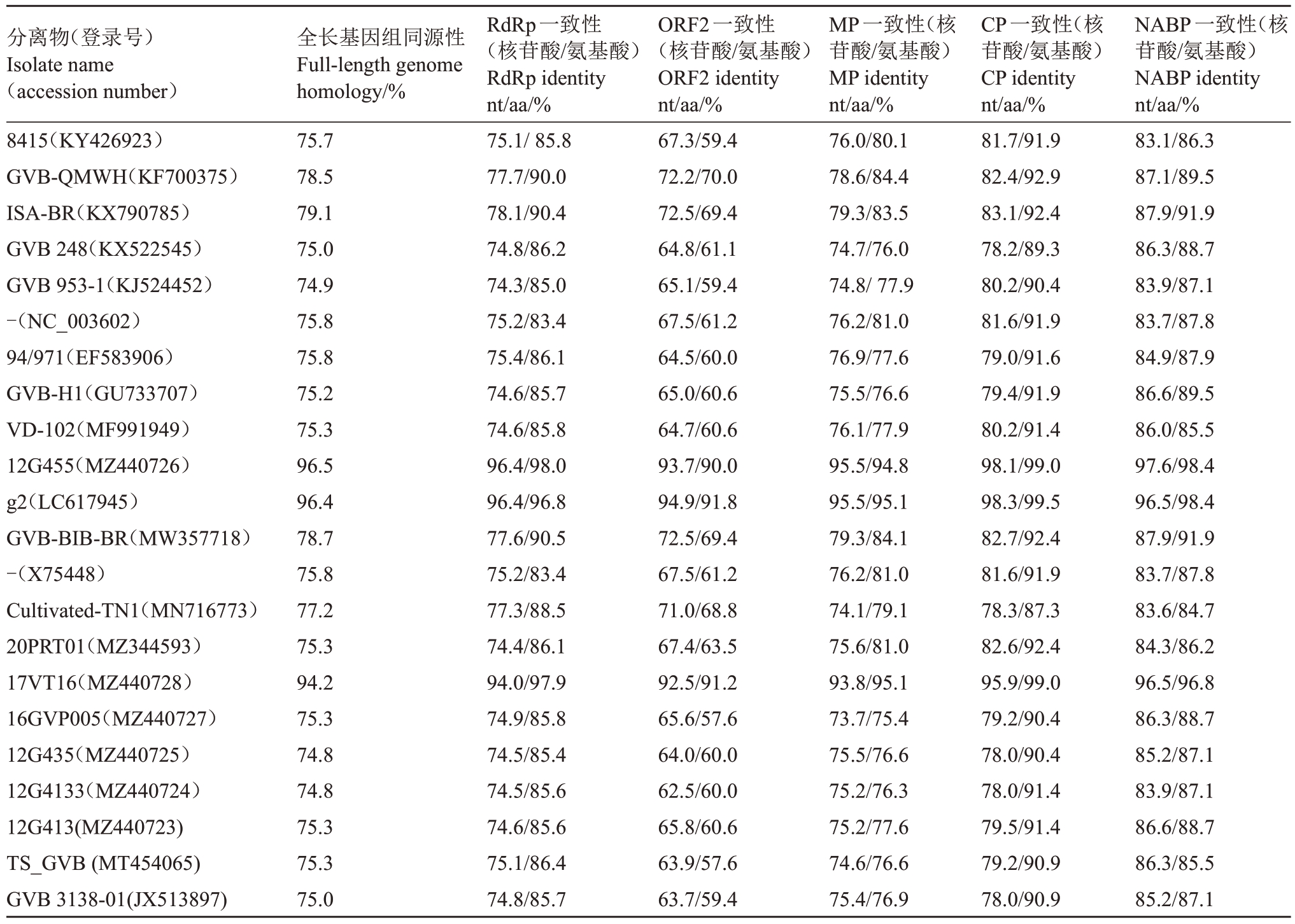
分离物(登录号)Isolate name(accession number)8415(KY426923)GVB-QMWH(KF700375)ISA-BR(KX790785)GVB 248(KX522545)GVB 953-1(KJ524452)-(NC_003602)94/971(EF583906)GVB-H1(GU733707)VD-102(MF991949)12G455(MZ440726)g2(LC617945)GVB-BIB-BR(MW357718)-(X75448)Cultivated-TN1(MN716773)20PRT01(MZ344593)17VT16(MZ440728)16GVP005(MZ440727)12G435(MZ440725)12G4133(MZ440724)12G413(MZ440723)TS_GVB(MT454065)GVB 3138-01(JX513897)全长基因组同源性Full-length genome homology/%75.7 78.5 79.1 75.0 74.9 75.8 75.8 75.2 75.3 96.5 96.4 78.7 75.8 77.2 75.3 94.2 75.3 74.8 74.8 75.3 75.3 75.0 RdRp一致性(核苷酸/氨基酸)RdRp identity nt/aa/%75.1/85.8 77.7/90.0 78.1/90.4 74.8/86.2 74.3/85.0 75.2/83.4 75.4/86.1 74.6/85.7 74.6/85.8 96.4/98.0 96.4/96.8 77.6/90.5 75.2/83.4 77.3/88.5 74.4/86.1 94.0/97.9 74.9/85.8 74.5/85.4 74.5/85.6 74.6/85.6 75.1/86.4 74.8/85.7 ORF2一致性(核苷酸/氨基酸)ORF2 identity nt/aa/%67.3/59.4 72.2/70.0 72.5/69.4 64.8/61.1 65.1/59.4 67.5/61.2 64.5/60.0 65.0/60.6 64.7/60.6 93.7/90.0 94.9/91.8 72.5/69.4 67.5/61.2 71.0/68.8 67.4/63.5 92.5/91.2 65.6/57.6 64.0/60.0 62.5/60.0 65.8/60.6 63.9/57.6 63.7/59.4 MP一致性(核苷酸/氨基酸)MP identity nt/aa/%76.0/80.1 78.6/84.4 79.3/83.5 74.7/76.0 74.8/77.9 76.2/81.0 76.9/77.6 75.5/76.6 76.1/77.9 95.5/94.8 95.5/95.1 79.3/84.1 76.2/81.0 74.1/79.1 75.6/81.0 93.8/95.1 73.7/75.4 75.5/76.6 75.2/76.3 75.2/77.6 74.6/76.6 75.4/76.9 CP一致性(核苷酸/氨基酸)CP identity nt/aa/%81.7/91.9 82.4/92.9 83.1/92.4 78.2/89.3 80.2/90.4 81.6/91.9 79.0/91.6 79.4/91.9 80.2/91.4 98.1/99.0 98.3/99.5 82.7/92.4 81.6/91.9 78.3/87.3 82.6/92.4 95.9/99.0 79.2/90.4 78.0/90.4 78.0/91.4 79.5/91.4 79.2/90.9 78.0/90.9 NABP一致性(核苷酸/氨基酸)NABP identity nt/aa/%83.1/86.3 87.1/89.5 87.9/91.9 86.3/88.7 83.9/87.1 83.7/87.8 84.9/87.9 86.6/89.5 86.0/85.5 97.6/98.4 96.5/98.4 87.9/91.9 83.7/87.8 83.6/84.7 84.3/86.2 96.5/96.8 86.3/88.7 85.2/87.1 83.9/87.1 86.6/88.7 86.3/85.5 85.2/87.1
2.3 GVB 2个分离物的系统进化树分析
对24个GVB分离物全基因组序列构建系统进化树,以葡萄病毒A(Grapevine virus A,GVA)(登录号:DQ787959)为外参基因。结果显示,供试24个GVB分离物共分为2个大组。本试验得到的2个GVB内蒙古分离物与美国(MN716773)、巴西(KX790785、MW357718)、中国(KF700375)、加拿大(MZ440726、MZ440728)、日本(LC617945)的GVB分离物聚为一支,并且与来自加拿大(MZ440726)的GVB分离物亲缘关系最近;来自加拿大(MZ440727、MZ440725、MZ440724、MZ440723、KY426923、MZ344593)、南非(GU733707、EF583906、KX522545、KJ524452)、印度(MT454065)、克 罗 地 亚(MF991949)、美 国(JX513897)和意大利(NC_003602、X75448)的GVB分离物聚为一支(图3)。

图3 基于24 个GVB 全长基因组序列构建的系统进化树
Fig.3 A phylogenetic tree based on complete genome of 24 GVB isolates
将24 个GVB 分离物的CP 基因序列构建进化树,以GVA(登录号:DQ787959)为外参基因。结果显示,24 个GVB 分离物共分为4 组。本试验获得的2 个GVB 内蒙古分离物与来自加拿大(MZ440726、MZ440728)、日本(LC617945)的分离物聚为一支,其中GVB Hohhot-1 和GVB Hohhot-2与来自加拿大(MZ440726)的分离物亲缘关系最近;来自巴西(KX790785、MW357718)的2 个分离物分为一支;来自中国(KF700375)的分离物单独分为一支;来自加拿大(KY426923、MZ440727、MZ440725、MZ440724、MZ440723、MZ344593)、南非(EF583906、GU733707、KX522545、KJ524452)、克罗地亚(MF991949)、美国(JX513897、MN716773)、印度(MT454065)和意大利(NC_003602、X75448)的分离物分为一支(图4)。

图4 基于24 个GVB CP 基因组序列构建的系统进化树
Fig.4 A phylogenetic tree based on CP genome of 24 GVB isolates
2.4 GVB分离物重组分析
通过RDP4 软件对包括GVB Hohhot-1 和GVB Hohhot-2 在内的共24 条GVB 全长基因组序列 进 行 重 组 分 析,经 RDP、GENECONV、BOOTSCAN、MACCHI、CHIMAERA、SISCAN、3SEQ 7 种方法在24 条GVB 全长基因组序列中均没有检测到重组事件。
3 讨 论
本研究通过RT-PCR 方法并结合RACE 技术成功获得了2个GVB全长基因组。截至目前,在我国仅仅报道了1 个GVB 中国分离物的全基因组序列(表2),该分离物报道于辽宁省兴城市[20]。本试验获得的2 个GVB 内蒙古分离物与我国已报道的中国分离物聚为一支,且与南非(EF583906、GU733707)、美国(JX513897)和意大利(X75448)分离物共处一支。通过表3和表4可以看出,ORF2在核苷酸和氨基酸水平上与其他分离物序列一致性最低,变异幅度最大,这与Goszczynski[8]和Hu 等[20]的研究结果一致。通过24 个GVB 分离物的CP 基因序列一致性比对结果发现,CP基因转录氨基酸序列有较高的同源性,该结果与Shi等[7]、Goszczynski[8]和Hu等[20]研究结果一致。但是,系统发育分析证明24个GVB分离物基于全基因组序列与CP基因序列分别构建进化树有一定差距,我国(KF700375)的分离物单独分为一支,确切原因还不能确定。此外,GVB Hohhot-1和GVB Hohhot-2分离物与来源于加拿大分离物(MZ440724)的基因组核苷酸一致性最低(74.7%、74.8%),却与加拿大分离物(MZ440726)基因组核苷酸一致性最高(96.5%、96.4%)的结果,以及系统进化树分成的2个大组中分别聚集的分离物都证明了GVB不具有地域分布规律。
表4 GVB Hohhot-2 与其他22 个GVB 分离物核苷酸与氨基酸一致性
Table 4 The nucleotide and amino acid identities of GVB Hohhot-2 and 22 other GVB isolates

分离物(登录号)Isolate name(accession number)8415(KY426923)GVB-QMWH(KF700375)ISA-BR(KX790785)GVB 248(KX522545)GVB 953-1(KJ524452)-(NC_003602)94/971(EF583906)GVB-H1(GU733707)VD-102(MF991949)12G455(MZ440726)g2(LC617945)GVB-BIB-BR(MW357718)-(X75448)Cultivated-TN1(MN716773)20PRT01(MZ344593)17VT16(MZ440728)16GVP005(MZ440727)12G435(MZ440725)12G4133(MZ440724)12G413(MZ440723)TS_GVB(MT454065)GVB 3138-01(JX513897)全长基因组同源性Full-length genome homology/%75.6 78.3 78.8 75.0 74.8 75.8 75.6 75.1 75.2 96.4 96.2 78.4 75.8 77.0 75.3 94.0 75.1 74.8 74.7 75.0 75.1 74.9 RdRp一致性(核苷酸/氨基酸)RdRp identity nt/aa/%75.1/85.5 77.6/89.7 78.0/90.1 74.8/86.0 74.1/84.7 75.2/83.1 75.2/85.8 74.5/85.4 74.5/85.4 96.3/97.6 96.4/96.4 77.5/90.2 75.2/83.1 77.2/88.2 74.5/85.8 93.8/97.5 74.9/85.5 74.4/85.1 74.4/85.2 74.4/85.4 74.9/86.1 74.7/85.3 ORF2一致性(核苷酸/氨基酸)ORF2 identity nt/aa/%67.7/60.6 71.6/68.8 72.0/68.8 65.2/60.5 65.9/59.4 68.1/61.8 64.1/59.4 65.4/60.0 65.1/60.0 93.3/89.4 94.5/90.6 72.0/68.8 68.1/61.8 70.4/68.2 67.8/63.5 92.2/90.6 65.6/57.1 64.4/59.4 62.9/59.4 66.2/60.0 64.1/57.6 64.0/58.8 MP一致性(核苷酸/氨基酸)MP identity nt/aa/%75.8/78.8 77.8/82.2 78.5/82.6 74.6/75.4 75.0/77.3 75.9/79.1 76.8/77.6 75.0/75.1 76.0/77.6 95.3/94.8 94.9/93.8 78.5/83.2 75.9/79.1 73.8/78.5 75.1/79.4 93.5/94.4 73.2/74.5 75.5/75.7 75.3/75.7 74.7/76.0 74.5/76.0 75.4/76.0 CP一致性(核苷酸/氨基酸)CP identity nt/aa/%80.9/90.9 81.4/91.9 82.4/91.4 77.5/88.3 79.7/89.8 80.7/90.9 79.0/90.6 78.5/90.9 79.5/90.4 98.1/99.0 97.6/98.5 82.1/91.4 80.7/90.9 77.7/86.8 81.6/91.4 95.6/99.0 78.7/89.3 77.2/89.3 77.3/90.4 78.7/90.4 79.2/89.8 77.2/89.8 NABP一致性(核苷酸/氨基酸)NABP identity nt/aa/%82.3/83.9 86.3/87.1 87.1/89.5 84.9/87.1 83.1/85.5 82.9/85.4 84.1/86.3 85.8/87.9 85.2/83.9 97.3/96.0 96.2/96.0 87.1/89.5 82.9/85.4 83.3/83.9 84.0/85.4 96.2/94.4 85.5/87.1 84.4/85.5 83.1/85.5 85.8/87.1 85.5/85.5 84.4/85.5
通过观察已报道的GVB 分离物基因组序列比对结果发现了短而连续的区域,这些区域的氨基酸序列显示出该病毒变种之间极显著的差异,而且在Goszczynski[8]的研究中进一步证明了ORF5 编码的RNA 结合蛋白在这些区域的氨基酸序列中发生了置换,这些氨基酸替换对于病毒的变异都有着巨大影响。病毒的自然变异是一个很缓慢的过程,如果改变环境或受到刺激将会加速其变异,而且葡萄大部分都是通过嫁接或无性繁殖方式进行繁殖,嫁接在加快其繁殖效率的同时,也常常会导致病毒的迅速传播,这都可能是GVB变异的原因。而且地理位置的差异是否会导致GVB的变异仍不能确定,这也许是不同国家分离株适应不同环境的结果,因此下一步还可以深入研究GVB基因组变异及致病性,探讨防控新策略和新方法,以减轻GVB对我国葡萄产业的危害,促进我国葡萄产业健康优质发展。
Davino 等[21]对ORF5 采用农杆菌注射法进行功能验证,证明GVB 的ORF5 是一个沉默抑制子,但内容还不够全面,因此可以深入研究GVB ORF5的基因沉默功能及形式功能的具体区域。对于植物来说,基因沉默是普遍存在的抗病毒防御机制,在世界上已发现的基因沉默抑制子包括苹果褪绿叶斑病毒(Apple chlorotic leaf spot virus,ACLSV)的运动蛋白P50[22]、黄 瓜 花 叶 病 毒(Cucumber mosaic virus,CMV)编码的2b 蛋白[23],草莓镶脉病毒(Strawberry vein band virus,SVBV)P6[24]、番茄丛矮病毒(Tomato bushy stunt virus,TBSV)编码的p19[25]等,因此深入研究ORF5 的基因沉默功能有助于深入认识GVB ORF5 抵御寄主植物的防御机制,对解析寄主植物和双链DNA 病毒之间防御和反防御机理均具有重要意义。
4 结 论
2个GVB内蒙古分离物基因组全长均为7609 nt,含有5个ORF,与22个已报道的GVB分离物全基因核苷酸一致性分别为74.9%~96.5%和74.8%~96.4%,24 个GVB 分离物基于全基因组序列与CP基因序列构建的进化树分别分成2组和4组,与加拿大分离物(登录号:MZ440726)的亲缘关系最近,在24 个GVB 全长基因组序列间无重组事件。本研究为我国GVB分类地位、病毒进化规律以及后续开展侵染性克隆奠定了一定基础。
[1] 朱红娟,胡国君,范旭东,张尊平,任芳,董雅凤.我国部分地区沙地葡萄茎痘相关病毒分离物外壳蛋白序列变异分析[J].植物保护学报,2014,41(2):169-175.ZHU Hongjuan,HU Guojun,FAN Xudong,ZHANG Zunping,REN Fang,DONG Yafeng.Genetic variation analysis of the coat protein of Grapevine rupestris stem pitting associated virus isolates from China[J].Journal of Plant Protection,2014,41(2):169-175.
[2] 王泽琼.引起葡萄皱木复合症的两种线形病毒的分子特性研究[D].武汉:华中农业大学,2011.WANG Zeqiong.Study on molecular characterization of two filamentous viruses associated with grapevine rugose wood complex[D].Wuhan:Huazhong Agricultural University,2011.
[3] 胡国君,董雅凤,张尊平,范旭东,任芳.基于巢式反转录-聚合酶链式反应技术检测葡萄病毒A 和葡萄病毒B[J].植物保护学报,2019,46(4):937-938.HU Guojun,DONG Yafeng,ZHANG Zunping,FAN Xudong,REN Fang.Identification of Grapevine virus A and Grapevine virus B by using nested reverse transcription-polymerase polymerase chain reaction[J].Journal of Plant Protection,2019,46(4):937-938.
[4] CHITARRA W,CUOZZO D,FERRANDINO A,SECCHI F,PALMANO S,PERRONE I,BOCCACCI P,PAGLIARANI C,GRIBAUDO I,MANNINI F,GAMBINO G.Dissecting interplays between Vitis vinifera L.and grapevine virus B(GVB)under field conditions[J].Molecular Plant Pathology,2018,19(12):2651-2666.
[5] NAKAUNE R,INOUE K,NASU H,KAKOGAWA K,NITTA H,IMADA J,NAKANO M.Detection of viruses associated with rugose wood in Japanese grapevines and analysis of genomic variability of Rupestris stem pitting-associated virus[J].Journal of General Plant Pathology,2008,74(2):156-163.
[6] HU G J,DONG Y F,ZHANG Z P,FAN X D,FANG R,ZHU H J.Detection and sequence analysis of grapevine virus B isolates from China[J].Acta Virologica,2014,58(2):180-184.
[7] SHI B J,HABILI N,GAFNY R,SYMONS R H.Extensive variation of sequence within isolates of grapevine virus B^+[J].Virus Genes,2004,29(2):279-285.
[8] GOSZCZYNSKI D E.Divergent molecular variants of Grapevine virus B (GVB) from corky bark (CB)-affected and CB-negative LN33 hybrid grapevines[J].Virus Genes,2010,41(2):273-281.
[9] 任芳,张尊平,范旭东,胡国君,李正男,董雅凤.葡萄病毒B CP 基因植物表达载体构建及烟草遗传转化[J].植物保护,2018,44(3):11-16.REN Fang,ZHANG Zunping,FAN Xudong,HU Guojun,LI Zhengnan,DONG Yafeng.Construction of a plant expression vector of the Grapevine virus B coat protein gene and transformation into Nicotiana tabacum[J].Plant Protection,2018,44(3):11-16.
[10] KUMAR S,SINGH L,FERRETTI L,BARBA M,ZAIDI A A.,HALLAN V.Evidence of Grapevine leafroll associated virus-1-3,Grapevine fleck virus and Grapevine virus B Occurring in Himachal Pradesh,India[J].Indian Journal of Virology,2013,24(1):66-69.
[11] 刘永清,王国平.葡萄病毒种类调查与检测技术研究[J].中外葡萄与葡萄酒,2004(4):27-28.LIU Yongqing,WAMG Guoping.Techniques for detecting grapevine virus[J].Sino-Overseas Grapevine &Wine,2004(4):27-28.
[12] 刘晓,BOSCIA D,RAIMONDI T,BROGGIO M,陈建,李洪雯,王建辉,刘建军.四川葡萄病毒病田间普查及血清学鉴定[J].西南农业学报,2004,17(1):52-56.LIU Xiao,BOSCIA D,RAIMONDI T,BROGGIO M,CHEN Jian,LI Hongwen,WANG Jianhui,LIU Jianjun.Field investigation and serological detection of grapevine viruses in Sichuan Province[J].Southwest China Journal of Agricultural Sciences,2004,17(1):52-56.
[13] 王国平,洪霓,张尊平,张少瑜,姜修风.辽宁、山东葡萄与核果病毒病的田间调查[J].中国果树,1996(4):39-41.WANG Guoping,HONG Ni,ZHANG Zunping,ZHANG Shaoyu,JIANG Xiufeng.Field investigation of grape and stone fruit virus diseases in Liaoning and Shandong[J].China Fruits,1996(4):39-41.
[14] ADAMS M J,ANTONIW J F,BAR-JOSEPH M,BRUNT A A,CANDRESSE T,FOSTER G D,MARTELLI G P,MILNE R G,ZAVRIEV S K,FAUQUET C M.The new plant virus family Flexiviridae and assessment of molecular criteria for species demarcation[J/OL].Archives of Virology,2004,149(8):1672.
[15] 王泽琼,洪霓,王国平.葡萄病毒B 的检测与分子鉴定[C]//中国植物病理学会2006 年学术年会论文集.北京:中国科学技术出版社,2006:206-207.WANG Zeqiong,HONG Ni,WANG Guoping.Detection of Grapevine virus B and its molecular identification[C]// Proceedings of 2006 Academic Annual Meeting of Chinese Society of Plant Pathology.Beijing:Science and Technology of China Press,2006:206-207.
[16] MARTELLI G P,CANDRESSE T,NAMBA S. Trichovirus,a new genus of plant viruses[J].Archives of Virology,1994,134(3/4):451-455.
[17] KUNIYUKI H,GIORIA R,REZENDE J,DE WILLINK C G,NOVO J P S,YUKI V A.Transmission of the grapevine virus B by the mealybug pseudococcus longispinus targioni-tozzetti(hemiptera:pseudococcidae) in Brazil[J].Summa Phytopathologica,2006,32(2):151-155.
[18] 刘晓萌,李小燕,张斌,张磊,孙平平,李正男.侵染葡萄的葡萄病毒属病毒研究进展[J].中国果树,2021(8):12-18.LIU Xiaomeng,LI Xiaoyan,ZHANG Bin,ZHANG Lei,SUN Pingping,LI Zhengnan.Advances in Vitivirus research members of the genus Vitivirus[J].China Fruits,2021(8):12-18.
[19] 闫雨婷.内蒙古自治区葡萄病毒鉴定和葡萄灰比诺病毒内蒙古分离物全基因组序列分析[D].呼和浩特:内蒙古农业大学,2021.YAN Yuting.Identification of grapevine viruses in Inner Mongolia autonomous region and full-genome sequence analysis of grapevine pinot Gris virus Inner Mongolia isolate[D].Hohhot:Inner Mongolia Agricultural University,2021.
[20] HU G J,DONG Y F,ZHANG Z P,FAN X D,REN F,ZHU H J.Complete nucleotide sequence of a new isolate of Grapevine virus B from China[J].Journal of Plant Pathology,2014,96(2):403-406.
[21] DAVINO S,RUIZ-RUIZ S,SERRA P,FORMENT J,FLORES R.Revisiting the cysteine-rich proteins encoded in the 3'-proximal open reading frame of the positive-sense single-stranded RNA of some monopartite filamentous plant viruses:Functional dissection of p15 from grapevine virus B[J].Archives of Virology,2020,165(10):2229-2239.
[22] YAEGASHI H,TAMURA A,ISOGAI M,YOSHIKAWA N.Inhibition of long-distance movement of RNA silencing signals in Nicotiana benthamiana by Apple chlorotic leaf spot virus 50 kDa movement protein[J].Virology,2008,382(2):199-206.
[23] DONG K,WANG Y,ZHANG Z,CHAI L X,TONG X,XU J,LI D W,WANG X B.Two amino acids near the N-Terminus of Cucumber mosaic virus 2b play critical roles in the suppression of RNA silencing and viral infectivity[J].Molecular Plant Pathology,2016,17(2):173-183.
[24] 冯明峰.草莓镶脉病毒RNA 沉默抑制子功能分析[D].合肥:安徽农业大学,2016.FENG Mingfeng.Function analysis of suppressor of RNA silencing encoded by strawberry vein banding virus[D].Hefei:Anhui Agricultural University,2016.
[25] 吴根土.两种植物病毒编码的RNA 沉默抑制子在寄主体内的功能研究[D].杭州:浙江大学,2014.WU Gentu.Function analysis of two viral suppressors of RNA silencing in hosts[D].Hangzhou:Zhejiang University,2014.