蔷薇科植物在进化过程中为了避免自花授粉,普遍存在自交不亲和现象[1]。研究表明其自交不亲和由雌蕊S-RNase基因和花粉SFB(S haplotype specific F-box gene)或SFBB(S locus F-box brothers)基因相互作用实现[2]。有研究显示苹果亚科(Maloideae)梨属(Pyrus)植物砂梨[3](Pyrus pyrifolia)、新疆梨[3](P.sinkiangensis)、西洋梨[3](P.communis)、秋子梨[3](P. ussuriensis)、白梨(P. bretschneideri)[3]、野生梨(P. pyraster)[4],苹果属(Malus)植物苹果(M. domestica)[5]、野苹果(M. sylvestris)[5]、新疆野苹果(M.sieversii)[5]、窄叶海棠(M. angustifolia)[5]、楸子(M.prunifolia)[5]、陇东海棠(M. kansuensis)[5]、锡金海棠(M.sikkimensis)[5]、毛山荆子(M.mandshurica)[5]、海棠(M.spectabilis)[5]、变叶海棠(M.toringoides)[5]、花叶海棠(M.transitoria)[5]、山荆子(M.baccata)[6]、丽江山荆子(M.rockii)[6]、锡金海棠(M.sikkimensis)[6]、八棱海棠(M. micromalus)[6]、杂种海棠(M. hybrid)[7],花楸属(Sorbus)植物欧亚花楸(S.aucuparia)[8],山楂属(Crataegus)植物山楂(C.pinnatifida)[9]、单子山楂(C. monogyna)[10],枇杷属(Eriobotrya)植物枇杷(E.japonica)[11]已克隆出了S-RNase基因;李亚科(Prunoideae)李属(Prunus)植物李(P.salicina)[12]、扁桃(P.dulcis)[12]、杏(P.armeniaca)[12]、梅(P.mume)[12]、甜樱桃(P. avium)[12]、酸樱桃(P. cerasus)[12]、大岛樱(P.speciosa)[12]、樱桃(P. pseudocerasus)[13]以及桃(P.persica)[14]也克隆出了S-RNase基因。
蔷薇科S-RNase 基因的分离技术已经比较成熟,其作用机制和进化研究也已经比较深入[1-14],但蔷薇科花粉自交不亲和控制基因的研究进展较慢。花粉自交不亲和基因研究最先在李亚科植物中取得实质性进展。Ushijima等[15]从扁桃中分离出在花粉中特异表达且具有较高序列多态性的SFB基因,Entani 等[16]从梅中分离出了具有较高序列多态性在花粉中特异表达的SLF(S-locus F-box)基因,比较发现二者是同一基因。研究者通过自交亲和的甜樱桃、扁桃品种与自交不亲品种进行对比发现,自交亲和品种的SFB 基因发生变异从而无法编码完整蛋白,从侧面印证了SFB是花粉自交不亲和决定基因[17-19]。
蔷薇科苹果亚科梨属花粉自交不亲和基因研究进展缓慢。1993 年Sassa 等[20]研究发现控制梨雌蕊和花粉的自交不亲和基因是不同的基因,2007 年Sassa等[21]才从苹果和日本梨中分离出MdSFBB3-a、PpSFBB4-a 等10 个花粉自交不亲和相关SFBB 基因。2010 年Franceschi 等[22]从2 个西洋梨品种中分离出SFBBα-AF1、SFBBα-MRB1等15个SFBB基因,并对其在染色体上位置以及连锁情况进行了研究,二者在研究中均发现梨S基因座包含多个花粉相关基因。2009年国内学者张琳等[23]、2010年乌云塔娜等[24]先后从中国梨品种中分离出新的SFBB-gamma基因,并分析发现该基因具有高度的同源性。2015年Aguiar等[14]以及2019年Claessen等[25]对梨自交不亲和机制进行研究,发现其不同于李亚科自交不亲和单一花粉S 基因的模式。截至目前,梨属植物砂梨、白梨、秋子梨、新疆梨以及西洋梨中均克隆出了SFBB 基因[20-24],但梨花粉SFBB 基因的实际数量和所起的作用尚不明确。为了研究梨SFBB基因序列变异、基因多态性以及进化特性等特征,笔者从3个梨品种中分离鉴定出一个新的梨SFBB 基因。同时,对所有已分离鉴定的能够和梨S-RNase 基因一一对应起来的SFBB 基因全长序列进行比较分析。新的梨花粉自交不亲和基因的克隆丰富了梨花粉自交不亲和基因的资源,对现有梨花粉自交不亲和相关基因的分析评价有助于进一步找出梨自交不亲和基因现象中起主导作用的花粉S基因。
1 材料和方法
1.1 试验材料
春季采摘云南宝珠梨(P.pyrifolia‘Yunnan baozhu’)、福安尖把梨(P. ussuriensns Maxim.‘Fu’an Jianba’)、皮胎梨(P. ussuriensns Maxim.‘Pitai’)的嫩叶置于液氮灌中带回备用。
1.2 试验方法
1.2.1 基因特异扩增 试剂材料有TaKaRa 公司的ExTaq、pMD18-T Vector,梨叶片基因组DNA 采用CTAB(Cetyltriethy lammonnium bromide)法提取备用。根据已有梨SFBB-gamma基因的保守性设计5’端特异引物PF2:5’-tgtgtaattcatgtgcatgg-3’,3’端包含终止密码子的反向引物PR:5’-tggaacgtttccctcaactc-3’。PCR条件:40 ng DNA,0.4µmol·L-1引物,200µmol·L-1 dNTP,1×PCR Buffer,1 U of ExTaq(TaKaRa),灭菌纯水加至20 μL,进行PCR 扩增:94 ℃变性30 个循环15 s,55 ℃退火15 s,72 ℃延伸1.5 min,最后在72 ℃延伸3 min。PCR产物被克隆到质粒载体中测序。
1.2.2 基因序列分析 利用DNAStar软件进行基因DNA序列分析,并利用NCBI进行同源性搜索分析。1.2.3 梨SFBB 基因和对应S-RNase 基因系统发育和序列分析 从NCBI中搜索已登录的能够相互对应的梨SFBB和S-RNase基因全长序列,找出其编码序列,利用MEGA 6.06软件自带程序分别进行多序列比对,并进行人工调整后采用邻接法(NJ)构建以1000 次Bootsrap 做可靠性验证的梨SFBB 基因和对应S-RNase 基因编码区的系统发育树,分别找出序列适用最优模型,借助模型和算法分别计算二者序列间的分化以及遗传多态性,对以上结果进行比较分析,用同样的方法对梨SFBB基因4个主要群体和对应的S-RNase基因进行分析。
2 结果与分析
2.1 梨新SFBB-gamma基因序列分析
据前人的研究[26-28],云南宝珠梨、福安尖把梨和皮胎梨品种的S 基因型分别为S22S42、S16S22和S22S43,设计的特异引物从3 个梨品种中均只能克隆出1 个大小为1227 bp 的基因,且3 个基因序列完全一致,该基因编码区序列大小为1191 bp,DNAstar MegAlign By jotun Hein Method显示编码的氨基酸序列和已有的梨SFBB-gamma 序列的同源率为96.0%~99.0%,最高为PpSFBB2-gamma(BAF79627.1),仅有4 处氨基酸的差异,最低的为SFBB21-gamma(ABZ81686.1)和SFBB28-gamma(ABZ81685.1),也仅有16 处氨基酸差异。3 个梨品种具有共同的S22-RNase 基因,因此将克隆的基因命名为SFBB22-gamma(EU081887.1)。推导梨SFBB22-gamma基因编码氨基酸序列符合梨SFBB-gamma基因共有的一级结构特征:编码区序列大小为1191 bp,编码396个氨基酸,ProtParam预测分子质量为45.47 ku,理论等电点4.67,酸性氨基酸残基总数为60,碱性氨基酸残基总数为35,表明其为酸性蛋白质;分子式C2053H3108N500O619S24;不稳定性指数为40.62,属于不稳定蛋白,脂肪系数为85.86,总平均亲水性为-0.160,为亲水蛋白。其中N 端包含由50 个氨基酸组成的F-box。蛋白质二级结构分析表明,该基因编码蛋白的β-折叠和线圈的比例都高于α-螺旋。蛋白质结构预测SFBB22-gamma 基因有5 个α-螺旋和24 个β-折叠,α-螺旋主要分布于F-box 区域,β-折叠则散布于整个蛋白质,和P.dulcis中SFBk蛋白预测的二级结构相似[29]。
2.2 梨SFBB基因和对应S-RNase基因数据分析
2.2.1 梨SFBB 基因和对应S-RNase 基因进化分析
目前,NCBI 数据库中共登录能和梨S-RNase 基因对应起来的SFBB 基因全长CDS 序列52 个,其中SFBB-alpha 15 个,SFBB-beta 10 个,SFBB-gamma 24 个,SFBB-epsilon 3 个,对应的梨S-RNase 基因全长CDS 序列共30 个。不同的梨SFBB 基因NJ 进化树分别聚成4个亚类(图1),而亚类内部除了epsilon均为西洋梨数据无法看出,其余亚类没有出现明显的以种为界限的聚类现象;对应梨S-RNase 基因也没有明显地以种为单位的聚类现象(图1),SFBB 4个亚类间的同源率为73.8%~99.9%;对应S-RNase间的同源率为76.0%~99.7%。二者进化树没有出现明显相似的聚类。

图1 梨SFBB 基因和对应S-RNase 基因的NJ 系统发育树
Fig.1 NJ phylogenetic tree of pear SFBB genes and corresponding S-RNase genes
若将梨SFBB基因4个亚类归为一个整体来看,则梨SFBB 编码区序列长1227 bp,包含409 个密码子,544 个变异位点,占比44.34%,683 个保守位点,466 个简约信息位点,78 个单突变位点。序列中不同核苷酸占比,A:30.67%、T:30.67%、C:19.33%、G:19.33%,A+T:61.34%,C+G:38.66%。转换和颠换的比植(R)为1.85。
对应梨S-RNase编码区序列长711 bp,包含237个密码子,有421 个变异位点,占比59.21%,287 个保守位点,345 个简约信息位点,76 个单突变位点。序列中不同核苷酸占比,A:32.66%、T:25.40%、C:21.87%、G:20.07%,A+T 占比58.06%,C+G 为41.94%。R植为0.95。
二者变异位点占比梨S-RNase 基因大于对应SFBB 基因,前者是后者的1.34 倍,R 植后者是前者的1.95倍,二者C+G、A+T含量相差不大。
2.2.2 梨SFBB 基因和对应S-RNase 基因序列分化分析 梨SFBB 基因和对应S-RNase 基因编码区序列所有碱基成对遗传距离分布见图2,梨属植物SFBB 基因编码区序列成对的平均遗传距离为0.147,而对应S-RNase 基因编码区序列成对的平均遗传距离为0.211,后者是前者的1.44倍。由图2可知,梨SFBB 基因遗传距离分布中0.000~0.030 的占比远高于S-RNase 在此范围的占比,这部分遗传距离是梨SFBB-alpha、SFBB-gamma 以及SFBB-epsilon 基因3 个不同的亚类内部的遗传距离。此外,SFBB 基因遗传距离占比最多的为>0.180~0.210,而S-RNase 占比最多的为>0.201~0.240,遗传距离较大部分占比(>0.240~0.330)的S-RNase 要比SFBB多。
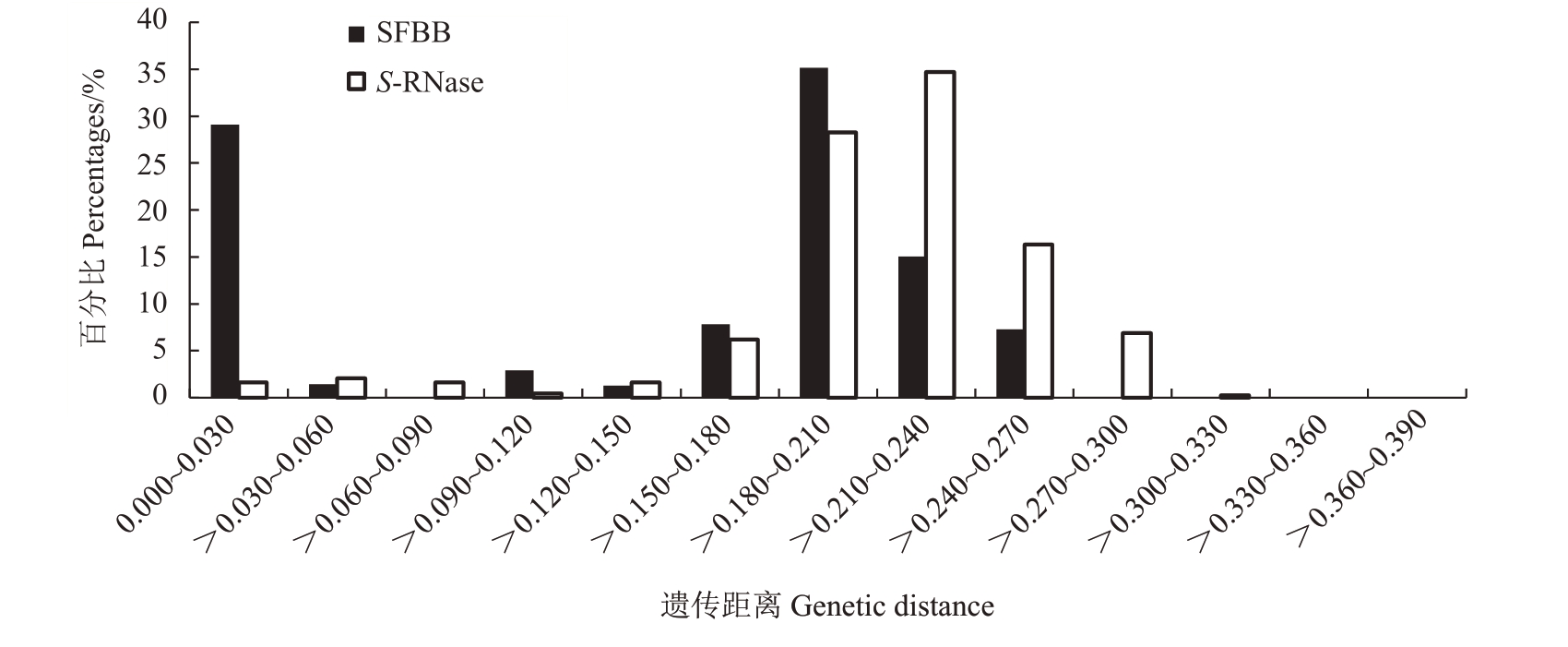
图2 梨SFBB 基因和对应S-RNase 基因编码区序列所有碱基成对遗传距离分布
Fig.2 Pairwise genetic distance of whole nucleotide substitutions in coding regions of pear SFBB gene and S-RNase gene
为了探寻影响梨属植物SFBB 基因和对应SRNase 基因的进化因素,利用MEGA 软件Selection程序分别估算二者密码子对应的非同义替换率与同义替换率的差植(dN-dS),结果见表1。dN-dS=0,序列之间分歧是中性选择的结果;dN-dS>0是正选择效应;dN-dS<0 是纯化选择的结果。由表1 可以看出,二者受正选择效应的位点占比接近,SFBB受中性选择的位点占比稍高于S-RNase,而受纯化选择的位点占比正好相反。SFBB 连续阳性位点数(至少2 个及以上的相邻位点dN-dS>0)是S-RNase 的1.74倍,但若考虑序列的长度因素,二者的连续阳性位点占比基本一致。
表1 梨SFBB 基因与S-RNase 基因核苷酸非同义突变率与同义突变率情况
Table 1 Nucleotide non-synonymous mutation rate and synonymous mutation rate of pear SFBB gene and S-RNase gene
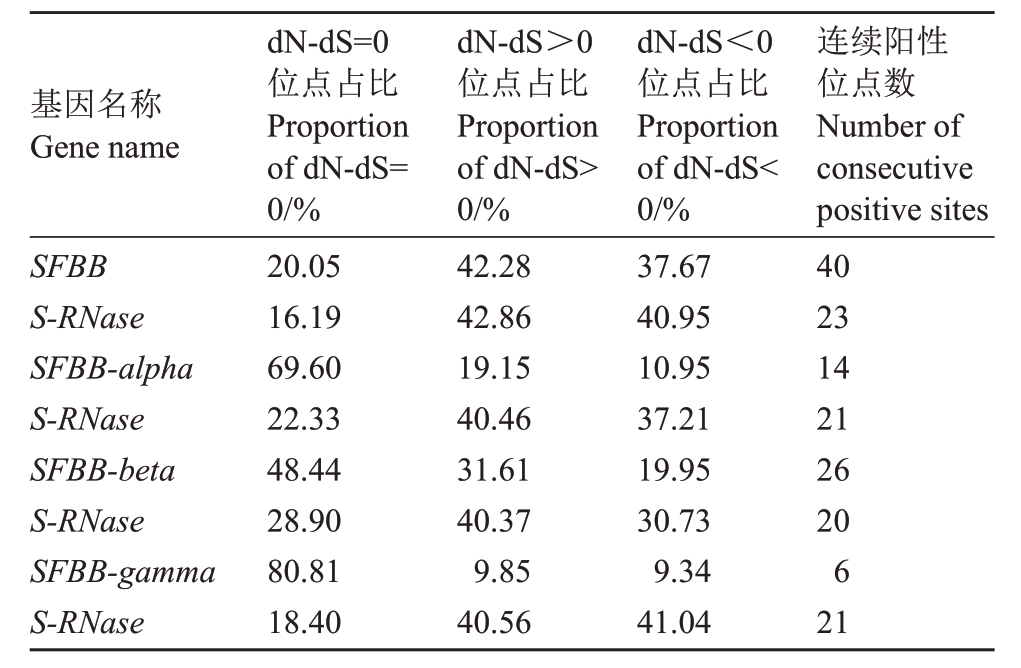
基因名称Gene name SFBB S-RNase SFBB-alpha S-RNase SFBB-beta S-RNase SFBB-gamma S-RNase dN-dS=0位点占比Proportion of dN-dS=0/%20.05 16.19 69.60 22.33 48.44 28.90 80.81 18.40 dN-dS>0位点占比Proportion of dN-dS>0/%42.28 42.86 19.15 40.46 31.61 40.37 9.85 40.56 dN-dS<0位点占比Proportion of dN-dS<0/%37.67 40.95 10.95 37.21 19.95 30.73 9.34 41.04连续阳性位点数Number of consecutive positive sites 40 23 14 21 26 20 6 21
2.2.3 梨SFBB 基因和对应S-RNase 基因位点的遗传多态性 梨SFBB 基因和对应S-RNase 基因编码区遗传多态性见表2,变异碱基位点由于受序列数的影响不能作为比较对象。从排除了序列数干扰能够反映遗传多态性的两个指标Θ 和π 数植来看,SRNase基因的二者的数植均大于SFBB基因,前者分别是后者的1.52 和1.43 倍,这一点也可以通过表中的S/m 反映出来,因此梨S-RNase 基因的遗传多态性高于SFBB基因。同时二者的Tajima检验统计量D均为正植,表明二者均受平衡选择的影响。
表2 梨SFBB 基因和对应S-RNase基因编码区遗传多态性
Table 2 Genetic polymorphisms of pear SFBB gene and corresponding S-RNase gene coding region
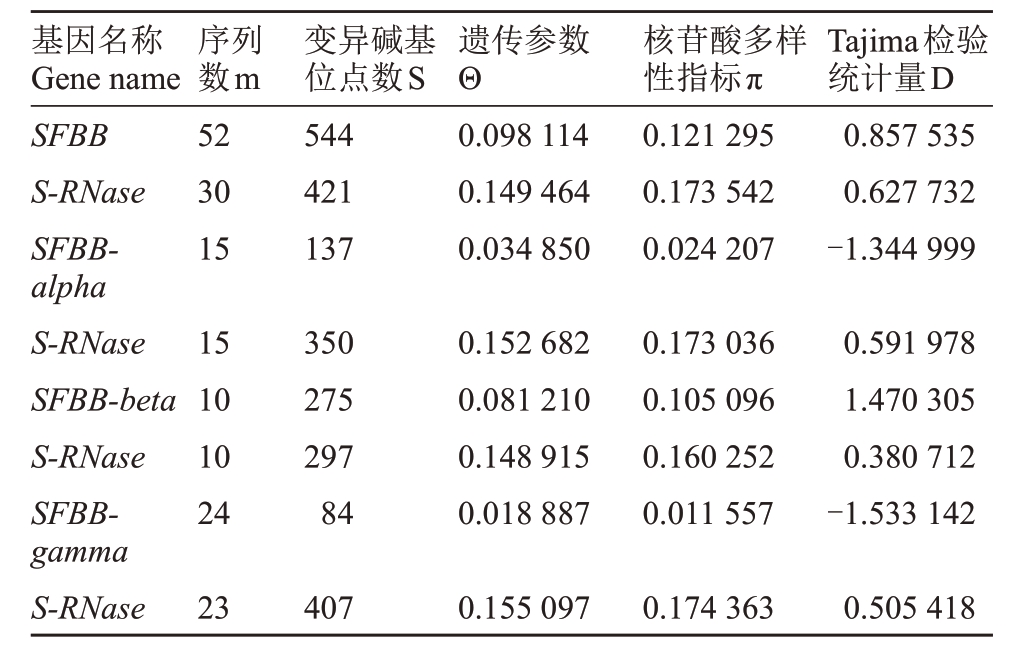
基因名称Gene name SFBB S-RNase SFBBalpha S-RNase SFBB-beta S-RNase SFBBgamma S-RNase序列数m 52 30 15 15 10 10 24 23变异碱基位点数S 544 421 137 350 275 297 84 407遗传参数Θ 0.098 114 0.149 464 0.034 850 0.152 682 0.081 210 0.148 915 0.018 887 0.155 097核苷酸多样性指标π 0.121 295 0.173 542 0.024 207 0.173 036 0.105 096 0.160 252 0.011 557 0.174 363 Tajima检验统计量D 0.857 535 0.627 732-1.344 999 0.591 978 1.470 305 0.380 712-1.533 142 0.505 418
2.3 梨SFBB-alpha 基因及对应S-RNase 基因特征分析
2.3.1 梨SFBB-alpha 基因及对应S-RNase 基因理化性质 用Protparam进行蛋白质理化性质预测,梨SFBB-alpha 基因编码蛋白平均由393 个氨基酸组成,平均分子质量45.53 ku,平均理论等电点5.01,平均酸性蛋白质残基总数为54,平均碱性蛋白质残基总数为38,表明其为酸性蛋白质,平均分子式C2069H3047N508O608S21,平均不稳定性指数为43.33,属于不稳定蛋白,平均脂肪系数为82.91,总平均亲水性为-0.219,为亲水蛋白。
对应的S-RNase 基因编码蛋白平均由228 个氨基酸组成,平均分子质量26.06 ku,平均理论等电点9.07,平均酸性蛋白质残基总数为17,平均碱性蛋白质残基总数为26,表明其为碱性蛋白质,平均分子式C1165H1817N315O336S15,平均不稳定性指数为38.53,属于稳定蛋白,平均脂肪系数为74.71,总平均亲水性为-0.451,为亲水蛋白。
二者比较显示SFBB-alpha 基因分子质量是SRNase基因的1.75倍,二者蛋白分别为酸性和碱性,理论上具有酸碱相互作用和异性电荷相吸引的化学和物理促进相互作用机制。蛋白质稳定性虽然相反,但二者预测平均数植相差不大,存在实际稳定性一致的可能性,亲水性方面均为亲水蛋白,二者的蛋白质脂肪系数均比较高,脂肪系数是一个蛋白质中脂肪侧链所占的相对植,包括异亮氨酸、亮氨酸、丙氨酸、缬氨酸所占的比重,常作为蛋白质热稳定性增加的阳性因素,说明二者蛋白热稳定性近似。
2.3.2 梨SFBB-alpha基因及对应S-RNase基因进化分析 梨SFBB-alpha 基因及对应S-RNase 基因NJ进化树见图3,二者对应的基因在自己的进化树中拓扑结构不一致。

图3 梨SFBB-alpha 基因和对应S-RNase 基因的NJ 系统发育树
Fig.3 NJ phylogenetic tree of pear SFBB-alpha genes and corresponding S-RNase gene
梨SFBB-alpha 基因编码区序列长1209 bp,有403 个密码子,137 个变异位点,占比11.33%,1057个保守位点,45 个简约信息位点,87 个单突变位点。序列中不同核苷酸占比,A:30.53%、T:30.53%、C:19.47%、G:19.47%,A+T 占比61.06%,C+G 为38.94%。R为1.81。
对应的梨S-RNase编码区序列长705 bp,235个密码子,350个变异位点,占比49.65%,346个保守位点,256个简约信息位点,94个单突变位点。序列中核苷酸占比,A:29.16%、T:29.16%、C:20.84%、G:20.84%,A+T 占比58.32%,C+G 为41.68%。R 为0.86。
二者变异位点占比梨S-RNase 基因大于SFBBalpha 基因,前者是后者的4.38 倍,R 植后者是前者的2.11倍,C+G、A+T含量相差不大。
2.3.3 梨SFBB-alpha基因和对应S-RNase基因序列分化分析 梨属植物SFBB-alpha 和对应的梨SRNase基因编码区序列成对的遗传距离计算结果如图4所示。SFBB-alpha总平均遗传距离0.025,考虑所有碱基替换分布情况:>0.015~0.020、>0.020~0.025、>0.025~0.030 分别占比29.52%、25.71%和20.95%、其他>0.010~0.015 占比8.57%、>0.050~0.055 占比9.52%,其余占比不足5%。对应的梨SRNase总平均遗传距离0.208,是对应SFBB-alpha基因平均遗传距离的8.32 倍,>0.150~0.200、>0200~0.250、>0.250~0.300 分别占比25.74%、43.81%、20.95%,其余占比不足10%,图形分布和SFBB-alpha相差较大。
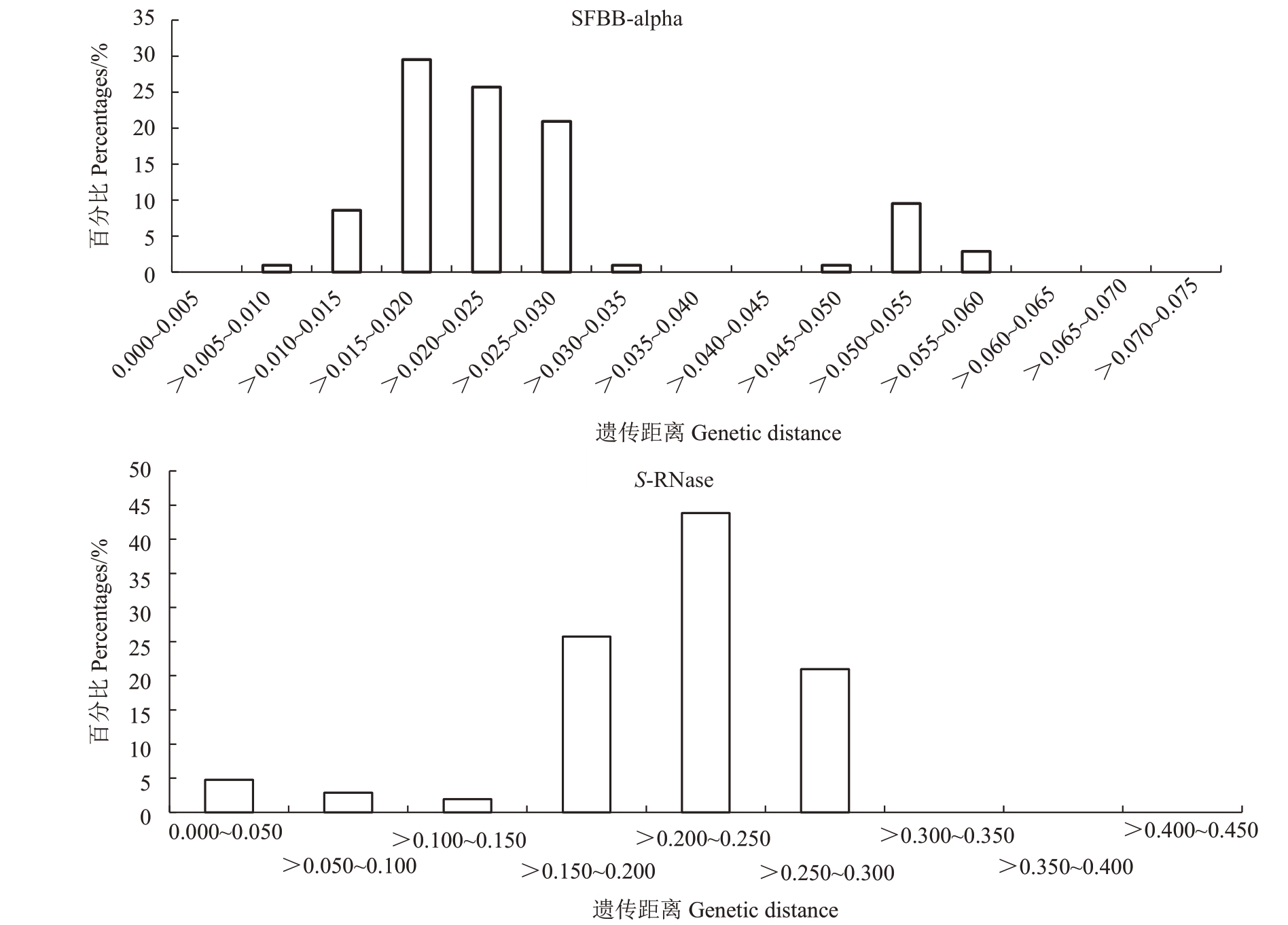
图4 梨SFBB-alpha 基因和对应S-RNase 基因编码区序列所有碱基成对遗传距离分布
Fig.4 Pairwise genetic distance of whole nucleotide substitutions in coding regions of pear SFBB-alpha genes and S-RNase genes
梨SFBB-alpha基因和对应S-RNase基因所有密码子对应dN-dS 结果见表1。由表1 可以看出二者受正选择效应的位点SFBB-alpha是S-RNase的1/2,受中性选择的位点SFBB-alpha 则是S-RNase 的3倍,而受纯化选择的位点SFBB-alpha 则是S-RNase的1/3。连续阳性位点数SFBB-alpha 是S-RNase 的2/3。
2.3.4 梨SFBB-alpha基因和对应S-RNase基因位点的遗传多态性 梨属不同植物SFBB-alpha 和对应S-RNase 基因位点的遗传分化情况结果如表2 所示。从反映遗传多态性的两个指标Θ 和π 数植来看,梨S-RNase 基因二者的数植均远远大于对应的SFBB-alpha 基因,前者分别是后者的4.38 和7.16倍,因此S-RNase 基因的遗传多态性高于SFBB 基因。同时二者的Tajima 检验统计量D 一正一负,表明二者受不同选择的影响。
2.4 梨SFBB-beta 基因及对应S-RNase 基因特征分析
2.4.1 梨SFBB-beta 基因及对应S-RNase 基因理化性质 梨SFBB-beta基因编码蛋白质理化性质预测显示其由395个氨基酸组成,平均分子质量45.62 ku,平均理论等电点5.32,平均酸性氨基酸残基总数为52,平均碱性氨基酸残疾总数为37,表明其为酸性蛋白质,平均分子式C2059H3135N521O605S22,平均不稳定系数为46.26,属不稳定蛋白,平均脂肪系数为87.84,总平均亲水性为-0.200,为亲水蛋白。对应SRNase基因编码蛋白质理化性质预测结果显示其由228 个氨基酸组成,平均分子质量26.07 ku,平均理论等电点9.08,平均酸性氨基酸残基总数17,平均碱性氨基酸残基总数25,表明其为碱性蛋白质,平均分子式C1169H1820N316O334S14,平均不稳定系数为41.32,属于不稳定蛋白,平均脂肪系数为76.00,总平均亲水性为-0.436,为亲水蛋白。
梨SFBB-beta基因编码蛋白平均分子质量是SRNase 基因编码蛋白的1.75 倍,二者蛋白分别为酸性和碱性,理论上二者具有酸碱相互作用和异性电荷相吸引的化学和物理促进相互作用机制。蛋白不稳定系数相近,且均属于不稳定蛋白,亲水性方面均为亲水蛋白,蛋白热稳定性近似。
2.4.2 梨SFBB-beta 基因及对应S-RNase 基因进化分析 梨SFBB-beta基因及对应S-RNase基因NJ进化树见图5,二者对应的基因在自己的进化树中拓扑结构不一致。
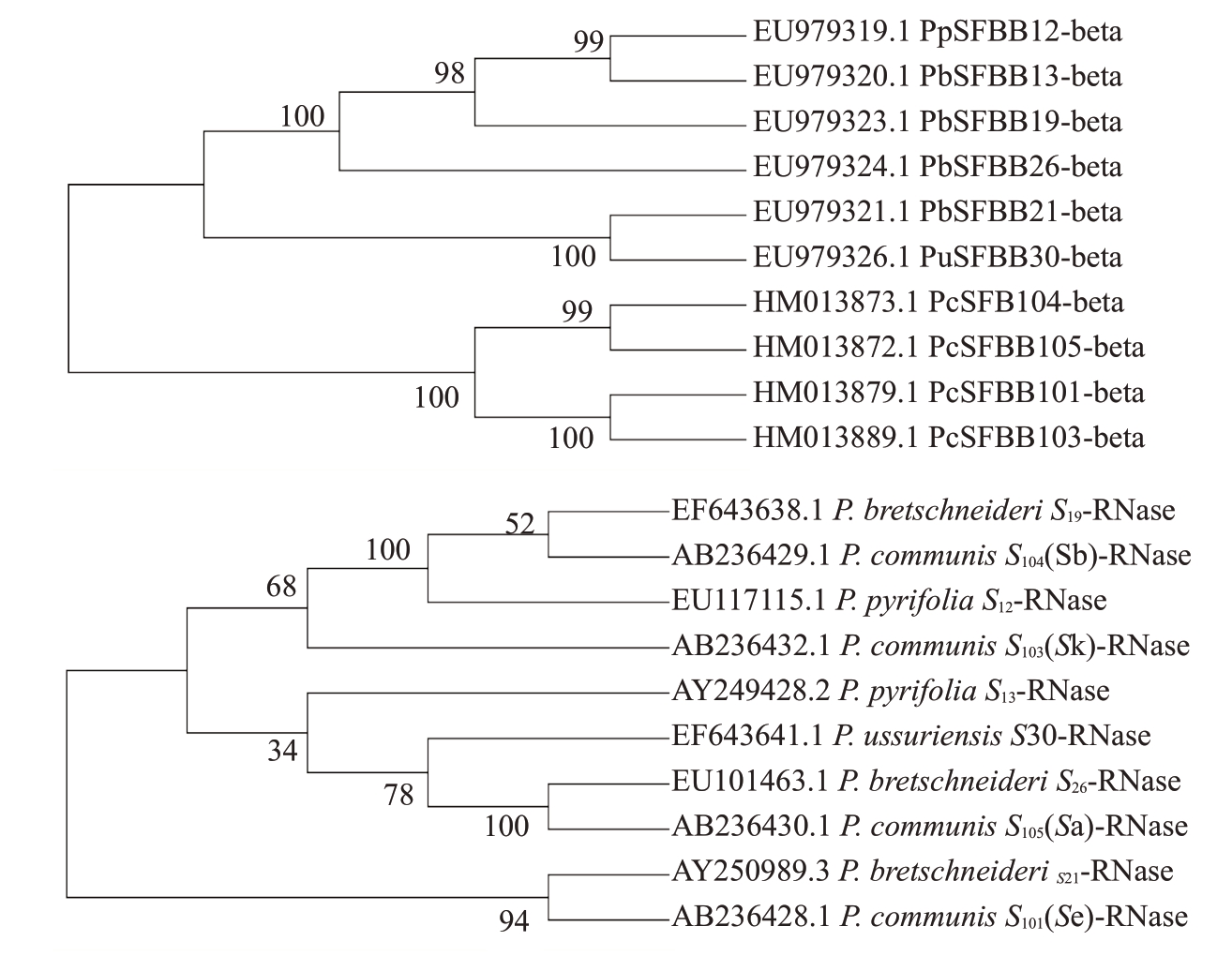
图5 梨SFBB-beta 基因和对应S-RNase 基因的NJ 系统发育树
Fig.5 NJ phylogenetic tree of pear SFBB-beta genes and S-RNase genes
梨SFBB-beta 编码区序列长1197 bp,399 个密码子,有275 个变异位点,占比22.97%,922 个保守位点,235个简约信息位点,39个单突变位点。序列中不同核苷酸占比,A:30.72%、T:30.72%、C:19.28%、G:19.28%,A+T 占 比61.44%,C+G 为38.56%。R为1.99。
对应的S-RNase编码区序列长705 bp,235个密码子,有297 个变异位点,占比42.13%,396 个保守位点,162 个简约信息位点,135 个单突变位点。序列中核苷酸占比,A:32.28%、T:25.76%、C:22.09%、G:19.87%,A+T占比58.04%,C+G为41.96%。R为0.88。
二者变异位点占比S-RNase基因大于SFBB-alpha 基因,前者是后者的1.83 倍,后者R 植是前者的2.26倍,C+G、A+T含量相差不大。
2.4.3 梨SFBB-beta 基因和对应S-RNase 基因序列分化分析 梨SFBB-beta 序列成对的总平均遗传距 离0.119。 其 中>0.160~0.180 占 比35.56% ,0.000~0.020 占 比13.33% ,>0.100~0.120 占 比11.11% 、>0.020~0.040、>0.140~0.160、>0.180~0.200 占比8.89%,>0.040~0.060、>0.120~0.140 占比6.67%,其余占0.00%。对应的梨S-RNase序列成对的总平均遗传距离0.191,是梨SFBB-beta 的1.61倍。其中>0.200~0.220占比42.22%,>0.180~0.200占比17.78%,>0.220~0.240 占比15.56%,>0.160~0.180占比8.89%,>0.240~0.260占比6.67%,其余占比不足10%。图形分布和SFBB-beta 非常相似(图6)。
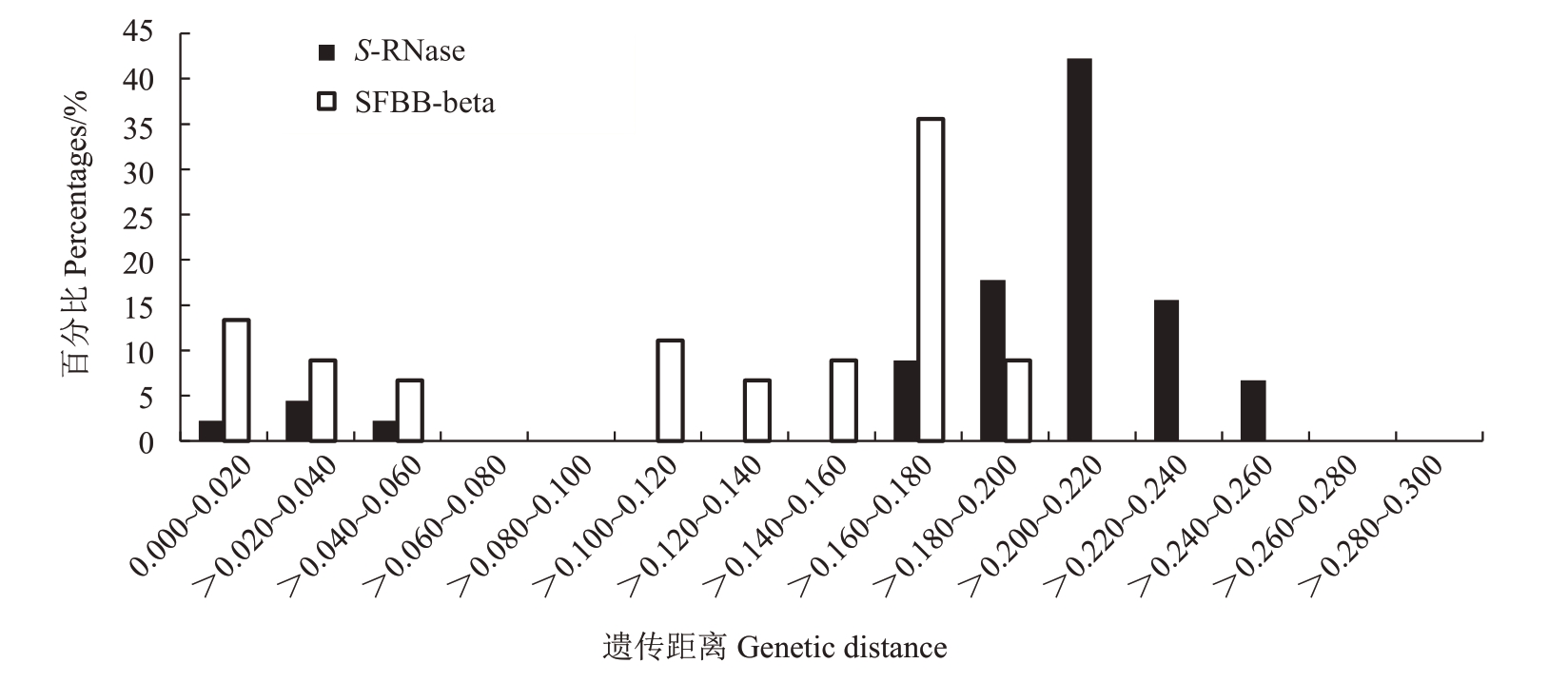
图6 梨SFBB-beta 基因和对应S-RNase 基因编码区序列所有碱基成对遗传距离分布
Fig.6 Pairwise genetic distance of whole nucleotide substitutions in coding regions of pear SFBB-beta genes and S-RNase genes
梨SFBB-beta 和对应S-RNase 所有密码子对应dN-dS 见表1,二者受正选择效应的位点SFBB-beta是S-RNase 的4/5,受中性选择的位点SFBB-beta 则是S-RNase 的1.68 倍,而受纯化选择的位点SFBBbeta则是S-RNase的3/5。SFBB-alpha与S-RNase连续阳性位点数比为26∶20。
2.4.4 梨SFBB-beta 基因和对应S-RNase 基因位点的遗传多态性 从反映梨属不同植物SFBB-beta 和对应S-RNase基因位点遗传多态性的两个指标Θ和π 数植来看(表2),S-RNase 基因的二者的数植均大于对应的SFBB-beta 基因,分别是其的1.83 倍和1.52 倍。因此,S-RNase 基因的遗传多态性高于SFBB 基因。同时二者的Tajima 检验统计量D 为正植,表明二者受相同选择的影响。
2.5 梨SFBB-gamma 基因及对应S-RNase 基因特征分析
2.5.1 梨SFBB-gamma基因及对应S-RNase基因理化性质 梨SFBB-gamma 基因编码蛋白平均由396个氨基酸组成,分子质量45.45 ku,平均理论等电点4.66,平均酸性氨基酸残基总数60,平均碱性氨基酸残基总数35,为酸性蛋白,平均分子式C2054C3108N498O618S24,平均不稳定系数为43.76,为不稳定蛋白,平均脂肪系数为86.05,总平均亲水性-0.147,为亲水蛋白。
对应S-RNase 基因编码蛋白平均由228 个氨基酸组成,平均分子质量26.02 ku,平均理论等电点9.12,平均酸性氨基酸残基总数17,平均碱性氨基酸残基总数26,为碱性蛋白,平均分子式C1161C1812N316O335S15,不稳定系数为37.98,属于稳定蛋白,平均脂肪系数为73.94,总平均亲水性-0.446,为亲水蛋白。
SFBB-gamma 基因分子质量是S-RNase 基因的1.75倍,二者蛋白分别为酸性和碱性,理论上具有酸碱相互作用和异性电荷相吸引的化学和物理促进相互作用机制。蛋白质稳定性虽然相反,但二者预测平均数植相差不大,存在实际稳定性一致的可能性,亲水性方面均为亲水蛋白,热稳定性也相似。
2.5.2 梨SFBB-gamma基因及对应S-RNase基因进化分析 梨SFBB-gamma基因及对应S-RNase基因NJ 进化树见图7,二者对应的基因在自己的进化树中拓扑结构不一致。由于SFBB-gamma基因的同源性太高,导致其进化树绝大多数分枝自展植不高。
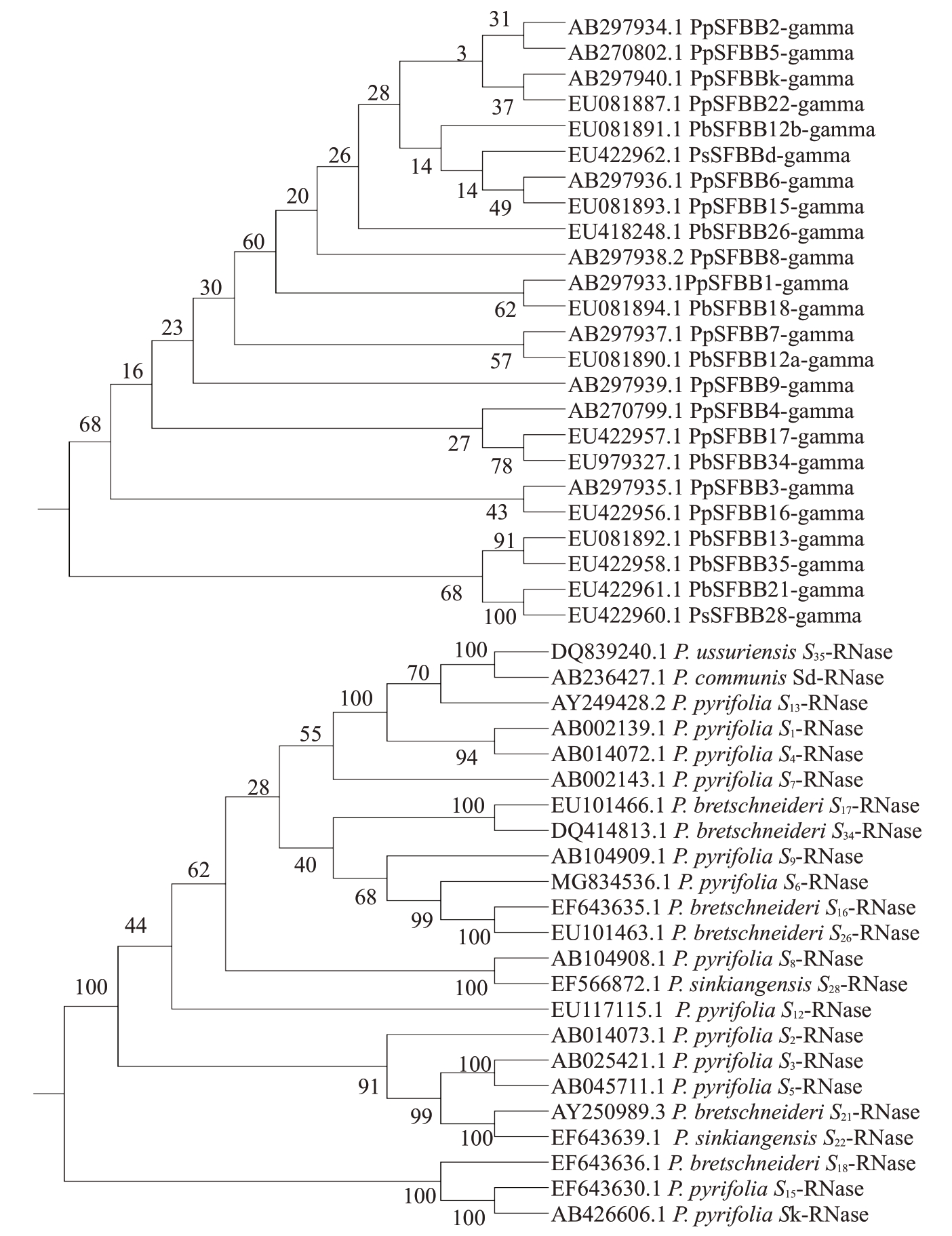
图7 梨SFBB-gamma 基因和对应S-RNase 基因的NJ 系统发育树
Fig.7 NJ phylogenetic tree of pear SFBB-gamma genes and S-RNase genes
梨SFBB-gamma 编码区序列长1191 bp,397 个密码子,有84 个变异位点,占比7.05%,1107 个保守位点,34 个简约信息位点,50 个单突变位点。序列中不同核苷酸占比,A:30.71%、T:30.71%、C:19.29%、G:19.29%,A+T 占比61.42%,C+G 为38.58%。R为3.68。
对应S-RNase编码区序列长711 bp,237个密码子,有407 个变异位点,占比57.24%,298 个保守位点,297 个简约信息位点,109 个单突变位点。序列中核苷酸占比,A:28.99%、T:28.99%、C:21.01%、G:21.01%,A+T 占比57.98%,C+G 为42.02%。R 为0.93。
二者变异位点占比S-RNase 基因大于对应SFBB-beta 基因,前者是后者的8.12 倍,R 植后者是前者的3.96倍,C+G、A+T含量相差不大。
2.5.3 梨SFBB-gamma基因和对应S-RNase基因序列分化分析 梨SFBB-gamma序列成对总平均遗传距离0.012。>0.009~0.012 占比25.36%、>0.006~0.009 占比23.19%、>0.012~0.015 占比14.86%、>0.015~0.018 占 比13.77% 、>0.003~0.006 占 比11.23%、>0.018~0.021 占比7.61%、>0.021~0.024占比3.99%,其余为0。对应S-RNase 序列成对的遗传距离总平均遗传距离0.212,是SFBB-gamma 的17.67 倍,其中>0.200~0.240 占比42.69%、>0.160~0.200占比24.51%、>0.240~0.280占比21.34%、其余占比11.46%(图8)。
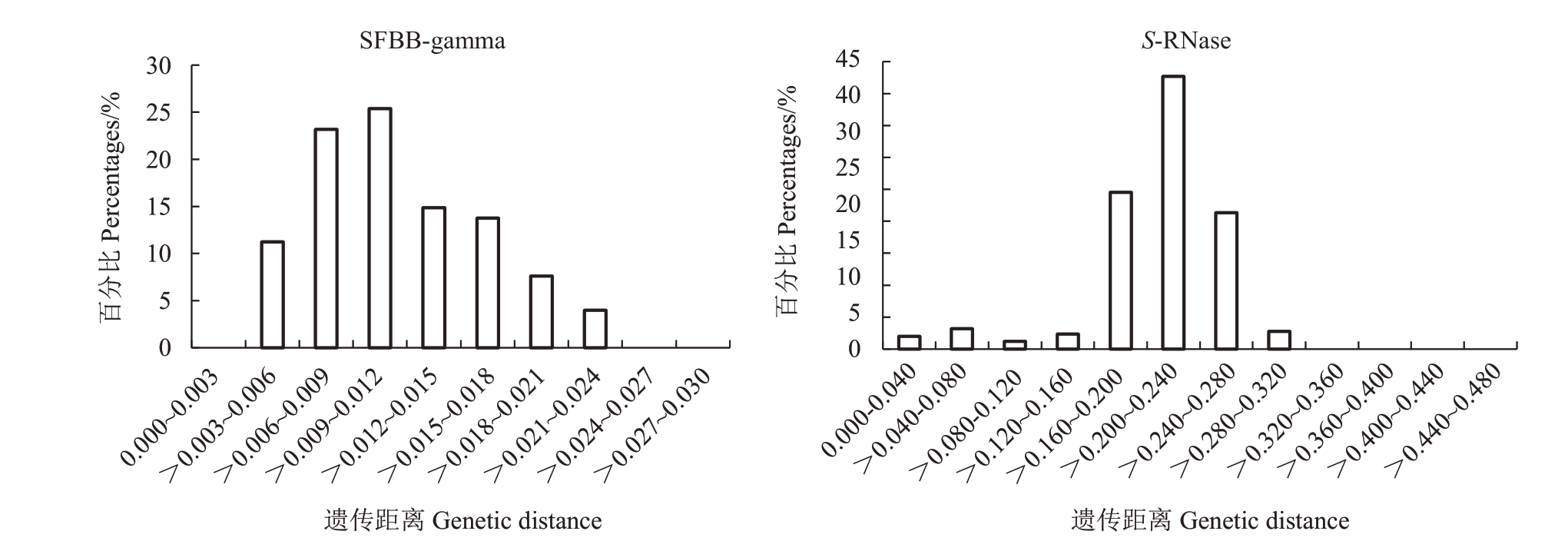
图8 梨SFBB-gamma 基因和对应S-RNase 编码区序列所有碱基成对遗传距离分布
Fig.8 Pairwise genetic distance of whole nucleotide substitutions in coding regions of pear SFBB-gamma genes and S-RNase genes
梨SFBB-gamma和对应S-RNase所有密码子对应的dN-dS 结果见表1,二者受正选择效应的位点SFBB-gamma 是S-RNase 的1/4,受中性选择的位点SFBB-gamma 则是S-RNase 的4.4 倍,而受纯化选择的位点SFBB-gamma则是S-RNase的1/5。梨SFBBgamma 与对应S-RNase 的连续阳性位点数比为6∶21。
2.5.4 梨SFBB-gamma基因和对应S-RNase基因位点的遗传多态性 梨属不同植物SFBB-gamma和对应S-RNase 基因位点的遗传分化情况结果如表2 所示。从反映遗传多态性的两个指标Θ 和π 数植来看,S-RNase 基因的二者的数植均远远大于对应的SFBB-gamma 基因,分别是其8.21 倍和15.09 倍,因此S-RNase基因的遗传多态性高于SFBB-gamma基因。同时二者的Tajima 检验统计量D 一正一负,表明二者受不相同选择的影响。
2.6 梨SFBB-epsilon 基因及对应S-RNase 基因特征分析
由于梨SFBB-epsilon 基因及对应S-RNase 基因目前只有3 个,因此仅能比较理化性质和基因进化情况,其余由于数据较少,无法比较。
2.6.1 梨SFBB-epsilon 基因及对应S-RNase 基因理化性质 梨SFBB-epsilon 基因编码蛋白平均由390个氨基酸组成,平均分子质量45.25 ku,理论等电点5.30,平均酸性氨基酸残基总数51,平均碱性氨基酸残基总数28,为酸性蛋白,平均分子式C2054H3111N506O591S28,不稳定系数42.71,属于不稳定蛋白,脂肪系数86.08,总平均亲水系数-0.149,属于亲水蛋白。
对应S-RNase 基因编码蛋白平均由230 个氨基酸组成,平均分子质量26.09 ku,平均理论等电点9.12,平均酸性氨基酸残基总数18,平均碱性氨基酸残基总数27,为碱性蛋白,平均分子式C1174H1830N315O338S14,不稳定系数为34.84,属于稳定蛋白,脂肪系数74.41,总平均亲水性-0.478,属于亲水蛋白。
梨SFBB-epsilon 基因编码蛋白分子质量是SRNase 基因编码蛋白的1.73 倍,二者蛋白分别为酸性和碱性,理论上具有酸碱相互作用和异性电荷相吸引的化学和物理促进相互作用机制。蛋白质稳定性虽然相反,但二者预测平均数植相差不大,存在实际稳定性一致的可能性,亲水性方面均为亲水蛋白,蛋白热稳定性相似。
2.6.2 梨SFBB-epsilon 基因及对应S-RNase 基因进化分析 梨SFBB-epsilon 编码区序列长1173 bp,391 个密码子,有19 个变异位点,占比1.62%,1154个保守位点,1 个简约信息位点,1 个单突变位点。序列中不同核苷酸占比,A+T 占比30.66%,C+G 为19.34%。R植为2.18。阳性位点占比3.85%、连续阳性位点1 处,等于0 占比95.13%、小于0 占比1.02%。平均成对遗传距离0.011。
对应S-RNase编码区序列长699 bp,233个密码子,有175 个变异位点,占比25.04%,515 个保守位点,0个简约信息位点,175个单突变位点,序列中不同核苷酸占比,A+T 占比29.38%,C+G 为20.62%。R 为0.97。阳性位点占比29.74%、连续阳性位点15处,等于0占比50%、小于0占比20.26%。平均成对遗传距离0.204。
二者变异位点占比S-RNase基因大于SFBB-beta 基因,前者是后者的18.36 倍,R 植后者是前者的2.25倍,C+G、A+T含量相差不大。
3 讨 论
笔者在本研究中利用苹果亚科梨属SFBB-gamma基因的保守序列特征设计特异引物,从3个梨品种中扩增出了SFBB22-gamma 全长序列,针对SFBB-gamma设计的特异引物未能在3 个梨品种中扩增出其他SFBB-gamma 基因,这种现象在以前研究中[23-25]也常有出现,这制约了更多的梨SFBB基因被分离鉴定,一定程度上影响了后续对SFBB 基因特征的研究。
目前,NCBI 上登录的能与梨S-RNase 和对应SFBB全长基因有alpha(α)、beta(β)、gamma(γ)以及epsilion(ε)4 个类群。本研究的结果发现alpha、beta、gamma 以及epsilion 现有序列均自己聚成一类,显示各序列内有一定的保守性,这一结论还需要更多新的同类基因序列分离出来加以验证。理化性质预 测SFBB-alpha、SFBB-beta、SFBB-gamma 以 及SFBB-spsilion 基因编码的蛋白均为酸性蛋白和SRNase编码的碱性蛋白均具有正负电荷相吸引的物理机制促进和酸碱反应的化学促进机制,这和Li等[30]在研究矮牵牛(Petunia hybrida)自交不亲和位点S-RNase 和SLF 情况一致,同时二者编码蛋白均为亲水蛋白,热稳定性也相似,SFBB编码的蛋白分子质量约是S-RNase 基因编码蛋白的1.75 倍,均存在理论上相互作用的可能性。
在已分离鉴定的蔷薇科植物花粉S 基因中,李亚科的李属、茄科矮牵牛属以及车前科金鱼草属的花粉SFB 基因是位于S 位点区域S-RNase 基因下游的第一个F-box基因[15,31-32]。苹果亚科梨属、苹果属、花楸属中花粉S基因的研究表明,这些物种S位点区域S-RNase 基因单倍型内含有两个以上F-box 基因(被称为SFBB 基因)[20-23,33]。具体实际数量尚不清楚,目前也很难假设有多少未被发现。现有和梨SRNase 基因一一对应的SFBB 基因全长序列统计结果也支持上述观点。
已有的花粉和雌蕊S 基因进化研究的情况显示,蔷薇科李亚科李属植物花粉SFB 基因与雌蕊SRNase 基因在序列多样性、阳性选择证据和共享的祖先多态性方面表现出共同进化的特征,茄科矮牵牛属和车前科金鱼草属的雌蕊S-RNase基因和花粉S(SLF/SFB)基因之间没有太多共同进化的特征[34]。最近的研究表明,蔷薇科的自交不亲和包括两个不同的系统:蔷薇科李亚科李属表现出的一种自我识别系统,即其他的SFB识别自我S-RNase;而苹果亚科苹果属、梨属以及花楸属显示了一种非自我识别系统[14,33]。在对花楸属欧亚花楸的自交不亲和模式研究中发现许多SFBB蛋白可能参与自交不亲和,每个识别非自身S-RNases 的子集,即一个SFBB 基因可以识别不止一个S-RNase 基因。为何梨亚科花粉S基因的模式更接近亲缘关系较远的茄科、车前科等植物而非亲缘关系近的李亚科,有研究表明毛茛科、茄科、车前科和蔷薇科多个物种S位点极其古老,可能与被子植物的起源和扩张有关。因此,推测SFB或SFBB基因的分化至少早于毛茛科、茄科、车前科和蔷薇科植物[35]。
根据多数学者认为的自交不亲和位点进化理论,花粉S 基因预计与雌蕊S 基因存在共同进化[33,35]。因此这两个基因应该表现出长期共同进化的共同特征。通过对比梨属雌蕊S-RNase和其对应的在S 位点区域内花粉SFBB 基因的4 个类群52 个成员在序列变异、基因多态性以及进化方面的特性找出更符合的花粉S 基因。研究表明梨属植物SFBB-alpha、SFBB-beta、SFBB-gamma 以及SFBBepsilon 的结构具有较高的保守性,进一步研究分析表明梨SFBB-alpha、SFBB-gamma 以及SFBB-epsilon表现出与对应S-RNase基因共进化的证据较少,这些SFBB 基因表现出较低的序列变异,较少的阳性选择以及较低的基因多态性。SFBB-beta 基因显示出较高的序列多样性水平,尽管其仍低于SRNase显示的序列多样性水平。这与矮牵牛花SLF表现出的特征相一致[36-37],Chen等[38]和Vieira等[39]认为有害突变的强烈选择作用于SLF 基因,从而限制矮牵牛SLF 的变异性,但这并不影响其作为花粉S基因的作用。若把SFBB 基因4 个类群作为整体来看,其和对应S-RNase的情况反而好于SFBB-alpha、SFBB-gamma和SFBB-epsilon,甚至比SFBB-beta基因更接近S-RNase 序列特征,这种分析方式在以往学者的类似研究中还未出现。是否4种亚类均参与自交不亲和还是SFBB-beta独自参与自交不亲和还需要更多的研究加以验证。
4 结 论
设计特异引物可以直接扩增出梨SFBB基因全长,目前这一技术在通用性方面还有待于改进。找出一种可以媲美扩增梨S-RNase全长序列的方法是研究梨SFBB 基因的关键所在。现有能和梨SRNase 基因一一对应的全长序列SFBB 基因有4 个成员,其每组成员间序列相对比较保守,现有序列数据显示其在序列多态性和变异位点方面均小于对应S-RNase,从理论角度讲是否某一SFBB基因参与自交不亲和,其余SFBB 基因不直接参与还是所有SFBB 基因均参与其中都有一定的证据,但还需要更多的试验数据进一步加以验证。
[1] SASSA H.Molecular mechanism of the S-RNase-based gametophytic self-incompatibility in fruit trees of Rosaceae[J]. Breeding Science,2016,66(1):116-121.
[2] 何敏,谷超,吴巨友,张绍铃. 果树自交不亲和机制研究进展[J].园艺学报,2021,48(4):759-777.HE Min,GU Chao,WU Juyou,ZHANG Shaoling. Recent advances on self-incompatibility mechanism in fruit trees[J].Acta Horticulturae Sinica,2021,48(4):759-777.
[3] 梁文杰,谭晓风,乌云塔娜.梨自交不亲和基因克隆及其进化分析[J].果树学报,2021,38(10):1621-1637.LIANG Wenjie,TAN Xiaofeng,WUYUN Tana. Cloning and phylogenetic analysis of S- RNase genes in genus Pyrus plants[J].Journal of Fruit Science,2021,38(10):1621-1637.
[4] WOLKO Ł,ANTKOWIAK W,SIPS M,SŁOMSKI R. Self-incompatibility alleles in Polish wild pear [Pyrus pyraster (L.)Burgsd.]:A preliminary analysis[J]. Journal of Applied Genetics,2010,51(1):33-35.
[5] 唐亮,马香,李明霞,周志钦.苹果属S-RNase 基因的进化研究[J].中国农业科学,2013,46(13):2717-2729.TANG Liang,MA Xiang,LI Mingxia,ZHOU Zhiqin.Evolutionary studies of S-RNase genes in genus Malus[J]. Scientia Agricultura Sinica,2013,46(13):2717-2729.
[6] GU C,WANG L,KORBAN S S,HAN Y P. Identification and characterization of S-RNase genes and S-genotypes in Prunus and Malus species[J]. Canadian Journal of Plant Science,2015,95(2):213-225.
[7] RYAN S,SARA S,PAOLO D F,LUCA D,STEFANO M.Characterization of a novel self-incompatibility allele in Malus and S-genotyping of select crabapple cultivars[J]. Scientia Horticulturae,2018,240:186-195.
[8] OLIVIER R,JOSHUA R K. Population structure at the S-locus of Sorbus aucuparia L. (Rosaceae:Maloideae)[J]. Molecular Ecology,2007,16(6):1315-1325.
[9] MA J Z,WANG H J,WANG D S,LI X Y,LIU J Z,XIAO X,ZHANG L B,WU J K.Self-compatibility of Crataegus pinnatifida Bge.‘Zizhenzhu’is associated with the mutation of a pistilpart non-S factor[J].Scientia Horticulturae,2020,268:109362.
[10] RASPE' O,KOHN J R. S-allele diversity in Sorbus aucuparia and Crataegus monogyna (Rosaceae:Maloideae)[J]. Heredity,2002,88:458-465.
[11] ZHANG Y,QIN L H,WANG H K,CHEN X P,WANG S H.Identification of S genotypes in loquat (Eriobotrya japonica Lindl.) based on allele specific PCR[J]. Scientia Horticulturae,2017,225:736-742.
[12] ASHKANI J,REES D J G.A comprehensive study of molecular evolution at the self-Incompatibility locus of Rosaceae[J]. Journal of Molecular Evolution,2016,82(2/3):128-145.
[13] GU C,ZHANG S L,HUANG S X,HENG W,LIU Q Z,WU H Q,WU J. Identification of S-genotypes in Chinese cherry cultivars(Prunus pseudocerasus Lindl.)[J]. Tree Genetics & Genomes,2010,6(4):579-590.
[14] AGUIAR B,VIEIRA J,CUNHA A E,FONSECA N A,IEZZONI A,VAN NOCKER S,VIEIRA C P. Convergent evolution at the gametophytic self- Incompatibility system in Malus and Prunus[J].PLoS One,2015,10(5):e0126138.
[15] USHIJIMA K,SASSA H,DANDEKAR A M,GRADZIEL T M,TAO R,HIRANO H. Structural and transcriptional analysis of the self-incompatibility locus of almond:Identification of a pollen-expressed F-box gene with haplotype-specific polymorphism[J].The Plant Cell,2003,15(3):771-781.
[16] ENTANI T,IWANO M,SHIBA H,CHE F S,ISOGAI A,TAKAYAMA S. Comparative analysis of the self-incompatibility (S-) locus region of Prunus mume:Identification of a pollenexpressed F-box gene with allelic diversity[J]. Genes to Cells,2003,8(3):203-213.
[17] SONNEVELD T,TOBUTT K R,VAUGHAN S P,ROBBINS T P. Loss of pollen-S function in two self-compatible selections of Prunus avium is associated with deletion/mutation of an S haplotype-specific F-box gene[J].The Plant Cell,2005,17(1):37-51.
[18] MARCHESE A,BOSKOVIC R I,CARUSO T,RAIMONDO A,CUTULI M,TOBUTT K R.A new self-compatibility haplotype in the sweet cherry‘Kronio’,S5′,attributable to a pollenpart mutation in the SFB gene[J]. Journal of Experimental Botany,2007,58(15/16):4347-4356.
[19] USHIJIMA K,YAMANE H,WATARI A,KAKEHI E,IKEDA K,HAUCK N R,IEZZONI A F,TAO R. The S haplotype-specific F-box protein gene,SFB,is defective in self-compatible haplotypes of Prunus avium and P. mume[J].The Plant Journal,2004,39(4):573-586.
[20] SASSA H,HIRANO H,IKEHASHI H. Identification and characterization of stylar glycoproteins associated with self-incompatibility genes of Japanese pear,Pyrus serotina Rehd. [J]. Molecular Genetics and Genomics,1993,241(1/2):17-25.
[21] SASSA H,KAKUI H,MIYAMOTO M,SUZUKI Y,HANADA T,USHIJIMA K,KUSABA M,HIRANO H,KOBA T. S locus F-box brothers:Multiple and pollen-specific F-box genes with S haplotype- specific polymorphisms in apple and Japanese pear[J].Genetics,2007,175(4):1869-1881.
[22] FRANCESCHI P D,PIERANTONI L,DONDINI L,GRANDI M ,SANZOL J,SANSAVINI S. Cloning and mapping multiple S-locus F-box genes in European pear (Pyrus communis L.)[J].Tree Genetics&Genomes,2011,7(2):231-240.
[23] 张琳,谭晓风,胡姣,乌云塔娜,袁德义,何小勇,龙洪旭,李秀根. 中国白梨PbSFBB13-gamma 基因的分子克隆与序列特征[J].林业科学,2009,45(11):36-43.ZHANG Lin,TAN Xiaofeng,HU Jiao,WUYUN Tana,YUAN Deyi,HE Xiaoyong,LONG Hongxu,LI Xiugen. Molecular cloning and characterization of PbSFBB13-gamma from Chinese white pear(Pyrus bretschneideri)[J]. Scientia Silvae Sinicae,2009,45(11):36-43.
[24] 乌云塔娜,李洪果,李振国,包梅荣,谭晓风.梨4 个SFBB-γ基因的分离及遗传多态性分析[J]. 林业科学,2010,46(1):147-151.WUYUN Tana,LI Hongguo,LI Zhenguo,BAO Meirong,TAN Xiaofeng. Isolation and genetic polymorphism analysis of pear SFBB-r genes[J].Scientia Silvae Sinicae,2010,46(1):147-151.
[25] CLAESSEN H,KEULEMANS W,POEL B V D,STORME N D.Finding a compatible partner:Self-incompatibility in European pear(Pyrus communis); molecular control,genetic determination,and impact on fertilization and fruit set[J]. Frontiers in Plant Science,2019,10:407.
[26] 王立新,梁文杰,谭晓风,李秀根,张琳.6 个梨品种S 基因型鉴定及新基因S45-RNase 序列分析[J].果树学报,2010,27(5):689-693.WANG Lixin,LIANG Wenjie,TAN Xiaofeng,LI Xiugen,ZHANG Lin. Identification of S-genotypes of six pear cultivars and sequence analyses of S45-RNase[J].Journal of Fruit Science,2010,27(5):689-693.
[27] 邓建军,谭晓风,包梅荣,乌云塔娜,张琳,曹玉芬.11 个中国梨品种S 基因型的鉴定[J].中南林业科技大学学报,2010,30(3):63-66.DENG Jianjun,TAN Xiaofeng,BAO Meirong,WUYUN Tana,ZHANG Lin,CAO Yufen.S-genotypes of 11 Chinese pear cultivars[J]. Journal of Central South University of Forestry &Technology,2010,30(3):63-66.
[28] 张校立,艾沙江·买买提,徐叶挺,邓莉,王继勋.梨S 基因与S基因型鉴定的研究进展[J].西北农业学报,2018,27(8):1077-1087.ZHANG Xiaoli,Aishajiang·Maimaiti,XU Yeting,DENG Li,WANG Jixun. Present advance of S-gene genotype and S-genotypes in pear[J].Acta Agriculturae Boreali-Occidentalis Sinica,2018,27(8):1077-1087.
[29] ABDALLAH D,BARAKET G,PEREZ V,HANNACHI A S,HORMAZA J I.Self-compatibility in peach[Prunus persica(L.)Batsch]:Patterns of diversity surrounding the S-locus and analysis of SFB alleles[J].Horticulture Research,2020,7(1):170.
[30] LI J H,ZHANG Y,SONG Y Z,ZHANG H,FAN J B,LI Q,ZHANG D F,XUE Y B. Electrostatic potentials of the S-locus F-box proteins contribute to the pollen S specificity in self-incompatibility in Petunia hybrida[J].The Plant Journal,2017,89(1):45-57.
[31] LAI Z,MA W S,HAN B,LIANG L Z,ZHANG Y S,HONG G F,XUE Y B. An F-box gene linked to the self-incompatibility(S) locus of Antirrhinum is expressed specifically in pollen and tapetum[J].Plant Molecular Biology,2022,50(1):29-42.
[32] WANG Y,TSUKAMOTO T,YI K W,WANG X,HUANG S,MCCUBBIN A G,KAO T H.Chromosome walking in the Petunia inflata self-incompatibility(S-)locus and gene identification in an 881-kb contig containing S2-RNase[J].Plant Molecular Biology,2004,54(5):727-742.
[33] AGUIAR B,VIEIRA J,CUNHA A E,FONSECA N A,REBOIRO-JATO D,REBOIRO-JATO M,FDEZ-RIVEROLA F,RASPÉ O,VIEIRA C P.Patterns of evolution at the gametophytic self- incompatibility Sorbus aucuparia (Pyrinae) S pollen genes support the non-self recognition by multiple factors model[J]. Journal of Experimental Botany,2013,64(8):2423-2434.
[34] NEWBIGIN E,PAAPE T,KOHN J R.RNase-based self-incompatibility:Puzzled by pollen S[J]. Plant Cell,2008,20(9):2286-2292.
[35] ZHAO H,ZHANG Y,ZHANG H,SONG Y Z,ZHAO F,ZHANG Y,ZHU S H,ZHANG H K,ZHOU Z D,GUO H,LI M M,LI J H,GAO Q,HAN Q Q,HUANG H Q,COPSEY L,LI Q,CHEN H,COEN E,ZHANG Y J,XUE Y B. Origin,loss and regain of self-incompatibility in angiosperms[J]. The Plant Cell,2022,34(1):579-596.
[36] SIJACIC P,WANG X,SKIRPAN A L,WANG Y,DOWD P E,MCCUBBIN A G,HUANG S,KAO T H. Identification of the pollen determinantof S-RNase-mediated self-incompatibility[J].Nature,2004,429(6989):302-305.
[37] QIAO H,WANG F,ZHAO L,ZHOU J L,LAI Z,ZHANG Y S,ROBBINS T P,XUE Y B. The F-box protein AhSLF-S2 controls the pollen function of S-RNase-based self-incompatibility[J].Plant Cell,2004,16(9):2307-2322.
[38] CHEN G,ZHANG B,ZHAO Z H,SUI Z H,ZHANG H,XUE Y B.‘A life or death decision’for pollen tubes in S-RNasebased selfincompatibility[J]. Journal of Experimental Botany,2010,61(7):2027-2037.
[39] VIEIRA J,FONSECA N A,VIEIRA C P. RNase-based gametophytic self-incompatibility evolution:Questioning the hypothesis of multiple independent recruitments of the S-pollen gene[J].Journal of Molecular Evolution,2009,69(1):32-41.