猕猴桃(Actinidia)为猕猴桃科(Actinidiaceae)猕猴桃属(Actinidia Lindl.)雌雄异株多年生藤本植物,共有54个种和21个变种[1]。它作为20世纪野生果树人工驯化栽培最有成就的四大果树之一[2],既是一种古老植物,又是一种新兴水果,同时由于具有丰富的维生素含量和独特的口感常被誉为“水果之王”[3]。品种是产业发展的“芯片”,由于杂交育种可以实现优异多基因聚合的育种目标,从而可以培育更加符合生产者、消费者及经营者要求的优良品种,所以猕猴桃育种途径从传统野生直接选种越来越多的转向杂交育种。在杂交育种过程中,从杂交分离后代中挑选优良的重组基因型是其关键步骤,传统依靠表型选择的育种方法耗时费力并且难度大,培育一个新品种需花费10~15 a时间,并且多数表型性状是易受环境影响的数量性状,表现不稳定,从而降低了表型选育的准确度。随着PCR 技术和现代测序技术的发展,产生了分子标记辅助选择(markerassisted selection,MAS)育种技术,由于该方法是基于基因型而不是表现型来选择育种所需的性状,因此受到广大育种学家及遗传学家的青睐。猕猴桃属植物染色体倍性复杂,从二倍体到八倍体均有发现[4],该属植物存在丰富的核基因组遗传多样性和叶绿体、线粒体基因组遗传变异[5],分子标记具有丰富的多态性,在资源评价及早期性状鉴定等方面可产生较好的效果[6],因此,可在育种工作中采用定向杂交育种与分子辅助育种相结合的方法,将分子标记与性状进行关联分析,从而快速筛选出具有特定优良性状的品种[7]。标记辅助育种的关键是找到和表型连锁的标记,构建遗传连锁图谱(genetic linkage map)并进行数量性状位点(quantitative trait locus,QTL)定位,可有效建立起标记与表型之间的连锁关系,是实施MAS的有效途径。
遗传连锁图谱,也称遗传图谱(genetic map)或连锁图谱(linkage map),是指以遗传标记(已知性状的基因或特定DNA序列)间重组频率为基础的染色体或基因位点的相对位置线性排列图[8]。以分子标记技术作为基础,猕猴桃遗传图谱的构建从由形态、生理生化标记构建的低密度遗传图谱,转为由分子标记构建的高密度遗传图谱[9];可将遗传图谱中已知位点的分子标记与未知的QTL进行连锁分析,把一个或多个QTL 定位到同一染色体上已知位点的分子标记附近,从而确定QTL在基因组中的位置。遗传图谱密度越高,越有利于后续猕猴桃重要农艺性状的QTL 定位,因此,猕猴桃遗传连锁图谱的构建以及QTL精细定位的研究,为提高优质猕猴桃新品种选育效率提供了理论与方法。
1 猕猴桃遗传连锁图谱构建的分子标记基础
分子标记技术以个体间核苷酸序列变异为基础,是生物个体遗传变异的直接反映,可以检测出生物个体之间在核苷酸序列水平上的差异[10],与传统的形态学标记、细胞学标记相比,它存在诸多优点:不受气候、环境等因素影响,在植物的各个组织、器官以及各发育阶段都能进行检测,多态性高[11]。目前,该技术被广泛应用于遗传多样性分析、种质资源鉴定、遗传图谱构建、系统进化研究及分子标记辅助育种等方面[12]。
1.1 分子标记在猕猴桃种质资源遗传多样性评价中的应用
对猕猴桃种质资源进行准确鉴定及评价是对其合理利用的前提。SSR(simple sequence repeats)标记,也称为微卫星DNA(microsatellite DNA)是由几个核苷酸(一般为1~6个)为基本重复单位的多次串联重复DNA 序列,以其重复性较好、易于获取及多态性丰富等优点,在猕猴桃资源鉴定及评价等研究领域被大量应用。前人研究表明,共显性SSR 标记在构建猕猴桃指纹图谱以及分析种质资源的遗传多样性方面都有较好效果[13]。1998 年,Huang 等[14]从富含(AC/GT)和(AG/CT)重复序列的两个小的插入型文库中分离并测序了263 个含SSR 的克隆,针对其中203个SSR位点设计的引物成功在质粒和中华猕猴桃(A.chinensis)基因组DNA中扩增出产物,同时报道了40对SSR引物的序列,为猕猴桃后续SSR标记开发提供了参考[14];郑轶琦等[15]进一步从中筛选出9对稳定性较好的引物分别在48个猕猴桃品种(品系)中进行PCR扩增,成功获得了每个品种特有的指纹图谱。姜春芽等[16]筛查了猕猴桃EST 数据库,并从中开发了SSR标记,表明该标记可对猕猴桃种质的多态性进行分析;之后,王丹丹等[17]利用21对EST-SSR 分子标记构建了常见的42 份软枣猕猴桃(A.arguta)种质的数字指纹图谱和模式指纹图谱数据库,并进行了遗传多样性分析,将42 份种质划分为4 个类群,聚类结果与种质的特性及来源具有较高的一致性。汤佳乐等[18]和邹梁峰等[19]也先后利用SSR分子标记进行了江西省境内的猕猴桃资源遗传多样性分析。
除SSR 标记外,其他类型的分子标记在分析猕猴桃遗传多样性方面也发挥着重要作用。例如利用RAPD[20- 21],AFLP[22- 23],ISSR[24- 25],SPAR[26- 28],SNP[29]、SCoT[30]等分子标记,构建了各种猕猴桃指纹图谱并对猕猴桃种质资源进行了分类和鉴定。上述研究在分子水平上揭示了猕猴桃属植物的遗传多样性以及各品种或种质间的亲缘关系,为种质资源的合理开发及利用奠定了基础。
1.2 分子标记在猕猴桃性状早期鉴定中的应用
猕猴桃雌雄异株及染色体倍性水平多样化特征,在很大程度上阻碍其一些重要经济性状的遗传学研究。用简便易行、高效快捷的分子标记技术对猕猴桃属植物进行遗传学研究十分必要,开发出与目标性状连锁的分子标记进行标记辅助选择可以有效提高育种的效率及准确性。
猕猴桃属于功能性的雌雄异株植物[31],在实际生产中雌株的经济价值远大于雄株,但开花以前难以从其他形态上进行性别区分,所以利用分子标记技术在童期进行性别早期鉴定一直是该树种分子辅助育种研究的最大热点。例如Harvey 等[32]和Gill等[33]通过集团分离分析法(Bulk Segregate Analysis,BSA),利用中华猕猴桃(A.chinensis)实生群体,开发出雄性特异的SmY 和雌性特异的SmX 2 个SCAR 标记;Zhang 等[34]利用山梨猕猴桃(A.rufa 与中华猕猴桃(A.chinensis)的种间杂交F1代群体,开发了A001、A002 和A003 等3 个性别特异性SSR 标记。不过,前述两个标记的开发均是以二倍体猕猴桃为试材,由于猕猴桃倍性复杂,因此郭丹丹等[35]对两组性别标记的通用性在已知性别的四倍体软枣猕猴桃和二倍体中华猕猴桃材料上进行了验证,结果除了SmY在中华猕猴桃中有较好的通用性,其余标记在两种猕猴桃中均未表现出良好的通用性,其进一步利用软枣猕猴桃杂交群体通过全基因组重测序及BSA 分析技术,开发了2 个雄性特异性性别标记,在四倍体软枣猕猴桃上验证准确率均可达97%[12]。
另外,徐小彪等[36]进行了中华猕猴桃矮型性状EST-SSR 连锁标记的筛选,获得了鉴别矮型与普通型植株的标记引物EST-Ad042;易盼盼等[37]获得了与猕猴桃抗溃疡病基因连锁的SSR 分子标记UDK97-428116;汤佳乐等[38]获得了野生毛花猕猴桃(A.eriantha)叶片及果实中与AsA 含量关联的SSR标记。
1.3 分子标记在猕猴桃种质真实性鉴定中的应用
在猕猴桃育种过程中,由于机械混杂、生物学混杂等,杂交F1代实生群体中容易出现异型株而影响分子标记开发的准确性,因此孙世航等[39]以软枣猕猴桃为材料,筛选了可对杂交子代进行真实性鉴定的InDel引物,为后续利用群体进行分子标记开发奠定了基础。
品种的纯度与真实性直接影响农作物的产量和品质,也是作为杂交亲本利用的前提。对种质纯度与真实性鉴定的本质就是对基因型的鉴定[40],目前鉴定方法主要包括形态学法、生物化学法以及分子标记法,但前两种方法容易受到环境及材料本身影响,而分子标记鉴定法不受外部条件影响,相较于前两种方法准确性更高。分子标记鉴定法利用对遗传物质多态性的直接分析来判断基因的排布规律和性状的表现规律,是通过比较分析各品种间基因组DNA 的差异来鉴别品种[41]。猕猴桃产业中品种乱杂现象相对较多,田间测试鉴定难以满足时效性及准确性的要求,并且费时费力,因此王丹丹等[42]采用SSR、EST-SSR 和SRAP 三种分子标记鉴定了8 个软枣猕猴桃区试品种的纯度,表明ESTSSR 引物多态性、稳定性好,可用于后续研究应用。目前,中国农业科学院郑州果树研究所等四家单位联合制定的农业行业标准《中华猕猴桃品种鉴定SSR 分子标记法》NY/T 3639—2020[43],已经正式颁布实施。
2 猕猴桃遗传连锁图谱构建
遗传连锁图谱的理论基础是在细胞减数分裂过程中,非同源染色体上的基因自由组合,同源染色体上的连锁基因发生交换和重组[44]。遗传图谱的密度依赖于标记的类型,分子标记作为一种新型的标记技术,其不受环境因素影响、稳定、高效等优点在构建图谱中有较大优势,由于DNA分子标记可以从微观上检测不同个体间基因或基因型的差异来反应个体间宏观上的表型差异,因此在构建遗传图谱过程中使用分子标记不受发育阶段和环境的影响,这将大大提高图谱构建效率,并且相较于传统的形态、生物化学标记,DNA分子标记多态性高并且数量无限多,存在许多等位变异并几乎能遍及整个基因组,分子标记这些特点都使得遗传图谱的构建越来越高效、准确,并且构建的图谱密度越来越高。高密度的遗传连锁图谱是进行QTL定位的基础,也是进行图位克隆、分子标记辅助育种等的前提。目前常用的图谱构建软件有JoinMAP4.0、MapMarker/EXP3.0等。
2.1 猕猴桃作图群体的选择
用于构建遗传图谱的材料通常是目的性状分离的群体,群体亲本的选择直接影响到遗传图谱构建的难易程度及图谱的适用范围。如果亲本间差异过大、亲缘关系过远,会使染色体的配对和重组受到抑制,从而导致连锁的基因座之间重组率降低,甚至会造成后代不育,影响作图群体的构建,即使群体构建完成,也可能出现严重的偏分离现象[45];亲本间差异过小,会使亲本间DNA 多态性降低,影响图谱密度及后续QTL定位精度。因此,用于构建图谱的作图群体亲本间遗传差异要适中,选择合适的分离群体是构建遗传图谱必不可少的条件。
要构建合适的群体需要根据植物本身的特征来决定。如果作图的对象可以自交,而且能够获得纯系,那么可以选择F2代、重组自交系(recombinant inbred lines,RIL)、回交群体(backcross,BC)或者双单倍体(doubled haploid lines,DH)来构建作图群体[46];如果作图对象本身自交不亲和或者近交衰退,不能获得纯系,则可选择F1代和BC群体。对于果树,由于其杂合度高、世代周期长,加上部分果树存在较严重的自交退化现象,很难获得自交纯系,因此较多的采用F1代作图群体。猕猴桃自身高度杂合,F1代即可产生大量性状分离,并且童期较长,所以目前常用F1代群体作为作图群体。
2.2 猕猴桃遗传连锁图谱构建的研究现状
随着分子标记技术的发展,自1986 年来,许多作物都已经建立了分子遗传图谱,并且逐渐饱和[47],例如Mccouch 等[48]在1988 年构建出第一张水稻RFLP图谱之后,Wu等[49]用超过6000个标记构建出一张高密度水稻遗传图谱。作物遗传图谱的发展为构建猕猴桃图谱提供了借鉴,当前猕猴桃分子育种领域也逐步构建出一些遗传连锁图谱,从2001年第一张猕猴桃遗传连锁图谱的构建,国内外至今至少构建了10 个(组)猕猴桃的遗传连锁图谱(表1),这些图谱当中有的是基于父母本分别构建两张遗传图谱,有的是构建一张父母本整合遗传图谱;图谱构建所用的分子标记主要包括SSR、AFLP 和SNP;随着分子标记技术不断发展,猕猴桃遗传图谱的标记数、图谱长度、标记间的平均距离等都取得了明显进步,而且在多倍体猕猴桃中也逐步开始建立较完善的遗传图谱。
表1 猕猴桃遗传连锁图谱比较
Table 1 Comparison of genetic linkage map of kiwifruit
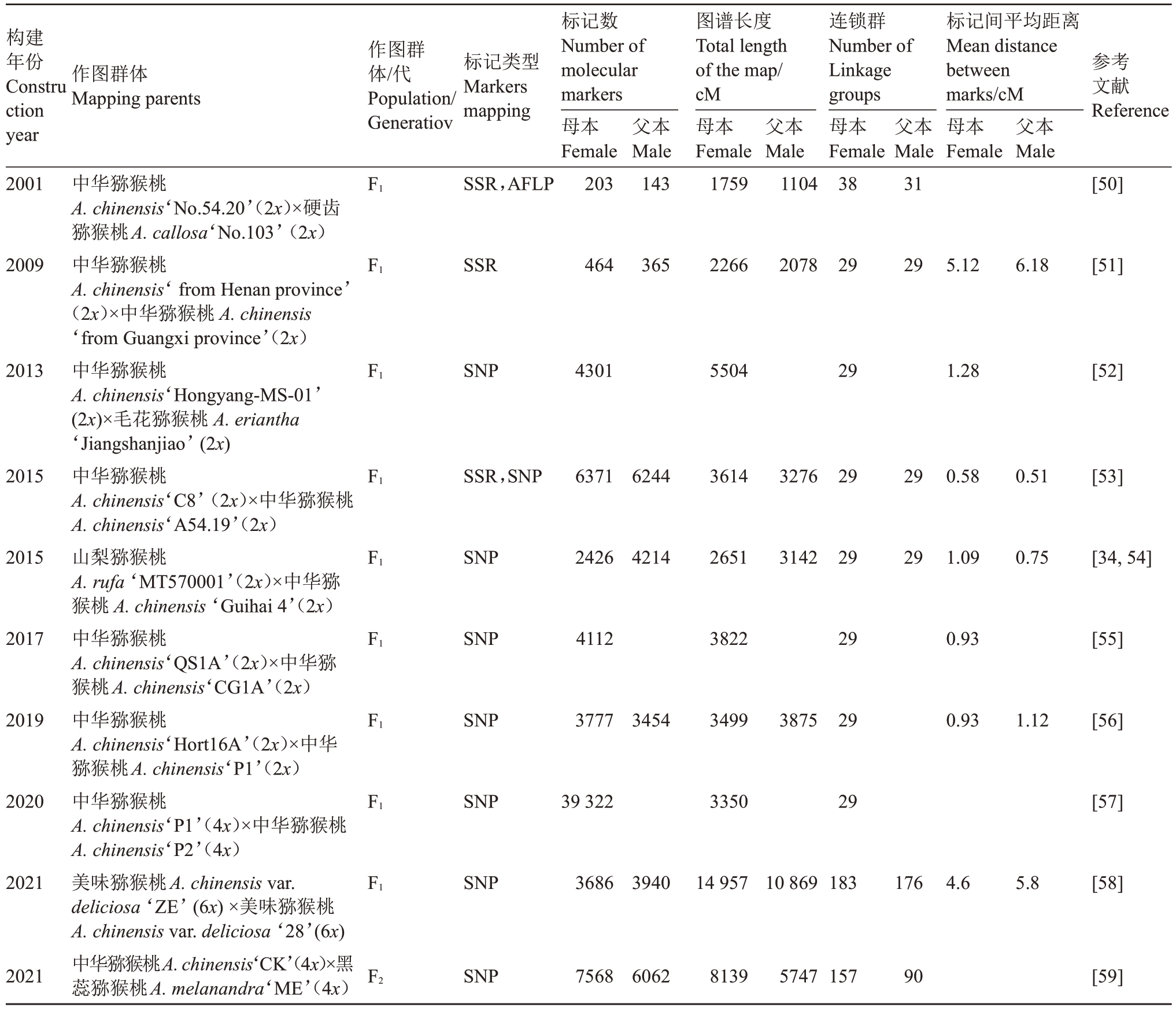
构建年份Constru ction year作图群体Mapping parents作图群体/代Population/Generatiov标记类型Markers mapping标记间平均距离Mean distance between marks/cM母本Female父本Male参考文献Reference 2001 F1 SSR,AFLP标记数Number of molecular markers母本Female 203父本Male 143图谱长度Total length of the map/cM母本Female 1759父本Male 1104连锁群Number of Linkage groups母本Female 38父本Male 31[50]2009 F1 SSR 464 365 2266 2078 29 29 5.12 6.18[51]2013 F1 SNP 4301 5504 29 1.28[52]2015 F1 SSR,SNP 6371 6244 3614 3276 29 29 0.58 0.51[53]2015 F1 SNP 2426 4214 2651 3142 29 29 1.09 0.75[34,54]2017 F1 SNP 4112 3822 29 0.93[55]2019 F1 SNP 3777 3454 3499 3875 29 0.93 1.12[56]2020 F1 SNP 39 322 3350 29[57]2021 F1 SNP 3686 3940 14 957 10 869183 176 4.6 5.8[58]2021中华猕猴桃A.chinensis‘No.54.20’(2x)×硬齿猕猴桃A.callosa‘No.103’(2x)中华猕猴桃A.chinensis‘from Henan province’(2x)×中华猕猴桃A.chinensis‘from Guangxi province’(2x)中华猕猴桃A.chinensis‘Hongyang-MS-01’(2x)×毛花猕猴桃A.eriantha‘Jiangshanjiao’(2x)中华猕猴桃A.chinensis‘C8’(2x)×中华猕猴桃A.chinensis‘A54.19’(2x)山梨猕猴桃A.rufa‘MT570001’(2x)×中华猕猴桃A.chinensis‘Guihai 4’(2x)中华猕猴桃A.chinensis‘QS1A’(2x)×中华猕猴桃A.chinensis‘CG1A’(2x)中华猕猴桃A.chinensis‘Hort16A’(2x)×中华猕猴桃A.chinensis‘P1’(2x)中华猕猴桃A.chinensis‘P1’(4x)×中华猕猴桃A.chinensis‘P2’(4x)美味猕猴桃A.chinensis var.deliciosa‘ZE’(6x)×美味猕猴桃A.chinensis var.deliciosa‘28’(6x)中华猕猴桃A.chinensis‘CK’(4x)×黑蕊猕猴桃A.melanandra‘ME’(4x)F2 SNP 7568 6062 8139 5747157 90[59]
最初构建的猕猴桃遗传连锁图谱是2001 年Testolin 等[50]利用中华猕猴桃和硬齿猕猴桃(A.callosa)杂交群体,以AFLP 和SSR 标记为框架分别构建了父、母本两张遗传图谱,但由于当时技术所限,这两张图谱密度较低,并且连锁群(LG)数目与猕猴桃属染色体基数的29 条不相同。2009 年Fraser等[51]利用644个SSR标记构建了中华猕猴桃的整合图谱,雌雄图谱均为29 个连锁群,该图谱也是迄今为止构建的密度最高的SSR 图谱,在该图谱中定位了SmX、SmY 等性别相关的标记,利用性别连锁标记鉴定出X 和Y 染色体,并且认为LG17 的亚末端是性别决定区域。第三张图谱是利用简化基因组测序方法构建的基于SNP标记的图谱,这张图谱辅助了基因组拼接过程[52],但是仍然有25%的测序序列未能组装到基因组上。第四张图谱同样利用二倍体中华猕猴桃种内杂交群体建立遗传图谱,通过双酶切简化基因组测序识别出SNP 位点和与限制性核酸内切酶识别位点相关的DNA(Restriction site Associated DNA, RAD)位点,并用其构建遗传图谱,辅助拼接了红阳参考基因组中未锚定的序列[53]。第五张图谱是Zhang 等[34]、刘春燕[54]利用RAD-seq 技术构建了山梨猕猴桃和中华猕猴桃两张种间高密度遗传图谱,该图谱能将猕猴桃基因组草图中无法组装的440 个Scafflold 锚定到基因组上,极大地提高了基因组组装水平。第六、七张图谱都是在二倍体中华猕猴桃种内杂交群体中,利用SNP 标记构建的遗传连锁图谱,Liu 等[55]将构建的图谱与参考基因组进行比对分析,发现母本图谱与参考基因组图谱之间存在高度保守性,Tahir 等[56]将构建的图谱用于后续抗溃疡病相关的QTL 定位。
随着猕猴桃遗传图谱构建的深入研究,近年来,逐步开始构建多倍体猕猴桃的高密度遗传图谱。Tahir 等[57]首次利用四倍体中华猕猴桃P1×P2为材料在杂合多倍体猕猴桃中建立了高密度SNP图谱,并表明四倍体猕猴桃多数连锁群为多倍体遗传特点、少数连锁群为二倍体遗传特点,符合节段性异源多倍体的特征。Popowski 等[58]以六倍体美味猕猴桃(A.deliciosa)ZE×28 杂交群体为材料,利用序列基因分型技术(GBS 技术)和SNP 标记构建出六倍体美味猕猴桃的高密度遗传连锁图谱,用于后续果实性状的QTL 定位;母本图谱中在29 条染色体中的17 条上拥有6 个连锁群,与预期的6 个同源染色体对应,在父本图谱中29 条染色体中的25条有6 个同源染色体。最近,Macnee 等[59]先利用四倍体黑蕊猕猴桃(A.melanandra)与四倍体中华猕猴桃杂交获取F1子代,再从中选出雌株与四倍体中华猕猴桃进行回交,并在回交群体中分别利用7568 和6062 个SNP 标记构建出母本和父本的遗传图谱,最后进行了果实性状相关的QTL 定位研究。
3 猕猴桃遗传图谱构建的QTL 定位应用
遗传图谱的应用主要是在QTL的定位和分析、分子标记辅助育种、图位克隆、比较基因组研究等方面。目前,国内外猕猴桃QTL的定位和分析主要体现在猕猴桃的生长发育特性、细菌性溃疡病抗性和果实品质形成等研究方面。
3.1 在生长发育特性方面的应用
目前,雌雄异株植物的进化研究建立了“两突变”理论模型[60-61],并得到了广泛认可,但人们对性别相关特定基因进化过程仍知之甚少。Akagi 等[62]从山梨猕猴桃和中华猕猴桃种间杂交F1代群体中定位到一个猕猴桃性别决定基因C类型的细胞分裂素响应调节因子(type-C cytokinin response regulator),该基因是心皮发育的显性抑制基因,名为Shy Girl(Sy-Gl);该基因定位于Y 染色体特异区域,其特异性复制和随后顺式元件的演化在猕猴桃性别决定基因的获取中具有重要作用。之后,Akagi等[63]又定位到第二个Y 编码的性别决定基因,将其命名为Friendly Boy(FrBy),该基因用于维持雄性功能,且独立于Sy-GI基因;Y染色体编码的SyGl和FrBy在猕猴桃性别发育中,分别作为雌性化抑制因子和雄性化维持因子独立发挥作用,这为猕猴桃雌雄同株育种提供了一种新思路。
在前述二倍体猕猴桃研究基础上,Tahir 等[57]利用四倍体猕猴桃群体雌雄花的表型数据,成功定位到一个与性别关联度较高的QTL,位于LG25 上臂9.1 cM,同时,在Y染色体上识别到一个雄性特异区域,该区域此前在二倍体猕猴桃研究中已被定位到25号连锁群Y染色体上,目前该区域被用于性别早期预测。
3.2 在细菌性溃疡病抗性方面的应用
猕猴桃细菌性溃疡病是由丁香假单胞菌(PSA)引起的病害,国内外很多猕猴桃产区均有发生,目前最根本的预防手段是进行高抗品种选育。Tahir等[56]采用中华猕猴桃高感PSA的品种Hort16A与抗性优系P1杂交建立F1代群体,通过高密度遗传图谱和强化表型分析法,在Hort16A的LG27上定位到一个控制PSA抗性的主效QTL,并在该位点上发现了敏感性和抗性的候选等位基因,在P1图谱中共定位到6个微效QTLs,分别位于LG3、14、15、22、24和28上;验证了4 个QTLs(LG27、14、22、28)的互作效应,表明Hort16A和P1 QTL的加性效应增强了F1代的抗性。最近Tahir 等[57]进一步在四倍体群体中进行了PSA抗性相关的QTL定位,采用中华猕猴桃中抗PSA的优系P1为母本与高抗优系P2杂交建立的F1代群体,构建出一张四倍体种内杂交图谱,定位到4个田间抗PSA的QTLs,分别位于图谱LG1、2、4和LG7;通过对这些QTL 贡献的分析,表明高抗亲本P2有3个与抗PSA有关的QTLs,分别位于LG1、4、7上,而抗性弱的P1在LG2上也有一个与PSA抗性相关的微效QTL;此前研究表明,二倍体中华猕猴桃中定位的抗PSA 的QTL 位于PSA 敏感品种Hort16A 的LG27 上[56],这表明敏感亲本的等位基因对PSA抗性有不同程度的作用,但在四倍体中华猕猴桃中定位的PSA 抗性QTL 与二倍体材料中定位的区域没有重叠,表明四倍体中华猕猴桃中PSA抗性的起源与猕猴桃属中任何共同原始基因位点无关;进一步评估了QTL 组合的加性效应,表明定位到的4 个QTLs 在子代中联合作用可对PSA 抗性提供更强大的数量效应。
3.3 在果实品质形成方面的应用
以果实为主要经济产品的猕猴桃,果实的风味和品质尤为重要,传统育种通过对成熟果实植物学、生物学等性状的调查分析达到筛选优良株系的目的。随着分子标记的发展和遗传图谱的完善,可以将果实的表型数据与基因型数据相结合,通过QTL定位技术,直接从分子水平找到控制优良果实性状的目的基因,从而提高育种效率和准确性。刘春燕[54]利用中华猕猴桃和山梨猕猴桃杂交F1代群体,构建了两张亲本高密度遗传图谱后,对单果质量、果实纵横经以及果糖、葡萄糖、苹果酸、奎宁酸、柠檬酸含量等12个果实性状进行了调查和检测,并结合基因型数据,利用区间作图法对上述性状进行了QTL定位,在山梨猕猴桃图谱中检测到44 个QTL 位点,中华猕猴桃图谱中检测到57个QTL位点,但遗憾的是定位的QTL都是微效QTL,未定位出主效的QTL位点。成熟的猕猴桃散发出香气是由萜类物质挥发所造成,Zeng 等[64]在二倍体中华猕猴桃种内杂交群体中,利用QTL 定位技术在图谱LG27 和LG29a 上定位到2 个与萜类物质产生相关的QTL,其中LG29a 上的QTL 位点与一个复杂的萜类合成酶(TPS)编码基因簇重合,该研究采用感官描述的判别分析方法在二倍体中华猕猴桃中定位到与萜类物质产生相关的QTL,为利用分子标记辅助选择育种改善猕猴桃风味提供了合理的方法和途径。
Popowski等[58]在六倍体美味猕猴桃种内杂交群体中,构建出双亲高密度连锁图谱,并在此基础上对单株产量、单果质量、果实硬度及干物质含量进行了QTL定位;共检测到8个与果实品质相关的QTL,其中2个来自母本(其中LG5上与单果质量相关、LG6上与干物质含量相关),6个来自父本(其中LG5、6、20 上与单株产量相关,LG13 上与单果质量相关,LG24 上与干物质含量相关,LG15 上与果实硬度相关);这是首次在六倍体猕猴桃群体中构建连锁图谱并进行QTL定位,证明了利用GBS技术在六倍体群体中进行QTL定位方法的可行性,为后续多倍体猕猴桃群体遗传研究提供了借鉴。
猕猴桃属于浆果,果实外果皮性状对其抗性、货架期及贮藏性能极为重要。大多数猕猴桃外果皮由生活细胞组成,被一层由角质素组成的蜡质角质层所覆盖,而有的是由单层木栓细胞组成的周皮,周皮是取代活性表皮的次生保护组织。为研究猕猴桃果皮类型的遗传特性,Macnee等[59]以正常表皮的黑蕊猕猴桃为父本与具有周皮层的中华猕猴桃为母本杂交获得了F1子代,由于F1代雌株果实全部具有正常活性表皮,因此,从F1代中选出雌株与具有周皮的中华猕猴桃回交,得到了具有周皮与正常表皮分离的回交群体,并对回交群体进行果皮相关性状的QTL定位;在父母本图谱上定位到了全果活性表皮、全果周皮、角质层厚度、角质层覆盖率、褐变等性状相关的QTL,通过对果实表皮类型分离及QTL定位分析可知,猕猴桃果皮发育不仅受角质层强度的影响还和表皮的褐变倾向有关,两者为竞争关系,坚硬的角质层可以抑制褐变,而强烈的褐变倾向可以形成连续的周皮层;另外,还对果皮被毛数量和密度、果实长度等相关性状进行了QTL 定位。该项研究是目前在猕猴桃果实性状方面完成度最高的QTL 定位研究,摸清了猕猴桃果皮类型的遗传规律及分子机制,并找到一个控制褐斑产生的候选基因,证明了与猕猴桃果实周皮形成相关的QTL与褐斑形成相关,也与角质层强度相关。
4 展 望
目前在猕猴桃属植物中已经发现不少与育种目标性状紧密连锁的分子标记,但有些标记的多态性仍然较低,并且之前的研究大都将目的基因的分子标记与育种工作分开进行,无法很好地应用于实践。今后应不断提高分子标记的稳定性和多态性,加大新型分子标记的开发力度,同时应将分子标记技术的开发与常规育种相结合进行,使其尽快服务于育种工作。
相较于大田作物而言,猕猴桃遗传连锁图谱的研究起步较晚,虽已构建了一些遗传图谱,但相比之下密度低、数量少,并且构建成本较高。其中影响图谱质量的主要因素为作图群体的类型,猕猴桃染色体基数大、倍性复杂、杂合度高、世代周期长,雌雄异株而且雄株性状早期不可预见,因此构建高层次、数量较大且亲本间差异较为适合的作图群体较为困难。今后应注重雄株性状早期预判研究并应用于杂交父本选择,同时将杂交育种与图谱构建相结合,针对猕猴桃属植物特点,大规模开发具有自主知识产权的分子标记,以建立密度高、分布均匀、实用价值高的猕猴桃分子标记连锁图谱。
目前常用的基因分型平台有测序和基因芯片。基因芯片只能检测已知的序列特征,是一个“封闭”的系统;高通量DNA测序可以发现和寻找新的信息,是一个“开放”的系统。但基因芯片杂交结果快速、直观,目前已经拥有完整的标准化流程,可以检测多倍体基因型的拷贝剂量,短时间内可对大量样品进行检测。因此,今后可将两种优势互补的高通量方法联合利用,以解决目前多倍体猕猴桃基因分型难的问题,为高效构建遗传连锁图谱提供技术支撑。
近年来分子标记技术的发展为深入研究数量性状的遗传规律创造了条件,但客观讲目前关于猕猴桃数量性状的系统性研究仍处于探索阶段。除群体构建的因素,最主要的原因就是对于多倍体猕猴桃现有的作图分析方法和定位到的QTL 位点不够准确,目前基于线性模型计算出的数量性状基因表达能力不够完整,即某一数量性状实际可能是多个性状的综合表现。针对上述问题,今后一段时间猕猴桃QTL 研究的主要内容应该包括:选择更合理的数学遗传模型来进行QTL 作图分析,开展QTL 精细定位与基因测序、克隆、功能验证相结合等等。
[1]LI J Q,LI X W,SOEJARTO D D.A revision of the genus Actin-idia from China[C].Acta Horticulturae,2007,753(1):41-44.
[2]WARRINGTON I J,WESTON G C.Kiwifruits:science and management[M].Auckland:Ray Riehards Publisher,1990:183-204.
[3]齐秀娟,郭丹丹,王然,钟云鹏,方金豹.我国猕猴桃产业发展现状及对策建议[J].果树学报,2020,37(5):754-763.QI Xiujuan,GUO Dandan,WANG Ran,ZHONG Yunpeng,FANG Jinbao.Development status and suggestions on Chinese kiwifruit industry[J].Journal of Fruit Science,2020,37(5):754-763.
[4]徐小彪,张秋明.中国猕猴桃种质资源的研究与利用[J].植物学通报,2003,20(6):648-655.XU Xiaobiao,ZHANG Qiuming.Researches and utilizations of germplasm resource of kiwifruit in China[J].Chinese Bulletin of Botany,2003,20(6):648-655.
[5]李作洲.猕猴桃属植物的分子系统学研究[D].武汉:中国科学院研究生院(武汉植物园),2006.LI Zuozhou.Molecular phylogeny of the genus Actinidia based on nuclear DNA genetic markers and cytoplasm DNA sequence analysis[D].Wuhan:Chinese Aeademy of Sciences(Wuhan Botanical Garden),2006.
[6]卢立媛,刘振盼,孙阳,张永华,尤文忠.软枣猕猴桃研究进展[J].特产研究,2020,42(5):89-93.LU Liyuan,LIU Zhenpan,SUN Yang,ZHANG Yonghua,YOU Wenzhong.Research progress of kiwiberry[J].Special Wild Economic Animal and Plant Research,2020,42(5):89-93.
[7]张敏,王贺新,娄鑫,赵丽娜,闫东玲.世界软枣猕猴桃品种资源特点及育种趋势[J].生态学杂志,2017,36(11):3289-3297.ZHANG Min,WANG Hexin,LOU Xin,ZHAO Lina,YAN Dongling.The development status and breeding trend of hardy kiwifruit cultivars in the world[J].Chinese Journal of Ecology,2017,36(11):3289-3297.
[8]朱德威,陈庆富.普通小麦遗传图谱研究现状与展望[J].种子,2010,29(3):64-69.ZHU Dewei,CHEN Qingfu.Status and prospect of common wheats’genetic maps[J].Seed,2010,29(3):64-69.
[9]张校立,艾沙江·买买提,薛华柏,闫鹏,王继勋.梨遗传连锁图谱研究进展[J].果树学报,2018,35(S1):1-8.ZHANG Xiaoli,Aishajiang · Maimaiti,XUE Huabai,YAN Peng,WANG Jixun.Recent advances in research on the genetic linkage maps in pears[J].Journal of Fruit Science,2018,35(S1):1-8.
[10]邹自征,陈建华,栾明宝,郭劲霞,王超,王晓飞,许英,孙志民.应用RSAP、SRAP 和SSR 分析苎麻种质亲缘关系[J].作物学报,2012,38(5):840-847.ZOU Zizheng,CHEN Jianhua,LUAN Mingbao,GUO Jinxia,WANG Chao,WANG Xiaofei,XU Ying,SUN Zhimin.Genetic relationship of ramie germplasm revealed by RSAP,SRAP,and SSR markers[J].Acta Agronomica Sinica,2012,38(5):840-847.
[11]董晓莉,汤浩茹,甘玲,李明章.DNA 分子标记在猕猴桃上的应用[J].果树学报,2005,22(6):682-686.DONG Xiaoli,TANG Haoru,GAN Ling,LI Mingzhang.Advances in research on application of DNA molecular markers in Actinidia[J].Journal of Fruit Science,2005,22(6):682-686.
[12]郭丹丹.软枣猕猴桃(Actinidia arguta)性别相关分子标记的开发及应用[D].北京:中国农业科学院,2019.GUO Dandan.Development and application of the sex-linked molecular maekers in Actinidia arguta[D].Beijing:Chinese Academy of Agricultural Sciences,2019.
[13]WEISING K,FUNG R W M,KEELING D J,ATKINSON R G,GARDNER R C.Characterisation of microsatellites from Actinidia chinensis[J].Molecular Breeding,1997,3(2):159-160
[14]HUANG W G,CIPRIANI G,MORGANTE M,TESTOLIN R.Microsatellite DNA in Actinidia chinensis:isolation,characterisation,and homology in related species[J].Theoretical and Applied Genetics,1998,97(8):1269-1278.
[15]郑轶琦,李作洲,黄宏文.猕猴桃品种SSR 分析的初步研究[J].武汉植物学研究,2003,21(5):444-448.ZHENG Yiqi,LI Zuozhou,HUANG Hongwen.Preliminary study on SSR analysis in kiwifruit cultivars[J].Journal of Wuhan Botanical Research,2003,21(5):444-448.
[16]姜春芽,徐小彪,廖娇,辜青青,刘善军,陈金印.基于EST 的猕猴桃SSR 标记的建立[J].果树学报,2010,27(4):622-625.JIANG Chunya,XU Xiaobiao,LIAO Jiao,GU Qingqing,LIU Shanjun,CHEN Jinyin.Establishment of SSR markers based on EST database from Actinidia[J].Journal of Fruit Science,2010,27(4):622-625.
[17]王丹丹,张彦文.软枣猕猴桃栽培品种DNA 指纹图谱的构建及遗传多样性分析[J].东北师大学报(自然科学版),2017,49(3):104-111.WANG Dandan,ZHANG Yanwen.Establishment of DNA fingerprinting and analysis of genetic diversity among Actinidia arguta cultivars[J].Journal of Northeast Normal University(Natural Science Edition),2017,49(3):104-111.
[18]汤佳乐,黄春辉,吴寒,郎彬彬,曲雪艳,徐小彪.野生毛花猕猴桃果实表型性状及SSR 遗传多样性分析[J].园艺学报,2014,41(6):1198-1206.TANG Jiale,HUANG Chunhui,WU Han,LANG Binbin,QU Xueyan,XU Xiaobiao.Genetic diversity of wild Actinidia eriantha germplasm based on fruit traits and SSR markers[J].Acta Horticulturae Sinica,2014,41(6):1198-1206.
[19]邹梁峰,廖光联,李西时,黄春辉,钟敏,陶俊杰,曲雪艳,徐小彪.72 份猕猴桃种质基于SSR 标记的聚类分析及指纹图谱构建[J].中国南方果树,2019,48(1):83-88.ZOU Liangfeng,LIAO Guanglian,LI Xishi,HUANG Chunhui,ZHONG Min,TAO Junjie,QU Xueyan,XU Xiaobiao.Cluster analysis and fingerprinting of 72 kiwifruit germplasm based on SSR markers[J].South China Fruits,2019,48(1):83-88.
[20]邹游,丁建,申瑛,赵建,杨志荣,吴成.11 个猕猴桃品种间的遗传多样性分析[J].应用与环境生物学报,2007,13(2):172-175.ZOU You,DING Jian,SHEN Ying,ZHAO Jian,YANG Zhirong,WU Cheng.Analysis of random amplified polymorphic DNA in 11 Actinidia varieties[J].Chinese Journal of Applied and Environmental Biology,2007,13(2):172-175.
[21]陈延惠,李洪涛,朱道圩,胡青霞,萧蓉萍,薛淑丽,李静.RAPD 分子标记在猕猴桃种质资源鉴定上的应用[J].河南农业大学学报,2003,37(4):360-364.CHEN Yanhui,LI Hongtao,ZHU Daoyu,HU Qingxia,XIAO Rongping,XUE Shuli,LI Jing.Application of RAPD molecular marker on identification of germplasm resources in Actinidia[J].Journal of Henan Agricultural University,2003,37(4):360-364.
[22]龚俊杰.猕猴桃属植物AFLP 分析及其系统发育关系的研究[D].武汉:华中农业大学,2003.GONG Junjie.Phylogenetic relationships among taxa of the genus Actinidia based on AFLP marks[D].Wuhan:Huazhong Agricultural University,2003.
[23]陈华.AFLP 及其在猕猴桃种质鉴别与分类上的应用[J].肇庆学院学报,2007,28(5):60-63.CHEN Hua.AFLP and its application in germplasm identification and classification of Actinidia[J].Journal of Zhaoqing University,2007,28(5):60-63.
[24]张安世,韩臣鹏,齐秀娟,张中海.基于ISSR 标记的猕猴桃品种遗传多样性分析及指纹图谱构建[J].植物资源与环境学报,2017,26(3):19-26.ZHANG Anshi,HAN Chenpeng,QI Xiujuan,ZHANG Zhonghai.Genetic diversity analysis and fingerprinting construction of cultivars of Actinidia spp.based on ISSR marker[J].Journal of Plant Resources and Environment,2017,26(3):19-26.
[25]邹游,黄敏,侯若彤,吴成,杨志荣.ISSR 标记技术在猕猴桃遗传研究中的运用[J].西南师范大学学报(自然科学版),2008,33(1):111-115.ZOU You,HUANG Min,HOU Ruotong,WU Cheng,YANG Zhirong.Analysis on inter-simple sequence repeats in 14 Actinidia varieties[J].Journal of Southwest China Normal University(Natural Science Edition),2008,33(1):111-115.
[26]王卫.猕猴桃遗传多样性的SRAP 分析及GPX 和cAPX 基因片段的克隆[D].福州:福建师范大学,2013.WANG Wei.Using SRAP markers to analyze genetic diversity of kiwifruit and cloning the gene of GPX and cAPX[D].Fuzhou:Fujian Normal University,2013.
[27]张安世,司清亮,齐秀娟,张中海.猕猴桃种质资源的SRAP遗传多样性分析及指纹图谱构建[J].江苏农业学报,2018,34(1):138-144.ZHANG Anshi,SI Qingliang,QI Xiujuan,ZHANG Zhonghai.Genetic diversity and fingerprints of Actinidia germplasm resource based on SRAP markers[J].Jiangsu Journal of Agricultural Sciences,2018,34(1):138-144.
[28]井赵斌,徐明,雷玉山.猕猴桃SRAP-PCR 体系的建立及品种资源亲缘关系研究[J].园艺学报,2016,43(2):337-346.JING Zhaobin,XU Ming,LEI Yushan.Construction and application of SRAP-PCR system to analyze genetic relationship of Actinidia[J].Acta Horticulturae Sinica,2016,43(2):337-346
[29]周锦,刘义飞,黄宏文.基于EST 数据库进行SNP 分子标记开发的研究进展及在猕猴桃属植物中的应用研究[J].热带亚热带植物学报,2011,19(2):184-194.ZHOU Jin,LIU Yifei,HUANG Hongwen.Progress on development of EST derived SNP markers and its applications in Actinidia chinensis species complex[J].Journal of Tropical and Subtropical Botany,2011,19(2):184-194.
[30]张安世,张中海,齐秀娟,刘莹,骆扬.猕猴桃SCoT 遗传多样性分析及指纹图谱的构建[J].植物研究,2017,37(2):259-265.ZHANG Anshi,ZHANG Zhonghai,QI Xiujuan,LIU Ying,LUO Yang.Genetic diversity and fingerprints with SCoT markers in Actinidia[J].Bulletin of Botanical Research,2017,37(2):259-265.
[31]LI H L.A taxonomic review of the genus Actinidia[J].Journal of the Arnold Arboretum,1952,33(1):1-61.
[32]HARVEY C F,GILL G P,FRASER L G,MCNELIAGE M A.Sex determination in Actinidia.1.Sex-linked markers and progeny sex ratio in diploid A.chinensis[J].Sexual Plant Reproduction,1997,10(3):149-154.
[33]GILL G P,HARVEY C F,GARDNER R C,FRASER L G.Development of sex-linked PCR markers for gender identification in Actinidia[J].Theoretical and Applied Genetics,1998,97(3):439-445.
[34]ZHANG Q,LIU C Y,LIU Y F,VANBUREN R,YAO X H,ZHONG C H,HUANG H W.High-density interspecific genetic maps of kiwifruit and the identification of sex-specific markers[J].DNA Research,2015,22(5):367-375.
[35]郭丹丹,钟云鹏,方金豹,黄武权,任建杰,齐秀娟.猕猴桃性别分子标记在软枣猕猴桃中的通用性验证[J].果树学报,2019,36(5):549-556.GUO Dandan,ZHONG Yunpeng,FANG Jinbao,HUANG Wuquan,REN Jianjie,QI Xiujuan.Validation of kiwifruit sex molecular markers in Actinidia arguta[J].Journal of Fruit Science,2019,36(5):549-556.
[36]徐小彪,姜春芽,廖娇,辜青青,刘善军,陈金印.中华猕猴桃矮型性状EST-SSR 连锁标记的筛选[J].园艺学报,2010,37(4):553-558.XU Xiaobiao,JIANG Chunya,LIAO Jiao,GU Qingqing,LIU Shanjun,CHEN Jinyin.Screening of EST-SSR marker linked to dwarf character in Actinidia chinensis planch[J].Acta Horticulturae Sinica,2010,37(4):553-558.
[37]易盼盼,樊红科,雷玉山,王飞.猕猴桃抗溃疡病基因连锁SSR 分子标记初步研究[J].西北农林科技大学学报(自然科学版),2015,43(4):91-98.YI Panpan,FAN Hongke,LEI Yushan,WANG Fei.Priliminary study on SSR marker of gene linkage against Pseudomonas ayringae pv. actinidae[J].Journal of Northwest A&F University(Natural Science Edition),2015,43(4):91-98.
[38]汤佳乐,吴寒,郎彬彬,曲雪艳,黄春辉,徐小彪.野生毛花猕猴桃叶片和果实AsA 含量的SSR 标记关联分析[J].园艺学报,2014,41(5):833-840.TANG Jiale,WU Han,LANG Binbin,QU Xueyan,HUANG Chunhui,XU Xiaobiao.Association analysis on leaf and fruit AsA content and SSR markers of wild Actinidia eriantha[J].Acta Horticulturae Sinica,2014,41(5):833-840.
[39]孙世航,林苗苗,齐秀娟,孙雷明,钟云鹏,方金豹.应用InDel标记进行软枣猕猴桃杂交子代真实性鉴定[J].果树学报,2018,35(1):32-37.SUN Shihang,LIN Miaomiao,QI Xiujuan,SUN Leiming,ZHONG Yunpeng,FANG Jinbao.Application of InDel markers on progeny identification in Actinidia arguta[J].Journal of Fruit Science,2018,35(1):32-37.
[40]郑成超,温孚江.DNA 分子标记技术与作物品种纯度鉴定[J].山东农业大学学报,1997,28(4):499-505.ZHENG Chengchao,WEN Fujiang.Application prospects of DNA molecular markers in identifying the purity of crop seeds[J].Journal of Shandong Agricultural University,1997,28(4):499-505.
[41]辛景树,郭景伦,张软斌.几种常用分子标记技术在种子纯度和品种真实性鉴定方面的比较与分析[J].种子,2005,24(1):58-60.XIN Jingshu,GUO Jinglun,ZHANG Ruanbin.Comparison and analysis of several commonly used molecular marker techniques for seed purity and varietal authenticity identification[J].Seed,2005,24(1):58-60.
[42]王丹丹,杨东霞.8 种软枣猕猴桃种质纯度鉴定[J].西北农业学报,2016,25(11):1656-1662.WANG Dandan,YANG Dongxia.Purity research and analysis of eight Actinidia arguta cultivars[J].Acta Agriculturae Borealioccidentalis Sinica,2016,25(11):1656-1662.
[43]中华人民共和国农业农村部.中华猕猴桃品种鉴定SSR 分子标记法.NY/T 3639—2020[S].北京:中国农业出版社,2020.Ministry of Agriculture and Rural Affairs of the People’s Republic of China.Identification of Actinidia chinensis cultivars using SSR markers method.NY/T 3639—2020[S].Beijing:China Agriculture Press,2020.
[44]王霖.小麦遗传连锁图谱构建及主要农艺和品质性状QTL 定位[D].泰安:山东农业大学,2012.WANG Lin.Construction of wheat molecular genetic map and QTL analysis for agronomic and quanlity traits[D].Taian:Shandong Agricultural University,2012.
[45]张坤普.小麦分子遗传图谱的构建及数量性状基因定位[D].泰安:山东农业大学,2008.ZHANG Kunpu.Construction of wheat molecular genetic map and QTL analysis[D].Taian:Shandong Agricultural University,2008.
[46]方宣钧,吴为人,唐纪良.作物DNA 标记捕助育种[M].北京:科学出版社,2000,30-55.FANG Xuanjun,WU Weiren,TANG Jiliang.Crop DNA marker trapping for breeding[M].Beijing:Science Press,2000,30-55.
[47]周元昌,陈启锋,吴为人,李维明.作物QTL 定位研究进展[J].福建农业大学学报,2000,29(2):138-144.ZHOU Yuanchang,CHEN Qifeng,WU Weiren,LI Weiming.A review of QTL research in crops[J].Journal of Fujian Agricultural University,2000,29(2):138-144.
[48]MCCOUCH S R,KOCHERT G,YU Z H,WANG Z Y,KHUSH G S,COFFMAN W R,TANKSLEY S D.Molecular mapping of rice chromosomes[J].Theoretical and Applied Genetics,1988,76(6):815-829.
[49]WU J Z,MAEHARA T,SHIMOKAWA T,YAMAMOTO S,HARADA C,TAKAZAKI Y,ONO N,MUKAI Y,KOIKE K,YAZAKI J,FUJII F,SHOMURA A,ANDO T,KONO I,WAKI K,YAMAMOTO K,YANO M,MATSUMOTO T,SASAKI T.A comprehensive rice transcript map containing 6591 expressed sequence tag sites[J].Plant Cell,2002,14(3):525-535.
[50]TESTOLIN R,HUANG W G,LAIN O,MESSINA R,VECCHIONE A,CIPRIANI G.A kiwifruit (Actinidia spp.) linkage map based on microsatellites and integrated with AFLP markers[J].Theoretical and Applied Genetics,2001,103(1):30-36.
[51]FRASER L G,TSANG G K,DATSON P M,SILVA H N D,HARVEY C F,GILL G P,CROWHURST R N,MCNEILAGE M A.A gene-rich linkage map in the dioecious species Actinidia chinensis (kiwifruit) reveals putative X/Y sex-determining chromosomes[J].BMC Genomics,2009,10(1):102.
[52]HUANG S X,DING J,DENG D J,TANG W,SUN H H,LIU D Y,ZHANG L,NIU X L,ZHANG X,MENG M,YU J E,LIU J,HAN Y,SHI W,ZHANG D F,CAO S Q,WEI Z J,CUI Y L,XIA Y H,ZENG H P,BAO K,LIN L,MIN Y,ZHANG H,MIAO M,TANG X F,ZHU Y Y,SUI Y,LI G W,SUN H J,YUE J Y,SUN J Q,LIU F F,ZHOU L Q,LEI L,ZHENG X Q,LIU M,HUANG L,SONG J,XU C H,LI J W,YE K Y,ZHONG S L,LU B R,HE G H,XIAO F M,WANG H L,ZHENG H K,FEI Z J,LIU Y S.Draft genome of the kiwifruit Actinidia chinensis[J].Nature Communications,2013,4(4):2640.
[53]SCAGLIONE D,FORNASIERO A,PINTO C,CATTONARO F,SPADOTTO A,INFANTE R,MENESES C,MESSINA R,LAIN O,CIPRIANI G,TESTOLIN R.A RAD-based linkage map of kiwifruit ( Actinidia chinensis Pl.) as a tool to improve the genome assembly and to scan the genomic region of the gender determinant for the marker-assisted breeding[J].Tree Genetics&Genomes,2015,11(6):115.
[54]刘春燕.猕猴桃种间高密度遗传图谱的构建及果实性状QTLs 定位[D].武汉:中国科学院研究生院(武汉植物园),2016.LIU Chunyan.Construction of high-density interspecific genetic maps and identification of QTLs for fruits in kiwifruit[D].Wu-han:Chinese Aeademy of Sciences(Wuhan Botanical Garden),2016.
[55]LIU C Y,LI D W,ZHOU J H,ZHANG Q,TIAN H,YAO X H.Construction of a SNP-based genetic linkage map for kiwifruit using next-generation restriction-site-associated DNA sequencing(RADseq)[J].Molecular Breeding,2017,37(11):139.
[56]TAHIR J,HOYTE S,BASSETT H,BRENDOLISE C,CHATTERJEE A,TEMPLETON K,DENG C,CROWHURST R,MONTEFIORI M,MORGAN E,WOTTON A,FUNNELL K,WIEDOW C,KNAEBEL M,HEDDERLEY D,VANNESTE J,MCCALLUM J,HOEATA K,NATH A,CHAGNÉ D,GEA L,GARDINER S E.Multiple quantitative trait loci contribute to resistance to bacterial canker incited by Pseudomonas syringae pv. actinidiae in kiwifruit (Actinidia chinensis)[J].Horticulture Research,2019,6(1):101.
[57]TAHIR J,BRENDOLISE C,HOYTE S,LUCAS M,THOMSON S,HOEATA K,MCKENZIE C,WOTTON A,FUNNELL K,MORGAN E,HEDDERLEY D,CHAGNÉ D,BOURKE P M,MCCALLUM J,GARDINER S E,GEA L.QTL Mapping for resistance to cankers induced by Pseudomonas syringae pv.Actinidiae(Psa)in a tetraploid Actinidia chinensis kiwifruit population[J].Pathogens,2020,9(11):967.
[58]POPOWSKI E,THOMOSON S J,KNÄBEL M,TAHIR J,CROWHURST R N,DAVY M,FOSTER T M,SCHAFFER R J,TUSTIN D S,ALLAN A C,MCALLUM J,CHAGNÉ D.Construction of a high-density genetic map for hexaploid kiwifruit (Actinidia chinensis var. deliciosa) using genotyping by sequencing[J].G3-Genes|Genomes|Genetics,2021,11(7):142.
[59]MACNEE N,HILARIO E,TAHIR J,CURRIE A,WARREN B,REBSTOCK R,HALLETT I C,CHAGNÉ D,SCHAFFER R J,BULLEY S M.Peridermal fruit skin formation in Actinidia sp.(kiwifruit) is associated with genetic loci controlling russeting and cuticle formation[J].BMC Plant Biology,2021,21(1):334.
[60]CHARLESWORTH B,CHARLESWORTH D.A model for the evolution of dioecy and gynodioecy[J].The American Naturalist,1978,112(988):975-997.
[61]WESTERGAARD M.The mechanism of sex determination in dioecious flowering plants[J].Advances in Genetics,1958,9:217-281.
[62]AKAGI T,HENRY I M,OHTANI H,MORIMOTO T,BEPPU K,KATAOKA I,TAO R.A Y-encoded suppressor of feminization arose via lineage-specific duplication of a cytokinin response regulator in kiwifruit[J].The Plant Cell,2018,30(4):780-795.
[63]AKAGI T,PILKINGTON S M,VARKONYI-GASIC E,HENRY I M,SUGANO S S,SONODA M,FIRL A,MCNEILAGE M A,DOUGLAS M J,WANG T C,REBSTOCK R,VOOGD C,DATSON P,ALLAN A C,BEPPU K,KATAOKA I,TAO R.Two Y-chromosome-encoded genes determine sex in kiwifruit[J].Nature Plants,2019,5(8):801-809.
[64]ZENG Y L,WANG M Y,HUNTER D C,MATICH A J,MCATEE P A,KNÄBEL M,HAMIAUX C,POPWSKI E A,JAEGER S R,NIEUWENHUIZEN N J,YAUK Y K,ATKINSON R G.Sensory-directed genetic and biochemical characterization of volatile terpene production in kiwifruit[J].Plant Physiology,2020,183(1):51-66.