杏(Prunus armeniaca L.)在我国广泛栽植,栽培面积和产量均居世界首位。杏果实营养丰富,风味独特,深受消费者青睐。作为以鲜食为主的果树,生产优质、无病的果品是杏树栽培的首要目标。然而,在杏果实发育过程中由于经常受到各种病菌侵染,尤其是褐腐病、软腐病、白粉病、疮痂病和炭疽病等,严重影响果实外观和食用品质,降低商品价值[1]。目前,杏果实病害研究主要停留在果实生理机制、栽培管理措施等方面,对于果实抗病性的分子机制鲜有研究,阻碍了杏抗病品种的选择和遗传改良进程[2]。近年来,在多种植物中克隆到一类广谱的抗病基因家族NBS(Nucleotide Binding Site,NBS),其共同特点是编码的蛋白一般具有核苷酸结合位点,在植物识别和抵御病原方面起着非常重要的作用,因而成为植物抗病研究的重要方向[3]。在香蕉中克隆的RGAs(Resistance gene analogues)属于无TIR结构域(Toll-interleukin-1 receptor domain)的NBS基因,对香蕉抗病具有重要作用[4]。在猕猴桃中,NBS 基因所编码的RPS2(Resistance to Pseudomonas syringae 2)和RPS5(Resistance to Pseudomonas syringae 5)蛋白对丁香假单胞杆菌具有抗性,而该菌是猕猴桃细菌性溃疡病的关键致病菌[5-6]。NBS基因编码的RPP8(Recognition of Peronospora parasitica 8)蛋白与RPS5蛋白分别与苹果抗褐斑病和桃树抗流胶病有关[7]。可见,NBS 基因家族在植物抗病机制中发挥着重要作用,开展NBS基因家族鉴定将为果树抗病育种揭开崭新的一页,但受到全基因组序列信息的准确性和可获取性等方面的局限,果树这方面研究的进展较为缓慢。
杏基因组测序与组装的完成[8],为进行基因家族鉴定、分析和功能预测奠定了基础,也为深入研究杏果实抗病的分子机制提供了必要条件。笔者在本研究中选择果实抗病性存在差异的2个杏品种开展NBS基因家族对果实抗病性影响的研究,应用生物信息学的方法对NBS 基因进行鉴定、分类、调控模式与功能预测,并对2 个品种不同发育时期转录组进行分析,研究NBS相关基因差异表达与果实抗病性之间的关系,为探索杏果实抗病的分子机制提供理论指导,并为后续杏果实抗病育种奠定理论基础。
1 材料和方法
1.1 数据获取及NBS基因家族鉴定
分别在蔷薇科数据库GDR和Pfam数据库中下载串枝红杏基因组[8]序列、蛋白序列及注释文件和NB-ARC(PF00931)hmm 模型文件,用于NBS 基因家族鉴定。基因通常具有多种剪接形式,在本研究中选取最长转录本蛋白序列作为代表进行NBS 基因鉴定。使用Hmmsearch在蛋白质序列中搜索NBARC 结构域,选取E-value <1 e-50的蛋白序列使用MAFFT 进行多序列比对(最大迭代数设置为1000)[9],构建杏中NBS的结构域模型,应用该模型再次进行鉴定,选取E-value<1 e-50的蛋白序列作为NBS 基因家族的蛋白序列。为避免遗漏部分NBS基因,将鉴定到的NBS蛋白序列与杏基因组中所有基因的代表蛋白质序列进行blast比对,选取长度为300~2000 aa、相似度>80%、长度覆盖度>80%的蛋白序列。最终,将上述2 种方法获得的序列取并集作为NBS 基因的最终结果。将抗病基因的蛋白质序列通过NCBI CDD[10]数据库和Pfam 数据库进行比对,手动剔除不含NB-ARC 结构域的蛋白序列。以上NBS 基因家族鉴定过程使用的软件参数除特别说明外,均采用默认值。
1.2 NBS 基因进化、顺式作用元件与基序(motif)分析
为探索杏NBS抗病基因的进化关系,选取拟南芥NBS 基因作为外群构建杏NBS 基因家族的系统发育树。将NBS 蛋白序列使用MAFFT、采用默认参数进行多序列比对,最大迭代数设置为1000,利用JTT+F+R9 模型,构建分子进化树[11],Bootstrap 设置为1000,使用iTOL 进行可视化[12]。为评估NBS基因在进化过程所受的选择压力,将抗病基因蛋白质序列进行All-vs-All的Blastp比对,选择序列相似性和覆盖度>80%的结果作为同源基因对,然后使用KaKs Calculator的YN 模型计算这些旁系同源抗病基因对的Ka 和Ks[13]。为了解NBS 基因家族的表达调控机制,提取起始密码子上游2000 bp 范围序列,使用PlantCARE 进行顺式作用元件预测[14]。使用MEME 对抗病蛋白的Motif 进行预测[15]。以上软件除特别说明以外,均采用默认参数。
1.3 杏果实不同发育时间NBS基因家族表达分析
从国家基因库生命大数据平台下载串枝红和大白杏果实不同发育时期的转录组数据,转录组数据使用Fastp 进行质控[16],经Hisat2 比对至串枝红杏参考基因组[17],使用FeastureCounts获得各处理的表达矩阵[18],使用DESeq2 对NBS 基因进行差异表达分析[19]。以上软件均采用默认参数。
2 结果与分析
2.1 NBS基因家族结构域及进化分析
NBS抗病基因通常包括3个结构域:N端的CC(coiled-coil)、NBS(nucleotide binding site)和C 端的LRR(leucine-rich-repeat)结构域,在杏NBS 基因家族中,除了具有以上结构域外,还存在其他的结构域,包括RPW8、SRF-TF、SH3_15、NACHT、ABC2_membrane、CSTF_C、K-box、Oxidored_FMN、Colipase-like 等结构域,其中,PARG07386 和PARG27090 基因中含有RPW8 结构域,含有此结构域的基因对白粉病菌具有广谱抗性[5]。
NBS 基因家族进化树结果表明,根据N 端保守结构域的不同,可分为NL 型和CNL 型2类,其中NL 型29个,CNL 型126个,CNL 型抗病基因多于NL型,而CNL 型抗病基因根据序列的差异程度可进一步分为5个类型,CNL1、CNL2、CNL3、CNL4和CNL5(图1)。以拟南芥中NBS基因作为外群,对杏中NBS 基因进化关系分析发现,NL 类型抗病基因在进化上早于CNL 类型,而在CNL 类型中,含有TIR(toll interleukin-1 receptor)结构域的NBS 基因(PARG08776与PARG20738)与其他4个类型相比更古老。
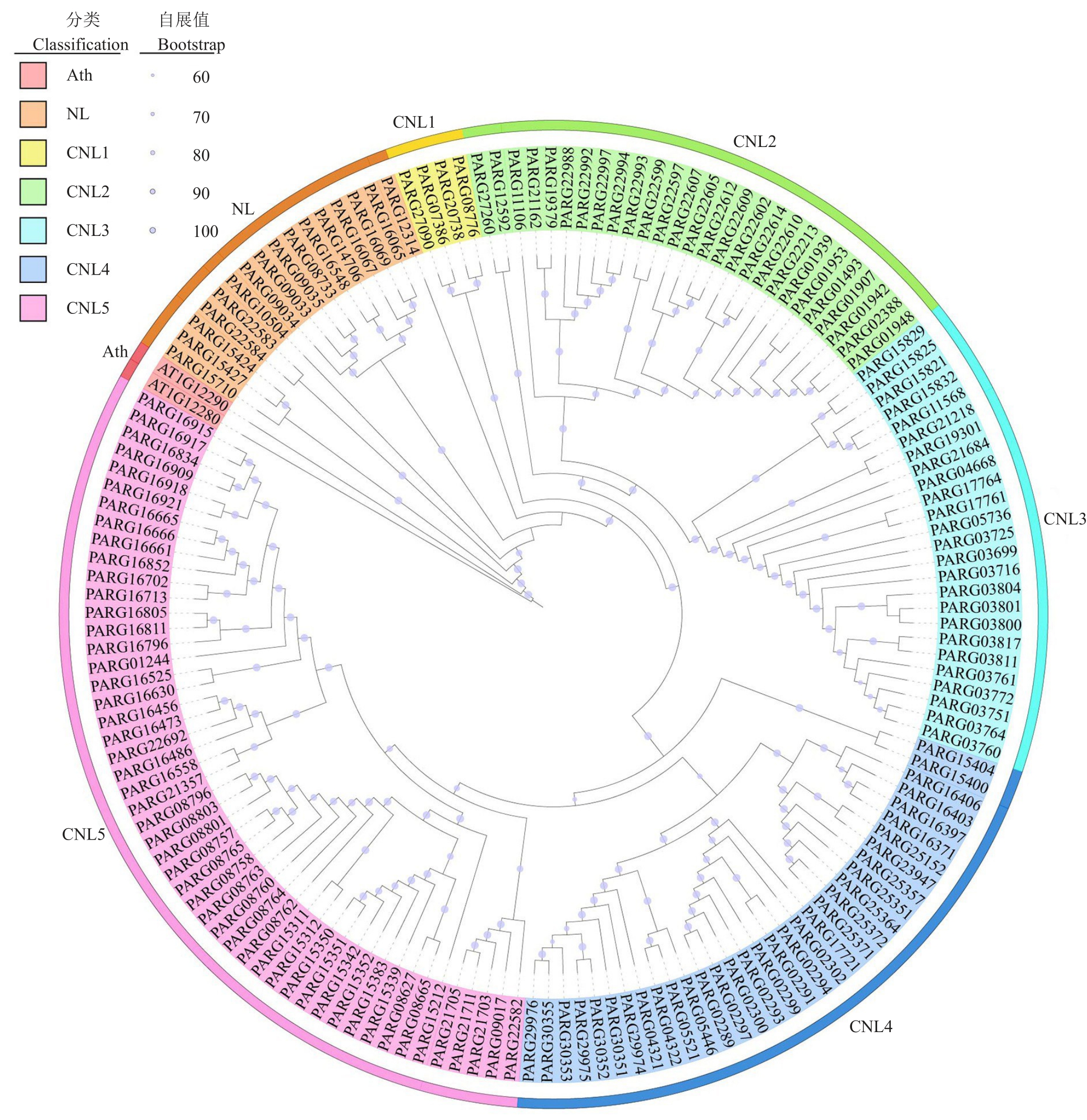
图1 杏NBS 基因家族系统发育树
Fig.1 Phylogenetic tree of apricot NBS gene family
2.2 NBS基因家族染色体定位及功能注释
NBS 基因在基因组上的分布规律如图2 所示,NBS 基因家族主要分布在1 号与2 号染色体上,分别包含39 个和31 个NBS 基因,共占到所有NBS 基因的45%,表明1号和2号染色体对杏抵御病害的贡献较大。不同类型的NBS 基因分布也存在差异,CNL型抗病基因主要分布在2号染色体上,而NL型主要在3 号染色体上,说明2 号染色体上的NBS 基因与3号染色体相比形成时间更晚。NBS基因大部分以基因簇的形式存在,其中,CNL型(44%)成簇存在的比率显著高于NL 型(21%),表明CNL 型抗病基因与NL 型相比更倾向于协同表达,有效抵御病原侵害。

图2 杏NBS 基因家族染色体定位和分布
Fig.2 Chromosomal localization and distribution of apricot NBS gene family
通过对杏NBS基因蛋白序列的注释发现,与抗病相关的蛋白主要为RPP13-like、RPP8、RGA、RPM1、RPS2、SUMM2、RPS5和ROQ11等,与4类病原有关,分别是寄生霜霉菌、丁香假单胞杆菌、致病疫霉菌和黄杆菌。寄生霜霉菌、丁香假单胞杆菌可能会引起果树霜霉病、溃疡病等病害,致病疫霉菌暂无报道,其余蛋白质与病原关系未知,仅明确其与抗病有关。
2.3 NBS基因家族基序分析
NBS基因家族的NB-ARC结构域通常包括8个保守基序,分别为P-loop(phosphate-binding loop)、RNBS-A(resistance NBS-A)、Kinase-2、RNBS-B、RNBS-C、RNBS-D、GLPL 和MHDL。杏NBS 基因家族中鉴定到10个Motif(图3),包括P-loop、RNBSA、Kinase-2、RNBS-B、GLPL和MHDL,N端的CC-1(coiled-coil-1)和CC-2,C 端的LRR,以及杏特异的CNBS-1(CC-NBS-1),但没有鉴定到RNBS-C 和RNBS-D,其中,在90%以上的NBS抗病基因中都鉴定到了P-loop、Kinase-2、RNBS-B 和GLPL 这4 个Motif,75%的NBS抗病基因鉴定到了RNBS-A,85%的NBS抗病基因鉴定到了MHDL。以上结果表明,NBS基因家族的motif相对比较保守。此外,在GLPL 和MHDL 之间鉴定到了杏特有的CNBS-1,CNBS-1在CNL型抗病基因中更保守,占82%,而在NL型抗病基因中只占55%;此外,在CNL 型抗病基因的N 端还鉴定到了杏CC 结构域特有的2 个Motif,分别为CC-1 和CC-2,其中CC-2 更为保守,占CNL型抗病基因的97%,而CC-1只占到67%。

图3 杏NBS 基因家族中保守motif 分布
Fig.3 Distribution of conserved motifs of apricot NBS gene family
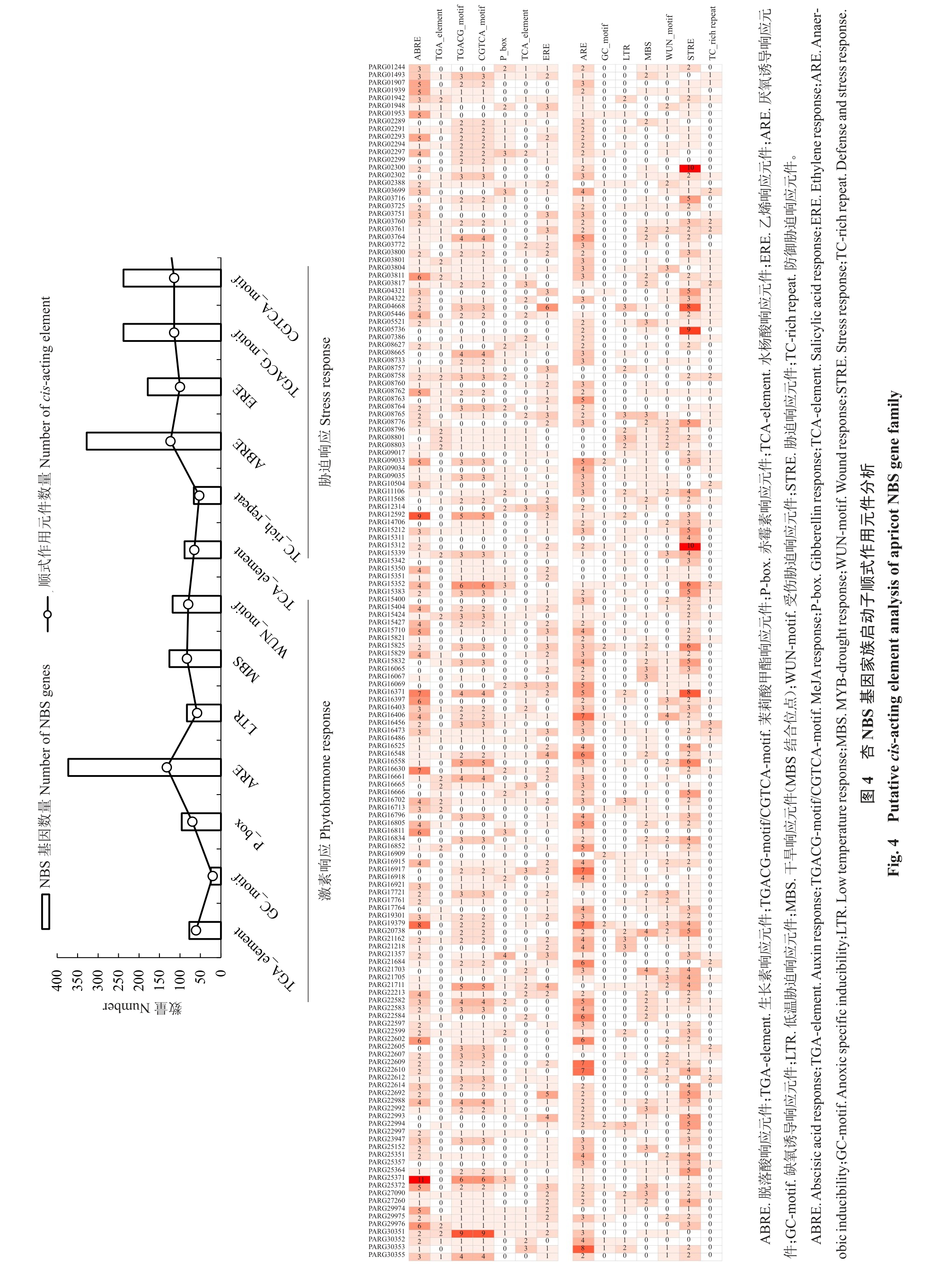
2.4 NBS基因家族顺式作用元件分析
杏NBS基因家族表达调控机制比较复杂,存在多种环境和激素信号的响应元件,大致可以分为植物激素响应元件和非生物胁迫响应元件。激素响应元件在NBS 抗病基因中普遍存在,79%的抗病基因有脱落酸响应元件,74%的抗病基因有茉莉酸甲酯响应元件,表明NBS基因家族基因表达主要受激素调控的影响;非生物胁迫响应元件中,85%抗病基因预测到了厌氧诱导元件,84%的抗病基因预测到了胁迫响应元件(图4)。此外,还预测到了水杨酸、赤霉素、生长素、乙烯等激素响应元件和光响应元件以及干旱、低温等环境胁迫响应元件,表明NBS 抗病基因可以响应植物体内多种代谢通路的调控。
2.5 NBS基因家族选择压力分析
Ka、Ks 分析结果表明(图5),65 对同源基因的非同义突变率Ka 主要分布在0.1 以下,同义突变率Ks 主要分布在0.05~0.15 之间,Ka/Ks 主要分布在0.5~1.0之间。在Ks=0.2时出现大量同源基因对,表明NBS 基因家族在进化中发生过加倍事件。61 对基因对Ka/Ks 值小于1,表明这些基因受到纯化选择。有4 对基因的Ka/Ks 值大于1,其中,3 个CNL型,1个NL型,CNL型中PARG16921-PARG16918与抗霜霉病有关,PARG08758-PARG08765与根腐病有关,PARG03801-PARG03800 与果树褐斑病、绿皱果病、白粉病和霜霉病有关,表明杏在进化过程中受这些病原的侵染高于其他病原,从而受到正选择。
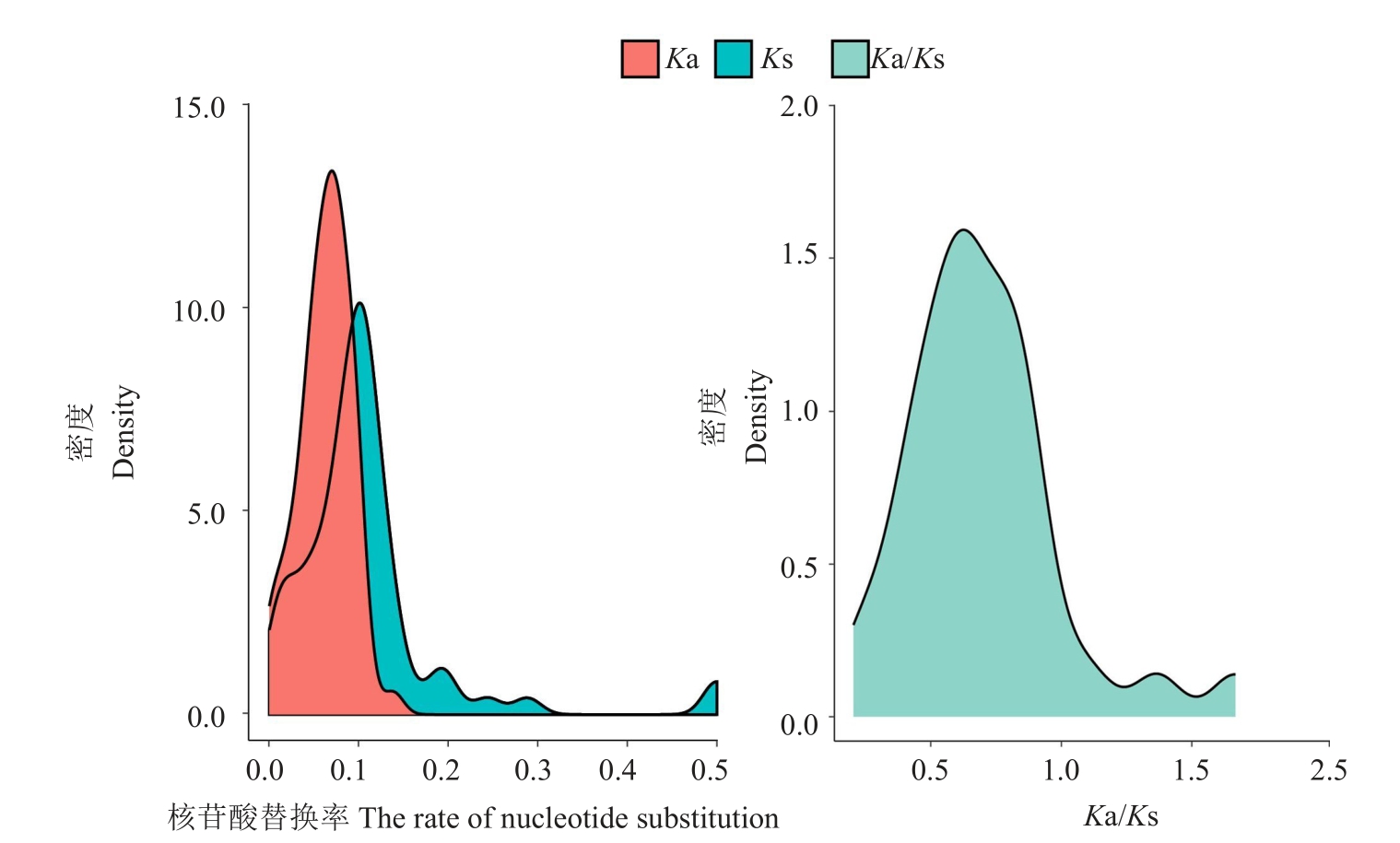
图5 杏NBS 基因家族Ka、Ks 分析
Fig.5 Ka and Ks analysis of apricot NBS gene family
2.6 NBS基因在果实发育过程中的差异表达分析
如图6所示,串枝红和大白杏在果实发育的4个时期均表达的基因分别有86个与84个,在大白杏果实中,2个基因在软核期(G1)特异表达(PARG15350和PARG15400),在硬核期(G2)、转色期(CT)和成熟期(FR)特异表达基因各有1个,分别是PARG02388、PARG21703 和PARG01953(图6-C)。在串枝红果实中,1 个基因(PARG15350)在G1 时期特异表达,其他时期无特异表达基因,但NBS 基因表达量差异较大(图6-A~C),表明NBS基因表达模式在品种间差异不大,但在表达量上存在较大差异。

图6 串枝红和大白杏果实发育4 个时期NBS 基因家族的表达分析
Fig.6 Transcriptome analysis of NBS gene family in 4 fruit development stages between Chuanzhihong and Dabaixing fruit
A.串枝红杏和大白杏NBS 基因家族果实发育4 个时期表达情况,1、2、3、4 分别代表串枝红G1 果实软核期、G2 果实硬核期、CT 果实转色期、FR 果实成熟期;a、b、c、d 分别代表大白杏的G1、G2、CT、FR 时期。B.串枝红杏各时期表达数量分析。C.大白杏各时期表达数量分析。
A.Expression pattern of NBS gene family in the 4 fruit developmental stages,including G1(soft kernel stage),G2(hard kernel stage),CT(fruit veraison stage),FR (mature stage),in the apricot cultivars including Chuanzhihong and Dabaixing.B.Venn diagram of Chuanzhihong NBS gene expression in different stages.C.Venn diagram of Dabaixing NBS gene expression in different stages.
NBS 基因家族杏果实不同发育时期差异表达分析表明,8 个基因(PARG04668、PARG09017、PARG15825、PARG15832、PARG16406、PARG17721、PARG22597 和PARG30352)在串枝红果实发育的4个时期表达量都显著高于大白杏(图6-A)。除了上述8 个差异基因外,各个时期内还有一些差异基因在串枝红中表达显著高于大白杏(图6-A),分别是G1 时期4 个(PARG03817、PARG11106、PARG15350和 PARG29976),G2时期3 个(PARG03716、PARG21218 和 PARG22609),CT时期2 个(PARG21684 和PARG22582)和FR时期7 个(PARG05521、PARG08801、PARG08803、PARG16630、PARG17764、PARG25364和PARG30351)。通过以上分析表明,不同发育时期差异表达基因并不完全相同,不同品种间果实抗病性差异主要与抗致病疫霉病原和抗霜霉病原的基因有关,尤其是4 个时期均显著差异表达的8 个NBS 基因,可能与杏果实的抗病性关联更为密切。
3 讨论
不同物种中NBS 基因数量与类型存在显著差异,杏基因组中共鉴定155个NBS抗病基因,而苹果(Malus domestica)中却鉴定出1000 个[20],这可能与苹果基因组发生过多次加倍事件及其设置过滤参数较为宽松有关(1 e-4)。NBS的进化树结果表明,NL型抗病基因较CNL型而言更为古老,推测CNL的N端的CC结构域是在与病原的对抗过程中逐渐演化而来。CNL 类型数量远大于NL,这与其他蔷薇科NBS抗病基因家族相符[21]。
杏NBS 抗病基因在不同染色体上的分布存在显著差异,主要分布在1、2和3号染色体上,这些染色体发生变异时易导致抗病基因丢失,致使植物易遭受病原侵害,因此,在进行抗病育种时应重点关注。此外,CNL 型抗病基因以基因簇的方式存在,基因簇促进了抗病信号转导,有利于NBS基因协作抵抗病原[22]。另外,李痘病抗性的PPVres(plum pox virus resistance locus)QTL 位于1 号染色体上[23],在这个QTL区域发现PARG04361基因,表明该基因可能与李痘病毒抗性有关,这还有待进一步研究。
顺式作用元件与基序均可与转录因子结合而调控基因表达,NBS基因的顺式作用元件较多且大多属于激素调控元件,如脱落酸、水杨酸等,这与植物激素通过协同或拮抗方式参与抗病信号网络调控的研究相吻合[24],此外还存在部分非生物胁迫响应元件,表明一些抗病基因不仅传递生物胁迫信号,而且具有传递非生物胁迫信号的功能。杏NBS 基因家族中,共鉴定到10个motif保守结构域,其中6个(分别是P-loop、RNBS-A、Kinase-2、RNBS-B、GLPL、MHDL)为NBS 基因通常具有的结构域,这与拟南芥和苹果等NBS 抗病基因的鉴定结果类似[21,25],另外,杏中独有CNBS-1 motif 结构域,其是否具有特殊作用还有待进一步研究。
2个杏品种果实发育过程中NBS基因的表达模式类似,表明NBS 基因表达受品种的影响较小,但不同品种间NBS基因在表达量上存在较大差异,可能与抵抗某些病原有关。通过对不同品种间NBS基因的差异分析发现,4个时期共有8个差异表达基因,均是串枝红高于大白杏,可能与串枝红果实抗病性高于大白杏有关,这些基因主要与抗致病疫霉病原、霜霉病原有关,在今后果实抗病研究中应重点关注;另外,不同时期特有的差异基因16个,分别是G1 4个,G2 3个,CT 2 个和FR 7个,表明这些基因的表达与果实发育特定时期有关,也可能受到相应病原刺激而被激活;其他无表达差异的基因可能未受到相关病原刺激而未出现表达差异。
4 结论
在串枝红杏基因组中共鉴定到155个NBS抗病基因,主要分为CNL 型(126 个)和NL 型(29 个)。NBS 基因的顺式作用元件较多且大多属于激素调控元件,基序分析发现1个杏中特有的CNBS-1 motif结构域。Ks分析结果显示,NBS基因家族发生过加倍事件导致部分NBS基因发生扩增。8个基因在串枝红果实发育的4个时期表达量均显著高于同时期的大白杏,这些NBS基因可能与串枝红果实抗病性高于大白杏有关,在果实抗病中应予以重点关注,研究结果为下一步杏果实抗病育种提供了理论指导。
[1] CAB International. Prunus armeniaca datasheet:Invasive species compendium[EB/OL].Wallingford,UK:CAB International,2019:[2021-9-19].https://www.cabi.org/isc/datasheet/44249.
[2] NAIDOO S,SLIPPERS B,PLETT J M,COLES D,OATES C N.The road to resistance in forest trees[J].Frontiers in Plant Science,2019,10:273.
[3] JONES J D,DANGL J L.The plant immune system[J].Nature,2006,444(7117):323-329.
[4] PERAZA-ECHEVERRIA S,DALE J L,HARDING R M,SMITH M K,COLLET C.Characterization of disease resistance gene candidates of the nucleotide binding site (NBS) type from banana and correlation of a transcriptional polymorphism with resistance to Fusarium oxysporum f.sp. cubense race 4[J].Molecular Breeding,2008,22(4):565-579.
[5] 文欢,刘永立.‘红阳’猕猴桃全基因组NBS-LRR 类基因家族的生物信息学分析[J].果树学报,2015,32(3):413-419.WEN Huan,LIU Yongli.Bioinformatic analysis of the NBSLRR gene family in kiwifruit‘Hongyang’[J].Journal of Fruit Science,2015,32(3):413-419.
[6] POTTINGER S E,INNES R W.RPS5-mediated disease resistance:fundamental insights and translational applications[J].Annual Review of Phytopathology,2020,58:139-160.
[7] MCDOWELL J M,DHANDAYDHAM M,LONG T A,AARTS M G M,GOFF S,HOLUB E B,DANGL J L.Intragenic recombination and diversifying selection contribute to the evolution of downy mildew resistance at the RPP8 locus of Arabidopsis[J].The Plant Cell,1998,10(11):1861-1874.
[8] JIANG F C,ZHANG J H,WANG S,YANG L,LUO Y F,GAO S H,ZHANG M L,WU S Y,HU S N,SUN H Y,WANG Y Z.The apricot (Prunus armeniaca L.) genome elucidates Rosaceae evolution and beta-carotenoid synthesis[J].Horticulture Research,2019,6(1):128.
[9] KATOH K,STANDLEY D M.MAFFT multiple sequence alignment software Version 7:Improvements in performance and usability[J].Molecular Biology and Evolution,2013,30(4):772-780.
[10] LU S N,WANG J Y,CHITSAZ F,DERBYSHIRE M K,GEER R C,GONZALES N R,GWADZ M,HURWITZ D I,MARCHLER G H,SONG J S,THANKI N,YAMASHITA R A,YANG M Z,ZHANG D C,ZHENG C J,LANCZYCKI C J,MARCHLER-BAUER A.CDD/SPARCLE:The conserved domain database in 2020[J].Nucleic Acids Research,2020,48(D1):265-268.
[11] MINH B Q,SCHMIDT H A,CHERNOMOR O,SCHREMPF D,WOODHAMS M D,VON HAESELER A,LANFEAR R.IQ-TREE 2:New models and efficient methods for phylogenetic inference in the genomic Era[J].Molecular Biology and Evolution,2020,37(5):1530-1534.
[12] LETUNIC I,BORK P.Interactive tree of life(iTOL)v5:An online tool for phylogenetic tree display and annotation[J].Nucleic Acids Research,2021,49(W1):293-296.
[13] WANG D,ZHANG Y,ZHANG Z,ZHU J,YU J.KaKs_Calculator 2.0:A toolkit incorporating gamma-series methods and sliding window strategies[J].Genomics Proteomics and Bioinformatics,2010,8(1):77-80.
[14] LESCOT M,DEHAIS P,THIJS G,MARCHAL K,MOREAU Y,VAN DE PEER Y,ROUZE P,ROMBAUTS S.PlantCARE,a database of plant cis-acting regulatory elements and a portal to tools for in silico analysis of promoter sequences[J].Nucleic Acids Research,2002,30(1):325-327.
[15] BAILEY T L,JOHNSON J,GRANT C E,NOBLE W S.The MEME suite[J].Nucleic Acids Research,2015,43(W1):39-49.
[16] CHEN S F,ZHOU Y Q,CHEN Y R,GU J.Fastp:An ultra-fastall-in-one FASTQ preprocessor[J].Bioinformatics,2018,34(17):884-890.
[17] KIM D,LANGMEAD B,SALZBERG S L.HISAT:A fast spliced aligner with low memory requirements[J].Nature Methods,2015,12(4):357-360.
[18] PERTEA M,PERTEA G M,ANTONESCU C M,CHANG T C,MENDELL J T,SALZBERG S L.StringTie enables improved reconstruction of a transcriptome from RNA-seq reads[J].Nature Biotechnology,2015,33(3):290-295.
[19] LOVE M I,HUBER W,ANDERS S.Moderated estimation of fold change and dispersion for RNA-seq data with DESeq2[J].Genome Biology,2014,15(12):550.
[20] PERAZZOLLI M,MALACARNE G,BALDO A,RIGHETTI L,BAILEY A,FONTANA P,VELASCO R,MALNOY M.Characterization of resistance gene analogues (RGAs) in apple(Malus × domestica Borkh.) and their evolutionary history of the Rosaceae family[J].PLoS One,2014,9(2):e83844.
[21] 贾艳晓.蔷薇科和葫芦科植物中NBS 基因的进化分析与比较[D].南京:南京大学,2015.JIA Yanxiao.Genome-wide comparative analysis of NBS-encoding genes in Rosaceae and Cucurbitaceae[D].Nanjing:Nanjing University,2015.
[22] VAN WERSCH S,LI X.Stronger when together:Clustering of plant NLR disease resistance genes[J].Trends in Plant Science,2019,24(8):688-699.
[23] DECROOCQ S,CHAGUE A,LAMBERT P,ROCH G,AUDERGON J M,GEUNA F,CHIOZZOTTO R,BASSI D,DONDINI L,TARTARINI S,SALAVA J,KRŠKA B,PALMISANO F,KARAYIANNIS I,DECROOCQ V.Selecting with markers linked to the PPVres major QTL is not sufficient to predict resistance to plum pox virus (PPV) in apricot[J].Tree Genetics and Genomes,2014,10(5):1161-1170.
[24] TON J,FLORS V,MAUCH-MANI B.The multifaceted role of ABA in disease resistance[J].Trends in Plant Science,2009,14(6):310-317.
[25] MEYERS B C,KOZIK A,GRIEGO A,KUANG H H,MICHELMORE R W.Genome-wide analysis of NBS-LRR-encoding genes in Arabidopsis[J].The Plant Cell,2003,15(4):809-834.