实时荧光定量PCR(real time quantitative PCR,RT-qPCR)是目前基因表达研究最常用的方法,广泛用于基因表达水平分析和转录组数据验证。该技术对PCR反应过程中的指数增长阶段进行实时跟踪,对每个循环扩增的PCR产物进行测量,从而精确确定模板的起始量[1],可以对表达水平较低的基因进行定量检测。较目前可用的其他技术,其具有定量准、灵敏度高、特异性强、成本低和效率高等特征[2]。由于各样品在RNA 提取、反转录和PCR 等过程中存在差异,需要进行适当的归一化来控制实验误差[3]。常采用对样本大小、总RNA 进行标准化以及测量参考基因等方法。其中,最常用的方法之一是将靶标基因的表达相对于参考基因的核糖体RNA(rRNA)或信使RNA(mRNA)进行标准化[4]。此类参考基因(也称为管家基因)能大量表达,且不与目标基因共同调控,在生物体的各个组织及各个生长阶段表达水平都较为稳定。另一方面,这些基因的表达应根据与该技术相关的实验误差(由于样品处理和上样等)而变化,以便通过归一化校正这些误差[5]。目前,植物常用的内参基因有Actin(ACT)、βtubulin (TUB)、Glyceraldehyde-3-phosphate dehydrogenase(GAPDH)、 Ubiquitin conjugating enzyme(UBC)、Elongation factor-1α(EF-1α)和18S ribosome(18S)等[6]。大量研究报告显示,管家基因的表达也会随着组织和实验条件的不同而发生很大的变化[6]。比如,Gopalam 等[7]对丹参(Salvia hispanica)稳定内参基因的分析结果表明,在不同的非生物胁迫处理条件下,以及在生殖阶段和营养阶段的最优内参基因不同;Pu 等[8]对白三叶草(Trifolium repens L.)在5 种不同的非生物胁迫(干旱,盐,热,冷和重金属)条件下,从两个组织(叶和根)中取样,分析得到在不同胁迫下,最优内参基因的选择不同;Artico等[6]在对陆地棉(Gossypium hirsutum)23个组织样本的候选基因表达稳定性测试中分析得到,在陆地棉不同组织、花发育和果实发育中,最优内参基因差异很大;叶新如等[9]对冬瓜(Benincasa hispida Cogn.)不同组织和不同非生物胁迫条件下的内参基因的表达情况进行分析,发现在不同组织和不同胁迫处理下最优内参基因存在差异。为了保证RT-qPCR 结果的可靠性,需要根据试验材料的不同部位、生长发育阶段及试验条件等因素筛选合适的内参基因[10-11]。
山核桃(Carya cathayensis Sarg.)为胡桃科山核桃属多年生落叶乔木,是我国特色坚果和木本油料树种,现主要分布在浙、皖交界的天目山区[12-13]。山核桃种仁含有大量的初级和次级代谢营养成分,具有极高的保健价值,比如具有健脑、预防心脑血管疾病、抗氧化及调节免疫等作用[14-15]。其浓郁的香味和独特的风味深受消费者喜爱,具有很高的经济效益,是山区农民脱贫致富奔小康主要的经济来源。为了更深入地研究山核桃的生长、发育、果实品质调控及逆境响应等的内在分子机制,课题组前期已完成山核桃的全基因组测序及组装[16],同时也利用Actin作为内参基因分析了一些山核桃基因的表达[15],然而,最佳的山核桃内参基因目前未见报道。因此,笔者在本研究中以山核桃的根、茎、叶、柱头、果皮和不同发育时期的胚,干旱、盐浓度和激素喷施下的叶及嫁接山核桃的砧木和接穗作为试验材料,利用ge-Norm、NormFinder、BestKeeper 和RefFinder 4 种软件对ADP 核糖基化因子(ADP-ribosylation factor,ARF)、肌动蛋白(Actin1/7,ACT1/7)、金属蛋白酶1(Metalloprotease,MPS)、泛素结合酶(Ubiquitin conjugating enzyme 9,UBC9)、亲 环 素(Cyclophilin,CYP)、60S 核 糖 体 蛋 白(60S ribosomal protein,60SRP)、延伸因子1α (Elongation factor1α,EF-1α)、甘油醛-3-磷酸脱氢酶(Glyceraldehyde-3-phosphate dehydrogenase,GAPHD)和组蛋白(Histone,HIS)等10 个管家基因家族中的19 个候选内参基因的稳定性进行综合分析,旨在筛选出山核桃中最稳定的内参基因,为后续山核桃的分子生物学研究提供理论支撑。
1 材料和方法
1.1 试验材料
不同组织样品(根、茎、叶、柱头、果皮及胚)采集于浙江农林大学果木园(119°43′42″E,30°15′16″N,海拔:38 m)的山核桃。每个样品3 个生物学重复。样品采集后立即保存于液氮,存于-80 ℃冰箱贮存备用。
选取生长状况良好的1 年生山核桃苗(来自浙江农林大学温室大棚),使用100 mmol·L-1的NaCl模拟盐胁迫水培处理,分别在处理0 h、12 h 和24 h后采集叶片立即保存于液氮,后存于-80 ℃冰箱贮存备用。使用25%(φ,后同)的聚乙二醇8000(PEG8000)模拟干旱胁迫水培处理,分别在处理0、12、24 h 后,采集叶片立即保存于液氮,后存于-80 ℃冰箱贮存备用。
选用以2 年生实生山核桃苗(来自浙江农林大学温室大棚)为砧木,当年生嫩枝(来自浙江农林大学温室大棚)为接穗进行嫁接,2 a 后,喷施10 种不同的植物激素包括吲哚3-乙酸(IAA,100 μmol·L-1)、吲哚丁酸(IBA,100 μmol·L-1)、萘乙酸(NAA,100 μmol·L-1)、N-(1-萘基)邻氨甲酰苯甲酸(NPA,10 μmol·L- 1)、激动素(KT,100 μmol·L- 1)、脱落酸(ABA,100 μmol·L-1)、水杨酸(SA,200 μmol·L-1)、赤霉素(GA3,200 μmol·L-1)、乙烯(ETH,1 mmol·L-1)和茉莉酸(MeJA,100 mmol·L-1)于山核桃叶片表面,其中蒸馏水作为对照处理。处理1 h 后补喷1次,每次喷施激素或蒸馏水直到第一滴液体从叶面流下为止。3 h 后,采集山核桃叶片,立即保存于液氮,存于-80 ℃冰箱贮存备用。
以2 a实生山核桃苗(来自浙江农林大学温室大棚)为砧木,当年生嫩枝(来自浙江农林大学温室大棚)为接穗进行嫁接,分别于0、3、7、14 d采集砧木和接穗,立即保存于液氮,存于-80 ℃冰箱贮存备用。
1.2 总RNA提取及反转录cDNA合成
各样品RNA 提取方法参照TaKaRa MiniBEST Plant RNA Extraction Kit说明书,1.5%琼脂糖凝胶电泳检测RNA 完整性,NanoDrop 1000 核酸蛋白测定仪测定RNA 浓度及质量。参照PrimerScriptTMRT Master Mix(Perfect Real Time)说明书,合成cDNA,得到的cDNA-20 ℃保存,用于后续试验。
1.3 内参基因的筛选及引物设计
基于内参基因相关文献和山核桃转录组测序数据,选取了ARF、ACT1、ACT7、MPS、UBC9、CYP、60SRP、EF-1α、GAPHD和HIS 10个持家基因家族中的19 个候选内参基因。利用Primer Premier 5 软件设计引物(表1),所有引物由杭州有康生物技术有限公司合成。
表1 19 个候选基因的RT-qPCR 引物序列
Table 1 RT-qPCR primer sequences of 19 candidate genes

基因名称Gene name CcARF-1退火温度TM/℃55.2产物长度Product length/bp 157 CcARF-2 53.8 182 CcACT1-1 54.1 184 CcACT1-2 58.3 190 CcACT7-1 53.2 158 CcACT7-2 54.9 164 CcACT7-3 54.1 192 CcMPS 55.3 192 CcUBC9-1 54.5 194 CcUBC9-2 53.4 187 CcUBC9-3 53.0 168 CcUBC9-4 53.4 159 CcUBC9-5 55.7 192 CcCYP 55.7 181 Cc60SRP 51.4 172 CcEF-1α-1 54.1 153 CcEF-1α-2 53.1 154 CcGAPDH 54.2 162 CcHIS正向引物(5’-3’)Forward primer(5’-3’)反向引物(5’-3’)Reverse primer(5’-3’)F:TGTGGTTGAAGCTAGGGATG R:GCGTTGACGGAGTGAGTG F:GGATGAACTGCGAGATGC R:AGAGCCAATCCAAACCCT F:AAAACGAACTTACCGCTTCA R:CCTGCTCTTTGGGACATT F:TGCAAGCCTTCCTGCTGA R:CTCCACCTGAATACCATCCAAC F:GCGACAATGGAACTGGAA R:CCTCTTTGACTGGGCTTC F:TATGTTGCCCTTGACTACGA R:GCAGCTTCCATTCCGATC F:GGAATGGGCCAGAAAGAT R:TGGTGCCTCGGTAAGAAG F:ATGCTTGTTTCATCCGTGTT R:TACGCTGAACCTCATTGTCTC F:ATCCCTTTAAGCCACCCA R:AGCAATCTCAGGCACCAA F:ATAAATAGCAATGGCAGCAT R:TCCTCGCAGTTGTCTCGT F:TCCTCCAGACTACCCATTT R:TCCGTCAGAAGCGAACAG F:TACCCATTCAAACCTCCC R:GTCCGTCAGAAGTGAACAGATA F:GGCCCTGTTGCTGAAGAC R:GTCAAGGCAAATGCTCCC F:GGACCAGGGCTTCTGTCA R:TCTTGGGCGTTCCATTCT F:ATTGAGAAGGCAGGTGGA R:TTGATCGTACATAGGGTTTT F:AAGATTGGTGGCATTGGA R:CACGTTGAAGCCGACATT F:GGCTGACTGTGCTGTCCT R:GGTTGTGGCATCCATTTT F:GACCACCCATTCCTACACC R:ATGCCATTCAACTTTCCTTT F:TACGAGAAATCCGCAAGTAC R:TCAAACAGTCCAACGAGGT 53.1 172
1.4 候选内参基因的RT-qPCR
在实时定量PCR 仪(BIO-RAD CFX96)上进行RT-qPCR扩增。反应体系:TB Green Taq(2×)5 μL,ddH2O 3.8 μL,上下游引物(浓度为10 μmol·L-1)各0.2 μL,待测cDNA 样品0.8 μL,共10 μL。反应程序:在95 ℃下预变性30 s,在95 ℃下变性5 s,然后在60 ℃下退火30 s,此过程循环40 次,最后在55~95 ℃下生成熔解曲线,检测引物特异性。每个RT-qPCR 反应均进行3 次生物学和技术重复。利用BioRad CFX Manager软件获取数据。
1.5 引物特异性检测
以山核桃不同组织混合cDNA 为模板对19 个候选内参基因进行普通PCR 扩增,扩增方法参照PrimeSTAR® Max DNA Polymerase 使用说明。对获得的PCR产物进行琼脂糖凝胶(1.5%)电泳检测,并通过RT-qPCR反应获取样品熔解曲线图,判断引物特异性。
1.6 数据处理与分析
求3 次生物学重复循环数阈值(cycle threshold,CT)值的平均值,导入BestKeeper程序计算相关系数(R)、标准偏差(standard derivatives,SD)和变异系数(Coefficient of Variation,CV)等值,根据判定原则,确定较稳定的内参基因。计算候选基因的相对表达量2-(CT 样品-CT 最小),然后将数据导入geNorm 和Norm-Finder 软件中计算基因表达稳定值,对不同研究条件下合适的参考基因的稳定性进行排名,稳定性值越低,基因越稳定。geNorm软件可得到最适数据归一化内参基因数目的柱状图,确定试验所需的最适内参基因数目。RefFinder 程序集成了Delta CT,BestKeeper,NormFinder 和geNorm 的分析结果[17-18],筛选出最适内参基因。利用SPSS20 软件对数据进行单因素方差分析,基于p值判断差异显著性。
2 结果与分析
2.1 引物特异性分析
1.5%琼脂糖凝胶电泳显示19 个候选参考基因均扩增产生预期大小的目的条带,且条带单一,无引物二聚体或非特异性扩增,说明所筛选的引物特异性良好(图1)。此外,RT-qPCR 熔解曲线均只显示单个信号峰,说明RT-qPCR 反应的特异性高,结果可靠(图2)。
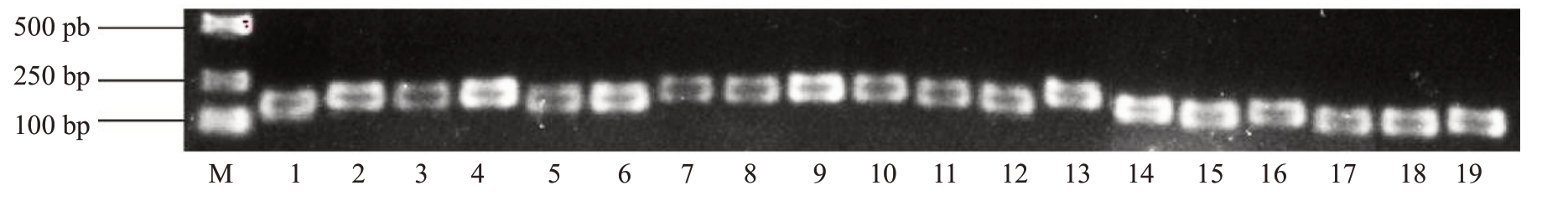
图1 山核桃19 个候选内参基因的常规PCR 扩增条带
Fig.1 PCR amplification of 19 candidate internal reference genes in hickory(Carya cathayensis)
M.Marker;1-19.ARF-1,ARF-2,ACT1-1,ACT1-2,ACT7-1,ACT7-2,ACT7-3,MPS,UBC9-1,UBC9-2,UBC9-3,UBC9-4,UBC9-5,60SRP,EF-1α-1,EF-1α-2,CYP,GAPHD and HIS.

图2 19 个候选内参基因的RT-qPCR 溶解曲线
Fig.2 Dissolution curves of 19 candidate internal reference genes for RT-qPCR analysis
2.2 候选内参基因在山核桃不同组织的稳定性分析
2.2.1 CT 值分析 19 个候选内参基因在山核桃不同组织器官中的CT 值大小及变化趋势均有一定程度的变化(图3),CT值越小,基因表达水平越高。19个候选基因在山核桃6 个组织中的平均CT 值范围在22.49~26.06。平均表达量最高的基因UBC9-5的CT 值为22.49,平均表达量最低的基因EF-1α-2 的CT 值为26.06,其与表达量倒数第二的基因MPS 相比,差异显著(p<0.001)。
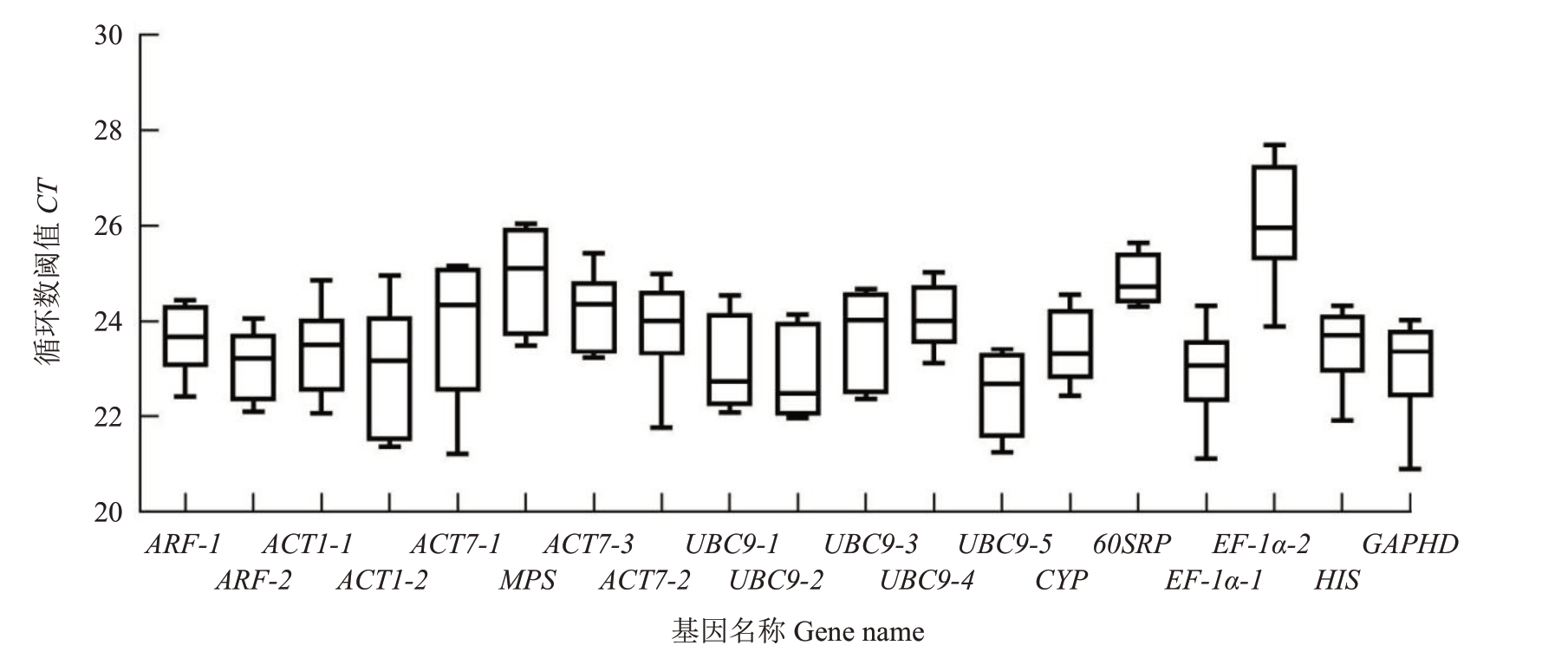
图3 19 个候选内参基因在山核桃不同组织中的CT 值
Fig.3 CT values of 19 candidate internal reference genes in different tissues of hickory(Carya cathayensis)
2.2.2 geNorm 分析 利用geNorm 软件计算出19个候选内参基因在山核桃不同组织表达的稳定性值(M),其中,M 值越小稳定性越好,M 值>1.5 的基因表达不稳定。由表2 可知,在山核桃不同组织中,ARF-1 基因的M 值最低,为0.769,即表达最稳定;GAPDH 基因M 值超过1.5,为不稳定的基因。基于本软件共有18 个候选内参基因的M 值小于阈值1.5,都可以考虑将其作为内参基因。由于geNorm软件将每个候选内参基因与其他内参基因的配对表达水平比值经过对数转换计算标准差,更适用于准确定量的内参基因数目。该软件可计算引入新基因后标准化因子的配对变异值V(Variations),Vn/Vn+1的值可确定所需内参基因的最佳数目。Vn/Vn+1默认临界值为0.15,Vn/Vn+1<0.15,n 即为所需内参基因数目。由图4可得,V2/3的比值为0.102,小于0.15,说明在山核桃的不同组织中,ARF-1和60SRP这2个内参基因组合的稳定性可以满足要求。
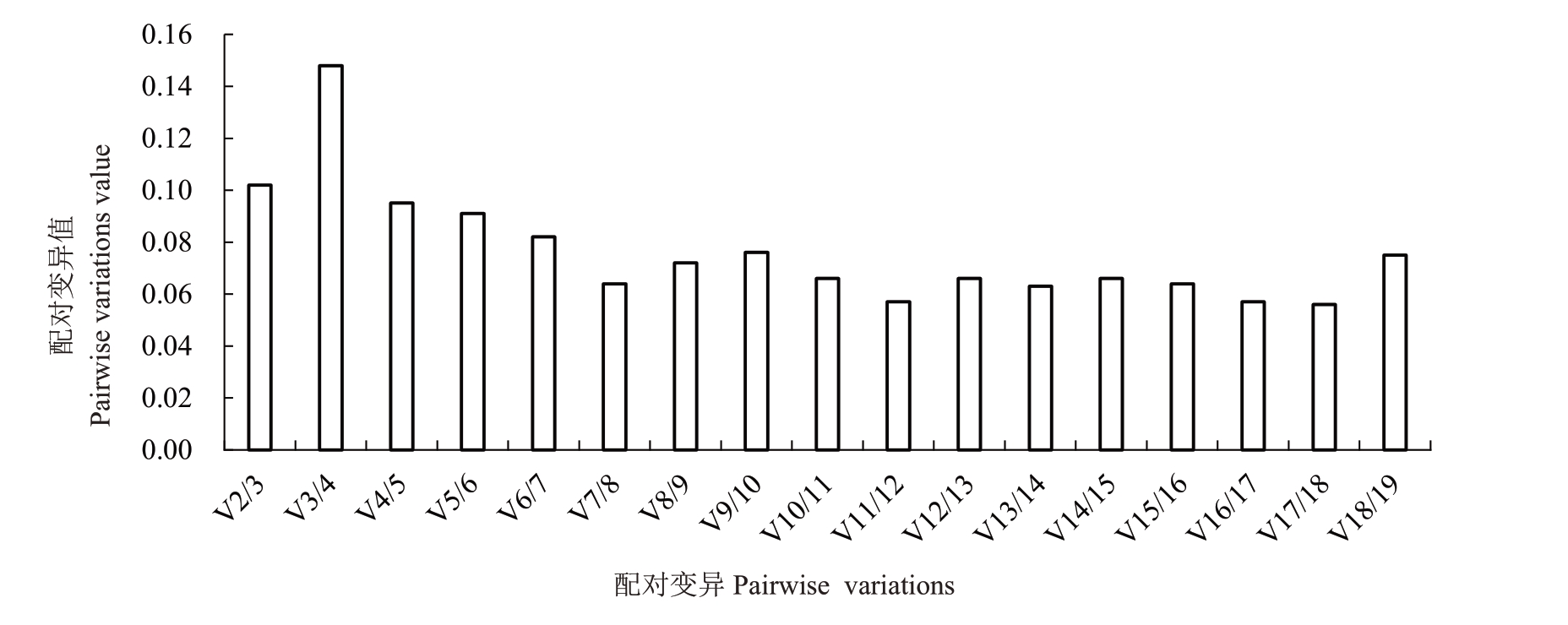
图4 利用geNorm 软件分析内参基因的最佳数目
Fig.4 Determination of optimal number of internal reference genes for normalization using geNorm software
V2/3 表示稳定性最好的2 个基因的变异值与稳定性最好的3 个基因的变异值的比值,其他以此类推。
V2/3 represented the ratio of the variation value of the two genes with the best stability to the variation value of the three genes with the best stability,and so on.
表2 用geNorm 分析19 个候选内参基因在山核桃不同组织中的表达稳定性Table 2 geNorm analysis of expression stability of 19 candidate internal reference genes in different tissues of Carya cathayensis
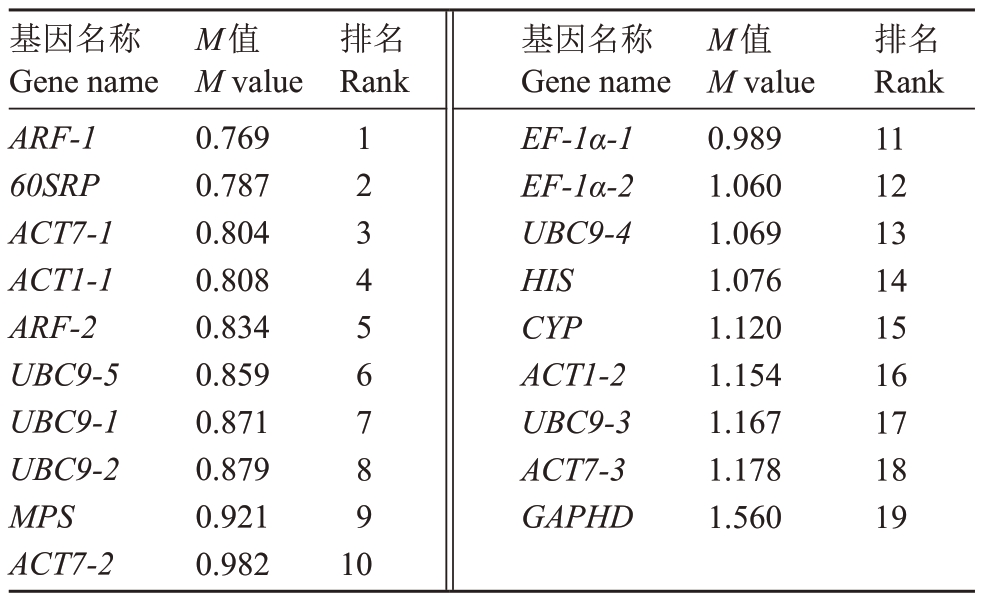
基因名称Gene name ARF-1 60SRP ACT7-1 ACT1-1 ARF-2 UBC9-5 UBC9-1 UBC9-2 MPS ACT7-2 M值M value 0.769 0.787 0.804 0.808 0.834 0.859 0.871 0.879 0.921 0.982排名Rank 1 2 3 4 5 6 7 8 9基因名称Gene name EF-1α-1 EF-1α-2 UBC9-4 HIS CYP ACT1-2 UBC9-3 ACT7-3 GAPHD M值M value 0.989 1.060 1.069 1.076 1.120 1.154 1.167 1.178 1.560排名Rank 11 12 13 14 15 16 17 18 19 10
2.2.3 NormFinder 分析 NormFinder 与geNorm 相似,通过对计算出的候选内参基因表达稳定性值进行排序,筛选出最适的内参基因,稳定性值越小,内参基因的稳定性越好。表3显示在山核桃不同组织中ARF-1和60SRP仍为最稳定的基因。
表3 用NormFinder 分析19 个候选内参基因在山核桃不同组织中的表达稳定性
Table 3 Analysis of expression stability of 19 candidate internal reference genes in different tissues of Carya cathayensis with NormFinder
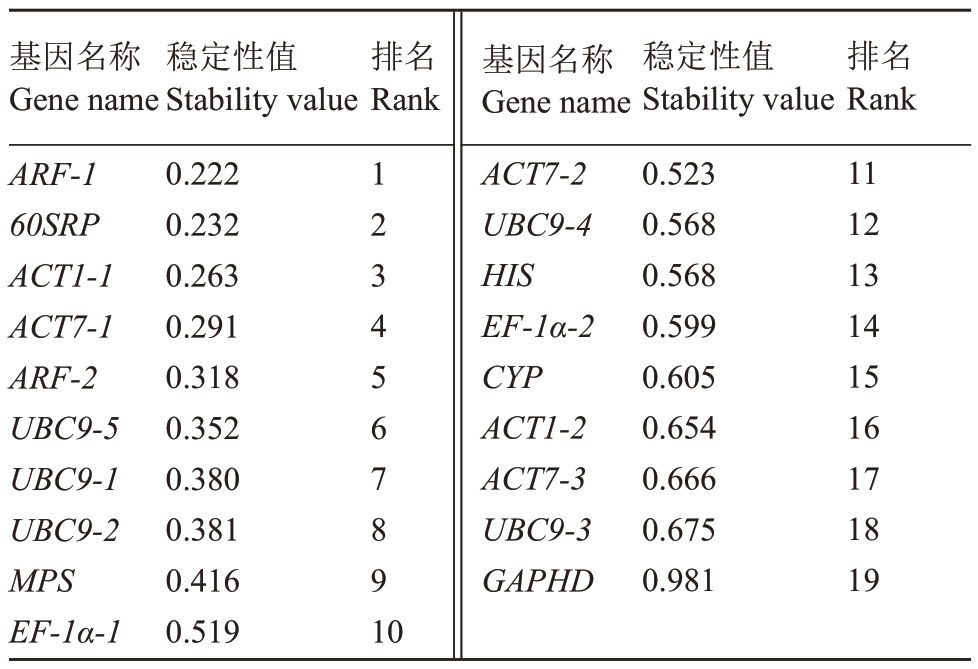
基因名称Gene name ARF-1 60SRP ACT1-1 ACT7-1 ARF-2 UBC9-5 UBC9-1 UBC9-2 MPS EF-1α-1稳定性值Stability value 0.222 0.232 0.263 0.291 0.318 0.352 0.380 0.381 0.416 0.519排名Rank 1 2 3 4 5 6 7 8 9 10基因名称Gene name ACT7-2 UBC9-4 HIS EF-1α-2 CYP ACT1-2 ACT7-3 UBC9-3 GAPHD稳定性值Stability value 0.523 0.568 0.568 0.599 0.605 0.654 0.666 0.675 0.981排名Rank 11 12 13 14 15 16 17 18 19
2.2.4 BestKeeper 分析 BestKeeper 可直接根据内参基因的CT 值计算标准偏差(standard deviation,SD)、变异系数(coefficient of variation,CV),其中,SD值和CV越小,基因的稳定性越好,若SD值>1,基因不稳定。从BestKeeper 软件分析结果(表4)可知,在19 个内参基因中,60SRP 的SD 值最小,为0.433,即表达稳定性最高。ACT1-2的SD值超过1,为不稳定基因。
表4 19 个候选内参基因在山核桃不同组织中的表达稳定性的BestKeeper 分析
Table 4 BestKeeper analysis of expression stability of 19 candidate internal reference genes in different tissues of hickory(Carya cathayensis)
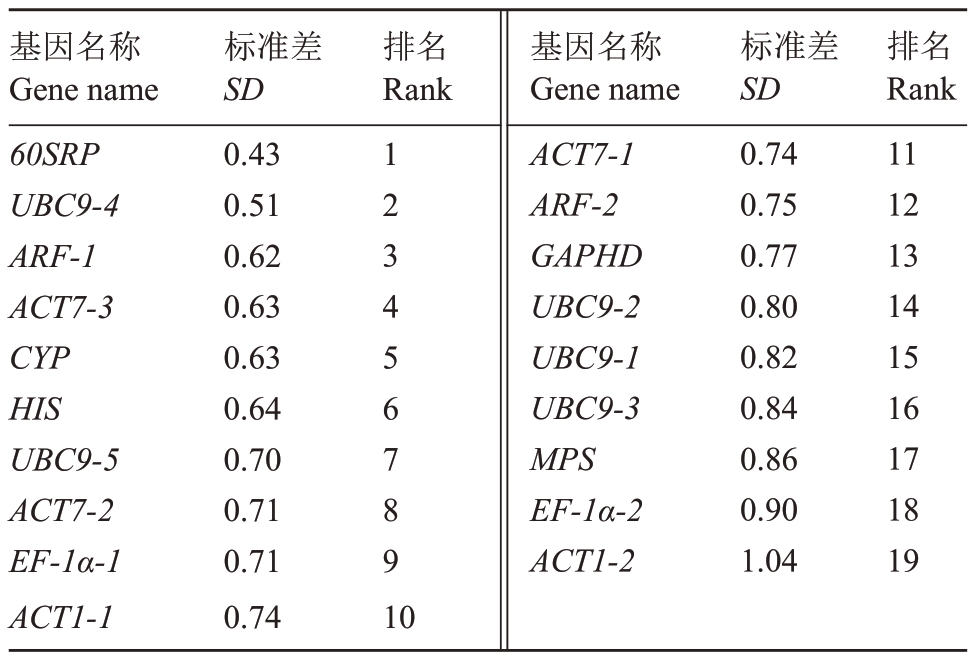
基因名称Gene name 60SRP UBC9-4 ARF-1 ACT7-3 CYP HIS UBC9-5 ACT7-2 EF-1α-1 ACT1-1标准差SD 0.43 0.51 0.62 0.63 0.63 0.64 0.70 0.71 0.71 0.74排名Rank 1 2 3 4 5 6 7 8 9 10基因名称Gene name ACT7-1 ARF-2 GAPHD UBC9-2 UBC9-1 UBC9-3 MPS EF-1α-2 ACT1-2标准差SD 0.74 0.75 0.77 0.80 0.82 0.84 0.86 0.90 1.04排名Rank 11 12 13 14 15 16 17 18 19
2.2.5 RefFinder 分析 RefFinder 程序集成了Delta CT,BestKeeper,NormFinder 和geNorm 4 种 方法[17-18]。表5 中显示了候选参考基因的综合排名,ARF-1 和60SRP 基因为综合稳定性最高的两个基因。又因geNorm 软件V2/3的比值小于0.15,说明在山核桃的不同组织中,ARF-1和60SRP这2个内参基因组合的稳定性可以满足要求。
表5 19 个候选内参基因在山核桃不同组织中的表达稳定性的RefFinder 分析
Table 5 RefFinder analysis of expression stability of 19 candidate internal reference genes in various tissues of hickory(Carya cathayensis)
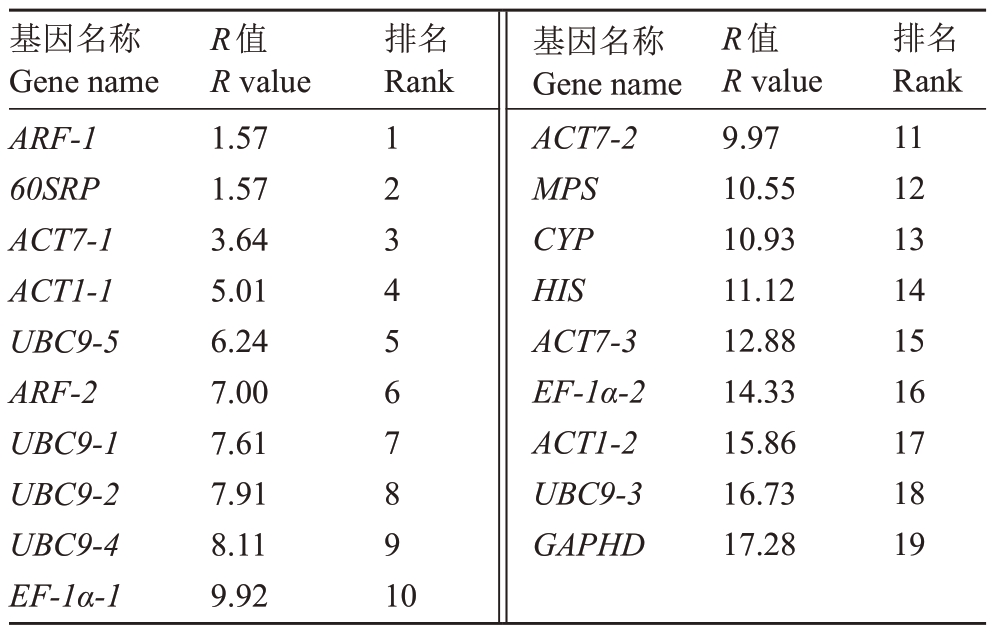
基因名称Gene name ARF-1 60SRP ACT7-1 ACT1-1 UBC9-5 ARF-2 UBC9-1 UBC9-2 UBC9-4 EF-1α-1 R值R value 1.57 1.57 3.64 5.01 6.24 7.00 7.61 7.91 8.11 9.92排名Rank 1 2 3 4 5 6 7 8 9 10基因名称Gene name ACT7-2 MPS CYP HIS ACT7-3 EF-1α-2 ACT1-2 UBC9-3 GAPHD R值R value 9.97 10.55 10.93 11.12 12.88 14.33 15.86 16.73 17.28排名Rank 11 12 13 14 15 16 17 18 19
2.3 候选内参基因在山核桃胁迫、激素和嫁接条件下的稳定性
根据19 个候选内参基因在山核桃不同组织的稳定性,选取稳定性最高的5 个基因(ARF-1、60SRP、ACT7-1、ACT1-1 和UBC9-5)在胁迫、激素和嫁接条件下的稳定性进行分析。
2.3.1 CT值分析 5个候选内参基因在山核桃不同处理下的CT 值大小及变化趋势均显示一定程度的变化(图5)。在盐胁迫和干旱条件下,山核桃叶的5个候选基因的平均CT 值范围在23.73~26.42,平均表达量最高的基因为UBC9-5,最低的基因为60SRP;在对山核桃叶不同激素处理下,平均CT 值范围在21.39~23.71,平均表达量最高的基因为UBC9-5,最低的基因为ARF-1;在山核桃砧木和接穗嫁接条件下,平均CT值范围在22.24~27.23,平均表达量最高的基因为ACT1-1,最低的基因为60SRP;在综合所有组织和处理的条件下,平均CT值范围在22.32~25.25,平均表达量最高的基因为UBC9-5,最低的基因为60SRP。

图5 5 个候选内参基因在山核桃盐胁迫和干旱处理(A)、不同激素处理(B)、嫁接处理(C)及综合所有组织及处理下(D)的CT 值
Fig.5 CT value of 5 candidate reference genes under salt and drought stress(A),different hormone treatments(B),grafting treatments(C)and comprehensive tissue and treatments(D)of hickory(Carya cathayensis)
误差棒上的不同字母表示差异显著性(p <0.05)。
Different letters on the error bar are significantly different from each another(p <0.05).
2.3.2 geNorm 分析 由geNorm 分析结果(表6)可知,5个候选基因在山核桃叶响应盐胁迫和干旱时,其中60SRP 基因的M 值最低,为0.265,即表达最稳定;在对山核桃叶进行不同激素处理后,UBC9-5 为最稳定的基因,M=0.268;在山核桃砧木和接穗嫁接后,60SRP 基因的表达不随时间的变化而变化,M=0.526;综合所有组织和处理的条件下,ACT7-1 和UBC9-5为最稳定的基因,M=0.556。
表6 5 个山核桃候选内参基因在不同处理下表达稳定性的geNorm 分析
Table 6 geNorm analysis of expression stability of five candidate internal reference genes of hickory(Carya cathayensis)under various treatments
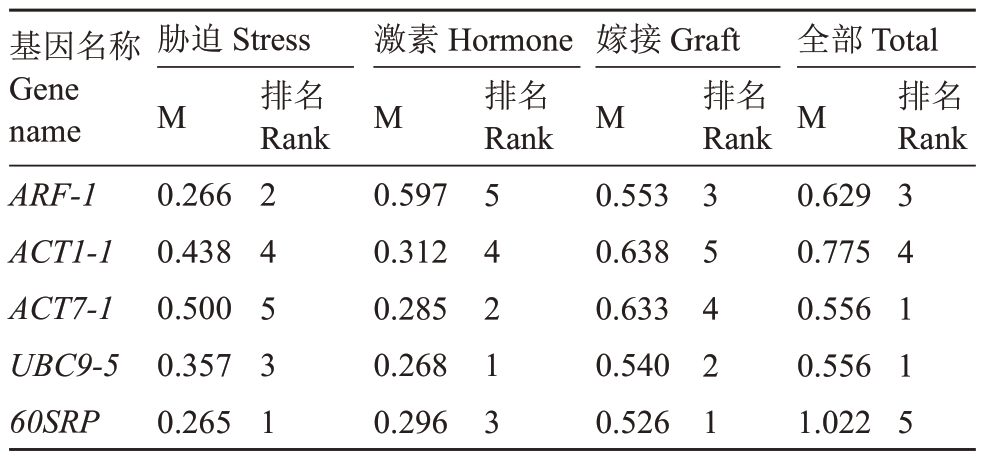
基因名称Gene name ARF-1 ACT1-1 ACT7-1 UBC9-5 60SRP胁迫Stress M 0.266 0.438 0.500 0.357 0.265排名Rank排名Rank排名Rank排名Rank 2 4 5 3 1激素Hormone M 0.597 0.312 0.285 0.268 0.296 5 4 2 1 3嫁接Graft M 0.553 0.638 0.633 0.540 0.526 3 5 4 2 1全部Total M 0.629 0.775 0.556 0.556 1.022 3 4 1 1 5
使用geNorm 软件计算引入新基因后标准化因子的配对变异值(图6),在盐和干旱胁迫及激素处理条件下,V2/3的比值均小于阈值1.5,分别为0.098和0.059,表明2个内参基因组合的稳定性可以满足要求。在山核桃砧木和接穗嫁接后,V2/3的比值为0.153,大于阈值,所以需要引入第三个内参基因确保结果的准确性。
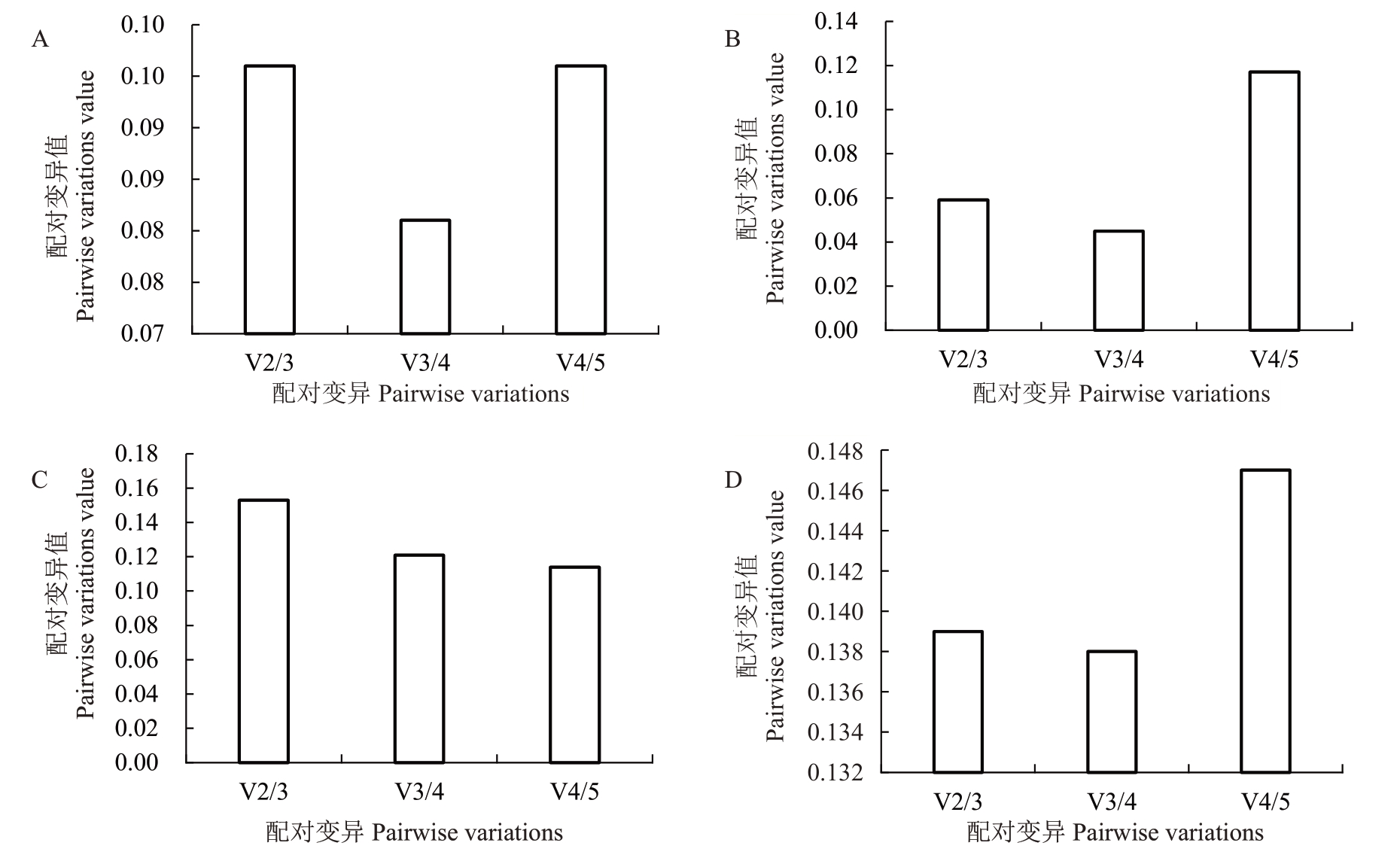
图6 利用geNorm 软件分析各种处理下山核桃内参基因的最佳数目
Fig.6 Analysis of the optimum number of internal reference genes of hickory(Carya cathayensis)under various treatments using geNorm
A.盐胁迫和干旱处理;B 不同激素处理;C 嫁接处理;D.综合所有组织和处理。
A.Salt and drought stress;B.Different hormone treatments;C.Grafting treatments;D.Comprehensive tissue and treatments.
2.3.3 NormFinder 分析 NormFinder 分析结果(表7)表明,在山核桃叶的盐胁迫和干旱条件下,ARF-1基因最稳定;在对山核桃叶不同激素处理下,UBC9-5 为最稳定基因;在山核桃砧木和接穗嫁接后,60SRP 基因表达最稳定;在综合所有组织和处理的条件下,ACT7-1为最稳定的基因。NormFinder分析结果与geNorm结果基本一致。
表7 5 个山核桃候选内参基因响应不同处理的表达稳定性的NormFinder 分析
Table 7 Analysis of expression stability of five candidate internal reference genes of hickory(Carya cathayensis)in response to different treatments by NormFinder
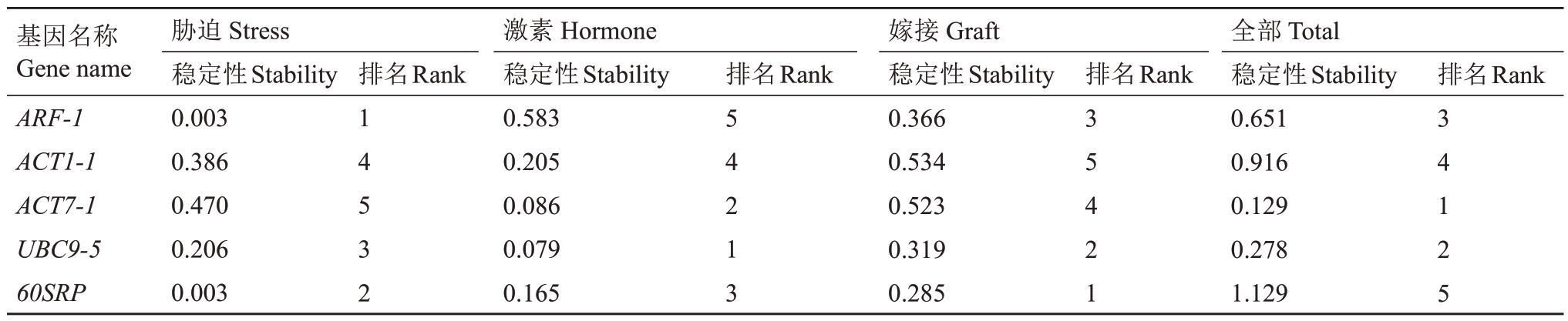
基因名称Gene name ARF-1 ACT1-1 ACT7-1 UBC9-5 60SRP胁迫Stress稳定性Stability 0.003 0.386 0.470 0.206 0.003排名Rank 排名Rank 排名Rank 排名Rank 1 4 5 3 2激素Hormone稳定性Stability 0.583 0.205 0.086 0.079 0.165 5 4 2 1 3嫁接Graft稳定性Stability 0.366 0.534 0.523 0.319 0.285 3 5 4 2 1全部Total稳定性Stability 0.651 0.916 0.129 0.278 1.129 3 4 1 2 5
2.3.4 BestKeeper 分析 从BestKeeper 软件分析结果(表8)可知,在盐胁迫和干旱条件下,山核桃叶的ACT7-1基因的表达最稳定;在对山核桃叶不同激素处理下,ACT7-1 同样为最稳定的基因;在山核桃砧木和接穗嫁接后,UBC9-5 基因表达最稳定,而ACT7-1稳定性最差;在综合所有组织和处理的条件下,ARF-1为最稳定的基因。
表8 5 个山核桃候选内参基因在不同处理下表达的稳定性的BestKeeper 分析
Table 8 Analysis of expression stability of five candidate internal reference genes of hickory(Carya cathayensis)under different treatments using BestKeeper
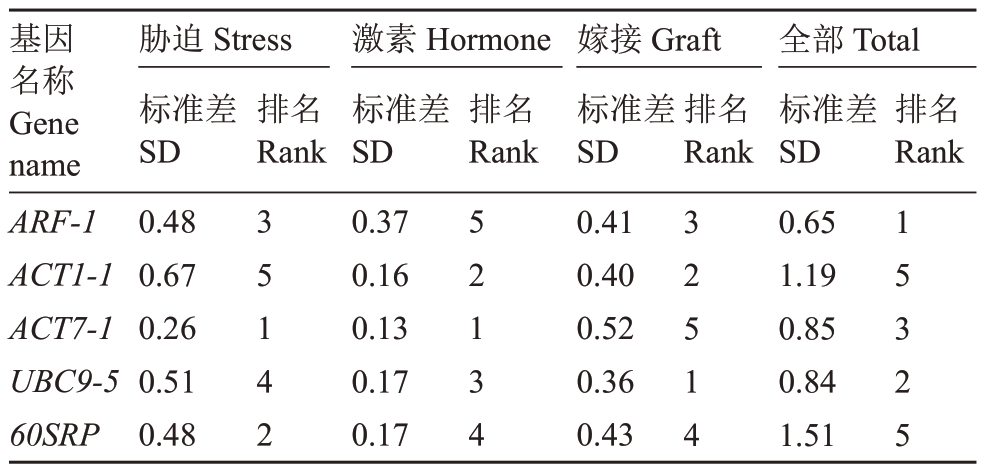
基因名称Gene name ARF-1 ACT1-1 ACT7-1 UBC9-5 60SRP胁迫Stress标准差SD 0.48 0.67 0.26 0.51 0.48排名Rank排名Rank排名Rank排名Rank 3 5 1 4 2激素Hormone标准差SD 0.37 0.16 0.13 0.17 0.17 5 2 1 3 4嫁接Graft标准差SD 0.41 0.40 0.52 0.36 0.43 3 2 5 1 4全部Total标准差SD 0.65 1.19 0.85 0.84 1.51 1 5 3 2 5
2.3.5 RefFinder分析 RefFinder综合分析结果(表9)显示:在山核桃叶的盐胁迫和干旱条件下,60SRP基因的表达最稳定;在对山核桃叶不同激素处理下UBC9-5 为最稳定基因;在山核桃砧木和接穗嫁接后,60SRP 和UBC9-5 基因表达最稳定;在综合所有组织和处理的条件下,UBC9-5为最稳定的基因。
表9 利用RefFinder 分析5 个候选内参基因在不同处理下表达的稳定性
Table 9 Reffinder was used to analyze the expression stability of 5 candidate internal reference genes under different treatments
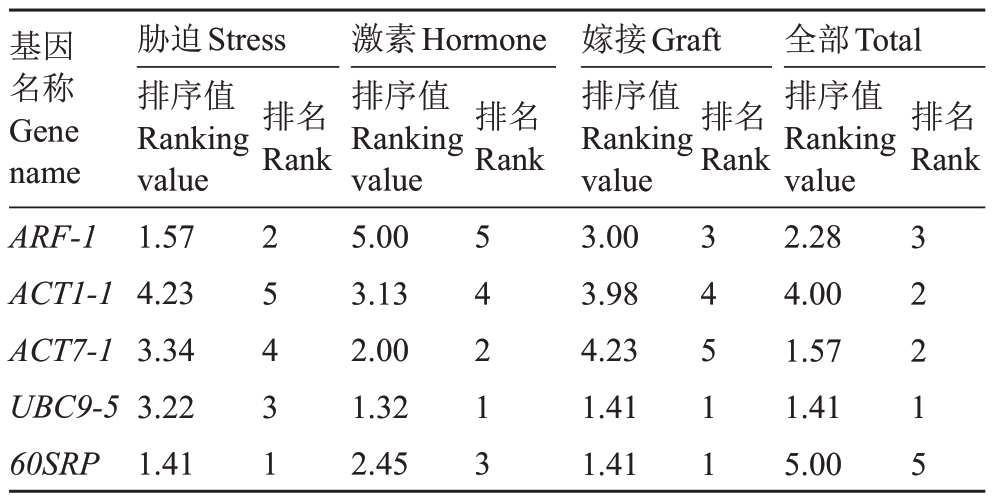
基因名称Gene name ARF-1 ACT1-1 ACT7-1 UBC9-5 60SRP胁迫Stress排序值Ranking value 1.57 4.23 3.34 3.22 1.41排名Rank排名Rank排名Rank排名Rank 2 5 4 3 1激素Hormone排序值Ranking value 5.00 3.13 2.00 1.32 2.45 5 4 2 1 3嫁接Graft排序值Ranking value 3.00 3.98 4.23 1.41 1.41 3 4 5 1 1全部Total排序值Ranking value 2.28 4.00 1.57 1.41 5.00 3 2 2 1 5
3 讨 论
选择合适的内参基因来减少RT-qPCR 试验中的误差造成的影响是非常有必要的[19],参考基因标准化是可靠的RT-qPCR 分析的重要组成部分。使用参考基因是mRNA 定量数据最常作为内部对照进行标准化的方法。内参基因的使用必须对特定组织或细胞进行试验验证其效用,针对多个参考基因进行标准化,使用的单一参考基因必须经过充分验证[20]。没有一个基因在所有试验条件下都是稳定表达的,相似的,本研究发现山核桃的最优内参基因在不同组织及各种激素和胁迫处理下也存在差异,且与其他非模式植物如丹参(Salvia hispanica)、白三叶草(Trifolium repens L.)、陆地棉(Gossypium hirsutum)及冬瓜(Benincasa hispida Cogn.)等的最优内参基因有相似之处[6-10,21]。ARF基因是小G蛋白超家族的亚家族成员,在高尔基体小囊泡形成以及细胞信号传导中起重要作用[22]。马小英等[23]研究表明,在大麦(Hordeum vulgare ssp.vulgare)的30%PEG干早胁迫与盐胁迫条件下ARF 均可作为稳定的内参基因使用,这与本研究结果相近,表明ARF 基因在各物种中的保守性较好。60SRP基因是核糖体60S亚基合成的重要基因,是核糖体的重要组成部分[24],张芷睿等[25]的研究表明,60SRP 为大豆(Glycine max)叶片组织中最稳定的内参基因,笔者在本研究中发现60SRP在山核桃根、茎、叶、柱头、果皮及胚各组织中的表达最为稳定。UBC 基因是泛素化过程中的关键酶基因,在植物生长发育、非生物应激反应、植物免疫反应和DNA修复等方面起重要作用[26],刘青芝等[27]在对东方泽泻[Alisma orientale(Sam.)Juzep]不同激素处理下的内参基因稳定性研究中发现,UBC基因的表达稳定性均较高,笔者在山核桃中也得到相似结论,在山核桃叶片外源喷施不同激素后,UBC9-5 的表达最为稳定。这些结果进一步为随组织差异及试验条件变化的最优内参基因选择提供了依据。
此外,不同的评估方法对最优内参基因的准确性评价也存在一定的影响,需运用多种分析方法综合评估以确保准确性。geNorm、NormFinder、Best-Keeper 和RefFinder 是常用的内参基因分析的4 种程序。在本研究中,应用geNorm、NormFinder、Best-Keeper和RefFinder 4种数据评估方法,分析发现ge-Norm、NormFinder 的分析结果基本一致,而Best-Keeper 分析结果与前2 种方法均有明显差异,这可能是由于BestKeeper 软件在分析时直接使用CT 值这一原始数据,与geNorm 和NormFinder 软件相比有效性较差。此结果与大量先前的研究结果一致[28-29]。RefFinder 可进一步对geNorm,NormFinder和BestKeeper 的数据进行综合评估,选择稳定的单个基因或基因组合作为内部对照,是一个全面的评估程序[30]。笔者在本研究中发现在山核桃的不同组织中,ARF-1 和60SRP 这2 个内参基因稳定性最好,在不同处理下,最优的内参基因存在差异:在盐胁迫和干旱条件下,60SRP基因表达最稳定;在对山核桃叶进行不同激素处理下,UBC9-5 为最稳定基因;在山核桃砧木和接穗嫁接后,60SRP和UBC9-5基因表达最稳定。这与目前的一些研究结果是一致的,稳定性最好的内参基因因不同植物、不同组织及不同胁迫处理而不同[19-24]。这为今后分析山核桃组织差异性和激素胁迫响应差异性基因表达提供了内参基因选择依据。
4 结 论
根据19 个候选基因在山核桃不同组织中表达稳定性的差异,选出了稳定性排名前五的5个基因,分别为ARF-1、60SRP、ACT7-1、ACT1-1 和UBC9-5,其中ARF-1和60SRP基因为表达稳定性最好的两个基因;在盐胁迫和干旱条件下,山核桃叶的60SRP基因表达最稳定;在对山核桃叶进行不同激素处理下,UBC9-5 为最稳定基因;在山核桃砧木和接穗嫁接后,60SRP和UBC9-5基因表达最稳定。在综合所有组织和处理的条件下,UBC9-5为最稳定的基因。本研究为利用RT-qPCR 分析山核桃基因表达中的内参基因选择提供了重要的理论参考。
[1]FREEMAN W M,WALKER S J,VRANA K E.Quantitative RT PCR: pitfalls and potential[J].Biotechniques,1999,26(1):112-125.
[2]GACHON C,MINGAM A,CHARRIER B.Real-time PCR:what relevance to plant studies?[J].Journal of Experimental Botany,2004,55(402):1445-1454.
[3]SMITHA P K,VISHNUPRIYAN K,KAR A S,ANIL KUMAR M,BATHULA C,CHANDRASHEKARA K N,DHAR S K,DAS M.Genome wide search to identify reference genes candidates for gene expression analysis in Gossypium hirsutum[J].BMC Plant Biology,2019,19(1):1-11.
[4]KIRSCHNECK C,BATSCHKUS S,PROFF P,KÖSTLER J,SPANIER G,SCHRÖDER A.Valid gene expression normalization by RT-qPCR in studies on hPDL fibroblasts with focus on orthodontic tooth movement and periodontitis[J].Scientific Reports,2017,7(1):1-13.
[5]CHERVONEVA I,LI Y Y,SCHULZ S, CROKER S, WILSON C,WALDMAN S A,HYSLOP T.Selection of optimal reference genes for normalization in quantitative RT-PCR[J].BMC Bioinformatics,2010,11(1):1-15.
[6]ARTICO S,NARDELI S M,BRILHANTE O,GROSSI-DE-SA M F,ALVES-FERREIRA M.Identification and evaluation of new reference genes in Gossypium hirsutum for accurate normalization of real-time quantitative RT-PCR data[J].BMC Plant Biology,2010,10(1):1-12.
[7]GOPALAM R,RUPWATE S D,TUMANEY A W.Selection and validation of appropriate reference genes for quantitative realtime PCR analysis in Salvia hispanica[J].PLoS One,2017,12(11):e0186978.
[8]PU Q,LI Z,NIE G,ZHOU J,LIU L,PENG Y.Selection and validation of reference genes for quantitative real-time PCR in white clover(Trifolium repens L.)involved in five abiotic stresses[J].Plants,2020,9(8):996.
[9]叶新如,朱海生,林珲,刘建汀,王彬,陈敏氡,温庆放.冬瓜实时荧光定量PCR 内参基因的筛选与评价[J].核农学报,2019,33(3):473-481.YE Xinru,ZHU Haisheng,LIN Hui,LIU Jiangting,WANG Bin,CHEN Mindong,WEN Qingfang.Screening and evaluation of reference genes for RT-qPCR in wax gourd (Benincasa hispida Cogn.)[J].Journal of Nuclear Agricultural Sciences,2019,33(3):473-481
[10]CHEN Z S,HAN N N,LI J H,HUANG G H,WAN H.Selection of reference genes for expression analysis using RT-qPCR in the dissemination system of Heliothis virescens ascovirus 3 h(HvAV-3h)[J].Scientific Reports,2017,7(1):1-10.
[11]吕运舟,董筱昀,黄利斌.黄山栾树实时荧光定量PCR 内参基因的筛选[J].分子植物育种,2019,17(2):553-560.LÜ Yunzhou,DONG Xiaoyun,HUANG Libin.The screening of reference genes for real-time fluorescent quantitative PCR of Koelreuteria bipinnata[J].Molecular Plant Breeding,2019,17(2):553-560.
[12]程慧,孙志超,王红红,沈超,郭文磊,王正加.3 种山核桃砧木对干旱胁迫的生理响应[J].安徽农业大学学报,2016,43(6):919-925.CHENG Hui,SUN Zhichao,WANG Honghong,SHEN Chao,GUO Wenlei,WANG Zhengjia.Varied physiological responses to drought stress among three rootstocks of Carya cathayensis[J].Journal of Anhui Agricultural University,2016,43(6):919-925.
[13]王正加,张斌,夏国华,黄有军,洪丹丹,黄坚钦.山核桃×薄壳山核桃花粉直感效应与后代分析[J].果树学报,2010,27(6):908-913.WANG Zhengjia,ZHANG Bin,XIA Guohua,HUANG Youjun,HONG Dandan,HUANG Jianqin.Analysis of the progeny of Carya cathayensis×C.illinoensis and the xenia effect[J].Journal of Fruit Science,2010,27(6):908-913.
[14]HUANG X X,LI W,YOU B,TANG W P,GAN T S,Feng C,LI C F,YANG R.Serum metabonomic study on the antidepressant-like effects of ellagic acid in a chronic unpredictable mild stress- induced mouse model[J].Journal of Agricultural and Food Chemistry,2020,68(35):9546-9556.
[15]黄春颖,黄有军,吴建峰,黄仁,栾雨濛,张深梅,王正加,张启香,黄坚钦.SAD 和FAD 家族基因调控山核桃不饱和脂肪酸组分配比[J].园艺学报,2018,45(2):250-260.HUANG Chunying,HUANG Youjun,WU Jianfeng,HUANG Ren,LUAN Yumeng,ZHANG Shenmei,WANG Zhengjia,ZHANG Qixiang,HUANG Jianqin.SAD and FAD genes regulate the ratio of unsaturated fatty acid components in Carya cathayensis[J].Acta Horticulturae Sinica,2018,45(2):250-260.
[16]HUANG Y J,XIAO L H,ZHANG Z R,ZHANG R,WANG Z J,HUANG C Y,HUANG REN,LUAN Y M,FAN T Q,WANG J H,SHEN C,ZHANG S M,WANG X W,RANDALL J,ZHENG B S,WU J S,ZHANG Q X,XIA G H,XU C M,CHEN M,ZHANG L S,JIANG W K,GAO L Z,CHEN Z D,LESLIE C A,GRAUKE L J.,HUANG J Q.The genomes of pecan and Chinese hickory provide insights into Carya evolution and nut nutrition[J].GigaScience,2019,8(5):1-17.
[17]SARAVANA K R M,GAO L X,YUAN H W,XU D B,LIANG Z,TAO S C,GUO W B,YAN D L,ZHENG B S, EDQVIST J.Auxin enhances grafting success in Carya cathayensis (Chinese hickory)[J].Planta,2018,247(3):761-772.
[18]NIU K,SHI Y,MA H.Selection of candidate reference genes for gene expression analysis in Kentucky Bluegrass(Poa pratensis L.)under abiotic stress[J].Frontier in Plant Science,2017,8:193.
[19]HUANG L K,YAN H D,JIANG X M,YIN G H,ZHANG X Q,QI X,ZHANG Y,YAN Y H,XIAO M,PENG Y.Identification of candidate reference genes in perennial ryegrass for quantitative RT-PCR under various abiotic stress conditions[J].PLoS One,2014,9(4):e93724.
[20]SCHMITTGEN T D,ZAKRAJSEK B A.Effect of experimental treatment on housekeeping gene expression: validation by realtime,quantitative RT-PCR[J].Journal of Biochemical and Biophysical Methods,2000,46(1/2):69-81.
[21]BUSTIN S A,BEAULIEU J F,HUGGETT J,JAGGI R,KIBENGE F S B,OLSVIK P A,PENNING L C,TOEGEL S.MIQE précis: Practical implementation of minimum standard guidelines for fluorescence-based quantitative real-time PCR experiments[J].BMC Molecular Biology,2010,11(1):74.
[22]BROWN H A,GUTOWSKI S,MOOMAW C R,SLAUGHTER C,STERNWELS P C.ADP-ribosylation factor,a small GTP-dependent regulatory protein,stimulates phospholipase D activity[J].Cell,1993,75(6):1137-1144.
[23]马小英,贾方兴,赵颖岚,赵杰才.干旱和盐胁迫中大麦实时定量PCR 内参基因的筛选[J].分子植物育种, 2016, 14(11):3093-3101.MA Xiaoying,JIA Fangxing,ZHAO Yinglan,ZHAO Jiecai.Reference genes screening for Quantitative Real-Time PCR in barley under drought and salt stress[J].Molecular Plant Breeding,2016,14(11):3093-3101.
[24]ESPINAR- MARCHENA F,RODRÍGUEZ- GALÁN O,FERNÁNDEZ- FERNÁNDEZ J,LINNEMANN J,DE LA CRUZ J.Ribosomal protein L14 contributes to the early assembly of 60S ribosomal subunits in Saccharomyces cerevisiae[J].Nucleic Acids Research,2018,46(9):4715-4732.
[25]张芷睿,张耀华,王秋实,于慧,杨素欣.大豆实时荧光定量PCR 内参基因的筛选与验证[J].植物生理学报,2020,56(9):1963-1973.ZHANG Zhirui,ZHANG Yaohua,WANG Qiushi,YU Hui,YANG Suxin.Screening and validation of reference genes for real-time quantitative PCR in soybean[J].Plant Physiology Journal,2020,56(9):1963-1973.
[26]LIU W G,TANG X,QI X H,FU X,GHIMIRE S,MA R,LI S G,ZHANG N,SI H J.The ubiquitin conjugating enzyme:an important ubiquitin transfer platform in ubiquitin-proteasome system[J].International Journal of Molecular Sciences,2020,21(8):2894.
[27]刘青芝,谷巍,孙云飞,吴启南,巢建国,桑晓华,王小浩,刘琪.东方泽泻实时荧光定量PCR 内参基因的筛选及AoSS 基因的组织表达特性分析[J].南方农业学报,2018,49(8):1467-1475.LIU Qingzhi,GU Wei,SUN Yunfei,WU Qinan,CHAO Jianguo,SANG Xiaohua,WANG Xiaohao,LIU Qi.Selection of reference genes of Alisma orientale(Sam.)Juzep by real-time fluorescence quantitative PCR and expression characters of AoSS in tissues[J].Journal of Southern Agriculture,2018,49(8):1467-1475.
[28]DELPORTE M,LEGRAND G,HILBERT J L,GAGNEUL D.Selection and validation of reference genes for quantitative realtime PCR analysis of gene expression in Cichorium intybus[J].Frontier in Plant Science,2015,6:651.
[29]LI L,LI N,FANG H,QI X,ZHOU Y.Selection and validation of reference genes for normalisation of gene expression in Glehnia littoralis[J].Scientific Reports,2020,10(1):1-12.
[30]XIE F L,XIAO P,CHEN D L,XU L,ZHANG B H.miRDeep-Finder: A miRNA analysis tool for deep sequencing of plant small RNAs[J].Plant Molecular Biology,2012,80(1):75-84.