核桃(Juglans regia L.)别名胡桃、羌桃,是一种经济价值很高的经济林木,在我国具有悠久的栽培历史。目前,国内外广泛栽培的核桃品种叶片和果皮为绿色、种皮呈黄白色或浅黄色,性状单一。我国作为核桃的原产地之一,种质资源类型十分丰富[1-2]。红仁核桃是一种较为稀少的种质,目前在中国和美国都有发现,并育成数个品种。其叶片、果皮、种皮等组织均可呈红色,具有一定的经济价值。研究证实,红色性状的表现是因为这些组织中具有花青苷的合成与积累[3-4]。
花青苷是黄酮类代谢物,广泛分布于植物组织器官中,是植物组织呈现颜色的重要色素[5]。花青苷的生物合成受内在因素(主要是内源糖信号和激素)、环境因素(如温度、光照等)以及一系列酶反应和转录因子的调控。查尔酮合成酶(chalcone synthetase,CHS)、查尔酮异构酶(chalcone isomerase,CHI)、二氢黄酮醇4-还原酶(dihydroflavonol 4-reductase,DFR)、花青苷合成酶(anthocyanidin synthase,ANS)等是由结构基因编码的花青苷合成关键酶。MYB、bHLH、WD40 等转录因子以及它们形成的MBW 复合物则调控着这些关键酶的时空表达[6]。
查尔酮合成酶(CHS)是类黄酮生物合成途径中的第一个关键酶,其催化的3分子的丙二酰-CoA和1分子的对香豆酰-CoA结合形成查尔酮的反应更是整个过程中的重要限速步骤[7]。首个CHS 基因的cDNA 序列于1983 年从欧芹细胞悬浮培养物中获得,之后多种植物中的CHS 基因陆续被克隆[8]。大量研究表明,CHS能够调控花青苷的空间表达及累积水平。甘薯IbCHS1 主要在紫肉甘薯中表达,其表达水平与花青苷积累呈显著正相关[9];苹果果实发育过程中CHS 与F3H、DFR、ANS、UFGT 等基因协同表达,表达水平与花青素积累呈正相关[10];红色桃和油桃果实中的CHS 和DFR 是花青素合成的关键调节基因[11];我国的红色砂梨品种‘美人酥’果实中分离得到了PpCHS,其花青素积累水平与Py-MYB10 和多生物合成基因水平协调表达密切相关[12]。
CHS 在植物花青苷合成和代谢的调控网络中起着重要作用,但其在红仁核桃的功能与表达尚不清楚。日前,Zhang等[13]获得了核桃染色体水平的优质基因组序列,Guo等[14]开发并建立了胡桃科(PJU)的门户网站,;鱼尚奇等[15]对纸皮核桃内果皮硬化期样品进行转录组测序,筛选出了内果皮硬化期差异表达的基因并进行了生物信息功能预测。这些基础研究为胡桃科比较基因组学的研究以及核桃品种的遗传改良提供了重要的理论参考。因此,笔者基于红仁核桃自然杂交后代不同表型(红叶和绿叶)转录组测序,结合核桃最新基因组数据,对核桃CHS 基因时空表达与结构、功能进行分析,为探明JrCHSs基因调控红仁核桃花青苷的生物合成机制及核桃分子育种提供理论基础。
1 材料和方法
1.1 试验材料
红仁核桃RW-1(J.regia L.)是课题组前期发现并命名的一个太行山野生红色特异种质资源[16]。为了保持遗传背景的一致性,试验选取红仁核桃自然杂交后代2 a(年)生苗的不同表型(红叶和绿叶)株系为试材。叶片共3 个取样时期,依照红叶颜色变化分为叶片发育早期[即幼叶期(2019年4月6日),叶片全红色;命名为时期1]、叶片发育中期[即新梢旺长、果实迅速膨大期(2019 年5 月29 日),叶片红绿相间,命名为时期2]、叶片发育后期[即果实成熟早期(2019 年6 月21 日),叶片成熟近于老化,趋于全绿色,命名为时期3]。试材均种植于河南农业大学园艺学院果树试验站,南北向定植,株行距2 m×3 m,常规管理。选择生长一致、长势良好的植株,采样后立即放入液氮速冻,-80 ℃冰箱保存备用以进行转录组测序。
1.2 花青素含量测定分析
将样品真空冷冻干燥后研磨成粉末,称取100 mg的粉末,溶解于1.0 mL提取液(70%甲醇,φ)中,4 ℃冰箱过夜,期间涡旋3次。离心(1000 g,10 min)后,吸取上清,用微孔滤膜(0.22 μm pore size)过滤样品,并保存于进样瓶中,用于LC-MS/MS 分析[17],并以矢车菊素-3-O-葡萄糖苷的相对含量代表各样品的花青素相对含量[18]。
1.3 RNA提取及转录组测序
叶片总RNA提取、纯度检查和完整性评估按照课题组之前描述的方法进行[16]。提取的总RNA 经DNase Ⅰ处理后用于RNA 测序文库的构建。使用TruSeq Stranded mRNA 样品制备试剂盒(Illumina,sandiego,CA,USA)制备RNA测序文库。转录组测序由Illumina HiSeq 2500 测序平台完成,测序结果与核桃参考基因组进行比对分析。
1.4 红仁核桃花青苷合成相关差异表达CHS 基因的筛选
根据转录组测序结果获得各基因功能的注释及其在红仁核桃自然杂交后代不同表型叶片各发育时期的表达水平,利用TBtools 构建核桃CHSs 基因表达图谱[19]。筛选在不同样品中显著差异表达的CHS基因为研究对象,从测序结果中下载得到其核苷酸序列与氨基酸序列进行基因结构与功能分析。
1.5 核桃CHSs基因qRT-PCR分析
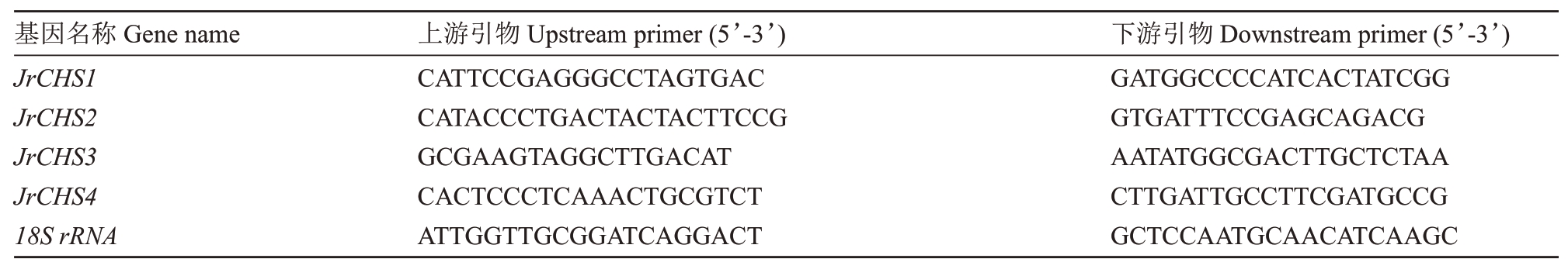
以不同表型叶片各发育时期的总RNA为模板,参照FastQuant RT(One-Step gDNA Removal;Trans-Gen,Beijing,China)说明书合成第一链cDNA。荧光定量引物由Primer Premier 5.0 设计,引物序列见表1。qRT-PCR 采用ChamQ Universal SYBR qPCR Master Mix 试剂盒(Vazyme,Nanjing,China)于LightcyClerR 480 Ⅱ检测系统(Applied Biosystems,Foster City,CA,USA)进行。以18S rRNA(XM_019004991.1)作为内参基因。用2-ΔΔCt法分析不同基因的表达水平[20]。
表1 qRT-PCR 引物序列
Table 1 Primers used for qRT-PCR
?
1.6 核桃JrCHSs基因结构与功能分析
利用测序得到的CHSs 基因cDNA 序列获取其编码区长度与氨基酸数量,并在NCBI 数据库中通过BLAST 查找其基因ID。利用ExPASy 工具预测CHS蛋白等电点(pI)和相对分子质量,Cell-PLoc在线软件分析蛋白亚细胞定位。使用TBtools 软件对核桃CHS 基因进行了结构与染色体定位的预测分析,蛋白保守基序分析使用MEME(Multiple Expectation Maximization for Motif Elicitation)在线软件。利用NCBI 数据库CD-Search 分析核桃CHS 的结构域;亲疏水性与蛋白跨膜分析分别使用ProtScale和TMpred软件进行,蛋白质二级及三级结构的预测分别利用SOPMA 和SWISS MODEL 完成。利用MEGA 7.0 软件进行多物种CHS 蛋白序列比对分析,采用NJ法构建系统进化树。选取CHS基因上游2000 bp 片段在PlantCARE 软件中预测分析启动子顺式作用元件。
1.7 数据分析
所有数据的采集和计算均设3 组生物学重复。其中,利用SPSS 17.0和Excel 2007进行数据处理与统计分析;采用单因素方差分析(p <0.05)、独立样本t检验(p <0.01)进行差异显著性分析。
2 结果与分析
2.1 红仁核桃自然杂交后代不同表型叶片颜色观察及其花青素相对含量
红仁核桃自然杂交后代不同颜色叶片表型观察如图1-A所示。时期1,2种叶片的颜色表型差异最大,自然杂交后代红叶(SR)为全红色,而自然杂交后代绿叶(SG)为嫩绿色。随着叶片的生长发育,红叶颜色变化显著,时期2为红绿相间,时期3趋于全绿色;而绿叶颜色变化较小,始终为绿色。
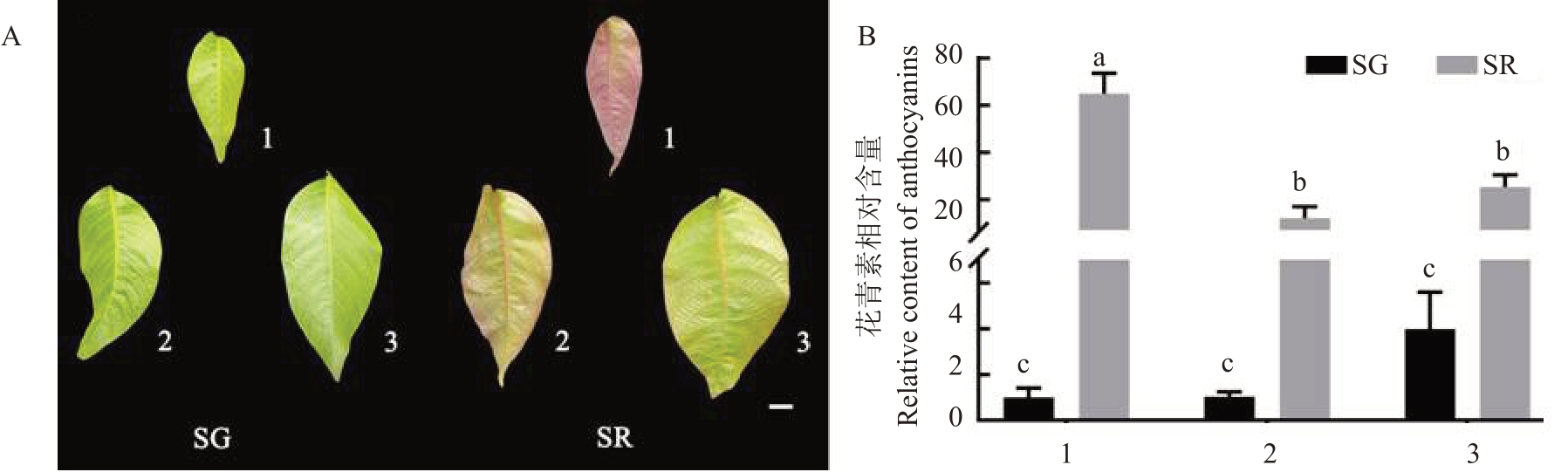
图1 红仁核桃自然杂交后代不同颜色表型叶片及其花青素相对含量
Fig.1 The different color phenotype leaves of natural hybrid progenies of red walnut
A.红仁核桃自然杂交后代不同颜色叶片表型;B.不同表型叶片在各发育时期的花青素相对含量;SG.自然杂交后代绿叶;SR.自然杂交后代为红叶;1.叶片发育早期,为全红色;2.叶片发育中期,红绿相间;3.叶片发育后期,叶片成熟近于老化,趋于全绿色;比例尺=1 cm。不同小写字母表示在p <0.05 差异显著。下同。
A.Phenotype of different color leaves in natural hybrid progenies of red walnut;B.Relative content of anthocyanins in leaves of different phenotypes at different developmental stages; SG. Seedling progenies-Green leaves; SR. Seedling progenies-Red leaves; 1.The early stage, the leaves are all red;2.The middle stage,the leaves are red and green;3.The later stage,the leaves are close to aging and tend to be all green;Bar=1 cm.The different small letters indicate significant difference at p <0.05.The same below.
两种叶片在不同发育时期的花青素相对含量如图1-B所示。红叶(SR)在各个时期的花青素相对含量均显著高于绿叶(SG),与两种叶片颜色表型差异相符;随着叶片的生长发育,红叶中的花青素相对含量呈先下降后上升的趋势,且时期1 的花青素相对含量显著高于其他时期;绿叶中的花青素相对含量呈持续上升趋势,但差异不显著。
2.2 基于转录数据红仁核桃花青苷合成相关差异表达CHS基因的筛选及qRT-PCR分析
为筛选获得红仁核桃花青苷合成相关差异表达CHS 基因,对红仁核桃自然杂交后代不同表型叶片进行了转录组测序。18 个叶片样品共获得134 Gb Clean Data,各样品Clean Data 超过6.47 Gb,Q30 碱基百分比超过95.22%,与参考基因组的比对效率为92.23%~95.27%。这些数据均表明测序结果较为可靠,与参考基因组比对较好,文库质量较高,可为后续结构与功能分析的准确性提供保障。
分析转录组测序结果,共有17个基因被功能注释为“Chalcone synthetase”。以P value<0.05、FC≥1.5 为标准,筛选获得4 个CHSs(gene35863、gene4994、gene39336、gene32601)的表达量在不同表型叶片之间存在显著差异。如图2-A 所示,4 个CHSs的表达量均在时期1的红叶中显著高于绿叶,因此可将上述4个基因作为研究对象进行下一步分析与功能研究。进一步探究各基因表达模式,发现4个JrCHSs基因在两种颜色叶片的不同发育时期均有表达,且均在时期1的表达量存在显著差异,与该时期表型相符;时期2,除gene35863 在红叶中表达量略高于绿叶外,gene4994、gene39336、gene32601均在绿叶中的表达量高于红叶,与该时期红叶变绿的表型呈正相关;在时期3,除gene4994在绿叶中表达量略高于红叶外,gene32601、gene39336、gene35863均在红叶中的表达量高于绿叶。
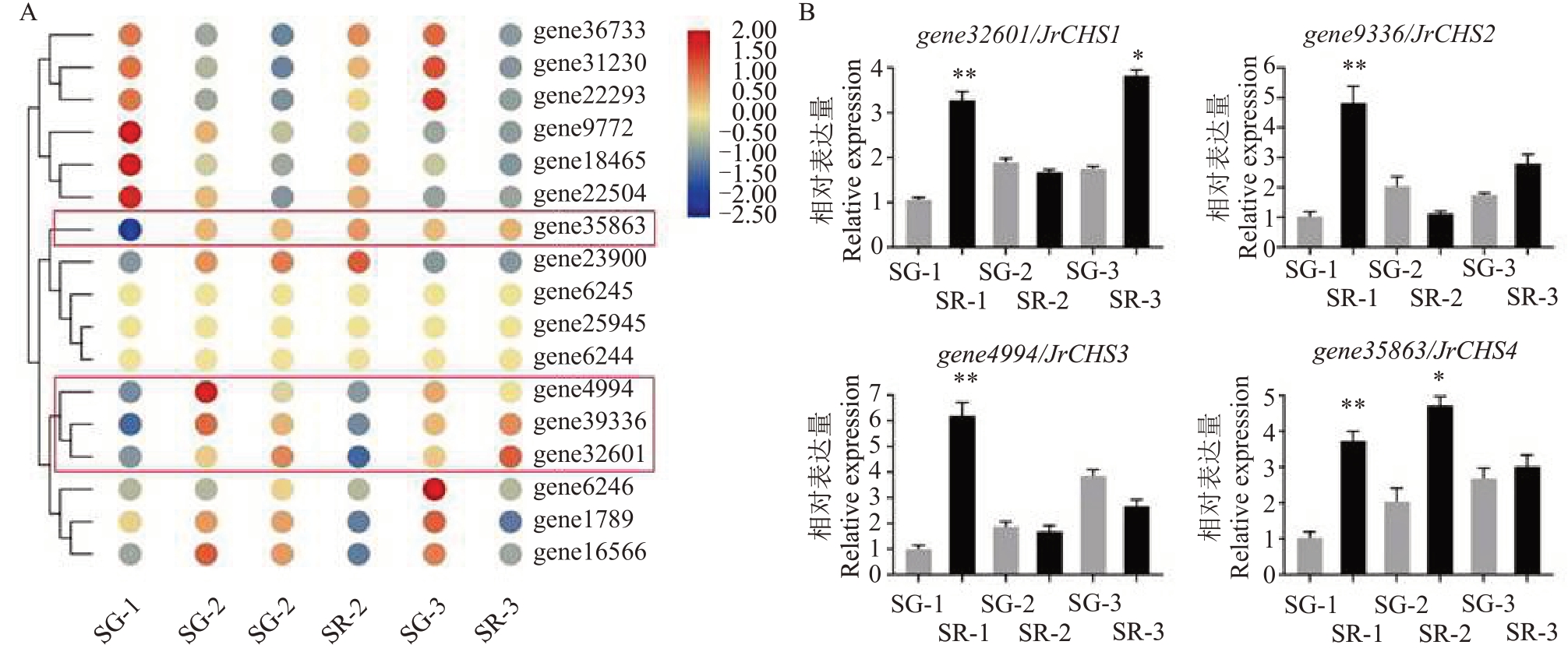
图2 红仁核桃自然杂交后代不同表型叶片中CHSs 基因表达谱及候选基因qRT-PCR 分析
Fig.2 CHS genes expression profile and qRT-PCR analysis of candidate genes in different phenotypic leaves of red walnut progeny
A.红仁核桃自然杂交后代不同表型叶片中CHSs 基因表达谱,比例尺表示RPKM 值的log2 转换值;B.候选CHS 基因的qRT-PCR 分析,在同一时期的不同表型叶片之间进行显著性差异比较,“*”表示在p <0.05 差异显著,“**”表示在p <0.01 差异显著。
A. CHS genes expression profile in different phenotypic leaves of red walnut progeny,the scale bars represent the log2 transformations of the RPKM values. B. qRT-PCR analysis of candidate CHS genes,significant differences were compared among different phenotypic leaves in the same stage,“*”indicated significant difference at p <0.05,“**”indicated significant difference at p <0.01.
对上述基因进行qRT-PCR分析(图2-B)的结果表明,gene32601在时期3的红叶(SR-2)中表达量最高,而gene35863、gene4994、gene39336 均在时期1的红叶(SR-1)中表达量最高,且4个基因均在时期1的红叶中表达量极显著高于绿叶。随着叶片的生长发育,gene35863、gene4994、gene39336 在时期2、3两种叶片中的表达量差异减小且不显著,而gene32601 的表达量在时期1、3 两种叶片间均存在显著差异。qRT-PCR进一步验证了转录组数据结果的可靠性,可对候选基因进行后续功能验证。
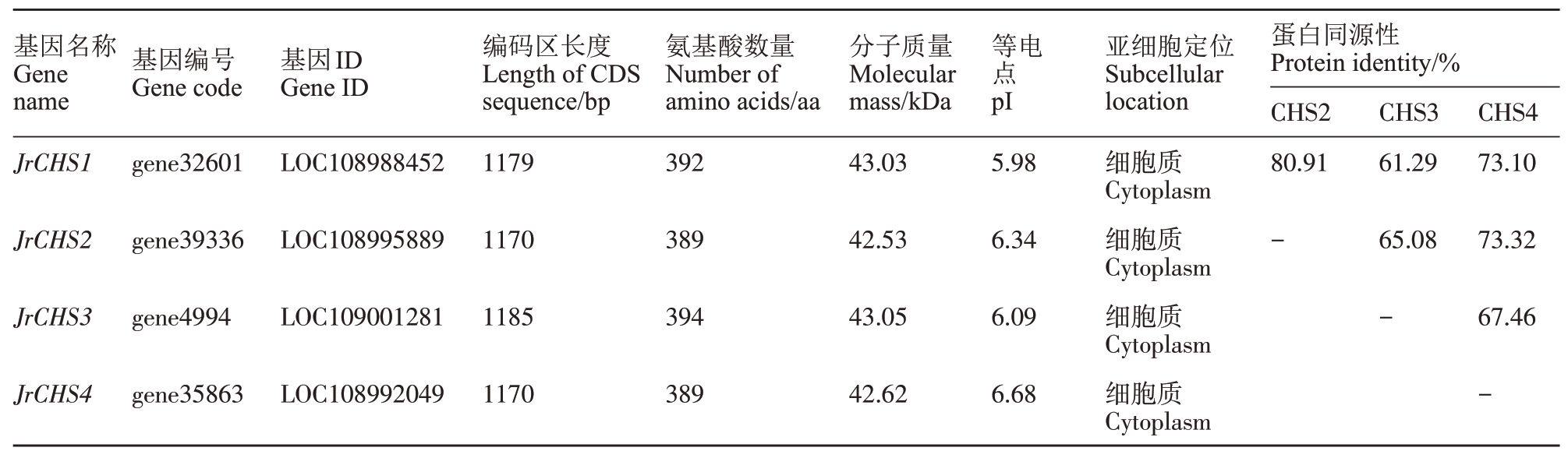
2.3 核桃CHS 基因的染色体定位及蛋白理化性质分析
根据CHSs在染色体上的位置信息,绘制核桃染色体物理图谱并进行基因定位(图3-A),gene-32601、gene39336、gene4994、gene35863分别位于1、2、3、7号染色体上的不同位点,gene32601、gene4994和gene35863 分别位于1、3 和7 号染色体上臂,而gene39336 位于2 号染色体下臂。将4 个核桃CHS基因根据其在染色体上的定位信息依次命名为JrCHS1~JrCHS4,其基因信息及理化性质如表2 所示。4 个JrCHS 基因CDS 序列长度分别为1179、1170、1185 和1170 bp;4 个CHS 蛋白氨基酸数目分别为392、389、394和389;蛋白相对分子质量分别为43.03、42.53、43.05 和42.62 kDa;等电点分析表明均偏酸性。对4 个JrCHS 进行亚细胞定位预测,发现均主要分布于细胞质中。此外,JrCHS 蛋白之间有较高的同源性(61.29%~80.91%),且JrCHS1 和JrCHS2蛋白同源性明显高于其他序列。

图3 核桃CHSs 基因结构、蛋白保守基序与结构域及染色体定位分析
Fig.3 Analysis of CHSs gene structure,protein conserved motif,domain and chromosome location in walnut
A.JrCHS 基因在染色体上的位置,Chr 1、2、3、7 分别代表4 条染色体,基因位置和染色体长度用左侧的标尺(bp)测量;B.JrCHS 基因内含子-外显子结构 .CDS
.CDS .内含子;C.JrCHS 蛋白保守基序,分别用不同颜色表示;D.motifs 序列图;E.JrCHS 蛋白保守结构域。
.内含子;C.JrCHS 蛋白保守基序,分别用不同颜色表示;D.motifs 序列图;E.JrCHS 蛋白保守结构域。
A. The position of JrCHS genes on chromosome, and chr1, 2, 3 and 7 represent 4 chromosomes respectively, gene position and chromosome length are measured with left ruler (bp); B. The intron exon structure of JrCHS genes, . CDS,“
. CDS,“ ”. Intron; C. The conserved motifs of JrCHS proteins,which is indicated by different colors;D.Protein conserved motif logos;E.The conservative domain of JrCHS proteins.
”. Intron; C. The conserved motifs of JrCHS proteins,which is indicated by different colors;D.Protein conserved motif logos;E.The conservative domain of JrCHS proteins.
表2 核桃CHS 基因信息与蛋白理化性质
Table 2 Gene information and physicochemical properties of CHSs in walnut
?
2.4 核桃CHS基因结构、蛋白保守基序及功能结构域分析
对4 个核桃CHSs 基因进行结构分析(图3-B),其内含子-外显子分布结果表明,JrCHSs 的基因结构较为相似,均只含有1 个内含子,且分布差异较小。JrCHS 蛋白的保守基序分析结果见图3-C~D,4 个JrCHS 蛋白保守基序结构中均含有5 个motif,且蛋白基序位置均相同。利用氨基酸序列进行保守结构域分析(图3-E),发现JrCHS1 和JrCHS2 含有PLN03173 型结构域,而JrCHS3 和JrCHS4 含PLN03172 型结构域,均属于CHS 功能结构域。
2.5 核桃CHS蛋白跨膜结构与亲疏水性分析
通过在线预测各基因编码蛋白的跨膜结构,发现JrCHS1~JrCHS4 蛋白均位于膜外,不存在跨膜区域。蛋白亲疏水性结果显示,4 个JrCHS 的亲疏水性分值的平均值均小于0,则表明JrCHS1~JrCHS4均为亲水性蛋白。其中,JrCHS1~JrCHS4 蛋白均为第119 位氨基酸(谷氨酰胺,Gln)亲水性最大,分值分别为-2.489、-2.489、-2.667,-2.556;JrCHS1、JrCHS3 和JrCHS4 蛋白均为第342 位氨基酸(缬氨酸,Val)疏水性最大,且分值均为2.422,而JrCHS2蛋白为第191 位氨基酸(半胱氨酸,Cys)疏水性最大,分值为2.611。
2.6 核桃CHS蛋白二级结构与三级结构预测分析
核桃CHS蛋白二级结构预测结果如表3所示,4个JrCHS 蛋白的二级结构均由4 种构象组成,包含α-螺旋(Alpha helix)、延伸链(Extended strand)、β-转角(Beta turn)以及无规则卷曲(Random coil),其中α-螺旋比例最高,分别为46.27%、47.30%、42.39%和44.99%,其次是无规则卷曲,分别为31.36%、31.88%、35.28%和32.90%,延伸链和β-转角比例较低,分别为15.17%和7.20%、14.40%和6.43%、15.74%和6.60%以及15.42%和6.68%。蛋白三级结构预测建模结果表明,4个蛋白空间结构较为相似,均由单螺旋结构蛋白亚基构成。
表3 核桃CHS 蛋白二级结构比例
Table 3 Secondary structure proportion of CHS protein in walnut %

?
2.7 核桃CHS 蛋白系统进化关系与多序列比对分析
利用4 个核桃CHS 蛋白与拟南芥Arabidopsis thaliana AtCHS(CAI30411.1)、AtTT4(AAF23561.1),葡萄Vitis vinifera VvCHS1 (AB015872)、VvCHS2(AB066275)、VvCHS3 (AB066274),柑橘Citrus Sinensis CsCHS (BAA81664.1),石榴Punica granatum PgCHS1 (AHZ97870.1)、PgCHS2 (ALT22073.1),木薯Manihot esculenta MeCHS(XP021609389.1),猕猴桃Actinidia chinensis AcCHS(KF157394.1);蜜柑Citrus unshiu CuCHS(FJ887898.1);蓝莓Vaccinium ashei VaCHS(AB694902.1);龙眼Dimocarpus longan DlCHS(AEO36981.1);荔枝Litchi chinensis LcCHS(ADB44076.1);柠檬Citrus limon ClCHS(AKR05585.1);甜樱桃Prunus avium PaCHS(AJO67963.1);苹果 Malus domestica MdCHS(ACJ54530.1);鸡爪槭Acer palmatum ApCHS(AFM74036.1);毛果杨Populus trichocarpa PtCHS(XP_002321081.1);巨桉Eucalyptus grandis EgCHS(XP_010028921.1);金银花 Lonicera japonica LjCHS(AGE10597.1);碧桃Prunus persica PpCHS(AJA79072.1);金花茶Camellia nitidissima CnCHS(ADZ28512.1);黄秋葵Abelmoschus esculentus AeCHS(AGW22222.1);杜鹃花Rhododendron simsii RsCHS(CAC88858.1);盐肤木Rhus chinensis RcCHS(AGH13332.1);陆地棉Gossypium hirsutum GhCHS(ABS52573.1)以及荷花Nelumbo nucifera NnCHS(XP_010249317.1)等24 个物种的CHS 蛋白构建系统发育树(图4-A),发现32 个蛋白可聚为3个类群。拟南芥作为一种模式植物,与其他物种亲缘关系较远,单独为一类。核桃JrCHS1、JrCHS2 归于同一类别,与金花茶CnCHS、杜鹃花RsCHS 以及蓝莓VaCHS亲缘关系较近;核桃JrCHS3、JrCHS4归于一组,其中JrCHS3 与毛果杨PtCHS、荔枝LcCHS以及龙眼DlCHS 亲缘关系较近,而JrCHS4 与木薯MeCHS 及石榴PgCHS1、PgCHS2 同源性较高,推测其功能也相似。多序列比对分析(图4-B)表明,各物种CHS 蛋白的整体一致性为78.01%,相似度较高,具有相同保守结构。

图4 核桃与其他物种CHS 蛋白系统进化树分析与多序列比对
Fig.4 Phylogenetic tree analysis and multiple sequence alignment of CHS protein system in walnut and other species
A.核桃与其他物种CHS 蛋白系统进化树;B.多序列比对结果,蓝色表示50%一致性的氨基酸序列,红色表示75%一致性的氨基酸序列,黑色表示100%一致性的氨基酸序列。
A.Phylogenetic tree of CHS protein system in walnut and other species;B.Multiple sequence alignment results of JrCHS proteins.The blue boxes indicate 50%identity of amino acids,the red boxes indicate 75%identity of amino acids,the black boxes indicate 100%identity of amino acids.
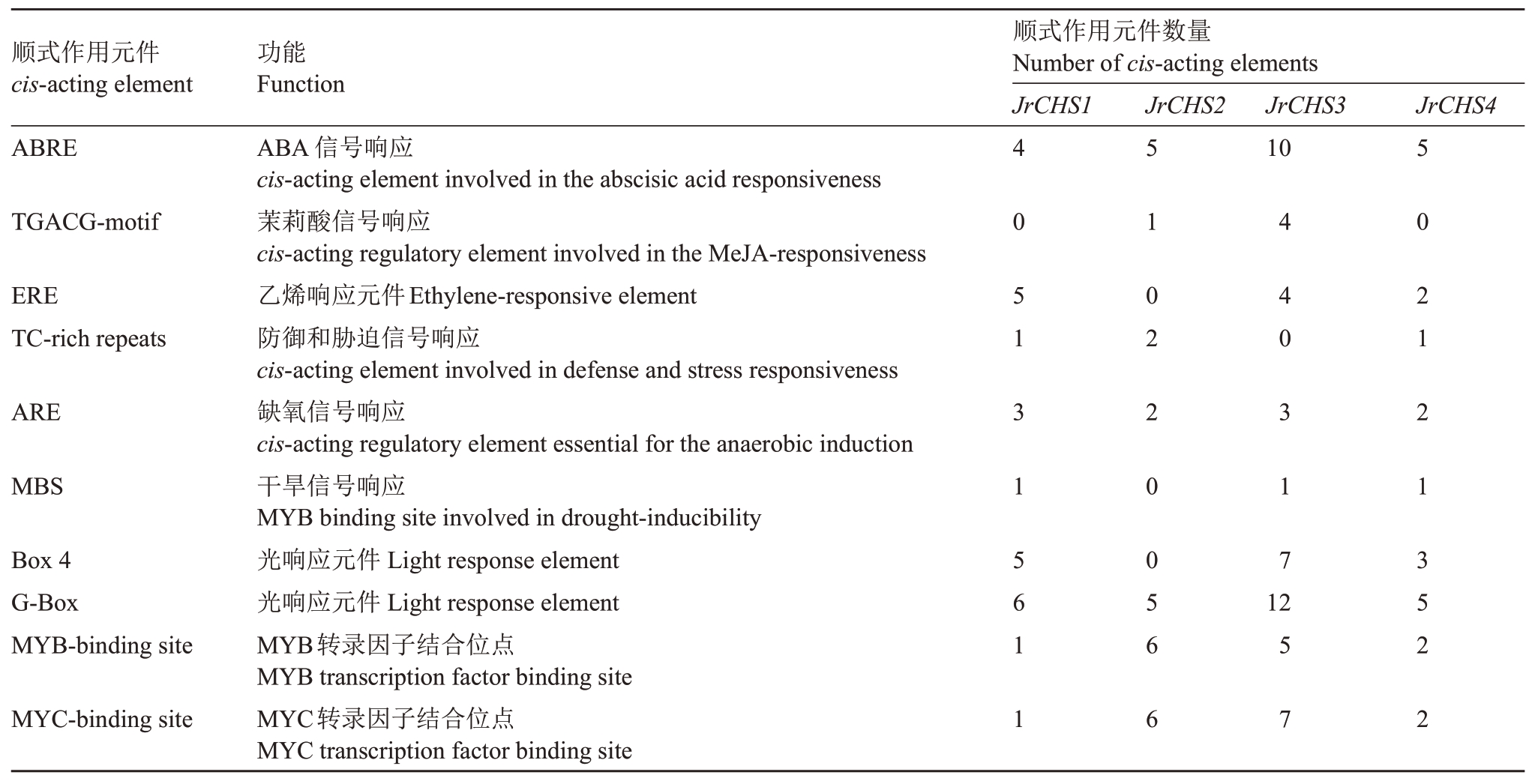
2.8 核桃CHS 基因启动子序列关键顺式作用元件分析
启动子是决定基因转录起始的重要作用因子,其序列中包含多个基本作用元件,发挥着不同的功能。为深入探究核桃CHS 基因功能,选取起始密码子上游2000 bp 的序列作为启动子区,利用PlantCare 软件预测4 个JrCHS 基因上游启动子元件,最终选取10 个数量最多的关键顺式作用元件进行分析(表4)。结果显示,各基因启动子除含有核心启动子序列TATA-box、增强子元件CAAT-box 等典型真核生物的启动子元件外,还包含大量激素响应作用元件(ABRE、TGACG-motif、ERE)、胁迫响应作用元件(TCrich repeats、ARE、MBS)、光响应元件(Box 4、GBox)以及转录因子结合位点作用元件(MYBbinding site、MYC-binding site)等。 此外,各JrCHS 基因启动子序列中均包含多个MYB 转录因子结合位点(CAACAG/CAACCA/CAACTG)和MYC 转录因子结合位点(CATTTG/CATGTG/TCTCTTA),表明JrCHSs 基因作为花青苷生物合成途径重要的结构基因,可与MYB、MYC 等调控因子结合共同作用于红仁核桃花青苷的合成与积累。
表4 核桃CHSs 基因启动子序列关键顺式作用元件分析
Table 4 Pivotal cis-element analysis in promoter regions of CHS genes in walnut
?
3 讨 论
CHS 基因是一个超基因家族,广泛存在于各种植物中。桃[21]、葡萄[22]和苹果[23]等多种果树中的CHS基因已被克隆出来。研究表明,CHS 基因除在拟南芥、欧芹和金鱼草中发现是单拷贝基因外,其他物种的CHS 是多基因家族[24],‘巨峰’葡萄中含有4 个CHS 基因[25],番茄[26]、大豆[27]中均含有8 个CHS 基因。本研究基于转录组测序数据,共获得17个核桃CHS 基因,进一步筛选得到了4 个在红仁核桃自然杂交后代不同表型叶片之间存在显著差异表达的JrCHSs(JrCHS1~JrCHS4),对其基本信息及编码蛋白的理化性质进行预测分析。其中,4 个JrCHS 基因的基因结构,均只含有2个外显子和1个内含子,与百合[28]、观赏桃[29]等植物中CHS 的基因结构相符。4个JrCHS基因编码蛋白的保守基序结构极为相似,这为后续验证各基因在花青苷生物合成途径中的调控作用提供了一定的理论基础。
为研究4个JrCHS基因在红仁核桃花青苷生物合成途径中的作用,构建了蛋白系统发育树,发现JrCHS1、JrCHS2与金花茶CnCHS、杜鹃花RsCHS及蓝莓VaCHS 亲缘关系较近,JrCHS3 与毛果杨PtCHS、荔枝LcCHS 以及龙眼DlCHS 亲缘关系较近,JrCHS4与木薯MeCHS及石榴PgCHS1、PgCHS2亲缘关系较近,已有研究表明CnCHS、VaCHS、LcCHS、DlCHS、MeCHS 及PgCHSs 均参与花青苷的生物合成[30-35],因此推测4个JrCHS基因在红仁核桃花青苷的合成与积累中能够起到一定的调控作用。此外,对JrCHSs 启动子顺式作用元件进行分析,发现各基因启动子序列中均含有大量关键作用元件。ABA能够促进类黄酮合成,而内源激素促进类黄酮合成大多数是通过提高酶活性或促进成熟来完成[36]。JrCHSs 的启动子序列中包含ABA 响应的顺式作用元件(ABRE),推测其可能受到ABA 等内源激素的调节而影响花青苷的合成与积累[37]。MYB与MYC(bHLH)是植物花青苷合成代谢途径中重要的转录调控因子,其单独或共同作用均可调控花青苷结构基因启动子活性来促进或抑制该基因的表达[38]。本研究中4 个JrCHSs 启动子中含有多个MYB-binding site、MYC-binding site作用元件,表明其活性均受到单个或多个转录因子作用以调控其表达水平,从而影响花青苷的生物合成与积累[39]。本研究对启动子基本作用元件的预测与分析,为后续基因功能的研究提供了基础。
植物体内花青苷的合成与积累是一个复杂的系统工程,在其代谢过程中的每个阶段均由大量直接与间接相关的基因参与调控,不同基因在植物体不同组织及不同发育时期的表达模式并不相同[6]。周军等[25]报道,葡萄CHS4基因在开花后表达量升高然后迅速降低,至果实将近成熟时又一次升高,果实成熟后降低。为探明相关基因在红仁核桃花青苷积累不同阶段的表达情况与颜色表型及其花青素含量之间的关系,分析红仁核桃自然杂交后代不同表型(红叶SR 和绿叶SG)转录组数据结果,发现4 个JrCHS基因在两种颜色叶片的不同发育时期均有表达,且均在时期1 红叶中的表达量显著高于绿叶,这与该时期颜色表型差异最大及红叶中花青素含量显著大于绿叶相符,而时期2除JrCHS4在红叶中表达量略高于绿叶外,JrCHS1~JrCHS3 均在绿叶中的表达量高于红叶,可认为是该时期红叶中花青苷的合成受阻,降解加速,导致了CHS基因的表达量降低,从而表型向绿色转变,其花青素含量也随之减少。时期3,红叶表型转变结束为绿色,而叶脉仍呈红色,此时花青苷合成与积累趋于稳定,JrCHSs表达量及其花青素含量在红叶中仍高于绿叶。综上所述,4 个JrCHS基因的表达水平与叶片颜色表型变化及其花青素含量存在正相关。qRT-PCR分析结果进一步验证了转录组结果,同时为证实4 个JrCHS 基因与红仁核桃花青苷生物合成调控相关提供了试验依据。此外,实时定量分析显示JrCHS1 的表达量在时期1、3 两种叶片间均存在显著差异,其发挥的作用还需进一步探究。
红仁核桃是一种少见的普通核桃树种的变异类型,涉及大量花青苷的合成与积累。CHS作为花青苷代谢途径中的关键作用酶,催化香豆酰辅酶A 和丙二酰辅酶A 生成柚皮素查尔酮,在其他酶的协同作用下合成一系列黄酮类化合物,调控着植物体内花青苷的生物合成[40]。然而,CHS 基因的功能与其代谢活跃程度、植物体发育阶段、外界环境胁迫等也有密切关系,对于单个CHS基因的调控机制有待更深入研究。此外,花青苷生物合成过程由多基因、多种代谢途径共同参与作用,CHS 等结构基因还会与3种转录因子MYB、bHLH和WD40形成的MBW复合物结合来促进或抑制花青苷代谢[41]。
4 结 论
本试验基于红仁核桃自然杂交后代不同表型(红叶和绿叶)转录组测序结果,筛选得到4 个显著差异表达CHS基因JrCHS1~JrCHS4,其均与其他物种花青苷合成相关基因具有较高同源性,且各基因启动子序列中均包含多个MYB、MYC 转录因子结合位点作用元件。JrCHSs 的表达水平与叶片颜色表型变化及其花青素含量呈正相关,初步推断为红仁核桃花青苷生物合成相关差异表达基因。
[1] 敬丹,骆翔,陈利娜,夏小丛,杨选文,李好先,王企,曹尚银.核桃油酸脱氢酶基因JrFAD2 的克隆及表达分析[J].果树学报,2020,37(10):1475-1486.JING Dan,LUO Xiang,CHEN Lina,XIA Xiaocong,YANG Xuanwen,LI Haoxian,WANG Qi,CAO Shangyin.Cloning and expression analysis of oleate dehydrogenase gene JrFAD2 in walnut[J].Journal of Fruit Science,2020,37(10):1475-1486.
[2] 马庆国,乐佳兴,宋晓波,周晔,裴东.新中国果树科学研究70年:核桃[J].果树学报,2019,36(10):1360-1368.MA Qingguo,YUE Jiaxing,SONG Xiaobo,ZHOU Ye,PEI Dong. Fruit scientific research in New China in the past 70 years: Walnut[J]. Journal of Fruit Science,2019,36(10): 1360-1368.
[3] 王克建,郝艳宾,齐建勋,胡小松.红色核桃仁种皮提取物紫外-可见光谱和质谱分析[J].光谱学与光谱分析,2009,29(6):1668-1671.WANG Kejian,HAO Yanbin,QI Jianxun,HU Xiaosong.Analysis of the extraction of red pellicle of walnut (Juglans regia L.)by ultraviolet-visible spectra and HPLC-ESI-MSn[J].Spectroscopy and Spectral Analysis,2009,29(6):1668-1671.
[4] 李永洲,尚军华,周奕菲,吴文江,揭波,吴国良.UPLC-PDAMS/MS 测定红瓤核桃中花青苷类物质[J].食品科学,2018,39(6):207-214.LI Yongzhou,SHANG Junhua,ZHOU Yifei,WU Wenjiang,JIE Bo,WU Guoliang. Determination of anthocyanins in redfleshed walnut by ultra performance liquid chromatography-electrospray ionization tandem mass spectrometry[J].Food Science,2018,39(6):207-214.
[5] 刘晓芬,李方,殷学仁,徐昌杰,陈昆松.花青苷生物合成转录调控研究进展[J].园艺学报,2013,40(11):2295-2306.LIU Xiaofen,LI Fang,YIN Xueren,XU Changjie,CHEN Kunsong.Recent advances in the transcriptional regulation of anthocyanin biosynthesis[J]. Acta Horticulturae Sinica,2013,40(11):2295-2306.
[6] MISYURA M,COLASANTI J,ROTHSTEIN S J.Physiological and genetic analysis of Arabidopsis thaliana anthocyanin biosynthesis mutants under chronic adverse environmental conditions[J].Journal of Experimental Botany,2013,64(1):229-240.
[7] 万东璞,于卓,吴燕民,丁梦琦,李金博,周美亮.花青素代谢调控植物彩叶研究进展[J]. 中国农业科技导报,2020,22(2):30-38.WAN Dongpu,YU Zhuo,WU Yanmin,DING Mengqi,LI Jinbo,ZHOU Meiliang. Regulation of anthocyanin metabolism on colored leaves of plants[J]. Journal of Agricultural Science and Technology,2020,22(2):30-38.
[8] WANG C H,ZHI S,LIU C Y,XU F X,ZHAO A C,WANG X L,TANG X,LI Z G,HUANG P,YU M D.Isolation and characterization of a novel chalcone synthase gene family from mulberry[J].Plant Physiology and Biochemistry,2017,115:107-118.
[9] 徐靖,朱家红,王效宁,韩义胜,唐力琼,朱红林.甘薯查尔酮合成酶基因IbCHS1 的克隆和表达分析[J]. 分子植物育种,2018,16(6):1752-1757.XU Jing,ZHU Jiahong,WANG Xiaoning,HAN Yisheng,TANG Liqiong,ZHU Honglin. Cloning and expression analysis of chalcone synthase gene IbCHS1 in Ipomoea batatas[J]. Molecular Plant Breeding,2018,16(6):1752-1757.
[10] KONDO S,HIRAOKA K,KOBAYASHI S,HONDA C,TERAHA-RA N.Changes in the expression of anthocyanin biosynthetic genes during apple development[J]. Journal of the American Society for Horticultural Science,2002,127(6): 971-976.
[11] TSUDA T,YAMAGUCHI M,HONDA C. Expression of anthocyanin biosynthesis genes in the skin of peach and nectarine fruit[J]. Journal of the American Society for Horticultural Science,2004,129(6):857-862.
[12] YU B,ZHANG D,HUANG C H,QIAN M J,ZHENG X Y,TENGY W,SU J,SHU Q.Isolation of anthocyanin biosynthetic genes in red Chinese sand pear (Pyrus pyrifolia Nakai.) and their expression as affected by organ/ tissue,cultivar,bagging and fruit side[J].Scientia Horticulturae,2012,136:29-37.
[13] ZHANG J P,ZHANG W T,JI F Y,QIU J,SONG X B,BU D C,PAN G,MA Q G,CHEN J X,HUANG R M,CHANG Y Y,PEI D. A high-quality walnut genome assembly reveals extensive gene expression divergences after whole-genome duplication[J].Plant Biotechnology Journal,2020,18(9):1-3.
[14] GUO W L,CHEN J H,LI J,HUANG J Q,WANG Z J,LIM K J. Portal of Juglandaceae:A comprehensive platform for Juglandaceae study[J].Horticulture Research,2020,7:35.
[15] 鱼尚奇,贾昌路,宋岩,刘春花,郭永翠,张文涛,陈立平,张锐. 纸皮核桃内果皮硬化期差异表达基因筛选及功能预测[J].果树学报,2019,36(4):410-420.YU Shangqi,JIA Changlu,SONG Yan,LIU Chunhua,GUO Yongcui,ZHANG Wentao,CHEN Liping,ZHANG Rui.Screening and functional prediction of differential expression genes at lignification stage of endocarp in Zhipi walnut[J]. Journal of Fruit Science,2019,36(4):410-420.
[16] LI Y Z,LUO X,WU C Y,CAO S Y,ZHOU Y F,JIE B,CAO Y L,MENG H J,WU G L. Comparative transcriptome analysis of genes involved in anthocyanin biosynthesis in red and green walnut(Juglans regia L.)[J].Molecules,2018,23(1):25-42.
[17] CHEN W,GONG L,GUO Z L,WANG W S,ZHANG H Y,LIU X Q,YU S B,XIONG L Z,LUO J. A novel integrated method for large-scale detection,identification,and quantification of widely targeted metabolites: application in the study of rice metabolomics[J].Molecular Plant,2013,6(6):1769-1780.
[18] 位路路,林杨,王月华,孟宪军.黑果腺肋花楸花色苷提取工艺优化及其抗氧化活性和组成鉴定[J]. 食品科学,2018,39(12):239-246.WEI Lulu,LIN Yang,WANG Yuehua,MENG Xianjun. Optimization of extraction of anthocyanins from berries of Aronia melanocarpa and their antioxidant activity and composition[J]. Food Science,2018,39(12):239-246.
[19] CHEN C J,CHEN H,ZHANG Y,THOMAS H R,FRANK M H,HE Y H,XIA R. TBtools- an integrative toolkit developed for interactive analyses of big biological data[J]. Molecular Plant,2020,13(8):1-26.
[20] LIVAK K J,SCHMITTGEN T D.Analysis of relative gene expression data using real-time quantitative PCR and the 2- Δ Δ CT method[J].Methods,2001,25:402-408.
[21] 张蕾,朱立新,徐川,崔春梅,盛宏亚,李瑞,王红清.查尔酮合酶基因对桃果实花色苷代谢的影响[J]. 园艺学报,2015,42(1):31-37.ZHANG Lei,ZHU Lixin,XU Chuan,CUI Chunmei,SHENG Hongya,LI Rui,WANG Hongqing.The effect of silencing chalcone synthase on anthocyanin metabolism in peach[J].Acta Horticulturae Sinica,2015,42(1):31-37.
[22] 袁华招,赵密珍,吴伟民,于红梅,王庆莲.葡萄CHS 和STS 基因家族生物信息学鉴定和表达分析[J].植物遗传资源学报,2016,17(4):756-765.YUAN Huazhao,ZHAO Mizhen,WU Weimin,YU Hongmei,WANG Qinglian. Genome-wide identification and expression analysis of CHS and STS gene families in grape (Vitis vinifera L.)[J].Journal of Plant Genetic Resources,2016,17(4):756-765.
[23] CHEN L,GUO Y R,ZHANG X R,MI R F. Effects of 5-aminolevulinic acid on the content of total flavonoids and expression of CHS and CHI genes in young apples[J].Agricultural Biotechnology,2015,4(3):39-42.
[24] SASLOWSKY D,WINKEL-SHIRLEY B.Localization of flavonoid enzymes in Arabidopsis roots[J]. Plant Journal for Cell &Molecular Biology,2010,27(1):37-48.
[25] 周军,陶建敏,彭日荷,熊爱生,蔡斌,徐锦涛,金晓芬,张斌,高峰,高建杰,章镇,姚泉洪.巨峰葡萄查尔酮合成酶基因4 的克隆及表达特性的RT-PCR 分析[J]. 南京农业大学学报,2010,33(2):39-44.ZHOU Jun,TAO Jianmin,PENG Rihe,XIONG Aisheng,CAI Bin,XU Jintao,JIN Xiaofen,ZHANG Bin,GAO Feng,GAO Jianjie,ZHANG Zhen,YAO Quanhong.Cloning and expression analysis of CHS4 of Kyoho grape by RT-PCR[J].Journal of Nan jing Agricultural University,2010,33(2):39-44.
[26] 阮美颖,万红建,叶青静,王荣青,姚祝平,周国治,俞锞,袁伟,刘云飞,杨悦俭.番茄查尔酮合成酶基因的鉴定及生物信息学分析[J].分子植物育种,2013,11(3):379-384.RUAN Meiying,WAN Hongjian,YE Qingjing,WANG Rongqing,YAO Zhuping,ZHOU Guozhi,YU Ke,YUAN Wei,LIU Yunfei,YANG Yuejian. Identification and bioinformatics analysis of chalcone synthase genes in tomato[J]. Molecular Plant Breeding,2013,11(3):379-384.
[27] 牛天敏,马会勤,陈尚武.大豆查尔酮合成酶(CHS)基因的克隆、表达及其在雪莲提取液中的代谢产物分析[J].中国生物工程杂志,2007,27(2):58-63.NIU Tianmin,MA Huiqin,CHEN Shangwu. Cloning and expression of chalcone synthase(CHS)of Glycine max L.and analysis of it metabolize produce in the extracts from Saussurea spp.[J].China Biotechnology,2007,27(2):58-63.
[28] 杨丽,刘雅莉,王跃进,徐伟荣.百合查尔酮合成酶(CHS)基因的克隆与分析[J].西北植物学报,2006,26(5):933-936.YANG Li,LIU Yali,WANG Yuejin,XU Weirong. Cloning and analysis of chalcone synthase genes in Lilium[J].Acta Botanica Boreali-Occidentalia Sinica,2006,26(5):933-936.
[29] 林玲,汤浩茹,陈清,鲁敏,贾慧峰,李英改,张晓楠.观赏桃查尔酮合成酶基因的克隆及序列分析[J]. 园艺学报,2012,39(3):581-587.LIN Ling,TANG Haoru,CHEN Qing,LU Min,JIA Huifeng,LI Yinggai,ZHANG Xiaonan. Cloning and sequence analysis of chalcone synthase gene in ornamental peach[J]. Acta Horticulturae Sinica,2012,39(3):581-587.
[30] 周兴文,李纪元,朱宇林. 金花茶查尔酮合成酶基因CnCHS的克隆及遗传转化研究[J].植物研究,2015,35(3):327-332.ZHOU Xingwen,LI Jiyuan,ZHU Yulin. Cloning and genetic transformation of CnCHS gene from Camellia nitidissima[J].Bulletin of Botanical Research,2015,35(3):327-332.
[31] SUN Y T,LI M,MITRA S,MUHAMMAD R H,DEBNATH B,LU X C,JIAN H X,QIU D L.Comparative phytochemical profiles and antioxidant enzyme activity analyses of the southern highbush blueberry (Vaccinium corymbosum) at different developmental stages[J].Molecules,2018,23(9):2209.
[32] LAI B,HU B,QIN Y H,ZHAO J T,WANG H C,HU G B.Transcriptomic analysis of Litchi chinensis pericarp during maturation with a focus on chlorophyll degradation and flavonoid biosynthesis[J].BMC Genomics,2015,16:225-243.
[33] 王锦玲.龙眼叶片黄酮类化合物合成关键酶基因和DHAR 基因cDNA 全长的克隆[D].福州:福建农林大学,2011.WANG Jinling. Cloning of the full length cDNA of flavonoids synthesis key genes and DHAR gene from Longan leaves [D].Fuzhou:Fujian Agriculture and Forestry University,2011.
[34] 赵景梅,黄东益,张青,许云.紫参薯查尔酮合成酶及异构酶基因的克隆与表达分析[J]. 热带作物学报,2018,39(5): 920-925.ZHAO Jingmei,HUANG Dongyi,ZHANG Qing,XU Yun.Clonging and expression analysis of DaCHS and DaCHI gene in Dioscorea alata[J]. Chinese Journal of Tropical Crops,2018,39(5):920-925.
[35] 冯立娟,尹燕雷,杨雪梅,唐海霞,李英朋,鹿英.石榴查尔酮合成酶蛋白的生物信息学分析[J]. 山东农业科学,2019,51(2):7-12.FENG Lijuan,YIN Yanlei,YANG Xuemei,TANG Haixia,LI Yingpeng,LU Ying. Bioinformatics analysis of CHS protein in pomegranate[J].Shandong Agricultural Sciences,2019,51(2):7-12.
[36] 程水源,王燕,李俊凯,费永俊,朱桂才.内源激素含量与银杏叶中类黄酮含量的关系[J].林业科学,2004,40(6):45-49.CHENG Shuiyuan,WANG Yan,LI Junkai,FEI Yongjun,ZHU Guicai. Study on the relationship between the endogenous hormones and flavonoids in Ginkgo biloba leaf[J]. Scientia Silvae Sinicae,2004,40(6):45-49.
[37] 刘航程,孙爽,徐秀琴,邱黎斌,苏青玲,张晓玉,仇悦璇,赖艳,张蔚,胡永峰.‘月月粉’和野蔷薇花青素合成酶基因的鉴定与表达分析[J]. 分子植物育种,2020,http://kns.cnki.net/kcms/detail/46.1068.S.20200226.1303.002.html.LIU Hangcheng,SUN Shuang,XU Xiuqin,QIU Libin,SU Qingling,ZHANG Xiaoyu,QIU Yuexuan,LAI Yan,ZHANG Wei,HU Yongfeng.Identification and expression analysis of anthocyanin biosynthesis genes in Rosa chinensis and Rosa multiflora[J].Molecular Plant Breeding,2020,http://kns.cnki.net/kcms/detail/46.1068.S.20200226.1303.002.html.
[38] YUAN Y,MA X H,SHI Y M,TANG D Q.Isolation and expression analysis of six putative structural genes involved in anthocyanin biosynthesis in Tulipa fosteriana[J]. Scientia Horticulturea,2013,153:93-102.
[39] SABLOWSKI R W,MOYANO E,CULIANFZ-MACIA F A,SCHUCH W,MARRTIN C,BEVAN M. A flower- specific MYB protein activates transcription of phenylpropanoid biosynthetic genes[J].The EMBO Journal,1994,13(1):128-137.
[40] 李文静,孙艳香,付亚娟,苏彦苹,王聪艳,侯晓强,张新业.菊芋查尔酮合成酶基因的克隆与表达分析[J].西北农业学报,2020,29(4):1-10.LI Wenjing,SUN Yanxiang,FU Yajuan,SU Yanping,WANG Congyan,HOU Xiaoqiang,ZHANG Xinye.Cloning and expression analysis of chalcone synthase gene from Helianthus tuberosus[J/L]. Acta Agriculturae Boreali- Occidentalis Sinica,2020,29(4):1-10.
[41] MA D W,REICHELT M,YOSHIDA K,GERSHENZON J,CONSTABEL C P. Two R2R3-MYB proteins are broad repressors of flavonoid and phenylpropanoid metabolism in poplar[J].The Plant Journal,2018,96(5):949-965.